
中国海洋湖沼学会主办。
文章信息
- 王玲, 李琪, 孔令锋, 于红. 2018.
- WANG Ling, LI Qi, KONG Ling-Feng, YU Hong. 2018.
- 基于COI基因的中国沿海青蚶野生群体遗传结构及种群动态研究
- POPULATION GENETIC STRUCTURE AND DEMOGRAPHIC HISTORY OF BARBATIA VIRESCENS ALONG CHINESE COAST BASED ON MITOCHONDRIAL COI SEQUENCES
- 海洋与湖沼, 49(1): 87-95
- Oceanologia et Limnologia Sinica, 49(1): 87-95.
- http://dx.doi.org/10.11693/hyhz20170300058
-
文章历史
- 收稿日期:2017-03-17
- 收修改稿日期:2017-05-31
青蚶(Barbatia virescens)隶属于软体动物门(Mollusca)、双壳纲(Bivalvia)、列齿目(Taxodonta)、蚶科(Arcidae)、须蚶属(Barbatia), 主要分布于我国浙江嵊山以南至海南沿海, 日本、越南、菲律宾等国也有分布(徐凤山等, 2008)。青蚶主要栖息于潮间带到数十米深的浅水区, 以足丝附着于岩礁缝隙中(蔡如星等, 1991)。青蚶作为蚶科贝类中的新兴品种, 肉味鲜美、营养丰富, 具有较大的养殖开发潜力。目前对于青蚶的研究相对较少, 仅在青蚶的人工育苗、精子超微结构、血红蛋白亚基结构等方面有报道(Suzuki et al, 1989; 竺俊全等, 2004; 张鹏等, 2011), 其种群遗传结构及其遗传多样性现状还不明确。物种种群的遗传结构是物种种质资源保护的重要科学依据, 因此, 开展青蚶不同地理种群的分子遗传学研究, 了解其遗传结构, 对我国青蚶种质资源的保护及可持续开发利用具有重要意义。
线粒体DNA (Mitochondria DNA, mtDNA)因其具有单亲遗传、进化速度快、核苷酸替代率高、分子量小(Brownw et al, 1979; Whitmoer et al, 1994)的特点, 已作为一种重要的分子标记, 广泛应用于群体遗传学和系统发生的研究中(Hallerman, 2003)。其中, 线粒体COI基因作为最常用的分子标记, 在青蛤(Cyclina sinensis)、长牡蛎(Crassostrea gigas)、菲律宾蛤仔(Ruditapes philippinarum)等海洋双壳贝类的遗传多样性及种群结构研究中发挥了重要作用(Ni et al, 2012; Li et al, 2015; Cordero et al, 2017)。
本研究对中国沿海青蚶6个地理群体进行了形态变异分析, 利用线粒体COI基因探讨了其遗传结构与种群历史动态, 以期阐明青蚶的遗传多样性现状, 为合理有效利用和保护青蚶种质资源提供参考资料。
1 材料与方法 1.1 样品的采集6个青蚶群体的样品分别采集自浙江嵊泗(SS)、浙江南麂岛(NJD)、福建霞浦(XP)、福建平潭(PT)、广东阳江(YJ)和广西北海(BH)(图 1)。每个群体数量为9-28个, 共计112个。样品采集后用95%酒精固定保存。
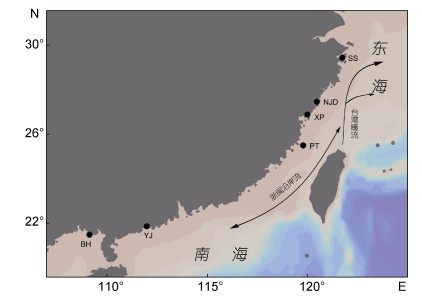 |
| 图 1 青蚶群体采样分布点 Fig. 1 Map of sampling sites for B.virescens |
根据青蚶的外部形态, 测量各个体的形态指标。采用游标卡尺作为测量工具(精度0.01mm), 测量壳长(L)、壳宽(W)、壳高(H)、壳顶宽(w)、外韧带长(l)、背缘长(x)6个指标, 每个指标测3次后取平均值。
1.3 DNA的提取、扩增及测序分别取青蚶闭壳肌肌肉约100mg, 采用苯酚氯仿法提取基因组DNA(Li et al, 2002), 置于-20℃冰箱保存。用1%的琼脂糖凝胶电泳检测DNA的完整性, 紫外分光光度计测定DNA的浓度和纯度。
本研究设计了1对通用性较高的青蚶COI引物: COIQ-F:TCATATTCGGCTAAATCTT, COIQ-R:CATC TTTCCACACCTCAA对青蚶群体进行扩增。PCR反应总体系为30μL, 包括:模板DNA 100ng, 3μL 10× PCR buffer, 2.0mmol/L MgCl2, 25mmol/L dNTPs, 正反向引物各10μmol/L, 1.5U Taq DNA聚合酶(TaKaRa)。反应条件为: 94℃预变性5min, 94℃变性30s, 48℃退火30s, 72℃延伸1min, 35个循环, 最终72℃延伸5min。扩增产物送深圳华大基因科技有限公司进行纯化测序。
1.4 数据统计与分析 1.4.1 形态学分析在SPSS22.0中采用单因素方差分析(ANOVA)对6个青蚶群体的形态差异进行比较和分析。将每个个体所测参数的平均数除以自身的壳长, 得到校正值后再进行数据处理以消除个体大小对形态特征的影响(张尧庭等, 1982; 余建英等, 2003)。
1.4.2 序列分析测序获得的双向序列利用SeqMan (Swindell et al, 1997)根据峰值图进行比对拼接。将拼接矫正后的DNA序列导入MEGA6.0 (Tamura et al, 2013)进行ClusterW序列比对, 辅助人工检查结果的可靠性, 确定序列的长度、遗传距离及碱基组成等。在DnaSP v5 (Librado et al, 2009)中统计群体的总单倍型数及总多态位点数。青蚶群体的遗传多样性参数[每个群体的单倍型数(H)、单倍型多样度(h)、核苷酸多样度(π)及两两序列比对平均核苷酸差异数(k)]的计算在Arlequin3.5 (Excoffier et al, 2010)中完成。
1.4.3 群体遗传结构分析利用Arlequin3.5进行分子变异方差分析(AMOVA), 组间、组内群体间、群体内个体间的协方差的显著性通过1000次重抽样来检验。基于Tumura-Nei模型下的群体分化指数(Fst)分析两两群体间的遗传差异, Fst值的显著性通过1000次重抽样检验, 并对多重检验进行班费罗尼校正(Rice, 1989)。利用Popart1.7 (http://popart.otago.ac.nz)基于TCS法建立单倍型网络关系图。在MEGA6.0中利用邻接法(neighbor-joining method)以Kimura 2 parameter模型构建单倍型系统发育进化树, 各支点的支持率采用bootstrap法, 重复1000次。
1.4.4 历史动态分析种群历史动态分析在Arlequin3.5中进行。利用Tajima’s D检验(Tajima, 1989)和Fu’s Fs检验(Fu, 1997)的统计值及其显著性检测中性假说与否成立, 利用核苷酸不配对分布(Mismatch Distribution)分析检测群体是否经历扩张事件。根据公式t=τ/(2μk)估算种群扩张时间, 其中τ是突然扩张模型下的参数, k为该研究中的序列长度, μ为整个序列长度的突变速率, 计算所得到的t值即为群体扩张到现在经历的时间。演化速率采用0.7%每百万年(Marko, 2002)。
2 结果 2.1 形态差异比较6个地理群体青蚶形态特征的描述性统计值和Tukey多重比较分析结果见表 1和2。单因子方差分析结果表明, 对于壳高(H), 北海群体与阳江、平潭群体间有显著差异(P < 0.05);对于壳宽(W), 阳江群体与南麂岛、霞浦、平潭群体有显著差异(P < 0.05);对于壳顶宽(w), 嵊泗和平潭群体与其他四个群体有显著差异(P < 0.05);对于外韧带长(l), 南麂岛和阳江群体分别与其他五个群体有显著差异(P < 0.05)。
| 性状 | 群体 | |||||
| 嵊泗(n=9) | 南麂岛(n=9) | 霞浦(n=28) | 平潭(n=23) | 阳江(n=22) | 北海(n=21) | |
| L (cm) | 2.392±0.039 (1.622—2.923) | 2.563±0.049 (1.732—3.360) | 2.952±0.025 (2.553—3.562) | 3.289±0.026 (2.774—3.969) | 1.792±0.037 (1.252—2.846) | 2.756±0.074 (1.849—3.897) |
| H (cm) | 1.408±0.031 (0.839—1.886) | 1.407±0.028 (0.879—1.896) | 1.659±0.016 (1.343—1.940) | 1.776±0.016 (1.422—2.145) | 1.144±0.034 (0.662—1.836) | 1.554±0.039 (1.090—2.135) |
| W (cm) | 9.113±0.019 (0.581—1.217) | 1.041±0.022 (0.682—1.491) | 1.147±0.010 (0.984—1.386) | 1.216±0.016 (0.963—1.466) | 6.632±0.020 (0.385—1.142) | 1.140±0.035 (0.610—1.553) |
| w (cm) | 0.388±0.007 (0.293—0.528) | 0.544±0.013 (0.314—0.746) | 0.614±0.011 (0.452—0.941) | 0.495±0.011 (0.206—0.830) | 0.336±0.011 (0.162—0.597) | 0.599±0.021 (0.235—0.910) |
| l (cm) | 0.849±0.015 (0.589—1.070) | 0.968±0.022 (0.626—1.467) | 0.995±0.013 (0.738—1.245) | 1.110±0.016 (0.828—1.414) | 0.501±0.021 (0.311—0.940) | 0.969±0.031 (0.528—1.671) |
| x (cm) | 1.669±0.029 (1.280—2.173) | 1.702±0.039 (1.224—2.499) | 1.929±0.019 (1.525—2.307) | 2.188±0.020 (1.827—2.701) | 1.296±0.032 (0.906—1.749) | 1.777±0.047 (1.175—2.489) |
| 注: L:壳长; W:壳宽; H:壳高; w:壳顶宽; l:外韧带长; x:背缘长 | ||||||
| 变量 | 显著性水平 | 平均值及其多重比较 | |||||
| 嵊泗 | 南麂岛 | 霞浦 | 平潭 | 阳江 | 北海 | ||
| W/L | α=0.05 | 0.5847±0.05904ab | 0.5500±0.04917ab | 0.5641±0.05686ab | 0.5414±0.04494bc | 0.6347±0.11872bc | 0.5677±0.04021a |
| H/L | α=0.05 | 0.3790±0.02810ac | 0.4056±0.03338ab | 0.3897±0.03484ab | 0.3706±0.04651ab | 0.3652±0.04577c | 0.4118±0.05102a |
| w/L | α=0.05 | 0.1622±0.04901b | 0.2118±0.03066a | 0.2088±0.03839a | 0.1505±0.03003b | 0.1865±0.04412a | 0.2166±0.05318a |
| l/L | α=0.05 | 0.3645±0.10372a | 0.3776±0.04100b | 0.3384±0.04485a | 0.3380±0.04649a | 0.5010±0.06990c | 0.3530±0.07092a |
| x/L | α=0.05 | 0.6993±0.05025ab | 0.6637±0.06556ab | 0.6552±0.05989b | 0.6663±0.04808ab | 0.7232±0.10373a | 0.6494±0.07005ab |
| 注:平均值为X ±SD, 字母完全不同表示差异显著 | |||||||
将测序所得序列经SeqMan矫正去除引物序列及部分端序列后得到587bp的等长同源序列。利用Mega6.0对青蚶112条COI序列进行分析, 其中A、T、C、G的平均含量分别为16.0%、41.4%、15.3%、27.3%, AT含量(57.4%)高于GC含量(42.6%), 各群体碱基组成相近。
112条同源序列中共检测出18个多态性位点, 其中单突变位点14个, 简约信息位点4个。共定义了17个单倍型, 包括3个共享单倍型, 占总数的17.6%。其中Hap_1拷贝数最多, 为84次, 占序列总数的75.0%, 同时也是所有群体共享的单倍型; Hap_2是第二多的单倍型, 共有9个拷贝, 占序列总数的8.0%, 为三个群体共享(平潭、阳江、北海); Hap_12为三个群体共享的单倍型(嵊泗、霞浦、平潭), 但每个群体仅有一个个体。其余均为每个群体的特有单倍型。总群体的平均单倍型多样性指数为0.5472, 平均核苷酸多样性为0.000974, 平均核苷酸差异数为0.571469。青蚶6个群体的遗传学参数如表 3。单倍型多样度最高的为嵊泗群体(h=0.5833), 最低的为霞浦群体(h=0.2063);核苷酸多样度最高的为嵊泗群体(π=0.001514), 最低的为霞浦群体(π=0.00036)。总体来看, 青蚶群体有中等水平的单倍型多样性和较低水平的核苷酸多样性, 遗传多样性最高的群体为嵊泗群体, 最低的为霞浦群体。
| 群体 | 样本数目 | 单倍型数H | 多态位点数s | 单倍型多样度h | 平均核苷酸差异数k | 核苷酸多样性指数π |
| 嵊泗 | 9 | 4 | 4 | 0.5833±0.1833 | 0.888889±0.492170 | 0.001514±0.001316 |
| 南麂岛 | 9 | 3 | 2 | 0.4167±0.1907 | 0.444444±0.433519 | 0.000757±0.000838 |
| 霞浦 | 28 | 4 | 3 | 0.2063±0.1005 | 0.214286±0.266020 | 0.000365±0.000505 |
| 平潭 | 23 | 6 | 5 | 0.5178±0.1218 | 0.592885±0.492170 | 0.001010±0.000935 |
| 阳江 | 22 | 3 | 2 | 0.4805±0.0935 | 0.545455±0.467423 | 0.000929±0.000888 |
| 北海 | 21 | 6 | 6 | 0.4952±0.1298 | 0.742857±0.572329 | 0.001266±0.001088 |
青蚶群体间遗传距离及群体间遗传分化系数见表 4。群体间遗传距离分布在0.0006-0.0014之间, 遗传距离最高的为嵊泗与阳江群体及嵊泗与北海群体间, 均为0.0014, 北海与其他群体之间的遗传距离相对较高; 而霞浦与其他群体之间的遗传距离相对较低, 最低的为霞浦与南麂岛群体之间, 遗传距离为0.0006。
| 嵊泗 | 南麂岛 | 霞浦 | 平潭 | 阳江 | 北海 | |
| 嵊泗 | 0.0011 | 0.0009 | 0.0013 | 0.0014 | 0.0014 | |
| 南麂岛 | 0.00027 | 0.0006 | 0.0009 | 0.0011 | 0.0010 | |
| 霞浦 | 0.04615 | 0.03240 | 0.0007 | 0.0008 | 0.0008 | |
| 平潭 | 0.01226 | 0.00489 | 0.01609 | 0.0010 | 0.0012 | |
| 阳江 | 0.13740* | 0.14453* | 0.20522** | 0.07121* | 0.0012 | |
| 北海 | 0.01709 | –0.00084 | 0.02792* | 0.01412 | 0.09934** | |
| 注: *表示差异显著0.01 < P≤0.05, **表示差异极显著P≤0.01 | ||||||
基于COI基因的两两群体之间的Fst值分布在-0.00084-0.20522, 其中阳江与霞浦间的遗传分化系数最大, 南麂岛与北海间的遗传分化系数最小。阳江与其他5个群体之间存在显著或极显著的低程度的遗传分化(Fst=0.07121-0.20522), 除此之外, 霞浦与北海群体之间也存在显著差异(P < 0.05)。其他群体相互间的遗传分化表现为差异不显著(P > 0.05)。
AMOVA分析结果显示(表 5), 青蚶群体间遗传变异大多数来自于群体内个体间, 占87.40%, 且具有显著的统计值(P=0.0004), 较少部分的遗传差异来自于组间, 占11.15%(P=0.1655), 而来自于组内群体间的遗传差异只占1.45% (P=0.0701)。由此表明所研究的青蚶群体的遗传分化主要表现为群体内分化。
| 分组 | 变异来源 | 自由度df | 方差总和 | 方差组分 | 变异百分比(%) | Φ | P |
| (阳江) & (嵊泗, 南麂岛, 霞浦, 平潭, 北海) | 组间 | 1 | 1.556 | 0.03381 Va | 11.15 | ΦCT= 0.1115 | 0.1655 |
| 组内群体间 | 4 | 1.362 | 0.00439 Vb | 1.45 | ΦSC=0.0163 | 0.0701 | |
| 群体内个体间 | 106 | 28.091 | 0.26501 Vc | 87.40 | ΦST=0.1260 | 0.0004 | |
| 总计 | 111 | 31.009 | 0.30321 | ||||
| 注: Va、Vb、Vc分别表示组间、组内群体间、群体内个体间的方差变异组分; ΦCT、ΦSC、ΦST分别表示组间、组内群体间、群体内个体间的遗传分化指数 | |||||||
图 2为青蚶群体的单倍型网络关系图, 由图可知, Hap_1位于单倍型中央, 为所有群体共享, 且拷贝数占序列总数的四分之三, 呈现明显的“星状”拓扑结构。各单倍型间碱基差异小, 不同群体的单倍型未呈现地理分支, 没有表现出显著的地理系谱结构。17个单倍型的NJ系统树(图 3)也没有表现出清晰的网状结构, 支持单倍型网络图的结论。
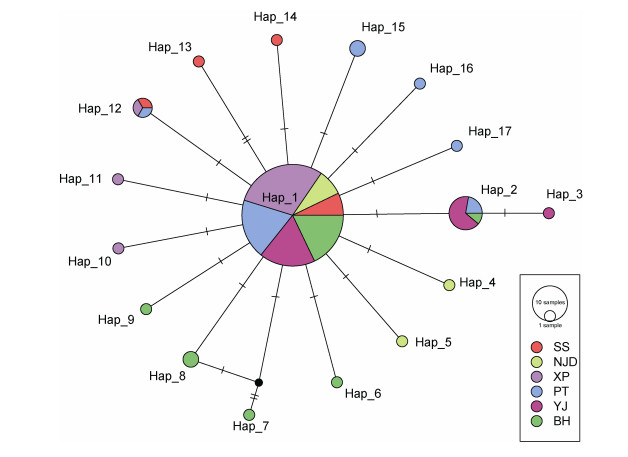 |
| 图 2 青蚶17个单倍型的网络关系图 Fig. 2 Haplotype network for the17 haplotypes of B.virescens 注: SS:嵊泗; NJD:南麂岛; XP:霞浦; PT:平潭; YJ:阳江; BH:北海 |
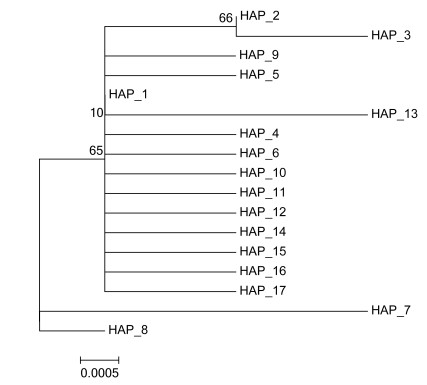 |
| 图 3 青蚶线粒体COI基因不同单倍型邻接树 Fig. 3 Haplotype neighbor-joining tree based on mtDNA COI gene of B.virescens |
检验群体在中性条件下是否经历了扩张事件方法为:如果Tajima’s D检验和Fu’s Fs检验的检验值至少有一个为负值且显著偏离中性, 则表明该群体经历了扩张事件。两种中性检验结果显示(表 6): Tajima’s D检验中, 所有群体的检验值均为负值, 除南麂岛和阳江群体外均显著偏离中性, Fu’s Fs检验中, 除阳江群体外其余群体检验值均为负值且显著偏离中性, 表明青蚶群体在中性模式下经历了群体扩张事件。核苷酸不配对分布分析表明, 观测值呈现近似的单峰分布(图 4), 平方离差之和(Sum of squared deviations, SSD)也不显著(P > 0.05), 符合群体扩张模型下的预期分布。采用蚶科线粒体进化速率(0.7%每百万年)估算可知, 青蚶群体的扩张时间约为26万年前。
| 群体 | Fu’s Fs检测(P) | Tajima’s D检测(P) | 平方离差之和SSD (P) |
| 嵊泗 | -1.2827(0.0440*) | -1.6097(0.0350*) | 0.0007(0.9230) |
| 南麂岛 | -1.0811(0.0390*) | -1.3624(0.0960) | 0.0087(0.6960) |
| 霞浦 | -3.2664(0.0000*) | -1.7332(0.0170*) | 0.0018(0.4360) |
| 平潭 | -3.7468(0.0020*) | -1.6497(0.0330*) | 0.0267(0.1423) |
| 阳江 | 0.0213(0.4270) | -0.0132(0.4710) | 0.0078(0.3470) |
| 北海 | -3.1288(0.0040*) | -1.7419(0.0240*) | 0.0087(0.3910) |
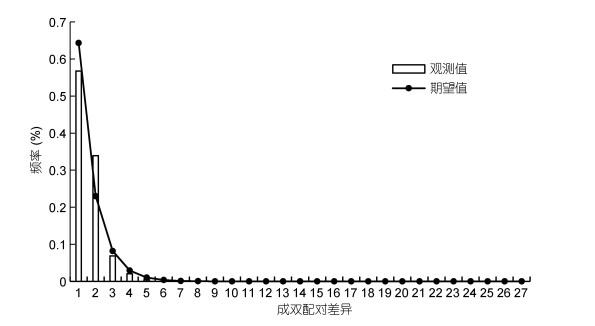 |
| 图 4 青蚶群体的COI基因核苷酸错配分布 Fig. 4 Differences in the observed pairwise sequence and the expected mismatch distributions in population expansion model of the COI gene haplotypes for the B. virescens populations |
遗传多样性是生物多样性的重要组分之一, 遗传多样性的高低决定了物种生存适应和发展进化的潜力。一个物种的遗传多样性的高低与其对环境的适应能力成正比, 即遗传多样性越高的物种对环境的适应能力越强, 反之亦然。正确认识物种的遗传多样性水平, 对物种遗传资源的保护、资源的可持续发展、遗传育种等方面都是至关重要的。
形态学性状可以在一定程度上对物种进行鉴定, 初步描述不同地理群体间的差异。但仅通过形态学分析得到的结论是不全面的, 因此在进行物种分类和遗传变异检测中, 应将形态学方法与遗传学方法结合起来。已有研究利用多变量形态度量学方法对魁蚶(Scapharca broughtonii)、毛蚶(Scapharca subcrenata)、泥蚶(Tegillarca granosa)等蚶科贝类进行不同地理群体形态差异分析, 表明不同群体间的不同性状具有显著差异(张永普等, 2004; 梁超等, 2011; 宋菲菲等, 2012)。本研究中青蚶6个地理种群的单因素方差分析表明, 对于进行比较的性状, 不同种群之间存在显著差异。而分子数据结果证实, 虽然不同地理群体间形态差异显著, 但不同地理群体间COI基因序列差异小, 不具有明显的地理谱系分化, 表明青蚶不同形态个体间遗传差异小, 环境影响是导致形态差异的主要因素。Laudien(2003)的研究也表示, 形态特征虽然由遗传因子决定, 但受物种的栖息环境影响, 包括温度、盐度、饵料等, 如果环境因子差别较大并长期作用于种群, 就会导致不同地理种群在形态上产生显著差异, 而群体间的基因交流则导致遗传均质性的产生。
单倍型多样性和核苷酸多样性是评价遗传多样性的重要指标。本研究中我们对中国南方沿海6个青蚶群体的COI基因进行扩增, 获得587bp的序列, 检测到18个多态位点和17个单倍型。结果表明, 青蚶具有中等水平的单倍型多样性(0.5472)和较低水平的核苷酸多样性(0.000974), 符合Grant等(1998)提出的四种单倍型多样性和核苷酸多样性组合中的“高h, 低π”的遗传多样性类型。这种情况在许多海洋无脊椎动物中都曾出现(Ni et al, 2012; Zhou et al, 2016)。而相比于蚶科中其他物种, 如泥蚶(Ni et al, 2012)、毛蚶(陈辰, 2015)等, 青蚶的遗传多样性参数较低, 112个个体中仅检测到17个单倍型, 暗示了青蚶种群的遗传多样性处于相对较低的水平。具有低水平遗传多样性的物种易受到环境及人为因素影响, 在不良环境下的生存也会受到威胁。因此, 对于青蚶的种质资源的保护应该得到相应的重视。
3.2 种群遗传结构AMOVO分析发现两两群体间的Fst值较低且多数为不显著, 表明不同群体间没有形成显著的遗传分化, 只有阳江群体与其他群体间存在低程度的遗传分化。总体来看, 青蚶群体没有形成显著的遗传结构, 遗传分化程度低, 群体间存在基因交流。
一些海洋生物通常表现出在其较大空间范围具有较低水平的遗传分化, 不具有显著的遗传结构的特点。大多数海洋无脊椎动物具有浮游期, 浮游期幼虫在海流推动下扩散, 使不同地理群体间保持基因交流, 呈现出低水平的遗传分化, 表现出遗传均质性(Palumbi et al, 1994; Lee et al, 2009)。
海流对生物的遗传分化的影响是十分重要的。存在于东海和南海的海洋环流对物种的遗传分布会造成一定程度的影响。我国浙江以南沿海的海洋环流系统主要包括两大部分, 一是黑潮的支流, 沿中国台湾北上入东海, 即台湾暖流; 二是季节性的浙闽沿岸流, 在冬季和夏季分别自北向南和自南向北流动, 促使不同海区间的联系加强(苏纪兰等, 2005)。青蚶生活史中具有10d左右的浮游期(张鹏等, 2011), 在此期间, 在海流的推动下, 各群体间基因交流频繁, 可能是导致其没有形成显著遗传格局的原因。
此外, 已有研究报道港湾岛屿也会对种群的遗传分化造成一定的影响, 由于其会对幼虫的扩散产生一定的阻碍作用, 因此会导致物种产生不同程度的遗传分化(Zhan et al, 2009; Ni et al, 2011; 李双等, 2015)。本研究中阳江群体与其他群体间存在显著的低程度的分化, 可能与阳江所处的地理位置有一定的关系。广东省位于我国大陆南端沿海, 港湾岛屿数仅次于浙江福建二省, 阳江地处广东西南沿海, 周围多港湾及岛屿, 这些港湾和岛屿可能对该海域的青蚶幼虫的扩散形成天然的屏障, 一定程度上阻碍了其与其他群体间的基因交流。
3.3 种群动态本研究采用中性检验和核苷酸不配对分析来研究青蚶群体的历史动态。实验结果揭示青蚶群体经历了群体扩张事件, 扩张时间约为26万年前。该时间对应于第四纪晚冰期的第3次间冰期。第四纪晚期冰期-间冰期对于东海的生物种群的分布格局具有重大影响。该时期, 由于冰川作用, 海平面下降, 东海面积缩小, 导致大量生物栖息地丧失, 许多海洋生物被迫向深海避难所迁移, 形成了均质性的祖先群体。冰期后, 海平面上升, 避难所的种群重新扩张, 占据新的栖息地从而导致新的遗传格局的产生(Wang et al, 1994; Shen et al, 2011; Ni et al, 2012)。本研究中青蚶群体具有典型的“星状”单倍型网络关系图, 序列间差异小, 单倍型的NJ系统树也不具有明显分支, 因此我们推测所研究的青蚶具有同一祖先群体, 来自于冰期时期东海避难所, 在更新世末期发生了种群的扩张。
4 结论本研究利用线粒体COI基因对中国南方沿海6个青蚶群体进行遗传结构及种群动态分析。结果显示:不同群体间在某些形态特征上存在显著差异, 这些差异主要是由地理环境特性决定的; 所研究的青蚶群体遗传多样性水平较低, 应采取相应措施对其遗传多样性进行保护; 整体来看青蚶没有形成显著的遗传结构, 仅阳江群体表现出显著的低程度的遗传分化; 种群历史动态分析表示, 青蚶在约26万年前经历种群扩张, 推测其具有同一祖先群体。
| 余建英, 何旭宏, 2003. 数据统计分析与SPSS应用. 北京: 人民邮电出版社, |
| 宋菲菲, 王其翔, 刘名, 等, 2012. 不同地区毛蚶群体的形态差异的比较研究. 中国海洋大学学报(自然科学版), 42(11): 40–45 |
| 张鹏, 黄贤克, 王铁杆, 等, 2011. 青蚶人工育苗的初步研究. 上海海洋大学学报, 20(6): 838–844 |
| 张永普, 林志华, 应雪萍, 2004. 不同地理种群泥蚶的形态差异与判别分析. 水产学报, 28(3): 339–342 |
| 张尧庭, 方开泰, 1982. 多元统计分析引论. 北京: 科学出版社, |
| 李双, 李琪, 于红, 等, 2015. 浙闽沿海葡萄牙牡蛎群体遗传结构及种群历史分析. 中国水产科学, 22(6): 1260–1269 |
| 苏纪兰, 袁业立, 2005. 中国近海水文. 北京: 海洋出版社, 367 |
| 陈辰, 2015. 毛蚶群体遗传学研究. 青岛: 中国海洋大学博士学位论文, 62-68 http://cdmd.cnki.com.cn/Article/CDMD-10423-1015716141.htm |
| 竺俊全, 杨万喜, 2004. 毛蚶与青蚶精子超微结构及其所反映的蚶科进化关系. 动物学研究, 25(1): 57–62 |
| 徐凤山, 张素萍, 2008. 中国海产双壳类图志. 北京: 科学出版社, 52 |
| 梁超, 杨爱国, 刘志鸿, 等, 2011. 4个地理群体魁蚶(Scapharca broughtonii)的形态差异与判别分析. 海洋科学, 35(11): 108–113 |
| 蔡如星, 黄惟灏, 1991. 浙江动物志-软体动物. 杭州: 浙江科学技术出版社, 157 |
| Brown W M, George M, Wilson A C, 1979. Rapid evolution of animal mitochondrial DNA. Proc Natl Acad Sci USA, 76(4): 1967–1971 DOI:10.1073/pnas.76.4.1967 |
| Cordero D, Delgado M, Liu B Z, et al, 2017. Population genetics of the Manila clam (Ruditapes philippinarum) introduced in North America and Europe. Sci Rep, 7: 39745 DOI:10.1038/srep39745 |
| Excoffier L, Lischer H E L, 2010. Arlequin suite Ver 3. 5:a new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Res, 10(3): 564–567 DOI:10.1111/men.2010.10.issue-3 |
| Fu Y X, 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics, 147(2): 915–925 |
| Grant W A S, Bowen B W, 1998. Shallow population histories in deep evolutionary lineages of marine fishes:Insight from sardines and anchovies and lessons for conservation. J Heredity, 89(5): 415–426 DOI:10.1093/jhered/89.5.415 |
| Hallerman E, 2003. Population Genetics:Principles and Applications for Fisheries Scientist. Bethesda, MD: American Fisheries Society, 59-100 |
| Laudien J, Flint N S, Van Der Bank F H, et al, 2003. Genetic and morphological variation in four populations of the surf clam Donax serra (R ding) from southern African sandy beaches. Biochem Systemat Ecol, 31(7): 751–772 DOI:10.1016/S0305-1978(02)00252-1 |
| Lee H J E, Boulding E G, 2009. Spatial and temporal population genetic structure of four northeastern Pacific littorinid gastropods:the effect of mode of larval development on variationat one mitochondrial and two nuclear DNA markers. Mol Ecol, 18(10): 2165–2184 DOI:10.1111/mec.2009.18.issue-10 |
| Li Q, Park C, Kijima A, 2002. Isolation and characterization of microsatellite loci in the Pacific abalone, Haliotis discus hannai. J Shellf Res, 21(2): 811–816 |
| Li S, Li Q, Yu H, et al, 2015. Genetic variation and population structure of the Pacific oyster Crassostrea gigas in the northwestern Pacific inferred from mitochondrial COI sequences. Fisher Sci, 81(6): 1071–1082 DOI:10.1007/s12562-015-0928-x |
| Librado P, Rozas J, 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451–1452 DOI:10.1093/bioinformatics/btp187 |
| Marko P B, 2002. Fossil calibration of molecular clocks and the divergence times of geminate species pairs separated by the Isthmus of Panama. Mol Biol Evolut, 19(11): 2005–2021 DOI:10.1093/oxfordjournals.molbev.a004024 |
| Ni G, Li Q, Kong L F, et al, 2012. Phylogeography of bivalve Cyclina sinensis:Testing the historical glaciations and Changjiang river outflow hypotheses in Northwestern Pacific. PLoS One, 7(11): 1–11 |
| Ni L H, Li Q, Kong L F, 2011. Microsatellites reveal fine-scale genetic structure of the Chinese surf clam Mactra chinensis (Mollusca, Bivalvia, Mactridae) in Northern China. Mar Ecol, 32(4): 488–497 DOI:10.1111/mae.2011.32.issue-4 |
| Palumbi S R, 1994. Genetic divergence, reproductive isolation, andmarine speciation. Annual Rev Ecol Systemat, 25(1): 547–572 DOI:10.1146/annurev.es.25.110194.002555 |
| Rice W R, 1989. Analyzing tables of statistical tests. Evolution, 43(1): 223–225 DOI:10.1111/evo.1989.43.issue-1 |
| Shen K N, Jamandre B W, Hsu C C, et al, 2011. Plio-Pleistocene sea level and temperature fluctuations in the northwestern Pacific promoted speciation in the globally-distributed flathead mullet Mugil cephalus. BMC Evolut Biol, 11(1): 83 DOI:10.1186/1471-2148-11-83 |
| Suzuki T, Shiba M, Furukohri T, et al, 1989. Hemoglobins from the two closely related clams Barbatia lima and Barbatia virescenes. Comparison of their subunit structures and N-terminal Sequence of the unusual two-domain chain:Biochemistry. Zool Sci, 6(2): 269–281 |
| Swindell S R, Plasterer T N, 1997. SEQMAN. In: Swindell S R, ed. Sequence data analysis guidebook. Methods In Molecular MedicineTM. Totowa, NJ: Springer, 70: 75-89 |
| Tajima F, 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics, 123(3): 585–595 |
| Tamura K, Stecher G, Peterson D, et al, 2013. MEGA6:molecular evolutionary genetics analysis version 6.0. Mol Biol Evolut, 30(12): 2725–2729 DOI:10.1093/molbev/mst197 |
| Wang P X, Sun X J, 1994. Last glacial maximum in China:comparison between land and sea. Catena, 23(3-4): 341–353 DOI:10.1016/0341-8162(94)90077-9 |
| Whitmoer D H, Thai T H, Craft C M, 1994. The largemouth bass ctyochrome b gene. J Fish Biol, 44(4): 637–645 DOI:10.1111/jfb.1994.44.issue-4 |
| Zhan A B, Hu J J, Hu X L, et al, 2009. Fine-scale population genetic structure of Zhikong scallop (Chlamys farreri):do local marine currents drive geographical differentiation. Mar Biotechnol, 11(2): 223–235 DOI:10.1007/s10126-008-9138-1 |
| Zhou N, Shen H D, Chen C, et al, 2016. Genetic structure of Onchidium"struma"(Mollusca:Gastropoda:Eupulmonata) from the coastal area of China based on mtCO Ⅰ. Mitochondrial DNA Part A:DNA Mapp Seq Anal, 27(2): 1319–1323 |
 2018, Vol. 49
2018, Vol. 49


