中国海洋湖沼学会主办。
文章信息
- 郭强强, 蔡伟聪, 李桂玲, 曾珺, 李健, 刘静雯. 2018.
- GUO Qiang-Qiang, CAI Wei-Cong, LI Gui-Ling, ZENG Jun, LI Jian, LIU Jing-Wen. 2018.
- 海洋球石藻(Emiliania huxleyi)磷脂酶C的结构特点及系统进化分析
- STRUCTURAL CHARACTERIZATION AND EVOLUTION OF PHOSPHOLIPASE C IN MARINE MICROALGA EMILIANIA HUXLEYI
- 海洋与湖沼, 49(1): 106-116
- Oceanologia et Limnologia Sinica, 49(1): 106-116.
- http://dx.doi.org/10.11693/hyhz20170200030
-
文章历史
- 收稿日期:2017-02-16
- 收修改稿日期:2017-03-21
2. 福建省高校食品微生物与酶工程重点实验室 厦门 361021
2. Fujian Provincial Key Laboratory of Food Microbiology and Enzyme Engineering, Xiamen 361021, China
磷脂酶C (PLC: EC3.1.4.3)广泛存在于原核生物和真核生物中, 可以水解磷脂生成二酰甘油(diacyl glycerol, DAG)和肌醇三磷酸(inositol trisphosphates, IP3), 这两个小分子作为第二信使在动、植物信号转导中具有重要作用(Zheng et al, 2012; Singh et al, 2013; 王法微等, 2014)。PLC根据反应底物的不同可以分为磷脂酰肌醇特异性PLC (phosphoinositide-specific PLC, PI-PLC)和非特异性PLC (Non-specific PLC, NPC)。有关PLC研究较多的集中于动物中, 可分为6大类, 分别为PLCβ、PLCγ、PLCδ、PLε、PLCζ和PLCη (Suh et al, 2008)。在动物细胞中, PLC水解磷脂形成DAG和IP3, 其中DAG能够激活蛋白激酶C (protein kinase C, PKC), 进一步传递给有丝分裂原活化蛋白激酶, 从而在细胞增殖、分化和凋亡信号转导中起重要作用; IP3则结合在内质网膜的受体上, 激活Ca2+通道, 将内质网中储存的Ca2+释放到细胞质中, 而释放到胞浆中的大部分Ca2+由钙泵重新摄取回内质网, 以此维持内质网Ca2+浓度的平衡, 少量钙离子则被线粒体摄入, 这些过量的Ca2+会导致线粒体损伤并释放细胞色素C, 活化凋亡特征蛋白酶caspases, 从而诱导细胞凋亡(Kadenbach et al, 2004; Bardo et al, 2006)。植物中的PLC分类没有动物中那么复杂, 只存在一种亚型结构, 缺少PH结构域, 与动物中PLCζ结构类似。目前已发现拟南芥基因组中存在6种NPC、9种PI-PLC和4种普通的PLC (Nakamura et al, 2005; Tasma et al, 2008)。水稻基因组中含有9个PLC, 其中4个属于PI-PLC, 另外5个均为NPC (Singh et al, 2013)。植物中尚未发现PKC的同源蛋白, 也没有鉴定出明确的IP3受体。植物中的PLC水解产生的DAG在激酶的作用下转化成磷脂酸(phos-phatatic acid, PA), IP3则会被磷酸化成IP4、IP5和IP6, 这些磷酸化产物在植物激素信号转导中可能起主要作用(黄冬等, 2017)。浮游植物中的PLC则报道较少, 在莱茵衣藻、红藻、绿藻中只发现单拷贝的PLC基因, 这些单拷贝基因在进化演变中促成了PLC功能的多样性(Mikami, 2014)。
海洋球石藻(Emiliania huxleyi)是一种全球广泛分布且具有重要生态功能的真核浮游植物。其具有独特的生物矿化作用和高产二甲基硫化物(DMSP)能力, 这一特点使其成为影响全球碳、硫生物地化循环及气候变化的一个关键物种(Joassin et al, 2011; Martínez et al, 2012; Blanco-Ameijeiras et al, 2016)。自然海域中球石藻经常会形成大面积赤潮, 研究证实特异性病毒(E. huxleyi virus)感染和裂解是终止该藻赤潮的一个重要因素。有趣的是, 球石藻被病毒感染后能够大量合成鞘脂类代谢产物—神经酰胺类物质并导致宿主细胞凋亡, 从而调控宿主种群密度(Bidle et al, 2007; Evans et al, 2009; Michaelson et al, 2010; Rosenwasser et al, 2014)。可见, 球石藻细胞中鞘脂类物质的代谢在调节病毒与宿主间的相互作用中起着非常重要的作用。神经酰胺是细胞信号转导中的一个重要的信号分子, 可作为传递细胞外应激信号的第二信使将凋亡信号传至核内, 激活凋亡特征蛋白酶caspases, 从而诱发细胞凋亡; 也可以直接作用于线粒体, 改变其正常结构和功能从而导致细胞凋亡(罗磊, 2014)。在动物细胞中, 神经酰胺主要以结合态的鞘脂类形式存在于细胞膜上, 通过PLC的水解作用转化为游离的神经酰胺, 从而作为第二信使参与细胞信号转导(金道忠等, 2007)。细胞凋亡这一古老而又十分保守的死亡过程存在于所有的多细胞生物中。近年来对于单细胞生物, 特别是具有光合能力的浮游植物细胞凋亡的研究越来越受到关注。过去, 动物摄食和沉降被认为是浮游植物死亡的原因, 现在人们发现病毒感染以及逆境条件下的主动死亡(细胞凋亡或程序化死亡)也是引起浮游植物死亡的重要原因, 但对其程序性死亡的分子机制及其生态学意义仍不十分清楚(Gordeeva et al, 2004; Franklin et al, 2006; Schatz et al, 2014)。
本文采用生物信息学方法, 对海洋球石藻磷脂酶C进行理化性质和结构分析, 预测其可能的代谢调节功能, 探索其在浮游植物环境胁迫应答及微生物侵染过程中发挥的重要作用, 并为进一步开展浮游植物PLC信号通路特别是病毒介导的海洋球石藻细胞凋亡信号通路的研究奠定基础。
1 材料与方法 1.1 研究材料利用NCBI在线蛋白数据库搜索、过滤, 获得30条海洋球石藻(Emiliania huxleyi CCMP 1516) PLC (EhPLC)氨基酸序列信息, 对这些序列进行系统的生物信息分析。
1.2 方法 1.2.1 EhPLC一级结构及理化性质的分析蛋白质一级结构是指蛋白质多肽链氨基酸的组成及排列顺序, 利用不同的在线软件对EhPLC理化性质、疏水性、信号肽及磷酸化位点等进行预测分析。利用ExPaSy中的ProtParam (http://web.expasy.org/protparam/)对EhPLC氨基酸序列进行理化性质分析; 利用SignaIP4.1 (http://www.cbs.dtu.dk/services/SignalP/)对EhPLC氨基酸序列中的信号肽进行预测; 利用TMHMM程序(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白跨膜区; 利用ProtScale (http:// www.expasy.org/cgi-bin/protscale.pl)对EhPLC进行疏水性分析, 窗口尺寸调整为19;利用NetPhos3.0 Server程序(http://www.cbs.dtu.dk/services/NetPhos/)对 EhPLC氨基酸序列中的磷酸化位点进行分析, 预测其氨基酸序列中的苏氨酸(Thr)、酪氨酸(Tyr)和丝氨酸(Ser)等三种氨基酸被磷酸化修饰的可能性。
1.2.2 EhPLC二级结构及亚细胞定位利用SMART网站(http://smart.embl-heidelberg.de/)在线分析30种EhPLC的保守结构域的特征; 筛选其中具有代表性的8种EhPLC序列, 利用Prabi程序(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_hnn.pl)对其蛋白质二级结构进行预测; 利用Target P1.1数据库对EhPLC进行蛋白质转运肽预测; 利用ProtComp v9.0 (http://linux1.softberry.com/berry.phtml?group=programs&subgroup=proloc&topic=protcompan)预测EhPLC的亚细胞定位。
1.2.3 EhPLC三级结构预测利用在线工具ExPaSy中的Swiss-model (http://swissmodel.expasy.org)程序进行同源性比较, 选取同源性最高的模板在线生成模型, 然后通过PyMOL Viwer软件进行编辑, 最终绘制成蛋白质的三级结构模型图。
1.2.4 EhPLC序列比对及系统发育树的构建利用NCBI将EhPLC19的氨基酸序列进行Blast同源性分析, 选取同源性较高的不同物种, 再利用MEGA5.0对这些氨基酸序列进行序列比对, 用相邻链接法(NJ法)构建系统发育树。
2 结果与分析 2.1 搜索结果根据NCBI的基因数据库, 输入关键词Emilianai hyxleyi phospholipase C, 获得33个EhPLC的登录信息, 将搜索到的33个EhPLC氨基酸序列比对后发现其中有三对的氨基酸序列完全一致, 因此确定有30种EhPLC。将30种氨基酸序列由小到大排序, 并依次将其命名为EhPLC1、EhPLC2、EhPLC3、EhPLC4……等等, 序列信息如表 1。根据氨基酸个数相近和所含结构域的不同从中选取8种EhPLC作进一步分析。
| 名称 | Gene ID | Protein ID | 功能 | 基因序列长度(nt) | 氨基酸长度(aa) |
| EhPLC1 | XM_005782057.1 | XP_005782114.1 | phospholipase c | 501 | 166 |
| EhPLC2 | XM_005780273.1 | XP_005780330.1 | phospholipase c | 501 | 166 |
| EhPLC3 | XM_005759050.1 | XP_005759107.1 | phospholipase c | 527 | 176 |
| EhPLC4 | XM_005782058.1 | XP_005782115.1 | phospholipase c | 711 | 236 |
| EhPLC5 | XM_005757135.1 | XP_005757192.1 | phospholipase c | 747 | 248 |
| EhPLC6 | XM_005790884.1 | XP_005790941.1 | phospholipase c | 816 | 271 |
| EhPLC7 | XM_005776531.1 | XP_005776588.1 | phospholipase c | 822 | 273 |
| EhPLC8 | XM_005774657.1 | XP_005774714.1 | phospholipase c | 933 | 310 |
| EhPLC9 | XM_005779192.1 | XP_005779249.1 | phospholipase c | 1267 | 380 |
| EhPLC10 | XM_005770382.1 | XP_005770439.1 | phospholipase c | 1164 | 387 |
| EhPLC11 | XM_005772628.1 | XP_005772685.1 | phospholipase c | 1197 | 398 |
| EhPLC12 | XM_005758266.1 | XP_005758323.1 | phospholipase c | 1197 | 398 |
| EhPLC13 | XM_005774657.1 | XP_005774714.1 | phospholipase c | 1311 | 436 |
| EhPLC14 | XM_005787721.1 | XP_005787778.1 | phospholipase c | 1359 | 452 |
| EhPLC15 | XM_005776126.1 | XP_005776183.1 | phospholipase c | 2049 | 682 |
| EhPLC16 | XM_005763258.1 | XP_005763315.1 | phospholipase c | 2619 | 872 |
| EhPLC17 | XM_005771178.1 | XP_005771235.1 | phospholipase c | 2679 | 892 |
| EhPLC18 | XM_005792977.1 | XP_005793034.1 | phospholipase c | 2886 | 961 |
| EhPLC19 | XM_005782439.1 | XP_005782496.1 | phospholipase c | 3075 | 1024 |
| EhPLC20 | XM_005770577.1 | XP_005770634.1 | phospholipase c | 4914 | 1147 |
| EhPLC21 | XM_005768682.1 | XP_005768739.1 | phospholipase c | 3795 | 1264 |
| EhPLC22 | XM_005761145.1 | XP_005761202.1 | phospholipase c | 4662 | 1500 |
| EhPLC23 | XM_005781409.1 | XP_005781466.1 | phospholipase c | 4587 | 1528 |
| EhPLC24 | XM_005774402.1 | XP_005774459.1 | phospholipase c | 4641 | 1546 |
| EhPLC25 | XM_005777446.1 | XP_005777503.1 | phospholipase c | 4950 | 1649 |
| EhPLC26 | XM_005793571.1 | XP_005793628.1 | phospholipase c | 5943 | 1692 |
| EhPLC27 | XM_005756497.1 | XP_005756554.1 | phospholipase c | 5127 | 1708 |
| EhPLC28 | XM_005785467.1 | XP_005785524.1 | phospholipase c | 8102 | 1931 |
| EhPLC29 | XM_005785678.1 | XP_005785735.1 | phospholipase c | 6093 | 2030 |
| EhPLC30 | XM_005762079.1 | XP_005762136.1 | phospholipase c | 7341 | 2446 |
8种EhPLC均含碱性氨基酸和酸性氨基酸(表 2)。当蛋白质的不稳定系数大于40即为不稳定蛋白。表 2结果显示只有EhPLC20的不稳定系数小于40, 因此, 除EhPLC20外, 其余7种EhPLC均为不稳定蛋白。
| 蛋白名称 | 主要氨基酸(%) | 碱性/酸性氨基酸 | 理论等电点pI | 不稳定指数 |
| EhPLC14 | Ala 11.5 Thr 9.5 Leu 9.1 Gly 8.4 | 35/48 | 5.30 | 41.26 |
| EhPLC15 | Leu 10.9 Ala 10.4 Arg 9.4 Gly 8.4 | 80/83 | 6.77 | 51.56 |
| EhPLC16 | Leu 12.0 Ala 11.5 Ser 9.6 Gly 7.2 | 85/94 | 5, 92 | 46.52 |
| EhPLC17 | Leu 12.0 Ala 11.0 Arg 9.3 Ser 9.0 | 99/1115 | 5.93 | 51.46 |
| EhPLC18 | Leu 11.7 Ala 10.6 Ser 10.0 Arg 8.5 | 98/100 | 6.99 | 60.99 |
| EhPLC19 | Ala 11.8 leu 10.8 Pro 10.2 Arg 9.1 | 110/108 | 7.75 | 62.00 |
| EhPLC20 | Ser 16.2 Ala 15.9 Arg 11.5 Gly 9.8 | 167/72 | 11.08 | 38.45 |
| EhPLC21 | Ala 11.2 Leu 10.6 Ser 9.8 Arg 7.5 | 145/136 | 8.62 | 46.27 |
通过SignalP 4.1 Sever在线分析上述8种EhPLC氨基酸序列中的信号肽, 结果显示, EhPLC16和EhPLC21在N端都存在一个典型的信号肽, 其他6种不存在信号肽(以EhPLC16为例, 如图 1所示)。通过TMHMM Server分析, EhPLC18在N端存在一个跨膜区(如图 2), EhPLC19在第74—97氨基酸位置上存在一个跨膜结构域, 其他蛋白序列不存在跨膜区。
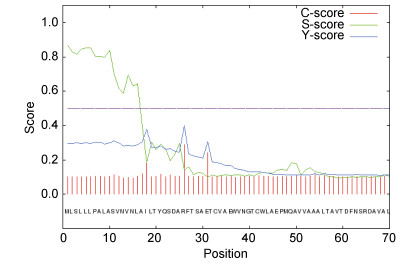 |
| 图 1 EhPLC16信号肽预测图 Fig. 1 The prediction of signal peptide of EhPLC16 注: C-score (Cleavage site score):剪切位置分值; S-score (Signal peptide score):信号肽分值; Y-score (combined cleavage site score):综合考虑C-score和S-score。在信号肽的位置, Y-score有高值, C-score有个高峰值 |
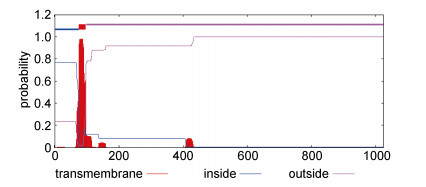 |
| 图 2 EhPLC19跨膜区预测图 Fig. 2 The prediction of transmembrane structure domain of EhPLC19 注:蓝线代表膜内区, 红线代表膜外区; 位置发生交换, 说明此处具有跨膜结构, 且预测概率接近1, 说明此处很可能存在一个跨膜区 |
通过ProtScale程序中的得分值法进行疏水性分析。结果显示, 该8种EhPLC的氨基酸疏水性平均值均为负值, 表明整体上都属于亲水性蛋白, 但在亲水程度上存在一定差异, 其中亲水性最强的是EhPLC20 (图 3)。
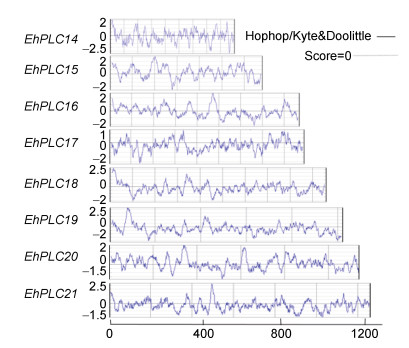 |
| 图 3 海洋球石藻磷脂酶C疏水性分析 Fig. 3 The hydrophobicity of EhPLC |
利用NetPhos对8种EhPLC酸化位点进行预测, 预测其磷酸化位点的可能性均大于0.5。8种EhPLC中丝氨酸磷酸化位点最多, 苏氨酸磷酸化位点次之, 酪氨酸磷酸化位点较少(表 3)。PLC在信号分子刺激下可结合GTP, 通过获得若干个磷酸基团而被激活, 在信号刺激减弱时这些磷酸基团将被去除。EhPLC如此多样性的磷酸化修饰表明在海洋球石藻中, 磷酸化与去磷酸化分子开关在调控EhPLC参与的信号传导中起着关键作用。
| 蛋白名称 | Thr | Ser | Tyr |
| EhPLC14 | 9 | 14 | 8 |
| EhPLC15 | 9 | 24 | 4 |
| EhPLC16 | 12 | 37 | 2 |
| EhPLC17 | 21 | 37 | 3 |
| EhPLC18 | 19 | 49 | 1 |
| EhPLC19 | 13 | 51 | 2 |
| EhPLC20 | 24 | 87 | 3 |
| EhPLC21 | 13 | 56 | 4 |
根据植物PI-PLC的基本结构: EF指型结构、X结构域、Y结构域及C2结构域, 利用SMART在线分析预测30条氨基酸序列, 结果如图 4所示, 其中10条序列不含PI-PLC的典型结构域; 有11条序列只有一个PI-PLC典型结构域, 其中EhPLC4、EhPLC9、EhPLC10只含有C2结构域, EhPLC11只含Y区, EhPLC15、EhPLC16、EhPLC28只含X区, EhPLC24、EhPLC 26含有X区和跨膜结构域, EhPLC29含有X区、ww结构域、跨膜区, EhPLC30含有X区、跨膜区、PbH1结构域; 有6个序列含有两个PLC典型结构域, EhPLC12含有Y区、C2区; EhPLC17、EhPLC22含有X区、Y区; EhPLC18含有X区、C2区、跨膜区, EhPLC23含有X区、C2区、FHA结构域; 有3个序列含有3个PLC典型结构域, 其中, EhPLC21含有X区、Y区、C2区, EhPLC19含有X区、Y区、C2区、跨膜区, EhPLC27含有X区、Y区、C2区、FHA结构域。
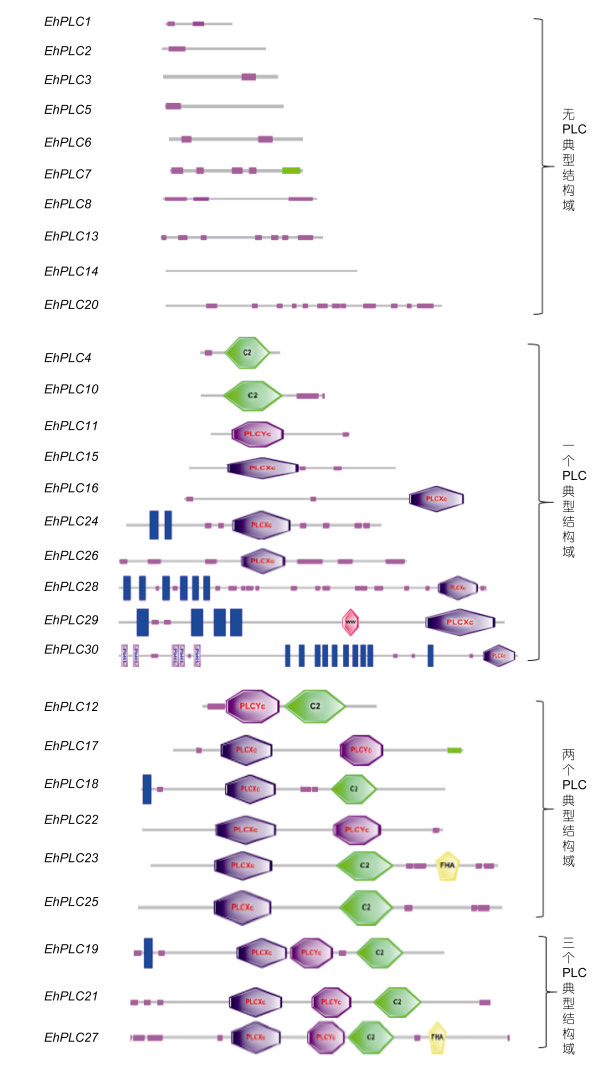 |
| 图 4 海洋球石藻磷脂酶C结构分析 Fig. 4 The conserved domain of EhPLC |
X-Y催化区中间的连接序列XY-linker与IP3结合; EF手型结构用于结合Ca2+; C2结构域的功能是结合磷脂, 其过程依赖Ca2+, 对维持催化区活性有辅助作用(Nomikos et al, 2007; Suh et al, 2008)。PLC结构的多样性使得不同的磷脂酶C所依赖的金属离子不同, 如大多微生物来源的PLC酶活需要Zn2+的参与(Hough et al, 1989)。EhPLC9特有的ww结构域存在于第1405—438氨基酸位置处。ww结构域是由38—40个氨基酸残基严密组织形成一个连贯、紧凑的结构域, 其中两个色氨酸残基为其结构的主要特征, 能特异性识别并结合富含脯氨酸的蛋白质基序和磷酸化的丝氨酸/色氨酸-脯氨酸肽段, 是介导蛋白质相互作用的一类小的蛋白质模块(Halder et al, 2011; Hergovich, 2012)。EhPLC26在氨基酸231—583位置处含有5个PbH1结构域, 可以结合多糖。FHA为叉头相关结构域(forkhead-associated domain), 是许多调控蛋白的磷酸肽识别结构域, 具有磷酸苏氨酸和磷酸酪氨酸专一性识别位点, 且对前者的亲和力会相对高一些(Hofmann et al, 1995)。
2.3.2 二级结构特征对8种EhPLC二级结构预测结果显示, 它们均含有α螺旋、β折叠及无规则卷曲, 无规则卷曲所含比例最大, β折叠所含比例最小(图 5)。具体而言, 含α螺旋比例最高是EhPLC16, 为43.46%, 最低的是EhPLC20, 为27.9%;含β折叠比例最高是EhPLC14, 为14.82%, 最低的是EhPLC18, 为9.78%;含无规则卷曲比例最高的是EhPLC14, 为55.88%, 最低的是EhPLC16, 为44.84%。
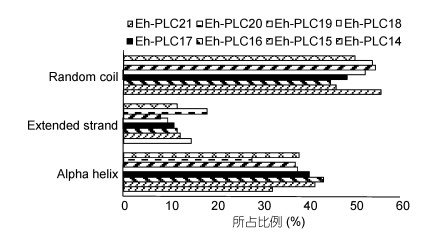 |
| 图 5 海洋球石藻磷脂酶C二级结构域预测 Fig. 5 Prediction of the secondary structure domain of EhPLC 注: Random coil:无规则卷曲; Extended strand: β折叠; Alpha helix: α螺旋 |
利用ProtComp v9.0程序进行分析, 结果显示, 所有EhPLC都主要定位于线粒体, 另外也有个别分布于过氧化物酶体和高尔基体中(表 4)。推测EhPLC主要在线粒体中行使其功能。EhPLC的细胞定位对确定其功能具有重要意义。禾谷炭疽菌中不同的PLC定位在线粒体、高尔基体、和细胞核等不同位置(韩长志, 2016), 莱茵衣藻PLC则定位于质膜(Awasthi et al, 2012)。
| 名称 | 核 | 线粒体 | 过氧化物酶体 | 高尔基体 | 内质网 | 溶酶体 | 液泡 | 细胞质 | 质膜 | 胞外 |
| EhPLC14 | 0 | 2.85 | 2.15 | 0.73 | 0.67 | 1.35 | 0 | 1.21 | 0.92 | 2.38 |
| EhPLC15 | 0 | 3.17 | 2.03 | 0 | 0.80 | 1.60 | 0 | 1.36 | 0.91 | 2.33 |
| EhPLC16 | 0 | 2.93 | 1.93 | 0.11 | 0.93 | 1.66 | 0 | 1.48 | 0.87 | 2.21 |
| EhPLC17 | 0 | 3.13 | 2.50 | 2.74 | 1.03 | 0.11 | 0 | 0.99 | 1.00 | 2.74 |
| EhPLC18 | 0 | 2.96 | 2.23 | 0.45 | 0.50 | 1.30 | 0 | 1.48 | 0.94 | 2.49 |
| EhPLC19 | 0 | 2.78 | 2.20 | 0.39 | 0.43 | 1.45 | 0 | 1.45 | 0.94 | 2.50 |
| EhPLC20 | 0 | 2.62 | 1.83 | 0.29 | 0.49 | 1.86 | 0 | 1.61 | 0.86 | 2.21 |
| EhPLC21 | 0 | 2.34 | 2.02 | 0.15 | 0.58 | 1.88 | 0 | 1.61 | 0.88 | 2.30 |
同源建模结果显示, 8种EhPLC主要有6种模板: 2pz0.1.A, 3qr0.1.A, 5fbh.1.A, 2fju.1.B, 3qr1.1.A及4qj3.1.B, 可预测的蛋白区域主要集中在多肽中间, 这与PLC保守结构域的位置相吻合。与模板相似度最高的为EhPLC19, 达54.05%。而其他与模板同源性均较低, 分析结果并不可靠, 所以选取EhPLC19进行同源建模, 结果如图 6所示, 图中红色а-螺旋结构是EhPLC19的C2结构域, 即与底物及Ca2+结合的部位。
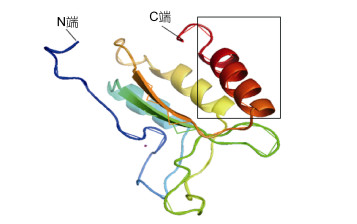 |
| 图 6 海洋球石藻EhPLC19三级结构预测图 Fig. 6 Fig.6 Prediction of the tertiary structure domain of EhPLC19 注:黑框中红色a-螺旋结构是EhPLC19的C2结构域, 即与底物及Ca2+结合的部位 |
以EhPLC19作为目标序列, 在GenBank中进行blast搜索氨基酸序列进行同源性比较分析。结果显示, 该序列与其他物种的PLC同源性较低, 与白斑狗鱼Esox lucius的同源性最高, 为39%, 与气单胞菌属Angomonas deanei的同源性为38%, 与利什曼虫Leishmania infantum JPCM5的同源性为37%, 与袋獾Sarcophilus harrisii的相似度为35%, 与旋毛虫Trichinella的同源性为31%, 以上结果表明不同物种在进化过程中PLC基因出现了较大的差异。同源比对结果中未出现植物物种的PLC, 说明海洋球石藻EhPLC的同源性与植物的相似度很低, 而与低等动物的相似度相对较高, 推测在进化过程中EhPLC的结构和功能也更接近动物。8种EhPLC则大致聚为三类, EhPLC16、EhPLC20为一类, EhPLC17、EhPLC15、EhPLC18、EhPLC19为一类, EhPLC4、EhPLC21为一类(图 7)。
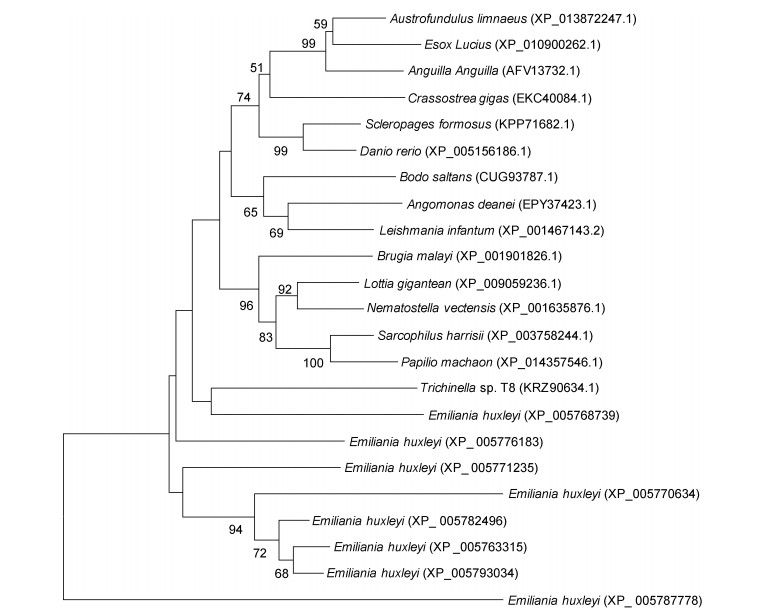 |
| 图 7 根据不同物种的PLC氨基酸序列绘制的系统发育树 Fig. 7 The distance tree based on the amino acid sequences of the PLCs from different organisms 注: Emiliania huxleyi (XP_005768739): EhPLC21; Emiliania huxleyi (XP_005776183): EhPLC15; Emiliania huxleyi (XP_005771235): EhPLC17; Emiliania huxleyi (XP_005770634): EhPLC20; Emiliania huxleyi (XP_005782496): EhPLC19; Emiliania huxleyi (XP_005763315): EhPLC16; Emiliania huxleyi (XP_005793034): EhPLC18; Emiliania huxleyi (XP_005787778): EhPLC14 |
PLC的功能在动物中已经研究得十分清楚, 动物细胞膜上的G蛋白偶联受体(R)接受配体信号, 通过G蛋白激活PI-PLC, 水解PIP2, 生成DAG和IP3 (李莉等, 2015)。它们分别结合并激活PKC和IP3的受体, 使内质网钙离子通道打开, 后者释放Ca2+到胞质。线粒体摄取Ca2+, 导致细胞色素C的释放, 最终使细胞凋亡。然而在植物中尚未鉴定出激活PLC的G蛋白, 也没有发现PLC下游信号中的两个重要调节因子, 即IP3的受体和PKC (Munnik et al, 2009)。植物体内生成的IP3会通过磷酸化形成IP4、IP5及IP6参与细胞信号转导, 植物中IP6是促进钙离子释放的主要因素(Lemtiri-Chlieh et al, 2003; Michell, 2008)。现在众多植物包括番茄、杨树、大豆、拟南芥和水稻等的基因组已经完全测序, 还有大量EST文库, 在这些数据库中并未发现IP3的受体基因。但是在衣藻和草履虫基因组中发现了IP3受体基因(Wheeler et al, 2008)。在硅藻(角毛藻, 三角褐指藻, 圆柱拟脆杆藻及多列拟菱形藻)基因组中搜索到G蛋白偶联受体的基因, 与人体蛋白序列比对含G蛋白偶联受体核心组件, 硅藻G蛋白α-和β-亚基与人同源物的序列相似性水平比已知的植物界G蛋白亚基和人G蛋白略高(Port et al, 2013)。海洋球石藻E. huxleyi CCMP1516全基因组注释显示, 基因组中含有蛋白激酶基因、IP3基因、G蛋白及G蛋白偶联受体基因。海洋球石藻中存在丰富多样的磷脂C种类及完善的G蛋白偶联受体介导的信号通路中的主要成员, 因此, 我们可以提出这样一个有趣的假设, 即病毒感染海洋球石藻后或许也可以同时启动神经酰胺以外的另一信号途径, 即G蛋白偶联的DAG/IP3双信号系统诱导宿主细胞凋亡。这也为我们进一步深入研究球石藻病毒诱导宿主细胞死亡的分子机制提供了一个新的思路。
神经酰胺可作为第二信使传递细胞凋亡和分化增殖的信号, 通过调控细胞周期的运行过程从而控制细胞的分裂和死亡。病毒感染海洋球石藻后通过掌控宿主鞘脂类合成途径大量合成病毒特有的新型鞘糖脂神经酰胺, 这些神经酰胺在细胞膜表面形成神经酰胺富集的膜结构, 最终形成膜筏(Michaelson et al, 2010; Rose et al, 2014)。病毒利用这种新颖的脂筏通过与宿主细胞质膜融合或内吞作用进入宿主细胞, 导致宿主细胞膜结构和功能的紊乱, 促进子代病毒的增殖和释放, 而这一过程可能涉及到宿主细胞周期调控的异常, 并最终诱导宿主细胞凋亡(Liu et al, 2011, 2015; Vermont et al, 2016)。最新研究发现, 病毒感染球石藻可以诱导宿主细胞另一种主动死亡程序即——细胞自噬过程(Schatz et al, 2014), 可见细胞凋亡和细胞自噬之间可能存在交叉作用。本实验室初步的研究结果显示, 病毒感染能够促使海洋球石藻宿主细胞中PLC在转录和蛋白水平表达量的增加并与细胞中神经酰胺的积累存在一定的相关性(数据未发表)。在哺乳动物钙离子与凋亡的研究中发现, PLC含量的增加, 使细胞内的IP3含量也增加, 通过内质网表面的受体, 打开Ca2+通道, 使Ca2+进入细胞质的同时, 线粒体内的Ca2+出现了升高, 线粒体内的Ca2+超载, 而后线粒体结构出现肿胀和裂解, 进而引起细胞的凋亡(赵凤命等, 2012)。因此我们可以提出一个假设:病毒感染藻细胞后, 主导宿主鞘脂类代谢促进磷脂酶C的积累并合成病毒特有的新型鞘脂类物质, 这两者是否都通过线粒体途径, 导致细胞色素C的释放, 活化metacasepase, 引起线粒体结构和功能改变, 进而促进凋亡的发生?综上所述, 本研究系统地分析了EhPLC结构特点及可能的功能, 为进一步开展EhPLC细胞定位及其参与的信号通路与环境胁迫之间关系的研究提供了重要的理论指导。
4 结论自然海域中, 病毒感染和裂解球石藻过程中伴随着大量球石粒CaCO3和DMSP的释放, 特别是其赤潮的瓦解有赖于病毒裂解而使其在全球碳、硫生物地化循环、气候变化过程中起着十分重要的作用。磷脂酶C作为一种重要的细胞信号分子, 在浮游植物环境胁迫应答及病原菌侵染过程中发挥重要作用。随着球石藻及其特异性病毒全基因组序列的注释和深入分析, 病毒与宿主间复杂的信号转导网络研究引起了学者们的极大兴趣。本研究基于前人研究成果, 对海洋球石藻PLC进行了系统的生物信息学分析, 明确该藻中存在30个典型的PLC并分属于四大类型; 它们在信号肽、亚细胞定位及二级结构组成等方面上存在一定差异, 推测上述蛋白在不同位置发挥不同作用; 多数EhPLC具有典型的PLCX、PLCY及C2等保守的结构域; 系统发育分析结果显示EhPLC与植物的相似度很低, 而与低等动物的相似度相对较高。该研究补充了对浮游植物PLC信号转导网络系统的认识, 并对进一步开展海洋球石藻PLC细胞定位及其参与的信号通路与环境胁迫之间关系的研究具有重要的理论意义。
| 王法微, 王骐, 邓宇, 等, 2014. 磷脂酶C基因家族研究进展. 生物技术通报,(12): 33–39 |
| 李莉, 井文, 章文华, 2015. 植物细胞中磷酸肌醇和磷脂酶C介导的信号转导. 植物生理学报, 51(10): 1590–1596 |
| 罗磊, 2014. 神经酰胺在外源化学物诱导线粒体依赖性细胞凋亡中的作用. 卫生研究, 43(6): 1031–1037 |
| 金道忠, 朱兴族, 2006. 神经酰胺代谢及凋亡信号调节. 生命科学, 18(5): 481–486 |
| 赵凤命, 张谈顺, 王仲兵, 等, 2012. 钙离子与细胞凋亡. 中国动物检疫, 29(2): 67–71 |
| 黄冬, 吴燕, 2017. 植物磷脂酶C的功能研究进展. 生命科学, 29(6): 575–581 |
| 韩长志, 2016. 禾谷炭疽菌磷脂酰肌醇特异性磷脂酶C生物信息学分析. 科学技术与工程, 16(2): 18–23 |
| Awasthi M, Batra J, Kateriya S, 2012. Disulphide bridges of phospholipase C of Chlamydomonas reinhardtii modulates lipid interaction and dimer stability. PLoS One, 7(6): e39258 DOI:10.1371/journal.pone.0039258 |
| Bardo S, Cavazzini M G, Emptage N, 2006. The role of the endoplasmic reticulum Ca2+ store in the plasticity of central neurons. Trends Pharmacol Sci, 27(2): 78–84 DOI:10.1016/j.tips.2005.12.008 |
| Bidle K D, Haramaty L, Barcelos J B E, et al, 2007. Viral activation and recruitment of metacaspases in the unicellular coccolithophore, Emiliania huxleyi. Proc Natl Acad Sci, 104(14): 6049–6054 DOI:10.1073/pnas.0701240104 |
| Blanco-Ameijeiras S, Lebrato M, Stoll H M, et al, 2016. Phenotypic variability in the coccolithophore Emiliania huxleyi. PLoS One, 11(6): e0157697 DOI:10.1371/journal.pone.0157697 |
| Evans C, Pond D W, Wilson W H, 2009. Changes in Emiliania huxleyi fatty acid profiles during infection with E. huxleyi virus 86:physiological and ecological implications. Aquat Microb Ecol, 55(3): 219–228 DOI:10.3354/ame01295 |
| Franklin D J, Brussaard C P D, Berges J A, 2006. What is the role and nature of programmed cell death in phytoplankton ecology?. Eur J Phycol, 41(1): 1–14 DOI:10.1080/09670260500505433 |
| Gordeeva A V, Labas Y A, Zvyagilskaya R A, 2004. Apoptosis in unicellular organisms:mechanisms and evolution. Biochemistry, 69(10): 1055–1066 |
| Halder G, Johnson R L, 2011. Hippo signaling:growth control and beyond. Development, 138(1): 9–22 |
| Hergovich A, 2012. Mammalian Hippo signalling:a kinase network regulated by protein-protein interactions. Biochem Soc Trans, 40(1): 124–128 DOI:10.1042/BST20110619 |
| Hofmann K, Bucher P, 1995. The FHA domain:a putative nuclear signalling domain found in protein kinases and transcription factors. Trends Biochem Sci, 20(9): 347–349 DOI:10.1016/S0968-0004(00)89072-6 |
| Hough E, Hansen L K, Birknes B, et al, 1989. High-resolution (1.5Å) crystal structure of phospholipase C from Bacillus cereus. Nature, 338(6213): 357–360 DOI:10.1038/338357a0 |
| Joassin P, Delille B, Soetaert K, et al, 2011. Carbon and nitrogen flows during a bloom of the coccolithophore Emiliania huxleyi:modelling a mesocosm experiment. J Mar Syst, 85(3-4): 71–85 DOI:10.1016/j.jmarsys.2010.11.007 |
| Kadenbach B, Arnold S, Lee I, et al, 2004. The possible role of cytochrome c oxidase in stress-induced apoptosis and degenerative diseases. Biochem Biophys Acta, 1655: 400–408 |
| Lemtiri-Chlieh F, MacRobbie E A C, Webb A A R, et al, 2003. Inositol hexakisphosphate mobilizes an endomembrane store of calcium in guard cells. Proc Natl Acad Sci, 100(17): 10091–10095 DOI:10.1073/pnas.1133289100 |
| Liu J W, Bratbak G, Zheng T L, et al, 2011. Effects of virus infection on expression of cell cycle regulatory proteins in the unicellular marine algae Emiliania huxleyi. Acta Oceanol Sin, 30(4): 89–95 DOI:10.1007/s13131-011-0138-z |
| Liu J W, Xu M M, Zheng T L, 2015. A minireview of marine algal virus-Coccolithoviruses. J Ocean Univ China, 14(2): 293–300 DOI:10.1007/s11802-015-2623-z |
| Martínez J M, Schroeder D C, Wilson W H, 2012. Dynamics and genotypic composition of Emiliania huxleyi and their co-occurring viruses during a coccolithophore bloom in the North Sea. FEMS Microbiol Ecol, 81(2): 315–323 DOI:10.1111/fem.2012.81.issue-2 |
| Michaelson L V, Dunn T M, Napier A, 2010. Viral trans-dominant manipulation of algal sphingolipids. Trends Plant Sci, 15(12): 651–655 DOI:10.1016/j.tplants.2010.09.004 |
| Michell R H, 2008. Inositol derivatives:evolution and functions. Nat Rev Mol Cell Biol, 9(2): 151–161 DOI:10.1038/nrm2334 |
| Mikami K, 2014. Structural divergence and loss of phosphoinositide-specific phospholipase C signaling components during the evolution of the green plant lineage:implications from structural characteristics of algal components. Front Plant Sci, 5: 380 |
| Munnik T, Testerink C, 2009. Plant phospholipid signaling:"in a nutshell". J Lipid Res, 50(S): S260–S265 |
| Nakamura Y, Awai K, Masuda T, et al, 2005. A novel phosphatidylcholine-hydrolyzing phospholipase C induced by phosphate starvation in Arabidopsis. J Biol Chem, 280(9): 7469–7476 DOI:10.1074/jbc.M408799200 |
| Nomikos M, Mulgrew-Nesbitt A, Pallavi P, et al, 2007. Binding of phosphoinositide-specific phospholipase C-ζ (PLC-ζ) to phospholipid membranes:potential role of an unstructured cluster of basic residues. J Biol Chem, 282(22): 16644–16653 DOI:10.1074/jbc.M701072200 |
| Port J A, Parker M S, Kodner R B, et al, 2013. Identification of G protein-coupled receptor signaling pathway proteins in marine diatoms using comparative genomics. BMC Genomics, 14: 503–518 DOI:10.1186/1471-2164-14-503 |
| Rose S L, Fulton J M, Brown C M, et al, 2014. Isolation and characterization of lipid rafts in Emiliania huxleyi:a role for membrane microdomains in host-virus interactions. Environ Microbiol, 16(4): 1150–1166 DOI:10.1111/emi.2014.16.issue-4 |
| Rosenwasser S, Mausz M A, Schatz D, et al, 2014. Rewiring host lipid metabolism by large viruses determines the fate of Emiliania huxleyi, a bloom-forming alga in the ocean. Plant Cell, 26(6): 2689–2707 DOI:10.1105/tpc.114.125641 |
| Schatz D, Shemi A, Rosenwasser S, 2014. Hijacking of an autophagy-like process is critical for the life cycle of a DNA virus infecting oceanic algal blooms. New Phytol, 204(4): 854–863 DOI:10.1111/nph.13008 |
| Singh A, Kanwar P, Pandey A, et al, 2013. Comprehensive genomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in Rice. PLoS One, 8(4): e62494 DOI:10.1371/journal.pone.0062494 |
| Suh P G, Park J I, Manzoli L, et al, 2008. Multiple roles of phosphoinositide-specific phospholipase C isozymes. BMB Rep, 41(6): 415–434 DOI:10.5483/BMBRep.2008.41.6.415 |
| Tasma I M, Brendel V, Whitham S A, et al, 2008. Expression and evolution of the phosphoinositide-specific phospholipase C gene family in Arabidopsis thaliana. Plant Physiol Biochem, 46(7): 627–637 DOI:10.1016/j.plaphy.2008.04.015 |
| Vermont A I, Martínez J M, Waller J D, et al, 2016. Virus infection of Emiliania huxleyi deters grazing by the copepod Acartia tonsa. J Plankton Res, 38(5): 1194–1205 DOI:10.1093/plankt/fbw064 |
| Wheeler G L, Brownlee C, 2008. Ca2+ signalling in plants and green algae-changing channels. Trends Plant Sci, 13(9): 506–514 DOI:10.1016/j.tplants.2008.06.004 |
| Zheng S Z, Liu Y L, Li B, et al, 2012. Phosphoinositide-specific phospholipase C9is involved in the thermotolerance of Arabidopsis. Plant J, 69(4): 689–700 DOI:10.1111/tpj.2012.69.issue-4 |
 2018, Vol. 49
2018, Vol. 49


