
中国海洋湖沼学会主办。
文章信息
- 房祖业, 陈晓东, 吴咏诗, 谭洁瑢, 张玮恩, 王正宇, 林婷婷, 查广才, 舒琥. 2018.
- FANG Zu-Ye, CHEN Xiao-Dong, WU Yong-Shi, TAN Jie-Rong, ZHANG Wei-En, WANG Zheng-Yu, LIN Ting-Ting, ZHA Guang-Cai, SHU Hu. 2018.
- 大刺鳅(Mastacembelus armatus)二、三、四碱基重复微卫星标记的筛选和特征分析
- SCREENING AND CHARACTERISTIC ANALYSIS ON DI-/TRI-/TETRA-NUCLEOTIDE-REPEATED MICROSATELLITES IN MASTACEMBELUS ARMATUS
- 海洋与湖沼, 49(1): 174-182
- Oceanologia et Limnologia Sinica, 49(1): 174-182.
- http://dx.doi.org/10.11693/hyhz20170600172
-
文章历史
- 收稿日期:2017-06-26
- 收修改稿日期:2017-07-30
2. 韩山师范学院食品工程与生物科技学院 潮州 521041
2. School of Food Engineering and Biotechnology, Hanshan Normal University, Chaozhou 521041, China
大刺鳅(Mastacembelus armatus)俗称辣锥、刀鳅、石锥等, 隶属于合鳃目(Synbranchiformes)、刺鳅科(Mastacembelidae)、刺鳅属(Mastacembelus) (Nelson, 2006)。在全球主要分布于东南亚、非洲等地区, 在我国主要分布在南部地区, 如福建、广东、广西、海南和云南等地(《福建鱼类志》编写组, 1985; 郑慈英, 1989; 中国水产科学研究院珠江水产研究所, 1991; 广西壮族自治区水产研究所等, 2006)。大刺鳅肉质鲜美, 营养丰富, 属于名贵淡水鱼类(蔡雪芹, 2006), 具有很高的经济价值。但是由于近年来长期的过度捕捞和江河环境污染, 使得大刺鳅野生资源遭到严重破坏, 面临着逐渐枯竭的严峻形势, 已被广东、福建、湖南等省列为重点保护野生水生动物(陈忠, 2005)。
微卫星分子标记拥有通用性好, 多态信息容量高, 突变率高, 重复性、共显性好等优点(孙效文等, 2008), 在鱼类遗传育种中得到广泛应用, 可见于鱼类种群结构(Carvalho et al, 1994)、亲缘关系鉴定(Brooker et al, 1994)、种群遗传多样性(史方等, 2009)、遗传图谱构建及QTL定位等(宋国华等, 2005; 刘金亮, 2011)方面。在鱼类研究中多以二碱基重复微卫星引物的应用为主, 如赵哲霞等(2014)对黄颡鱼(Pelteobagrus fulvidraco)进行遗传多样性研究所采用的20对微卫星引物均为二碱基重复, 海萨等(2008)对鲈形目河鲈(Perca fluviatilis)的二碱基重复微卫星引物进行筛选, 但是近年来在有尾目动物(Sequeira et al, 2005; 王慧等, 2012)和草鱼(Ctenopharyngodon idellus)、鲢鱼(Hypophthalmichthys molitrix)、鲤鱼(Cyprinus carpio)等鱼类研究中发现三、四碱基重复位点较二碱基重复具有更高的筛选效率和多态性(鲁翠云, 2009; 李文升等, 2011; 谭照君等, 2011)。林婷婷(2016)用二碱基重复微卫星引物对不同群体大刺鳅进行了遗传多样性分析, 舒琥等(2017)对华南地区7个大刺鳅野生群体的形态差异进行了分析, 但未见有应用三、四碱基重复微卫星引物对大刺鳅进行研究分析的报道。本文通过筛选出多态性高的二、三、四碱基重复微卫星引物对大刺鳅的遗传多样性进行研究, 并比较三种碱基重复微卫星引物的筛选效率和多态性, 为二、三、四碱基重复微卫星引物在鱼类遗传多样性研究提供理论基础, 为大刺鳅及经济鱼类的遗传多样性评估和种质资源保护提供参考。
1 材料与方法 1.1 样本采集和基因组DNA提取2015年8月—2016年8月, 在广东北江(平均体重17.88g, 平均体长19.44cm)和云南澜沧江(平均体重24.99g, 平均体长20.89cm)分别采集大刺鳅全鱼样本各30尾, 置于90%酒精保存并带回实验室。每个样本取背部肌肉25mg, 采用上海生工Ezup基因组提取试剂盒提取基因组DNA。用紫外分光光度计测定浓度和纯度, 定量至100ng/μL。
1.2 微卫星引物的设计选取完整性和纯度高的单个个体DNA送至广州基迪奥生物科技有限公司进行基因组高通量测序, 利用生物信息学方法, 根据各位点重复序列两端的侧翼序列选择性设计出105对引物, 重复次数在5—27之间, 二碱基重复次数在15—25之间, 三碱基重复次数在8—11之间, 四碱基重复次数在5—21之间, 引物由上海美吉生物医药科技有限公司合成。
1.3 大刺鳅微卫星引物的筛选选用北江群体的大刺鳅30尾的基因组DNA作为模板, 对引物进行初步筛选。PCR反应总体系为24μL, 其中Mix 12.5μL, 50ng/μL DNA模板2μL, 50μmol/L引物各1μL, ddH2O 7.5μL。反应条件为94℃预变性5min, 94℃变性40s, 以适宜温度退火45s(部分见表 1), 72℃延伸90s, 循环35次, 72℃继续延伸10min。PCR产物1.5%琼脂糖凝胶电泳检测。从105个引物中筛选出能稳定扩增且产物与预期大小一致的引物共50对, 其片段长度均在104—271bp。然后在8%非变性聚丙烯酰胺凝胶电泳下对50对引物进行多态性检测, 并通过调整扩增条件(如退火温度、Mg2+浓度或循环次数)筛选出多态性较高、带型较清晰、重复性较好的引物。
| 位点 | 引物序列(5′→3′) | 重复序列 | 片段长度(bp) | 退火温度(Tm) |
| ER2 | F:TATTGAATGTAAACGGCGGCR:GCTTTGAATGAGACGCACAC | (CA)17 | 104 | 56 |
| ER5 | F:AATTCCTACGAGCCACATGCR:AGTTCATTGGCACCCTGAAA | (GT)14 | 114 | 58 |
| ER6 | F:GGGGATATTGAGAAGGACAAAR:ACCAAAAAGGCAACCCTAGA | (AC)19 | 109 | 59 |
| ER9 | F:AGTTCTGTTAACCTCTAACCTCCAR:TAGCTGAGCGTGGTGCTTTA | (TG)22 | 204 | 58 |
| ER10 | F:TCGATAGCCCAATGTTCCTCR:TCCCGATCCATTCAGAAGAC | (CA)20 | 174 | 57 |
| ER15 | F:TGTTTTAGTGTGCCACAGGGR:TGCACATTTTCCCCTCTTTC | (AC)22 | 229 | 56 |
| ER18 | F:AAATAAGCAGAGGGGGTTGGR:AGACCTGGAGCAGCTTTCAA | (GT)16 | 137 | 56 |
| ER22 | F:GTGTTCAGCCCACTCACTCAR:GGAGCACTGTGCAGACAGAA | (CA)25 | 141 | 57 |
| ER27 | F:ATTCACCATGGGAACACAGGR:GTGCTATCCGTGTGAGGGTT | (AC)21 | 260 | 58 |
| ER29 | F:CCAGGACATCTACAGGCGATR:ATGTTTTGCTGCTGTTGCTG | (CA)21 | 152 | 58 |
| SAN1 | F:TTCACTGAAGAATGAACCAGTTGR:GCACATGCAGACATAAACTGC | (TTA)9 | 112 | 57 |
| SAN3 | F:TCTGCCAAATGGCTGTGTTAR:CCTGGCAACAGTGGGATAGT | (TTA)9 | 157 | 57 |
| SAN7 | F:GCCATAGAGGAGACTGTCAGCR:TCCGTGAAGAGTTTCAGCCT | (AAT)9 | 176 | 59 |
| SAN8 | F:TGTATCCAAAATGGGGGAAAR:GTGGGACAAAAGGAAGACCA | (ATT)11 | 122 | 55 |
| SAN9 | F:GAAGATGAAAGAACAGCGCCR:TTGCATTATACAGCTGGGGG | (AAT)9 | 209 | 57 |
| SAN15 | F:GGCTGTAGTTCCATGCACAGR:ACCACCATGTGGCCATTTAT | (TTC)10 | 271 | 57 |
| SAN16 | F:AGATGCAGAAGGATTGGAGGR:TGCCACAGGCTGTTGATAAT | (TAA)9 | 212 | 56 |
| SAN17 | F:AGCACAGCAACAGCAACAACR:CCCATAACACAGCAAAACAAAA | (CAG)9 | 115 | 56 |
| SAN19 | F:CCGATTATCAGGAAATGGGAR:GGGGTCAGTGTGCTGTTTTT | (TGA)9 | 201 | 56 |
| SAN20 | F:CGCTGGTTTCACCACTTTTTR:TGACGACTGCTGCTGCTAAT | (TAT)9 | 236 | 56 |
| SAN21 | F:GCAGTGACAATGATCCACGAR:AATGATAGATTCCCACAGGGG | (GCT)9 | 252 | 58 |
| SAN23 | F:ATTTGACCCCTCCCAATACCR:AATGATAGATTCCCACAGGGG | (AGG)9 | 155 | 58 |
| SAN26 | F:TGGTGTAAATGCACAGCCATR:TAAACGAATCAGTCCCCGAC | (TAA)8 | 196 | 56 |
| SAN29 | F:CAAACCAGCCTCCATCATTTR:AATGATAGATTCCCACAGGGG | (TTA)8 | 211 | 58 |
| SI1 | F:ACTTCGTCCACCAGGATGAGR:CGAAGCACATGAGAACCAGA | (AAGA)12 | 251 | 58 |
| SI2 | F:TAAGCTAGTGGCATGGCAAAR:TGAAGGTCGTGACTGATTGC | (ATAG)21 | 138 | 56 |
| SI6 | F:GGTGGAGGTACTCAAGCTGCR:CTTTCACCCGAGAGAAGCTG | (TCCA)7 | 168 | 60 |
| SI7 | F:GCCTTTGCAATGTTGGTTTTR:CCTGGTTAAGGAATAAGGGGG | (ATCC)7 | 185 | 56 |
| SI8 | F:AACTGCACCTTAAAAGCAATTACAR:CGACACAACACACGTCAGTG | (ATAG)6 | 156 | 57 |
| SI9 | F:GCCCACAGAAAGGTTGTGTTR:TGGTAACTGATCATTTTTGTATTTTCA | (TATT)7 | 155 | 56 |
| SI10 | F:TTAAAGCCTCCAAATCGCTGR:CCCATGAAGTTTCACATCCA | (AAAG)7 | 270 | 56 |
| SI11 | F:ACTTGTCAGATAGACGGGGCR:GGTGTGTGGAGACTACGCCT | (ATCA)5 | 234 | 60 |
| SI12 | F:TGGTTACGAAGAACATGGCAR:TGTTGTAGTGTGTGGCCCTG | (TTAT)6 | 185 | 57 |
| SI17 | F:CCTGTGACCCTGGTTAAGGAR:GAATGACCATTGTGATGAATCC | (GATG)8 | 248 | 57 |
| SI18 | F:GATGGCACACCAGCAGTAAAR:CCTGTGACCCTGGTTAAGGA | (ATCC)6 | 109 | 57 |
| SI20 | F:CCTTTCACCCAAGTGATGCTR:ATTTTTACCAGTGGCACGCT | (ATGG)6 | 142 | 56 |
| SI24 | F:TAAGGGACAGGTGGACGGTAR:CAGCGCTGTAGAACTTTGCTT | (TGAA)5 | 177 | 58 |
| SI25 | F:TCCTCCCACAGTCCAAAAACR:TGAATCGTTGTGTGTCAGGAA | (ATGG)5 | 265 | 57 |
筛选的引物用来合成5'上游荧光引物(FAM、HEX、TEMED)。用荧光引物对北江和澜沧江大刺鳅群体各30尾的模板DNA进行PCR扩增, 扩增体系为10μL反应体系: Mix 5.0μL、50μmol/L荧光引物F为0.5μL, 50μmol/L引物R为0.5μL, 50ng/μL模板DNA 1.0μL、ddH2O 3.0μL, 扩增后的荧光PCR产物送上海生工生物工程有限公司进行STR测序分析。
1.5 数据处理用PopGene软件统计各微卫星座位的等位基因频率(allele frequency, P)、观测等位基因数(observed number of alleles, Na)、有效等位基因数(effective number of alleles, Ne)、观测杂合度(observed heterozygosity, Ho)期望杂合度(expected heterozygosity, He) (Yeh et al, 1999)。用PIC-CALC计算微卫星位点的多态信息含量(polymorphism information content, PIC)。本实验将不同重复序列的微卫星的筛选率定义为:最终筛选得到的稳定且多态性高的相应重复微卫星位点数目/最初设计并用于筛选的相应重复微卫星的序列数× 100%。
2 结果与分析 2.1 二、三、四碱基重复微卫星引物筛选结果的比较在本研究设计的105对引物(二碱基重复20对、三碱基重复45对、四碱基重复40对)中, 有50对能稳定扩增出目的条带。实验结果显示有38个位点表现出多态性并且扩增条带清晰, 其中二碱基重复的10对微卫星引物均表现出多态性, 筛选效率为10/20×100% = 50%, 而三、四碱基重复均有14对, 因此三碱基重复的筛选效率为14/45×100% = 31.1%, 四碱基重复的筛选效率为14/40×100% = 35%。位点ER6在北江大刺鳅个体中的稳定性和多态性检测见图 1。
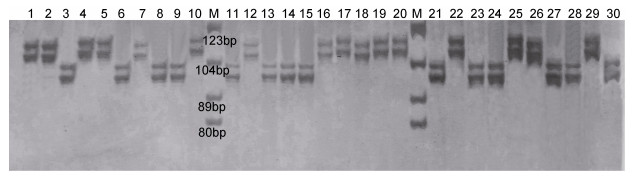 |
| 图 1 微卫星引物ER6在北江大刺鳅个体中的扩增结果 Fig. 1 The amplification results of Beijiang M. armatus using microsatellite primer ER6 注:图中M为DNA Marker; 1—30泳道为ER6位点对北江群体的30个大刺鳅个体的扩增图谱 |
从扩增稳定的引物中随机选择二、三、四碱基各10对, 对北江群体30尾大刺鳅进行多态性检测和分析, 结果显示, 30个微卫星位点共扩增出235个等位基因, 数量最多的是SI2共检测出20个等位基因; 等位基因数在3—20, 二碱基微卫星平均等位基因数(9.500)比三碱基重复(6.000)和四碱基重复(8.000)的高, 这些等位基因片段大小均在104—271bp; 有效等位基因数在1.544—12.766;观察杂合度(Ho)在0.233—1.000;期望杂合度变动范围为0.358—0.937;多态信息含量PIC在0.311—0.916。对北江野生群体的遗传结构进行评估, 结果显示, 等位基因数(Na)、有效等位基因数(Ne)、观察杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)这五项多态评估指标平均数分别为7.833、4.008、0.873、0.702、0.649, 而且基于以上五项评估指标对二、三、四碱基微卫星引物进行多态性比较结果:二碱基(PIC=0.735)高于四碱基(PIC=0.605)、三碱基(PIC= 0.606)。30对微卫星引物信息以及其对北江群体大刺鳅的遗传多样性评估见表 2。
| 位点 | 等位基因数(Na) | 有效等位基因数(Ne) | 观测杂合度(Ho) | 期望杂合度(He) | 多态信息含量(PIC) |
| ER2 | 8 | 2.765 | 1.000 | 0.649 | 0.578 |
| ER5 | 8 | 4.813 | 1.000 | 0.806 | 0.768 |
| ER6 | 8 | 4.380 | 0.967 | 0.785 | 0.741 |
| ER9 | 8 | 3.750 | 0.967 | 0.746 | 0.696 |
| ER10 | 9 | 5.294 | 1.000 | 0.825 | 0.787 |
| ER15 | 5 | 2.965 | 0.667 | 0.674 | 0.622 |
| ER18 | 15 | 10.112 | 1.000 | 0.916 | 0.893 |
| ER22 | 14 | 7.807 | 1.000 | 0.884 | 0.859 |
| ER27 | 10 | 6.949 | 0.944 | 0.868 | 0.840 |
| ER29 | 10 | 2.760 | 1.000 | 0.647 | 0.570 |
| SAN7 | 6 | 2.880 | 0.833 | 0.664 | 0.615 |
| SAN8 | 6 | 3.377 | 0.467 | 0.716 | 0.667 |
| SAN15 | 6 | 3.377 | 1.000 | 0.7158 | 0.657 |
| SAN16 | 5 | 2.605 | 0.700 | 0.627 | 0.574 |
| SAN17 | 7 | 3.285 | 0.967 | 0.707 | 0.643 |
| SAN19 | 4 | 2.643 | 0.967 | 0.632 | 0.557 |
| SAN20 | 10 | 3.352 | 0.933 | 0.714 | 0.676 |
| SAN21 | 7 | 3.529 | 0.967 | 0.729 | 0.670 |
| SAN23 | 5 | 2.280 | 0.967 | 0.572 | 0.467 |
| SAN29 | 4 | 2.400 | 0.733 | 0.593 | 0.530 |
| SI1 | 9 | 5.788 | 1.000 | 0.841 | 0.806 |
| SI2 | 20 | 12.766 | 1.000 | 0.937 | 0.916 |
| SI6 | 7 | 2.922 | 1.000 | 0.669 | 0.598 |
| SI10 | 7 | 2.368 | 0.767 | 0.588 | 0.533 |
| SI11 | 7 | 3.103 | 0.867 | 0.689 | 0.623 |
| SI12 | 6 | 2.456 | 0.633 | 0.603 | 0.558 |
| SI17 | 8 | 2.683 | 0.933 | 0.638 | 0.570 |
| SI18 | 4 | 1.544 | 0.233 | 0.358 | 0.311 |
| SI20 | 8 | 2.532 | 0.733 | 0.615 | 0.567 |
| SI24 | 4 | 2.765 | 0.933 | 0.649 | 0.565 |
从对北江群体分析的微卫星引物中随机选择二、三、四碱基各6对, 对澜沧江群体30尾大刺鳅进行多态性检测。用PopGene软件对这18对引物扩增的产物进行多态性评价分析, 结果显示, 18个微卫星位点等位基因数变动范围为2—20, 平均等位基因数为6.722, 其中SAN21位点仅获得的2个等位基因数, 位点SI1获得20个等位基因数, 有效等位基因数在1.455—7.258, 平均值为3.224, 观测杂合度(Ho)在0.367—1.000, 平均值为0.822;而期望杂合度(He)在0.318—0.862, 平均值为0.509。用PIC—CALC软件来分析多态信息含量, 多态信息含量PIC在0.283— 0.847, 平均值为0.572, 位点SAN16的PIC最低, 仅为0.283, 而SI2的PIC值最高, 为0.847。与北江一致的是基于五项评估指标, 二碱基重复微卫星的多态性均比三四碱基重复的高, 其中二碱基(PIC=0.627) > 四碱基(PIC=0.584) > 三碱基(PIC=0.506)。18对微卫星引物对澜沧江群体大刺鳅的遗传多样性评估见表 3, 以及两个群体的遗传多样性比较见表 4。
| 位点 | 等位基因数(Na) | 有效等位基因数(Ne) | 观测杂合度(Ho) | 期望杂合度(He) | 多态信息含量(PIC) |
| ER2 | 4 | 3.509 | 0.867 | 0.727 | 0.663 |
| ER5 | 6 | 2.980 | 0.933 | 0.676 | 0.605 |
| ER15 | 7 | 3.543 | 0.933 | 0.730 | 0.673 |
| ER18 | 10 | 5.522 | 0.767 | 0.833 | 0.796 |
| ER22 | 5 | 2.715 | 0.800 | 0.642 | 0.582 |
| ER27 | 5 | 2.195 | 0.967 | 0.554 | 0.445 |
| SAN7 | 9 | 3.056 | 0.933 | 0.684 | 0.617 |
| SAN8 | 10 | 4.511 | 0.967 | 0.722 | 0.750 |
| SAN15 | 5 | 1.822 | 0.567 | 0.459 | 0.414 |
| SAN16 | 3 | 1.484 | 0.400 | 0.332 | 0.283 |
| SAN17 | 6 | 2.885 | 0.967 | 0.664 | 0.598 |
| SAN21 | 2 | 2.000 | 1.000 | 0.509 | 0.375 |
| SI1 | 20 | 5.769 | 0.900 | 0.841 | 0.819 |
| SI2 | 11 | 7.258 | 1.000 | 0.862 | 0.847 |
| SI6 | 5 | 2.941 | 0.900 | 0.671 | 0.618 |
| SI10 | 6 | 2.490 | 0.867 | 0.609 | 0.531 |
| SI11 | 4 | 1.901 | 0.667 | 0.482 | 0.401 |
| SI18 | 3 | 1.455 | 0.367 | 0.318 | 0.285 |
| 群体 | 重复序列类型 | 座位数目 | 等位基因数(Na) | 有效等位基因数(Ne) | 观测杂合度(Ho) | 期望杂合度(He) | 多态信息含量(PIC) |
| 北江 | 二碱基重复 | 10 | 9.500 | 5.160 | 0.954 | 0.780 | 0.735 |
| 三碱基重复 | 10 | 6.000 | 2.973 | 0.853 | 0.667 | 0.606 | |
| 四碱基重复 | 10 | 8.000 | 3.893 | 0.810 | 0.659 | 0.605 | |
| 平均值 | 10 | 7.833 | 4.008 | 0.873 | 0.702 | 0.649 | |
| 澜沧江 | 二碱基重复 | 6 | 6.167 | 3.411 | 0.878 | 0.694 | 0.627 |
| 三碱基重复 | 6 | 5.833 | 2.626 | 0.806 | 0.562 | 0.506 | |
| 四碱基重复 | 6 | 8.167 | 3.636 | 0.783 | 0.630 | 0.584 | |
| 平均值 | 6 | 6.722 | 3.224 | 0.822 | 0.629 | 0.572 |
微卫星引物是指以微卫星核心序列两侧的侧翼序列设计的具有有效扩增DNA模板的一对碱基序列。本研究通过简化基因组测序, 筛选了105对微卫星引物, 通过不断优化PCR条件, 如退火温度, 循环次数等, 最终获得50对能在大刺鳅群体中稳定扩增的微卫星引物, 并测出其中有38对标记在大刺鳅群体中表现出多态性, 总筛选效率为36.2%, 筛选效率较高, 证明了该方法适用于大刺鳅微卫星位点的规模化筛选, 与韩承慧(2016)的看法一致。而刘瑞成等(2013)在宽口光唇鱼(Acrossocheilus monticola)的微卫星开发中, 163个合成的微卫星引物中仅得到18个多态性高的微卫星引物, 筛选效率仅为11.04%。本研究筛选出的二、三、四碱基微卫星引物重复次数都在30以内, 这与Ellegren(2002)提出的真核生物微卫星大部分都在30次重复以下一致。其中二碱基重复引物以15—19次重复为主, 三碱基重复引物以8—9次重复为主, 四碱基重复引物以5—7次重复为主。龚小玲等(2009)在对澳洲鳗鲡(Anguilla japonica)进行微卫星标记开发时发现, 微卫星重复序列的重复次数过高会影响PCR扩增效果。另有55对引物条件优化后进行聚丙烯酰胺电泳检测, 仍不能扩增产生稳定的特异性条带。可能是因为微卫星位点扩增DNA时容易形成发夹结构, 使得引物扩增不稳定或形成不了扩增产物, 导致引物质量不高。
3.2 大刺鳅二、三、四碱基重复微卫星的多态性比较及两个群体评估效果对北江和澜沧江群体进行遗传多样性分析的微卫星引物中, 二碱基重复引物的多态信息含量最高, 分别为0.735与0.627;其次是四碱基重复引物, 为0.605与0.584;最低是三碱基重复引物, 为0.606与0.506。从以上数据可看出, 二碱基引物在大刺鳅种群中应用效率最高, 验证了Brenner等(1993)提出的在鱼类基因组中, 二碱基微卫星引物在其基因组中数量最为丰富并且应用所占比重最大的说法(Crooijmans et al, 1997; Maguire et al, 2000; Tóth et al, 2000; Serapion et al, 2004)。另一方面, Weber(1990)表示, 当二碱基重复序列重复次数大于12次时, 微卫星标记或许能够表现出较高的PIC; 二碱基重复次数在16次或以上时, 可提供的PIC在0.5以上, 相应的多态性分析才可进行。本次研究在北江群体中使用的二碱基微卫星引物重复序列的重复次数均为19以上, PIC均超过0.5, 均可应用于种群间遗传多样性分析。
多态信息含量是评价种群遗传变异程度高低的重要指标, 当PIC > 0.5时, 该基因位点表现为高度多态性; 0.25 < PIC < 0.5时, 该位点表现为中度多态性; PIC < 0.25时, 该位点表现为低度多态性(Botstein et al, 1980), Feresu-Shonhiwa等(1998)认为PIC > 0.70时该微卫星标记是最理想的标记, 本研究筛选出来的30个微卫星位点中有28个位点表现出高度多态性, 占93.3%, 有8个位点(ER5、ER6、ER10、ER22、ER27、SI1、SI2) PIC > 0.7, 其中SI2具有最高的PIC为0.916;另有2个位点SAN23、SI18的PIC在0.311—0.467, PIC > 0.25, 表现为中度多态性, 占6.67%。因而本研究中所制备的微卫星将在大刺鳅的群体遗传多样性分析、遗传连锁分析等方面都会起到一定的作用。
遗传多样性丰富的生物能够更好地适应各种环境, 进行遗传多样性的分析, 是对物种进行种质资源保护的基础。本研究应用30对引物对北江群体扩增的平均等位基因数为7.833, 而18对引物扩增澜沧江群体的平均等位基因为6.722, 且二、三、四碱基重复微卫星引物对五项评估指标(Na、Ne、He、Ho、PIC)比较均为北江群体的遗传多样性比澜沧江的高, 这与王方(2012)认为澜沧江种群的遗传多样性较低的结果一致。其可能原因是北江群体有二次接触, 可显著提高群体的遗传多样性, 而澜沧江群体遗传多样性较低可能与长时间的地理隔离而不能进行必要的基因交流有关。此外还可能与生活环境遭到严重破坏和人为过度捕捞有关。两个群体的观测杂合度Ho均比期望杂合度He高, 表明大刺鳅遗传多样性高, 变异率高且存在杂合子过剩的可能。杨华强等(2016)提到, 与刀鲚(Coilia macrognathos Bleeker)、草鱼(C. idellus)、赤眼鳟(Squaliobarbus ourriculus)、翘嘴红鲌(Erythroculter ilishaeformis)、黄鳝(Monopterus albus)和河川沙塘鳢(Odontobutis potamophila)比较, 大刺鳅的遗传多样性水平较高。尽管如此, 仍存在野生资源少的问题, 主要还是人为因素造成的种群结构失调所致, 这也为今后对大刺鳅进行种质资源保护提供了方向。
3.3 筛选意义及展望前景本实验主要从Na、Ne、He、Ho、PIC这五项评估指标来分析种群的遗传多样性, 其中前三项为检测遗传变异的重要指标, 后两项是反映微卫星位点中杂合子基因型所占的比例大小, 从位点的遗传变异水平衡量群体的遗传变异水平。经这五项评估指标鉴定, 本实验筛选的二、三、四碱基重复微卫星引物均适用于大刺鳅的遗传多样性研究。结果表明大刺鳅二碱基重复微卫星引物较三、四碱基重复具有更高的筛选效率和遗传稳定性, 这与其他鱼类研究中三、四碱基微卫星标记具有更高的多态性, 基因分型效果较二碱基微卫星好(Hearneet al, 1992; Diwan et al, 1997; 鲁翠云等, 2009)的结果不同, 高焕等(2005)认为四碱基重复类型处于非编码区, 进化压力小可“自由突变”, 笔者认为虽然如此, 但四碱基存在着重复次数少、序列丰富度低和突变率低等劣势, 刘瑞成等(2013)认为二、四碱基重复微卫星引物是宽口光唇鱼(A. monticola)的主要微卫星重复类型, 三碱基重复则较少出现。离体复制中, 二碱基重复类型微卫星突变序列的突变率显著高于三碱基重复类型(Schlötterer et al, 1992; Hite et al, 1996)。鲁翠云(2009)在鲤鱼研究中得出四碱基重复多态性较好, 认为与其长期进化过程中基因组加倍有关。不同结果可能与物种的特异性有关, 也可能与实验选取引物数量及类型的随机性和样本的量有关。
本实验筛选的部分三、四碱基重复微卫星位点具有很高的多态性, 如ER5、ER6、ER10、ER22、ER27、SI1、SI2等是三、四碱基重复微卫星标记的最理想标记。本研究首次筛选并将三、四碱基重复微卫星应用于大刺鳅的种群分析中, 发现虽然三、四碱基微卫星相对于二碱基多态性较弱, 但依然适用于大刺鳅群体的遗传多样性分析、近交衰退和性状退化等方面的分析。运用所筛选的引物建立分子遗传选育技术的方法, 能够为遗传育种、种质资源保护研究、种苗放流等提供理论依据(李文升等, 2011), 实现大刺鳅的种质资源科学评价及保护利用。
| 广西壮族自治区水产研究所, 中国科学院动物研究所, 2006. 广西淡水鱼类志. 2版. 南宁: 广西人民出版社, 498499 |
| 王方, 2012. 中国南部地区大刺鳅种群遗传和亲缘地理研究. 广州: 华南师范大学硕士学位论文 http://d.wanfangdata.com.cn/Thesis/Y2208299 |
| 王慧, 张保卫, 史文博, 等, 2012. 商城肥鲵二碱基重复和四碱基重复微卫星DNA的结构特征及对筛选效率的影响. 生物多样性, 20(1): 51–58 |
| 中国水产科学研究院珠江水产研究所, 1991. 广东淡水鱼类志. 广州: 广东科技出版社, 519-520 |
| 史方, 徐念, 熊美华, 等, 2009. 利用微卫星标记评估乌江彭水水电站对泉水鱼的遗传多样性影响. 水生态学杂志, 2(2): 117–121 |
| 刘金亮, 2011. 东北雅罗鱼微卫星分子标记的筛选及雅罗鱼5个野生群体遗传多样性分析. 上海: 上海海洋大学硕士学位论文 http://cn.bing.com/academic/profile?id=dfed0de972e21f4b9050b81506f5ea0a&encoded=0&v=paper_preview&mkt=zh-cn |
| 刘瑞成, 张富铁, 但胜国, 等, 2013. 宽口光唇鱼微卫星位点的筛选与特征分析. 四川动物, 32(2): 161–166 |
| 孙效文, 张晓锋, 赵莹莹, 等, 2008. 水产生物微卫星标记技术研究进展及其应用. 中国水产科学, 15(4): 689–703 |
| 李文升, 刘翠, 鲁翠云, 等, 2011. 草鱼三、四核苷酸重复微卫星标记的分离与特征分析. 中国水产科学, 18(4): 742–750 |
| 杨华强, 李强, 舒琥, 等, 2016. 华南及邻近地区大刺鳅遗传多样性的ISSR分析. 水生生物学报, 40(1): 63–70 DOI:10.7541/2016.9 |
| 宋国华, 刘田福, 2005. 微卫星标记及其在实验动物中的应用. 中国比较医学杂志, 15(4): 244–248 |
| 陈忠, 2005. 大刺鳅的生物学及其养殖技术概要. 中国水产,(10): 22–25 DOI:10.3969/j.issn.1002-6681.2005.10.017 |
| 林婷婷, 2016. 大刺鳅微卫星标记开发及野生群体遗传多样性分析. 见: 2016年中国水产学会学术年会论文摘要集, 101 |
| 郑慈英, 1989. 珠江鱼类志. 北京: 科学出版社, 518-520 |
| 赵哲霞, 蒋珊, 王滨花, 等, 2014. 黄颡鱼属SSR分子鉴定及其遗传多样性. 南昌大学学报(理科版), 38(5): 498–501 |
| 高焕, 孔杰, 2005. 串联重复序列的物种差异及其生物功能. 动物学研究, 26(5): 555–564 |
| 海萨, 李家乐, 郭焱, 等, 2008. 河鲈微卫星引物筛选. 水生态学杂志, 1(6): 90–94 |
| 龚小玲, 李思发, 蔡完其, 等, 2009. 澳洲鳗鲡微卫星分子标记的筛选与检测. 中国水产科学, 16(1): 133–138 |
| 韩承慧, 马海涛, 姜海滨, 等, 2016. 许氏平鲉(Sebastes schlegeli)微卫星标记开发及野生、养殖群体遗传多样性分析. 海洋与湖沼, 47(1): 213–220 |
| 舒琥, 江小璐, 杨华强, 等, 2017. 华南地区7个大刺鳅野生群体的形态差异分析. 广州大学学报(自然科学版), 16(3): 8–14 |
| 鲁翠云, 毛瑞鑫, 李鸥, 等, 2009. 鲤鱼三、四核苷酸重复微卫星座位的筛选及特征分析. 农业生物技术学报, 17(6): 979–987 |
| 《福建鱼类志》编写组, 1985. 福建鱼类志下卷. 福州: 福建科学技术出版社, 447-448 |
| 蔡雪芹, 2006. 名优品种——大刺鳅. 海洋与渔业,(10): 17 |
| 谭照君, 张天奇, 鲁翠云, 等, 2011. 鲢三、四核苷酸重复微卫星标记的筛选及其特征分析. 上海海洋大学学报, 20(3): 328–335 |
| Botstein D, White R L, Skolnick M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Society of Human Genetics, 32(3): 314–331 |
| Brenner S, Elgar G, Sanford R, et al, 1993. Characterization of the pufferfish (Fugu) genome as a compact model vertebrate genome. Nature, 366(6452): 265–268 DOI:10.1038/366265a0 |
| Brooker A L, Cook D, Bentzen P, et al, 1994. Organization of microsatellites differs between mammals and cold-water teleost fishes. Canadian Journal of Fisheries and Aquatic Sciences, 51(9): 1959–1966 DOI:10.1139/f94-198 |
| Carvalho G R, Hauser L, 1994. Molecular genetics and the stock concept in fisheries. Reviews in Fish Biology and Fisheries, 4(3): 326–350 DOI:10.1007/BF00042908 |
| Crooijmans R P M A, Van der Poel J J, Groenen M A M, et al, 1997. Microsatellite markers in common carp (Cyprinus carpio L.). Animal Genetics, 28(2): 129–134 DOI:10.1111/age.1997.28.issue-2 |
| Diwan N, Cregan P B, 1997. Automated sizing of fluorescent-labeled simple sequence repeat (SSR) markers to assay genetic variation in soybean. Theoretical and Applied Genetics, 95(5-6): 723–733 DOI:10.1007/s001220050618 |
| Ellegren H, 2002. Microsatellite evolution:a battle between replication slippage and point mutation. Trends in Genetics, 18(2): 70 |
| Feresu-Shonhiwa F, Howard J H, 1998. Electrophoretic identification and phylogenetic relationships of indigenous tilapiine species of Zimbabwe. Journal of Fish Biology, 53(6): 1178–1206 DOI:10.1111/jfb.1998.53.issue-6 |
| Hearne C M, Ghosh S, Todd J A, 1992. Microsatellites for linkage analysis of genetic traits. Trends in Genetics, 8(8): 288–294 DOI:10.1016/0168-9525(92)90256-4 |
| Hite J M, Eckert K A, Cheng K C, 1996. Factors affecting fidelity of DNA synthesis during PCR amplification of d(C-A)n·d(C-T)n microsatellite repeats. Nucleic Acids Research, 24(12): 2429–2434 DOI:10.1093/nar/24.12.2429 |
| Maguire T L, Edwards K J, Saenger P, et al, 2000. Characterisation and analysis of microsatellite loci in a mangrove species, Avicennia marina (Forsk. ) Vierh. (Avicenniaceae). Theoretical and Applied Genetics, 101(1-2): 279–285 DOI:10.1007/s001220051480 |
| Nelson J S, 2006. Fishes of the World. 4th ed. Hoboken: Wiley and Sons, 316-317 |
| Schl tterer C, Tautz D, 1992. Slippage synthesis of simple sequence DNA. Nucleic Acids Research, 20(2): 211–215 DOI:10.1093/nar/20.2.211 |
| Serapion J, Kucuktas H, Feng J N, et al, 2004. Bioinformatic mining of type Ⅰ microsatellites from expressed sequence tags of channel catfish (Ictalurus punctatus). Marine Biotechnology, 6(4): 364–377 DOI:10.1007/s10126-003-0039-z |
| Sequeira F, Rocha S, Ferrand N, et al, 2005. Isolation and characterization of seven microsatellite loci in Chioglossa lusitanica (Urodela:Salamandridae). Molecular Ecology Notes, 5(2): 212–214 DOI:10.1111/men.2005.5.issue-2 |
| Tóth G, Gáspári Z, Jurka J, 2000. Microsatellites in different eukaryotic genomes:survey and analysis. Genome Research, 10(7): 967–981 DOI:10.1101/gr.10.7.967 |
| Weber J L, 1990. Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms. Genomics, 7(4): 524–530 DOI:10.1016/0888-7543(90)90195-Z |
| Yeh F C, Yang R C, Boyle T, 1999. Microsoft window-based freeware for population genetic analysis. version 1.31. Alberta: University of Alberta, |
 2018, Vol. 49
2018, Vol. 49


