
中国海洋湖沼学会主办。
文章信息
- 袁文成, 叶金明, 黄鹤忠, 肖攀, 路瑶, 李泽, 朱传坤. 2018.
- YUAN Wen-Cheng, YE Jin-Ming, HUANG He-Zhong, XIAO Pan, LU Yao, LI Ze, ZHU Chuan-Kun. 2018.
- 翘嘴鳜(Siniperca chuatsi)EST-SSR标记与生长性状相关性及4个选育群体遗传结构研究
- STUDY ON CORRELATION OF EST-SSR MARKERS WITH GROWTH TRAITS AND GENETIC STRUCTURE OF 4 BREEDING POPULATIONS IN MANDARIN FISH SINIPERCA CHUATSI
- 海洋与湖沼, 49(1): 224-231
- Oceanologia et Limnologia Sinica, 49(1): 224-231.
- http://dx.doi.org/10.11693/hyhz20171000257
-
文章历史
- 收稿日期:2017-10-10
- 收修改稿日期:2017-11-09
2. 扬州市水产生产技术指导站 扬州 225007;
3. 江苏省区域现代农业与环境保护协同创新中心 淮安 223300
2. Aquatic Products Production Technical Guidance Station of Yangzhou City, Yangzhou 225007, China;
3. Jiangsu Collaborative Innovation Center of Regional Modern Agriculture & Environmental Protection, Huai'an 223300, China
翘嘴鳜(Siniperca chuatsi)隶属鲈形目、鳜亚科、鳜属, 是我国鳜属的7个种中个体最大、生长速度最快的种类, 历来为我国重要的名优鱼类养殖对象。但近年来翘嘴鳜野生原始种质资源锐减、种质退化、生长和抗逆性能下降等问题变得越来越严重, 迫切需要开展鱼类选育和遗传改良的研究(Liu et al, 2004)。微卫星(即简单序列重复simple sequence repeat, SSR)具有重复性好、多态性高、共显性强以及在基因组中数量丰富和分布广泛的特点。通过转录组测序和验证获得来源于基因编码区的表达序列微卫星分子标记(EST-SSR)可以在相关功能基因上定位, 从而经济高效地开发出QTL定位的EST-SSR标记并应用于分子标记辅助育种(Zhang et al, 2012), EST-SSR标记相比基因组SSR(G-SSR)标记辅助育种的效率更高(Gupta et al, 2004; Skaala et al, 2004), 已成为开展物种性状选育(Nie et al, 2013; Xanthopoulou et al, 2017)和群体遗传结构分析(Ding et al, 2015)研究的理想工具。近年来, 微卫星分子标记技术在翘嘴鳜的遗传和育种研究方面也得到了较好的应用, 匡刚桥等(2007)采用FIASCO (Fast Isolation by AFLP Sequences Containing repeats)法构建翘嘴鳜G-SSR富集文库, 分离了微卫星DNA序列并对其特征进行分析; Yang等(2014)利用G-SSR开展了翘嘴鳜亲子关系的研究; 宋易等(2016)对5代翘嘴鳜选育群体开展了2个单核苷酸多态性标记和4个微卫星标记优势基因型的数目以及遗传相似性和遗传距离的研究, 以验证这些微卫星标记用于辅助育种的富集效果和可靠性。为了进一步开发与翘嘴鳜生长性状相关的EST-SSR分子标记并评估不同选育群体的遗传变异状况, 本文基于作者前期翘嘴鳜转录组测序并开发的EST-SSR多态性分子标记(袁文成等, 2015)的基础上, 通过单标记分析技术筛选了与翘嘴鳜体长、体高、体厚、体重4个生长性状相关的EST-SSR分子标记, 并利用此分子标记研究不同选育群体的遗传结构。以期为翘嘴鳜生长性状分子标记辅助育种和种质资源保护提供基础数据。
1 材料与方法 1.1 材料用于翘嘴鳜标记-性状相关性研究的材料来自于相同批次长江口群体和太湖群体苗种且在同一池塘养殖7月龄的翘嘴鳜健康个体, 随机选取60尾, 进行体长、体重等表型性状指标测量以及分子生物学检测。翘嘴鳜选育群体遗传结构分析的材料分别取自于扬州市董氏特种水产有限公司养殖基地的F2代鄱阳湖选育群体(简称BYHP)和F2代洞庭湖选育群体(简称HZHP)以及苏州市申航生态科技发展股份有限公司养殖基地的F2代太湖选育群体(简称THP)和F2代长江口选育群体(简称CJKP)。样本数量为每个群体中各随机选取健康个体30尾。
1.2 EST-SSR引物采用本实验室在翘嘴鳜转录组测序数据基础上自主设计并经多态性验证的13对EST-SSR引物(表 1), 引物由上海生工公司合成。
| 标记号 | SSR类型 | 引物序列(5'—3') | 产物大小(bp) | 退火温度Ta(℃) |
| C5150 | (AC)9 | F: CTGCACATGGCCAAGACTG | 217 | 56 |
| R:TACCTGTGTGTCCCTTGGC | ||||
| C12350 | (GGTT)5 | F:GTCGGGTCAGGCATTTAGC | 242 | 57 |
| R: ACACCACCTGCCAGACTTC | ||||
| C15209 | (GT)7 | F:TTGACCCACCAGAGCAAGG | 341 | 57 |
| R:TCAGACAACCCTGTCGAGC | ||||
| C10127 | (GT)9 | F: GCTGGTGCAGGTGGATTTC | 377 | 57 |
| R:GTGGGTGAGAAGCTTTGCC | ||||
| C5528 | (GT)7 | F:TGTCACTGCGACCATCCTC | 383 | 60 |
| R: CGATAACCGGGTAGCCCTC | ||||
| C43527 | (GCT)5 | F:GGGCTGTGATGGTTTGACG | 162 | 57 |
| R:AGTATCAACCACCGAGGCG | ||||
| C14847 | (GT)8 | F:TGATGGAGTGCACAGTCAAG | 354 | 56 |
| R:CTTGTCATTGGCGCTGAGG | ||||
| C18691 | (GCT)6 | F:GACGAGTCAGTGCGACAAC | 177 | 57 |
| R:CTTTGCGTCTTCCTGGCTG | ||||
| C9810 | (GT)9 | F:CTCGGGAGGCTTCTGGAC | 384 | 56 |
| R:TCCGTTTGTGCCAACCATC | ||||
| C19016 | (GT)8 | F:TCTTGTCTGCCGGTGAGC | 427 | 56 |
| R:GTGCTTGCGAGACATACTCC | ||||
| C32533 | (AC)8 | F:GCGAAGGTGGATCATTGCC | 184 | 56 |
| R:TCTCGACCCTGAGTCTTGC | ||||
| C6340 | (AGG)5 | F:CCATGATCAACGCTGTGCC | 383 | 61 |
| R:TCCTGGCTTCTTCCTCCAG | ||||
| C29449 | (ACC)6 | F:TGAGAACGGCTGCTACCTC | 388 | 57 |
| R:GGTTGTTGATGGAGAACGGG |
测量和计算每尾鱼的体重、体长、体厚及体高4个表型性状后剪取其鳍条组织, 保存于95%酒精中备用。基因组DNA提取采用酚-氯仿抽提法, DNA含量、纯度及完整性测定采用核酸蛋白测定仪(Thermo Fisher)和琼脂糖凝胶电泳法, 用无菌水稀释至100ng/μL后, –20℃保存备用。
1.3.2 微卫星扩增反应及电泳按照Zhang等(2012)的方法, 将13对微卫星引物(表 1)在PCR仪(TaKaRa)上扩增, 采用PCR反应体系20μL (其中:无菌水7μL, 2×ES Taq MasterMix 10μL, 模板DNA 50ng, 10μmol/L上下游引物各1μL), 扩增程序: 95℃ 2min预变性, 30个循环[变性(95℃)、退火(50—60℃)和延伸(72℃)各30s], 最后延伸(72℃)5min。参照高东等(2009)的方法, PCR产物在6%的非变性聚丙烯酰胺凝胶电泳90min(电压150V)后进行硝酸银染色, 电泳图用凝胶成像系统拍摄, 利用Gel-Pro Analyzer 4.5软件进行读带和分型。
1.3.3 种群遗传结构的检测按1.3.2方法, 利用13对微卫星分子标记引物(表 1)对HZHP、CJKP、BYHP和THP4个选育群体进行基因组扫描, 获得稳定扩增、条带清晰的多态性电泳图, 用于种群遗传结构分析。
1.4 数据分析与统计 1.4.1 基因标记、基因型与生长性状的相关分析在SPSS 16.0软件中首先利用Kolmogorov-Smirnov法对翘嘴鳜体长、体重、体厚和体重4个生长性状的频率分布进行正态分布检验; 其次利用广义线性模型(GLM)的最小二乘方差分析法分别分析13个SSR标记与翘嘴鳜4种生长性状间的相关性, 获得显著相关的SSR标记; 最后采用ANOVA方差分析法进行显著相关SSR标记的不同基因型与4种生长性状间的多重比较, 从而获得与生长性状显著相关的基因型。
1.4.2 遗传结构分析获取扩增电泳条带图以后, 利用软件Gel-Pro Analyzer 4.5获取每尾翘嘴鳜的基因型数据。采用PopGen32软件对4个选育群体的每个微卫星标记统计其等位基因数(Na)、有效等位基因数(Ne)、等位基因频率(P)、观测杂合度(Ho)及期望杂合度(He), 并计算多态性信息含量(PIC) (Botstein et al, 1980)。
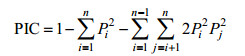
式中, Pi、Pj分别为第i个和第j个群体中的等位基因在群体中的频率, n为某一基因座上的等位基因数。
根据Lynch(1999)的Nei指数法计算4个翘嘴鳜选育群体间的遗传相似度(S)和遗传距离(D)。S =2Nxy/ (Nx + Ny)。式中, Nxy为两个体共享的图带数, Nx和Ny分别为x和y个体拥有的电泳条带数。
2 结果与分析 2.1 表型性状的分布检测翘嘴鳜体重、体长、体厚和体高4个生长性状的测量数据均表现出连续变化的特点, 表明这些性状均是数量性状(吴滟等, 2011); 由Kolmogorov-Smirnov检测表明, 4种生长性状符合正态分布(P > 0.05) (表 2)。
| 性状 | 均值 | 标准差 | 最大值 | 最小值 | P值 |
| 体重(kg) | 0.5562 | 0.1024 | 0.9425 | 0.3667 | 0.32 |
| 体长(cm) | 26.8654 | 2.7545 | 33.8747 | 16.3542 | 0.24 |
| 体厚(cm) | 4.6638 | 0.6543 | 6.0870 | 3.3075 | 0.20 |
| 体高(cm) | 9.6878 | 0.9372 | 13.536 | 6.0546 | 0.25 |
统计分析结果表明, 13个多态性微卫星标记(表 1)中的5个标记与翘嘴鳜生长性状表现出显著相关性(表 3)。其中, 标记C10127分别与体厚和体长极显著相关(P < 0.01)和显著相关(P < 0.05);标记C19016与体重显著相关(P < 0.05);标记C14847与体厚显著相关(P < 0.05);标记C12350分别与体重、体厚和体长、体高显著相关(P < 0.05)和极显著相关(P < 0.01);标记C18691分别与体高和体重、体厚显著相关(P < 0.05)和极显著相关(P < 0.01)。
| 标记号 | 基因型 | 个体数 | 体长(cm) | 体高(cm) | 体厚(cm) | 体重(kg) |
| C10127 | AA | 23 | 29.059±0.964a | 9.7594±0.412a | 4.602±0.284a | 0.559±0.035a |
| AB | 15 | 25.657±0.857b | 9.7631±0.536a | 4.188±0.245ab | 0.525±0.045a | |
| AC | 7 | 25.425±0.756b | 9.4569±0.654a | 3.968±0.324b | 0.487±0.037a | |
| BB | 12 | 27.625±0.868ab | 9.654±0.547a | 4.354±0.350ab | 0.504±0.056a | |
| CC | 3 | 27.784±1.472ab | 9.373±0.640a | 4.324±0.674ab | 0.496±0.054a | |
| C19016 | AA | 13 | 29.025±1.230a | 9.786±0.495a | 4.631±0.261a | 0.562±0.074a |
| AB | 15 | 25.056±0.094b | 8.435±0.510a | 4.036±0.234a | 0.403±0.032b | |
| AC | 13 | 26.654±0.095ab | 9.462±0.624a | 4.278±0.324a | 0.515±0.045ab | |
| BB | 19 | 26.953±0.084ab | 9.556±0.427a | 4.476±0.265a | 0.527±0.038ab | |
| C14847 | AB | 31 | 26.685±0.635a | 9.402±0.401a | 4.057±0.102a | 0.475±0.042a |
| BB | 29 | 27.054±0.658a | 9.654±0.451a | 4.765±0.125b | 0.524±0.054a | |
| C12350 | AA | 6 | 28.845±1.245ab | 9.845±0.627a | 4.242±0.351ab | 0.520±0.086a |
| AB | 19 | 27.654±0.856ab | 9.395±0.425a | 4.140±0.285a | 0.498±0.056a | |
| AC | 17 | 25.234±0.651b | 9.412±0.335a | 4.168±0.202a | 0.508±0.052a | |
| BB | 6 | 28.314±1.628ab | 9.435±0.542a | 4.224±0.367a | 0.512±0.075a | |
| BC | 5 | 31.354±1.864a | 12.686±0.758b | 5.685±0.402b | 0.882±0.131b | |
| CC | 7 | 25.764±1.145b | 9.584±0.581a | 4.567±0.357ab | 0.556±0.054a | |
| C18691 | AA | 32 | 26.425±0.754a | 9.056±0.301a | 4.102±0.124a | 0.465±0.037a |
| AB | 13 | 26.541±0.756a | 9.320±0.362ab | 4.198±0.146a | 0.464±0.038a | |
| BB | 15 | 28.685±1.132a | 10.858±0.351b | 5.251±0.184b | 0.726±0.048b | |
| 注:同一栏中相同列的不同上标字母代表数值间存在显著差异(P < 0.05) | ||||||
将翘嘴鳜5个微卫星分子标记的不同基因型分别与生长性状进行多重比较的结果显示:在C10127标记上, 个体AA型的体厚显著高于AC型(P < 0.05), 个体AA型的体长显著高于AC、AB型(P < 0.05)。因此, AA型是体厚、体长性状的优势基因型, 而AC型和AB型分别是体厚、体长和体长性状的劣势基因型。在C19016标记上, 个体AB型的体长显著低于AA型个体(P < 0.05), AA型个体的体重显著高于AB型(P < 0.05), 说明AB基因型是体长性状的劣势基因型, AA型为体重的优势基因型。在C14847标记上, 个体BB型的体厚显著大于AB型(P < 0.05), 表明BB型和AB型分别是体厚性状的优势基因型和劣势基因型; 而基因型间的体长、体高和体厚性状无显著差异(P > 0.05)。在C12350标记上, BC型的体长显著大于AC型和CC型个体(P < 0.05), 体高和体重都显著大于AA型、AB型、AC型、BB型和CC型个体(P < 0.05), 体厚显著大于AB型、AC型和BB型个体(P < 0.05), 说明BC型是体长、体高、体重和体厚4种性状的优势基因型。标记C18691与增强子结合蛋白β有关, 该蛋白是增强子结合蛋白家族一员, 在生长发育、能量代谢和细胞分化等多个方面发挥作用。该标记上BB型个体的体厚、体重和体高分别显著大于AA型、AB型和AA型个体(P < 0.05), 推测BB型为体厚、体重和体高性状的优势基因型, AA型为劣势基因型。
2.3 翘嘴鳜4个选育群体遗传多样性13个多态性微卫星标记引物(表 1)对4个选育群体的120尾个体进行了PCR和聚丙烯酰胺凝胶电泳, 均获得了具有多态性的扩增条带, 图 1和图 2显示了部分标记在翘嘴鳜选育群体中的扩增结果。每个标记平均得到等位基因2.2115个, 其中, 平均有效等位基因数为1.9405个。4个选育群体的等位基因数、有效等位基因数、观测杂合度、期望杂合度及多态信息含量见表 4, 由表 4可知, 4个选育群体遗传多样性高低依次为THP > CJKP > HZHP > BYHP。
 |
| 图 1 标记C15209在翘嘴鳜选育群体中的扩增结果 Fig. 1 PCR amplification results of C15209 loci in S. chuatsi |
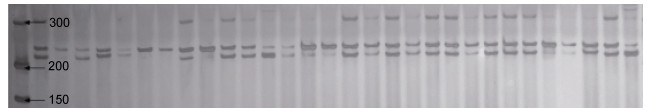 |
| 图 2 标记C12350在翘嘴鳜选育群体中的扩增结果 Fig. 2 PCR amplification results of C12350 loci in S. chuatsi |
| 标记 | THP | BYHP | HZHP | CJKP | ||||||||||||||||
| Na | Ne | Ho | He | PIC | Na | Ne | Ho | He | PIC | Na | Ne | Ho | He | PIC | Na | Ne | Ho | He | PIC | |
| C5150 | 2 | 1.9652 | 0.4745 | 0.7854 | 0.4305 | 2 | 1.8751 | 0.4226 | 0.4786 | 0.3562 | 2 | 1.5624 | 0.5258 | 0.5607 | 0.3856 | 2 | 1.6361 | 0.5219 | 0.5324 | 0.3752 |
| C12350 | 3 | 2.8548 | 0.4643 | 0.7625 | 0.4628 | 3 | 2.1356 | 0.4853 | 0.5867 | 0.3857 | 3 | 2.1867 | 0.4516 | 0.5062 | 0.3954 | 2 | 2.3514 | 0.5236 | 0.5305 | 0.3745 |
| C15209 | 2 | 1.7654 | 0.4534 | 0.6634 | 0.3864 | 3 | 2.0517 | 0.4682 | 0.5044 | 0.3852 | 3 | 1.8954 | 0.5025 | 0.5238 | 0.3251 | 3 | 2.2257 | 0.6238 | 0.6532 | 0.4545 |
| C10127 | 2 | 1.7586 | 0.6581 | 0.5243 | 0.3952 | 2 | 1.8425 | 0.4350 | 0.4861 | 0.3235 | 2 | 1.5571 | 0.5124 | 0.5950 | 0.3567 | 3 | 2.6524 | 0.5623 | 0.578 | 0.4251 |
| C43527 | 2 | 1.9657 | 0.5162 | 0.7857 | 0.4021 | 2 | 1.4255 | 0.4056 | 0.2994 | 0.2586 | 2 | 1.4846 | 0.5840 | 0.5727 | 0.3305 | 2 | 1.5852 | 0.7541 | 0.7641 | 0.4527 |
| C14847 | 2 | 1.9623 | 0.5152 | 0.8241 | 0.4125 | 2 | 1.6567 | 0.4652 | 0.4861 | 0.3341 | 3 | 2.0642 | 0.4427 | 0.4804 | 0.3026 | 2 | 1.5574 | 0.4587 | 0.4622 | 0.3652 |
| C18691 | 2 | 1.9687 | 0.6753 | 0.7845 | 0.3856 | 2 | 1.5842 | 0.5856 | 0.6025 | 0.3956 | 2 | 1.8162 | 0.6543 | 0.7218 | 0.4309 | 2 | 1.4658 | 0.6204 | 0.6324 | 0.3854 |
| C6340 | 2 | 1.9568 | 0.5264 | 0.6320 | 0.3805 | 2 | 1.7415 | 0.5341 | 0.5524 | 0.3625 | 2 | 1.5321 | 0.5642 | 0.5684 | 0.3214 | 2 | 1.4575 | 0.536 | 0.6524 | 0.3985 |
| C32533 | 2 | 1.6251 | 0.7065 | 0.6358 | 0.4128 | 2 | 1.7428 | 0.6350 | 0.6532 | 0.3665 | 2 | 1.5354 | 0.5451 | 0.5624 | 0.3026 | 2 | 1.5242 | 0.6028 | 0.6707 | 0.4032 |
| C9810 | 3 | 2.5564 | 0.7084 | 0.6523 | 0.4852 | 2 | 1.7564 | 0.4265 | 0.4962 | 0.3324 | 2 | 1.5271 | 0.5456 | 0.5745 | 0.3548 | 2 | 1.4528 | 0.5234 | 0.5260 | 0.3454 |
| C19016 | 2 | 1.8767 | 0.7181 | 0.5264 | 0.3851 | 2 | 1.7567 | 0.4658 | 0.4958 | 0.3628 | 3 | 2.5657 | 0.624 | 0.6341 | 0.4686 | 3 | 2.4262 | 0.6527 | 0.5828 | 0.3541 |
| C5528 | 2 | 1.8645 | 0.6272 | 0.7568 | 0.3568 | 2 | 1.6548 | 0.4856 | 0.5024 | 0.3762 | 2 | 1.5384 | 0.5367 | 0.5623 | 0.3654 | 2 | 1.6751 | 0.5042 | 0.5374 | 0.3325 |
| C29449 | 2 | 1.8643 | 0.6874 | 0.5365 | 0.3652 | 2 | 1.6527 | 0.6574 | 0.6853 | 0.4056 | 2 | 1.7818 | 0.5688 | 0.5742 | 0.3708 | 2 | 1.9846 | 0.5234 | 0.5538 | 0.3452 |
| 平均 | 2.1538 | 1.9988 | 0.5947 | 0.6823 | 0.4067 | 2.1538 | 1.7597 | 0.4978 | 0.5253 | 0.3573 | 2.3077 | 1.7729 | 0.5429 | 0.5720 | 0.3623 | 2.2302 | 1.8457 | 0.5698 | 0.5905 | 0.3855 |
| 注: Na:等位基因数; Ne:有效等位基因数; Ho:观测杂合度; He:期望杂合度; PIC:多态信息含量 | ||||||||||||||||||||
由表 5可知, THP和CJKP两个选育群体间的遗传距离最小(0.0696)、遗传相似度最大(0.8133), 表明这两个群体亲缘关系最近; 而BYHP和HZHP两个群体的遗传距离最大(0.1247), 遗传相似度最小(0.6326), 表明它们之间的亲缘关系较远。
| Pop ID | HZHP | CJKP | BYHP | THP |
| HZHP | * | 0.7230 | 0.6326 | 0.7226 |
| CJKP | 0.0766 | * | 0.6972 | 0.8133 |
| BYHP | 0.1247 | 0.1037 | * | 0.7342 |
| THP | 0.0772 | 0.0696 | 0.0725 | * |
通过分子标记的基因型与个体数量性状表型间关联的差异显著性检验, 可开展标记-性状连锁分析(叶华等, 2014)。若根据其关联分析, 找到一个或多个分子标记与某一性状存在显著的遗传相关性, 则可认为存在一个数量性状分子标记, 从而实现育种技术由传统的表型选择向分子标记选择育种的革新(王高富等, 2006)。因此, 分子标记与性状间的关联分析是近年来遗传育种的研究热点。但是, 目前有关翘嘴鳜的研究主要集中在人工繁育和病害防治(Ding et al, 2010; 刘希良等, 2013; Tanaka et al, 2013)、不同地理群体间遗传结构及遗传多样性研究(杨受保等, 2003; 方展强等, 2005)和微卫星分子标记开发(Yang et al, 2012; Huang et al, 2013)等方面。本文首次采用基于转录组测序基础上开发的13个多态性EST-SSR分子标记开展了与翘嘴鳜体重、体高、体长和体厚4个生长性状关联分析的研究, 找到了5个与上述翘嘴鳜4个生长性状显著相关的微卫星分子标记, 其中, 标记C10127和C12350均与体长性状显著和极显著相关, 标记C14847和C10127均与体厚性状显著和极显著相关, 标记C19016和C12350均与体重性状显著相关, 表明翘嘴鳜的生长性状与多个EST-SSR标记相关, 提示存在多因一效或表型与基因连锁的现象(李仕贵等, 2002; 郑先虎等, 2011)。实验结果还显示翘嘴鳜存在一因多效的现象, 如:标记C12350与体重、体长、体高和体厚4个生长性状均具有显著相关性; 标记C18691与体厚、体重和体高存在极显著和显著相关。这与草鱼的研究结果相类似(王解香等, 2012)。导致这些一因多效的原因, 一方面是由于这些生长性状之间本身存在着相关性; 另一方面提示这些一因多效的标记可能是与控制生长性状的主效基因紧密连锁或处于同一染色体的不同座位的标记。由于这些与翘嘴鳜多个生长性状相关的EST-SSR标记已获得了所在EST序列的转录组测序信息以及相关基因序列或相对应的蛋白质信息, 因此, 可为翘嘴鳜QTL定位和分子标记辅助育种提供重要的遗传学基础。
3.2 基于EST-SSR分子标记的翘嘴鳜不同选育群体的遗传结构群体遗传多样性水平的高低与基因丰富度和遗传潜力的大小正相关, 观测杂合度、期望杂合度、等位基因数和多态信息含量都是其常用指标(侯宁等, 2007)。本研究基于13对多态性EST-SSR引物对4个翘嘴鳜选育群体进行基因组扫描结果显示(表 4), 平均有效等位基因数(Ne)位于1.7597—1.9988之间, 其值小于2.15, 表明4个群体均存在等位基因缺失现象(万玉美等, 2011)。作为衡量群体遗传结构和遗传变异程度的最适指标之一的杂合度, 可以反映一个群体内所包含的所有等位基因的分布情况和丰富程度, 也可反映群体内个体的均匀度和变异程度, 揭示群体、物种或家系的生存和适应能力以及进化程度, 杂合度的高低与其生命力和适应能力成正相关(Botstein et al, 1980)。本研究4个翘嘴鳜选育群体平均的观测杂合度(Ho)和期望杂合度(He)分别位于0.4978—0.5947和0.5253—0.6823之间, 均低于对翘嘴鳜野生群体的研究结果(0.6754和0.7487)(朱韬等, 2013)。导致上述结果的原因, 可能是由于本试验所用SSR引物均与经济性状相关, 在同一群体内人工选育至F2代以后, 逐渐降低了这些经济性状的遗传变异程度, 使选育群体经济性状遗传的富集度增加、多样性降低。一般而言, 多态信息含量(PIC) < 0.25为低度多态性, 0.25 < PIC < 0.5为中度多态性, 而PIC> 0.5为高度多态性(Nei, 1987; Liu et al, 2008)。本研究结果显示, 4个翘嘴鳜选育群体的平均PIC位于0.3573— 0.4067之间, 为中度多态水平, 表明仍具有一定的选育潜力。遗传距离和相似性指数是用于检验群体间亲缘关系的重要指标。一级和二级亲缘关系的遗传相似性指数分别为0.5和0.25 (Nei et al, 1979)。本研究4个翘嘴鳜选育群体的遗传相似性指数均处于0.6326—0.8133之间, 表明同一物种在不同地理种群的选育群体之间仍然处于一级亲缘关系水平上。因此, 今后可在不同地理种群间构建选育群体或家系, 并且通过增加不同地理种群的数量和野生亲本数量等手段, 来提高选育群体的遗传多样性, 以获得遗传结构丰富和性状优良的选育种质。
4 结论(1) 对60尾翘嘴鳜的体长、体高、体厚及体重4种生长性状进行标记-性状相关性研究结果表明:有13个中的5个翘嘴鳜EST-SSR标记分别与生长性状显著相关, 包括:分别与体厚和体长极显著相关(P < 0.01)和显著相关(P < 0.05)的标记C10127, AA型为体长、体厚的优势基因型, 而AC型为劣势基因型; 与体重显著相关(P < 0.05)的标记C19016, AA型为体重的优势基因型, AB型为体长的劣势基因型; 与体厚显著相关(P < 0.05)的标记C14847, BB型为体厚的优势基因型; 分别与体长、体高和体重、体厚极显著相关(P < 0.01)和显著相关(P < 0.05)的标记C12350, BC型为体长、体厚、体高和体重性状的优势基因型; 分别与体厚、体重和体高极显著相关(P < 0.01)和显著相关(P < 0.05)的C18691标记, BB型为体厚、体重和体高性状的优势基因型。
(2) 对4个翘嘴鳜选育群体各30尾个体进行遗传结构分析的结果显示:遗传多样性高低依次为太湖选育群体 > 长江口选育群体 > 洪泽湖选育群体 > 鄱阳湖选育群体, 其平均的等位基因数、有效等位基因数、观测杂合度、期望杂合度和多态信息含量分别为2.2115、1.9405、0.55513、0.5925和0.3775, 属于中度多态水平。4个翘嘴鳜选育群体的遗传相似性指数均处于0.6326—0.8133之间, 表明同一物种在不同地理种群的选育群体之间仍然处于一级亲缘关系。其中, 鄱阳湖选育群体与洪泽湖选育群体的遗传距离最大(0.1247), 遗传相似度最小(0.6326), 表明它们之间的亲缘关系较远。
| 万玉美, 王蕾, 谭照君, 等, 2011. 红鳍东方鲀两个群体的遗传结构及与经济性状的相关性分析. 淡水渔业, 41(5): 9–16 |
| 王高富, 吴登俊, 2006. 凉山半细毛羊微卫星标记与羊毛性状的相关分析. 遗传, 28(12): 1505–1512 DOI:10.3321/j.issn:0253-9772.2006.12.005 |
| 王解香, 白俊杰, 于凌云, 2012. 草鱼EST-SSRs标记的筛选及其与生长性状相关分析. 淡水渔业, 42(1): 3–8 |
| 方展强, 陈军, 郑文彪, 等, 2005. 鳜野生群体与养殖群体的RAPD分析. 大连水产学院学报, 20(1): 16–19 |
| 叶华, 刘洋, 刘贤德, 等, 2014. 大黄鱼微卫星标记与生长性状的相关分析. 西南大学学报(自然科学版), 36(3): 27–33 |
| 匡刚桥, 刘臻, 鲁双庆, 等, 2007. FIASCO法筛选鳜鱼微卫星标记. 中国水产科学, 14(4): 608–614 |
| 朱滔, 梁旭方, 彭敏燕, 等, 2013. 翘嘴鳜EST-SSR标记的开发及3个群体遗传多态性分析. 暨南大学学报(自然科学与医学版), 23(3): 347–352 |
| 刘希良, 宾石玉, 王开卓, 等, 2013. 翘嘴鳜的人工繁殖与胚胎发育观察. 广西师范大学学报(自然科学版), 31(2): 100–106 |
| 李仕贵, 马玉清, 何平, 等, 2002. 不同环境条件下水稻生育期和株高的QTL分析. 作物学报, 28(4): 546–550 |
| 杨受保, 祖国掌, 程久发, 2003. 鳜鱼遗传多样性的RAPD指纹分析. 水产养殖, 24(4): 33–35 |
| 吴滟, 付春鹏, 蒋速飞, 等, 2011. 中华绒螯蟹微卫星标记与生长性状相关性的初步分析. 水生生物学报, 35(2): 197–202 |
| 宋易, 梁旭方, 田昌绪, 等, 2016. 生长相关分子标记在翘嘴鳜五代中的富集. 水生生物学报, 40(5): 951–957 DOI:10.7541/2016.123 |
| 郑先虎, 匡友谊, 鲁翠云, 等, 2011. 镜鲤体长、体高、体厚性状QTL定位分析. 遗传, 33(12): 1366–1373 |
| 侯宁, 张研, 鲁翠云, 等, 2007. 微卫星DNA标记分析德国镜鲤的遗传潜力. 遗传, 29(12): 1509–1518 DOI:10.3321/j.issn:0253-9772.2007.12.016 |
| 袁文成, 黄鹤忠, 李文龙, 等, 2015. 翘嘴鳜(Siniperca chuatsi)转录组EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 46(2): 403–409 DOI:10.11693/hyhz20140900248 |
| 高东, 杜飞, 朱有勇, 2009. 低背景、高分辨率PAGE简易银染法. 遗传, 31(6): 668–672 |
| Botstein D, White R L, Skolnick M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 32(3): 314–331 |
| Ding W C, Chen J, Shi Y H, et al, 2010. Rapid and sensitive detection of infectious spleen and kidney necrosis virus by loop-mediated isothermal amplification combined with a lateral flow dipstick. Archives of Virology, 155(3): 385–389 DOI:10.1007/s00705-010-0593-4 |
| Ding Y J, Zhang J H, Lu Y F, et al, 2015. Development of EST-SSR markers and analysis of genetic diversity in natural populations of endemic and endangered plant Phoebe chekiangensis. Biochemical Systematics and Ecology, 63: 183–189 DOI:10.1016/j.bse.2015.10.008 |
| Gupta P K, Rustgi S, 2004. Molecular markers from the transcribed/expressed region of the genome in higher plants. Functional & Integrative Genomics, 4(3): 139–162 |
| Huang W, Liang X F, Qu C M, et al, 2013. Development and characterization of novel polymorphic microsatellite loci in Siniperca scherzeri Steindachner and Siniperca chuatsi (Basilewsky). Molecular Biology Reports, 40(2): 751–756 DOI:10.1007/s11033-012-1958-4 |
| Liu Q S, Feng D Y, Zhang H M, et al, 2008. Genetic diversity of Chinese Shar-pei Dog using microsatellite DNA markers. Zoological Research, 29(1): 31–36 DOI:10.3724/SP.J.1141.2008.00031 |
| Liu Z J, Cordes J F, 2004. DNA marker technologies and their applications in aquaculture genetics. Aquaculture, 238(1-4): 1–37 DOI:10.1016/j.aquaculture.2004.05.027 |
| Lynch M, 1999. The similarity index and DNA fingerprinting. Molecular Biological Evolution, 7(5): 478–484 |
| Nei M, Li W H, 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceedings of the National Academy of Sciences of the United States of America, 76(10): 5269–5273 DOI:10.1073/pnas.76.10.5269 |
| Nei M, 1987. Molecular Evolutionary Genetics. New York:Columbia University Press, 121 |
| Nie Q, Yue X, Chai X L, et al, 2013. Three vibrio-resistance related EST-SSR markers revealed by selective genotyping in the clam Meretrix meretrix. Fish & Shellfish Immunology, 35(2): 421–428 |
| Skaala , H yim B, Glover K, et al, 2004. Microsatellite analysis in domesticated and wild Atlantic salmon (Salmo salar L. ):allelic diversity and identification of individuals. Aquaculture, 240(1): 131–143 |
| Tanaka N, Izawa T, Kuwamura M, et al, 2013. The first case of infectious spleen and kidney necrosis virus (ISKNV) infection in aquarium-maintained mandarin fish, Siniperca chuatsi (Basilewsky), in Japan. Journal of Fish Diseases, 37(4): 401–405 |
| Xanthopoulou A, Ganopoulos I, Psomopoulos F, et al, 2017. De novo comparative transcriptome analysis of genes involved in fruit morphology of pumpkin cultivars with extreme size difference and development of EST-SSR markers. Gene, 622: 50–66 DOI:10.1016/j.gene.2017.04.035 |
| Yang M, Liang X F, Tian C X, et al, 2012. Isolation and characterization of fifteen novel microsatellite loci in golden mandarin fish (Siniperca scherzer i) Steindachne. Conservation Genetics Resources, 4(3): 599–601 DOI:10.1007/s12686-012-9601-1 |
| Yang M, Tian C X, Liang X F, et al, 2014. Parentage determination of mandarin fish (Siniperca chuatsi) based on microsatellite DNA markers. Biochemical Systematics and Ecology, 54: 285–291 DOI:10.1016/j.bse.2014.03.003 |
| Zhang H Y, Wei L B, Miao H M, et al, 2012. Development and validation of genic-SSR markers in sesame by RNA-seq. BMC Genomics, 13(1): 316 DOI:10.1186/1471-2164-13-316 |
 2018, Vol. 49
2018, Vol. 49


