
中国海洋湖沼学会主办。
文章信息
- 刘阳, 韩慧宗, 王腾腾, 孙娜, 张明亮, 王斐, 冯艳微, 杜荣斌, 姜海滨. 2018.
- LIU Yang, HANG Hui-Zong, WANG Teng-Teng, SUN Na, ZHANG Ming-Liang, WANG Fei, FENG Yan-Wei, DU Rong-Bin, JIANG Hai-Bin. 2018.
- 许氏平鲉(Sebastes schlegelii)速生选育群体的生长性能与遗传特征分析
- ANALYSIS OF GROWTH PERFORMANCES AND GENETIC CHARACTERISTICS OF FAST-GROWING BREEDING POPULATIONS OF SEBASTES SCHLEGELⅡ
- 海洋与湖沼, 49(2): 432-443
- Oceanologia et Limnologia Sinica, 49(2): 432-443.
- http://dx.doi.org/10.11693/hyhz20170900243
-
文章历史
- 收稿日期:2017-09-27
- 收修改稿日期:2018-01-07
2. 山东省海洋资源与环境研究院 山东省海洋生态修复重点实验室 烟台 264006;
3. 烟台大学海洋学院 烟台 264005
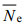 )为2.9849-7.9598,平均观测杂合度(
)为2.9849-7.9598,平均观测杂合度(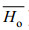 )和期望杂合度(
)和期望杂合度(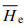 )分别为0.7230-0.8405和0.6315-0.8716,平均多态信息含量(
)分别为0.7230-0.8405和0.6315-0.8716,平均多态信息含量(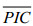 )为0.6472-0.8478。经两代家系选育的R-F3和RC-F3平均等位基因数显著降低,但观测杂合度(Ho)和期望杂合度(He)均值仍达到0.7230、0.7549和0.6527、0.6315,还有维持了较高的遗传多样性水平,遗传潜力较大。Hardy-Weinberg平衡检测结果和遗传偏离指数(d)表明各群体在大约10个位点上表现出一定程度的杂合子缺失现象。群体间平均遗传分化系数(Fst)为0.1279,各群体间存在中等遗传分化程度。R与R-F1分别与RC-F3遗传距离最远(0.9959、0.9848),预测利用两组群体中生长优良个体分别进行杂交育种有可能获得杂种优势。
)为0.6472-0.8478。经两代家系选育的R-F3和RC-F3平均等位基因数显著降低,但观测杂合度(Ho)和期望杂合度(He)均值仍达到0.7230、0.7549和0.6527、0.6315,还有维持了较高的遗传多样性水平,遗传潜力较大。Hardy-Weinberg平衡检测结果和遗传偏离指数(d)表明各群体在大约10个位点上表现出一定程度的杂合子缺失现象。群体间平均遗传分化系数(Fst)为0.1279,各群体间存在中等遗传分化程度。R与R-F1分别与RC-F3遗传距离最远(0.9959、0.9848),预测利用两组群体中生长优良个体分别进行杂交育种有可能获得杂种优势。2. Shandong Provincial Key Laboratory of Restoration for Marine Ecology, Shandong Marine Resource and Environment Research Institute, Yantai 264006, China;
3. School of ocean, Yantai University, Yantai 264005, China
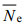 ) was from 2.9849 to 7.9598, average observed heterozygosity (
) was from 2.9849 to 7.9598, average observed heterozygosity (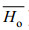 ) and expected heterozygosity (
) and expected heterozygosity (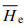 ) were 0.7230-0.8405 and 0.6315-0.8716, respectively, average polymorphism information contention (
) were 0.7230-0.8405 and 0.6315-0.8716, respectively, average polymorphism information contention (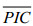 ) ranged 0.6472 to 0.8478. The average effective number of alleles was significantly reduced in R-F3 and RC-F3. But average observed heterozygosity and expected heterozygosity were still 0.7230, 0.7549 and 0.6527, 0.6315. Therefore, R-F3 and RC-F3remained in a high level of genetic diversity, and had a great genetic potential. By analyzing the P value and genetic deviation index (d), we revealed 10 primers existing heterozygote deficiency in all populations. The average coefficient of genetic differentiation (Fst) was 0.1279, suggesting that the differentiation was intermediate. The genetic distance between R-F1 and RC-F3and between R and RC-F3(0.9959, 0.9848) were the longest in cluster analysis. We predicted that there will be heterosis in the two groups mating pattern.
) ranged 0.6472 to 0.8478. The average effective number of alleles was significantly reduced in R-F3 and RC-F3. But average observed heterozygosity and expected heterozygosity were still 0.7230, 0.7549 and 0.6527, 0.6315. Therefore, R-F3 and RC-F3remained in a high level of genetic diversity, and had a great genetic potential. By analyzing the P value and genetic deviation index (d), we revealed 10 primers existing heterozygote deficiency in all populations. The average coefficient of genetic differentiation (Fst) was 0.1279, suggesting that the differentiation was intermediate. The genetic distance between R-F1 and RC-F3and between R and RC-F3(0.9959, 0.9848) were the longest in cluster analysis. We predicted that there will be heterosis in the two groups mating pattern.
许氏平鲉(Sebastes schlegelii)又称黑鲪, 隶属鲉形目(Scorpaeniformes)、鲉科(Scorpaenidae)、平鲉属(Sebastes), 是黄、渤海海区常见的近海冷温型底层岩礁性鱼类, 生殖方式为卵胎生, 雄性与雌性一般分别需要3年和4年达到性成熟(Wourms, 1991)。具有肉质细嫩、味道鲜美、繁殖能力与适应性强、洄游范围小、自然越冬等优点, 已成为我国北方沿海网箱养殖、海洋放牧与海钓业的重要经济鱼类。目前网箱养殖的许氏平鲉达到500—750g/尾的上市规格, 大约需要3年的养殖时间, 并且苗种来源以海捕野生苗或带仔亲鱼室内人工繁育方式获得, 存在生长速度慢、养殖周期长和成活率低等问题, 而北方沿海对生长速度快的优良苗种需求量巨大, 因此, 亟需开展许氏平鲉人工良种选育工作, 满足养殖需求和提高养殖效率。
微卫星标记作为现代分子标记辅助遗传育种高效而有力的分子标记, 因其数量广、多态性高、易检测、共显性等特点(Nandakumar et al, 2004), 被广泛应用于大菱鲆(Scophthalmus maximus) (Chen et al, 2007)、牙鲆(Paralichthys olivaceus) (Liu et al, 2017)、半滑舌鳎(Cynoglossus semilaevis) (Liao et al, 2009)等各种海水鱼类的遗传结构分析、纯系与亲子鉴定、遗传连锁图谱构建与QTL定位等研究领域。目前, 在许氏平鲉微卫星标记开发与应用方面, 主要集中在利用磁珠富集法、简化基因组测序法和EST序列筛选法等方法进行微卫星标记的开发(et al, 2013; Ma et al, 2015; 薛蕊, 2015), 以及雌亲家系鉴定及遗传图谱的构建(初冠囡, 2012)等方面。
鱼类人工选育维持选育群体遗传多样性, 能提高选育后代对环境适应力与抗病力。封闭选育群体随着选育世代推进易受随机遗传漂变, 如瓶颈效应等作用而导致后代稀有等位基因丢失和遗传多样性降低, 增加近交衰退风险(Hartl et al, 1997)。长期人工选育的选择响应需要足够的遗传变异水平, 也要兼顾无关性状基因保持良好的多态性(Gamfeldt et al, 2007)。研究证明, 一般人工选育可能会造成群体遗传多样性的降低, 如Appleyard等(2006)利用微卫星分子标记技术对长牡蛎野生群体和人工4代选育群体的研究发现, 人工选育群体比野生群体的等位基因丰富度下降了13.9%—28.0%, 表明长牡蛎群体产生了瓶颈效应, 应该减少近亲交配、重新引种和进行遗传检测。田岳强等(2015)对大菱鲆连续选育三个世代进行遗传多样性分析发现, 随世代选育推进, 遗传多样性参数逐代下降, 部分位点基因型趋向纯合, 但多数位点仍保持较高遗传多样性。而在许氏平鲉选育群体的遗传多样性研究方面报道较少, 薛蕊(2015)对许氏平鲉17个快速生长选育系的遗传结构分析发现, 选育系的杂合度有所下降, 但其遗传多样性仍维持在较高水平, 仍具有较大选育潜力。而对许氏平鲉群体选育结合多代家系选育的生长性能及遗传多样性分析仍未见报道。
本研究以开展许氏平鲉遗传改良和培育优良新品系为主要目标, 选择荣成和长岛的野生许氏平鲉为基础群体, 采用群体选育(群体内和群体间)和家系选育相结合的方法, 得到2个群体选育和4个家系选育群体。对4个家系选育群体的生长性能进行了比较分析, 并利用20对高多态性的微卫星标记来评估许氏平鲉基础群体和选育群体的遗传多样性和遗传结构, 查明其遗传多样性水平与遗传潜力, 以期为下一步许氏平鲉良种选育工作提供理论指导。
1 材料与方法 1.1 样品采集本研究采集的许氏平鲉基础群体、群体和家系选育群体样本信息见表 1。2个基础群体分别采自荣成的野生群体(2005年, R)和长岛的野生群体(2007年, C), 挑选健康、活力强、个体大的性成熟个体作为亲本, 在室内养殖池自由交尾, 2006年建立荣成群体内选育子1代(R-F1), 2008年以荣成和长岛野生群体为基础群体建立群体间选育子1代(RC-F1), 2011年与2017年分别建立群体内的家系选育子2代(R-F2)、子3代(R-F3); 2013年和2017年依次建立群体间的家系选育子2代(RC-F2)、子3代(RC-F3), 家系选育均为半同胞雌亲家系。2017年集体产仔进行样本采集, 分别在30、60、90和120日龄取样测量体质量、体长和体高3个形态性状指标, 每个选育家系各取100尾, 共计1600尾。群体和家系建立的方法参照姜海滨等(2014), 选育后代培育条件一致, 均在烟台泰华海洋科技有限公司(国家级黑鲪原种场)进行养殖。
| 群体 | 群体类型 | 来源 | 群体 | 群体类型 | 来源 |
| R | 野生群体 | 荣成野生许氏平鲉 | C | 野生群体 | 长岛野生许氏平鲉 |
| R-F1 | 群体内选育 | 荣成♀×荣成♂ | RC-F1 | 群体间选育 | 荣成×长岛 |
| R-F2 | 家系选育 | 荣成半同胞家系2代 | RC-F2 | 家系选育 | 荣成和长岛半同胞家系2代 |
| R-F3 | 家系选育 | 荣成半同胞家系3代 | RC-F3 | 家系选育 | 荣成和长岛半同胞家系3代 |
每个群体随机选取32尾, 共计256尾, 活体剪取部分胸鳍, 存放于70%的乙醇中, 暂存于–20℃冰箱中备用。基因组DNA提取方法采用传统的饱和酚-氯仿-异戊醇的方法。提取完成后用0.8%的琼脂糖凝胶电泳检测其完整性, 通过NanoDrop OneC分光光度计检测DNA质量与浓度。稀释至50ng/μL以备后续遗传多样性分析使用。
1.3 微卫星引物设计与筛选本研究共选用20对高多态性的微卫星引物, 其中9对源自简化基因组测序法, 其余来自本课题组已开发的引物(贾超峰等, 2014; 韩承慧等, 2016), 这些引物扩增稳定、条带清晰, 适用于选育群体遗传特征分析, 序列信息见表 2。引物均由上海生工生物工程有限公司合成。PCR反应体系为25μL, 包含17.8μL ddH2O, 2.5μL 10×PCR Buffer (Mg2+), 0.5μL 10mmol/L dNTPs, 正反引物各1μL, 1U Taq酶, 2μL模板DNA。反应程序为: 94℃预变性5min; 94℃变性40s、退火40s、72℃延伸1min, 35个循环; 72℃延伸10min, 4℃下保存。PCR产物用8%非变形聚丙烯酰胺凝胶电泳检测, 用10bp DNA ladder标记等位基因位置, 使用银染法染色, 用Bio-5000 PLUS扫描仪扫描电泳图谱, 扩增产物片段大小由Gel-Pro analyzer 32凝胶分析软件读取。
| 位点 | 核心序列 | 引物序列(5'—3') | 退火温度(℃) | 产物大小(bp) | GenBank序列号 |
| HJ2-28 | (TG)13…(GCGTGT)3 | F:TGGAACCGAAGGAAGAGT | 62 | 163 | KM079278 |
| R:CTGCCATCATGGAGGAGA | |||||
| HJ3-11 | (GGA)5(GGC)7 …(GGA)4 |
F:GAACTCTTCTGTCTACGTCCAG | 62 | 148 | KM079279 |
| R:TGTCTTCAATCTTGGGTGG | |||||
| HJ6-19 | (GT)10 | F:TTGCAGACAGAATAAAATTCACGG | 60 | 114 | KT260128 |
| R:TGGCTTTACAACCATATCACTCCC | |||||
| HJ6-23 | (TGC)10 | F:GACGAGCTTCTTCCCATCAGG | 60 | 140 | KT260129 |
| R:TGACCACACCATCACTAGAAGAGC | |||||
| HJ6-25 | (GT)13 | F:CAACCCTCACTTCTGACAGATTCA | 60 | 140 | MG001942 |
| R:CTTCCTATGGAGCCTTGGATTTCT | |||||
| HJ7-13 | (CA)11 | F:AAGGCTTTAGGGGTGGTTAGTCTG | 60 | 142 | MG001943 |
| R:TCACCCTCTAACACGCTCTCTCTT | |||||
| HJ7-62 | (TG)16 | F:TGGTGTGAATGCTAACCTGCTAAC | 60 | 152 | MG001944 |
| R:CATGCAGCAGATCAGTACCTCTGT | |||||
| HJ8-9 | (GT)11 | F:CTGTGAAGCCACTGACTCCATTTA | 60 | 132 | MG001945 |
| R:CCAAGAGAGACATCTGGACAGTCA | |||||
| HJ8-11 | (AC)9 | F:AGTACGACAGACTGCTGGAGGAAC | 60 | 152 | MG001946 |
| R: CATCTCTGTGCATGGCATCATT | |||||
| HJ8-12 | (CAG)8 | F:GCCTCTGAAACAGCATAGACATCC | 60 | 121 | MG001947 |
| R:ATAATGAGCCATTGCATCACGTC | |||||
| HJ8-45 | (TC)7 | F:AGACAAAGTCACGGGAAAGTGAAC | 60 | 141 | MG001948 |
| R:GGGCATTCTGACAATATGACTTCC | |||||
| HJ8-83 | (TAGA)6 | F: AGGTCATTGGAAAACGGATAATCA | 60 | 133 | MG001949 |
| R:TGTCTGGATAACCTGCTGCTGTAA | |||||
| HJ9-13 | (AC)9 | F:CACCATCACTTCCACACTTACTGG | 60 | 120 | MG001950 |
| R:ATAACTGCGAAGCATGTGTGTGAT | |||||
| HJ9-83 | (AGAT)8 | F:TCTGTGGTTTCTCAATTTTGGGTT | 60 | 155 | MG001951 |
| R:GCTTGGATAACCTGCTGCTGTAAC | |||||
| HJ9-92 | (CTG)7 | F:GACTCTGGTCTCTGGTCTCTGGTC | 60 | 150 | MG001952 |
| R:ATCTACCGTGACTGCAGCTCATTT | |||||
| HJ10-13 | (AGAA)6 | F:TTGGAAAATGGTGTGCCCTATAAC | 60 | 159 | MG001953 |
| R:AAAAAGGATGTCTTCCATGAGCAC | |||||
| HJ10-20 | (TG)15 | F:TATTCACGTTTTCTTTTTCACCGC | 60 | 144 | MG001954 |
| R:AATATAGATTTCTTGGATGGGCCG | |||||
| HJ10-24 | (AC)11 | F:TCTGATCTGTCCCTATCCCCTAGA | 60 | 113 | MG001955 |
| R:TTCGAATAATCTAGAAAGCAGTGCG | |||||
| HJ10-36 | (GACAG)6 | F:GACCTGAACATCTCCATGTGAGTC | 60 | 159 | MG001956 |
| R:AAAACTTTTCCATCTCAGAGTTTGC | |||||
| HJ10-56 | (TG)12 | F:GTGGTAAACAGGCTGATCCTGAAA R:CACAAACAAACTGTTGAGAGGACG |
60 | 158 | MG001957 |
对不同生长阶段许氏平鲉选育家系进行绝对增重率(absolute growth rate, AGR)和特定生长率(specific growth rate, SGR)的计算, 公式如下:
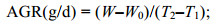
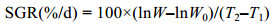
其中, W和W0分别表示终末体质量和初始体质量, T2和T1分别表示生长终末时间和初始时间。
采用SPSS 17.0软件, 使用ANOVA单因素方差分析和Duncan’s多重比较分析选育家系间体质量、体长、体高、AGR和SGR的差异性, 差异的显著性设置为P < 0.05。
使用PopGene32软件统计8个群体每个微卫星位点的等位基因数(N)、有效等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)、香农指数(I), 计算群体间的遗传相似性指数(Fis)及遗传距离(Ds); 使用Cervus 3.0软件计算多态信息含量(PIC); 使用GenePop软件计算P值, 检验Hardy-Weinberg平衡和连锁不平衡情况, 统计结果若P < 0.05则为差异显著; 使用FASTA计算选育群体间的遗传分化系数(Fst)和基因流(Nm); 使用MEGA 7.0软件采取UPGMA方法, 根据群体的遗传距离进行聚类分析, 作NJ聚类图和UPGMA聚类图。
2 结果 2.1 许氏平鲉选育家系不同日龄的生长性能比较许氏平鲉选育家系间的体质量、体长、体高以及各个生长阶段绝对增重率、特定生长率的比较结果见图 1(a—e)。由图 1可知, (1)经过120日龄养殖, 在体质量、体长和体高的平均值方面, R-F3均显著大于其他家系(P < 0.05), R-F3比R-F2分别提高21.59%、6.89%、4.92%, RC-F3比RC-F2提高30.99%、12.09%、11.70%。生长速度方面, 第3代家系选育均优于第2代家系选育, 群体内的家系选育优于群体间。(2) 4个选育家系的绝对增重率均随着生长天数的增加而上升, R-F3的上升幅度均最高, 分别为18.77%和26.78%; RC-F2的上升幅度均最低, 分别为14.47%和11.29%。第Ⅰ阶段各家系差异性不显著(P > 0.05), 第Ⅱ阶段R-F3和RC-F3均显著大于R-F2和RC-F2 (P < 0.05), 第Ⅲ阶段R-F3显著大于其他家系(P < 0.05)。(3)各家系特定生长率均随着天数的增加而先上升后下降, 但在每个阶段家系间差异性不显著(P > 0.05)。
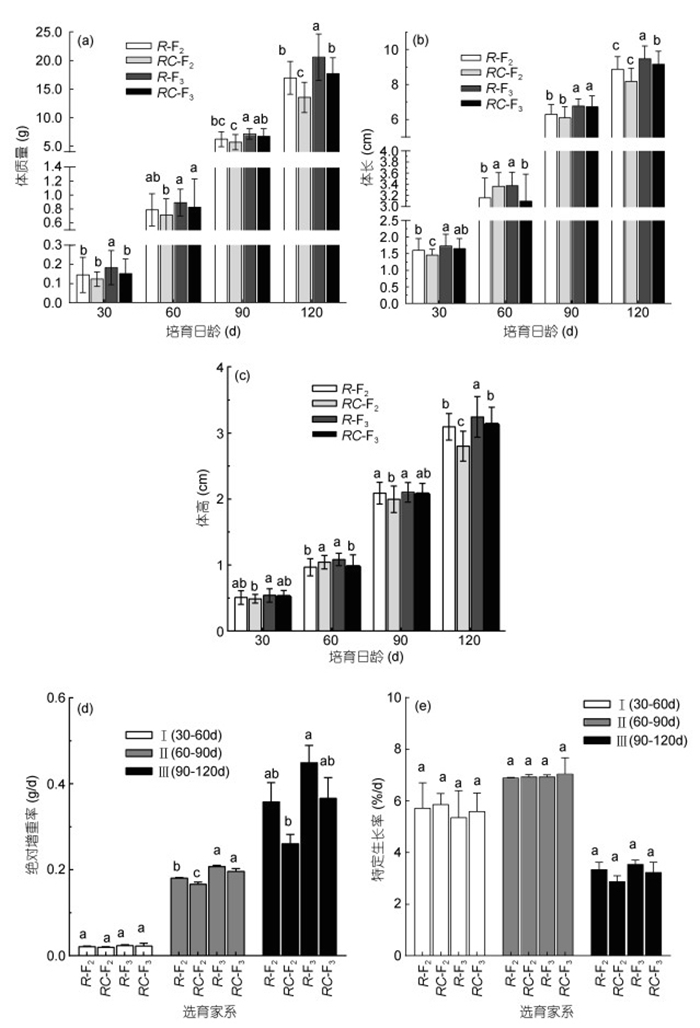 |
| 图 1 许氏平鲉4个选育家系体质量(a)、体长(b)、体高(c)、绝对增重率(d)和特定生长率(e)的比较 Fig. 1 Comparison in body weight (a), length (b), body height (c), AGR (d), and SGR (e) for the 4 breeding families of S. schlegelii 注:不同字母表示差异性显著(P < 0.05) |
本研究利用的20对微卫星引物均能在许氏平鲉样品中稳定的扩增出对应序列。图 2给出了HJ8-83标记在许氏平鲉群体中的聚丙烯酰胺凝胶电泳图。各个位点在群体中的遗传统计指标列于表 3。20个微卫星位点在8个群体中总共获得398个等位基因, 等位基因数(A)范围8—35个, 平均值为19.90, 等位基因数最多和最少分别为HJ7-13和HJ8-12。有效等位基因数范围为3.1471—8.5460, 平均值为6.2981。观测杂合度(Ho)和期望杂合度(He)分别介于0.3190— 0.9610和0.7230—0.9360, 每个位点的多态信息含量介于0.6780—0.9300之间。
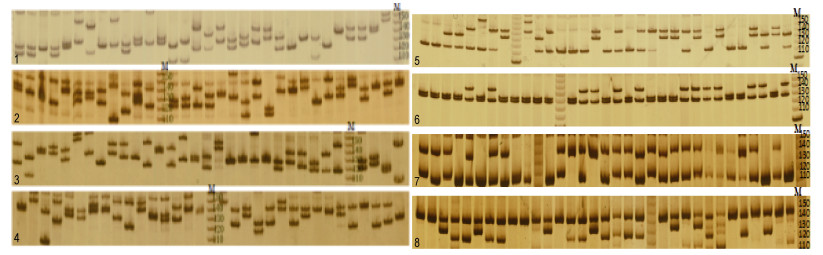 |
| 图 2 微卫星引物HJ8-83在许氏平鲉8个群体中的电泳图 Fig. 2 Electrophoresis patterns of primer HJ8-83 in the 8 populations of S. schlegelii 注: M为10bp Marker; 1:荣成野生群体R; 2:长岛野生群体C; 3:荣成群体内选育子一代R-F1; 4:荣成和长岛群体间选育子一代RC-F1; 5:荣成群体内的家系选育子2代R-F2; 6:群体间的家系选育子2代RC-F2; 7:群体内的家系选育子3代R-F3; 8:群体间的家系选育子3代RC-F3 |
| 位点 | 样本数n | 等位基因数A | 有效等位基因数Ne | 观测杂合度Ho | 期望杂合度He | 多态信息含量PIC |
| HJ2-28 | 256 | 27 | 6.9686 | 0.9380 | 0.9360 | 0.9300 |
| HJ3-11 | 245 | 11 | 4.9764 | 0.4570 | 0.7630 | 0.7280 |
| HJ6-19 | 249 | 25 | 8.5460 | 0.8550 | 0.9250 | 0.9180 |
| HJ6-23 | 255 | 30 | 8.4497 | 0.6750 | 0.9310 | 0.9250 |
| HJ6-25 | 248 | 23 | 5.9450 | 0.8990 | 0.8600 | 0.8460 |
| HJ7-13 | 251 | 35 | 6.2834 | 0.9200 | 0.8860 | 0.8740 |
| HJ7-62 | 246 | 17 | 6.5832 | 0.9590 | 0.8880 | 0.8750 |
| HJ8-9 | 256 | 16 | 7.0006 | 0.9340 | 0.8550 | 0.8370 |
| HJ8-11 | 244 | 20 | 6.4681 | 0.9470 | 0.9200 | 0.9130 |
| HJ8-12 | 254 | 8 | 3.1471 | 0.3940 | 0.7230 | 0.6780 |
| HJ8-45 | 256 | 9 | 4.3884 | 0.5080 | 0.7580 | 0.7190 |
| HJ8-83 | 254 | 23 | 7.1820 | 0.7130 | 0.9270 | 0.9210 |
| HJ9-13 | 248 | 18 | 5.1481 | 0.5440 | 0.8340 | 0.8160 |
| HJ9-83 | 234 | 27 | 7.4988 | 0.7260 | 0.9300 | 0.9240 |
| HJ9-92 | 251 | 25 | 7.3997 | 0.5020 | 0.8850 | 0.8740 |
| HJ10-13 | 254 | 9 | 3.4519 | 0.3190 | 0.7380 | 0.6920 |
| HJ10-20 | 248 | 21 | 6.7129 | 0.9480 | 0.8680 | 0.8540 |
| HJ10-24 | 256 | 20 | 7.8736 | 0.9340 | 0.9020 | 0.8920 |
| HJ10-36 | 255 | 16 | 6.6919 | 0.6980 | 0.8700 | 0.8550 |
| HJ10-56 | 256 | 18 | 5.2467 | 0.9610 | 0.9060 | 0.8970 |
| 平均值Mean | 250.80 | 19.90 | 6.2981 | 0.7416 | 0.8653 | 0.8484 |
各群体总的遗传多样性统计结果见表 4。平均有效等位基因数(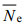
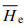
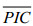
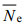
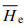
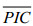
| 群体 | 平均等位基因数A | 平均有效等位基因数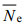 |
平均观测杂合度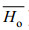 |
平均期望杂合度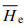 |
平均多态信息含量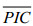 |
平均香农-威纳指数I |
| R | 12.00 | 7.9598 | 0.8356 | 0.8716 | 0.8418 | 2.1797 |
| C | 10.30 | 6.6488 | 0.8405 | 0.7912 | 0.8050 | 2.0003 |
| R-F1 | 10.50 | 6.5465 | 0.7947 | 0.8409 | 0.8061 | 1.9919 |
| RC-F1 | 9.55 | 6.7950 | 0.8178 | 0.7974 | 0.8071 | 1.9885 |
| R-F2 | 4.30 | 3.3845 | 0.7590 | 0.7001 | 0.7485 | 1.2836 |
| RC-F2 | 3.65 | 3.0611 | 0.7849 | 0.6598 | 0.7984 | 1.0944 |
| R-F3 | 3.95 | 3.2616 | 0.7230 | 0.6527 | 0.6580 | 1.1438 |
| RC-F3 | 3.60 | 2.9849 | 0.7549 | 0.6315 | 0.6472 | 1.0426 |
基于Hardy-Weinberg平衡定律对8个群体中的每个位点基因平衡状态和遗传偏离指数(d)进行检测(表 5), 在α=0.05和α=0.01显著或极显著水平上, 有7个位点在所有群体中呈Hardy-Weinberg平衡。基础群体R和C分别有5个和6个位点显著偏离Hardy- Weinberg平衡, 其中有HJ3-11、HJ9-92、HJ10-13、HJ10-36四个位点在R和C均出现显著偏离现象; 选育群体R-F1和RC-F1分别有8个和7个位点显著偏离Hardy-Weinberg平衡, 在两个群体中有6个共同位点出现偏离, 为HJ3-11、HJ6-23、HJ8-45、HJ8-83、HJ9-92、HJ10-13。在选育家系R-F2、RC-F2、R-F3、RC-F3分别有3、6、5、4个位点显著偏离Hardy-Weinberg平衡。总体来看, 这些位点符合Hardy-Weinberg平衡, 适用于本研究。8个群体的20个位点中, 有37.5%的位点遗传偏离指数(d)为负值, R、C、R-F1和RC-F1四个群体表现出杂合子缺失的位点分别有8、7、9、6个; 在R-F2、RC-F2、R-F3、RC-F3四个选育家系表现出杂合子缺失的位点分别为8、6、9、7个, 基础群体和选育群体家系的杂合子缺失数量差异不大。
| 位点 | 群体 | |||||||||||||||
| R | C | R-F1 | RC-F1 | R-F2 | RC-F2 | R-F3 | RC-F3 | |||||||||
| P | d | P | d | P | d | P | d | P | d | P | d | P | d | P | d | |
| HJ2-28 | 1.0000 | 0.1040 | 1.0000 | 0.0669 | 1.0000 | 0.1539 | 0.8686 | 0.1294 | 1.0000 | 0.2793 | 1.0000 | 0.1957 | 1.0000 | 0.3405 | 1.0000 | 0.9689 |
| HJ3-11 | 0.0000** | –0.5364 | 0.0150* | 0.3356 | 0.0134* | –0.2866 | 0.0077** | 0.3689 | 0.3893 | –0.1153 | — | — | 0.4437 | –0.0790 | 0.1622 | –0.1096 |
| HJ6-19 | 0.9426 | 0.0444 | 0.0602 | –0.1010 | 0.0018** | –0.2152 | 0.1992 | –0.1097 | 1.0000 | 0.2940 | 1.0000 | 0.1623 | 1.0000 | 0.2898 | 1.0000 | 0.1957 |
| HJ6-23 | 0.7014 | 0.0448 | 0.0815 | 0.1237 | 0.0000** | –0.2989 | 0.0316* | 0.0915 | 0.9738 | 0.1036 | 0.0016** | –0.1784 | 0.8860 | –0.2133 | 0.0012** | –0.3696 |
| HJ6-25 | 1.0000 | 0.1440 | 0.0579 | –0.0556 | 0.9980 | 0.1456 | 0.0718 | –0.0387 | 1.0000 | 0.5090 | 1.0000 | 0.3567 | 1.0000 | 0.4229 | 1.0000 | 0.4318 |
| HJ7-13 | 1.0000 | 0.0594 | 0.9964 | 0.1532 | 0.8678 | 0.1023 | 0.9986 | 0.1725 | 1.0000 | 0.3177 | 1.0000 | 0.3142 | 1.0000 | 0.9689 | 1.0000 | 0.2399 |
| HJ7-62 | 1.0000 | 0.1183 | 1.0000 | 0.2015 | 1.0000 | 0.3641 | 1.0000 | 0.2483 | 1.0000 | 0.2855 | 1.0000 | 0.9689 | 0.0036** | 0.1509 | 0.0024** | 0.2015 |
| HJ8-9 | 1.0000 | 0.2093 | 0.9084 | 0.0172 | 1.0000 | 0.1915 | 0.4425 | 0.0523 | 1.0000 | 0.3194 | 1.0000 | 0.2293 | 1.0000 | 0.9689 | 1.0000 | 0.3387 |
| HJ8-11 | 1.0000 | 0.1478 | 1.0000 | 0.0808 | 1.0000 | 0.1058 | 0.7855 | 0.1432 | 1.0000 | 0.3298 | 1.0000 | 0.4318 | 1.0000 | 0.2460 | 1.0000 | 0.2768 |
| HJ8-12 | 0.0005** | –0.5240 | 0.1854 | 0.3011 | 0.0123* | –0.3476 | 0.0946 | 0.1831 | 0.0582 | –0.7897 | 0.0025** | –0.1703 | 0.0000** | –0.2422 | 0.0256* | –0.4318 |
| HJ8-45 | 0.0684 | –0.1625 | 0.0001** | –0.1042 | 0.0000** | –0.4234 | 0.0091** | –0.2500 | — | –0.1854 | 0.0133* | –0.3152 | 0.0025** | — | 1.0000 | 0.9689 |
| HJ8-83 | 0.2542 | –0.0480 | 0.0310* | –0.2427 | 0.0422* | –0.0720 | 0.0022** | –0.1265 | 0.0086** | –0.0379 | 0.8038 | 0.0153 | 0.1747 | –0.1170 | 1.0000 | 0.5860 |
| HJ9-13 | 1.0000 | 0.1023 | 0.3945 | –0.0120 | 0.8890 | 0.0396 | 0.3215 | 0.0061 | 0.0000** | –0.7373 | 0.0235* | –0.0832 | 0.0000** | –1.0000 | 0.4056 | –0.2435 |
| HJ9-83 | 0.2880 | –0.0297 | 0.1401 | 0.0414 | 0.9332 | 0.0954 | 0.0800 | 0.1267 | 0.0000** | –0.0499 | 0.0981 | –0.0553 | 0.4099 | –0.2636 | 0.9764 | –0.1676 |
| HJ9-92 | 0.0000** | –0.2876 | 0.0000** | –0.4464 | 0.0000** | –0.3606 | 0.0000** | –0.3585 | 0.2387 | –0.1291 | 0.0137* | –0.7643 | 0.0722 | –0.1551 | 1.0000 | –0.3123 |
| HJ10-13 | 0.0000** | –0.5288 | 0.0000** | 0.4706 | 0.0000** | –0.9154 | 0.0043** | 0.5407 | 0.9939 | –0.5995 | 1.0000 | 0.1665 | 0.0010** | –0.3587 | 1.0000 | 0.3404 |
| HJ10-20 | 1.0000 | 0.1064 | 1.0000 | 0.1192 | 1.0000 | 0.0915 | 1.0000 | 0.1046 | 1.0000 | 0.3289 | 1.0000 | 0.9666 | 1.0000 | 0.3235 | 1.0000 | 0.9689 |
| HJ10-24 | 1.0000 | 0.1199 | 1.0000 | 0.0769 | 1.0000 | 0.2413 | 1.0000 | 0.1101 | 1.0000 | 0.4318 | 1.0000 | 0.4318 | 1.0000 | 0.4318 | 0.0086** | 0.2538 |
| HJ10-36 | 0.0111* | –0.2628 | 0.0000** | –0.3550 | 0.5224 | –0.0949 | 0.0000** | –0.2500 | 0.9841 | 0.2762 | 0.0023** | 0.1085 | 0.9995 | –0.2591 | 1.0000 | –0.4651 |
| HJ10-56 | 1.0000 | 0.1494 | 1.0000 | 0.0045 | 1.0000 | 0.2857 | 0.8686 | 0.1294 | 1.0000 | 0.2585 | 1.0000 | 0.9689 | 1.0000 | 0.3280 | 1.0000 | 0.3405 |
| 注: *表示差异显著(P < 0.05); **表示差异极显著(P < 0.01) | ||||||||||||||||
各位点的遗传分化系数(Fst)和基因流(Nm)见表 6, 各群体间存在中度遗传分化(0.05 < Fst < 0.15)的位点为14个; 有较大遗传分化(0.15 < Fst < 0.25)的位点有6个; 各群体平均基因分化系数为0.1279, 表明12.79%的遗传分化来自群体间, 而87.21%的遗传分化来自群体内。Nm和Fst呈负相关关系, 因此基因流所反映的群体遗传分化与基因分化系数相似。根据基因流的大小, 在各群体间遗传分化较小(Nm > 4)的位点仅有HJ6-19, 群体间分化较大的位点也仅有1个(HJ8-45), 其余18个位点在各基础群体和选育群体及家系之间存在一定程度的分化。
| 位点 | Fst | Nm | 位点 | Fst | Nm |
| HJ2-28 | 0.0893 | 2.5499 | HJ8-45 | 0.2299 | 0.8374 |
| HJ3-11 | 0.2152 | 0.9117 | HJ8-83 | 0.1277 | 1.7072 |
| HJ6-19 | 0.0533 | 4.4414 | HJ9-13 | 0.1382 | 1.5594 |
| HJ6-23 | 0.1836 | 1.1114 | HJ9-83 | 0.1085 | 2.0541 |
| HJ6-25 | 0.0983 | 2.2922 | HJ9-92 | 0.1359 | 1.5902 |
| HJ7-13 | 0.0958 | 2.3584 | HJ10-13 | 0.1999 | 1.0007 |
| HJ7-62 | 0.0993 | 2.2668 | HJ10-20 | 0.1204 | 1.8266 |
| HJ8-9 | 0.1080 | 2.0642 | HJ10-24 | 0.0998 | 2.2552 |
| HJ8-11 | 0.0640 | 3.6583 | HJ10-36 | 0.1643 | 1.2719 |
| HJ8-12 | 0.1826 | 1.1188 | HJ10-56 | 0.0826 | 2.7770 |
| 平均值 | 0.1279 | 1.7054 |
根据Nei(1978)计算群体间的遗传距离和遗传相似系数(表 7), 所有群体间的遗传相似系数在0.3694—0.7797之间, 遗传距离为0.2489—0.9959。C和RC-F1的遗传相似指数最高(0.7797), 遗传距离最小(0.2489), 说明C和RC-F1群体的亲缘关系最近; R和RC-F3群体的遗传相似系数最低(0.3694), 遗传距离最大(0.9959), 说明R和RC-F3群体的亲缘关系最远。其次R-F1和RC-F3遗传距离也较远, 达到0.9848。
| 群体 | R | C | R-F1 | RC-F1 | R-F2 | RC-F2 | R-F3 | RC-F3 |
| R | — | 0.6173 | 0.7226 | 0.5693 | 0.5645 | 0.4301 | 0.4318 | 0.3694 |
| C | 0.4823 | — | 0.6732 | 0.7797 | 0.5789 | 0.5098 | 0.5661 | 0.5546 |
| R-F1 | 0.3249 | 0.3957 | — | 0.5912 | 0.5110 | 0.4320 | 0.3765 | 0.3735 |
| RC-F1 | 0.5634 | 0.2489 | 0.5256 | — | 0.7021 | 0.5747 | 0.6234 | 0.5857 |
| R-F2 | 0.5719 | 0.5467 | 0.6714 | 0.3536 | — | 0.6219 | 0.6866 | 0.6993 |
| RC-F2 | 0.8436 | 0.6736 | 0.8392 | 0.5539 | 0.4750 | — | 0.5925 | 0.6459 |
| R-F3 | 0.8399 | 0.5690 | 0.9769 | 0.4725 | 0.3759 | 0.5234 | — | 0.7248 |
| RC-F3 | 0.9959 | 0.5896 | 0.9848 | 0.5349 | 0.3577 | 0.4371 | 0.3219 | — |
依据Nei遗传距离构建群体间聚类图(UPGMA聚类图和NJ聚类图)见图 3。因聚类方法有所差异, 两种聚类图显示了各自的特点, 虽有不同, 但都能真实地反应群体间遗传关系。经统计发现, R、C基础群体与R-F1、RC-F1选育群体一代都被聚为一类, R-F2、RC-F2、R-F3、RC-F3四个选育家系聚为一类; R与R-F1单独聚为一支, R-F3与RC-F3聚为一支, 再与RC-F2聚为一支, 然后与R-F2聚为一支。
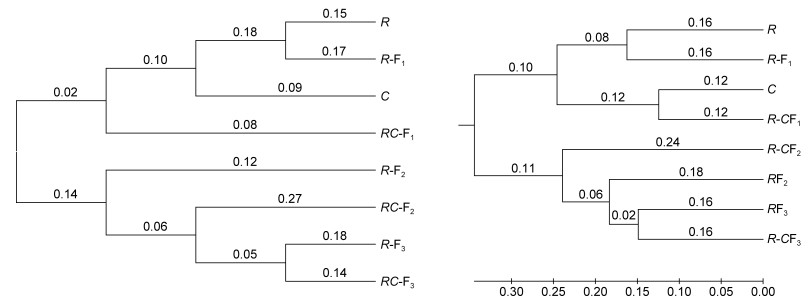 |
| 图 3 许氏平鲉8个群体亲缘关系的NJ聚类图(左)和UPGMA聚类图(右) Fig. 3 NJ dendrogram (left) and UPGMA dendrogram (right) of 8 S. schlegelii populations based on Nei's unbiased genetic distances |
选择育种是以育种性状为目标, 在原始资源群体中选择优良变异群体或个体, 不断选择和固定优良经济性状, 从而形成新品系或者品种(刘小林等, 2003)。许氏平鲉繁殖方式较特殊, 是“一雌多雄”交尾方式, 而且是卵胎生鱼类(Yoshida, 2001)。本研究首先采用群体内选育和不同地理群体间杂交选育相结合的方法, 然后分别构建两代同母异父半同胞家系来进行良种选育。姜海滨等(2014)研究表明半同胞家系的生长性状能较好地反映母本和相对应父本遗传因子差别及对后代性状的影响。俄罗斯鲟(Acipenser gueldenstaedtii) (罗坤等, 2015)、凡纳滨对虾(Litopenaeus vannamei) (Argue, 2002)等品种上均采用了群体选育和家系选育相结合的方式进行优良品种的选育。本研究中, 选育子3代比选育子2代生长速度分别提高了21.59%和30.99%, 随着养殖天数的增加, 选育子3代比选育子2代的绝对增重率提高显著, 两种路线选育效果上十分明显, 在生长速度、绝对增重率和特定生长率方面群体内家系选育子3代优于群体间家系选育子3代。
杂种优势在遗传学上表现为杂交子代在存活率、繁殖力及生长性能等方面均优于双亲均值, 在选育中利用同种不同地理群体杂交可能获得杂交优势。Falconer等(1996)认为进行群体选育的基础群体的基因频率不同, 其杂交后代将可能产生杂种优势。王卫军等(2015)对中国、日本和韩国3个长牡蛎F4代选育群体采用完全双列杂交后建立3个正交群体、3个反交群体和3个自交群体, 研究发现多数杂交子代比自交子代在生长性状上存在一定程度的杂种优势。然而, 不同地理种群的杂交可能产生杂交衰退的现象, 在尼罗罗非鱼(Oreochromis niloticus)、溪红点鲑(Salvelinus fontinalis)等(Bentsen et al, 1998; Granier et al, 2011)的群体杂交中发现, 并不是所有杂交系均能表现出超过纯系的生长优势, 有时杂交衰退和杂种优势同时存在。本研究中群体内世代选育家系(R-F3)的体质量、体高和体长的生长速度显著高于群体间杂交的选育家系(RC-F3), 而杂交系在生长性能方面并未表现出杂种优势, 孙际佳等(2016)对翘嘴鳜的家系选育中发现杂交系(HCJC和JCHC)的生长速度均显著小于纯种家系(JCJC和HCHC), 其结果与本研究相似。一方面可能是因为自身物种杂种优势与转录调节方式和群体特殊的遗传结构有关(Bougas et al, 2010), 另一方面可能与许氏平鲉“一雌多雄”的交尾方式有关, 多父本交配机制可能使其后代保持较高的杂合性, 因而杂交效果较难预测。
3.2 许氏平鲉选育群体的遗传多样性分析鱼类基础选育群体的遗传多样性水平的高低是衡量遗传潜力的重要指标, 遗传多样性水平越高, 对其进行种质资源挖掘和利用的潜力也越大。整个育种体系需在达到连续遗传改良的目的和维持较高遗传多样性水平之间保持相对平衡, 从而增强后代对环境的适应力和抗病力。微卫星标记是评定水产动物群体遗传多样性水平的重要手段之一(Liu et al, 2004)。本研究中20对微卫星位点PIC值均大于0.5(范围在0.6780—0.9300), 表现为高度多态(Ortí et al, 1997), 表明这些标记可用于许氏平鲉群体遗传分析。Leberg(2002)研究认为有效等位基因数(Ne)、期望杂合度(Ho)是衡量群体遗传多样性大小的指标, 本研究中选育群体1代R-F1和RC-F1的与基础群体(R和C)的Ne相差无几, 表明群体选育均保持很高的遗传多样水平。而选育家系随着世代推进, R-F3和RC-F3的Ne下降到3.2616和2.9849, 说明家系选育对后代群体的遗产结构产生影响, 但He仍分别为0.6527和0.6315, 说明家系选育后代有效等位基因数有所降低, 但这与韩承慧等(2016)检测的养殖许氏平鲉群体多样性(Ne=0.6365, He=0.5840)相比, He值相对更大, 表明选育家系仍保持较高的遗传多样性, 具有较大遗传潜力。本研究对群体内和群体间的家系选育群体遗传多样性检测时, 两者差异不明显, 原因可能是采集的样本为雌亲家系后代所导致, 与其他鱼类育种中的选育系遗传多样性有区别。
本研究检测了许氏平鲉群体显著偏离Hardy- Weinberg平衡的情况, 在所选择的20个微卫星标记中, 有10个位点在不同群体中出现了偏离Hardy- Wernberg平衡的现象。Rousset等(1995)研究发现杂合子缺失、无非随机交配群体、无效等位基因及人工选择等均能引起Hardy-Wernberg平衡的偏离。在本研究中8个群体20个位点中, 存在37.5%的位点遗传偏离指数(d)为负值, 表明杂合子缺失是造成许氏平鲉群体偏离平衡的一个原因。而选育群体与基础群体间的杂合子缺失数量差异不大, 可能原因是实验样本选取数量较少, 也可能存在着无效等位基因, 在实际进行数据处理时将其作为纯合子计算, 这对最终的数据会产生一定影响。
3.3 许氏平鲉选育群体家系间的遗传分化本研究利用遗传分化指数来评定群体间遗传分化程度。群体间遗传分化变异系数的范围为0到1, 参照Balloux等(2002)研究标准, 0.05—0.15表示群体分化程度为中等, 本研究在8个许氏平鲉群体的20个微卫星位点中的平均遗传分化变异系数为0.1279, 表明各群体之间存在中度的遗传分化。Crawford等(1998)得出由微卫星计算得到分化时间的长短, 能客观反映群体间的遗传变异。该研究中遗传距离结果表明基础群体R和群体内选育子1代R-F1分别与群体间家系选育子3代RC-F3的遗传距离最远(0.9959、0.9848)。建议在以后的选育过程选择R与RC-F3或R-F1与RC-F3群体中生长优良后代进行杂交, 后代可能产生较明显的杂种优势。以上结果为以后许氏平鲉选育群体优良性状的整合利用提供了参考。
4 结论本研究采用群体选育与家系选育方法, 获得2个群体选育群体和4个家系选育群体, 一方面对选育家系的生长性能进行比较分析, 表明选育子3代生长速度显著快于子2代, 群体内家系选育生长速度优于群体间家系选育, 生长上未表现出杂种优势; 另一方面通过对选育后代进行遗传多样性和遗传结构分析, 表明经过一代群体选育和两代家系选育后仍维持较高的遗传多样性水平。本研究为选育出快速生长许氏平鲉新品系提供理论基础。
| 王卫军, 李琪, 杨建敏, 等, 2015. 长牡蛎(Crassostrea gigas)三个选育群体完全双列杂交后代生长性状分析. 海洋与湖沼, 46(3): 628–635 DOI:10.11693/hyhz20141100314 |
| 田岳强, 2015. 大菱鲆选育世代遗传多样性分析及生长性状微卫星标记筛选. 上海: 上海海洋大学硕士学位论文, 26-29 |
| 刘小林, 相建海., 2003. 重要经济贝类选择育种及遗传力研究进展. 海洋科学, 27(6): 15–20 |
| 孙际佳, 何建国, 刘丽, 等, 2016. 翘嘴鳜选育群体的生长和遗传特征分析. 中国水产科学, 23(2): 425–435 |
| 初冠囡, 2012. 许氏平鲉(Sebastes schlegelii)微卫星标记筛选及遗传连锁图谱构建. 青岛: 中国海洋大学博士学位论文, 65-97 |
| 罗坤, 夏永涛, 王斌, 等, 2015. 俄罗斯鲟(Acipenser gueldenstaedtii)家系构建及不同家系间生长性能比较. 海洋与湖沼, 46(2): 463–469 DOI:10.11693/hyhz20140600158 |
| 姜海滨, 马海涛, 刘丽娟, 等, 2014. 黑鲪半同胞家系选育的初步研究. 海洋科学, 38(10): 70–75 DOI:10.11759/hykx20130814001 |
| 贾超峰, 马海涛, 姜海滨, 等, 2014. 许氏平鲉微卫星标记的开发及评价. 上海海洋大学学报, 23(3): 345–350 |
| 韩承慧, 马海涛, 姜海滨, 等, 2016. 许氏平鲉(Sebastes schlegelii)微卫星标记开发及野生、养殖群体遗传多样性分析. 海洋与湖沼, 47(1): 213–220 |
| 薛蕊, 2015. 许氏平鲉(Sebastes schlegelii)EST-SSR标记开发及快速生长选育系遗传结构分析. 上海: 上海海洋大学硕士学位论文, 23-29 |
| Appleyard S A, Ward R D., 2006. Genetic diversity and effective population size in mass selection lines of Pacific oyster (Crassostrea gigas). Aquaculture, 254(1-4): 148–159 DOI:10.1016/j.aquaculture.2005.10.017 |
| Argue B J, Arce S M, Lotz J M, et al, 2002. Selective breeding of Pacific white shrimp (Litopenaeus vannamei) for growth and resistance to Taura Syndrome Virus. Aquaculture, 204(3-4): 447–460 DOI:10.1016/S0044-8486(01)00830-4 |
| Balloux F, Lugon-Moulin N., 2002. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 11(2): 155–165 DOI:10.1046/j.0962-1083.2001.01436.x |
| Bentsen H B, Eknath A E, Vera M S P D, et al, 1998. Genetic improvement of farmed tilapias:growth performance in a complete diallel cross experiment with eight strains of Oreochromis niloticus. Aquaculture, 160(1-2): 145–173 DOI:10.1016/S0044-8486(97)00230-5 |
| Bougas B, Granier S, Audet C, et al, 2010. The transcriptional landscape of cross-specific hybrids and its possible link with growth in brook charr (Salvelinus fontinalis Mitchill). Genetics, 186(1): 97–107 DOI:10.1534/genetics.110.118158 |
| Chen S L, Ma H Y, Jiang Y, et al, 2007. Isolation and characterization of polymorphic microsatellite loci from an EST library of turbot (Scophthalmus maximus) and cross-species amplification. Molecular Ecology Notes, 7(5): 848–850 DOI:10.1111/men.2007.7.issue-5 |
| Crawford A M, Littlejohn R P., 1998. The use of DNA markers in deciding conservation priorities in sheep and other livestock. Animal Genetic Resources Information, 23: 21–26 DOI:10.1017/S1014233900004909 |
| Falconer D S, Mackay T F C, 1996. Introduction to Quantitative Genetics. 4th ed. New York, USA: Longman |
| Gamfeldt L, K llstr m B., 2007. Increasing intraspecific diversity increases predictability in population survival in the face of perturbations. Oikos, 116(4): 700–705 DOI:10.1111/oik.2007.116.issue-4 |
| Granier S, Audet C, Bernatchez L., 2011. Heterosis and outbreeding depression between strains of young-of-the-year brook trout (Salvelinus fontinalis). Canadian Journal of Zoology, 89(3): 190–198 DOI:10.1139/Z10-108 |
| Hartl D L, Clark A G, 1997. Principles of Population Genetics. 3rd ed. Sunderland, MA, Britain: Sinauer Associates, 145-149 |
| Leberg P L., 2002. Estimating allelic richness:effects of sample size and bottlenecks. Molecular Ecology, 11(11): 2445–2449 |
| Liao X L, Ma H Y, Xu G B, et al, 2009. Construction of a genetic linkage map and mapping of a female-specific DNA marker in half-smooth tongue sole (Cynoglossus semilaevis). Marine Biotechnology, 11(6): 699–709 DOI:10.1007/s10126-009-9184-3 |
| Liu Y X, Yang R Q, Liu Y, et al, 2017. Use of Microsatellite DNA Profiling to Identify Japanese Flounder, Paralichthys olivaceus of Hatchery Origin. Journal of the World Aquaculture Society, 48(2): 353–359 DOI:10.1111/jwas.2017.48.issue-2 |
| Liu Z J, Cordes J F., 2004. DNA marker technologies and their applications in aquaculture genetics. Aquaculture, 238(1-4): 1–37 DOI:10.1016/j.aquaculture.2004.05.027 |
| Ma H T, Jia C F, Yang J M, et al, 2015. Development of novel microsatellite markers in the Korean rockfish Sebastes schlegeli. Genetics and Molecular Research, 14(2): 5099–5102 DOI:10.4238/2015.May.12.12 |
| Nandakumar N, Singh A K, Sharma R K, et al, 2004. Molecular fingerprinting of hybrids and assessment of genetic purity of hybrid seeds in rice using microsatellite markers. Euphytica, 136(3): 257–264 DOI:10.1023/B:EUPH.0000032706.92360.c6 |
| Ortí G, Pearse D E, Avise J C., 1997. Phylogenetic assessment of length variation at a microsatellite locus. Proceedings of the National Academy of Sciences of the United States of America, 94(20): 10745–10749 DOI:10.1073/pnas.94.20.10745 |
| Rousset F, Raymond M., 1995. Testing heterozygote excess and deficiency. Genetics, 140(4): 1413–1419 |
| Wourms J P., 1991. Reproduction and development of Sebastes in the context of the evolution of piscine viviparity. Environmental Biology of Fishes, 30(1-2): 111–126 DOI:10.1007/BF02296882 |
| Yasuike M, Noda T, Fujinami Y, et al, 2013. Tri-, tetra-and pentanucleotide-repeat microsatellite markers for the Schlegel's black rockfish Sebastes schlegelii:the potential for reconstructing parentages. Conservation Genetics Resources, 5(2): 577–581 DOI:10.1007/s12686-012-9856-6 |
| Yoshida K, Nakagawa M, Wada S., 2001. Pedigree tracing of a hatchery-reared stock used for aquaculture and stock enhancement based on DNA markers. Fish Genetics and Breeding Science, 30: 27–35 |
 2018, Vol. 49
2018, Vol. 49


