
中国海洋湖沼学会主办。
文章信息
- 张晓洁, 孔令锋, 李琪. 2018.
- ZHANG Xiao-Jie, KONG Ling-Feng, LI Qi. 2018.
- 中国沿海常见蜑螺科贝类的DNA条形码
- DNA barcoding in Neritidae Species (Gastropoda, Neritimorpha) along the coast of China
- 海洋与湖沼, 49(3): 614-623
- Oceanologia et Limnologia Sinica, 49(3): 614-623.
- http://dx.doi.org/10.11693/hyhz20171200320
-
文章历史
- 收稿日期:2017-12-20
- 收修改稿日期:2018-01-15
蜑螺科(Neritidae)隶属于软体动物门(Mollusca)、腹足纲(Gastropoda)、蜑螺亚纲(Neritimorpha)、珍珠蜑螺目(Cycloneritimorpha)(Bouchet et al, 2005)。该科贝类是一类广盐性腹足类动物, 生境多样(Tan et al, 2008), 绝大多数生活在海岸潮间带(Frey, 2008), 少数生活在淡水和河口地带(Thompson, 1980; Beesley et al, 1998), 为贝类当中极少数能够生活在海洋及河川的种类(Fukumori et al, 2014)。目前世界上共记录蜑螺科16属, 现生种280余种(Spencer et al, 2007)。中国已发现蜑螺40种左右, 主要分布于东南部海域(张素萍, 2008)。
蜑螺科以及整个蜑螺亚纲在海洋腹足类的形态结构和系统演化上是较独特的分支(Kano et al, 2002), 在软体动物分类学研究中一直备受关注。长期以来, 蜑螺科的分类主要基于形态学特征(Woods et al, 1986; Schütt et al, 1992; Frey, 2008; Jagt et al, 2008), 而大多数蜑螺科种类由于适应辐射进化出多样的贝壳形态以及生态习性, 仅依靠贝壳形态特征为依据容易产生鉴定错误和分类争议(Grüneberg, 1976, 1982, Dekker, 2000; 陈志云等, 2016), 例如分类以及生态研究中常常将漆黑蜑螺N. atramentosa Reeve, 1855和小黑蜑螺N. melanotragus E. A. Smith, 1884这两个种混淆(Underwood, 1976, 2004; Waters et al, 2005)。
与传统分类学方法相比, DNA条形码在物种的快速鉴定、发现新种和隐存种等方面有着显著优势。自2003年Hebert提出DNA条形码概念(Hebert et al, 2003), 越来越多的生物学家应用这种方法进行物种鉴定和系统发生研究。由于能够从生物体的部分零散组织中对物种进行快速鉴定, DNA条形码技术已经开始应用于食品鉴定与保护生物学领域(Galimberti et al, 2013; Sheth et al, 2017); DNA条形码也能够在生物的不同生长阶段鉴定物种, 而且这种能力已经在多种动物类群中得到证实(Azmir et al, 2017; Hofmann et al, 2017); 此外, DNA条形码技术在分子系统发育和群体遗传学方面的应用也正被广泛关注(Padula et al, 2017)。
在蜑螺的分类学研究中, 应用DNA条形码技术可以在很大程度上避免贝壳形态多样性带来的误导。Chee等(2016)、Chee(2015)利用DNA条形码技术研究了马来西亚蜑螺科Nerina属与Neritina属的物种多样性, 发现了3个新纪录种。本研究以中国东南沿海3属7种蜑螺为材料, 分析了COI和16S rRNA基因的遗传变异, 旨在探讨DNA条形码技术在蜑螺科贝类鉴定中的可行性。
1 材料与方法 1.1 样品采集本研究所用61个蜑螺科样品均采自中国东南沿海, 固定于95%酒精溶液中, 保存于中国海洋大学水产学院贝类遗传育种研究室。依据Okutani(2017)、WoRMS (世界海洋物种名录, World Register of Marine Species, http://www.marinespecies.org/index.php)等文献资料进行鉴定, 这些样品隶属于3属7种, 分别为渔舟蜑螺Nerita albicilla Linnaeus, 1758、齿纹蜑螺Nerita yoldii Récluz, 1841、日本蜑螺Nerita japonica Dunker, 1860、黑玉蜑螺Nerita incerta von dem Busch, 1844、紫游螺Neripteron violaceum (Gmelin, 1791)、奥莱彩螺Clithon oualaniense (Lesson, 1831)和多色彩螺Clithon sowerbianum (Récluz, 1843)。研究所用样品的采集时间、采集地点、单倍型和Genbank序列号等详细信息见表 1, 代表个体如图 1所示。
| 物种 | 样品编号 | 采集时间 | 采集地点 | COI单倍型编号 | GenBank序列号 | 16SrRNA单倍型编号 | GenBank序列号 |
| 渔舟蜑螺 Nerita albicilla |
YZDL1 | 20080510 | 海南文昌 | Hap1 | MF687991 | Hap1 | MF687964 |
| YZDL2 | 20080510 | 海南文昌 | Hap2 | MF687992 | Hap2 | MF687965 | |
| YZDL3 | 20130917 | 广东阳江 | Hap2 | MF687992 | Hap3 | MF687966 | |
| YZDL4 | 20080409 | 广西涠洲岛 | Hap2 | MF687992 | Hap3 | MF687966 | |
| YZDL5 | 20080409 | 广西涠洲岛 | Hap3 | MF687993 | Hap3 | MF687966 | |
| YZDL6 | 20080218 | 海南三亚 | Hap4 | MF687994 | Hap3 | MF687966 | |
| YZDL7 | 20080218 | 海南三亚 | Hap5 | MF687995 | Hap3 | MF687966 | |
| YZDL8 | 20080510 | 海南文昌 | Hap5 | MF687995 | Hap3 | MF687966 | |
| YZDL9 | 20080426 | 广西防城港 | Hap6 | MF687996 | Hap3 | MF687966 | |
| YZDL10 | 20080415 | 广西北海 | Hap7 | MF687997 | Hap3 | MF687966 | |
| YZDL11 | 20080403 | 广西北海 | Hap8 | MF687998 | Hap3 | MF687966 | |
| YZDL12 | 20130917 | 广东阳江 | Hap9 | MF687999 | Hap3 | MF687966 | |
| YZDL13 | 20080512 | 海南临高 | Hap10 | MF688000 | Hap4 | MF687967 | |
| YZDL14 | 20080426 | 广西防城港 | Hap11 | MF688001 | Hap4 | MF687967 | |
| YZDL15 | 20080409 | 广西涠洲岛 | Hap12 | MF688002 | Hap4 | MF687967 | |
| YZDL16 | 20080409 | 广西涠洲岛 | Hap13 | MF688003 | Hap4 | MF687967 | |
| YZDL17 | 20080419 | 广东湛江 | Hap13 | MF688003 | Hap4 | MF687967 | |
| YZDL18 | 20080403 | 广西北海 | Hap13 | MF688003 | Hap4 | MF687967 | |
| YZDL19 | 20080403 | 广西北海 | Hap13 | MF688003 | Hap4 | MF687967 | |
| YZDL20 | 20080403 | 广西北海 | Hap13 | MF688003 | Hap5 | MF687968 | |
| YZDL21 | 20080510 | 海南文昌 | Hap13 | MF688003 | Hap6 | MF687969 | |
| YZDL22 | 20080419 | 广东湛江 | Hap13 | MF688003 | Hap7 | MF687970 | |
| YZDL23 | 20130915 | 福建漳州东山 | Hap14 | MF688004 | Hap8 | MF687971 | |
| YZDL24 | 20130915 | 福建漳州东山 | Hap15 | MF688005 | Hap9 | MF687972 | |
| YZDL25 | 20080218 | 海南三亚 | Hap16 | MF688006 | Hap10 | MF687973 | |
| YZDL26 | 20080403 | 广西北海 | Hap17 | MF688007 | Hap11 | MF687974 | |
| YZDL27 | 20080409 | 广西涠洲岛 | Hap18 | MF688008 | Hap12 | MF687975 | |
| 齿纹蜑螺 Nerita yoldii |
CWDL1 | 20090824 | 福建福清 | Hap19 | MF688011 | Hap13 | MF687978 |
| CWDL2 | 20080426 | 广西防城港 | Hap20 | MF688012 | Hap13 | MF687978 | |
| CWDL3 | 20080414 | 广西北海 | Hap21 | MF688013 | Hap13 | MF687978 | |
| CWDL4 | 20070629 | 浙江嵊泗 | Hap22 | MF688014 | Hap13 | MF687978 | |
| CWDL5 | 20070627 | 浙江舟山 | Hap22 | MF688014 | Hap13 | MF687978 | |
| CWDL6 | 20130917 | 广东阳江 | Hap22 | MF688014 | Hap13 | MF687978 | |
| CWDL7 | 20070627 | 浙江舟山 | Hap22 | MF688014 | Hap14 | MF687979 | |
| CWDL8 | 20130917 | 广东阳江 | Hap23 | MF688015 | Hap14 | MF687979 | |
| CWDL9 | 20070615 | 浙江南麂 | Hap24 | MF688016 | Hap15 | MF687980 | |
| 日本蜑螺 Nerita japonica |
RBDL1 | 20100603 | 浙江南麂 | Hap25 | MF688010 | Hap16 | MF687977 |
| RBDL2 | 20070615 | 浙江南麂 | Hap25 | MF688010 | Hap16 | MF687977 | |
| RBDL3 | 20070615 | 浙江南麂 | Hap25 | MF688010 | Hap16 | MF687977 | |
| RBDL4 | 20070615 | 浙江南麂 | Hap25 | MF688010 | Hap16 | MF687977 | |
| RBDL5 | 20070615 | 浙江南麂 | Hap25 | MF688010 | Hap16 | MF687977 | |
| 黑玉蜑螺Nerita incerta | HYDL1 | 20080510 | 海南文昌 | Hap26 | MF688009 | Hap17 | MF687976 |
| 紫游螺 Neripteron violaceum |
ZYL1 | 20170422 | 广西防城港 | Hap27 | MF688017 | Hap18 | MF687981 |
| ZYL2 | 20170422 | 广西防城港 | Hap28 | MF688018 | Hap18 | MF687981 | |
| ZYL3 | 20170422 | 广西防城港 | Hap28 | MF688018 | Hap18 | MF687981 | |
| ZYL4 | 20170422 | 广西防城港 | Hap29 | MF688019 | Hap18 | MF687981 | |
| ZYL5 | 20170422 | 广西防城港 | Hap30 | MF688020 | Hap18 | MF687981 | |
| ZYL6 | 20170422 | 广西防城港 | Hap30 | MF688020 | Hap19 | MF687982 | |
| 奥莱彩螺 Clithon oualaniense |
ALCL1 | 20080401 | 广西北海 | Hap31 | MF687983 | Hap20 | MF687956 |
| ALCL2 | 20080401 | 广西北海 | Hap31 | MF687983 | Hap21 | MF687957 | |
| ALCL3 | 20170418 | 广西北海 | Hap31 | MF687983 | Hap21 | MF687957 | |
| ALCL4 | 20170418 | 广西北海 | Hap31 | MF687983 | Hap21 | MF687957 | |
| ALCL5 | 20080401 | 广西北海 | Hap32 | MF687984 | Hap21 | MF687957 | |
| ALCL6 | 20170418 | 广西北海 | Hap33 | MF687985 | Hap21 | MF687957 | |
| ALCL7 | 20170418 | 广西北海 | Hap34 | MF687986 | Hap21 | MF687957 | |
| ALCL8 | 20080401 | 广西北海 | Hap35 | MF687987 | Hap22 | MF687958 | |
| ALCL9 | 20080401 | 广西北海 | Hap35 | MF687987 | Hap23 | MF687959 | |
| ALCL10 | 20080401 | 广西北海 | Hap36 | MF687988 | Hap24 | MF687960 | |
| 多色彩螺 Clithon sowerbianum |
DSCL1 | 20170418 | 广西北海 | Hap37 | MF687989 | Hap25 | MF687961 |
| DSCL2 | 20170418 | 广西北海 | Hap37 | MF687989 | Hap26 | MF687962 | |
| DSCL3 | 20170418 | 广西北海 | Hap38 | MF687990 | Hap27 | MF687963 |
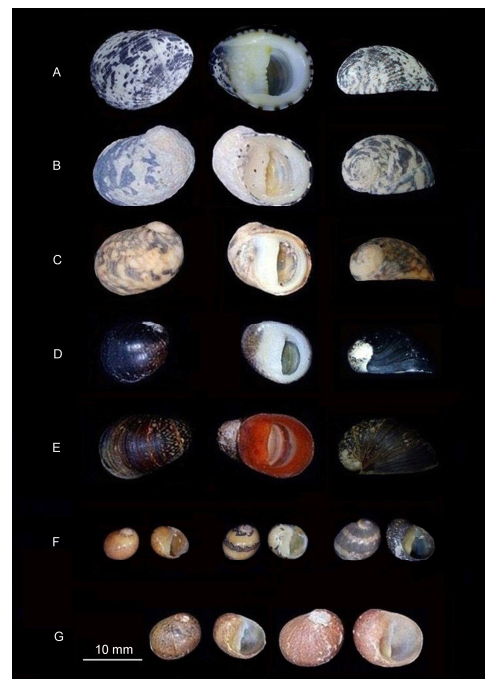 |
| 图 1 本研究中所使用的7个蜑螺科物种 Fig. 1 7 species of Neritidae in this study 注: A.渔舟蜑螺Nerita albicilla Linnaeus, 1758; B.齿纹蜑螺Nerita yoldii Récluz, 1841; C.日本蜑螺Nerita japonica Dunker, 1860; D.黑玉蜑螺Nerita incerta von dem Busch, 1844; E.紫游螺Neripteron violaceum (Gmelin, 1791); F.奥莱彩螺Clithon oualaniense (Lesson, 1831); G.多色彩螺Clithon sowerbianum (Récluz, 1843) |
采用CTAB法对样品DNA进行抽提(Winnepenninckx et al, 1993)。目的片段分别采用无脊椎动物COI基因片段的通用引物序列LCO1490/HCO2198 (Folmer et al, 1994)和16S rRNA基因片段的通用引物序列16Sar/16Sbr (Palumbi, 1996)进行扩增。COI通用引物对大部分样品并不适用, 对于那些使用COI通用引物无法成功扩增的样品, 本研究自行设计了特异性引物, 而所有样品的16S rRNA基因均能够被成功扩增, 引物信息如表 2所示。
| 物种 | 缩写 | 引物名称 | 序列(5′—3′) | 引物长度 | 引物来源 |
| 齿纹蜑螺 | N. yoldii | LCO1490 | GGTCAACAAATCATAAAGATATTGG | 25 | Folmer et al, 1994 |
| 多色彩螺 | C. sowerbianum | ||||
| 紫游螺 | N. violaceum | HCO2198 | TAAACTTCAGGGTGACCAAAAAATCA | 26 | Folmer et al, 1994 |
| 奥莱彩螺 | C. oualaniense | ||||
| 渔舟蜑螺 | N. albicilla | COIF-YZ | GCCCTAAGTCTTTTGATTCG | 20 | 本研究 |
| COIR-YZ | GCAAGAACAGGCAGAGAAAGT | 21 | |||
| 日本蜑螺 | N. japonica | COIF-RB | CTGCTTTGAGTCTTTTGATTCG | 23 | |
| 黑玉蜑螺 | N. incerta | COIR-RB | ATAACATTGTAATCGCACCTG | 21 | |
| 奥莱彩螺 | C. oualaniense | COIF-AL | GGTTGGTAGGAACTGCTCTTAG | 22 | |
| COIR-AL | GATCATACAAATAATGGAAGACG | 23 | |||
| 以上所有 | 16Sar | CGCCTGTTTATCAAAAACAT | 20 | Palumbi, 1996 | |
| 16Sbr | CCGGTCTGAACTCAGATCACGT | 22 | Palumbi, 1996 |
引物均由上海生工生物工程技术服务有限公司合成。扩增采用50μL反应体系, 含10×PCR Buffer (Mg2+ Plus), 2.5mmol/L dNTP, 10μmol/L正反引物, 5U/μL Taq DNA聚合酶(Takara), DNA模板约100ng。PCR反应参数为: 94℃预变性3min; 35次循环, 每次循环包括: 94℃变性30s, 48℃ (COI)/52℃ (16S rDNA)退火30s, 72℃延伸1min; 最后72℃延伸10min, 4℃保存。PCR产物经1.5%的琼脂糖电泳检测后进行纯化测序。纯化与测序由华大基因科技股份有限公司完成。
1.3 数据分析所得序列经SeqMan软件(DNASTAR®软件包)处理, 并结合人工校对, 确定每一位点准确无误, 最后将序列提交至GenBank数据库。使用BioEdit v.7.2.6.1 (Hall, 1999)软件中内置的Clustal W多序列比对程序对COI和16S rRNA基因序列分别进行比对, 并去掉正反向引物序列。用DnaSp 5.0 (Librado et al, 2009)软件计算样品的单倍型数(H)、平均核苷酸差异(K)及核苷酸多样性指数(π)。采用MEGA 6.0 (Tamura et al, 2013)软件分析序列碱基组成, 计算遗传距离, 应用邻接距离法(NJ, Neighbor-Joining)构建系统发育树, 选用K2P替代模型(Kimura-2-parameter, K2P)(Kimura, 1980), 进行1000次自检。由于NJ树不能真实反映物种的进化历史, 我们使用61个个体的COI和16S rRNA基因的拼接序列构建了最大似然树(Maximum Likelihood, ML)。ML树利用IQ-TREE (Nguyen et al, 2015)构建, COI和16S rRNA基因分区对应的模型均为GTR+ Γ, 通过1000次重复分析自检支持率(Bootstrap)。NJ树和ML树选取马蹄螺(Trochus maculatus, COI: GenBank, AB505301.1; 16S: Genbank, AB505344.1)作为外类群。
2 结果与分析 2.1 序列特征经PCR扩增、测序, COI和16S rRNA基因序列分别得到61条, 共122条序列。双向测序获得的COI基因片段长度在377—715bp之间, 16S rRNA基因片段长度在498—555bp之间。对剪切比对之后的同源序列进行分析, COI基因A、T、C、G平均含量分别为20.98%、40.80%、16.21%、22.02%, T含量明显高于其他三种碱基, G+C(38.23%)平均含量明显低于A+T(61.77%)含量。16S rRNA基因A、T、C、G平均含量分别为32.56%、29.38%、18.82%、19.25%, 其中A含量的总体水平最高, G+C(38.07%)平均含量同样明显低于A+T(61.93%)含量。碱基均出现明显的偏倚性, 符合线粒体碱基组成的特点, 这与帘蛤目(陈军等, 2010)、河蓝蛤(孙超等, 2014)等其他软体动物研究中的结果是相一致的。
获得的蜑螺科贝类序列分别定义了38个COI基因单倍型和27个16S rRNA基因单倍型(表 1), 不同种之间未出现共享单倍型。其中COI基因片段的平均核苷酸差异数为39.293, 核苷酸多样性指数为0.10736; 16S rRNA基因片段的平均核苷酸差异数为36.667, 核苷酸多态性指数为0.07687。COI基因片段的715个位点中, 保守位点数为476, 约占位点总数的66.6%;变异位点数为237个, 其中简约信息位点数为211个; 序列间所有位点的转换/颠换比率为2.77; 16SrRNA基因片段的560个位点中, 保守位点数为436, 约占位点总数的77.9%;变异位点数为123个, 其中简约信息位点数为101个; 序列间所有位点的转换/颠换比率为2.70。COI和16S rRNA基因各序列之间的差异度与遗传距离均成线性关系, 且转换大于颠换, 碱基替换未达到饱和。
2.2 条形码间隙条形码间隙的研究主要依赖于种内遗传差异和种间遗传差异的比较, 理想的条形码标准基因应该在种内、种间遗传距离水平上存在明显的条形码间隙(barcoding gap), 并可有效区分物种。由表 3可知, 基于COI基因序列的种内个体间遗传距离为0.00%—1.29%, 平均为0.67%, 种内遗传距离最大值发生在奥莱彩螺内; 属内种间遗传距离为4.62%—19.25%, 平均为13.02%, 种间最小遗传距离发生在齿纹蜑螺和日本蜑螺之间。基于16S rRNA基因序列的种内遗传距离为0.00%—0.48%, 平均为0.23%, 种内遗传距离最大值发生在奥莱彩螺内; 属内种间遗传距离为2.47%—8.48%, 平均为6.37%, 种间最小遗传距离发生在齿纹蜑螺和日本蜑螺之间(表 4)。两种基因序列在所研究蜑螺种类中, 种内遗传差异均小于种间遗传差异, 均存在明显的条形码间隙(barcoding gap), 但基于COI基因序列的DNA条形码间隙比基于16S rRNA基因序列的DNA条形码间隙显著。
| 物种 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | |
| 渔舟蜑螺 | N. albicilla | 0.72 | ||||||
| 齿纹蜑螺 | N. yoldii | 12.93 | 0.37 | |||||
| 日本蜑螺 | N. japonica | 16.71 | 4.62 | 0.00 | ||||
| 黑玉蜑螺 | N. incerta | 13.38 | 11.34 | 15.06 | - | |||
| 紫游螺 | N. violaceum | 19.06 | 15.32 | 16.11 | 19.48 | 0.52 | ||
| 奥莱彩螺 | C. oualaniense | 22.59 | 21.14 | 24.85 | 25.26 | 16.77 | 1.29 | |
| 多色彩螺 | C. sowerbianum | 22.24 | 17.35 | 20.08 | 21.14 | 13.94 | 19.25 | 1.13 |
| 物种 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | |
| 渔舟蜑螺 | N. albicilla | 0.42 | ||||||
| 齿纹蜑螺 | N. yoldii | 8.48 | 0.13 | |||||
| 日本蜑螺 | N. japonica | 8.08 | 2.47 | 0.00 | ||||
| 黑玉蜑螺 | N. incerta | 7.78 | 7.44 | 7.59 | - | |||
| 紫游螺 | N. violaceum | 15.40 | 17.02 | 17.28 | 16.29 | 0.07 | ||
| 奥莱彩螺 | C. oualaniense | 15.25 | 15.73 | 16.59 | 16.09 | 6.17 | 0.48 | |
| 多色彩螺 | C. sowerbianum | 15.62 | 17.86 | 18.12 | 17.11 | 4.11 | 4.55 | 0.28 |
基于COI和16S rRNA基因片段构建的邻接树(NJ树)见图 2和图 3。NJ树显示两种基因片段的聚类结果具有一致性, 且7种蜑螺均表现为单系群, 并且具有很高的支持度。基于COI和16S rRNA基因拼接序列构建的最大似然树(ML树)见图 4。7种蜑螺同样以较高的支持度形成单系群。
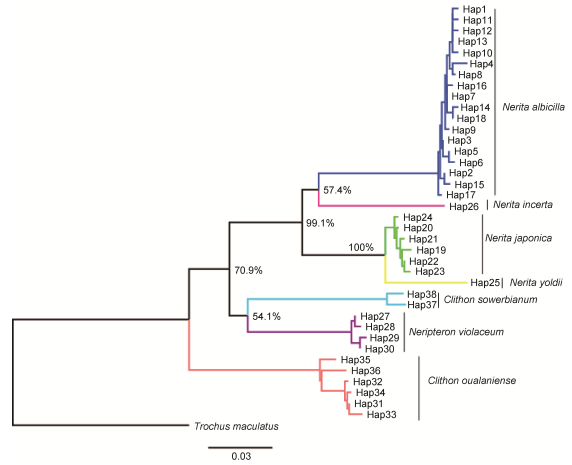 |
| 图 2 基于蜑螺COI基因片段的NJ树 Fig. 2 NJ tree of Neritidae species based on the partial COI gene 注:节点处数值表示Bootstrap 1000次重复抽样检测结果 |
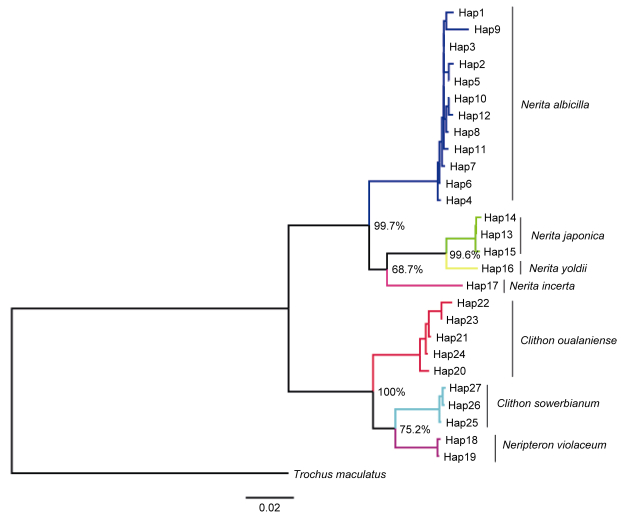 |
| 图 3 基于蜑螺16S rRNA基因片段的NJ树 Fig. 3 NJ tree of Neritidae species based on the partial 16SrRNA gene 注:节点处数值表示Bootstrap 1000次重复抽样检测结果 |
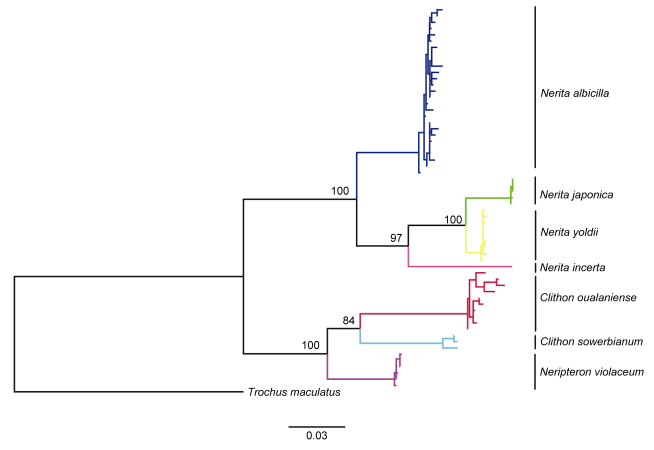 |
| 图 4 基于蜑螺COI和16S rRNA基因拼接序列的最大似然树(ML树) Fig. 4 Maximum likelihood tree of Neritidae species based on the concatenated COI and 16S rRNA gene 注:节点处数值为ML支持率 |
DNA条形码理论基于两个假设:一是种内遗传差异小于种间遗传差异, 即存在条形码间隙(barcoding gap); 二是在系统发生上种与种之间互为单系群(monophyletic group)(Hebert et al, 2004a)。对于条形码间隙, 过去研究通常使用Hebert等(2003)提出的“10倍法则”来对不同物种进行区分, 即种间遗传差异应达到种内遗传差异的10倍(李琪等, 2010; 陈军等, 2010)。本研究中, COI和16S rRNA两种基因序列在蜑螺科的7个物种中均存在明显条形码间隙, 同种个体间的遗传差异均明显小于属内不同物种之间的遗传差异, COI基因序列种间的遗传距离均比种内遗传距离高出10倍以上, 16S rRNA基因序列尽管不能完全符合“10倍法则”, 但也同样能够将本研究所涉及的蜑螺科贝类物种全部有效区分开。另外, 通过COI和16SrRNA基因构建的邻接树可以看出, 7个物种都以较高的支持度形成了单系群。因此, COI和16SrRNA两种基因序列都可以作为蜑螺科贝类DNA条形码标准基因用于物种鉴定。值得提及的是, 在对目标序列进行扩增时, 用于COI基因的Folmer等(1994)通用引物并不能对所有个体进行成功扩增, 而所有样品的16S rRNA基因使用Palumbi (1996)的通用引物均能够被成功扩增, 表明在开展蜑螺科物种的分子鉴定时, 使用16S rRNA基因序列可能更快速。
DNA条形码技术相对于传统的形态分类最大的优势在于, 可以很容易地鉴定缺少形态数据或因变异而导致形态上不易区分的种类(Hebert et al, 2004b)。蜑螺科同属的近缘种的贝壳形态、颜色十分相似, 如齿纹蜑螺N. yoldii和日本蜑螺N. japonica, 奥莱彩螺C. oualaniense和多色彩螺C. sowerbianum这两组同属的种类, 应用形态分类学很可能导致鉴定错误, 而本研究中应用COI和16S rRNA基因都可以将这两个组合准确区分。
基于COI和16S rRNA基因拼接序列的最大似然树显示, 蜑螺属Nerita 4个物种聚为一支(支持度100%), 这与Chee等(2016)、Fukumori等(2016)的研究结果相一致, 其中齿纹蜑螺N. yoldii和日本蜑螺N. japonica体现了很近的亲缘关系; 彩螺属Clithon和游螺属Neripteron所在的游螺亚科以100%的支持度形成单系, 与蜑螺属Nerita所在的蜑螺亚科Neritinae互为姊妹群。由于本研究物种数较少, 针对它们之间的亲缘关系和分类地位, 需要采集更多物种、应用多基因数据(如线粒体基因组和转录组)开展进一步分析。
4 结论本研究表明DNA条形码能够在种水平上将蜑螺科贝类进行区分, 这为应用DNA条形码来探讨我国沿海不同生境蜑螺科的物种多样性奠定了重要基础。未来可以通过强化标本采集, 结合传统形态学分类与DNA条形码等手段, 进一步明确中国沿海蜑螺科种属组成和区系特点。
| 孙超, 刘志鸿, 杨爱国, 等, 2014. 4种河蓝蛤线粒体COI和16S rRNA基因序列的种间遗传分析. 渔业科学进展, 35(1): 82–90 DOI:10.11758/yykxjz.20140112 |
| 李琪, 邹山梅, 郑小东, 等, 2010. DNA条形码及其在海洋生物中的应用. 中国海洋大学学报(自然科学版), 40(8): 43–47 |
| 张素萍, 2008. 中国海洋贝类图鉴. 北京: 海洋出版社, 46 |
| 陈军, 李琪, 孔令锋, 等, 2010. 基于COI序列的DNA条形码在中国沿海缀锦蛤亚科贝类中的应用分析. 动物学研究, 31(4): 345–352 |
| 陈志云, 连喜平, 谭烨辉, 等, 2016. 蜑螺科软体动物系统分类学研究进展. 海洋科学, 40(8): 168–173 |
| Azmir I A, Esa Y, Amin S M N, et al, 2017. Identification of larval fish in mangrove areas of Peninsular Malaysia using morphology and DNA barcoding methods. Journal of Applied Ichthyology, 33(5): 998–1006 DOI:10.1111/jai.2017.33.issue-5 |
| Beesley P L, Ross G J B, Wells A, 1998. Mollusca: The Southern Synthesis. Melbourne: CSIRO Publishing, 693-702 |
| Bouchet P, Rocroi J P, 2005. Classification and nomenclator of gastropod families. Malacologia, 47(1-2): 1–397 |
| Chee S Y, 2015. Limitations of cytochrome oxidase Ⅰ for the barcoding of Neritidae (Mollusca:Gastropoda) as revealed by Bayesian analysis. Genetics & Molecular Research, 14(2): 5677–5684 |
| Chee S Y, Mohd Nor S A, 2016. DNA barcoding reveals neritid diversity (Mollusca:Gastropoda) diversity in Malaysian waters. Mitochondrial DNA Part A:DNA Mapping, Sequencing, and Analysis, 27(3): 2282–2284 |
| Dekker H, 2000. The Neritidae (Gastropoda) from the circumarabian seas, with the description of two new species, a new subgenus and a new genus. Vita Marina, 47(2): 29–64 |
| Folmer O, Black M, Hoeh W, et al, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology, 3(5): 294–299 |
| Frey M A, 2008. A revised classification of the gastropod genus Nerita. The Veliger, 51(1): 1–7 |
| Fukumori H, Kano Y, 2014. Evolutionary ecology of settlement size in planktotrophic neritimorph gastropods. Marine Biology, 161(1): 213–227 DOI:10.1007/s00227-013-2330-5 |
| Fukumori H, Itoh H, Kano Y, 2016. The complete mitochondrial genome of the stream snail Clithon retropictus (Neritimorpha:Neritidae). Mitochondrial DNA Part B:Resources, 1(1): 820–821 DOI:10.1080/23802359.2016.1247659 |
| Galimberti A, De Mattia F, Losa A, et al, 2013. DNA barcoding as a new tool for food traceability. Food Research International, 50(1): 55–63 DOI:10.1016/j.foodres.2012.09.036 |
| Grüneberg H, 1976. Population studies on a polymorphic prosobranch snail (Clithon(Pictoneritina) oualaniensis Lesson). Philosophical Transactions of the Royal Society B:Biological Sciences, 275(940): 385–437 DOI:10.1098/rstb.1976.0088 |
| Grüneberg H, 1982. Pseudo-polymorphism in Clithon oualaniensis. Proceedings of the Royal Society B:Biological Sciences, 216(1203): 147–157 DOI:10.1098/rspb.1982.0067 |
| Hall T A, 1999. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 41: 95–98 |
| Hebert P D N, Cywinska A, Ball S L, et al, 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society B:Biological Sciences, 270(1512): 313–321 DOI:10.1098/rspb.2002.2218 |
| Hebert P D N, Stoeckle M Y, Zemlak T S, et al, 2004a. Identification of birds through DNA barcodes. PLoS Biology, 2(10): e312 DOI:10.1371/journal.pbio.0020312 |
| Hebert P D N, Penton E H, Burns J M, et al, 2004b. Ten species in one:DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator. Proceedings of the National Academy of Sciences of the United States of America, 101(41): 14812–14817 |
| Hofmann T, Knebelsberger T, Kloppmann M, et al, 2017. Egg identification of three economical important fish species using DNA barcoding in comparison to a morphological determination. Journal of Applied Ichthyology, 33(5): 925–932 DOI:10.1111/jai.2017.33.issue-5 |
| Jagt J W M, Kiel S, 2008. The operculum of Otostoma retzii (Nilsson.1827) (Gastropoda, Neritidae; Late Cretaceous) and its phylogenetic significance. Journal of Paleontology, 82(1): 201–205 DOI:10.1666/06-055.1 |
| Kano Y, Chiba S, Kase T, 2002. Major adaptive radiation in neritopsine gastropods estimated from 28S rRNA sequences and fossil records. Proceedings of the Royal Society B:Biological Sciences, 269(1508): 2457–2465 DOI:10.1098/rspb.2002.2178 |
| Kimura M, 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution, 16(2): 111–120 DOI:10.1007/BF01731581 |
| Librado P, Rozas J, 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451–1452 DOI:10.1093/bioinformatics/btp187 |
| Nguyen L T, Schmidt H A, Von Haeseler A, et al, 2015. IQ-TREE:a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution, 32(1): 268–274 DOI:10.1093/molbev/msu300 |
| Okutani T, 2017. Marine Mollusks in Japan. 2nd ed. Tokyo: Tokai University Press, |
| Padula V M, Causey D, López J A, 2017. Mitochondrial DNA phylogeography of least cisco Coregonus sardinella in Alaska. Journal of Fish Biology, 90(3): 1001–1020 DOI:10.1111/jfb.2017.90.issue-3 |
| Palumbi S R, 1996. Nucleic acids Ⅱ: the polymerase chain reaction. In: Hillis D M, Moritz C, Mable B K eds. Molecular Systematics. Sunderland: Sinauer Associates, 205-247 |
| Schütt H, Şeşen R, 1992. The genus Theodoxus in South-western Anatolia, Turkey (Gastropoda, Prosobranchia, Neritidae). Zoology in the Middle East, 6(1): 63–68 DOI:10.1080/09397140.1992.10637614 |
| Sheth B P, Thaker V S, 2017. DNA barcoding and traditional taxonomy:an integrated approach for biodiversity conservation. Genome, 60(7): 618–628 DOI:10.1139/gen-2015-0167 |
| Spencer H G, Waters J M, Eichhorst T E, 2007. Taxonomy and nomenclature of black nerites (Gastropoda: Neritimorpha: Nerita) from the South Pacific. Invertebrate Systematics, 21(3): 229–237 DOI:10.1071/IS06038 |
| Tamura K, Stecher G, Peterson D, et al, 2013. MEGA6:molecular Evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 30(12): 2725–2729 DOI:10.1093/molbev/mst197 |
| Tan S K, Clements R, 2008. Taxonomy and distribution of the neritidae (Mollusca:Gastropoda) in Singapore. Zoological Studies, 47(4): 481–494 |
| Thompson F G, 1980. Proserpinoid land snails and their relationships within the Archaeogastropoda. Malacologia, 20(1): 1–33 |
| Underwood A J, 1976. Food competition between age-classes in the intertidal neritacean Nerita atramentosa Reeve (Gastropoda:Prosobranchia). Journal of Experimental Marine Biology and Ecology, 23(2): 145–154 DOI:10.1016/0022-0981(76)90138-6 |
| Underwood A J, 2004. Landing on one's foot:small-scale topographic features of habitat and the dispersion of juvenile intertidal gastropods. Marine Ecology Progress Series, 268: 173–182 DOI:10.3354/meps268173 |
| Waters J M, King T M, O'Loughlin P M, et al, 2005. Phylogeographical disjunction in abundant high-dispersal littoral gastropods. Molecular Ecology, 14(9): 2789–2802 DOI:10.1111/mec.2005.14.issue-9 |
| Winnepenninckx B, Backeljau T, De Wachter R, 1993. Extraction of high molecular weight DNA from molluscs. Trends in Genetics, 9(12): 407 DOI:10.1016/0168-9525(93)90102-N |
| Woods A J C, Saul L R, 1986. New Neritidae from southwestern North America. Journal of Paleontology, 60(3): 636–655 DOI:10.1017/S0022336000022162 |
 2018, Vol. 49
2018, Vol. 49


