中国海洋湖沼学会主办。
文章信息
- 任晶莹, 王卫军, 徐涛, 赵强, 李彬, 孙国华, 杨建敏. 2018.
- REN Jing-Ying, WANG Wei-Jun, XU Tao, ZHAO Qiang, LI Bin, SUN Guo-Hua, YANG Jian-Min. 2018.
- 长牡蛎(Crassostrea gigas)多代近交与自交家系遗传差异及生长性状比较
- COMPARATION OF GENETIC DIVERSITY AND GROWTH OF PACIFIC OYSTER CRASSOSTREA GIGAS BETWEEN DIFFERENT INBREEDING SELECTION AND SELF FERTILIZATION SELECTION
- 海洋与湖沼, 49(3): 630-637
- Oceanologia et Limnologia Sinica, 49(3): 630-637.
- http://dx.doi.org/10.11693/hyhz20170900246
-
文章历史
- 收稿日期:2017-09-28
- 收修改稿日期:2018-01-05
2. 山东省海洋资源与环境研究院 烟台 264006;
3. 山东省渔业技术推广站 济南 250013;
4. 烟台市水产研究所 烟台 264000
2. Shandong Marine Resource and Environment Research Institute, Yantai 264006, China;
3. Fisheries Technology Extension Station of Shandong Province, Jinan 250013, China;
4. Yantai Fisheries Research Institute, Yantai 264000, China
长牡蛎(Crassostrea gigas)又称太平洋牡蛎, 属瓣鳃纲、珍珠贝目、牡蛎科, 自20世纪80年代从日本大规模引进养殖苗种后, 由于生长速度快、适应能力强、产量高等特点, 逐渐成为我国重要海水养殖品种(Miossec et al, 2009)。我国北方的长牡蛎苗种主要来源于人工育苗, 在进行家系选育时, 为了让优良性状尽快稳定地遗传, 常采用近交的方式建立纯系, 即有亲缘关系的个体进行交配, 但有研究(Longwell et al, 1973; Wada, 1986a, b)表明, 近交会不同程度上降低群体遗传多样性, 进而导致相关的繁殖和生长性状产生衰退。
自交为近交一种极端形式, 大多数牡蛎为雌雄异体, 少数为雌雄同体, 牡蛎绝大多数行异体受精, 但其自体受精即自交后代也可以正常发育和存活, 这就为本研究提供了可能性。国内外有部分研究者(Winkler et al, 2003; Zheng et al, 2012; 傅强, 2014)对扇贝近交、自交后代的数量性状和质量性状进行研究, 由于各研究遗传背景不同, 研究结果也不尽相同, 牡蛎受雌雄同体材料获得的限制, 涉及自交的研究相对较少。
微卫星(Simple Sequence Repeats, SSR)又称简单复序列, 由于其数量丰富、容易被检测, 可以区分物种的纯和度与杂合度且比较均匀地分布在基因组内等优点, 故常被用来进行遗传多样性的分析。本研究选择了8对引物, 对近交4代(F4代)家系、近交5代(F5代)家系和雌性同体自交1代(自交1代)家系进行遗传多样性分析, 比较F4代、F5代和自交1代的遗传多样性以及遗传结构, 以便更好地进行长牡蛎的新品种选育。
1 材料与方法 1.1 实验材料实验用长牡蛎(Crassostrea gigas)家系在莱州海益苗业有限公司进行。选育的快速生长性状近交4代(F4代)家系、近交5代(F5代)家系和雌性同体自交1代(自交1代)家系。其中F4代是经过连续四代兄妹交配获得的以生长性状为选育目标的近交系, F5代是以F4代为亲本, 其兄妹交配获得的近交系; 自交1代为在野生群体中发现的雌雄同体个体, 经自交获得; 野生对照组取崆峒岛海域的野生个体雌雄各5个, 取等量的卵子混合后, 与5个雄性个体的混合精子进行受精。各实验组和野生对照组于2014年6月2日同时构建。幼虫发育20天后, 出现眼点。眼点幼虫达总数1/3时, 投放栉孔扇贝壳作为附着基。室内附着10天后, 将附着好的附着基放入室外暂养池暂养30天后, 转移到崆峒岛海区进行稚贝的浮筏暂养。
60日龄时从F4代、F5代、自交1代和野生对照组中随机各选取30个牡蛎个体, 于95%酒精中固定, –20℃低温保存。生长性状指标分别在330日龄、510日龄和630日龄三个时间段测量, 三个家系和野生对照组分别取20—30个个体测其壳高、壳长、壳宽和湿重。
1.2 实验方法 1.2.1 DNA的提取及检测基因组DNA提取方法参照Li等(2006)的酚/氯仿抽提法, Thermo Scientific NanoDropTM OneC超微量紫外分光光度计检测DNA浓度和纯度。提取的模板DNA, –20℃贮存备用。
实验中所用的引物为本实验室已发表的论文(纪仁平等, 2015; 张荣良等, 2016), 核心序列及退火温度见表 1。PCR反应体系总体积为10μL, 包含模板DNA 1.0μL (50ng), 正反引物各1μL (10μmol/L), dNTP混合液1μL (2mol/L)、10×PCR Buffer 1μL、Taq酶0.05μL (5U/μL)、ddH2O 4.95μL。
| 微卫星位点 | 重复序列 | 引物序列(5′—3′) | 退火温度(℃) |
| ucd-Cg149 | (GA)n(GACA)N | F:TGATTAAACGTGGGTGATTCAG R:TTTCTGACTGTCCGTCTGTGA |
60 |
| ucd-Cg140 | (CT)N | F:TGCTCAATTCACAGCAATCAG R:TCTGACTGCTGAACAGCAAAAT |
60 |
| ucd-Cg112 | (TCA)n(TCG)N | F:TCAGTCATCTGAATCCTCATCC R:CTGCCGCAGATTTAGACAAA |
53 |
| ucdCg-151 | (GT)N | F:AGGTAATCCGCAAACCAGTG R:GCATTGCGTCAGGATTAGGT |
60 |
| ucdCg-181 | (GT)N(GA)n | F:CACCCCAAAGGACCACATAC R:TGTCAGCATGGGTAAGTCCA |
60 |
| ucdCg-170 | (GA)n(GT)n | F:TGGTGGTCAGTGAATGTGAGA R:CGGACAGTAGCCTTTTAACACA |
60 |
| ucdCg-109 | (CAT)n | F:GCTATGGTTGTCATCCTCGAA R:TGCCTTTATCGGTTTTGCTT |
53 |
| ucdCg-162 | (TTCA)N(ATCT)n(GTCT)N | F:CCAAATCACCGTTTTAGTTTGTT R:AGCGACACAGAGACCACCTT |
52 |
| 注:重复序列中的下标字母N表示连续的微卫星序列, n表示有间隔的微卫星序列 | |||
94℃预变性3min; 35个循环为: 94℃变性1min, 退火温度下退火30s, 72℃下延伸1min; 35个循环结束后, 继续72℃延伸5min, 最后4℃保存。
8%聚丙烯酰胺凝胶电泳和硝酸银染色检验PCR产物的多态性。用10bp DNA ladder (Invitrogen)作为对照(Marker), 检测等位基因位置, 避免不同凝胶之间条带位置的误差。
1.3 统计分析根据检验结果中每个个体的条带位置确定其基因型, 三个家系和野生对照组的各个微卫星位点的多态信息含量(polymorphism information content, PIC)用Cervus3.0进行计算, POPGENE Version1.32计算等位基因数(number of alleles, Na), 有效等位基因数(number of effective alleles, Ne), 观测杂合度(observed heterozygosity, Ho)和期望杂合度(expected heterozygosity, He), 使用GenePop软件计算P值(Raymond et al, 1995), 检验Hardy-Weinberg平衡和连锁不平衡情况, 结果用Bonferroni进行校正(Rice, 1989); 成体的生长数据由Spss16.0进行数据的差异性分析。
2 结果 2.1 4个长牡蛎实验组的微卫星遗传多样性对4个实验组的8个微卫星位点进行了分析, 野生对照组共获得98个等位基因。平均每个位点的等位基因数为12.25个, F4代、F5代分别获得16个等位基因, 平均每个位点获得的等位基因数为2个, 自交1代共获得16个等位基因, 平均每个位点获得的等位基因数为2个。野生对照组的平均期望杂合度和多态信息含量分别为0.859和0.8264, 而F5代的为0.441、0.357, 自交1代仅为0.397、0.297。4个实验组在8个位点上有16个群体的位点组合显著偏离HWE平衡(表 2)。具体数据和经校正的HWE平衡检验结果如表 2所示。总体而言, 野生群体遗传多样性最高, F4代次之, F5代和自交1代最低。
| 位点 | 野生对照 | F4代 | F5代 | 自交1代 | |
| ucd-Cg149 | Na | 17.000 | 2.000 | 2.000 | 2.000 |
| Ne | 11.392 | 1.763 | 1.966 | 1.946 | |
| Ho | 0.633 | 0.300 | 0.100 | 0.767 | |
| He | 0.928 | 0.440 | 0.525 | 0.494 | |
| PIC | 0.919 | 0.906 | 0.698 | 0.368 | |
| P | 0.603 | 0.000* | 0.000* | 0.001* | |
| ucd-Cg140 | Na | 15.000 | 2.000 | 2.000 | 2.000 |
| Ne | 9.183 | 1.867 | 1.965 | 1.991 | |
| Ho | 0.467 | 0.533 | 0.333 | 0.800 | |
| He | 0.906 | 0.472 | 0.599 | 0.506 | |
| PIC | 0.948 | 0.882 | 0.873 | 0.374 | |
| P | 0.491 | 0.312 | 0.008 | 0.001* | |
| ucd-Cg181 | Na | 8.000 | 2.000 | 3.000 | 3.000 |
| Ne | 3.390 | 1.105 | 1.873 | 2.15 | |
| Ho | 0.467 | 0.452 | 0.517 | 0.533 | |
| He | 0.717 | 0.667 | 0.568 | 0.733 | |
| PIC | 0.929 | 0.661 | 0.444 | 0.544 | |
| P | 0.069 | 0.001* | 0.141 | 0.137 | |
| ucd-Cg170 | Na | 7.000 | 2.000 | 1.000 | 2.000 |
| Ne | 4.306 | 1.986 | 1.000 | 1.180 | |
| Ho | 0.400 | 0.100 | 0.000 | 0.167 | |
| He | 0.781 | 0.096 | 0.000 | 0.155 | |
| PIC | 0.903 | 0.733 | 0.559 | 0.141 | |
| P | 0.002* | 0746 | 0.000* | 0.546 | |
| ucd-Cg162 | Na | 16.000 | 2.000 | 2.000 | 1.000 |
| Ne | 11.111 | 1.998 | 1.991 | 1.000 | |
| Ho | 0.267 | 0.300 | 0.400 | 0.000 | |
| He | 0.925 | 0.503 | 0.506 | 0.000 | |
| PIC | 0.948 | 0.903 | 0.551 | 0.000 | |
| P | 0.159 | 0.023 | 0.000* | 0.000* | |
| ucd-Cg151 | Na | 12.000 | 2.000 | 2.000 | 2.000 |
| Ne | 7.792 | 1.998 | 1.991 | 1.724 | |
| Ho | 0.933 | 0.900 | 0.933 | 0.600 | |
| He | 0.886 | 0.508 | 0.506 | 0.427 | |
| PIC | 0.956 | 0.859 | 0.659 | 0.332 | |
| P | 0.110 | 0.000* | 0.000* | 0.006* | |
| ucd-Cg109 | Na | 13.000 | 2.000 | 2.000 | 2.000 |
| Ne | 9.000 | 1.946 | 2.000 | 1.835 | |
| Ho | 0.567 | 0.500 | 0.472 | 0.433 | |
| He | 0.904 | 0.494 | 0.508 | 0.463 | |
| PIC | 0.932 | 0.879 | 0.542 | 0.351 | |
| P | 0.249 | 0.949 | 0.000* | 0.724 | |
| ucd-Cg112 | Na | 10.000 | 2.000 | 2.000 | 2.000 |
| Ne | 5.233 | 1.998 | 1.991 | 1.980 | |
| Ho | 0.567 | 0.967 | 0.933 | 0.900 | |
| He | 0.823 | 0.508 | 0.506 | 0.503 | |
| PIC | 0.929 | 0.784 | 0.375 | 0.372 | |
| P | 0.070 | 0.000* | 0.000* | 0.000* | |
| 平均值 | Na | 12.250 | 2.000 | 2.125 | 2.000 |
| Ne | 7.677 | 1.807 | 1.860 | 1.808 | |
| Ho | 0.538 | 0.533 | 0.530 | 0.449 | |
| He | 0.859 | 0.434 | 0.441 | 0.397 | |
| PIC | 0.826 | 0.363 | 0.357 | 0.297 | |
| 注:哈迪-温伯格平衡偏离水平: P<0.0062 | |||||
四个实验组的遗传距离和遗传相似性如表 3所示。从Nei遗传距离看, F4代、F5代和野生对照的遗传距离最大, 表明其亲缘关系较远。F4代和F5代遗传距离最小, 说明随着选育的进行, F4代和F5代的遗传结构逐步趋向稳定, 向一个方向进化。基于遗传距离采用UPGMA方法构建系统发育树(图 1), F4与F5首先聚到一支, 自交1代与野生聚到一支, 然后与F4代和F5代聚到一支。
| 组别 | F4代 | F5代 | 自交1代 | 野生对照 |
| F4代 | **** | |||
| F5代 | 0.0199 | **** | ||
| 自交1代 | 0.7350 | 0.7486 | **** | |
| 野生对照 | 0.8516 | 0.8602 | 0.6942 | **** |
 |
| 图 1 四个实验组的UPGMA聚类图 Fig. 1 UPGMA dendrogram of 4 groups |
三个家系和野生对照组的生长指标统计结果如表 4。经显著性检验, 总体而言, 三个家系各生长指标均高于野生对照组。三个家系间的壳长和壳宽没有显著性差异; 就壳高而言, 330日龄和630日龄时, F5代的壳高平均值分别为(55.5±8.4)mm、(84.5±4.7)mm, 明显高于F4代的(51.0±7.6)mm、(75.0±8.0)mm, 但与自交一代无显著性差异; 就湿重而言, 以630日龄时为例, F5代的平均值为(86.5±8.4)g, F4代和自交1代分别为(60.9±11.0)g、(60.1±15.5)g, F5代重量显著高于自交1代和F4代, 自交1代和F4代之间没有显著性差异, 510日龄与630日龄情况相同。
| 日龄 | 生长性状 | F4代 | F5代 | 自交1代 | 野生对照 |
| 330 | 壳高/mm | 51.0±7.6a | 55.5±8.4b | 51.4±7.3ab | 41.2±4.5c |
| 壳长/mm | 38.7±7.7a | 38.6±9.1a | 38.6±6.3a | 22.1±5.2b | |
| 壳宽/mm | 21.0±4.3a | 22.61±3.5a | 15.7±2.4b | 16.5±3.1b | |
| 湿重/g | 19.8±8.7a | 20.5±7.0a | 14.2±4.5b | 7.7±5.4c | |
| 510 | 壳高/mm | 71.5±10.3a | 73.3±16.2ab | 78.5±13.6b | 53.2±10.1c |
| 壳长/mm | 47.2±7.5a | 44.0±10.8a | 50.2±8.4a | 30.0±5.8b | |
| 壳宽/mm | 28.2±4.4a | 29.7±5.5a | 24.6±4.6a | 22.1±2.8b | |
| 湿重/g | 55.1±12.5a | 80.3±10.4b | 61.3±15.2a | 36.4±6.9c | |
| 630 | 壳高/mm | 75.0±8.0a | 84.5±4.7b | 81.1±10.3ab | 62.8±7.5c |
| 壳长/mm | 47.8±7.2ab | 44.8±10.4a | 49.8±7.4b | 38.9±4.7c | |
| 壳宽/mm | 29.0±4.1a | 31.6±7.0a | 26.6±4.5a | 25.7±3.1b | |
| 湿重/g | 60.9±11.0a | 86.5±8.4b | 60.1±15.5a | 49.6±6.7c | |
| 注:同一行中具有不同上标字母的代表差异显著 | |||||
各家系不同时间段各生长指标如图所示(图 2), 除了330日龄壳宽这一指标外, 野生对照组的所有生长指标都显著低于三个家系。三个家系壳长、壳宽两个指标在两年的生长过程中基本没有显著性差异, 壳高和湿重随着牡蛎的生长F5代明显高于F4代和自交1代。在一龄贝时期, 自交一代的壳高和湿重显著低于F5代和F4代, 随着牡蛎的生长, 二龄的自交1代牡蛎和F4代间基本没有了显著性差异。
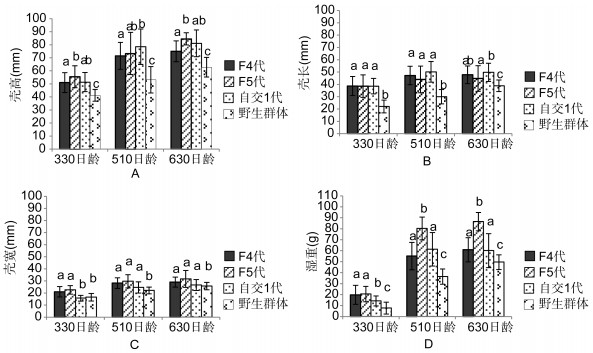 |
| 图 2 三个家系不同时间生长指标比较 Fig. 2 Average growth data of different point for different experimental groups 注: A.三个家系在三个时间点的壳高; B.三个家系在三个时间点的壳长; C.三个家系在三个时间点的壳宽; D.三个家系在三个时间点的湿重。所有数据按照平均值±标准差表示, 不同字母表示三个家系间有显著性差异 |
在良种选育过程中, 研究者们都希望优良性状基因能够快速纯化, 从而可以稳定地遗传。水产动物受性成熟年龄、每年繁育次数的限制, 这一过程一般要持续几年甚至十几年(王梅芳等, 2006)。在牙鲆等鱼类中, 雌核发育技术已渐趋成熟, 但贝类的人工雌核发育技术还处于研究阶段(潘英等, 2002), 因此对于贝类而言, 采用近交方式依然是提高基因纯和速度的有效方式, 且在牡蛎、贻贝、海湾扇贝、马氏珠母贝等品种的遗传改良上得到了应用(刘振等, 2013)。研究表明, 近交在使控制优良性状的基因纯合, 群体遗传机构稳定的同时, 也会使种群的遗传多样性下降, 而这会导致与生殖力或生理有关性状的基因表型平均值下降(张景晓等, 2014)。因此, 对近交和自交后代的遗传多样性进行检测, 了解其遗传多样性和遗传结构的变化可以更好地对实验室接下来的选育计划进行指导。
Barker(1994)在研究中提到, 评估遗传多样性时, 所用的微卫星标记至少应该有4个等位基因, 8对微卫星检测到的野生群体等位基因数目表明这8个微卫星能够较好地用于该研究。本研究中, F4代、F5代和自交1代家系与野生群体相比, 等位基因数、多态信息含量、期望杂合度明显下降。Appleyard等(2006)在对两个日本野生牡蛎组、塔斯马尼亚野生组和人工连续四代选育牡蛎组进行微卫星技术分析时发现, 野生组比人工选育组等位基因丰富度高13.9%—28%。韩智科(2011)对实验室的三疣梭子蟹选育家系F1到F4用16对微卫星分析发现, 从F1到F4, 遗传多样性指标逐渐下降。选育的过程其实就是对多样的基因库进行特定的选择, 基因多样性降低的同时, 与选育性状相关的基因会逐渐纯合, 有利于目标性状的稳定。
遗传杂合度是分析群体遗传多样性的重要指标, 王宇(2011)在海湾扇贝近交衰退的遗传分子机理的研究中利用10对微卫星分别分析了自交、近交和杂交家系, 发现近交和杂交家系杂合度较自交家系高, 这与本研究结果一致。在长牡蛎三代人工选育群体的微卫星分析研究中, 王庆志等(2012)发现, 经过连续3代的选育后, 与野生群体相比, 等位基因数和丰富度都有一定程度的降低, 但杂合度却并未明显降低。分析可能是因为在选择亲本繁殖和养殖的过程中, 未进行亲本的选择, 也就是未剔除生长发育不良的个体。而本研究根据选育目标, 在选择亲本建立家系的过程中, 会选择性腺发育丰满、个体较大的个体作为亲本, 这在一定程度上就淘汰了那些生长慢的小个体。同时, F5代170号引物位点的观测杂合度和期望杂合度都为1, 这表明, 这个位点在F5代已完全纯合。8个位点在F4代、F5代和自交1代中分别有4个、6个、5个位点偏离哈迪温伯格平衡, 分析原因可能是在人工选择的影响下, 与生长性状相关的基因逐渐纯和, 导致部分杂合子缺失; 也可能是样本量不够大, 在一定程度上影响了平衡。各个遗传多样性指标表明, 人工选育对选育系的遗传多样性和遗传结构产生了影响, 人工选择干预后, 选育群体遗传多样性明显降低, 并逐步趋向纯化, 同时, 其遗传结构逐步稳定。
3.2 生长性状在选育系遗传结构不断稳定的同时, 与适应性状相关基因的多样性也随着选育的不断进行逐渐降低。生长性状的数据表明, 总体而言, 三个家系各项生长指标都显著高于野生对照组, F5代成体生长的最好。其实, 雌雄异体的贝类研究近交衰退结果经常不一致, Beaumont等(1993)实验表明全同胞后代在壳的大小、肉的湿重和干重等方面都显著地小于杂交组, 而Lanna等(1980)研究发现长牡蛎全同胞家系幼虫的存活率并没有衰退。此外, 选择对近交起了作用, 日本珠母贝(Wada, 1986a, b)经过6代选择的近交系并没有出现近交衰退的现象, 而在美洲牡蛎家系研究中也出现了类似结果(Ford et al, 1987), 这表明连续多代近交会延缓或者遮盖部分近交衰退。本实验中, 在进行育种过程时, 通常会选择各生长指标良好的亲本, 这其实就是一种选择压力, 而这种选择, 在近交的过程中起到了筛选的作用, 从而使得F5代的壳高和湿重的生长性状明显好于F4代。而对自交一代而言, 壳长和壳宽两个生长指标三个家系之间基本没有显著性差异, 自交1代在壳高指标方面与F5代基本没有显著性差异, 在湿重方面自交一代的一龄贝显著低于F4代和F5代, 推测可能是近交所导致的衰退效应随着牡蛎的生长逐渐减弱或是因近交衰退生长不良的个体在自然选择的作用下被淘汰。于瑞海等(2011)研究了虾夷扇贝雌雄同体自体后代, 自交后代虽然成活率比对照组低, 但在生长上, 和对照组并无明显差别。这就为自体受精后代的选择育种提供了材料。实验室后期可以继续用获得的自交1代为实验材料, 进行其他相关研究。
4 结论本研究用来繁育的每一代亲本都是以体质优、生长快为指标, 从上一代中挑选出来的, 因此后代在生长方面表现出较好的水平。而在人工选择的干预下, 遗传多样性降会不可避免地降低, 同时, 与群体适应力相关的基因也会受到影响, 从而影响育种的潜力。从研究结果来看, 尽管F5代遗传多样性偏低, 但对成体牡蛎来说, 并没有出现明显的近交衰退, 所以在接下来的选育中, 可以继续保持一定的选育压力, 同时适当增加亲本数量, 避免由于遗传多样性引起的近交衰退。同时, 自交1代的后代遗传多样性降低程度相当于甚至低于F5代, 但成体牡蛎的生长形状并没有出现严重的衰退, Winkler等(2003)分析了紫扇贝(Argopecten purpuratus L.)自交后代和非自交后代幼虫的受精率和稚贝的生长情况, 发现自交后代受精率明显低于非自交后代, 但幼虫及稚贝的生长存活情况差异不大。这表明, 我们可以充分利用雌雄同体进行相关性状的品种选育, 从而缩短育种周期, 让育种促进生产发展。
| 于瑞海, 王昭萍, 赵雪琳, 等, 2011. 虾夷扇贝雌雄同体自体受精繁殖生物学的研究. 中国海洋大学学报, 41(11): 23–26 |
| 王宇, 2011. 海湾扇贝近交衰退的遗传分子机理的研究. 青岛: 中国科学院研究生院(海洋研究所)硕士学位论文, 24-29 |
| 王庆志, 李琪, 孔令锋, 2012. 长牡蛎3代人工选育群体的微卫星分析. 水产学报, 36(10): 1529–1536 |
| 王梅芳, 余祥勇, 刘永, 等, 2006. 马氏珠母贝雌雄同体和自体受精的研究. 水生生物学报, 30(4): 420–424 |
| 纪仁平, 李焕军, 冯艳微, 等, 2015. 长牡蛎(Crassostrea gigas)微卫星多重PCR体系构建及其在家系鉴定中的应用. 海洋与湖沼, 46(6): 1542–1548 |
| 张荣良, 王卫军, 冯艳微, 等, 2016. 长牡蛎(Crassostrea gigas)壳宽快速生长选育群体遗传多样性及遗传结构的微卫星标记分析. 渔业科学进展, 37(4): 90–96 DOI:10.11758/yykxjz.20150526004 |
| 张景晓, 李琪, 葛建龙, 2014. 近交对长牡蛎幼虫和稚贝生长与存活的影响. 水产学报, 38(12): 2005–2011 |
| 韩智科, 2011. 三疣梭子蟹(Portunus trituberculatus)近交家系遗传变异分析. 上海: 上海海洋大学硕士学位论文, 42-45 |
| 傅强, 2014. 虾夷扇贝(Patinopecten yessoensis)近交衰退的生物学效应及遗传机理研究. 青岛: 中国海洋大学博士学位论文, 45-46 |
| 潘英, 李琪, 王如才, 等, 2002. 海洋贝类雌核发育研究进展和展望. 水产学报, 26(5): 465–471 |
| Appleyard S A, Ward R D, 2006. Genetic diversity and effective population size in mass selection lines of Pacific oyster (Crassostrea gigas). Aquaculture, 254(1-4): 148–159 DOI:10.1016/j.aquaculture.2005.10.017 |
| Barker J S F, 1994. A global protocol for determining genetic distances among domestic livestock breeds. In: Proceedings of the 5th World Congress on Genetics Applied to Livestock Production. Guelph, Ontario, Canada: The Organizing Committee, 501-508 |
| Beaumont A R, Abdul-Matin A K M, Seed R, 1993. Early development, survival and growth in pure and hybrid larvae of Mytilus edulis and M. galloprovincialis. Journal of Molluscan Studies, 59(1): 120–123 |
| Ford S E, Haskin H H, 1987. Infection and mortality patterns in strains of oysters Crassostrea virginica selected for resistance to the parasiteHaplosporidium nelsoni (MSX). Journal of Parasitology, 73(2): 368–376 DOI:10.2307/3282092 |
| Lannan J E, Robinson A, Breese W P, 1980. Broodstock management of Crassostrea gigas:Ⅱ. Broodstock conditioning to maximize larval survival. Aquaculture, 21(4): 337–345 |
| Li G, Hubert S, Bucklin K, et al, 2003. Characterization of 79 mic; rosatellite DNA markers in the Pacific; oyster Crassostrea gigas. Molecular Ecology Notes, 3(2): 228–232 DOI:10.1046/j.1471-8286.2003.00406.x |
| Longwell A C, Stiles S S, 1973. Gamete cross incompatibility and inbreeding in the commercial American oyster, Crassostrea virginica Gmelin. Cytologia, 38(3): 521–533 DOI:10.1508/cytologia.38.521 |
| Miossec L, Le Deuff R M, Goulletquer P, 2009. Alien species alert:Crassostrea gigas (Pacific oyster). ICES Cooperative Research Report, 299: 1–42 |
| Raymond M, Rousset F, 1995. GENEPOP (Version1.2):Population genetic software for exact tests and ecumenicism. Journal of Heredity, 86(3): 248–249 DOI:10.1093/oxfordjournals.jhered.a111573 |
| Rice W R, 1989. Analyzing tables of statistical tests. Evolution, 43(1): 223–225 DOI:10.1111/evo.1989.43.issue-1 |
| Wada K T, 1986a. Genetic selection for shell traits in the Japanese pearl oyster, Pinctada fucata martensii. Aquaculture, 57(1-4): 171–176 DOI:10.1016/0044-8486(86)90194-8 |
| Wada K T, 1986b. Genetic variability at four polymorphic loci in Japanese pearl oysters, Pinctada fucata martensii, selected for six generations. Aquaculture, 59(2): 139–146 DOI:10.1016/0044-8486(86)90126-2 |
| Winkler F M, Estévez B F, 2003. Effects of self-fertilization on growth and survival of larvae and juveniles of the scallop Argopecten purpuratus L. Journal of Experimental Marine Biology and Ecology, 292(1): 93–102 DOI:10.1016/S0022-0981(03)00147-3 |
| Zheng H P, Li L, Zhang G F, 2012. Inbreeding depression for fitness-related traits and purging the genetic load in the hermaphroditic bay scallop Argopecten irradians irradians (Mollusca:Bivalvia). Aquaculture, 366: 27–33 |
 2018, Vol. 49
2018, Vol. 49


