
中国海洋湖沼学会主办。
文章信息
- 田儒品, 张海琪, 许晓军, 何中央, 丁雪燕, 苏胜齐. 2018.
- TIAN Ru-Pin, ZHANG Hai-Qi, XU Xiao-Jun, HE Zhong-Yang, DING Xue-Yan, SU Sheng-Qi. 2018.
- 中华鳖(Pelodiscus sinensis)MITF基因的生物信息学及表达差异研究
- BIOINFORMATICS AND EXPRESSION DIFFERENCES ANALYSIS OF MITF GENE IN PELODISCUS SINENSIS
- 海洋与湖沼, 49(3): 662-670
- Oceanologia et Limnologia Sinica, 49(3): 662-670.
- http://dx.doi.org/10.11693/hyhz20171100300
-
文章历史
- 收稿日期:2017-11-29
- 收修改稿日期:2018-01-23
2. 浙江省淡水水产研究所 湖州 313001;
3. 浙江省水产引种育种中心 杭州 310012
2. Zhejiang Institute of Freshwater Fisheries, Huzhou 313001, China;
3. Zhejiang Fisheries Introduction and Breeding Center, Hangzhou 310012, China
脊椎动物的皮肤和毛发颜色的形成涉及到黑色素细胞的分化和迁移、黑色素的合成和运输生物过程, 其黑色素合成是一个由酶催化的复杂过程。通过对脊椎动物黑色素细胞的发育、迁移、分化及黑色素的合成、转运的生物机制的研究有助于了解动物的肤色和毛色形成机制。小眼畸形相关转录因子(microphthalmia- associated transcription factor, MITF)是一种具有典型螺旋-环-螺旋-亮氨酸拉链结构的转录因子, 是黑色素细胞最重要的转录因子之一。目前有关MITF的报道主要集中在牛、羊、猪、马、鸡等许多物种, 参与生物体的生长、发育、分化和功能调节及色素沉积过程等各个方面(Gao et al, 1997; Opdecamp et al, 1997; Hallsson et al, 2004; Steingrímsson et al, 2004; Lang et al, 2005; Osawa et al, 2005; Wellbrock et al, 2005; 周泓燕, 2015)。其主要通过MC1R/ASIP/a-MSH和Wnt/β-Catenin及c-Kit/SCF及UV信号通路调控黑色素的合成(Lee et al, 2011; 张浩等, 2016)。近年来, 特异性表达于黑素细胞和黑素瘤细胞的MITF基因已成为研究热点, MITF蛋白不仅在NCC分化为黑素细胞过程中起到重要调控作用, 而且是调控酪氨酸酶(TYR)基因家族转录最关键的转录因子(Hou et al, 2008)。
中华鳖(Pelodiscus sinensis)作为我国重要的水产经济爬行动物, 2016年全国的养殖产量约35.4万吨, 产值逾250亿元, 已逐渐成为我国渔业品种结构优化、农民增收致富的一个重要品种。中华鳖没有明确的亚种分类单元, 因分布广泛导致形成一定的地理群体, 在外部形态上存在一定的差异(李思发等, 2004)。如中华鳖日本品系背部呈黄绿色, 腹部呈黄白色, 中心有一块较大的三角形黑色花斑, 四周有若干对称黑色花斑(何中央等, 2016)。清溪乌鳖则腹部呈乌黑或灰黑色, 且能稳定遗传, 是中华鳖遗传育种研究的良好材料(张永正等, 2008; Zhang et al, 2015a, b, 2017; Xu et al, 2016)。中华鳖“浙新花鳖”较日本品系的腹部黑色花斑更大、黑色斑点更多。中华鳖腹部体色变异复杂, 如能明确其变异分子机制, 可以通过遗传育种手段定向改变中华鳖腹部体色, 形成特定品种特征, 提高杂交品种/群体的表型一致性, 满足市场对高品质商品鳖的需求。但目前尚未见关于中华鳖MITF的相关文献报道。本研究通过构建中华鳖cDNA文库和转录组测序, 获得中华鳖MITF基因序列, 并进行生物信息学分析及组织表达差异研究, 为进一步研究中华鳖MITF基因表达的分子机理奠定基础。
1 材料与方法 1.1 实验材料本实验所用中华鳖(Pelodiscus sinensis)来源于浙江清溪鳖业股份有限公司, 包括中华鳖日本品系和清溪乌鳖及其正交种中华鳖“浙新花鳖”, 数量各10只, 2龄, 体质量为140—400g。分别取心脏、肝脏、脾脏、肺、肾、肌肉、眼、皮肤等8种组织, 液氮冷冻保存, 用于后续分析。
1.2 实验方法 1.2.1 总RNA的提取按照RNAiso Plus (TaKaRa, Japan)使用说明书提取总RNA。用1.2%琼脂糖凝胶电泳检测RNA的完整性和丰度, 并检测RNA的浓度及OD值。
1.2.2 cDNA文库构建及转录组测序采用Illumina公司的RNA Sample Preparation Kit进行中华鳖肝脏cDNA文库构建。取4μg RNA, 用带有Oligo(dT)的珠子分离纯化其中的mRNA, 然后将得到的mRNA处理成小片段。以mRNA小片段为模板, 用随机引物进行逆转录生成ds DNA, 末端补平后, 在3'末端加A, 然后连接接头, 用纯化试剂盒对产物进行2次纯化, 进行12个循环的PCR扩增, 扩增产物进行琼脂糖凝胶电泳, 用Mini Elute Gel Extraction Kit (QIAGEN)切胶回收长度200—700bp的条带, 得到测序用双末端文库。用高通量测序平台Illumina GAⅡx进行DNA双末端测序, 获得原始序列数据。
1.2.3 MITF基因的生物信息学分析获得RNA- seq原始数据后, 使用Tophat v2.0.5与中华鳖基因组数据库参考基因组进行比对, 筛选出中华鳖MITF基因序列信息。使用NCBI (https://blast.ncbi.nlm.nih. gov/Blast.cgi) Blast进行同源序列搜索; 蛋白质相似性搜索使用Blastx程序(https://blast.ncbi.nlm.nih.gov/ Blast.cgi); 用(http://www.bioinformatics.org/sms/orf_find.html)分析开放阅读框并推导氨基酸序列; 用String软件(https://string-db.org/cgi/Network.pl)预测了与中华鳖Mitf相互作用的蛋白质; 用Protscale软件(http://web.expasy.org/protscale/)预测中华鳖MITF蛋白疏水性; 使用SOPMA (http://Npsa-pbil.ibcp.frlcgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线分析软件, 预测MITF基因蛋白质的二级结构; 使用SWISS-MODEL (http://swissmodel, Expasy.org/)服务器进行三级结构在线预测; 使用DNAMAN软件进行氨基酸序列比对; 用MAGE6.0软件构建NJ系统进化树。
1.2.4 组织表达荧光定量PCR分析根据中华鳖MITF基因序列, 参照GenBank中中华鳖的近缘物种绿海龟(Chelonia mydas)、西部锦龟(Chrysemys picta bellii)等的序列, 用Primer Premier 5.0设计引物(表 1), 同时选择β-actin基因为内参基因, 对MITF基因表达水平进行均一化, 引物由上海生工生物工程有限公司合成。对8种组织进行MITF基因表达分析, 反应体系共20µL, 其中cDNA 2µL, 上下游引物各2µL, THUNDERBIRD SYBR qPCR Mix 10μL, 无RNA酶水4μL。在LightCycler® 96上进行定量扩增, 使用试剂盒, 反应参数:预变性95℃ 600s;扩增95℃ 10s, 56℃ 15s, 72℃ 15s;共45个循环, 溶解95℃ 10s, 65℃ 60s, 97℃ 10s。PCR扩增完成后对产物进行溶解曲线分析, 2–△△CT方法进行荧光定量的数据分析(Livak et al, 2001), 用SPSS16.0进行单因素方差分析(ANOVA)和Dunnect方法多重比较。
| 引物名称 | 引物序列 | 用途 |
| F | CAGTCAGACGCCAGCCATCAAC | qRT-PCR |
| R | AGCCATTGGACTGTTGGGTGC | |
| β-actin F | GAGACCTGACAGACTACCT | qRT-PCR |
| β-actin R | AGGATGATGAAGCAGCAGCAGT |
提取的中华鳖总RNA经1.5%琼脂糖凝胶电泳检测, 结果见图 1, 电泳结果表明, 获得的中华鳖RNA总纯度较高, 无DNA污染, 其28S和18S清晰可见, 28S : 18S接近2 : 1。紫外分光光度检测显示, A280/A260值为1.8—2.0, 反转录前浓度调整浓度至1000ng/µL, 符合后续实验。
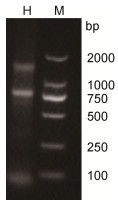 |
| 图 1 中华鳖RNA电泳图 Fig. 1 RNA of P. sinensis 注: M为DL2000 |
利用中华鳖日本品系、清溪乌鳖及中华鳖“浙新花鳖”肝脏组织的RNA, 分别构建了3个转录组的cDNA文库。经转录组测序后, 三个文库分别产生了53143118、56219804和58720302个raw reads, 分别发现7282026、7942102和8565638个reads, 得到高质量的clean数据共14.43Gb用于比对分析, 结果见表 2和表 3。本研究对三种中华鳖转录组数据中Unigenes色素相关的基因的表达量进行分析, 结果显示在清溪乌鳖和中华鳖“浙新花鳖”黑色素表达相达量Top10的基因中, 绝大部分富集于酪氨酸代谢通路和黑色素合成通路, 说明这些基因可能都参与了中华鳖黑色皮肤细胞活动和黑色素形成。同时, 这些基因中表达上调较高的为酪氨酸酶相关基因、小眼畸形相关转录因子基因、溶酶体转运调节蛋白基因等基因。
| 样品 | 原始序列 | 原始序列碱基数 | 有效序列 | 有效序列碱基数 | 有效碱基比例 |
| 中华鳖日本品系 | 53143118 | 5367454918 | 45861092 | 4631970292 | 86.3% |
| 清溪乌鳖 | 56219804 | 5678200204 | 48277702 | 4876047902 | 85.90% |
| 中华鳖“浙新花鳖” | 58720302 | 5930750502 | 50154664 | 5065621064 | 85.4% |
| 样品 | 有效读数 | 有效碱基数 | 匹配读数 | 匹配率 | 仅匹配到一个位置的读数 | 单一匹配率 | 重复匹配率 | 误判率 |
| 中华鳖日本品系 | 45861092 | 4631970292 | 38193853 | 83.3% | 26641286 | 69.8% | 30.2% | 0.3% |
| 清溪乌鳖 | 48277702 | 4876047902 | 38654927 | 80.1% | 26173303 | 67.7% | 32.3% | 0.4% |
| 中华鳖“浙新花鳖” | 50154664 | 5065621064 | 40784302 | 81.3% | 28305678 | 69.4% | 30.6% | 0.4% |
转录组测序获得的中华鳖MITF基因序列信息, 翻译氨基酸信息见图 2。与西部锦龟Chrysemys picta bellii (XM_008179493.1)、绿海龟Chelonia mydas (XM_007061484.1)、美国短吻鳄Alligator mississippiensis (XM_019499760.1)、非洲爪蟾Xenopus laevis (NM_001100277.1)、人Homo sapiens (XM_017006444.1)等5个物种相比, 同源性分别为93%、93%、85%、58%和76%。
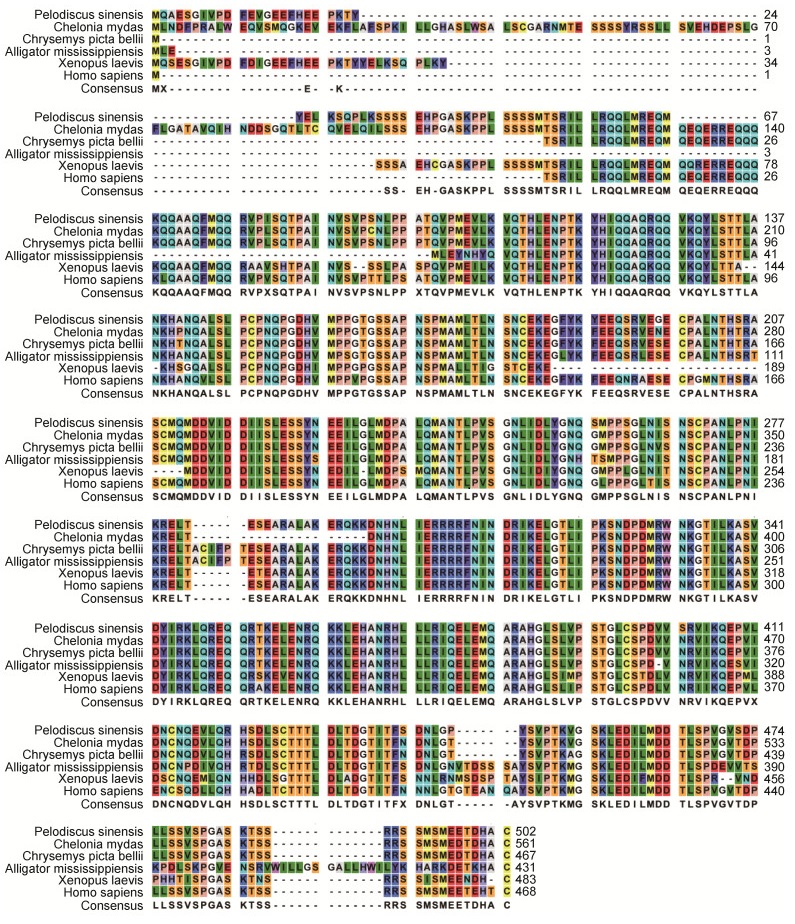 |
| 图 2 中华鳖与其他物种的MITF氨基酸序列比对 Fig. 2 Alignment of the deduced amino acid sequences of P. sinensis MITF and other vertebrates |
使用SOPMA在线分析软件, 预测中华鳖MITF的二级结构, 如图 3。结果显示: α-螺旋(Alpha helix)占25.37%, 延伸链(Extended strand)占20.90%, β-转角(Beta-turn)占7.76%, 无规则卷曲(Random coil)占45.97%。使用SWISS-MODEL三级结构在线预测服务器预测中国鳖MITF蛋白质三级结构, 如图 4。如图所示此基因有明显的α-螺旋(Alpha helix)结构, 这可能就是中国鳖MITF基因的功能位点。
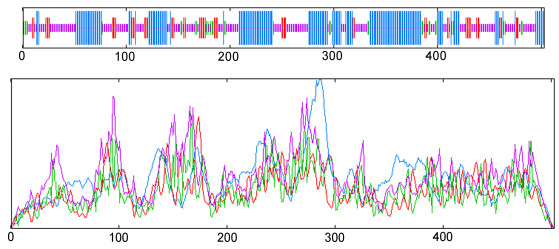 |
| 图 3 中华鳖Mitf蛋白二级结构预测 Fig. 3 The predicted protein secondary structure of P. sinensis MITF gene |
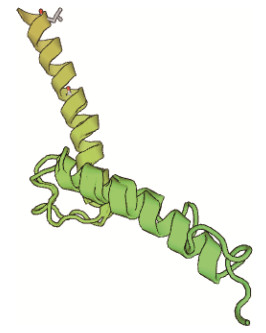 |
| 图 4 中华鳖Mitf蛋白三级结构模式图 Fig. 4 The tertiary structure ideograph of P. sinensis MITF gene protein |
利用String软件预测了与中华鳖MITF相互作用的蛋白质, 搜索条件限制在11个蛋白质, 其中与有丝分裂原激活蛋白激酶1(MAPK1)的关系最密切(图 5)。
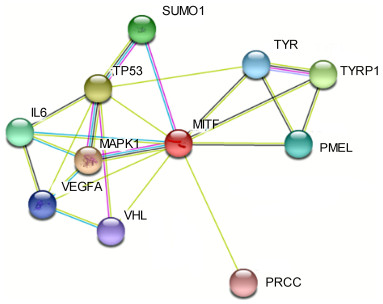 |
| 图 5 中华鳖MITF蛋白相互作用预测 Fig. 5 Predicted protein-protein interaction of the P. sinensis MITF |
根据Protscale软件预测中华鳖MITF蛋白亲/疏水性, 第49、50位氨基酸同处亲/疏水性分值最高点, 表明C端含有1个强的疏水区域; 位于第232位的分值最低, 表明N端含有1个强的亲水区域(图 6)。
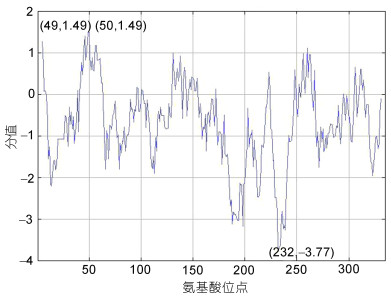 |
| 图 6 中华鳖MITF蛋白亲水性预测 Fig. 6 Predicted hydrophobicity profile of the P. sinensis MITF |
利用GenBank数据, 开展中华鳖等11种动物的MITF基因系统发育分析, 用MEGA6.0软件进行比对, 构建进化树见图 7。由图 7可见, 11个物种可明显分为四大类, 第一大类为龟鳖类, 西部锦龟Chrysemys picta bellii (XM 008179493.1)和绿海龟Chelonia mydas (XM 007061484.1)先聚为一小分支后与中华鳖聚成一类; 第二类为鳄类, 美国短吻鳄Alligator mississippiensis (XM 019499760.1)和印度鳄Gavialis gangeticus (XM 019504773.1)先聚为一小分支后与河口鳄Crocodylus porosus (XM 019536665.1)聚在一起; 第三类是鸵鸟类, 鹬鸵Apteryx australis mantelli (XM 013941846.1)和非洲鸵鸟Struthio camelus australis (XM_009670021.1)聚在一起; 第四类为高等鸟类, 白尾鹲Phaethon lepturus (XM 010285926.1)和猫头鹰Tyto alba (XM 009964751.1)先聚为一分支后再与帝企鹅Aptenodytes forsteri (XM 009284368.2)聚在一起。这种系统发育关系与传统的分类基本一致。由进化树可以看出, 中华鳖与西部锦龟、绿海龟MITF基因分子进化距离最近, 亲缘关系最密切。
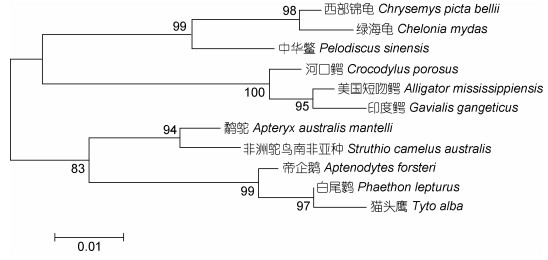 |
| 图 7 基于MITF基因构建的部分脊椎动物系统进化树 Fig. 7 The neighbor-joining tree of P. sinensis MITF |
MITF基因在清溪乌鳖不同组织间的相对表达量差异如表 4所示。实验发现, MITF在中华鳖肌肉中的mRNA表达量最高, 并显著高于其他组织, 其次是心脏, 同时心脏中mRNA的表达量显著性高于、脾、肺、肾、眼、皮肤等组织, 脾脏表达量最低。除心脏、肌肉外, 其他的组织间的表达差异性均不显著(P>0.05)。其按表达量从高到低的顺序, 依次为肌肉>心脏>皮肤>眼>肾>肝脏>肺>脾脏。
| 组织名 | 表达量 |
| 心脏 | 1.16±0.10b |
| 肝 | 0.24±0.05a |
| 脾 | 0.07±0.01a |
| 肺 | 0.14±0.01a |
| 肾 | 0.22±0.07a |
| 肌肉 | 3.54±0.30c |
| 眼 | 0.38±0.47a |
| 皮肤 | 0.24±0.05a |
形态特征是生物遗传变异最直观的表型之一, 为选育良种提供依据。水生动物的形态特征主要由遗传因子决定, 但同时又与其行为、食性和生活环境等有密切关系。通过外部表现型差异分析, 进一步从生化遗传、分子遗传分析亲本与子代遗传变异, 可为培育优良的新品种提供理论依据。本研究选择的3种中华鳖新品种, 在腹部体色上存在显著差异, 尤以清溪乌鳖的腹部全黑色最为独特。MITF基因是调控黑色素合成通路下游关键因子, TYR、TYRP-1、TYRP-2、Dct基因的启动子上存在可供MITF识别的M-box (GTCATGTGCT)或E-box (TCATGTG)调控元件, MITF通过bHLHZipd结构与酪氨酸家族基因启动子相结合, 并刺激酪氨酸家族基因的表达, 调控其活性, 促进黑色素的生成(Yasumoto et al, 1994; Bertolotto et al, 1998; Carreira et al, 2005)。目前对羊、鸡等经济动物的MITF等研究较多(Hou et al, 2008), 对水生动物的研究还相对较少。Zhao等(2012a)研究发现, 与普通海参相比, 白化海参体表中MITF表达水平显著降低, 表皮黑素细胞数量减少, 黑色素含量也较少。在白化海参的后代中, MITF表达水平在受精后32d显著降低, 表皮黑素细胞数量减少, 发育不足。他们认为MITF的低表达可能会导致白化海参体表中黑素细胞数量减少和合成黑色素的能力减弱。本研究对三种中华鳖转录组数据中Unigenes色素相关的基因的表达量进行分析, 结果显示在清溪乌鳖和中华鳖“浙新花鳖”黑色素表达相达量Top10的基因中, 绝大部分富集于酪氨酸代谢通路和黑色素合成通路, 说明这些基因可能都参与了中华鳖黑色皮肤细胞活动和黑色素形成。同时, 这些基因中表达上调较高的为酪氨酸酶相关基因、小眼畸形相关转录因子基因、溶酶体转运调节蛋白基因等基因。清溪乌鳖和中华鳖“浙新花鳖”的cAMP的表达量高于中华鳖日本品系, 这预示着该通路在中华鳖黑色素合成的过程十分重要。
3.2 中华鳖MITF基因的组织表达差异性MITF基因在大多数细胞类型中表达, 并且在神经嵴衍生和神经上皮衍生的含黑色素的色素细胞中具有主要作用(Arnheiter, 2010)。Zhao等(2012b)证明了MITF在青鳉胚芽基因表达中扮演着转录激活因子的角色。Steingrímsson等(1994)验证了在杂合情况下, 一些隐性的MITF突变有时也会在小鼠腹部或尾部产生小的斑点。MITF基因表达存在显著的组织特异性, 如在绵羊毛色相关基因的研究中MITF基因的mRNA表达量为黑色皮肤组织显著高于棕黑色和白色(Han et al, 2015); 对白绒乌鸡的研究表明MITF基因在皮肤组织中表达量最高, 且具有组织特异性, 对白绒乌鸡体内黑色素沉积起正向调控作用, 其高表达可能有助于乌鸡体内黑色素的形成(郑嫩珠等, 2015)。本研究采用荧光定量PCR分析了清溪乌鳖8种组织中MITF基因的表达差异情况, 结果发现其存在显著的组织特异性, 其在肌肉中的表达量最高, 这也间接说明了其在黑色素形成中起着正向调节的作用, 其具体的作用机制有待于进一步深入研究。
4 结论本研究通过测序得到了中华鳖的小眼畸形相关转录因子(MITF)的全长序列, 并对其编码的长度为503个氨基酸的蛋白质进行生物信息学分析, 发现该蛋白具有明显的α-螺旋结构, 蛋白相互作用与有丝分裂原激活蛋白激酶1的关系最为密切, 进化分析表明中华鳖MITF基因与西部锦龟、绿海龟进化关系最近。组织特异性分析表明MITF基因在中华鳖肌肉组织中表达最高, 可能在黑色素形成中起重要的调控作用。
| 李思发, 蔡完其, 刘至治, 等, 2004. 中华鳖七群体体形和腹部黑斑图案的差异比较. 水产学报, 28(1): 15–22 |
| 何中央, 张海琪, 蔡引伟, 等, 2016. 中华鳖高效养殖致富技术与实例. 北京: 中国农业出版社, 28-35 |
| 张浩, 徐超, 宋兴超, 等, 2016. 黑色素细胞调控小眼畸形相关转录因子的研究进展. 畜牧与兽医, 48(8): 113–117 |
| 张永正, 张海琪, 何中央, 等, 2008. 中华鳖(Pelodiscus sinensis)5个不同地理种群细胞色素b基因序列变异及种群遗传结构分析. 海洋与湖沼, 39(3): 234–239 |
| 周泓燕, 2015. MITF基因对动物毛色中黑色素细胞的作用. 中国畜牧兽医文摘, 31(3): 46 |
| 郑嫩珠, 辛清武, 朱志明, 等, 2015. 白绒乌鸡MITF基因的cDNA克隆、表达及其对黑色素沉积的影响. 中国农业科学, 48(18): 3711–3718 DOI:10.3864/j.issn.0578-1752.2015.18.014 |
| Arnheiter H, 2010. The discovery of the microphthalmia locus and its gene, Mitf. Pigment Cell & Melanoma Research, 23(6): 729–735 |
| Bertolotto C, Abbe P, Hemesath T J, et al, 1998. Microphthalmia gene product as a signal transducer in cAMP-induced differentiation of melanocytes. The Journal of Cell Biology, 142(3): 827–835 DOI:10.1083/jcb.142.3.827 |
| Carreira S, Goodall J, Aksan I, et al, 2005. MITF cooperates with Rb1 and activates p21Cip1 expression to regulate cell cycle progression. Nature, 433(7027): 764–776 DOI:10.1038/nature03269 |
| Gao E W, Wang Y, Alcorn J L, et al, 1997. The basic helix-loop-helix-zipper transcription factor USF1 regulates expression of the surfactant protein-A gene. Journal of Biological Chemistry, 272(37): 23398–23406 DOI:10.1074/jbc.272.37.23398 |
| Hallsson J H, Haflidadóttir B S, Stivers C, et al, 2004. The basic helix-loop-helix leucine zipper transcription factor Mitf is conserved in Drosophila and functions in eye development. Genetics, 167(1): 233–241 DOI:10.1534/genetics.167.1.233 |
| Han J L, Yang M, Guo T T, et al, 2015. Molecular characterization of two candidate genes associated with Coat color in Tibetan sheep (Ovis arise). Journal of Integrative Agriculture, 14(7): 1390–1397 DOI:10.1016/S2095-3119(14)60928-X |
| Hou L, Pavan W J, 2008. Transcriptional and signaling regulation in neural crest stem cell-derived melanocyte development:do all roads lead to MITF?. Cell Research, 18(12): 1163–1176 DOI:10.1038/cr.2008.303 |
| Lang D, Lu M M, Huang L, et al, 2005. Pax3 functions at a nodal point in melanocyte stem cell differentiation. Nature, 433(7028): 884–887 DOI:10.1038/nature03292 |
| Lee H D, Lee W H, Roh E, et al, 2011. Manassantin A inhibits cAMP-induced melanin production by down-regulating the gene expressions of MITF and tyrosinase in melanocytes. Experimental Dermatology, 20(9): 761–763 DOI:10.1111/exd.2011.20.issue-9 |
| Livak K J, Schmittgen T D, 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-DDCT method. Methods, 25(4): 402–408 DOI:10.1006/meth.2001.1262 |
| Opdecamp K, Nakayama A, Nguyen M T, et al, 1997. Melanocyte development in vivo and in neural crest cell cultures:crucial dependence on the Mitf basic-helix-loop-helix-zipper transcription factor. Development, 124(12): 2377–2386 |
| Osawa M, Egawa G, Mak S S, et al, 2005. Molecular characterization of melanocyte stem cells in their niche. Development, 132(24): 5589–5599 DOI:10.1242/dev.02161 |
| Steingrímsson E, Copeland N G, Jenkins N A, 2004. Melanocytes and the microphthalmia transcription factor network. Annual Review of Genetics, 38: 365–411 DOI:10.1146/annurev.genet.38.072902.092717 |
| Steingrímsson E, Moore K J, Lamoreux M L, et al, 1994. Molecular basis of mouse microphthalmia (mi) mutations helps explain their developmental and phenotypic consequences. Nature Genetics, 8(3): 256–263 DOI:10.1038/ng1194-256 |
| Wellbrock C, Marais R, 2005. Elevated expression of MITF counteracts B-RAF-stimulated melanocyte and melanoma cell proliferation. The Journal of Cell Biology, 170(5): 703–708 DOI:10.1083/jcb.200505059 |
| Xu J H, Zhao J, Li Y Q, et al, 2016. Evaluation of differentially expressed immune-related genes in intestine of Pelodiscus sinensis after intragastric challenge with lipopolysaccharide based on transcriptome analysis. Fish & Shellfish Immunology, 56: 417–426 |
| Yasumoto K I, Yokoyama K, Shibata K, et al, 1994. Microphthalmia-associated transcription factor as a regulator for melanocyte-specific transcription of the human tyrosinase gene. Molecular and Cellular Biology, 14(12): 8058–8070 DOI:10.1128/MCB.14.12.8058 |
| Zhang C, Xu X J, Zhang H Q, et al, 2015a. PCR-RFLP identification of four Chinese soft-shelled turtle Pelodiscus sinensis strains using mitochondrial genes. Mitochondrial DNA, 26(4): 538–543 DOI:10.3109/19401736.2013.869674 |
| Zhang H Q, Xu X J, He Z Y, et al, 2017. De novo transcriptome analysis reveals insights into different mechanisms of growth and immunity in a Chinese soft-shelled turtle hybrid and the parental varieties. Gene, 605: 54–62 DOI:10.1016/j.gene.2016.12.003 |
| Zhang H Q, Zhang C, Xu X J, et al, 2015b. Differentiation of four strains of Chinese soft-shelled turtle (Pelodiscus sinensis) based on high-resolution melting analysis of single nucleotide polymorphism sites in mitochondrial DNA. Genetics and Molecular Research, 14(4): 13144–13150 DOI:10.4238/2015.October.26.10 |
| Zhao H B, Li M Y, Purwanti Y I, et al, 2012a. MITF is a transcriptional activator of medaka germ genes in culture. Biochimie, 94: 759–767 DOI:10.1016/j.biochi.2011.11.007 |
| Zhao H L, Yang H S, Zhao H, et al, 2012b. Differences in MITF gene expression and histology between albino and normal sea cucumbers (Apostichopus japonicus Selenka). Chinese Journal of Oceanology and Limnology, 30(1): 80–91 DOI:10.1007/s00343-012-1043-9 |
 2018, Vol. 49
2018, Vol. 49


