
中国海洋湖沼学会主办。
文章信息
- 黄天晴, 徐革锋, 谷伟, 王炳谦, 张玉勇, 郑先虎, 姚作春, 赵成, 鲁翠云. 2018.
- HUANG Tian-Qing, XU Ge-Feng, GU Wei, WANG Bing-Qian, ZHANG Yu-Yong, ZHENG Xian-Hu, YAO Zuo-Chun, ZHAO Cheng, LU Cui-Yun. 2018.
- 用微卫星分析细鳞鲑(Brachymystax lenok)连续3代选育群体的遗传结构
- GENETIC STRUCTURE OF THREE CONSECUTIVE BREEDING GENERATIONS OF BRACHYMYSTAX LENOK REVEALED BY MICROSATELLITE MARKERS
- 海洋与湖沼, 49(4): 858-865
- Oceanologia et Limnologia Sinica, 49(4): 858-865.
- http://dx.doi.org/10.11693/hyhz20180100022
-
文章历史
- 收稿日期:2018-01-30
- 收修改稿日期:2018-02-23
细鳞鲑(Brachymystax lenok)隶属于鲑形目(Salmoniformes), 鲑科(Salmonidae), 细鳞鲑属, 在我国主要分布于东北部的黑龙江、鸭绿江、图门江、绥芬河等流域(李思忠, 1984)。细鳞鲑肉质细嫩鲜美, 必需氨基酸含量远高于鲤(Cyprinus carpio L.)、鲫(Carassius auratus)等淡水鱼类(徐革锋等, 2010), 具有较高的营养和经济价值。为了保护和利用细鳞鲑资源, 中国水产科学研究院黑龙江水产研究所从乌苏里江及牡丹江采捕细鳞鲑亲鱼, 于2003—2010年连续进行了人工繁殖及养殖试验, 掌握了细鳞鲑全人工繁育技术(牟振波等, 2013), 对细鳞鲑的繁殖生理(牟振波等, 2008; 徐革锋等, 2009a)、苗种的早期发育(李永发等, 2009; 徐革锋等, 2013)及生长生理(Liu et al, 2015; Xu et al, 2015; 徐革锋等, 2016)开展了一系列的研究, 实现了细鳞鲑规模化苗种繁育及养殖, 在此基础上开展了细鳞鲑良种的选育, 目前已经培育至F3, 使其逐步成为广泛养殖的名特优品种之一。
相对而言, 细鳞鲑遗传方面的研究开展较少, 徐革锋等(2009b)分析了乌苏里江、鸭绿江和牡丹江细鳞鲑的染色体核型; 王荻等(2009)用AFLP标记的分析结果表明牡丹江细鳞鲑群体的多态性比例最高(67.80%), 鸭绿江群体次之(55.81%), 而乌苏里江群体最低(33.39%); 夏颖哲等(2006)和张艳萍等(2014)分别用线粒体D-loop区序列分析了细鳞鲑及秦岭细鳞鲑(Brachymystax lenok tsinlingensis)的遗传结构, 线粒体全基因组序列的比对结果显示两者98.3%的核苷酸序列相同(Si et al, 2012)。近年来, 微卫星标记(microsatellite)因其多态性高、呈现共显性遗传等特点成为研究群体遗传分化的首选标记, 但细鳞鲑的微卫星标记资源相对有限(Liu et al, 2014; Yu et al, 2014), 制约了其在细鳞鲑种质鉴定、遗传保护方面的应用。本研究从遗传标记积累较多的近缘种虹鳟(Oncorhynchus mykiss)的微卫星标记中, 筛选了适用于细鳞鲑遗传分析的标记, 并分析了细鳞鲑连续3代选育群体的遗传结构及差异, 以期为细鳞鲑种质资源保护以及遗传选育提供物质基础。
1 材料与方法 1.1 实验材料细鳞鲑(Brachymystax lenok)群体样本均采自中国水产科学研究院黑龙江水产研究所丹东繁育基地。野生细鳞鲑于2002年采捕于乌苏里江虎头江段, 培育成熟后于2003—2010年实现了全人工繁殖(牟振波等, 2013), 并以2009年生产的苗种建立细鳞鲑F1选育基础群体, 2011年和2014年繁殖获得F2和F3选育基础群体。2015年4月, 随机采集每代选育群体32尾, 其中F1(7龄)平均体重为2415±765g, 平均体长为60.5±7.4cm; F2(4龄)平均体重为1018±380g, 平均体长45.6±7.7cm; F3(1龄)平均体重为139±30.6g, 平均体长为20.8±1.8cm。剪取部分鳍条组织用以提取基因组DNA(李超等, 2014)。
1.2 微卫星引物利用GenBank数据库中检索的虹鳟微卫星引物150对分析随机4个细鳞鲑样本, 检测标记的适用性、稳定性及多态性。64对引物在细鳞鲑中获得了稳定、清晰的目的条带, 38对引物在个体间表现出多态性, 最终选择22个微卫星标记检测了全部96个样本的基因型。引物信息及在虹鳟高密度遗传图谱(Rexroad Ⅲ et al, 2008)上的位置见表 1。
| 标记 | GenBank 注册号 |
正向引物序列(5'—3) | 反向引物序列(5'—3) | 重复序列 | 片段长度 (bp) |
等位基因数 (Na) |
虹鳟连锁群 |
| OMM1042 | BY722064 | GCCTGCCTCTCTGCTAGTAGTCA | TAGCCTACTCAGCAGTGAGAGGTATAG | (CTAT)21 | 127—238 | 18 | 12 |
| OMM1145 | AF375040 | CTCCTTCGGTGAAGTAAACCT | TTCGGTAAGTTCATGTCGC | (GA)13 | 134—158 | 8 | 9 |
| OMM1192 | AF469977 | GGGCTCATGGAAGCATTAACGC | TCGCATCCACAAAGGTAGGTAGGC | (GT)15 | 290—300 | 2 | 12 |
| OMM1231 | AF470011 | TCCACCTGCTCTGACCTCTACTCA | GCAGCCAGAGAACAGTAAGCATGT | (CTAT)11 | 232—279 | 12 | 1 |
| OMM1236 | AF470016 | GGACAGATTCACGGGTGTCT | ATCGGTTGTTAACTAGTGTGGC | (ATCT)18 | 240—324 | 26 | 7 |
| OMM1263 | AF470029 | CTGCATTCCAATACTCCACAG | TGGACGAACACTGGATCAG | (CA)29 | 261—284 | 2 | 3 |
| OMM1329 | G73564 | GGGAAGTGTTCACCATTACACAAG | CATCCAGGAACGCACCTTTA | (catc)7 | 126—240 | 9 | 8 |
| OMM1372 | BY005159 | CACTTCATGATGCCGAAAGCAG | CCCCCATCATGACTCCTTCTAGTT | (GA)11 | 217—234 | 4 | sex |
| OMM1727 | BY212207 | GGCCATCATCCATATCAG | GGAGGAGGGCTACAAGTC | (CAT)8 | 224—245 | 8 | 4 |
| OMM1762 | BY212231 | CCTCTAATTTCACTCGGATG | CCTTTGTCTGTCTTAAACCC | (AG)20 | 147—208 | 13 | 28 |
| OMM3042 | BY722029 | ATTCAAAGGGCCTCATGCAT | TGCATGTGTTCCCTTGGAAA | (CA)18 | 153—159 | 3 | 11 |
| OMM3048 | BY078106 | CACAAATGAAAAGGGCAGAG | GCCTCTAAAGCAAACCATCA | (CA)24 | 192—203 | 5 | 7 |
| OMM3065 | BV078105 | GCCCAAACCATGACAACAACT | GGTGTTCATGCTTTTGCAGACT | (GT)26 | 118—122 | 2 | 17 |
| OMM3080 | BY678008 | TCAGGGACATCATAGTAGGGA | TACCCACCCACAOCTGACTTAT | (gt)6 | 175—181 | 3 | 2 |
| OMM3081 | BY678009 | CCTCCCTTTCTCGACCTCC | CAGCTCTCTCGCGTTCTCC | (CT)l3 | 196—234 | 5 | 3 |
| OMM3126 | BY683039 | AGACAGCGAGGCTTACTACTG | GTCCTCGCTACTGGCATAA | (gt)18 | 263—296 | 9 | 17 |
| OMM5006 | CA347214 | AAGGCATCATTGGTGATAACAAGG | GGGACGATGCTTTGGCTAAGA | (ga)19 | 220—239 | 4 | 27 |
| OMM5044 | CA349174 | GCCCACATAAACACAGACGCA | GTCCGGTGGTTGAGCAGTACA | (CA)27 | 168—188 | 8 | 20 |
| OMM5106 | CA348902 | GGTATGATGCCTCTGAATGAACAGTAT | ACCAGTTGGTGTTTAACTCATATCAGC | (CA)16 | 271—355 | 20 | 19 |
| OMM5136 | BY211865 | CCAGCACTTTCTGTCTCATA | CTCCACAGGCCTTATTACTT | (AT)17 | 171—200 | 12 | sex |
| OMM5192 | BX317661 | CCGACGCAGGACATACATAC | GGGATGGTAAGGCAGTCAGA | (CA)31 | 312—331 | 5 | 14 |
| BX076085 | BX076085 | AAGACAGGAGATGAAGACACCG | ATATGTCGTGGGAAACATGTAGG | (GT)12 | 178—182 | 3 | sex |
利用基因组DNA提取试剂盒(Axygen)提取细鳞鲑鳍条组织的基因组DNA, 经过紫外分光光度计(260/280)测定浓度和纯度后, 调整浓度至50ng/μL。采用毛细管电泳技术检测96个样本的基因型, 在正向引物的5′端标记蓝色(FAM)和绿色(HEX)荧光, 建立15μL PCR扩增反应体系, 包括1.5mmol/L MgCl2、50mmol/L KCl、10mmol/L Tris-Cl (pH 8.3)、200μmol/L dNTP、微卫星引物、0.3μmol/L、100ng DNA模板及1U Taq DNA聚合酶。PCR反应程序为94℃ 3min; 94℃ 30s, 56℃ 30s, 72℃ 30s, 25个循环; 72℃ 5min。反应结束后, 用3730XL遗传分析仪(ABI)进行毛细管凝胶电泳, 利用GeneMapper V4.1软件进行图像收集和数据分析。
1.4 数据分析使用软件“鱼类种质资源遗传分析装置(ZL200710144749.3)”进行数据转换, 用PopGene32 (Version 1.32)软件计算每个微卫星标记的等位基因数(observed number of alleles, Na)、等位基因频率(allele frequency, P)、有效等位基因数(effective number of alleles, Ne)、观测杂合度(observed heterozygosity, Ho)和期望杂合度(expected heterozygosity, He)。标记的多态性信息含量(polymorphism information content, PIC)按照Botstein等(1980)的方法计算, 公式如下:
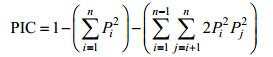
式中, n为某一位点上等位基因数, Pi、Pj分别为第i和第j个等位基因在群体中的频率, j=i+1。
用GenePop(Version 4.2)软件进行χ2检验估计群体Hardy-Weinberg平衡偏离。
群体的遗传分化系数(Fst)、基因流系数(Nm)以及群体间的遗传相似性指数(I)及遗传距离(Ds)也由PopGene32软件计算, 运行PHYLIP (version3.696)软件的Neighbor程序, 绘制群体间基于UPGMA的聚类图。
2 结果 2.1 PCR扩增结果用随机4个细鳞鲑样本初步筛选150个虹鳟微卫星标记, 在56℃退火温度下, 64个标记扩增出清晰、稳定的目的条带, 38个标记表现出不同程度的多态性。选择其中22个微卫星标记用毛细管电泳技术分析了细鳞鲑连续3代选育群体96个样本的基因型, 结果共检测到181个等位基因, 片段大小为118—355bp, 每个标记扩增到2—26个等位基因, 平均为8.227(表 1), 其中16个标记(72.73%)检测到4个以上的等位基因, 检测到等位基因数(Na)最多的标记为OMM1236, 扩增出26个等位基因; Na最少的标记为OMM1192、OMM1263和OMM3065, 仅检测到2个等位基因。标记OMM1329在全部96个细鳞鲑样本的毛细管电泳图谱见图 1。
 |
| 图 1 细鳞鲑3代选育群体在微卫星标记OMM1329的毛细管电泳图谱 Fig. 1 The capillary electrophoretogram of the three-generation breeding populations of B. lenok at OMM1329 注: 1—32: F1群体; 33—64: F2群体; 65—96: F3群体 |
细鳞鲑F1选育群体在22个微卫星标记的等位基因数(Na)为2—17, 平均为6.500;有效等位基因数(Ne)为1.032—10.722, 平均为3.649。F2选育群体的Na为2—21, 平均为6.773; Ne为1.032—12.412, 平均为3.356。F3选育群体的Na为1—17, 平均为6.773; Ne为1.000—11.571, 平均为3.624。成对样本的T检验结果显示Na和Ne在细鳞鲑连续3代选育群体间差异不显著。细鳞鲑F1选育群体的观测杂合度(Ho)为0.031—0.969, 平均为0.530;期望杂合度(He)为0.031—0.921, 平均为0.566。F2选育群体的Ho为0.031—0.875, 平均为0.462; He为0.031—0.934, 平均为0.494。F3选育群体的Ho为0.000—0.969, 平均为0.474; He为0.000—0.928, 平均为0.519。成对样本的T检验结果显示细鳞鲑F2和F3选育群体的Ho和He显著低于F1选育群体(P<0.05), 而F2和F3差异不显著。细鳞鲑F1选育群体的多态信息含量(PIC)为0.030—0.900, 平均为0.525; F2选育群体的PIC为0.030—0.919, 平均为0.459; F3选育群体的PIC为0.000—0.907, 平均为0.481。可见, F1选育群体处于高度多态水平(PIC≥0.5) (Botstein et al, 1980), 而F2和F3选育群体虽然保持了中度多态水平(0.25≤PIC<0.5) (Botstein et al, 1980), 但是与F1相比多态信息含量已显著降低。
用GenePop(Version 4.2)软件分析了细鳞鲑连续3代选育群体的Hardy-Weinberg遗传偏离, 结果表明细鳞鲑3代选育群体整体保持了遗传平衡状态(multi-loci test, P=1), 但经Bonferroni校正后, F1选育群体在OMM1762和OMM3048极限著偏离遗传平衡(P<0.0005), F2选育群体在OMM1145、OMM1329和OMM3048极限著偏离遗传平衡(P<0.0005), F3选育群体在OMM3048和OMM5192极限著偏离遗传平衡(P<0.0005)。除了F2选育群体在OMM1329表现为杂合子显著过剩外, 其余均表现为杂合子显著不足。详细统计数据见表 2。
| 标记 | F1 | F2 | F3 | |||||||||||
| Na/Ne | Ho/He | PIC | PHWE | Na/Ne | Ho/He | PIC | PHWE | Na/Ne | Ho/He | PIC | PHWE | |||
| OMM1042 | 14/7.699 | 0.875/0.884 | 0.858 | 0.419 | 14/8.225 | 0.844/0.892 | 0.867 | 0.914 | 14/9.102 | 0.969/0.904 | 0.880 | 0.040 | ||
| OMM1145 | 7/4.947 | 0.594/0.810 | 0.771 | 0.011 | 7/4.064 | 0.594/0.766 | 0.723 | 0.000** | 8/4.785 | 0.563/0.804 | 0.764 | 0.041 | ||
| OMM1192 | 2/1.032 | 0.031/0.031 | 0.030 | 1.000 | 2/1.032 | 0.031/0.031 | 0.030 | 1.000 | 1/1.000 | 0.000/0.000 | 0.000 | / | ||
| OMM1231 | 11/8.192 | 0.969/0.892 | 0.866 | 0.372 | 11/7.699 | 0.875/0.884 | 0.856 | 0.091 | 10/8.292 | 0.875/0.893 | 0.868 | 0.861 | ||
| OMM1236 | 17/10.039 | 0.906/0.915 | 0.892 | 0.074 | 21/12.412 | 0.844/0.934 | 0.919 | 0.011 | 17/11.571 | 0.969/0.928 | 0.907 | 0.779 | ||
| OMM1263 | 2/1.358 | 0.312/0.268 | 0.229 | 0.324 | 2/1.133 | 0.125/0.119 | 0.110 | 0.746 | 2/1.098 | 0.094/0.091 | 0.085 | 0.822 | ||
| OMM1329 | 5/3.012 | 0.656/0.679 | 0.618 | 0.681 | 6/2.236 | 0.625/0.562 | 0.496 | 0.000** | 8/2.513 | 0.625/0.612 | 0.531 | 0.999 | ||
| OMM1372 | 3/2.042 | 0.281/0.518 | 0.444 | 0.003 | 4/2.077 | 0.312/0.527 | 0.436 | 0.154 | 4/2.301 | 0.406/0.574 | 0.483 | 0.368 | ||
| OMM1727 | 6/1.907 | 0.438/0.483 | 0.446 | 0.994 | 5/1.538 | 0.375/0.355 | 0.330 | 0.991 | 7/1.665 | 0.406/0.406 | 0.382 | 0.999 | ||
| OMM1762 | 9/3.287 | 0.531/0.707 | 0.657 | 0.000** | 8/2.214 | 0.531/0.557 | 0.514 | 0.239 | 8/3.205 | 0.625/0.699 | 0.634 | 0.024 | ||
| OMM3042 | 3/1.292 | 0.250/0.230 | 0.213 | 0.905 | 3/1.135 | 0.125/0.121 | 0.114 | 0.991 | 3/1.210 | 0.188/0.177 | 0.166 | 0.964 | ||
| OMM3048 | 4/2.216 | 0.219/0.558 | 0.500 | 0.000** | 5/1.853 | 0.250/0.468 | 0.436 | 0.000** | 4/1.645 | 0.219/0.398 | 0.366 | 0.000** | ||
| OMM3065 | 2/1.358 | 0.312/0.268 | 0.229 | 0.324 | 2/1.168 | 0.156/0.146 | 0.134 | 0.670 | 2/1.064 | 0.062/0.062 | 0.059 | 0.898 | ||
| OMM3080 | 3/1.415 | 0.344/0.298 | 0.265 | 0.744 | 3/1.250 | 0.219/0.203 | 0.188 | 0.939 | 3/1.336 | 0.188/0.256 | 0.237 | 0.059 | ||
| OMM3081 | 3/2.359 | 0.562/0.585 | 0.486 | 0.831 | 4/2.263 | 0.594/0.567 | 0.459 | 0.948 | 5/2.392 | 0.469/0.591 | 0.504 | 0.830 | ||
| OMM3126 | 7/2.872 | 0.594/0.662 | 0.621 | 0.446 | 7/3.408 | 0.781/0.718 | 0.671 | 0.953 | 8/2.589 | 0.531/0.624 | 0.578 | 0.760 | ||
| OMM5006 | 4/1.527 | 0.406/0.351 | 0.320 | 0.929 | 4/1.211 | 0.188/0.177 | 0.167 | 0.999 | 4/1.253 | 0.156/0.205 | 0.194 | 0.142 | ||
| OMM5044 | 8/2.829 | 0.656/0.657 | 0.622 | 0.834 | 7/2.266 | 0.656/0.568 | 0.532 | 0.986 | 8/2.609 | 0.688/0.626 | 0.583 | 0.999 | ||
| OMM5106 | 17/10.722 | 0.938/0.921 | 0.900 | 0.888 | 18/9.225 | 0.875/0.906 | 0.883 | 0.382 | 16/11.130 | 0.844/0.925 | 0.903 | 0.043 | ||
| OMM5136 | 10/6.282 | 0.844/0.854 | 0.822 | 0.274 | 11/4.339 | 0.656/0.782 | 0.745 | 0.727 | 10/5.491 | 0.844/0.831 | 0.794 | 0.921 | ||
| OMM5192 | 3/1.336 | 0.219/0.256 | 0.237 | 0.146 | 2/1.098 | 0.094/0.091 | 0.085 | 0.822 | 4/1.379 | 0.188/0.279 | 0.256 | 0.000** | ||
| BX076085 | 3/2.550 | 0.719/0.618 | 0.526 | 0.512 | 3/1.994 | 0.406/0.506 | 0.402 | 0.382 | 3/2.107 | 0.531/0.534 | 0.416 | 0.998 | ||
| 均值 | 6.500/3.649 | 0.530/0.566 | 0.525 | / | 6.773/3.356 | 0.462/0.494 | 0.459 | / | 6.773/3.624 | 0.474/0.519 | 0.481 | / | ||
| 注:PHWE,Hardy-Weinberg平衡检验概率值; *, P<0.0023; **, P<0.0005 | ||||||||||||||
用PopGene32软件计算的细鳞鲑连续3代选育群体间近交系数(Fst)为0.0053—0.0329, 平均为0.0149, 表明1.49%的遗传差异来自群体间, 说明群体间遗传分化较弱。连续3代选育群体的基因流(Nm)为7.3393—47.2500, 平均为16.5748, 表明群体间有广泛的遗传交流。群体间遗传距离为0.0219—0.0252, 遗传相似系数为0.9751—0.9782, F1与F2间遗传距离最小(0.0219), 遗传相似系数最大(0.9782); F1与F3间遗传距离最大(0.0252), 遗传相似系数最小(0.9751) (表 3)。利用UPGMA法构建的群体遗传关系聚类图如图 2所示。
| 群体 | F1 | F2 | F3 |
| F1 | **** | 0.9782 | 0.9751 |
| F2 | 0.0219 | **** | 0.9775 |
| F3 | 0.0252 | 0.0228 | **** |
 |
| 图 2 依据群体间的遗传距离构建的UPGMA聚类图 Fig. 2 The clustering of populations built by UPGMA method according to their genetic distances |
细鳞鲑作为我国珍稀的土著冷水性鱼类, 其自然资源量急剧下降, 在中国濒危动物红皮书中已被列为易危物种(汪松, 1998), 保护和合理开发细鳞鲑势在必行。目前, 细鳞鲑在完成规模化人工繁殖苗种的基础上, 开展了以生长和怀卵量为目标的选育, 已获得F3苗种。维持群体的遗传多样性水平是群体持续利用的前提, 遗传多样性越高, 性状改良潜力及育种价值也就越大。本研究用22个虹鳟微卫星标记评估细鳞鲑连续3代选育群体的遗传结构, 3代平均观测杂合度(Ho)分别为0.530、0.462和0.474, 期望杂合度(He)平均为0.566、0.494和0.519, F1群体的Ho和He均高于Jia等(2008)用13个虹鳟微卫星标记分析的乌苏里江野生细鳞鲑群体的杂合度(Ho=0.5056; He=0.5465), 但F2和F3的杂合度已显著低于野生群体。与F1群体相比, F2群体的Ho和He分别降低了12.83%和12.72%, F3群体降低了10.57%和8.30%, 表明细鳞鲑已经受到人工选择的影响, 导致部分位点纯合子比例增加, 尤其在等位基因数较少的标记如OMM1263、OMM3065、OMM3080等。累代选育致使群体遗传多样性降低在其他鱼类也有发现, 大黄鱼F4选育群体的Ho从F1的0.779下降到0.532, 下降了31.71%(赵广泰等, 2010); 翘嘴鳜F4选育群体的Ho从F1的0.405下降到0.229, 下降了43.46%(郑荷子等, 2013), 说明鱼类在选育初期, 遗传多样性呈现较大幅度的降低, 而选育代数较多的“新吉富”罗非鱼F13选育群体的Ho从F10的0.5744下降到0.5256, 仅下降了8.50%(于爱清等, 2011), 也说明随着选择代数的增加, 遗传多样性趋于稳定。多态信息含量(PIC)直观反应了群体的遗传多样性水平, 细鳞鲑连续3代选育群体的PIC在0.459—0.525, 表明群体均处于中高度多态水平(PIC≥0.25) (Botstein et al, 1980), 尚具有一定的遗传选育潜力, 而显著降低的遗传参数也提示我们在今后的选育工作中, 应该注重维持和保护选育群体的遗传多样性, 制定合理的选育种策略, 以确保顺利进行选育工作。
遗传分化系数(Fst)是反映群体间遗传分化程度的重要参数。Wright(1978)将群体间遗传分化程度划分为轻度(Fst=0—0.05)、中度(Fst=0.05—0.15)、较大(Fst=0.15—0.25)和极大(Fst>0.25)。本研究结果显示细鳞鲑连续3代选育群体在22个微卫星标记的Fst为0.0053—0.0329, 平均为0.0149, 表明群体间遗传分化较弱, 但是F1与F3间的遗传距离(0.0252)略远于F1与F2间(0.0219)。由于细鳞鲑选育代数较少, 后续累代选育群体间的遗传分化趋势尚无法预测。但在选育代数较多的罗非鱼中的研究结果显示F0与F8、F9之间的Fst平均值(0.06840)大于F0与F6、F7之间(0.05103), 而F0与F8、F9之间遗传距离(0.0691和0.0722)相对于F0与F6、F7之间(0.0581和0.0595)也有加大的趋势, 表明随着选育世代的增加, 后续世代与F0的遗传距离逐代增大, 遗传相似性逐代减小, 也表明9代选育在罗非鱼世代间形成了中度的遗传分化(颉晓勇等, 2007)。鉴于此, 在人工定向选育过程中, 细鳞鱼选育群体趋于遗传纯化的趋势无法避免。因此, 在细鳞鲑的开发利用过程中, 建议采用保护与选育并举的策略, 同时建立保种和育种2个群体, 保种群体最大限度地保持细鳞鲑种质资源库的遗传多样性, 并适当补充野生资源, 在保种的同时也可补充后续育种群体遗传多样性的不足; 育种群体在进一步选育中, 保持适当比例的选择压力以便使选育群体的优良性状得到持续提高, 同时应采用分子标记辅助选育, 使与目标性状相关的位点尽快纯合固定, 而其它位点尽可能保持多态, 并适当增加每代繁育亲本的数量, 减少近交繁殖可能带来的负面影响。
4 结论综上所述, 本研究选用虹鳟微卫星标记分析了连续3代细鳞鲑选育群体的遗传结构, 鉴定出22对可用于细鳞鲑遗传育种研究的微卫星标记, 分析结果也为细鳞鲑的持续选育、种质保护及持续利用提供参考。
| 于爱清, 李思发, 蔡完其, 2011. "新吉富"罗非鱼选育F10~F13遗传变异微卫星分析. 上海海洋大学学报, 20(1): 1–7 |
| 王荻, 徐革锋, 刘洋, 等, 2009. 细鳞鱼三个野生种群的遗传多样性AFLP分析. 农业生物技术学报, 17(4): 638–644 |
| 牟振波, 李永发, 徐革锋, 等, 2013. 细鳞鱼全人工繁育技术的研究. 水产学杂志, 26(1): 15–18 |
| 牟振波, 徐革锋, 杨双英, 2008. 细鳞鱼卵巢滤泡细胞的发育及功能. 中国水产科学, 15(1): 167–171 |
| 李超, 鲁翠云, 郑先虎, 等, 2014. 一种保存鱼类鳍条的便捷方法. 水产学杂志, 27(1): 22–24 |
| 李永发, 丛宇, 徐革锋, 等, 2009. 细鳞鱼发眼卵孵化及仔、稚鱼培育. 水产学杂志, 22(4): 34–35 |
| 李思忠, 1984. 中国鲑科鱼类地理分布的探讨. 动物学杂志, 19(1): 76–79 |
| 汪松, 1998. 中国濒危动物红皮书:鱼类. 北京: 科学出版社, 35-37 |
| 张艳萍, 杜岩岩, 王太, 等, 2014. 秦岭细鳞鲑群体遗传结构. 生态学报, 34(17): 4950–4956 |
| 郑荷子, 易提林, 梁旭方, 等, 2013. 翘嘴鳜连续4代选育群体遗传多样性及遗传结构分析. 淡水渔业, 43(6): 8–12 |
| 赵广泰, 刘贤德, 王志勇, 等, 2010. 大黄鱼连续4代选育群体遗传多样性与遗传结构的微卫星分析. 水产学报, 34(4): 500–507 |
| 夏颖哲, 盛岩, 陈宜瑜, 2006. 利用线粒体DNA控制区序列分析细鳞鲑种群的遗传结构. 生物多样性, 14(1): 48–54 |
| 徐革锋, 叶远涛, 刘洋, 等, 2010. 雌雄细鳞鱼肌肉营养成分比较分析. 水产学杂志, 23(2): 29–33 |
| 徐革锋, 刘洋, 李永发, 等, 2013. 细鳞鲑早期发育过程中的消化系统发生. 中国水产科学, 20(4): 733–742 |
| 徐革锋, 刘洋, 郝其睿, 等, 2016. 不同蛋白质和脂肪水平对细鳞鲑幼鱼生长和肌肉氨基酸含量的影响. 中国水产科学, 23(6): 1311–1319 |
| 徐革锋, 李永发, 范兆廷, 等, 2009a. 细鳞鱼卵子发生过程中细胞器的形态变化与作用. 动物学杂志, 44(6): 81–88 |
| 徐革锋, 牟振波, 薛淑群, 等, 2009b. 不同流域细鳞鱼染色体遗传多态性分析. 水生生物学报, 33(5): 975–979 |
| 颉晓勇, 李思发, 蔡完其, 2007. 吉富品系尼罗罗非鱼选育过程中遗传变异的微卫星分析. 水产学报, 31(3): 385–390 |
| Botstein D, White R L, Skolnick M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 32(3): 314–331 |
| Jia Z Y, Zhang Y Y, Shi L Y, et al, 2008. Amplification of rainbow trout microsatellites in Brachymystax lenok. Molecular Ecology Resources, 8(6): 1520–1521 DOI:10.1111/men.2008.8.issue-6 |
| Liu Q, Li Y, Liang H W, et al, 2014. Isolation and characterization of fourteen novel microsatellite loci from Brachymystax lenok tsinlingensi. Journal of Genetics, 93(2): e35–e37 |
| Liu Y, Li Z J, Zhang T L, et al, 2015. Growth and energy budget of juvenile lenok Brachymystax lenok in relation to ration level. Chinese Journal of Oceanology and Limnology, 33(2): 347–355 DOI:10.1007/s00343-015-3349-x |
| Rexroad Ⅲ C E, Palti Y, Gahr S A, et al, 2008. A second generation genetic map for rainbow trout (Oncorhynchus mykiss). BMC Genetics, 9: 74 |
| Si S J, Wang Y, Xu G F, et al, 2012. Complete mitochondrial genomes of two lenoks, Brachymystax lenok and Brachymystax lenok tsinlingensis. Mitochondrial DNA, 23(5): 338–340 DOI:10.3109/19401736.2012.690749 |
| Wright S, 1978. Variability Within and Among Natural Populations. Chicago: The University of Chicago Press, 114 |
| Xu G F, Wang Y Y, Han Y, et al, 2015. Growth, feed utilization and body composition of juvenile Manchurian trout, Brachymystax lenok (Pallas) fed different dietary protein and lipid levels. Aquaculture Nutrition, 21(3): 332–340 DOI:10.1111/anu.2015.21.issue-3 |
| Yu J N, Kim S, Lim C E, 2014. Development of 21 novel microsatellite markers for conservation genetic studies of Brachymystax lenok tsinlingensis, an endangered species in Korea, using next-generation sequencing. Conservation Genetics Resources, 6(1): 213–216 DOI:10.1007/s12686-013-0059-6 |
 2018, Vol. 49
2018, Vol. 49


