
中国海洋湖沼学会主办。
文章信息
- 赖晓健, 林祥日, 罗碧莲, 江兴龙. 2018.
- LAI Xiao-Jian, LIN Xiang-Ri, LUO Bi-Lian, JIANG Xing-Long. 2018.
- 基于线粒体cyt b基因序列差异的组合PCR技术快速鉴定五种鳗鲡养殖种类的方法研究
- IDENTIFICATION OF FIVE CULTURAL ANGUILLA SPECIES BY COMBINED PCR TECHNIQUE BASED ON DIFFERENT SEQUENCES OF MITOCHONDRIA CYTOCHROME b GENE
- 海洋与湖沼, 49(4): 925-931
- Oceanologia et Limnologia Sinica, 49(4): 925-931.
- http://dx.doi.org/10.11693/hyhz20180400097
-
文章历史
- 收稿日期:2018-04-17
- 收修改稿日期:2018-05-14
2. 鳗鲡现代产业技术教育部工程研究中心 厦门 361021;
3. 厦门海洋职业技术学院 厦门 361100
2. Engineering Research Center of the Modern Industrial Technology for Eel, Ministry of Education, Xiamen 361021, China;
3. Xiamen Ocean Vocational College, Xiamen 361100, China
鳗鲡在分类学中隶属硬骨鱼纲(Osteichthyes), 辐鳍亚纲(Actinopterygii), 鳗鲡目(Anguilliformes), 鳗鲡科(Anguillidae), 鳗鲡属(Anguilla)。近二十多年来, 鳗鲡养殖业在我国占有很大的市场。目前, 鳗鲡养殖苗种完全依赖天然捕捞, 由于自然或人为因素, 容易导致不同种鳗苗混杂的现象。比如, 花鳗鲡(Anguilla marmorata)主要分布于西太平洋和印度洋的热带和亚热带地区, 在中国的东南省份和台湾地区以及太平洋中部诸岛等地均有发现(Wade et al, 2007)。太平洋双色鳗鲡(A. bicolor pacifica)属热带鳗鲡, 主要分布于西太平洋的赤道地区, 包括菲律宾的吕宋岛等地, 与花鳗鲡分布范围部分重叠, 在河口地区其鳗苗与前者往往被一同捕捞(Han et al, 2001), 导致鳗苗的自然混杂。另外, 由于不同种鳗苗的市场价格差异较大, 有商家将低价鳗苗参入高价鳗苗中, 从而获取更大的利益。而白仔鳗苗在形态上十分相似, 很难通过形态学进行辨别, 造成我国鳗鲡养殖业苗种资源比较混乱的局面, 而不同的鳗鲡种类具有不同的生物学特征和养殖环境要求(吴宁, 2010), 盲目养殖会增加风险。同时, 自然保护国际联盟组织已将欧洲鳗列为极度濒危物种, 将对其实施新的监管措施, 我国鳗鱼产品(如烤鳗)出口日本, 要提供检验检疫部门出具的鳗鱼物种鉴定报告(陈文炳等, 2017)。
目前用于鉴定鱼类的分子生物学技术还有许多, 有限制性片段长度多态性(RFLP) (Ludwig et al, 2002)、单链构象多态性分析(SSCP) (Rehbein et al, 1995)和特异性PCR (DeSalle et al, 1996; Birstein et al, 1998)等。但RFLP这种方法操作繁琐, 相对费时, 要求DNA的量大, 并且可能因为碱基突变而导致限制性酶切位点的改变。SSCP的结果易受多种因素影响, 并且当单链DNA碱基序列自身的差异或者突变对单链立体构象无影响时, 可能会因此产生假阴性结果而导致漏检(贾秀双, 2014)。特异性PCR的方法需要测序, 耗时长, 无法满足现场检测的要求。以上的几种方法不是操作繁琐, 耗时长, 成本高, 就是对技术人员、设备要求高等, 都不适用于生产应用, 所以急需一种能够在现场就快速、高效使用的鉴定方法。本研究通过比对日本鳗鲡(A. japonica)、花鳗鲡(A. mamorata)、美洲鳗鲡(A. rostrata)、太平洋双色鳗鲡(A. bicolor pacific)、欧洲鳗鲡(A. anguilla)线粒体细胞色素B (cytochrome b, cyt b)序列, 针对差异的片段设计4套特异性引物, 通过组合PCR扩增特定的片段, 以此鉴定鳗鲡的种类, 从而达到在现场快速鉴定这5种鳗鲡的目的。
1 材料与方法 1.1 实验材料日本鳗鲡、花鳗鲡、美洲鳗鲡(南美和北美种群)、太平洋双色鳗鲡和欧洲鳗鲡取自于集美大学水产学院海水试验场或购于鳗鲡养殖场。
1.2 提取DNA分别取3尾日本鳗鲡、花鳗鲡、美洲鳗鲡、太平洋双色鳗鲡和欧洲鳗鲡, 麻醉后, 测量全长与体重(表 1)。酒精消毒后减去表皮取肌肉, –80℃冰箱保存。根据DNA快速提取试剂盒(Tiangen)说明书步骤分别提取各种鳗鲡DNA, 用NanoDrop 2000系统和琼脂糖凝胶电泳检测DNA的浓度、纯度和质量。用灭菌超纯水稀释至10ng/μL, –20℃保存备用。
| 鳗鲡种类 | 日本鳗鲡 | 美洲鳗鲡(南) | 美洲鳗鲡(北) | 太平洋双色鳗鲡 | 花鳗鲡 | 欧洲鳗鲡 |
| 全长(cm) | 58.23±1.67 | 11.8±0.96 | 31.23±5.06 | 45.95±2.89 | 46.44±1.27 | 31.6±4.40 |
| 体重(g) | 214.55±16.90 | 0.08±0.003 | 79.01±44.96 | 201.53±18.44 | 184.95±3.85 | 66.52±41.07 |
| 注:美洲鳗鲡(南)指南美洲鳗鲡; 美洲鳗鲡(北)指北美洲鳗鲡; 它们属于美洲鳗鲡的不同地理种群 | ||||||
从NCBI上获取日本鳗鲡等五种鳗鲡的线粒体cyt b基因序列, 进行序列比对, 在差异片段用Primer Premier 5软件进行引物设计, 引物由上海生工生物科技有限公司合成。配置PCR反应体系(20μL): 2×Taq PCR Master Mix (Tiangen) 10μL, Primer (10μmol/L) 1μL, 鳗鲡DNA (10ng/μL) 1μL, H2O 7μL。温度梯度PCR扩增, PCR扩增程序为:预变性94℃ 5min, 94℃ 30s, 54—62℃ 30s, 72℃ 45s, 设置25、27、29和31共4个PCR扩增循环数, 总延伸72℃ 7min。根据PCR产物的琼脂糖胶电泳条带确定每对引物适合的退火温度和PCR扩增循环数。将每队引物的PCR产物测序, 得到的序列在NCBI Blast进行序列同源性比对分析, 确定PCR产物为目的基因片段。
1.4 组合PCR将经过PCR条件优化后选定的引物混合在同一个PCR体系中使用, 优化组合PCR温度和循环数, 筛选出最优的引物组合。
2 结果 2.1 cyt b序列分析和比对通过比对已有的5种鳗鲡cyt b基因序列可以发现5种鳗鲡cyt b基因核苷酸序列共有180个变异位点, 其中欧洲鳗鲡和美洲鳗鲡的cyt b基因核苷酸序列差异最小仅为47个, 同源性为95.88%, 而欧洲鳗鲡和太平洋双色鳗鲡cyt b基因核苷酸序列的差异最大为106bp, 同源性为90.70% (图 1)。选择其中5个核苷酸序列差异较大的片段, 分别为322—344bp, 451—472bp, 663—688bp, 768—789bp, 1086—1110bp (图 1), 根据这些片段设计PCR上下游引物。通过MEGA5.0软件构建5种鳗鲡cyt b基因序列NJ进化树, 发现花鳗鲡和太平洋双色鳗鲡先聚为一支, 再与日本鳗鲡聚在一起, 欧洲鳗鲡和美洲鳗鲡聚在一起(图 2), 进化树图显示的遗传距离和它们的地理分布的远近相似。
 |
| 图 1 5种鳗鲡cyt b基因序列比对 Fig. 1 The sequence and alignment of cyt b gene among five Anguilla species 注: AF006703.1, 日本鳗鲡; AF006715.1, 欧洲鳗鲡; AF006716.1, 美洲鳗鲡; AF006709.1, 太平洋双色鳗鲡; AF006705.1, 花鳗鲡。方框内为序列差异较大片段 |
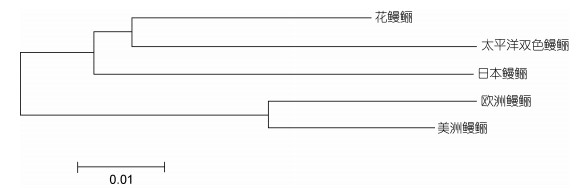 |
| 图 2 5种鳗鲡cyt b基因序列NJ进化树 Fig. 2 NJ clustering of five Anguilla species by cyt b gene sequence |
设计的每对引物经过PCR反应后, 选择能够特异性扩增得到相应物种cyt b基因序列片段的引物, 再进行梯度退火温度和梯度循环数进行条件优化后筛选出4对引物, 并确定了最优的PCR反应条件(表 2)。筛选出4对引物中, aj S1/A1特异性扩增日本鳗鲡cyt b基因序列, r-a S1/A2特异性扩增美洲鳗鲡cyt b基因序列, 由于花鳗鲡和太平洋双色鳗鲡亲缘关系较近, m-a S4/A4可以扩增花鳗鲡和太平洋双色鳗鲡cyt b基因序列, bp-m S5/A3能够特异性扩增太平洋双色鳗鲡cyt b基因序列, 因此可以将花鳗鲡和太平洋双色鳗鲡区分开。将5种鳗鲡DNA作为模板分别和这4对引物进行PCR反应, 通过PCR产物的琼脂糖凝胶电泳结果的阳性或阴性以及阳性条带的大小可以区分出这5种鳗鲡(表 3)。
| 引物 | 引物序列5′→3′ | 目的基因片段大小(bp) | 退火温度(℃) | 循环数 |
| aj S1 | TTATGGCTGATTCATCCGAAATT | 889 | 60.4 | 25 |
| aj A1 | CCTGCTAATGGGTTGAGTACTAAA | |||
| r-a S1 | TCTGCCGTTTCATACGTAGGA | 341 | 60.4 | 25 |
| r-a A2 | AACTATTGGGTTTGCAGGGGTA | |||
| bp-m S5 | TCCATACTTCTCATACAAAGACCAAT | 457 | 58 | 27 |
| bp-m A3 | CTAGTCAACCTACTAATGGGTTTAAT | |||
| m-a S4 | CCGCCGTCCCATACGTAGGAG | 678 | 58 | 27 |
| m-a A4 | TATTTTGTTTTCTAGTCAACCTACTAAT |
| 引物 | 日本鳗鲡 | 美洲鳗鲡 | 花鳗鲡 | 太平洋双色鳗鲡 | 欧洲鳗鲡 | |
| aj S1/A1 | + | — | — | — | — | |
| r-a S1/A2 | — | + | — | — | — | |
| bp-m S5/A3 | — | — | — | + | — | |
| m-a S4/A4 | — | — | + | + | — | |
| 注: +表示阳性, —表示阴性 | ||||||
在同时加入4对引物的组合PCR反应体系中, 以某一种鳗鲡DNA作为模板进行组合PCR反应。经过梯度退火温度和梯度循环数的条件优化, 确定组合PCR的最优条件:退火温度为58℃; 退火时间为45s;循环数为27。
组合PCR产物的琼脂糖凝胶电泳图可见欧洲鳗鲡无条带呈阴性, 其余均匀阳性条带; 日本鳗鲡为889bp, 南美洲鳗鲡与北美洲鳗鲡均为341bp, 吕宋鳗鲡(A. luzonensis)与花鳗鲡均为678bp, 太平洋双色鳗鲡扩增出两个条带, 一条大小为678bp, 另一条为457bp (表 4, 图 3)。组合PCR的结果和利用4对引物分别进行单个PCR的结果(表 3)一致。组合PCR产物各条带的测序结果显示各对引物扩增出相应种类的鳗鲡cyt b基因片段, 没有出现引物错配。通过组合4对引物的PCR反应和PCR产物的琼脂糖凝胶电泳图可以清楚地鉴别这5种常见鳗鲡种类。
| DNA模板 | 引物 | 结果 | |
| 阳性(+)、阴性(-) | 条带大小(bp) | ||
| 日本鳗鲡 | aj S1/A1 | + | 889 |
| 南美洲鳗鲡 | r-a S1/A2 | + | 341 |
| 北美洲鳗鲡 | bp-m S5/A3 | + | 341 |
| 花鳗鲡(吕宋鳗鲡) | + | 678 | |
| 太平洋双色鳗鲡 | m-a S4/A4 | + | 678和457 |
| 欧洲鳗鲡 | - | 无 | |
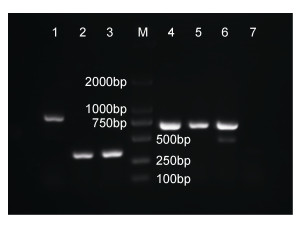 |
| 图 3 组合PCR产物琼脂糖胶片图 Fig. 3 Electrophoresis pattern of the combined PCR 注: 1.日本鳗鲡; 2.南美洲鳗鲡; 3.北美洲鳗鲡; 4.吕宋鳗鲡; 5.花鳗鲡; 6.太平洋双色鳗鲡; 7.欧洲鳗鲡; M. Marker |
本研究采样的3尾花鳗鲡有2尾PCR产物的测序结果显示属于吕宋鳗鲡的概率高于花鳗鲡。有研究者认为吕宋鳗鲡也是一种新鳗鲡种(Kuroki et al, 2012), 但是其仍存在很大的争议, 目前公认的鳗鲡种只有19种, 不包括吕宋鳗鲡的(Ege, 1939), 且有研究证明吕宋鳗鲡与花鳗鲡的亲缘关系较近(孙鹏, 2013)。而本次研究主要是鉴定日本鳗鲡、花鳗鲡、美洲鳗鲡、太平洋双色鳗鲡、欧洲鳗鲡, 吕宋鳗鲡可作为花鳗鲡的不同地理种群, 对本研究结果没有影响。
鳗鲡的种质鉴定研究已有报道。陈茂辉(2008)利用肉眼观察、镜检等技术观察鳗鲡的形态学特征, 并结合药检对美洲鳗鲡、花鳗鲡、日本鳗鲡、欧洲鳗鲡等进行种质鉴定。但由于形态学标记技术不够准确、稳定, 易受环境因素的影响, 因此这种方法用来对鳗鲡的种质鉴定并不可靠, 所以要结合分子生物学技术来进行更深入的研究。吴宁(2010)利用AFLP技术和线粒体DNA COⅠ和COⅡ分子标记技术成功地将美洲鳗鲡、日本鳗鲡、欧洲鳗鲡、非洲鳗鲡(A. mossambica)、花鳗鲡和太平洋双色鳗鲡进行鉴定区分, 陈文炳等(2017)利用16SrRNA基因序列PCR扩增和测序的方法鉴定了日本鳗鲡, 欧洲鳗鲡和美洲鳗鲡。但上述两种方法都有不足之处, AFLP技术的操作过程比较复杂, 对DNA的质量以及操作人员技术水平要求较高, 一般实验室开展此技术尚存在困难(刘云国, 2009), 而后者序列需要测序, 步骤繁琐, 耗时较长, 成本高。本研究将单独的4个PCR反应体系的引物通过优化组合建立成一个组合PCR反应, 通过实验探索出其合适的组合反应条件, 组合PCR得出的结果与单个PCR反应所得结果相同, 通过单个PCR反应产物的琼脂糖胶图即可鉴定鳗鲡的种类, 并且还能够省时、节约试剂和减少操作。本研究中DNA的提取通过使用快速DNA提取检测试剂盒, 提高了DNA提取速度, 通常只要5-10分钟。整个鉴定过程在2至3小时即可完成。同时所使用到的仪器也都是一些小型仪器(PCR仪、电泳仪和可见光透射仪等), 方便携带。所以本研究提供的组合PCR技术是一种能够在现场就快速、高效的检测, 适用于生产应用的鉴定方法。
线粒体DNA具有严格的母性遗传, 重组率低, 进化速度快, 并且线粒体DNA不同基因区域的进化速率不同, 因此不同的片段可进行不同水平的种类鉴定(齐兴柱等, 2007; 陈姝君等, 2008)。cyt b是线粒体自身编码的为数不多的蛋白质之一, 而且还是线粒体DNA上惟一结构和功能被了解得较为清楚的蛋白质编码基因, 具有适中的进化速度, 被认为是分析系统发育问题最可信的线粒体DNA标记之一, 因而广泛地运用于脊椎动物系统进化的研究(Zardoya et al, 1996)。本研究利用常见的5种养殖鳗鲡cyt b基因做进化树分析, 结果表明和利用线粒体DNA COⅠ(吴宁, 2010), 12S rRNA和cyt b (Lin et al, 2001), 16S rRNA和cyt b (Aoyama et al, 2001)的进行进化树分析的结果一致。马亚等(2012)和张殿昌等(2006)多位研究者曾使用PCR方法分析线粒体中的cyt b基因的部分片段, 成功区分了不同科、不同种的鱼类。
4 结论本实验针对日本鳗鲡、美洲鳗鲡、花鳗鲡、太平洋双色鳗鲡、欧洲鳗鲡这5种常见养殖鳗鲡线粒体cyt b序列的差异, 设计引物并通过PCR条件优化获得aj S1/A1、r-a S1/A2、bp-m S5/A3、m-a S4/A4这4对引物, 通过组合PCR方法可快速、有效的现场鉴定这5种鳗鲡。下一步实验可继续深入研究以区分出更多的鳗鲡种类, 完善组合PCR方法。本研究结果可提高养殖效益保障鳗农的合法利益, 促进鳗鲡养殖业的健康发展, 对鳗鲡加工与出口外贸也有重要的现实意义。
| 马亚, 岳志芹, 赵玉然, 等, 2012. 松江鲈鱼cyt b基因序列分析及种质鉴定. 生物技术通报,(8): 119–124 |
| 刘云国, 2009. 水产生物DNA分子标记技术. 北京: 科学出版社, |
| 齐兴柱, 尹绍武, 娄甜甜, 等, 2007. 海南产花鳗鲡细胞色素b基因的克隆及序列分析. 海南大学学报(自然科学版), 25(4): 397–401 |
| 孙鹏, 2013. COI条形码在鳗鲡种质鉴定和系统发育研究中的应用. 福州: 福建农林大学硕士学位论文 |
| 吴宁, 2010. 几种鳗鲡苗种的种质鉴定及其遗传多样性研究. 厦门: 集美大学硕士学位论文 |
| 张殿昌, 江世贵, 邵艳卿, 等, 2006. 3种野鲮亚科鱼类线粒体细胞色素b基因序列分析及种质鉴定. 农业生物技术学报, 14(3): 446–447 |
| 陈文炳, 缪婷玉, 彭娟, 等, 2017. 基于16S rRNA基因DNA条形码鉴定美洲鳗、欧洲鳗、日本鳗. 食品科学, 38(4): 283–289 DOI:10.7506/spkx1002-6630-201704046 |
| 陈茂辉, 2008. 浅谈常见几种鳗鲡苗的鉴定. 科学养鱼,(6): 7 |
| 陈姝君, 赫崇波, 木云雷, 等, 2008. 硬骨鱼类线粒体基因系统发育信息效率分析. 中国水产科学, 15(1): 12–21 |
| 贾秀双, 2014. 基于组合PCR的近缘物种鉴定方法的研究. 青岛: 中国海洋大学硕士学位论文 |
| Aoyama J, Nishida M, Tsukamoto K, 2001. Molecular phylogeny and evolution of the freshwater eel, genus Anguilla. Molecular Phylogenetics and Evolution, 20(3): 450–459 DOI:10.1006/mpev.2001.0959 |
| Birstein V J, Doukakis P, Sorkin B, et al, 1998. Population aggregation analysis of three caviar-producing species of sturgeons and implications for the species identification of black caviar. Conservation Biology, 12(4): 766–775 DOI:10.1046/j.1523-1739.1998.97081.x |
| DeSalle R, Birstein V J, 1996. PCR identification of black caviar. Nature, 381(6579): 197–198 DOI:10.1038/381197a0 |
| Ege V, 1939. A revision of the genus Anguilla Shaw:a systematic, phylogenetic, and geographical study. Oxford: Oxford University Press, 1-256 |
| Han Y S, Chang C W, He J T, et al, 2001. Validation of the occurrence of short-finned eel Anguilla bicolor pacifica in natural waters of Taiwan. Acta Zoologica Taiwanica, 12(1): 9–19 |
| Kuroki M, Miller M J, Aoyama J, et al, 2012. Offshore spawning for the newly discovered anguillid species Anguilla luzonensis (Teleostei:Anguillidae) in the Western North Pacific. Pacific Science, 66(4): 497–507 DOI:10.2984/66.4.7 |
| Lin Y S, Poh Y P, Tzeng C S, 2001. A phylogeny of freshwater eels inferred from mitochondrial genes. Molecular Phylogenetics and Evolution, 20(2): 252–261 DOI:10.1006/mpev.2001.0969 |
| Ludwig A, Debus L, Jenneckens I, 2002. A molecular approach to control the international trade in black caviar. International Review of Hydrobiology, 87(5-6): 661–674 DOI:10.1002/1522-2632(200211)87:5/6<661::AID-IROH661>3.0.CO;2-S |
| Rehbein H, Makie I M, Pryde S, et al, 1995. Fish species identification in canned tuna by DNA analysis (PCR-SSCP). Informationen fur die Fischwirtschaft aus der Fischereiforschng, 42(4): 209–212 |
| Wade B, Jamandre B W D, Shen K N, et al, 2007. Molecular phylogeny of Philippine freshwater eels Anguilla spp. (Actinopterygi:Anguilliformes:Anguillidae) inferred from mitochondrial DNA. The Raffles Bulletin of Zoology, 14: 51–59 |
| Zardoya R, Meyer A, 1996. Phylogenetic performance of mitochondrial protein-coding genes in resolving relationships among vertebrates. Molecular Biology and Evolution, 13(7): 933–942 DOI:10.1093/oxfordjournals.molbev.a025661 |
 2018, Vol. 49
2018, Vol. 49


