中国海洋湖沼学会主办。
文章信息
- 高天翔, 王志杨, 宋娜, 宋林. 2019.
- GAO Tian-Xiang, WANG Zhi-Yang, SONG Na, SONG Lin. 2019.
- 南沙群岛密斑马面鲀群体遗传多样性分析
- STUDIES ON POPULATION GENETIC DIVERSITY OF THAMNACONUS TESSELLATUS FROM NANSHA ISLANDS OF SOUTH CHINA SEA
- 海洋与湖沼, 50(1): 159-165
- Oceanologia et Limnologia Sinica, 50(1): 159-165.
- http://dx.doi.org/10.11693/hyhz20180300061
-
文章历史
- 收稿日期:2018-03-23
- 收修改稿日期:2018-07-21
2. 烟台大学海洋学院 烟台 264005;
3. 海水养殖教育部重点实验室(中国海洋大学) 青岛 266003;
4. 辽东学院 丹东 118001
2. Ocean College, Yantai University, Yantai 264005, China;
3. Key Laboratory of Mariculture(Ocean University of China), Ministry of Education, Qingdao 266003, China;
4. Eastern Liaoning University, Dandong 118001, China
马面鲀属Thamnaconus隶属于鲀形目Tetraodontiformes、单角鲀科Monachanthidae (苏锦祥等, 2002), 俗称面包鱼、剥皮鱼, 广泛分布于印度-西太平洋热带至暖温带海域。马面鲀属区分于同科其他属的主要形态学特征为腹鳍退化为1鳍棘, 腹鳍鳍棘不能活动, 第一背鳍具2鳍棘(苏锦祥等, 2002; 陈大刚等, 2015)。马面鲀属鱼类在西北太平洋的报道过的种类有5种, 分别是绿鳍马面鲀T. modestus、黄鳍马面鲀T. hypargyreus、密斑马面鲀T. tessellatus、马面鲀T. septentrionalis、拟马面鲀T. modestoides(苏锦祥等, 2002; 陈大刚等, 2015)。其中黄鳍马面鲀和绿鳍马面鲀均为我国近海常见的重要的经济鱼类, 而密斑马面鲀Thamnaconus tessellatus (Günther, 1880)较为少见, 主要分布于西太平洋热带海域, 在我国主要分布在南海海域。密斑马面鲀区分于黄鳍马面鲀和绿鳍马面鲀的典型特征为体侧密布大量黑褐色暗色斑点、尾鳍深色无黑色边缘(苏锦祥等, 2002; 陈大刚等, 2015)。目前, 对马面鲀属鱼类的研究主要包括种的鉴定和种间系统发育关系(Wu et al, 2008; 郜星晨, 2015)及重要经济种绿鳍马面鲀(郑元甲, 1997; 陈丕茂, 2000; An et al, 2013)、黄鳍马面鲀(钱世勤, 1998; 郑元甲, 2001; 李玉芳, 2014; Wang et al, 2016)的相关研究, 而对密斑马面鲀的研究极少, 仅限于渔业群落生态学研究中的物种报道。
线粒体DNA控制区因其具有较高的种内多态性成为研究群体水平遗传多样性的理想标记(Cann et al, 1984; Liu et al, 2007)。结合Avise总结的物种谱系地理结构的五种格局(Avise, 2000), 我们发现西北太平洋海洋鱼类存在3种系统地理格局(高天翔等, 2009)。近年来, 中国海鱼类的3种谱系地理格局已得到越来越多的验证(韩志强, 2008; Ni et al, 2014)。同属近缘种有相似的生活习性, 分布区重叠或邻近, 对同海域近缘种的谱系地理格局进行比较研究, 可充分阐明海洋环境的变迁和种的生活习性对各自谱系地理格局的影响(Ahti et al, 2016)。到目前为止, 绿鳍马面鲀和黄鳍马面鲀的分子系统地理学研究已有相关报道(An et al, 2013; Wang et al, 2016), 而密斑马面鲀的研究尚属空白。本研究基于线粒体DNA控制区第一高变区片段序列对采自南海海域的密斑马面鲀群体进行了分析, 旨在揭示其遗传多样性和遗传结构现状, 推测谱系历史动态, 阐明密斑马面鲀现有谱系地理格局形成及维持机制, 以期为其资源的保护和开发提供基础资料。
1 材料与方法 1.1 样品采集本研究于2013—2015年在南海生物多样性调查中共采集密斑马面鲀3个群体, 共86个个体(图 1, 表 1)。经形态鉴定后, 将样品冷冻保存至中国海洋大学渔业生态学实验室, 并取背部肌肉于95%的酒精中保存。
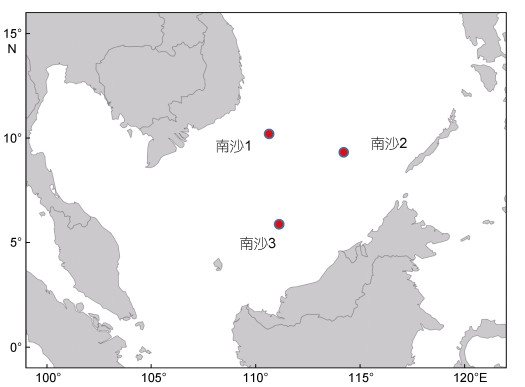 |
| 图 1 密斑马面鲀样品采集地点 Fig. 1 The sampling sites of T. tessellatus in this study |
| 群体简称 | 采样地点 | 位置 | 采样时间(年.月) | 样品数量 | 单倍型数 | 独享单倍型数 | 变异位点 | 单倍型多样度(h) | 核苷酸多样度(π) |
| NS1 | 南沙1 | 110°40′, 10°11′ | 2013.04 | 25 | 13 | 3 | 24 | 0.89±0.04 | 0.0102±0.0058 |
| NS2 | 南沙2 | 114°12′, 9°18′ | 2013.04 | 35 | 16 | 4 | 27 | 0.91±0.03 | 0.0116±0.0064 |
| NS3 | 南沙3 | 111°11′, 5°54′ | 2015.03 | 26 | 16 | 8 | 30 | 0.92±0.04 | 0.0132±0.0072 |
| 共计 | 86 | 28 | 15 | 38 | 0.91±0.02 | 0.0117±0.0064 |
采用传统酚-氯仿方法提取密斑马面鲀基因组DNA, 用超纯水将乙醇沉淀后的DNA溶解, –20℃保存备用。
1.3 PCR扩增与产物测序本研究中采用引物DL-S(5′-CCCACCACTA ACTCCCAAAGC′)和DL-R(5′-CTGGAAAGAACGC CCG GCATG-3′)对密斑马面鲀的线粒体DNA(mtDNA)控制区的高变区(Hypervariable region Ⅰ; HVRI)进行扩增。纯化、回收后的PCR产物送上海桑尼生物科技有限公司采用ABIPrism 3700自动测序仪进行双向测定。
1.4 数据分析测序结果使用DNASTAR软件进行编辑、比对, 去除引物序列, 对变异位点逐一核对, 截去5′端tRNA序列28bp, 得到423bp的线粒体控制区5′端片段。所有样本对应的序列经Mega 7.0 (Kumar et al, 2016)软件中的MUSCLE算法初步比对后, 进行人工校对。在DnaSP软件中统计简约信息位点、多态位点等的数量并定义单倍型, 根据单倍型所含个体归属群体的情况, 将所有单倍型分为群体独享单倍型和共享单倍型, 统计群体独享单倍型数, 共享单倍型依次按所含个体数、个体归属的群体数、群体自北向南的位置、实验样本号的次序编号整理。通过jModelTest (Posada, 2008)软件基于相关标准筛选出核苷酸最适模型, 利用Arlequin ver 3.5软件(Excoffier et al, 2010)计算了序列多样性指数包括单倍型多样度、核苷酸多样度、两两配对核苷酸差异平均值、转换位点数、颠换位点数等。
通过Arlequin软件计算两两群体间的ΦST值、两两群体间的净两两核苷酸差异平均值(以下简称“净核苷酸差异均值”)及群体内两两核苷酸差异平均值(以下简称“群体内核苷酸差异均值”), 对P值进行Bonferroni矫正, 并进行分子方差分析(AMOVA), 均置换检验10000次。由于本研究中密斑马面鲀群体间地理距离较近, 为检测群体间是否可进行随机交配产生基因流, 采用Arlequin软件中确切P检验(Raymond et al, 1995)对密斑马面鲀的群体进行检测。
通过Arlequin 3.5软件计算核苷酸不配对分布(mismatch distribution), 分别模拟群体数量(demographic / sudden expansion)和空间扩张(range / spatial expansion)。运用BEAST 1.8软件(Drummond et al, 2007)中贝叶斯天际线(Bayesian skyline plot; BSP)检测群体历史群体大小变动。
2 结果本研究中86尾密斑马面鲀的线粒体控制区序列的变异位点数为38个(表 1), 占序列总长的8.98%, 其中, 简约信息位点29个, 插入缺失位点2个、转换位点33个、颠换位点4个。密斑马面鲀的控制区序列的变异程度较大, 检测到了大量的简约信息位点和部分个体独享位点(9个), 插入缺失位点少, 转换颠换比较大, 多重置换发生较少, 适用于分子系统地理学研究。
密斑马面鲀序列对应着28个单倍型(表 2), 其中, 群体共享单倍型为13个, 群体独享单倍型为15个。群体共享单倍型共包含68尾密斑马面鲀的序列, 占总样本数的79.07%, 单倍型SH1包含的个体序列数最多, 为19条, 包含的个体涉及每个群体, 且在群体内的数量所占比例也较均衡, 分别为24.00%、17.14%、26.92%, 其次是单倍型SH2, 包含的个体序列数为16条, 主要分布于南沙2号和1号群体内, 其它共享单倍型包含的个体序列数都≤5条。除南沙3号群体的两个群体独享单倍型分别包含3条、2条个体序列外, 其余群体独享单倍型均仅包含1个个体的序列。在3个密斑马面鲀群体中, 单倍型多样度和核苷酸多样度相差不大, 总体的单倍型多样度为0.91±0.02, 核苷酸多样度为0.0117±0.0064, 单倍型多样度和核苷酸多样度均较高。群体内变异位点数为24—30个, 总体的变异位点数为38个。
| 共享单倍型 | 个体数 | NS1 | NS2 | NS3 |
| SH1 | 19 | 6 | 6 | 7 |
| SH2 | 16 | 6 | 8 | 2 |
| SH3 | 5 | 3 | 1 | 1 |
| SH4 | 4 | 1 | 3 | |
| SH5 | 4 | 3 | 1 | |
| SH6 | 4 | 3 | 1 | |
| SH7 | 3 | 1 | 2 | |
| SH8 | 3 | 1 | 1 | 1 |
| SH9 | 2 | 1 | 1 | |
| SH10 | 2 | 1 | 1 | |
| SH11 | 2 | 1 | 1 | |
| SH12 | 2 | 1 | 1 | |
| SH13 | 2 | 1 | 1 | |
| 共计 | 68 | 22 | 31 | 15 |
| NS1 | NS2 | NS3 | ||
| NS1 | 4.3046 | –0.0454 | 0.0012 | |
| NS2 | –0.0106 | 4.8973 | 0.0874 | |
| NS3 | 0.0001 | 0.0173 | 5.4834 | |
| 注:净核苷酸差异均值(右上)、群体内核苷酸差异均值(左上至右下的对角线上)及ΦST值(左下) | ||||
贝叶斯聚类分析(图 2)建议将所有单倍型分为2个类群, 此时边缘似然对数值最大, 为–332.8933, 但自大而小第2—5位的边缘似然对数值及类群数分别为–342.0236和3个类群、–355.6126和4个类群、–370.1723和5个类群、–371.3459和5个类群, 均差异较小, 需根据单倍型网络图、贝叶斯树和最大似然树再慎重考虑分组情况。
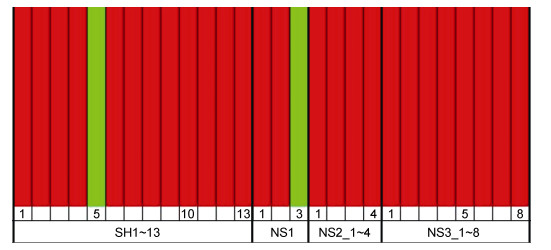 |
| 图 2 密斑马面鲀单倍型的贝叶斯种群镶嵌图 Fig. 2 The Bayesian clustering analysis of T. tessellatus haplotypes |
单倍型网络图(图 3)呈明显的星状分布, 主体中央单倍型为SH2, 仅单倍型SH5、NS1_3与其他相邻单倍型间的差异大于5bp, 其余各相邻单倍型间的核苷酸差异≤3bp, 未检测到与分布显著相关的谱系结构。
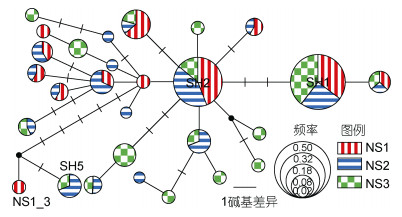 |
| 图 3 密斑马面鲀单倍型网络图 Fig. 3 The haplotype median-joining network of T. tessellatus |
贝叶斯树和最大似然树均显示出单倍型间松散的分布, 形成了大量多岐分支, 尤以贝叶斯树为甚(图 4)。密斑马面鲀的单倍型在贝叶斯树和最大似然树上仅能形成后验概率和支持率均较高的1—4个单倍型组成的分支, 未检测到显著的谱系结构。根据单倍型网络图、贝叶斯树和最大似然树结果, 在密斑马面鲀种内未检测到显著的谱系结构, 3个群体为1个谱系, 单倍型分支松散, 种内可能发生过群体扩张事件, 单倍型SH2可能是主要的祖先单倍型。
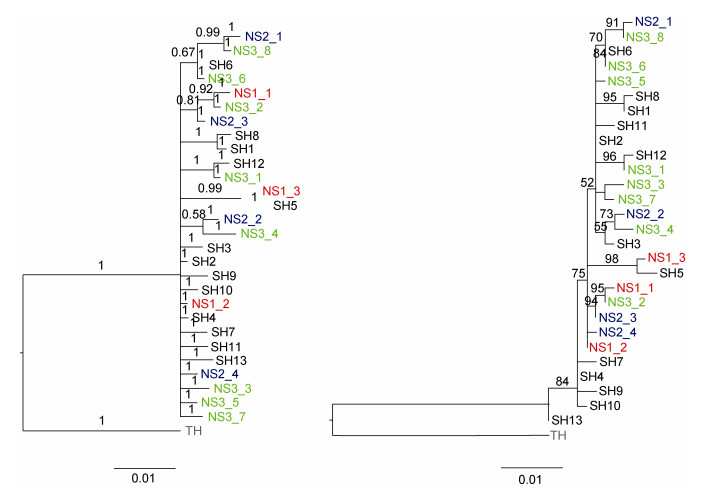 |
| 图 4 密斑马面鲀单倍型贝叶斯树(左)和最大似然树(右) Fig. 4 The Bayesian inference tree (left) and maximum likelihood tree (right) of T. tessellatus |
南沙的密斑马面鲀谱系的Tajima检验D值和符氏检验值均为负值, 分别为–1.2272和–10.5571, 且符氏检验的P值为0.0010, 为极显著, 故谱系序列的进化拒绝中性假说, 谱系可发生过群体扩张等事件。密斑马面鲀的核苷酸不配对分布(图 5)接近单峰型, Harpending偏离指数为0.0169且不显著, 尚未显著偏离单峰分布。空间扩张模型与观测值的拟合程度更佳, 空间扩张模型的方差总和(0.0025)显著小于种群扩张模型的方差总和(0.0049), 且统计检验均未显著偏离模型。空间扩张模型估算的τ值为3.56, 按控制区3%—10%每百万年的分歧速率推算出密斑马面鲀谱系扩张的时间约为28.19万年前至8.46万年前。
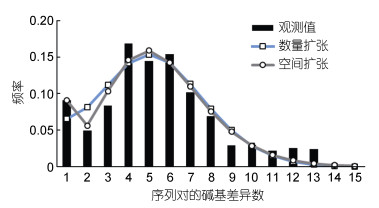 |
| 图 5 密斑马面鲀核苷酸不配对分布 Fig. 5 The mismatch distribution analysis of T. tessellatus |
贝叶斯天际线分析(图 6)表明密斑马面鲀谱系自约(150—500)万年前有效群体大小显著增长, 在约(120—400)万年前增速显著减缓并趋于稳定(采用控制区3%—10%每百万年的分歧速率)。
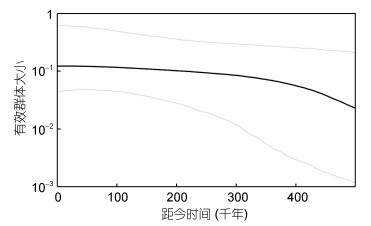 |
| 图 6 密斑马面鲀贝叶斯天际线分析 Fig. 6 The Bayesian skyline analysis of T. tessellatus |
物种的遗传多样性是长期进化的产物, 是其生存、发展和进化的前提(Laikre et al, 2005)。本研究中密斑马面鲀序列的单倍型多样度和核苷酸多样度均处于较高水平, 表明南沙群岛的密斑马面鲀具较高的遗传多样性暗示密斑马面鲀可能具有大而稳定的有效群体大小。这一结果也与已报道的绿鳍马面鲀(An et al, 2013)和黄鳍马面鲀(Wang et al, 2016)相似, 三种马面鲀属鱼类皆具有较高的遗传变异性, 这也暗示其可能具有较高的进化潜力, 对环境改变响应的进化能力也较强。另外, 密斑马面鲀作为一种尚未重点开发的经济种类, 经受渔业捕捞压力小, 群体的遗传多样性高, 因此可考虑适度开发利用其资源。
更新世冰期剧烈的气候变化对很多物种的分布范围造成了巨大的影响(Avise, 2000; Liu et al, 2007)。本研究中密斑马面鲀的群体历史动态分析说明其经历了更新世冰期后有效群体数量增大的历史事件, 更新世冰期海平面下降、温度骤降和黑潮势弱等因素可能导致密斑马面鲀的栖息地面积骤减, 甚至在冰盛期南海南部陆架已露出海面(Wang, 1999), 现今南海南部的密斑马面鲀显然是冰期后扩张重殖的群体。冰期海水温度的降低使密斑马面鲀的栖息环境更局限于赤道附近海域, 但尚不能推测出密斑马面鲀群体避难所的具体位置, 后续的研究须采集密斑马面鲀分布范围内的连续多个群体并基于多标记手段对其谱系地理结构进行更详尽的分析。
密斑马面鲀群体间未检测到显著的遗传结构, 整体的分子方差分析结果表明群体内的遗传差异显著大于群体间, 确切P检验的结果也暗示3个群体间可产生随机的基因流。本研究中的3个群体为1个可随机交配的种群, 可能主要由南海南部开阔的海域环境、复杂的环流系统(Liu, 2013)和马面鲀属鱼类较强的活动能力(苏锦祥等, 2002; 陈大刚等, 2015)等因素造成。目前对密斑马面鲀的生物学研究比较缺乏, 但系统发育分析的结果表明黄鳍马面鲀与密斑马面鲀亲缘关系较近, 在西北太平洋海域的重叠可能北至东海东部、南至赤道海域, 同域性极强(李玉芳, 2014), 因此密斑马面鲀呈现出与黄鳍马面鲀相似的系统地理格局(Wang et al, 2016)。同属的绿鳍马面鲀为暖温性鱼类, 虽然不同地理群体间也不存在显著的遗传结构, 但系统发育上具有较大分化, 与密斑马面鲀和黄鳍马面鲀呈现不同的系统地理格局。更新世冰期过后, 密斑马面鲀的避难群体发生栖息地扩张和有效群体大小的增长, 且不同群体间存在显著的基因流, 是其未形成显著的谱系结构的主要原因。许多高扩散性的鱼类如竹荚鱼Trachurus japonicus (Song et al, 2014)等也存在相同的谱系地理结构, 更新世冰期的海洋环境特征、冰期后东海和南海环境的演变和生活史特征在不同种谱系地理格局的形成中扮演了重要的作用。
4 结论本研究基于线粒体DNA控制区第一高变区序列对南沙群岛的3个密斑马面鲀群体进行了遗传学分析, 研究结果提示南沙群岛的密斑马面鲀群在遗传管理上应作为一个管理单元进行管理。较高的遗传多样度水平提示我们可考虑适度开发利用其资源。
| 苏锦祥, 李春生, 2002. 中国动物志硬骨鱼纲鲀形目海蛾鱼目喉盘鱼目鮟鱇目. 北京: 科学出版社 |
| 李玉芳, 2014.南海黄鳍马面鲀(Thamnaconus hypargyreus)种群分析.上海: 上海海洋大学硕士学位论文 |
| 陈大刚, 张美昭, 2015. 中国海洋鱼类. 青岛: 中国海洋大学出版社 |
| 陈丕茂, 詹秉义, 2000. 绿鳍马面鲀年龄生长与合理利用. 中国水产科学, 7(1): 35–40 DOI:10.3321/j.issn:1005-8737.2000.01.008 |
| 郑元甲, 1997. 东海区马面鲀资源变动及其原因的初步研究. 中国水产科学, 4(4): 18–24 DOI:10.3321/j.issn:1005-8737.1997.04.004 |
| 郑元甲, 李圣法, 宓崇道, 等, 2001. 东海与南海的黄鳍马面鲀种群研究. 中国水产科学, 8(1): 46–52 DOI:10.3321/j.issn:1005-8737.2001.01.011 |
| 郜星晨, 2015.基于线粒体COI序列的中国沿海鲀形目鱼类DNA条形码研究.广州: 暨南大学硕士学位论文 |
| 钱世勤, 1998. 东海黄鳍马面鲀生物学特性和资源利用状况. 中国水产科学, 5(3): 25–29 DOI:10.3321/j.issn:1005-8737.1998.03.005 |
| 高天翔, 任桂静, 刘进贤, 等, 2009. 海洋鱼类分子系统地理学研究进展. 中国海洋大学学报, 39(5): 897–902 |
| 韩志强, 2008.三种海洋鱼类分子系统地理学研究.青岛: 中国海洋大学博士学位论文 |
| Ahti P A, Coleman R R, DiBattista J D, et al, 2016. Phylogeography of Indo-Pacific reef fishes:sister wrasses Coris gaimard and C. cuvieri in the Red Sea, Indian Ocean and Pacific Ocean. Journal of Biogeography, 43(6): 1103–1115 DOI:10.1111/jbi.12712 |
| An H, Lee J, Park J, et al, 2013. Genetic structure of the Korean black scraper Thamnaconus modestus inferred from microsatellite marker analysis. Molecular Biology Reports, 40(5): 3445–3456 DOI:10.1007/s11033-012-2044-7 |
| Avise J C, 2000. Phylogeography:the History and Formation of Species. Harvard: Harvard University Press, |
| Cann R L, Brown W M, Wilson A C, 1984. Polymorphic sites and the mechanism of evolution in human mitochondrial DNA. Genetics, 106(3): 479–499 |
| Drummond A J, Rambaut A, 2007. BEAST:Bayesian evolutionary analysis by sampling trees. BMC Evolutionary Biology, 7(1): 214 DOI:10.1186/1471-2148-7-214 |
| Excoffier L, Lischer H E L, 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10(3): 564–567 DOI:10.1111/men.2010.10.issue-3 |
| Kumar S, Stecher G, Tamura K, 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 33(7): 1870–1874 DOI:10.1093/molbev/msw054 |
| Laikre L, Palm S, Ryman N, 2005. Genetic population structure of fishes:implications for coastal zone management. Ambio, 34(2): 111–119 DOI:10.1579/0044-7447-34.2.111 |
| Liu J X, Gao T X, Wu S F, et al, 2007. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish, Chelon haematocheilus (Temminck & Schlegel..1845). Molecular Ecology, 16(2): 275–288 |
| Liu J Y, 2013. Status of marine biodiversity of the China Seas. PLoS One, 8(1): e50719 DOI:10.1371/journal.pone.0050719 |
| Ni G, Li Q, Kong L F, et al, 2014. Comparative phylogeography in marginal seas of the northwestern Pacific. Molecular Ecology, 23(3): 534–548 |
| Posada D, 2008. jModelTest:phylogenetic model averaging. Molecular Biology and Evolution, 25(7): 1253–1256 DOI:10.1093/molbev/msn083 |
| Raymond M, Rousset F, 1995. An exact test for population differentiation. Evolution, 49(6): 1280–1283 DOI:10.1111/evo.1995.49.issue-6 |
| Song N, Jia N, Yanagimoto T, et al, 2013. Genetic differentiation of Trachurus japonicus from the Northwestern Pacific based on the mitochondrial DNA control region. Mitochondrial DNA, 24(6): 705–712 DOI:10.3109/19401736.2013.773982 |
| Wang P X, 1999. Response of Western Pacific marginal seas to glacial cycles:paleoceanographic and sedimentological features. Marine Geology, 156(1-4): 5–39 DOI:10.1016/S0025-3227(98)00172-8 |
| Wang Z Y, Zhang Y, Zhao L L, et al, 2016. Shallow mitochondrial phylogeographical pattern and high levels of genetic connectivity of Thamnaconus hypargyreus in the South China Sea and the East China Sea. Biochemical Systematics and Ecology, 67: 110–118 DOI:10.1016/j.bse.2016.05.013 |
| Wu Y J, Hsieh C H, Chen H M, et al, 2008. Identification of six common species of processed filefish using cytochrome b gene sequence and PCR-RFLP analysis. The Raffles Bulletin of Zoology, 19: 151–158 |
 2019, Vol. 50
2019, Vol. 50


