
中国海洋湖沼学会主办。
文章信息
- 李海涛, 周鹏, 何静, 黄彬彬, 陈凯彪. 2019.
- LI Hai-Tao, ZHOU Peng, HE Jing, HUANG Bin-Bin, CHEN Kai-Biao. 2019.
- 基于形态学和16S rRNA基因序列的轮螺科面盘幼虫物种鉴定
- SPECIES IDENTIFICATION OF ARCHITECTONICID VELIGER LARVAE BASED ON MORPHOLOGICAL AND MITOCHONDRIAL 16S rRNA GENE
- 海洋与湖沼, 50(1): 228-235
- Oceanologia et Limnologia Sinica, 50(1): 228-235.
- http://dx.doi.org/10.11693/hyhz20180700166
-
文章历史
- 收稿日期:2018-07-07
- 收修改稿日期:2018-09-12
物种的分类学特征通常基于成体来描述。因此, 对具有复杂生活史的海洋生物来说, 依据形态特征来鉴定其幼体或幼虫是极为困难的(Pradillon et al, 2007; Puillandre et al, 2009; Ko et al, 2013)。这是因为近缘种的幼体在形态上十分相似不易区分, 而且我们对于海洋无脊椎动物的幼体也缺乏了解(Levin, 1990)。基于分子生物学的物种鉴定技术(如DNA条形码)克服了对形态学的过度依赖(林森杰等, 2014), 能实现对不同发育阶段标本的鉴定(Barber et al, 2006; Puillandre et al, 2009), 是传统分类学和物种鉴定的重要补充。
自然界中绝大多数腹足类的螺壳是右旋的(dextral), 螺壳左旋(sinistral)的种类较少。螔螺科(Limacinidae)的螔螺属(Limacina)种类为中国海常见的螺壳左旋的腹足纲浮游动物。这是一类个体微小, 终生营浮游生活的腹足类。Tesch(1946)将一类与螔螺属具有相似左旋螺壳的浮游软体动物命名为强卷螺属(Agadina), 归到螔螺科之下。同时, Tesch(1946)也认识到Agadina并非为真正的翼足类(Pteropod), 推测这类动物可能是小塔螺科(Pyramidellidae)齿口螺属(Odostomia)种类的浮游幼虫。张福绥(1964)沿用了Tesch(1946)的观点, 将强卷螺属附于螔螺科之下。后续的研究表明, Tesch(1946)所描述的强卷螺实为成体营底栖生活的轮螺科(Architectonicidae)的面盘幼虫(Robertson, 1964; Bieler, 1993)。然而, 一些国内研究资料(徐兆礼, 2008; 罗文增等, 2016)仍将轮螺科的面盘幼虫记为强卷螺。
轮螺科是一类在浮游面盘幼虫期胚壳左旋而成体期螺壳右旋的特殊腹足类(Bieler, 1993)。鉴于轮螺科面盘幼虫左旋的螺壳极易识别, Robertson(1964)甚至认为仅通过面盘幼虫的形态特征即可将一些个体鉴定到属或种的水平。然而, 轮螺科种类较长的浮游幼体期(Robertson, 1964; Robertson et al, 1970)以及繁多的种类(Bieler, 1993), 都增加了面盘幼虫的鉴定难度。另外, 国内对轮螺科的分类学研究也极其不足, 种类的组成信息还不够完善。本研究分析了南海及其周边海域轮螺科面盘幼虫的形态特征, 同时结合线粒体16S RNA基因序列对其进行了物种鉴定, 更正了国内长期以来的分类错误, 研究结果可为今后轮螺科的分类学研究提供更多的基础资料。
1 材料和方法 1.1 实验材料实验样品于2014—2017年采自南海及其周边海域(图 1), 采样区域大致可分为南海北部和吕宋海峡海域(N区)及南海中南部海域(S区)。使用大型浮游生物网(网径0.505mm), 采用垂直拖网和水平拖网相结合的方式采集浮游动物样品, 样品经无水酒精固定后冰冻保存带回实验室, 体视镜下完成轮螺面盘幼虫的分选, 对每个个体进行编号并拍照。选取66个轮螺面盘幼虫(AL1—AL66)用于形态学和DNA序列分析, 同时选取了3个采自南海北部的配景轮螺(Architectonica perspectiva)成体(AD1—AD3)用于基因序列的分析, 标本的具体信息见表 1。
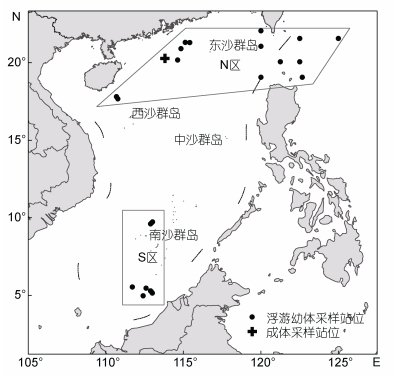 |
| 图 1 采样站点分布图 Fig. 1 Map of the sampling sites |
| 单倍型 | 样品编号 | 采样区域 | GenBank序列号 | 序列长度(bp) |
| H1 | AL1 | N区 | MH557974 | 384 |
| H2 | AL2、AL43 | N区 | MH557975 | 379 |
| H3 | AL3、AL4、AL18、AL50 | N区 | MH557976 | 391 |
| H4 | AL5 | N区 | MH557977 | 389 |
| H5 | AL6 | N区 | MH557978 | 387 |
| H6 | AL7 | N区 | MH557979 | 390 |
| H7 | AL8 | N区 | MH557980 | 403 |
| H8 | AL9 | N区 | MH557981 | 391 |
| H9 | AL10 | N区 | MH557982 | 391 |
| H10 | AL11 | N区 | MH557983 | 391 |
| H11 | AL12 | N区 | MH557984 | 382 |
| H12 | AL13 | N区 | MH557985 | 391 |
| H13 | AL14 | N区 | MH557986 | 388 |
| H14 | AL15 | N区 | MH557987 | 391 |
| H15 | AL16、AL44 | N区、S区 | MH557988 | 391 |
| H16 | AL17 | N区 | MH557989 | 380 |
| H17 | AL19 | N区 | MH557990 | 389 |
| H18 | AL20 | N区 | MH557991 | 392 |
| H19 | AL21、AL23、AL34、AL48 | N区、S区 | MH557992 | 389 |
| H20 | AL22 | N区 | MH557993 | 389 |
| H21 | AL24 | S区 | MH557994 | 390 |
| H22 | AL25 | S区 | MH557995 | 384 |
| H23 | AL26 | S区 | MH557996 | 383 |
| H24 | AL27 | S区 | MH557997 | 383 |
| H25 | AL28 | S区 | MH557998 | 383 |
| H26 | AL29 | S区 | MH557999 | 383 |
| H27 | AL30 | S区 | MH558000 | 387 |
| H28 | AL31 | S区 | MH558001 | 383 |
| H29 | AL32、AL33、AL35、AL39、AL52、AD1—AD3 | N区、S区 | MH558002 | 387 |
| H30 | AL36 | S区 | MH558003 | 386 |
| H31 | AL37 | S区 | MH558004 | 387 |
| H32 | AL38 | S区 | MH558005 | 387 |
| H33 | AL40 | S区 | MH558006 | 386 |
| H34 | AL41 | S区 | MH558007 | 386 |
| H35 | AL42、AL58、AL60、AL63、AL64 | N区、S区 | MH558008 | 391 |
| H36 | AL45 | N区 | MH558009 | 389 |
| H37 | AL46 | N区 | MH558010 | 381 |
| H38 | AL47 | N区 | MH558011 | 387 |
| H39 | AL49 | N区 | MH558012 | 390 |
| H40 | AL51 | N区 | MH558013 | 389 |
| H41 | AL53 | N区 | MH558014 | 391 |
| H42 | AL54 | N区 | MH558015 | 391 |
| H43 | AL55 | S区 | MH558016 | 391 |
| H44 | AL56 | S区 | MH558017 | 392 |
| H45 | AL57 | S区 | MH558018 | 391 |
| H46 | AL59 | S区 | MH558019 | 383 |
| H47 | AL61 | S区 | MH558020 | 391 |
| H48 | AL62 | S区 | MH558021 | 389 |
| H49 | AL65 | S区 | MH558022 | 391 |
| H50 | AL66 | S区 | MH558023 | 389 |
| 注: AL1—AL66为面盘幼虫编号; AD1—AD3为配景轮螺(A. perspectiva)成体编号; N区指南海北部和吕宋海峡海域; S区指南海中南部海域 | ||||
采用碱裂解法(李海涛等, 2015)提取DNA, 提取幼虫DNA时对溶液体积稍作改动, NaOH和Tris-Cl的使用体积分别减少为45μL和5μL。首先使用引物LCO-1490(5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′)和HCO-2198(5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′)(Folmer, 1994)对线粒体COI基因片段进行扩增, 但成功率较低。因此最终使用引物16sar-L(5′-CGC CTG TTT ATC AAA AAC AT-3′)和16sbr-H(5′-CCG GTC TGA ACT CAG ATC ACG T-3′) (Simon et al, 1994)扩增16S rRNA基因片段。
PCR反应体系总体积为50μL, 包括: 2×buffer 25μL, dNTP 400μmol/L, 引物各0.3μmol/L, KOD FX高保真PCR酶(TOYOBO公司)1.0 U, 模板DNA1 μL, 加去离子水补足至50μL。PCR反应条件为: 94℃预变性2min; 98℃变性15s, 53℃退火45s, 68℃延伸90s, 共35个循环; 最后68℃延伸10min。PCR产物经1%琼脂糖凝胶电泳检测后回收纯化, 然后进行双向测序, 测序引物与扩增引物相同。
1.3 数据分析拼接、校对后的测序结果(单倍型序列), 连同GenBank中下载的轮螺科16S rRNA基因序列, 在BioEdit软件中进行多重序列比对。使用MEGA 7计算碱基的组成, 基于Kimura 2-Parameter (K2P)参数模型计算遗传距离, 并采用邻接法(neighbor-joining)以Micromelo undatus (DQ093487)为外群构建系统进化树, 可靠性经过1000次自展(bootstrap)检验, 支持率大于70%的在系统树中标出。
同时采用GMYC(Generalized Mixed Yule Coalescent)模型(Fujisawa et al, 2013)和ABGD (Automatic Barcoding Gap Discovery)方法(Puillandre et al, 2012)进行分子可操作分类单元(molecular operational taxonomic units, MOTUs)估计。
1.3.1 GMYC模型在分析之前, 将外群移除; 利用BEAST v1.10.1(Drummond et al, 2007)构建超度量系统发育树; 采用uncorrelated log-normal clock模型, 1×108次MCMC(Markov Chain Monte Carlo)分析, 1000次burn in; 利用BEAST软件包中的TreeAnnotator分析超度量系统发育树, 并生成最大谱系置信树; 随后用Figtree v1.4.3将最大谱系置信树转化为APE(Analyses of Phylogenetics and Evolution)识别的格式(Newick); 最后利用R环境中的“SPLITS”软件包进行单阈值GMYC物种界定(Fujisawa et al, 2013), 利用gmyc函数获得零模型与GMYC模型之间的似然比值、种-种群的转折点, 以及转折点对应的MOTU的数量。
1.3.2 ABGD方法样本在线提交至ABGD网站(http://wwwabi.snv.Jussieu.fr./pubilc/abgd/abgdweb.html)进行划分, 所有参数使用网络版本的默认值。
2 结果 2.1 形态特征轮螺科面盘幼虫胚壳左旋, 壳面光滑, 无雕刻或生长纹。壳宽0.8—1.2mm。螺层约3层。壳口近圆形, 多数个体的边缘外翻, 形成卷边。壳淡琥珀色, 半透明, 软体部可见, 壳口和脐孔处常呈褐色。具厣, 角质状。根据壳顶和脐孔的形态特征, 轮螺科面盘幼虫可分为两种明显不同的形态类型(Morphortype)。形态类型Ⅰ(图 2a—图 2c):壳扁, 壳顶凹陷(图 2c); 脐孔规则, 形成圆而深的空腔(图 2b); 缝合线颜色不明显(图 2a)或呈淡褐色。形态类型Ⅱ(图 2d—图 2i):壳顶明显凸起(图 2f); 脐孔不规则, 浅或深(图 2e, 图 2g—图 2i); 部分个体脐孔处有一肛区龙骨(Anal Keel, 图 2e箭头示), 部分个体退化甚至完全缺失(图 2g); 缝合线大多呈明显的褐色(图 2d)。图 2h所示这一新的脐孔形态类型较为少见, 仅发现4个标本。从腹面观察(图 2b, 图 2e, 图 2g—图 2i), 可见软体部的黑色幼体器官(Black Larval Organ) (Bieler, 1993), 其位置和大小因个体而异。
 |
| 图 2 轮螺科浮游面盘幼虫的贝壳形态(比例尺=500μm) Fig. 2 Shell morphology of architectonicid veliger larvae (Scale bars=500μm) 注: a—c, 形态类型Ⅰ的贝壳形态; d—i, 形态类型Ⅱ的贝壳形态; a—b, 单倍型H5对应的标本; c, 单倍型H13对应的标本; d—e, 单倍型H2对应的标本(e中箭头示肛区龙骨); f—g, 单倍型H11对应的标本; h, 单倍型H19对应的标本; i, 单倍型H27对应的标本 |
16sar-L和16sbr-H引物能扩增所有轮螺科面盘幼虫和成体的16S rRNA基因。69条16S rRNA基因序列共定义了50种单倍型(H1—H50, GenBank登录号MH557974—MH558023), 其中6种为共享单倍型, 3个配景轮螺成体与5个面盘幼虫属同一单倍型H29(表 1)。单倍型长度为379—403bp(不含引物区), A、T、G、C碱基的含量分别为33.8%、33.1%、17.9%和15.2%, A+T的含量明显高于G+C的含量。
2.3 分子可操作分类单元估计本文获得的50种单倍型和3条来自GenBank的16S rRNA基因序列, 采用GMYC模型和ABGD方法可划分为相同的21个MOTUs, 本文仅显示GMYC模型划分的结果(图 3)。其中, 本文的50种单倍型和Psilaxis radiates(AY081999)形成19个MOTUs, 另外两条GenBank中的序列各自形成一个独立的MOTU。GMYC模型和ABGD方法划分的MOTUs与形态鉴定的结果略有不同, MOTU08和MOTU09(FJ917251)同属一个形态种配景轮螺。
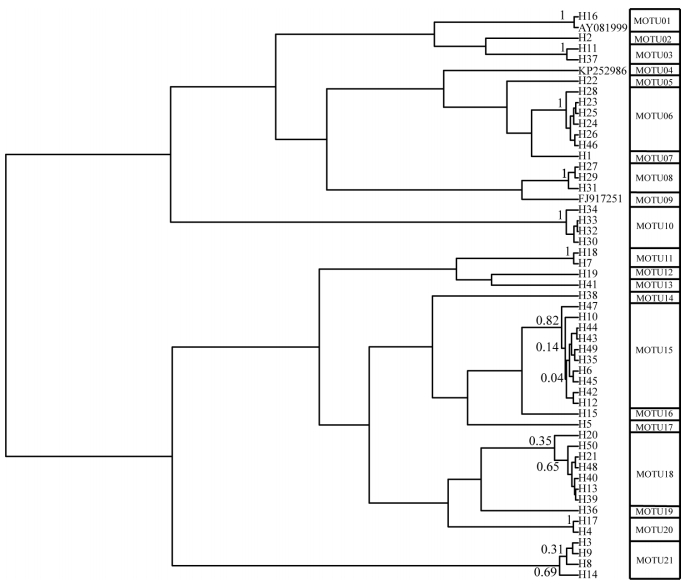 |
| 图 3 基于GYMC模型划分的MOTUs结果 Fig. 3 MOTUs revealed in GYMC model |
用本文获得16S rRNA基因的单倍型序列与GenBank中轮螺科的序列一起构建NJ系统进化树(图 4)。需要指出的是, 由于16S rRNA基因片段较短以及单个基因的缘故, 系统树可能无法反映各单倍型或物种间真实的系统发育关系, 故轮螺科的系统发育关系不是本文的研究内容。
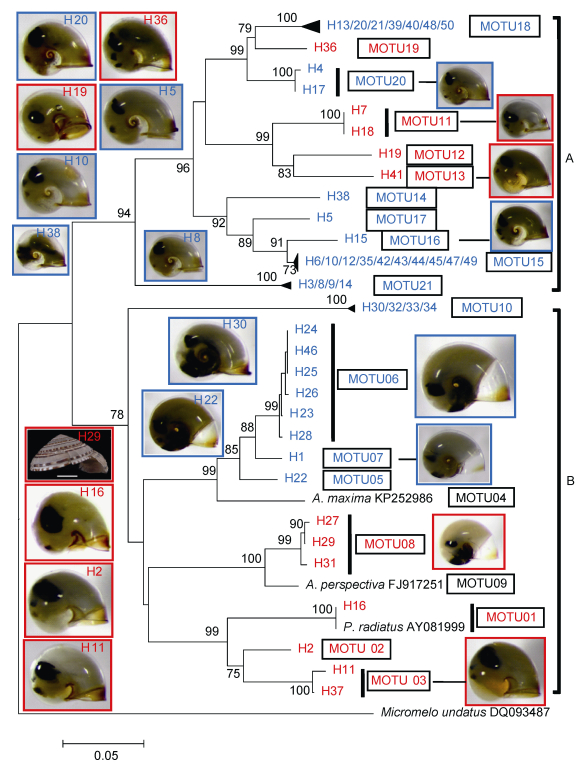 |
| 图 4 基于轮螺科面盘幼虫线粒体16S rRNA基因构建的系统发育树 Fig. 4 The neighbor-joining tree based on mitochondrial 16S rRNA gene sequences of architectonicid veliger larvae 注:蓝色单倍型编号、MOTUs编号和图片对应于形态类型Ⅰ的面盘幼虫; 红色单倍型编号、MOTUs编号和图片对应于形态类型Ⅱ的面盘幼虫和成体 |
16S rRNA基因的不同单倍型在系统树(图 4)中形成2个分支(A和B), 并获得较高的置信值。分支A包括MOTUs 11—21, 分支B包括MOTUs 1—10。从面盘幼虫的形态特征来看, 11个MOTUs属于形态类型Ⅰ; 8个MOTUs属于形态类型Ⅱ。形态类型Ⅱ的标本中, 仅MOTUs 1—3的部分个体存在肛区龙骨, 这3个MOTUs聚为一支且具有很高的支持率(99%), 其余MOTUs的个体不具有这个结构。形态类型Ⅱ中具有特殊脐孔形态(图 2h)的4个个体, 共享单倍型H19。MOTU08和MOTU05—07的标本分属不同的形态类型, 但它们却与同属(Architectonica)的2个种分别聚为一支。
从腹面观察, 软体部的黑色幼体器官的位置和大小在不同的MOTUs中存在明显差异。分支B中形态类型Ⅰ的个体(除MOTU07外), 在脐孔区存在一较大的黑色幼体器官, 而分支A中相同贝壳形态的个体则全部没有(图 4)。MOTU08的脐孔区也存在较大的黑色幼体器官, 而与之同属一个形态类型的MOTU01—03的标本中则未见(图 4)。MOTU05—07与大轮螺(A. maxima)聚为一支(支持率99%), 且这3个MOTUs的序列相似性较高, MOTU07由于在脐孔区不存在黑色幼体器官而与MOTU05和MOTU06能区分开来。这也从形态上证实了单倍型H1被独立划分为一个MOTU的合理性。MOTU14(单倍型H38)标本采样时可能已经死亡, 其肌肉和内脏组织已降解并在壳内形成空腔, 其黑色幼体器官无法辨认(图 4)。
由于GenBank中轮螺科种类的参考序列极少, 仅单倍型H16能确定为Psilaxis radiates, 该单倍型与参考序列(登录号AY081999)仅存1个碱基的差异。同时, 根据配景轮螺成体的序列, 可将MOTU08确定为配景轮螺, 但MOTU08的3种单倍型与GenBank中已有的来自澳大利亚的配景轮螺序列(FJ917251)存在较大的遗传差异。本文获得的配景轮螺3种单倍型遗传距离在0.003—0.009之间, 而它们与参考序列的遗传距离在0.046—0.049之间。轮螺科面盘幼虫MOTUs内的遗传距离在0—0.024之间, 平均值为0.005;各MOTUs间的遗传距离则在0.033—0.359之间, 平均值为0.237。MOTUs内和MOTUs间的遗传距离差异不明显。
3 讨论轮螺成体和面盘幼虫的线粒体16S rRNA基因序列分析结果表明, 这类螺壳左旋的标本属于轮螺科浮游幼虫, 而非营终生浮游的其他腹足类。在16S rRNA基因系统树中, 同一MOTU内的个体呈现相同形态特征, 这表明面盘幼虫的壳顶、脐孔和缝合线颜色这些贝壳特征, 以及黑色幼体器官的位置和大小这些软体部特征, 可作为区分轮螺科不同属或种的分类依据。肛区龙骨具有一定的不稳定性, 同属单倍型H2的2个个体, 肛区龙骨的发达程度不同(图 2和图 4); 同属MOTU03的单倍型H11和H37, 前者肛区龙骨完全消失, 而后者具有微弱的肛区龙骨(图 2和图 4)。同种的面盘幼虫具有相同的形态特征, 但不同种的面盘幼虫也可能具有相同的形态特征, 如MOTU15—18的个体(形态类型Ⅰ)在形态上就难以区分。
Robertson(1964)基于贝壳形态特征、个体大小、地理分布等多方面的因素考虑, 认为Tesch(1946)所记录的“Agadina sp.”就是Psilaxis krebsii(原文Philippia (Psilaxis) krebsii)的幼虫。本文MOTU03的标本(图 2和图 4), 其形态和大小与Tesch(1946)描述的“Agadina sp.”亦十分相似, 且在系统树(图 4)中与P. radiatus聚为一支。但Tesch(1946)所记录的“Agadina sp.”是否可确定为P. krebsii的幼虫, 可能还需进一步核实。毕竟不同种类的面盘幼虫形态可能非常相似, 特定海域的种类组成也可能未被完全了解。我们推测MOTU05—07也应属于轮螺属, 然而它们与配景轮螺的幼虫具有完全不同的形态特征。Robertson(1964)也观察到轮螺属面盘幼虫的肛区龙骨或不明显或微弱或是完全消失。可见, 轮螺属种类可能具有复杂多样的幼虫发育形式。若结合轮螺科成体的形态特征来研究浮游幼虫的形态发育, 也许更能揭示幼体形态特征的分类价值。如裸鳃目(Nudibranchia)枝鳃海牛属(Dendrodoris)不同种类的面盘幼虫具有相异的形态, 幼虫的形态特征能用于种类的区分(Hirose et al, 2015)。
Bieler(1993)对印度–太平洋的轮螺科进行了较为系统的分类学研究, 共记录了11属88种。而目前我国被记录的种类仅包括轮螺属和太阳螺属(Heliacus)共计11种(刘瑞玉, 2008), 种类数明显偏少。P. raditus的分布区域就包括我国南海北部近岸海域(Bieler, 1993), 但国内未见对该属或该种的记录。这表明国内对轮螺科的取样和分类尚显不足。可以推测, 本文涉及的面盘幼虫中可能还包括其他在我国未被记录的种类。不过, 轮螺浮游幼虫与成体的分布范围是否一致目前尚无法证实。轮螺长达数周至6个月的浮游幼体期使其能在海流的作用下漂流数千公里, 海流是影响这类幼虫分布的主要因素(Robertson, 1964; Robertson et al, 1970; Scheltema et al, 1996)。徐兆礼(2008)基于东海的环境数据认为轮螺科面盘幼虫(“强卷螺”)适应较低的温度, 南海的采样结果则不同, 5—7月南海中南部可采集到大量幼虫(未全部测序), 南海北部反而较少。基于特定海域或小尺度范围的数据分析, 可能无法得出准确的物种环境适应类型的结论。
成体参考序列的获得是对幼体进行DNA鉴定的前提。由于GenBank数据库中轮螺科的参考序列很少, 大多数种类无法通过DNA序列鉴定到种或属。随着DNA数据库的不断丰富和完善, DNA鉴定技术(DNA条形码技术)将越发显示其巨大的应用价值。DNA序列是联系生物成体和幼体的桥梁, 能实现已知成体和未知幼体的一一对应和匹配(Baldwin et al, 2009)。即使成体序列未知, 在成体难以获取的情况下, 通过其幼体来评估多样性也是一种有效的手段(Barber et al, 2006)。
| 刘瑞玉, 中国科学院海洋研究所, 2008. 中国海洋生物名录. 北京: 科学出版社, 493-494 |
| 李海涛, 何薇, 周鹏, 等, 2015. 伶鼬榧螺(Oliva mustelina)的分子鉴定及其形态变异. 海洋学报, 37(4): 117–123 DOI:10.3969/j.issn.0253-4193.2015.04.011 |
| 张福绥, 1964. 中国近海的浮游软体动物. Ⅰ.翼足类、异足类及海蜗牛类的分类研究. 海洋科学集刊, 5: 125–226 |
| 林森杰, 王路, 郑连明, 等, 2014. 海洋生物DNA条形码研究现状与展望. 海洋学报, 36(12): 1–17 DOI:10.3969/j.issn.0253-4193.2014.12.001 |
| 罗文增, 劳柏瑄, 2016. 台湾海峡浮游翼足类群聚之季节动态. 生物多样性, 24(7): 757–766 |
| 徐兆礼, 2008. 东海浮游翼足类(Pteropoda)环境适应类型的划分. 海洋与湖沼, 39(4): 381–387 DOI:10.3321/j.issn:0029-814X.2008.04.013 |
| Baldwin C C, Mounts J H, Smith D G, et al, 2009. Genetic identification and color descriptions of early life-history stages of Belizean Phaeoptyx and Astrapogon (Teleostei:Apogonidae) with comments on identification of adult Phaeoptyx. Zootaxa,: 1–22 |
| Barber P, Boyce S L, 2006. Estimating diversity of Indo-Pacific coral reef stomatopods through DNA barcoding of stomatopod larvae. Proceedings of the Royal Society B, 273(1597): 2053–2061 DOI:10.1098/rspb.2006.3540 |
| Bieler R, 1993. Architectonicidae of the Indo-Pacific (Mollusca, Gastropoda). Abhandlungen des Naturwissenschaftlichen Vereins in Hamburg, 30: 1–376 |
| Drummond A J, Rambaut A, 2007. BEAST:bayesian evolutionary analysis by sampling trees. BMC Evolutionary Biology, 7: 214 DOI:10.1186/1471-2148-7-214 |
| Folmer O, Black M, Hoeh W, et al, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology, 3(5): 294–299 |
| Fujisawa T, Barraclough T G, 2013. Delimiting species using single-locus data and the generalized Mixed Yule Coalescent approach:a revised method and evaluation on simulated data Sets. Systematic Biology, 62(5): 707–724 DOI:10.1093/sysbio/syt033 |
| Hirose M, Hirose E, Kiyomoto M, 2015. Identification of five species of Dendrodoris (Mollusca:Nudibranchia) from Japan, using DNA barcode and larval characters. Marine Biodiversity, 45(4): 769–780 DOI:10.1007/s12526-014-0288-2 |
| Ko H L, Wang Y T, Chiu T S, et al, 2013. Evaluating the accuracy of morphological identification of larval fishes by applying DNA barcoding. PLoS One, 8(1): e53451 DOI:10.1371/journal.pone.0053451 |
| Levin L A, 1990. A review of methods for labeling and tracking marine invertebrate larvae. Ophelia, 32(1-2): 115–144 DOI:10.1080/00785236.1990.10422028 |
| Pradillon F, Schmidt A, Peplies J, et al, 2007. Species identification of marine invertebrate early stages by whole-larvae in situ hybridisation of 18S ribosomal RNA. Marine Ecology Progress Series, 333: 103–116 DOI:10.3354/meps333103 |
| Puillandre N, Lambert A, Brouillet S, et al, 2012. ABGD, Automatic Barcode Gap Discovery for primary species delimitation. Molecular Ecology, 21(8): 1864–1877 DOI:10.1111/j.1365-294X.2011.05239.x |
| Puillandre N, Strong E E, Bouchet P, et al, 2009. Identifying gastropod spawn from DNA barcodes:possible but not yet practicable. Molecular Ecology Resources, 9(5): 1311–1321 DOI:10.1111/men.2009.9.issue-5 |
| Robertson R, 1964. Dispersal and wastage of larval Philippia krebsii (Gastropoda:Architectonicidae) in the north Atlantic. Proceedings of the Academy of Natural Sciences of Philadelphia, 116: 1–27 |
| Robertson R, Scheltema R S, Adams F W, 1970. The feeding, larval dispersal, and metamorphosis of Philippia (Gastropoda:Architectonicidae). Pacific Science, 24(1): 55–65 |
| Scheltema R S, Williams I P, Lobel P S, 1996. Retention around and long distance dispersal between oceanic islands by planktonic larvae of benthic gastropod mollusca. American Malacological Bulletin, 12: 67–75 |
| Simon C, Frati F, Beckenbach A, et al, 1994. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers. Annals of the Entomological Society of America, 87(6): 651–701 DOI:10.1093/aesa/87.6.651 |
| Tesch J J, 1946. The thecosomatous pteropods Ⅰ. the Atlantic. Dana Report, 28: 1–82 |
 2019, Vol. 50
2019, Vol. 50

