
中国海洋湖沼学会主办。
文章信息
- 乔艳, 周前进, 李孝军, 苗亮, 陈炯. 2019.
- QIAO Yan, ZHOU Qian-Jin, LI Xiao-Jun, MIAO Liang, CHEN Jiong. 2019.
- 环介导等温扩增联合横向流动试纸条检测简单异尖线虫/派氏异尖线虫方法的建立
- A LOOP-MEDIATED ISOTHERMAL AMPLIFICATION TECHNIQUE COMBINED WITH A LATERAL FLOW DIPSTICK FOR THE DETECTION OF ANISAKIS SIMPLEX SENSU STRICTO/ANISAKIS PEGREFFII IN COMMERCIAL FISH
- 海洋与湖沼, 50(2): 324-335
- Oceanologia et Limnologia Sinica, 50(2): 324-335.
- http://dx.doi.org/10.11693/hyhz20180800207
-
文章历史
- 收稿日期:2018-08-25
- 收修改稿日期:2018-10-20
2. 应用海洋生物技术教育部重点实验室 宁波 315211;
3. 舟山出入境检验检疫局 舟山 316000
2. Key Laboratory of Applied Marine Biotechnology of Ministry of Education, Ningbo University, Ningbo 315211, China;
3. Zhoushan Entry-Exit Inspection and Quarantine Bureau, Zhoushan 316000, China
简单异尖线虫复合种(Anisakis simplex species complex)包括三个姐妹单种, 分别是简单异尖线虫(A. sensu stricto), 派氏异尖线虫(A. pegreffii)和A.berlandi n. sp (Mattiucci et al, 2014), 其中简单异尖线虫和派氏异尖线虫能引起人异尖线虫病(Baird et al, 2014; Shimamura et al, 2016a)。简单异尖线虫和派氏异尖线虫幼虫通常寄生于许多硬骨鱼的内脏或肌肉组织中(Nieuwenhuizen et al, 2013; Baird et al, 2014; Nieuwenhuizen, 2016; Shimamura et al, 2016b)。人在食入生的、半熟或腌制的感染幼虫的海鱼, 会引起包括腹痛、恶心、腹泻、呕吐等胃肠道症状和一些过敏反应(Daschner et al, 2012; Mattiucci et al, 2013; Nieuwenhuizen et al, 2013)。在日本、意大利和韩国, 异尖线虫病比较常见(Audicana et al, 2002; Choi et al, 2009; Mattiucci et al, 2013; Heffler et al, 2016; Nieuwenhuizen, 2016)。随着生鲜饮食的逐渐普及, 异尖线虫病的发生率逐渐增加(Baird et al, 2014; Shimamura et al, 2016a)。我国进境动物检疫疫病名录(http://www.aqsiq.gov.cn/xxgk_13386/ywxx/dzwjy/201312/t20131205_390094.htm)将异尖线虫病列为二类传染病, 异尖线虫属(Anisakis)与对盲囊线虫属(Contracaecum)和宫脂线虫属(Hysterothylacium)一起被列为二类病原体, 这在一定程度上限制了海产品进出口贸易。因此, 建立快速检测简单异尖线虫和派氏异尖线虫技术对调查其空间分布和异尖线虫病流行病学评估至关重要。
到目前为止, 异尖线虫的检测主要依靠形态学方法, 如我国检疫机构的异尖线虫病诊断规程(国标SN/T 1509-2005)。随着分子生物学的快速发展, 基于聚合酶链式反应技术(polymerase chain reaction, PCR)的多种检测手段由于其灵敏度高和速度快等优点, 已被开发用于异尖线虫的诊断, 主要有PCR限制性片段长度多态性法(PCR-RFLP)(D’Amelio et al, 2000; Kijewska et al, 2000), 单链构象多态性方法(Zhu et al, 2007), 多重PCR(Umehara et al, 2008), 探针、荧光PCR法(Real time PCR, RT-PCR)(Lopez et al, 2010; Mossali et al, 2010; Godínez-González et al, 2017; Mattiucci et al, 2017; Paoletti et al, 2018)和分子系统发育学等方法(Nadler et al, 2005)。用于异尖线虫检测的靶标基因主要有线粒体DNA (cytochrome c oxidase, mtDNA cox), 核糖体DNA内转录间隔区(ribosomal DNA internal transcribed spacer, ITS)和核DNA EF1α-1(Mattiucci et al, 2016)。
Notomi等(2015)最初报道的环介导等温扩增(loop-mediated isothermal amplification, LAMP)由于其检测条件是恒定的温度, 检测更快速且更容易操作, 这引起了许多领域研究者的关注(Biswas et al, 2014; Notomi et al, 2015)。通常通过观察LAMP产物的琼脂糖凝胶电泳的梯形条带判断结果, 也可通过浊度计和核酸染料(SYBR Green I, 钙黄绿素等)等肉眼直接观察, 但是这些检测所用的设备比较昂贵(Tomita et al, 2008)。横向流动试纸条(lateral-flow dipstick, LFD)是一种快速检测核酸产物的方法, 且无需专门的仪器设备。主要原理是生物素标记的扩增产物与异硫氰酸荧光素(Fluorescein isothiocyanate, FITC)标记的DNA探针杂交, 杂交产物与金标记的抗FITC抗体结合, 可以在LFD的检测线上显示出来(Ding et al, 2010)。LAMP方法联合LFD检测(LAMP-LFD)最初应用于水生生物病毒性疾病的检测(Biswas et al, 2014), 目前已应用于动物致病菌、植物根结线虫等各种病原微生物(Tsai et al, 2012; Zhou et al, 2017)。本研究基于简单异尖线虫和派氏异尖线虫的rDNA-ITS2序列, 建立了LAMP-LFD的检测方法。同时, 利用LAMP-LFD方法对简单异尖线虫/派氏异尖线虫在东海经济海洋鱼类中的分布和流行情况进行了调查。
1 材料与方法 1.1 蠕虫及其基因组DNA的制备本研究中共涉及到12种蠕虫(表 1), 其中异尖科(Anisakidae)和棘头纲(Acanthocephala)蠕虫由本实验室分离自东海海鱼体内或者由舟山出入境检验检疫局提供。蠕虫首先用形态学方法鉴定到属, 利用NC5/NC2引物测序rDNA-ITS序列鉴定到种(引物序列见表 2)(Zhu et al, 1998)。此外, 还包括分离自养殖大黄鱼(Larimichthys crocea)体内的皮外寄生虫, 梅氏新贝尼登虫(Neobenedenia melleni)。实验所用蠕虫浸泡在70%乙醇中并保存在–20℃。
| 蠕虫 | 寄主 | 地理位置 | 其他信息(登录号等) |
| 简单异尖线虫 | 鳕鱼 | 舟山 | 舟山出入境检验检疫局(李孝军等, 2012a, b, 2013); KU991876 |
| 抹香鲸异尖线虫 | 鲐鱼 | ||
| 灰海鳗对盲囊线虫 | 鳕鱼 | ||
| 拟地新线虫 | 秋刀鱼 | ||
| 派氏异尖线虫 | 鲐鱼 | 宁波 | 本实验室分离并鉴定(Sheng et al, 2012); KU991879, KX228829, KU991878, KU991877, KU951441, KU991875 |
| 典型异尖线虫 | 鲐鱼 | ||
| 对盲囊属线虫 | 秋刀鱼 | ||
| 内弯宫脂线虫 | 鲐鱼 | ||
| 费氏宫脂线虫 | 鲐鱼 | ||
| 针蛔线虫 | 秋刀鱼 | ||
| 棘头虫 | 鲐鱼 | ||
| 梅氏新贝尼登虫 | 大黄鱼 |
| 引物 | 序列 | 用途 | 靶标基因 |
| FIP (F1c+F2)a | GTAATTCGACCCTCAGCCAGACACTAAGAATTCGAACGCACA | LAMP | rDNA-ITS2 |
| BIP (B1c+B2)b | CTGTGAAGCATTCGGCAAGCAACCGCTCGTCATATTGTC | ||
| F3 | GAATTGCAGACACATTGAGC | ||
| B3 | ATAGTAGATTCGGTGTTGACG | ||
| LF | TGCCATCGGGAATGAACC | ||
| LB | TTGCTGTTGTGTTGTTGGTG | ||
| HPc | GGTGAACTGTCTTCACGGTT | LFD | |
| Asi-F | TGGATCGATGAAGAACGCAG | PCR | 5.8S-ITS2-28S rDNA |
| Asi-R | AATCACGACTGAGCTGAGGT | ||
| NC5 | GTAGGTGAACCTGCGGAAGGATCATT | 18S-ITS1-5.8S-ITS2-28S rDNA (Zhu et al, 1998) | |
| NC2 | TTAGTTTCTTTTCCTCCGCT | ||
| 注: a: FIP是用生物素标记, 由F1c(下划线表示)和F2(粗体表示)组成, b: BIP由B1c(下划线表示)和B2(粗体表示)组成, c: HP由FITC标记 | |||
利用EZNA组织DNA提取试剂盒(OMEGA, 美国)提取简单异尖线虫和派氏异尖线虫第三期幼虫(third-stage larvae, L3)单虫的基因组DNA(genomic DNA, gDNA), 其他L3单条幼虫或成虫的gDNA用同样的方法提取制备(N. melleni的gDNA提取需要多个虫体)。另外, 同样提取以下11种海水鱼类的组织gDNA:白姑鱼(Argyrosomus argentatus), 黑鳃梅童鱼(Collichthys niveatus), 秋刀鱼(Cololabis saira), 大西洋鳕鱼(Gadus morhua), 
异尖线虫序列比对表明简单异尖线虫和派氏异尖线虫的rDNA-ITS2序列完全相同。设计引物Asi-F/Asi-R进行常规PCR, 扩增简单异尖线虫的rDNA-ITS2序列(引物信息见表 2)。PCR反应的总体积为25μL, 包括: dNTPs (2.5mmol/μL) 2μL, 10×PCR buffer 2.5μL, Asi-F/Asi-R (10pmol/μL)各1μL, Taq DNA聚合酶(5U/μL) (TaKaRa, 中国) 0.25μL, 模板DNA 2μL, 剩余体积用ddH2O补足。反应程序: 94℃预变性5min; 30个循环包括94℃变性30s, 56℃退火30s, 72℃延伸30s; 72℃延伸10min。产物经回收、纯化后, 克隆于pMD19-T载体中, 构建重组质粒pMD-rDNA-ITS2, 并进行测序鉴定。利用EZNA质粒提取试剂盒(OMEGA, 美国)提取阳性重组质粒, NanoDrop 2000 (Thermo Scientific, 美国)测定浓度并稀释至200ng/μL, 根据文献所述方法转换拷贝数即6×1011拷贝/μL(Whelan et al, 2003)。10倍梯度稀释重组质粒后作为LAMP-LFD灵敏度分析的DNA标准品。
1.3 LAMP引物和DNA探针的设计利用Primer Explorer Ver.4 (http://primerexplorer.jp/elamp4.0.0/index.html)在简单异尖线虫(GenBank No. KU99 1876)的rDNA-ITS2区域设计6条引物, 包括2条外引物F3/B3, 2条内引物FIP/BIP, 2条环引物LB/LF(见表 2)。FIP的5′末端用生物素标记。FITC标记的DNA探针HP设计在引物F1和B1c (与B1互补)之间。上述引物和探针的合成及相关标记均由英潍捷基(上海)贸易有限公司完成。
1.4 LAMP反应Bst DNA聚合酶的酶活性的最佳温度为60—65℃, 在LAMP反应中优选63℃和65℃ (Tomita et al, 2008; Zhou et al, 2014)。本研究选择63℃作为LAMP的反应温度。环引物可大幅度缩短LAMP的反应时间, 我们暂时选择60min作为LAMP反应时间(Zhou et al, 2014)。LAMP反应的体系: 20mmol/L Tris-HCl (pH 8.8), 6.5mmol/L MgSO4, 10mmol/L KCl, 10mmol/L (NH4)2SO4, 0.1% TritonX-100, 1.4mmol/L dNTPs, 0.8mol/L betaine (Sigma, 美国), 外引物F3/B3各0.2μmol/L, 内引物FIP/BIP各1.6μmol/L, 环引物LF/LB各0.4μmol/L, 8U Bst DNA聚合酶和2μL模板。0.1mmol/L的SYTO 9荧光染料加入混合物用于实时荧光LAMP测定。
1.5 LFD检测采用LFD (MileniaBiotec GmbH, 德国)检测LAMP产物。将20pmol HP加入到生物素标记的LAMP产物中, 63℃杂交5min。将8μL杂交产物与100μL测定缓冲液(MileniaBiotec, 德国)在新的1.5mL离心管中混合。然后, 将LFD垂直浸入混合液5min后, 观察结果。
1.6 LAMP-LFD特异性实验为了确定LAMP-LFD方法的特异性, 将另外7种异尖科线虫和其它3种蠕虫的gDNA作为模板进行LAMP反应。此外, 以上述11种海鱼的gDNA为模板进行荧光LAMP反应。所有LAMP反应在63℃下进行60min。LAMP产物通过LFD和2%琼脂糖凝胶电泳检测。实验平行重复10次。
1.7 LAMP-LFD灵敏度实验选取梯度稀释10倍的pMD-rDNA-ITS2质粒, 即6×107、6×106、6×105、6×104、6×103和6×102拷贝/μL。同时选取梯度稀释10倍的简单异尖线虫和派氏异尖线虫单虫gDNA, 即单虫gDNA的10–1、10–2、10–3、10–4、10–5和10–6倍。以上述为模板, 分别荧光LAMP反应, 在63℃下进行60min。产物通过LFD检测和2%琼脂糖凝胶电泳检测。实验平行重复10次。使用有效扩增起始时间(The time to positive value, Tp)的相对离散程度来反映LAMP-LFD方法的可重复性, 它定义为变异系数(coefficient of variation, CV), 即标准偏差(standard deviation, SD)与平均值之比(CV = SD/AVERAGE) (Zhou et al, 2014)。若CV < 0.10表明LAMP-LFD方法具有良好的重复性。
同时选取10倍梯度稀释的简单异尖线虫单虫的gDNA为模板, NC5/NC2为引物进行常规PCR反应(Zhu et al, 1998)。反应程序: 94℃预变性5min; 30个循环包括94℃变性30s, 54℃退火30s, 72℃延伸1min; 72℃延伸10min。产物通过2%琼脂糖凝胶电泳检测。实验平行重复10次。
1.8 LAMP时间优化根据之前的报道, Tp值随模板浓度降低而逐渐增加, 呈典型的线性相关(Zhou et al, 2014)。因此, 我们选用灵敏度浓度(最低检测浓度)的gDNA作为模板, 在63℃时分别反应25、30、35、40、45、50、55和60min。产物通过LFD检测和2%琼脂糖凝胶电泳检测, 以确定最适反应时间。实验平行重复10次。
1.9 实际样品检测2015—2017年期间, 采集来自东海海域(以舟山和宁波为主)11个物种的140尾海水鱼类, 包括白姑鱼、黑鳃梅童鱼、秋刀鱼、大西洋鳕鱼、红娘鱼

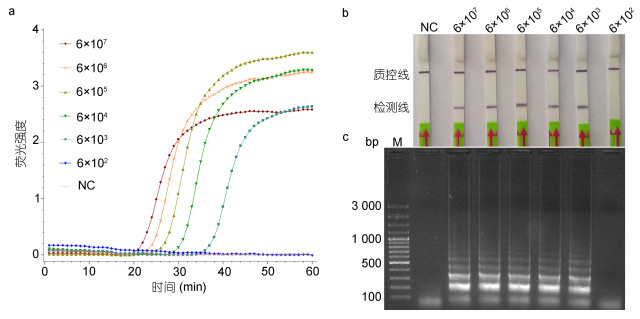
使用12种蠕虫gDNA为模板进行荧光LAMP-LFD反应, 确定该方法的特异性。结果表明, 以简单异尖线虫和派氏异尖线虫的gDNA为模板时, LAMP反应呈现典型的“S”形扩增曲线, 为阳性扩增(图 1A); 当以其它10种蠕虫包括抹香鲸异尖线虫(A. physeteris), 典型异尖线虫(A. typica), 对盲囊属线虫(Contracaecum spp.), 灰海鳗对盲囊线虫(C. muraenesoxi), 内弯宫脂线虫(Hysterothylacium aduncum), 费氏宫脂线虫(H. fabri), 针蛔线虫(Raphidascaris lophii), 拟地新线虫(Pseudoterranova decipiens), 棘头虫(Acanthocephala sp.)和梅氏新贝尼登虫的组织gDNA为模板时, 未观察到阳性扩增(图 1A)。当简单异尖线虫和派氏异尖线虫的LAMP产物通过LFD检测时, 检测线上出现明显的条带(图 1B); 而其它10种蠕虫的LAMP产物通过LFD检测时, 检测线上未见明显条带(图 1B)。这与琼脂糖凝胶电泳检测结果一致(图 1C)。此外, 当以11种海鱼组织gDNA为模板时也未出现阳性扩增(图 2A, B)。
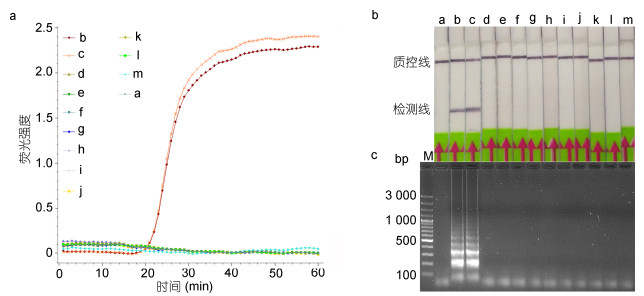 |
| 图 1 LAMP-LFD特异性分析 Fig. 1 The specificity test of LAMP-LFD 注: a:阴性对照, b:简单异尖线虫, c:派氏异尖线虫, d:抹香鲸异尖线虫, e:典型异尖线虫, f:对盲囊属线虫, g:灰海鳗对盲囊线虫, h:内弯宫脂线虫, i:费氏宫脂线虫, j:针蛔线虫, k:拟地新线虫, l:棘头虫, m:梅氏新贝尼登虫, M: GeneRuler 100bp plus DNA ladder (Fermentas, 美国) |
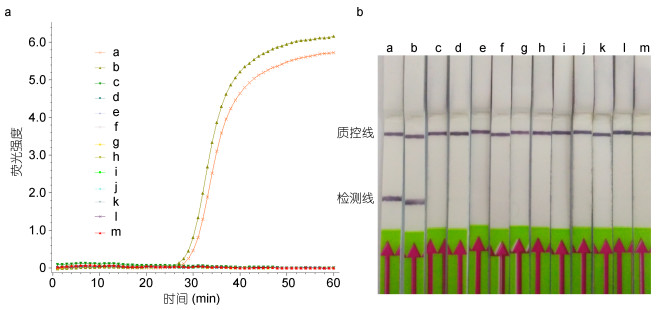 |
图 2 LAMP-LFD针对海鱼组织的特异性分析 Fig. 2 The specificity test of LAMP-LFD using fish tissue as template 注: a:简单异尖线虫, b:派氏异尖线虫, c:白姑鱼, d:黑鳃梅童鱼, e:秋刀鱼, f:大西洋鳕鱼, g: 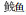 , h:红娘鱼, i: , h:红娘鱼, i:  j:真鲷, k:鲐鱼, l:竹荚鱼, m:带鱼 j:真鲷, k:鲐鱼, l:竹荚鱼, m:带鱼 |
首先, 利用6×107—6×102拷贝/μL的pMD-rDNA- ITS2质粒为模板, 分析LAMP-LFD的检测灵敏度。结果表明, 当模板浓度为6×107—6×103拷贝/μL时, 呈现典型的“S”形扩增曲线, 扩增结果为阳性(图 3A)。LAMP产物与探针HP杂交, 并在LFD检测线上出现明显的条带(图 3B)。当以6×103拷贝/μL的pMD-rDNA- ITS2为模板进行检测, 10次重复中的Tp值稍有变化, 平均Tp值为35.87, CV值为0.03 (图 6A, 表 3)。而当模板浓度为6×102拷贝/μL时, 未见阳性扩增(图 3A); LAMP产物通过LFD检测时, 检测线上未见明显条带(图 3B)。这与琼脂糖凝胶电泳检测结果一致(图 3C)。因此, LAMP-LFD的检测灵敏度为6×103拷贝/μL。
 |
| 图 3 LAMP-LFD灵敏度分析 Fig. 3 The sensitivity analysis of LAMP-LFD 注: NC:阴性对照, 6×107—6×102: pMD-rDNA-ITS2重组质粒, 单位:拷贝/μL, M: GeneRuler 100bp plus DNA ladder (Fermentas, 美国) |
 |
|
图 6 LAMP-LFD的重复性实验 Fig. 6 The repeatability test of LAMP-LFD 注: a:阴性对照, b—k: 6 |
| 模板类型 | 样品(n) | 阳性(n) | Tp平均值 | SD | CV |
| pMD-rDNA-ITS2 | 10 | 10 | 35.87 | 0.99 | 0.03 |
| 简单异尖线虫 | 10 | 10 | 27.26 | 0.32 | 0.01 |
| 派氏异尖线虫 | 10 | 10 | 27.31 | 0.33 | 0.01 |
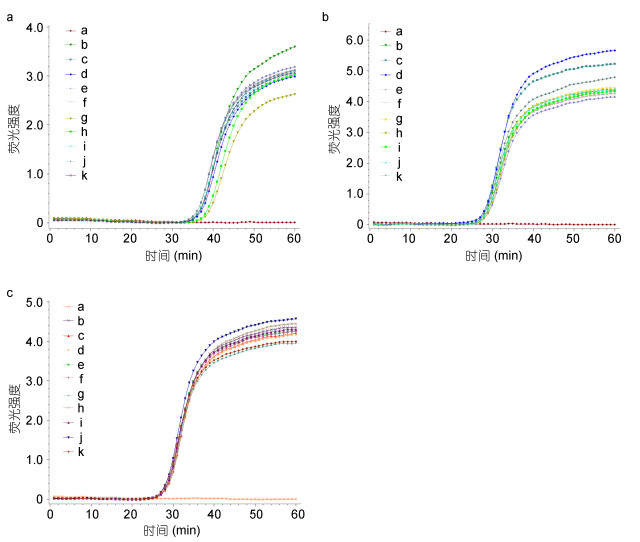
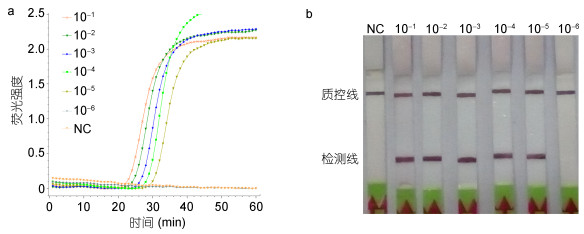
同时, 以简单异尖线虫和派氏异尖线虫单虫gDNA为模板, 分析了LAMP-LFD的灵敏度。当利用简单异尖线虫单虫的不同稀释倍数的gDNA(10–1—10–5倍)为模板, 进行荧光LAMP反应时, 呈现阳性扩增(图 4A), 且阳性扩增的起始时间与gDNA浓度呈线性化关系(图 4B)。以简单异尖线虫单虫gDNA的10–5倍为模板进行检测, 10次重复的Tp平均值为27.26, CV值为0.01(图 6B, 表 3)。LAMP产物与探针HP杂交, 并在LFD检测线上出现明显的条带(图 4C)。当以gDNA的10–6倍为模板时, 未出现阳性扩增(图 4A), 并在LFD检测线上为见明显的条带(图 4C)。这与琼脂糖凝胶电泳检测结果一致(图 4D)。以派氏异尖线虫单虫的不同稀释倍数的gDNA (10–1—10–5倍)为模板时, 结果呈阳性(图 5A, B)。以派氏异尖线虫单虫gDNA的10–5倍为模板进行检测, 10次重复的平均Tp值为27.31, CV值为0.01(图 6C, 表 3)。而当以gDNA的10–6倍为模板时, 结果呈阴性(图 5A, B)。而利用常规PCR只能检测到gDNA的10–3倍(图 4E)。因此, LAMP-LFD方法针对简单异尖线虫和派氏异尖线虫的检测灵敏度为单条虫体gDNA的10–5倍, 是常规PCR方法的100倍。
 |
| 图 4 LAMP-LFD针对单条简单异尖线虫L3的灵敏度分析 Fig. 4 The sensitivity analysis of LAMP-LFD to detect single A. simplex s. s.L3 注: NC:阴性对照, 10–1–10–6:简单异尖线虫L3 gDNA的不同稀释倍数, M: GeneRuler 100bp plus DNA ladder (Fermentas, 美国) |
 |
| 图 5 LAMP-LFD针对单条派氏异尖线虫L3的灵敏度分析 Fig. 5 The sensitivity analysis of LAMP-LFD to detect single A. pegreffii L3 注: NC:阴性对照, 10–1—10–6:派氏异尖线虫L3 gDNA的不同稀释倍数, M: GeneRuler 100bp plus DNA ladder (Fermentas, 美国) |
通过进一步优化时间, 确定LAMP有效扩增的最短时间, 使LAMP-LFD方法更加便捷。结果表明, 当利用简单异尖线虫L3检测灵敏度浓度的gDNA为模板进行LAMP反应, 观察到在35min获得有效扩增。当LAMP反应的时间为40—60min时, 观察到类似的扩增强度(图 7A, B), 且视觉上强于35min时的扩增(图 7A)。为确保测试的可靠性, LAMP反应的最佳时间过程设定为40min, 加之5min杂交和3—5min LFD可视化, 整个测试可以在50min内完成。
 |
| 图 7 LAMP-LFD时间优化 Fig. 7 Time optimization for LAMP-LFD 注: NC:阴性对照, 25—60min: LAMP反应时间, M: GeneRuler 100bp plus DNA ladder (Fermentas, 美国) |
通过形态学检查, 从140条海鱼中共分离出1573条异尖科线虫, 其感染率为77.1%。这些虫体包括简单异尖线虫/派氏异尖线虫, 内弯宫脂线虫, 费氏宫脂线虫和对盲囊属线虫(表 4)。由于感染严重的鱼类中可以分离出数十条甚至百余条线虫, 逐一鉴别分离的每条虫体费时费力。因此, 我们按照以下原则, 利用LAMP-LFD和rDNA-ITS1-5.8S-ITS2测序两种方法对进行鉴定:当分离到≤5条异尖线虫/鱼时, 所有虫体利用上述两种方法进行鉴定; 当分离出≥5条异尖线虫/鱼时, 随机选取5条虫体进行鉴定。遵循这一原则, 共计287条形态学检查为异尖科的虫体进一步使用上述两种分子生物学方法进行了鉴定。测序结果显示, 287条线虫均为异尖线虫科, 分别为简单异尖线虫/派氏异尖线虫(266条)、内弯宫脂线虫(15条)、费氏宫脂线虫(5条)以及对盲囊异尖线虫(1条), 简单异尖线虫/派氏异尖线虫占总异尖科线虫的92.7%(266/287)。LAMP-LFD结果表明, 共计266条鉴定为简单异尖线虫/派氏异尖线虫, 与rDNA-ITS1-5.8S-ITS2的测序结果完全一致(表 4)。
| 海鱼物种 | 异尖科线虫检测性海鱼(n)/所有被海鱼(n) | 异尖科线虫(n) | 异尖科线虫物种 | PCR测序法 | LAMP-LFD法 |
| 鲐鱼 | 9/9 | 94 | Asi/Ape | 20/20 | 20/20 |
| Had | 6/6 | 0/6 | |||
| Hfa | 1/1 | 0/1 | |||
| 秋刀鱼 | 10/15 | 224 | Asi/Ape | 17/17 | 17/17 |
| Contracaecum spp. | 1/1 | 0/1 | |||
| Had | 9/9 | 0/9 | |||
| Hfa | 3/3 | 0/3 | |||
| 真鲷 | 2/2 | 8 | Asi/Ape | 7/7 | 7/7 |
| 竹荚鱼 | 25/27 | 545 | Asi/Ape | 71/71 | 71/71 |
| 红娘鱼 | 2/2 | 4 | Asi/Ape | 4/4 | 4/4 |

|
8/8 | 379 | Asi/Ape | 25/25 | 25/25 |
| Hfa | 1/1 | 0/1 | |||

|
2/2 | 11 | Asi/Ape | 7/7 | 7/7 |
| 带鱼 | 9/12 | 90 | Asi/Ape | 22/22 | 22/22 |
| 黑鳃梅童鱼 | 17/32 | 50 | Asi/Ape | 36/36 | 36/36 |
| 白姑鱼 | 11/18 | 79 | Asi/Ape | 24/24 | 24/24 |
| 鳕鱼 | 13/13 | 89 | Asi/Ape | 33/33 | 33/33 |
| 总计 | 108/140 | 1573 | 287/287 | 266/287 | |
| 注: Asi:简单异尖线虫, Ape:派氏异尖线虫, Had:内弯宫脂线虫, Hfa:费氏宫脂线虫 | |||||
简单异尖线虫复合种寄生在全球各种海洋鱼类和哺乳动物中, 其中, A. berlandi n. sp.未有明确证据使人致病(Mattiucci et al, 2014), 但简单异尖线虫和派氏异尖线虫会引起人的异尖线虫病, 因此, 异尖线虫作为检疫对象在海鱼产品的进出口中受到广泛关注。鉴于检疫的统一要求, 即在鱼产品中不得检出单异尖线虫和派氏异尖线虫两个姐妹种, 我们建立了一种LAMP-LFD方法来检测简单异尖线虫/派氏异尖线虫。该方法包括40min的核酸扩增, 5min的探针杂交和3—5min的LFD检测, 总检测时间在50min内。该方法能稳定、快速且无需任何专业仪器检测简单异尖线虫/派氏异尖线虫, 对于现场快速检测具有较大的应用潜力。
近十年来, LAMP技术由于其便捷性, 快速性和高灵敏度等优点已经用于鉴定各种微生物(Li et al, 2017)。此外, 研究者利用该方法对多种病原体进行鉴别和诊断(Kikuchi et al, 2009; Liu et al, 2011; Zhou et al, 2017)。在本研究中, 利用rDNA在基因组中的多拷贝以及高度种内保守性和种间变异性的进化特征(Blaxter et al, 1998; Kim et al, 2006), 以rDNA-ITS2为检测靶标, 将LAMP方法结合LFD可视化检测简单异尖线虫/派氏异尖线虫。利用该方法, 简单异尖线虫/派氏异尖线虫检出阳性结果而其它被检测的蠕虫为阴性。
目前, 异尖线虫的检测多基于PCR技术(Zhu et al, 1998; D’Amelio et al, 2000; Kijewska et al, 2000; Chen et al, 2008; Umehara et al, 2008; Lopez et al, 2010; Mossali et al, 2010; Herrero et al, 2011; Piras et al, 2014; Godínez-González et al, 2017; Mattiucci et al, 2017; Paoletti et al, 2018)。利用PCR方法检测需要两步, 即核酸扩增和电泳观察, 耗时约3h(Chen et al, 2008)。研究者也将PCR-RFLP方法用于异尖线虫检测(D’Amelio et al, 2000; Kijewska et al, 2000; Piras et al, 2014; Zhu et al, 1998)。Hinf I和HhaI等限制酶是PCR产物分析的常用酶类, 消化扩增产物的时间通常超过3h, 导致整个检测时程较长。近年来, 研究者利用特异性探针或荧光素建立了RT-PCR技术检测异尖线虫(Lopez et al, 2010; Mossali et al, 2010; Herrero et al, 2011; Godínez-González et al, 2017; Mattiucci et al, 2017; Paoletti et al, 2018)。甚至, 针对不同靶标(mtDNA cox 2, rDNA-ITS和mtDNA cox 1)的基于RT-PCR的引物-探针系统可同时检测多种异尖线虫(Lopez et al, 2010; Mossali et al, 2010; Herrero et al, 2011)。最近, 成功开发的基于RT-PCR的特异性引物/探针系统, 可以区分Anisakis和Pseudoterranova属的重要病原物种以及H. aduncum(Mattiucci et al, 2017; Paoletti et al, 2018)。与PCR和PCR-RFLP分析相比, RT-PCR方法更加快捷, 但是成本更高、操作复杂且耗时。在本研究中建立的LAMP-LFD技术能够在50min内准确的将简单异尖线虫/派氏异尖线虫从其它线虫中鉴定出来, 无需特殊的仪器设备, 展示出良好的应用潜力。
分析灵敏度对于检测技术的实用性至关重要, 尤其在面对具有零容忍要求的检疫时(Nugen et al, 2008; Niemz et al, 2011)。基于PCR原理的一些分子检测技术已经在异尖线虫的口岸检疫中作为辅助手段加以使用, 通常是将虫体从鱼类样品中分离后, 将单条虫体用于实验室诊断(Zhu et al, 1998; D'Amelio et al, 2000; Kijewska et al, 2000; Chen et al, 2008; Piras et al, 2014; Zhou et al, 2017)。Chen等(2008)利用常规的PCR方法, 检测简单异尖线虫, 典型异尖线虫, 派氏异尖线虫, 内弯宫脂线虫和对盲囊线虫等单虫。利用限制性内切酶建立的PCR-RFLP技术也广泛地用于区分多种异尖科线虫的幼虫或成虫(Zhu et al, 1998; D'Amelio et al, 2000; Kijewska et al, 2000; Piras et al, 2014)。最近, 研究者开发了以mtDNA cox 2为检测靶标的RT-PCR引物-探针系统以鉴定简单异尖线虫和派氏异尖线虫L3(Godínez-González et al, 2017)。本研究建立的LAMP-LFD方法以简单异尖线虫或派氏异尖线虫的单条L3的gDNA为模板, 可以检测到单虫gDNA的10–5倍, 比常规的PCR检测具有更高的灵敏度(Chen et al, 2008)。
简单异尖线虫的形态种呈世界性分布, 大多数报道的病例是由简单异尖线虫和派氏异尖线虫引起(Piras et al, 2014; Casti et al, 2017)。Piras等(2014)通过PCR-RFLP方法在地中海西部采集的218个鱼体中分离出1286只幼虫(总感染率为28.1%), 其中1272只幼虫(99%)是派氏异尖线虫。最近, 撒丁岛市场采集的经济鱼类的流行病学调查显示, 异尖线虫的总体感染率为39.9%(Casti et al, 2017), 且均为派氏异尖线虫感染。而从大西洋鳕鱼中收集到的幼虫中有90.0%为简单异尖线虫, 10.0%为派氏异尖线虫(Casti et al, 2017)。在中国, 简单异尖线虫和派氏异尖线虫的分布可能是呈区域性的(Zhang et al, 2007; Zhang et al, 2013; Kong et al, 2015)。Zhang等(2007)从黄海鱼类中分离到200条L3, 其中13.5%为派氏异尖线虫。在南海采集的鱼类体内分离到237条L3, 仅有18条为派氏异尖线虫, 其余线虫为典型异尖线虫(Zhang et al, 2013)。然而, 在东海, Kong等(2015)研究表明简单异尖线虫和派氏异尖线虫是占主导地位的物种(所检虫体中派氏异尖线虫占84.8%, 简单异尖线虫占0.6%)。本研究结果表明, 异尖科幼虫在东海地区相当普遍, 感染率为77.1%。LAMP-LFD方法检测287条幼虫, 其中有266条(占92.7%)属于简单异尖线虫/派氏异尖线虫, 结果与形态学方法和rDNA-ITS1-5.8S-ITS2测序的结果一致, 表明简单异尖线虫/派氏异尖线虫在东海海域中占主导地位, 这与Kong等(2015)的报道保持一致。此外, A. berlandi n.sp与其它两个姐妹种具有相似的rDNA-ITS2序列, 这可能导致LAMP-LFD方法的阳性结果。然而, 这个姐妹种大多报道在南太平洋和北太平洋(Mattiucci et al, 2014), 在中国海洋硬骨鱼和哺乳动物中并未发现, 表明本LAMP-LFD方法在中国口岸检疫方面的应用前景较大。
4 结论本研究基于rDNA-ITS2序列建立了一种LAMP-LFD方法用于特异性地检测简单异尖线虫或派氏异尖线虫, 检测灵敏度达到单条L3 gDNA的10-5倍。该方法经核酸扩增, 探针杂交和LFD检测几个步骤, 整个检测可在50min内完成。田间样本的检测结果表明, 本方法简单、便捷、准确, 能够在简单异尖线虫和派氏异尖线虫的现场筛检和常规检疫中发挥作用。
李孝军, 陈宇, 唐行忠, 等. 2012a. 进境太平洋鳕鱼寄生的异尖科线虫rDNA-ITS序列的PCR扩增及分析. 中国畜牧兽医, 39(6): 90-92 |
李孝军, 耿新辉, 唐行忠, 等. 2012b. 一种异尖科线虫rDNA-ITS序列的PCR扩增及分析. 畜牧与兽医, 44(5): 65-67 |
李孝军, 施进, 陈宇, 等. 2013. 鲭鱼中寄生的1种异尖科线虫rDNA-ITS序列的PCR扩增及分析. 黑龙江畜牧兽医, (5): 91-93 |
Audicana M T, Ansotegui I J, De Corres L F et al, 2002. Anisakis simplex: dangerous-dead and alive? Trends Parasitol, 18(1): 20-25
|
Baird F J, Gasser R B, Jabbar A et al, 2014. Foodborne anisakiasis and allergy. Mol Cell Probes, 28(4): 167-174 DOI:10.1016/j.mcp.2014.02.003 |
Biswas G, Sakai M, 2014. Loop-mediated isothermal amplification (LAMP) assays for detection and identification of aquaculture pathogens:current state and perspectives. Appl Microbiol Biotechnol, 98(7): 2881-2895 DOI:10.1007/s00253-014-5531-z |
Blaxter M L, De Ley P, Garey J R et al, 1998. A molecular evolutionary framework for the phylum Nematoda. Nature, 392(6671): 71-75 DOI:10.1038/32160 |
Casti D, Scarano C, Piras M C et al, 2017. Occurrence of nematodes of the genus Anisakis in Mediterranean and Atlantic fish marketed in Sardinia. Ital J Food Saf, 6(1): 6185 |
Chen Q, Yu H Q, Lun Z R et al, 2008. Specific PCR assays for the identification of common anisakid nematodes with zoonotic potential. Parasitol Res, 104(1): 79-84 DOI:10.1007/s00436-008-1161-7 |
Choi S J, Lee J C, Kim M J et al, 2009. The clinical characteristics of Anisakis allergy in Korea. Korean J Intern Med, 24(2): 160-163 DOI:10.3904/kjim.2009.24.2.160 |
D'Amelio S, Mathiopoulos K D, Santos C P et al, 2000. Genetic markers in ribosomal DNA for the identification of members of the genus Anisakis (Nematoda:ascaridoidea) defined by polymerase-chain-reaction-based restriction fragment length polymorphism. Int J Parasitol, 30(2): 223-226 DOI:10.1016/S0020-7519(99)00178-2 |
Daschner A, Cuéllar C, Rodero M, 2012. The Anisakis allergy debate: does an evolutionary approach help? Trends Parasitol, 28(1): 9-15
|
Ding W C, Chen J, Shi Y H et al, 2010. Rapid and sensitive detection of infectious spleen and kidney necrosis virus by loop-mediated isothermal amplification combined with a lateral flow dipstick. Arch Virol, 155(3): 385-389 DOI:10.1007/s00705-010-0593-4 |
Godínez-González C, Roca-Geronès X, Cancino-Faure B et al, 2017. Quantitative SYBR Green qPCR technique for the detection of the nematode parasite Anisakis in commercial fish-derived food. Int J Food Microbiol, 261: 89-94 DOI:10.1016/j.ijfoodmicro.2017.05.012 |
Heffler E, Sberna M E, Sichili S et al, 2016. High prevalence of Anisakis simplex hypersensitivity and allergy in Sicily, Italy. Ann Allergy Asthma Immunol, 116(2): 146-150 DOI:10.1016/j.anai.2015.12.014 |
Herrero B, Vieites J M, Espi eira M, 2011. Detection of anisakids in fish and seafood products by real-time PCR. Food Control, 22(6): 933-939 DOI:10.1016/j.foodcont.2010.11.028 |
Kijewska A, Slominska M, Wegrzyn G et al, 2000. A PCR-RFLP assay for identification of Anisakis simplex from different geographical regions. Mol Cell Probes, 14(6): 349-354 DOI:10.1006/mcpr.2000.0322 |
Kikuchi T, Aikawa T, Oeda Y et al, 2009. A rapid and precise diagnostic method for detecting the Pinewood nematode Bursaphelenchus xylophilus by loop-mediated isothermal amplification. Phytopathology, 99(12): 1365-1369 DOI:10.1094/PHYTO-99-12-1365 |
Kim K H, Eom K S, Park JK, 2006. The complete mitochondrial genome of Anisakis simplex (Ascaridida:Nematoda) and phylogenetic implications. Int JParasitol, 36(3): 319-328 DOI:10.1016/j.ijpara.2005.10.004 |
Kong Q M, Fan L F, Zhang J H et al, 2015. Molecular identification of Anisakis and Hysterothylacium larvae in marine fishes from the East China Sea and the Pacific coast of central Japan. Int J Food Microbiol, 199: 1-7 DOI:10.1016/j.ijfoodmicro.2015.01.007 |
Li Y M, Fan P H, Zhou S S et al, 2017. Loop-mediated isothermal amplification (LAMP):A novel rapid detection platform for pathogens. Microb Pathog, 107: 54-61 DOI:10.1016/j.micpath.2017.03.016 |
Liu C Y, Song H Q, Zhang R L et al, 2011. Specific detection of Angiostrongylus cantonensis in the snail Achatina fulica using a loop-mediated isothermal amplification (LAMP) assay. Mol Cell Probes, 25(4): 164-167 DOI:10.1016/j.mcp.2011.04.002 |
Lopez I, Pardo M A, 2010. Evaluation of a real-time polymerase chain reaction (PCR) assay for detection of Anisakis simplex parasite as a food-borne allergen source in seafood products. J Agric Food Chem, 58(3): 1469-1477 DOI:10.1021/jf903492f |
Mattiucci S, Acerra V, Paoletti M et al, 2016. No more time to stay 'single' in the detection of Anisakis pegreffii, A. simplex (s. s.) and hybridization events between them: a multi-marker nuclear genotyping approach. Parasitology, 143(8): 998-1011
|
Mattiucci S, Cipriani P, Webb S C et al, 2014. Genetic and morphological approaches distinguish the three sibling species of the Anisakis simplex species complex, with a species designation as Anisakis berlandi n. sp. for A. simplex sp. C (Nematoda: Anisakidae). J Parasitol, 100(2): 199-214
|
Mattiucci S, Fazii P, De Rosa A et al, 2013. Anisakiasis and gastroallergic reactions associated with Anisakis pegreffii infection, Italy. Emerg Infect Dis, 19(3): 496-499 DOI:10.3201/eid1903.121017 |
Mattiucci S, Paoletti M, Colantoni A et al, 2017. Invasive anisakiasis by the parasite Anisakis pegreffii (Nematoda:Anisakidae):diagnosis by real-time PCR hydrolysis probe system and immunoblotting assay. BMC Infect Dis, 17: 530 DOI:10.1186/s12879-017-2633-0 |
Mossali C, Palermo S, Capra E et al, 2010. Sensitive detection and quantification of anisakid parasite residues in food products. Foodborne Pathog Dis, 7(4): 391-397 DOI:10.1089/fpd.2009.0428 |
Nadler S A, D'Amelio S, Dailey M D et al, 2005. Molecular phylogenetics and diagnosis of Anisakis, Pseudoterranova, and Contracaecum from northern Pacific marine mammals. J Parasitol, 91(6): 1413-1429 DOI:10.1645/GE-522R.1 |
Niemz A, Ferguson T M, Boyle D S, 2011. Point-of-care nucleic acid testing for infectious diseases. Trends Biotechnol, 29(5): 240-250 DOI:10.1016/j.tibtech.2011.01.007 |
Nieuwenhuizen N E, 2016. Anisakis-immunology of a foodborne parasitosis. Parasite Immunol, 38(9): 548-557 DOI:10.1111/pim.2016.38.issue-9 |
Nieuwenhuizen N E, Lopata A L, 2013. Anisakis-a food-borne parasite that triggers allergic host defences. Int J Parasitol, 43(12-13): 1047-1057 DOI:10.1016/j.ijpara.2013.08.001 |
Notomi T, Mori Y, Tomita N et al, 2015. Loop-mediated isothermal amplification (LAMP):principle, features, and future prospects. J Microbiol, 53(1): 1-5 |
Nugen S R, Baeumner A J, 2008. Trends and opportunities in food pathogen detection. Anal Bioanal Chem, 391(2): 451-454 DOI:10.1007/s00216-008-1886-2 |
Paoletti M, Mattiucci S, Colantoni A et al, 2018. Species-specific Real Time-PCR primers/probe systems to identify fish parasites of the genera Anisakis, Pseudoterranova and Hysterothylacium (Nematoda:Ascaridoidea). Fish Res, 202: 38-48 DOI:10.1016/j.fishres.2017.07.015 |
Piras M C, Tedde T, Garippa G et al, 2014. Molecular and epidemiological data on Anisakis spp. (Nematoda: Anisakidae) in commercial fish caught off northern Sardinia (western Mediterranean Sea). Vet Parasitol, 203(1-2): 237-240
|
Sheng Z L, LuX J, Chen J, 2012. Molecular cloning and characteristic analysis of a thioredoxin from Neobenedenia melleni. Afr J Biotechnol, 11(30): 7582-7591 |
Shimamura Y, Ishii N, Ego M et al, 2016a. Multiple acute infection by Anisakis:a case series. Intern Med, 55(8): 907-910 DOI:10.2169/internalmedicine.55.5628 |
Shimamura Y, Muwanwella N, Chandran S et al, 2016b. Common symptoms from an uncommon infection:gastrointestinal anisakiasis. Can J Gastroenterol Hepatol: 5176502 |
Tomita, N, Mori Y, Kanda H et al, 2008. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nat Protoc, 3(5): 877-882 DOI:10.1038/nprot.2008.57 |
Tsai S M, LiuH J, ShienJ H et al, 2012. Rapid and sensitive detection of infectious bursal disease virus by reverse transcription loop-mediated isothermal amplification combined with a lateral flow dipstick. J Virol Methods, 181(1): 117-124 DOI:10.1016/j.jviromet.2011.09.002 |
Umehara A, Kawakami Y, Araki J et al, 2008. Multiplex PCR for the identification of Anisakis simplex sensu stricto, Anisakis pegreffii and the other anisakid nematodes. Parasitol Int, 57(1): 49-53 |
Whelan J A, Russell N B, Whelan M A, 2003. A method for the absolute quantification of cDNA using real-time PCR. J Immunol Methods, 278(1-2): 261-269 DOI:10.1016/S0022-1759(03)00223-0 |
Zhang L P, Du X J, An R Y et al, 2013. Identification and genetic characterization of Anisakis larvae from marine fishes in the South China Sea using an electrophoretic-guided approach. Electrophoresis, 34(6): 888-894 DOI:10.1002/elps.201200493 |
Zhang L P, Hu M, Shamsi S et al, 2007. The specific identification of anisakid larvae from fishes from the Yellow Sea, China, using mutation scanning-coupled sequence analysis of nuclear ribosomal DNA. Mol Cell Probes, 21(5-6): 386-390 DOI:10.1016/j.mcp.2007.05.004 |
Zhou Q J, Cai Y, Gu J F et al, 2017. Rapid and sensitive detection of Meloidogyne mali by loop-mediated isothermal amplification combined with a lateral flow dipstick. EurJ Plant Pathol, 148(4): 755-769 DOI:10.1007/s10658-016-1130-4 |
Zhou Q J, Wang L, Chen J et al, 2014. Development and evaluation of a real-time fluorogenic loop-mediated isothermal amplification assay integrated on a microfluidic disc chip (on-chip LAMP) for rapid and simultaneous detection of ten pathogenic bacteria in aquatic animals. J Microbiol Methods, 104: 26-35 DOI:10.1016/j.mimet.2014.06.008 |
Zhu X Q, Gasser R B, Podolska M et al, 1998. Characterisation of anisakid nematodes with zoonotic potential by nuclear ribosomal DNA sequences. Int J Parasitol, 28(12): 1911-1921 DOI:10.1016/S0020-7519(98)00150-7 |
Zhu X Q, Podolska M, Liu J S et al, 2007. Identification of anisakid nematodes with zoonotic potential from Europe and China by single-strand conformation polymorphism analysis of nuclear ribosomal DNA. Parasitol Res, 101(6): 1703-1707 DOI:10.1007/s00436-007-0699-0 |
 2019, Vol. 50
2019, Vol. 50


