
中国海洋湖沼学会主办。
文章信息
- 刘硕博, 唐祖蓉, 申望, 叶莹莹, 陈晓磊, 祁鹏志, 郭宝英. 2019.
- LIU Shuo-Bo, TANG Zu-Rong, SHEN Wang, YE Ying-Ying, CHEN Xiao-Lei, QI Peng-Zhi, GUO Bao-Ying. 2019.
- 基于F型mtDNA D-Loop的厚壳贻贝(Mytilus coruscus)群体遗传多样性研究
- STUDY ON THE GENETIC DIVERSITY OF MYTILUS CORUSCUS IN THE EAST CHINA SEA
- 海洋与湖沼, 50(2): 355-364
- Oceanologia et Limnologia Sinica, 50(2): 355-364.
- http://dx.doi.org/10.11693/hyhz20180900231
-
文章历史
- 收稿日期:2018-09-30
- 收修改稿日期:2018-11-12
厚壳贻贝(Mytilus coruscus)隶属瓣鳃纲(Lamellibranchia)、异柱目(Anisomyaria)、贻贝总科(Mytilidae), 属暖温带种, 常栖息于近岸、内湾与岛礁区域, 主要分布于西北太平洋的日本北海道、韩国济州岛、我国的黄海、渤海、东海等地。厚壳贻贝味道鲜美、营养价值高, 是浙江沿海重要的海洋渔业资源。但近年来在经济利益的驱使下, 酷渔滥捕现象严重; 同时由于近海污染严重, 生态环境日益恶化, 厚壳贻贝资源萎缩迅速。现代遗传学观点认为, 一个物种的遗传多样性高低与其适应能力、生存能力和进化潜力密切相关(Rosen, 1986), 因此研究评价物种种质资源遗传多样性的水平是恢复和持续利用自然资源前提和基础。但目前与厚壳贻贝有关的研究多见于厚壳贻贝营养价值、贻贝多糖与脂类的药用价值、人工育苗等方面(姚滢等, 2005; 袁高峰等, 2007; 常抗美等, 2007; 郭宝英等, 2012), 而有关厚壳贻贝遗传多样性的研究报道很少。
线粒体DNA(mtDNA)是细胞中相对独立的核外遗传物质, 广泛存在于生物体的各种组织细胞中, 易于分离纯化, 其控制区又称为D环(D-Loop), 是线粒体基因组中最主要的一段非编码区, 具有碱基替换速率快、存在丰富的序列变异等特点, 特别适合于检测种群内及种群间的遗传多样性(位正鹏, 2008)。动物mtDNA一般都是母系遗传, 尽管偶然会在果蝇和大鼠个体基因组中发现比例非常低的父系mtDNA(大约10–4—10–3) (Rawson et al, 1995; Śmietanka et al, 2017)。而贻贝属mtDNA却广泛存在双单亲遗传现象(doubly uniparental inheritance, DUI), 即子代同时继承母系的雌性mtDNA (Female mtDNA, F mtDNA)或父系雄性mtDNA (male mtDNA, M mtDNA)。雌性成体中基本都是F mtDNA, 而雄性成体生殖腺中基本都是M mtDNA, 机体其它组织则为F mtDNA (Cao et al, 2004; Chakrabarti et al, 2006; Cogswell et al, 2006; Obata et al, 2006; Breton et al, 2007)。因此, 本研究以厚壳贻贝F mtDNA D-loop为标记, 对厚壳贻贝5个地理群体F mtDNA D-loop的序列多态性进行研究, 探讨东海区厚壳贻贝的遗传多样性及种群遗传结构, 以期为厚壳贻贝的遗传育种和种质资源保护提供参考。
1 材料与方法 1.1 样品采集厚壳贻贝样本采自浙江省舟山嵊山岛(SS)、宁波渔山岛(YS)、温州南麂岛(WZ)和福建省宁德湾(ND)、莆田南日岛(PT)。厚壳贻贝活体带回实验室, 取外套膜置于95%乙醇中–20℃冻存备用。
1.2 基因组DNA的提取取50mg贻贝外套膜, 剪碎, 加入300μL裂解液(50mmol/L Tris-HCl, pH 8.0; 25mmol/L EDTA; 1% SDS)和100μg蛋白酶K, 55℃水浴消化完全后用酚/氯仿法抽提基因组DNA, 干燥后加100μL T.E缓冲液(10mmol/L Tris-HCl, 1mmol/L EDTA)溶解备用。
1.3 厚壳贻贝雌性线粒体D-Loop片段的PCR扩增与测序根据厚壳贻贝已报道的F mtDNA D-loop核酸序列(GenBank accession: AF315574)设计了PCR扩增引物McDS1与McDR1(有义链引物McDS: 5′-AAG CAA TGT TGT GCC TAT GA-3′, 反义链引物McDR: 5′-TCT ATT ATG CCT TTG TTT TG-3′), 扩增目的片段预期长度大约1000bp, PCR扩增DNA聚合酶使用有3′—5′外切酶活性的Ex-Taq酶(TaKaRa公司)。PCR反应程序: 94℃预变性3min; 94℃变性1min, 60℃退火1min, 72℃延伸1min, 循环36次; 72℃后延伸5min。PCR产物回收按上海华舜生物工程有限公司Gel Extraction Mini Kit试剂盒进行, 回收的PCR产物送Invitrogen公司正反链双向测序。
1.4 数据分析测定的序列由人工校正后, 用ClustalX2.0软件(Thompson et al, 1997)进行序列比对。使用MEGA6软件(Tamura et al, 2007)计算变异位点(Variable sites)、简约信息位点(Parsimony informative sites)、碱基组成(Base composition)、转换/颠换值、群体内与群体间遗传距离, 单倍型系统树的构建采用邻接法(Neighbor joining, NJ), 选取基于Kimura 2-parameter遗传距离的模型, 自展值(Bootstrap)设为1000次, 替换模型选择Pairwise Deletion, 其余选项默认; DnaSP 5.0软件(Rozas et al, 2003)计算单倍型数量、单倍型多样性(Haplotype diversity, Hd)、核苷酸多态性(Nucleotide diversity, π)、平均核苷酸差异度(Average number of nucleotide differences, K)、基因流(Nm)、Tajima’s D中性检验及核苷酸不配对分析(mismatch distribution); 采用Arlequin3.5软件(Excoffier et al, 2005)进行分子方差分析(analysis of molecular variance, AMOVA), 计算群体间遗传分化系数(F-statistics, Fst)及其显著性(重复次数1000);采用Network5.1软件包(Bandelt et al, 1999)中的介邻接网络法(Median Joining, MJ)构建厚壳贻贝各单倍型的中介网络图。
2 结果 2.1 厚壳贻贝D-loop区序列特征及单倍型分布PCR扩增产物经电泳检测为大约1000bp的1条单一条带, 内嵌套引物测序后去除两端不准确部分, 最后在5个厚壳贻贝自然群体145个个体的F mtDNA D-loop区保留了准确的685—688bp序列。厚壳贻贝F mtDNA D-loop区A、T、C和G碱基平均含量分别为34.1% (33.8%—34.4%)、30.3% (29.9%—30.6%)、15.2% (14.7%—15.6%)和20.4% (20.1%—20.7%), 其中A+T含量(64.4%)明显高于G+C含量(35.6%), 表现出明显的AT偏倚性, 符合mtDNA D-loop区碱基组成特点。
序列比对的691个位点中存在82个变异位点, 占分析总位点数的11.9%, 其中简约信息位点(parsimony informative sites)30个、单突变位点(singleton variable sites)40个和插入/缺失(insertion/deletion, indel) 10个。转换(transition)位点53个, 颠换(transversion)位点16个, 3个转换/颠换同时存在位点, 转换/颠换比R=3.313, 表明厚壳贻贝F mtDNA D-loop区序列未达到突变饱和状态, 适合遗传变异研究。
145个厚壳贻贝个体的F mtDNA D-loop区共定义了65个单倍型, 其中单倍型Hap14和Hap48为5个群体共享单倍型, 分别占48/145和15/145, 另有10个单倍型(Hap11、Hap15、Hap17、Hap18、Hap19、Hap23、Hap25、Hap29、Hap52、Hap55)为部分群体间共享, 其余53个单倍型为各个群体所独享, 各单倍型在种群中的分布见表 1。
| 名称 | GenBank 序列号 | SS | YS | WZ | ND | PT | 合计 |
| Hap1 | 1ND22 | 1 | 1 | ||||
| Hap2 | 2PT28 | 1 | 1 | ||||
| Hap3 | 3PT17 | 1 | 1 | ||||
| Hap4 | 4PT25 | 1 | 1 | ||||
| Hap5 | 5WZ09, 6WZ20 | 2 | 2 | ||||
| Hap6 | 7YS16 | 1 | 1 | ||||
| Hap7 | 8YS26 | 1 | 1 | ||||
| Hap8 | 9YS07 | 1 | 1 | ||||
| Hap9 | 10YS18, 11YS19 | 2 | 2 | ||||
| Hap10 | 12YS03 | 1 | 1 | ||||
| Hap11 | 13PT04, 14SS09, 15YS05 | 1 | 1 | 1 | 3 | ||
| Hap12 | 16WZ17 | 1 | 1 | ||||
| Hap13 | 17PT12 | 1 | 1 | ||||
| Hap14 | 18YS28, 19SS11, 20WZ22, 21YS04, 22WZ16, 23WZ13, 24WZ02, 25WZ06, 26SS32, 27SS29, 28SS28, 29SS25, 32YS21, 33ND06, 34SS15, 35PT19, 36SS16, 37SS18, 38SS23, 39PT20, 40SS17, 41PT24, 42PT15, 43PT06, 44ND04, 45YS15, 46YS14, 47ND27, 48ND24, 49PT21, 60ND11, 73SS07, 74YS09, 75WZ24, 88ND18, 89ND26, 90PT03, 91YS12, 92PT22, 93SS03, 94SS26, 95SS33, 96WZ01, 97YS02, 98WZ04, 99WZ07, 100WZ10, 101YS24 | 14 | 9 | 10 | 8 | 7 | 48 |
| Hap15 | 30ND05, 31SS05 | 1 | 1 | 2 | |||
| Hap16 | 50YS13 | 1 | 1 | ||||
| Hap17 | 51ND13, 52WZ08 | 1 | 1 | 2 | |||
| Hap18 | 53ND19, 54SS22 | 1 | 1 | 2 | |||
| Hap19 | 55WZ11, 56YS20 | 1 | 1 | 2 | |||
| Hap20 | 57YS06 | 1 | 1 | ||||
| Hap21 | 58ND29 | 1 | 1 | ||||
| Hap22 | 59WZ14 | 1 | 1 | ||||
| Hap23 | 61SS02, 62YS25, 63YS30 | 1 | 2 | 3 | |||
| Hap24 | 64PT27 | 1 | 1 | ||||
| Hap25 | 65SS12, 66SS21, 67WZ05 | 2 | 1 | 3 | |||
| Hap26 | 68SS19 | 1 | 1 | ||||
| Hap27 | 69WZ23 | 1 | 1 | ||||
| Hap28 | 70PT08 | 1 | 1 | ||||
| Hap29 | 71PT09, 72YS10 | 1 | 1 | 2 | |||
| Hap30 | 76ND15 | 1 | 1 | ||||
| Hap31 | 77SS13 | 1 | 1 | ||||
| Hap32 | 78SS14 | 1 | 1 | ||||
| Hap33 | 79YS08 | 1 | 1 | ||||
| Hap34 | 80PT10 | 1 | 1 | ||||
| Hap35 | 81ND02 | 1 | 1 | ||||
| Hap36 | 82PT18 | 1 | 1 | ||||
| Hap37 | 83SS01 | 1 | 1 | ||||
| Hap38 | 84SS20 | 1 | 1 | ||||
| Hap39 | 85SS27 | 1 | 1 | ||||
| Hap40 | 86ND09 | 1 | 1 | ||||
| Hap41 | 87YS01 | 1 | 1 | ||||
| Hap42 | 102SS24 | 1 | 1 | ||||
| Hap43 | 103YS31 | 1 | 1 | ||||
| Hap44 | 104WZ18 | 1 | 1 | ||||
| Hap45 | 105ND01, 106ND12 | 2 | 2 | ||||
| Hap46 | 107SS30 | 1 | 1 | ||||
| Hap47 | 108YS27 | 1 | 1 | ||||
| Hap48 | 109ND23, 110WZ21, 111PT07, 112YS11, 113ND03, 114ND14, 115ND21, 116ND28, 117PT02, 132PT05, 133PT11, 134PT13, 135WZ15, 136YS17, 145SS08 | 1 | 2 | 2 | 5 | 5 | 15 |
| Hap49 | 118ND20 | 1 | 1 | ||||
| Hap50 | 119WZ12 | 1 | 1 | ||||
| Hap51 | 120YS23 | 1 | 1 | ||||
| Hap52 | 121ND07, 123ND25, 124WZ19 | 1 | 2 | 3 | |||
| Hap53 | 122PT26 | 1 | 1 | ||||
| Hap54 | 125ND10 | 1 | 1 | ||||
| Hap55 | 126ND16, 127SS04, 128SS06, 129WZ03 | 2 | 1 | 1 | 4 | ||
| Hap56 | 130SS10 | 1 | 1 | ||||
| Hap57 | 131YS29 | 1 | 1 | ||||
| Hap58 | 137SS31 | 1 | 1 | ||||
| Hap59 | 138ND08 | 1 | 1 | ||||
| Hap60 | 139ND17 | 1 | 1 | ||||
| Hap61 | 140PT16 | 1 | 1 | ||||
| Hap62 | 141PT01 | 1 | 1 | ||||
| Hap63 | 142PT23 | 1 | 1 | ||||
| Hap64 | 143YS22 | 1 | 1 | ||||
| Hap65 | 144PT14 | 1 | 1 | ||||
| 33 | 31 | 24 | 30 | 27 | 145 | ||
| 15 | 16 | 21 | 40 | 37% | 25.5 |
分析表明, 厚壳贻贝各群体的遗传多样性差异不明显, 5个群体的单倍型多样度(Hd)为0.824±0.068 (嵊山)—0.919±0.035 (宁德), 核苷酸多样度(π)为0.00440 (温州)—0.00610 (宁德), 在5个群体中, 宁德群体的遗传多样性相对最丰富; 将5个群体作为一个整体时, 单倍型多样性(Hd)为0.879±0.024, 核苷酸多态性(π)为0.00523, 呈现出较高的单倍型多样性和较低的核苷酸多样性。各群体内的遗传多样性指数详见表 2。
| 种群 | 样本数 | 变异位点数 | 单倍型数 | 单倍型多样(Hd) | 核苷酸多样度(π) | 平均核苷酸差异度(K) | Tajima’s D |
| 嵊山 | 33 | 32 | 18 | 0.824±0.068 | 0.00444 | 3.032 | –1.84933 (P < 0.05) |
| 渔山 | 31 | 36 | 20 | 0.916±0.043 | 0.00538 | 3.688 | –1.94751 (P < 0.05) |
| 温州 | 24 | 25 | 14 | 0.833±0.077 | 0.00440 | 3.011 | –1.59643 (0.10 > P > 0.05) |
| 宁德 | 30 | 34 | 17 | 0.919±0.035 | 0.00610 | 4.158 | –1.24280 (P > 0.10) |
| 莆田 | 27 | 29 | 17 | 0.899±0.044 | 0.00539 | 3.563 | –1.34786 (P > 0.10) |
| 总体 | 145 | 82 | 65 | 0.879±0.024 | 0.00523 | 3.563 | –2.32375 (P < 0.01) |
对厚壳贻贝5个群体间的遗传分化系数(Fst)和基因流(Nm)进行检测, 结果显示群体间Fst值都很低, 且Nm值都很高(Nm绝对值> 1), 表明5个群体间存在丰富的基因交流。但宁德群体与浙江沿海的3个群体(嵊山、渔山、温州)Fst值相对较高, 且差异显著(P < 0.05), 表明宁德群体与这3个群体间出现遗传分化。
对厚壳贻贝D-loop序列的变异进行分子方差分析(Analysis of Molecular Variance, AMOVA), 总方差剖分为群体间的方差(Va)和群体内的方差(Vb), 并进行显著性检验(表 4)。结果显示群体间方差(Va)占总变异的2.96%, 群体内的方差(Vb)占97.04%, 变异主要发生在群体内, 固定指数(Fixation index, Fst)为0.02960, 差异显著(P=0.04692±0.00523, P < 0.05), 支持厚壳贻贝群体间出现遗传分化, 存在一定程度的遗传结构。
| 嵊山 | 渔山 | 温州 | 宁德 | 莆田 | |
| 嵊山 | –17.37 | –14.94 | 2.31 | 9.17 | |
| 渔山 | –0.01412 (0.80180±0.0345) |
–15.56 | 2.81 | 18.20 | |
| 温州 | –0.01751 (0.71171±0.0417) |
–0.01751 (0.84685±0.0365) |
3.31 | 45.81 | |
| 宁德 | 0.09934 (0.00000±0.0000) |
0.08204 (0.01802±0.0121) |
0.06809 (0.04505±0.0152) |
438.64 | |
| 莆田 | 0.02756 (0.07207±0.0227) |
0.01370 (0.20721±0.0305) |
0.00470 (0.31532±0.0579) |
0.00049 (0.27027±0.0450) |
|
| 注:下三角为遗传分化系数Fst(P), 上三角为基因流Nm | |||||
| 变异来源 | 自由度d.f. | 平方和 | 方差组分 | 变异百分率(%) | 固定指数(Fst) |
| 群体间 | 4 | 13.846 | 0.05609Va | 2.96 | 0.02960 (P < 0.05) |
| 群体内 | 140 | 257.482 | 1.83916Vb | 97.04 | |
| 合计 | 144 | 271.328 | 1.89525 | ||
| 注: Va表示群体间方差组分, Vb表示群体内方差组分 | |||||
根据Kimura 2-parameter模型计算了5个群体内的平均遗传距离和群体间的平均遗传距离, 结果见表 5。群体间的为0.0043—0.0063, 表明5个群体间的平均遗传距离与群体内的处于同一水平, 群体间未出现明显的遗传分化。基于群体间的平均遗传距离采用NJ法聚类分析显示莆田、温州、渔山、嵊山4个群体间存在一定的地理距离关系, 而宁德群体则体现出地理隔离, 可能与宁德群体处于封闭的峡湾存在联系。
| 嵊山 | 渔山 | 温州 | 宁德 | 莆田 | |
| 嵊山 | 0.0044 | 0.0048 | 0.0043 | 0.0058 | 0.0051 |
| 渔山 | 0.0054 | 0.0048 | 0.0063 | 0.0055 | |
| 温州 | 0.0043 | 0.0056 | 0.0049 | ||
| 宁德 | 0.0054 | 0.0058 | |||
| 莆田 | 0.0061 | ||||
| 注:上三角为群体间的平均遗传距离, 对角线为群体内的平均遗传距离 | |||||
在MEGA 5.0软件中, 采用K 2-P距离模型, 以邻接法(Neighbor-joining, NJ)重建65个单倍型间系统发生关系如图 1所示, Bootstrap 1000次自举检验。结果显示65个单倍型聚为A、B两支, 网络分析适合于推断大样本且个体间的遗传距离比较近的种内水平的系统发育关系, 是分析mtDNA单倍型序列之间关系的有效方法。利用Network 5.1对厚壳贻贝雌性(F)线粒体D-loop单倍型进行网络分析。65个单倍型也聚为两个单倍型族群, 支持东海区5个厚壳贻贝群体F mtDNA存在2个母系来源; 族群内个体间联系紧密, 单倍型之间仅有一到两步突变, 演化关系呈星状, 提示厚壳贻贝在历史上曾发生种群扩张。
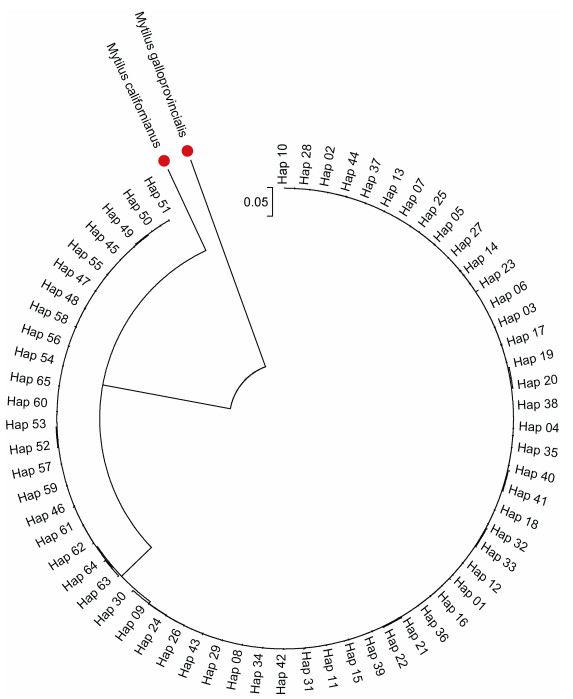 |
| 图 1 东海区厚壳贻贝5个群体mtDNA(D-loop)序列单倍型的NJ系统树 Fig. 1 The phylogenetic tree of M. coruscus female mitochondial D-loop haplotypes of the East China Sea (by neighbor joining method) 注:紫贻贝M. galloprovincialis、加州贻贝M. californianus作为外类群 |
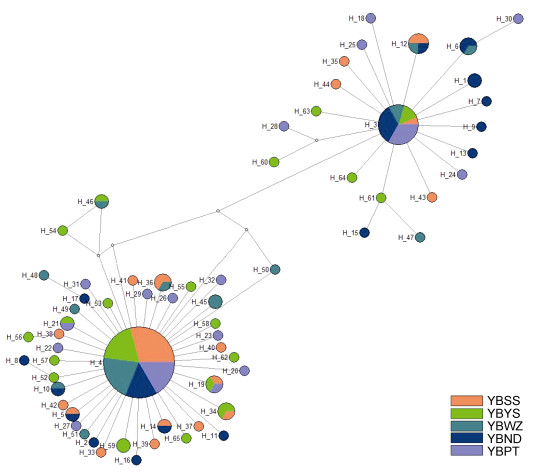 |
| 图 2 厚壳贻贝单倍型网络图 Fig. 2 The median joining network of M. coruscus female mitochondrial D-loop 注:圆圈大小与单倍型频率大小成比例 |
Tajima’s D中性检验5群体均为负值, 其中嵊山、渔山和温州3群体差异显著(P < 0.10), 提示这3群体不符合中性突变, 可能发生过群体扩张; 而宁德和莆田群体2群体则差异不显著(P > 0.10), 则符合中性突变, 在过去没有出现群体扩张, 群体大小较稳定。Fu and Li’s Fs = –4.97555 (P < 0.02)。
由于厚壳贻贝F mtDNA D-loop单倍型分为A/B 2个族群, 所以对两个族群分别进行歧点分布分析。结果显示两个族群单倍型碱基差异分布都呈单峰分布(图 3), 提示厚壳贻贝在浙江海域分布范围内曾经历过近期种群扩张事件(杨振雄等, 2014)。这一结果也得到了Tajima检验(A族: D =–2.12328, P < 0.05; A族: D =–1.98992, P < 0.05)和Fu检验(A族: Fs =–14.970; B族: Fs = –4.865)的支持。
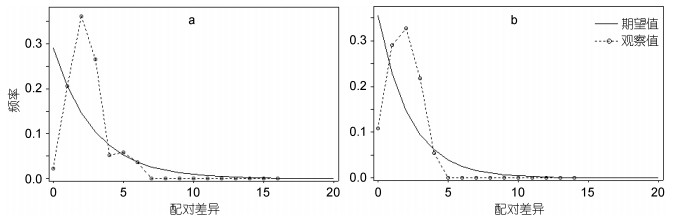 |
| 图 3 厚壳贻贝F mtDNA D-loop单倍型的歧点分布 Fig. 3 Mismatch distribution of the observed mitotypes of M. coruscus 注: a表示A族F mtDNA歧点分布, b表示B族F mtDNA歧点 |
海洋经济贝类是重要的海洋渔业资源之一, 在过度捕捞、栖息地生态环境恶化等生存压力作用下, 世界各地沿海重要经济贝类资源均不同程度呈下降趋势, 某些物种甚至处于濒危状态(Monteforte et al, 1992; Park et al, 1998; 苏天凤等, 2005)。因此有关重要经济贝类种群遗传结构和遗传多样性的研究也日益得到重视, 以期为贝类的种质资源管理、保护、遗传育种等提供参考数据(Nagashima et al, 2005; Mahidol et al, 2007)。
贝类浮游生活的幼体可随海流进行长距离扩散(Seed, 1969), 一般可以在较大尺度上进行基因交流。Cho等报道韩国沿海魁蚶(Scapharca broughtonii)属于同一种群, 而与中国山东群体、俄罗斯Vladivostok群体存在显著的差异(Cho et al, 2007); Mahidol等基于mtDNA 16S RNA序列数据研究显示泰国湾内的亚洲日月蛤(Amusium pleuronectes)群体没有显著遗传差异, 属于同一种群, 而与安达曼海种群发生遗传分化(Mahidol et al, 2007)。本研究中, 舟山、温州两地采样点虽然相距大约400km, 但厚壳贻贝群体之间遗传差异小(Gamma St: 0.01167)、并且基因交流充分(Nm: 42.33), 没有发生遗传分化, 也属于同一个地理种群。同时两地厚壳贻贝种群F mtDNA D-loop单倍型多态性均很高, 分别为0.93447、0.94737, 说明两地厚壳贻贝资源虽然由于过度捕捞等使资源量有所衰退, 但还保存了丰富遗传多样性。因此对于厚壳贻贝种质资源的保护, 我们建议在沿海厚壳贻贝自然分布区, 大尺度上选定一些岛礁予以保护, 严禁采集即可维持其现有的种质遗传多样性。
单倍型聚类分析与网络分析均将厚壳贻贝F mtDNA D-loop分为A/B两个单倍型族, 提示厚壳贻贝F mtDNA可能有两个来源; 并且网络关系图显示Hap_16与Hap_18分别为A/B两个单倍型族的祖先单倍型, 族群内单倍型间联系紧密, 单倍型之间仅有一到两步突变, 演化关系呈星状, 提示两地厚壳贻贝曾发生种群扩张; 两个族群单倍型碱基差异分布都呈单峰分布, 也支持厚壳贻贝发生种群扩张事件。厚壳贻贝属暖温带种, 我国沿海分布南限为厦门。在中全新世大西洋晚期至亚北方期阶段(6.5—2.4Ka BP), 浙江省气候波动频繁, 浙北舟山地区海滩岩形成时的温暖期海水温度比现今高3.25—4.25℃, 与现代广东南部沿海地区海水温度相当, 海滩岩中热带贝类化石占29.6%, 而现生贝类中没有热带贝类; 由中全新世至晚全新世, 气候总体趋冷, 但在1.1Ka BP左右, 有一个令人瞩目的高温阶段, 气温比现代大约高3.3℃; 1.1Ka BP后, 气温陡降3.2℃, 现代气候形成(孙金龙等, 2009; 徐辉龙等, 2010; 齐岩辛等, 2013)。因此, 我们认为:厚壳贻贝群体在气候变迁过程中可能多次退出浙江海域(海滩岩中未见厚壳贻贝化石记录), 最近的一次退出事件可能与1.1Ka BP左右的高温气候相关。而在随后的降温过程中, 海水温度又逐渐适宜厚壳贻贝生存, 发生奠基者效应(founder effect)并进行群体扩张, 由于扩张的时间较短, 虽然累计了单倍型多态性, 但单倍型核苷酸序列差异却很低。同时由北南下浙江海域的厚壳贻贝地理种群可能有两个, 因此在本研究中检测到两个厚壳贻贝F mtDNA单倍型族。
贝类浮游生活的幼体可随海流进行长距离扩散, 但扩散距离也受到过度捕捞、海岸礁石底质差异、近岸海流等因素影响, 因此贝类不同地域之间的遗传多样性、基因交流、群体遗传结构难以预计。
4 结论厚壳贻贝在浙江沿海潮下带礁石底质0—30m水深区间是优势物种, 但近年来的由于滥采和过度捕捞, 资源萎缩迅速, 种质资源亟待保护, 以免其重蹈大黄鱼的覆辙。本研究显示, 舟山海域厚壳贻贝遗传多样性丰富, 并且两个相距大约60km的岛屿群体没有产生遗传分化, 属于同一地理种群, 这可能与贝类在浮游幼虫阶段可随海流迁移, 散布范围广, 不同岛屿间基因交流频繁相关。因此, 对于厚壳贻贝种质资源的保护, 我们建议在沿海厚壳贻贝自然分布区, 大尺度上选定一些岛礁予以保护, 严禁采集即可维持其现有的种质遗传多样性。但具体在多大尺度上、选择哪些合适的岛屿作为保护区还有待进一步研究。
齐岩辛, 张岩, 陈美君, 等. 2013. 浙江海岛区地质遗迹资源及其价值. 地质调查与研究, 36(4): 311-317 |
孙金龙, 徐辉龙. 2009. 中国的海滩岩研究与进展. 热带海洋学报, 28(2): 103-108 DOI:10.3969/j.issn.1009-5470.2009.02.017 |
苏天凤, 朱彩艳, 江世贵. 2005. 海南岛大珠母贝遗传多样性分析. 海洋科学, 29(8): 20-22 DOI:10.3969/j.issn.1000-3096.2005.08.006 |
杨振雄, 毛阳丽, 宋娜, 等. 2014. 浙江和福建沿海厚壳贻贝Mytilus coruscus群体的COI序列比较分析. 海洋湖沼通报, (2): 82-88 |
位正鹏, 2008.翡翠贻贝(Perna viridis)线粒体COI片段序列特性和香港巨牡蛎(Crassostrea hongkongensis)线粒体基因组全序列研究.青岛: 中国海洋大学硕士学位论文
|
姚滢, 魏江洲, 王俊, 等. 2005. 厚壳贻贝多糖的提取和免疫学活性研究. 第二军医大学学报, 26(8): 896-899 DOI:10.3321/j.issn:0258-879X.2005.08.020 |
袁高峰, 姚婷, 励炯, 等. 2007. 厚壳贻贝脂溶性提取物抗炎生物活性研究. 浙江大学学报(农业与生命科学版), 33(2): 169-173 DOI:10.3321/j.issn:1008-9209.2007.02.010 |
徐辉龙, 孙金龙, 李亚敏. 2010. 华南沿海全新世海岸带碳酸盐胶结岩的分类及其研究意义. 海洋地质与第四纪地质, 30(3): 53-60 |
郭宝英, 祁鹏志, 李继姬, 等. 2012. 紫贻贝(Mytilus edulis)养殖群体的遗传多样性及遗传结构分析. 海洋与湖沼, 43(1): 52-56 |
常抗美, 吴剑锋. 2007. 厚壳贻贝人工繁殖技术的研究. 南方水产科学, 3(3): 26-30 DOI:10.3969/j.issn.2095-0780.2007.03.005 |
Bandelt H J, Forster P, R hl A, 1999. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 16(1): 37-48 DOI:10.1093/oxfordjournals.molbev.a026036 |
Breton S, Beaupré H D, Stewart D T et al, 2007. The unusual system of doubly uniparental inheritance of mtDNA: Isn't one enough? Trends in Genetics, 23(9): 465-474
|
Cao L Q, Kenchington E, Zouros E, 2004. Differential segregation patterns of sperm mitochondria in embryos of the blue mussel (Mytilus edulis). Genetics, 166(2): 883-894 DOI:10.1534/genetics.166.2.883 |
Chakrabarti R, Walker J M, Stewart D T et al, 2006. Presence of a unique male-specific extension of C-terminus to the cytochrome c oxidase subunit Ⅱ protein coded by the male-transmitted mitochondrial genome of Venustaconcha ellipsiformis (Bivalvia:Unionoidea). FEBS Letters, 580(3): 862-866 DOI:10.1016/j.febslet.2005.12.104 |
Cho E S, Jung C G, Sohn S G et al, 2007. Population genetic structure of the ark shell Scapharca broughtonii Schrenck from Korea, China, and Russia based on COI Gene sequences. Marine Biotechnology, 9(2): 203-216 DOI:10.1007/s10126-006-6057-x |
Cogswell A T, Kenchington E L R, Zouros E, 2006. Segregation of sperm mitochondria in two- and four-cell embryos of the blue mussel Mytilus edulis:implications for the mechanism of doubly uniparental inheritance of mitochondrial DNA. Genome, 49(7): 799-807 DOI:10.1139/g06-036 |
Excoffier L, Laval G, Schneider S, 2005. Arlequin (version 3. 0):an integrated software package for population genetics data analysis. Evolutionary Bioinformatics, 1: 47-50 |
Mahidol C, Na-Nakorn U, Sukmanomon S et al, 2007. Mitochondrial DNA diversity of the Asian moon scallop, Amusium pleuronectes (Pectinidae), in Thailand. Marine Biotechnology, 9(3): 352-359 DOI:10.1007/s10126-006-6137-y |
Monteforte M, Cari o M, 1992. Exploration and evaluation of natural stocks of pearl oysters Pinctada mazatlanica and Pteria sterna (Bivalvia:Pteriidae):la Paz bay, South Baja California, Mexico. Ambio, 21(4): 314-320 |
Nagashima K, Sato M, Kawamata K et al, 2005. Genetic structure of Japanese scallop population in Hokkaido, analyzed by mitochondrial haplotype distribution. Marine Biotechnology, 7(1): 1-10 DOI:10.1007/s10126-004-3046-9 |
Obata M, Kamiya C, Kawamura K et al, 2006. Sperm mitochondrial DNA transmission to both male and female offspring in the blue mussel Mytilus galloprovincialis. Development, Growth & Differentiation, 48(4): 253-261 |
Park S M, Lim J H, Kim J P, 1998. Effect of environmental factors on the growth, glycogen and hemoglobin content of cultured Arkshell, Scapharca broughtonii. J Kor Fish Sci Tech, 31(2): 176-185 |
Rawson P D, Hilbish T J, 1995. Evolutionary relationships among the male and female mitochondrial DNA lineages in the Mytilus edulis species complex. Molecular Biology and Evolution, 12(5): 893-901 |
Rosen R, 1986. Adaptability: the significance of variability from molecule to ecosystem: Michael Conrad. Plenum Press, New York, 1983. U.S., 83pp. Bulletin of Mathematical Biology, 48(5-6): 701-703
|
Rozas J, Sánchez-Delbarrio J C, Messeguer X et al, 2003. DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioinformatics, 19(18): 2496-2497 DOI:10.1093/bioinformatics/btg359 |
Seed R, 1969. The ecology of Mytilus edulis L. (Lamellibranchiata) on exposed rocky shores:I. Breeding and settlement. Oecologia, 3(3-4): 277-316 |
Śmietanka B, Burzyński A, 2017. Disruption of doubly uniparental inheritance of mitochondrial DNA associated with hybridization area of European Mytilus edulis and Mytilus trossulus in Norway. Marine Biology, 164(11): 209 DOI:10.1007/s00227-017-3235-5 |
Tamura K, Dudley J, Nei M et al, 2007. MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4. 0. Molecular Biology and Evolution, 24(8): 1596-1599 DOI:10.1093/molbev/msm092 |
Thompson J D, Gibson T J, Plewniak F et al, 1997. The Clustal_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 25(24): 4876-4882 DOI:10.1093/nar/25.24.4876 |
 2019, Vol. 50
2019, Vol. 50


