中国海洋湖沼学会主办。
文章信息
- 吴仁协, 肖瑶, 牛素芳, 翟云, 张浩冉, 陈伟勇, 黎晓. 2019.
- WU Ren-Xie, XIAO Yao, NIU Su-Fang, ZHAI Yun, ZHANG Hao-Ran, CHEN Wei-Yong, LI Xiao. 2019.
- 基于SLAF-seq技术的黑棘鲷微卫星标记开发及其在鲷科鱼类中的通用性研究
- DEVELOPMENT OF MICROSATELLITE MARKERS FOR ACANTHOPAGRUS SCHLEGELII BASED ON SLAF-SEQ TECHNOLOGY AND GENERALITY IN THE FAMILY SPARIDAE
- 海洋与湖沼, 50(2): 365-377
- Oceanologia et Limnologia Sinica, 50(2): 365-377.
- http://dx.doi.org/10.11693/hyhz20180900230
-
文章历史
- 收稿日期:2018-09-28
- 收修改稿日期:2018-12-05
黑棘鲷(Acanthopagrus schlegelii)是鲷科鱼类最为重要的经济种类之一, 广泛分布于西北太平洋的暖温性中下层水域, 我国南北沿海均产(刘静等, 2016)。黑棘鲷生长迅速、适应能力强, 肉质鲜美、外形美观, 具有较高的食用价值和观赏价值, 是我国重要的海洋捕捞鱼类和增养殖对象(杨慧荣等, 2006)。由于过度捕捞、渔业环境恶化以及人工养殖等原因, 黑棘鲷野生群体资源急剧衰减, 其养殖群体的种质资源状况也不容乐观(许思思, 2011)。为了恢复黑棘鲷资源、增加补充群体数量, 我国沿海各地纷纷开展了大规模的黑棘鲷增殖放流活动(沈新强等, 2007; 梁君等, 2010)。这些措施对黑棘鲷的渔业资源恢复起到了积极作用, 但也可能产生养殖群体与自然群体种质混杂和种群遗传多样性水平下降等不利方面(施晓峰等, 2015; 曹艳, 2016), 加大了对其种质资源评估与分析的复杂性。因此, 必须采用更加精确的评估方法和开发高灵敏度的分子标记, 才能有效评估增殖放流对野生群体遗传特征的影响, 以精确评价增殖放流成效和潜在的生态遗传风险。
微卫星标记(Microsatellites), 一般由2—6个碱基为核心单位组成的短串联排列, 广泛存在于真核生物核基因中。微卫星标记不但具有多态性丰富且等位基因条带易识别等优点(鲁双庆等, 2003), 而且又可克服RAPD、RFLP等分子标记易混淆大小相同但来自不同位点的等位基因的缺点(郭宝英等, 2007), 在家系亲权分析、家系鉴别及系谱认证、个体/家系溯源等方面显示出显著的优势和应用价值(李伟亚等, 2012; 刘永新等, 2015), 已成为目前物种增殖放流效果评估的最常用分析工具(Gonzalez et al, 2009; Wang et al, 2016)。以往的微卫星标记开发主要采用基于传统的富集文库法(Enriched Genomic Library), 不但费时费力、成本高昂, 而且开发效率较低, 通常一次只能筛选出几十个至几百个位点, 难以在短时间内一次性即可开发出大批量的高多态性微卫星标记(孙波等, 2009; Grover et al, 2016)。近年来, 随着高通量测序平台的快速发展和广泛应用, 通过SLAF-seq (Specific-Locus Amplified Fragment Sequencing)技术可以在短时间内获取遍布于物种基因组DNA中海量序列信息(Sun et al, 2013), 进而依据其序列开发出特异的分子标记, 可筛选出大量高多态性的分子标记。SLAF-seq技术在保障有效标记数量和质量的同时, 还能大幅度降低测序成本, 是一种高效、低成本的分子标记开发方法(张浩冉等, 2017)。目前SLAF-seq技术已成功用于多种动物的微卫星标记开发, 如沼泽山雀(Parus palustris) (万冬梅等, 2016)、沙带鱼(Lepturacanthus savala) (张浩冉等, 2018)、带鱼(Trichiurus japonicus) (张浩冉等, 2019)、蓝圆鲹(Decapterus maruadsi) (翟云等, 2018; Niu et al, 2018)等。
目前, 有关黑棘鲷的微卫星标记开发已有一些报道, 如Jeong等(2003, 2007)通过构建基因组酶切片段文库共开发了8个二碱基重复标记, Liu等(2007)基于6种鲷科近缘种微卫星位点的跨物种扩增筛选出16个适用于黑棘鲷的二碱基重复标记, 罗新惠(2010)、徐亘博(2010)、Yang等(2015)、Mao等(2016)各自采用磁珠富集法(FIASCO)共开发了73个二、三碱基重复标记。然而, 这些开发研究都是采用传统的富集文库法, 所开发的微卫星标记类型均为短重复序列(二、三碱基), 缺乏长重复序列(四至六碱基)类型标记。可见, 已开展的黑棘鲷微卫星标记开发研究其方法较为陈旧、标记类型单一, 还难以满足黑棘鲷增殖放流遗传效果评估所需的大批量且多样化的微卫星标记。为此, 本研究采用基于高通量测序平台的SLAF-seq技术来筛选黑棘鲷基因组DNA中的微卫星位点, 以开发出高多态性、多样化的微卫星标记, 并在9种鲷科鱼类中进行微卫星标记的跨物种扩增检测, 以期为黑棘鲷的种群遗传资源分析提供数量丰富、类型多样的分子标记和有效的技术支撑, 也为其近缘种的系统进化和群体遗传学研究提供新的分析手段和研究角度。
1 材料与方法 1.1 实验材料和基因组DNA提取实验所用的32尾野生黑棘鲷于2014年3月采集于湛江市遂溪县江洪和广东雷州珍稀海洋生物国家级自然保护区近岸。取鱼体背部肌肉后浸泡在95%的酒精溶液中, 并保存于–20℃条件下。采用蛋白酶-苯酚氯仿法提取样品基因组DNA (Sambrook et al, 2001)。
1.2 SLAF-seq测序和微卫星引物合成选取一份基因组DNA质量较好的肌肉样送至北京百迈客生物科技有限公司进行SLAF-seq测序, 以获得黑棘鲷全基因组范围内的微卫星序列。利用生物信息学方法确定酶切方案, 在酶切片段SLAF标签3'端加A处理基础上, 进行连接Dual-index测序接头、PCR扩増、混样、切胶以及文库质检等步骤的处理(张浩冉等, 2019), 最后上机(Illumina HiSeq 2500, Illumina Inc., San Diego, CA)对样品进行“全基因组随机测序”。随后, 通过微卫星识别软件MISA ( http://pgrc.ipk-gatersleben.de/misa/misa.html/)对二碱基重复次数6次以上、三至六碱基重复次数5次以上的微卫星位点进行搜集与统计, 并进行分类整理。为了获得具有较好的重复性和稳定性的微卫星标记, 按照如下标准挑选测序公司给出的微卫星引物: (1) PCR产物预计长度在180—300bp; (2)引物在距离微卫星重复序列上下游10bp以外; (3)引物长度在17—23bp, 上、下游引物间长度差异应该 < 4bp; (4)引物对GC含为45%—60%, 退火温度为50—65℃; (5)短重复序列的微卫星核心序列重复次数≥8, 长重复序列的微卫星核心序列重复次数≥5。在满足这些条件下随机挑选157个二至六碱基的微卫星位点引物送至生工生物工程(上海)股份有限公司进行引物序列合成。
1.3 PCR反应及微卫星多态性引物筛选采用4个黑棘鲷基因组DNA对合成的157对微卫星引物进行初步筛选, 以获得可扩增出具有多态性片段的微卫星引物。PCR反应的试剂配比体系(20µL)和扩增反应程序见张浩冉等(2018)报道的方法。扩增产物采用8%聚丙烯酰胺凝胶电泳(160V, 60—70min)、1%硝酸银染液染色3—5min, 显色后观察拍照。每对微卫星引物能扩增出2条或2条以上的具有特异性等位基因条带的统计为多态性引物。
1.4 微卫星荧光引物合成和等位基因分型检测参考Schuelke(2000)的M13标记法, 对筛选出的多态性微卫星引物进行荧光修饰, 即在每条正向引物的5′端加上一段M13序列(5′-AGGGTTTTCCC AGTCACG-3′或5′-GAGCGGATAACAATTTCACAC- 3′), 同时合成一条具有同样序列的M13通用引物, 在其5'端分别标记FAM或HEX荧光基团, 引物修饰和合成由上海捷瑞生物工程有限公司完成。在三引物PCR扩增体系条件下(Schuelke, 2000), 采用梯度PCR (52—62℃)重新摸索每对荧光修饰引物的最适退火温度, 再用32个黑棘鲷基因组DNA进行微卫星标记多态性的PCR扩增。PCR反应总体系为20µL, 三引物PCR反应的试剂配比和扩增反应程序参照张浩冉等(2018)的研究报道, 各对引物的PCR反应退火温度见表 1。扩增产物经凝胶电泳和染色后, 选择扩增特异性强、产量高的PCR产物送至武汉天一辉远生物科技有限公司进行微卫星等位基因的分型检测。
采用黄鳍棘鲷(Acanthopagrus latus)(n=6, 湛江近岸)、太平洋棘鲷(Acanthopagrus pacificus)(n=4, 湛江近岸)、澳洲棘鲷(Acanthopagrus australis)(n=2, 湛江近岸)、台湾棘鲷(Acanthopagrus taiwanensis)(n=1, 台湾近岸)、平鲷(Rhabdosargus sarba)(n=6)、蓝点赤鲷(Pagrus caeruleostictus)(n=2, 北海近岸)、真赤鲷(Pagrus major)(n=6, 湛江近岸)、二长棘犁齿鲷(Evynnis cardinalis)(n=7, 湛江近岸)和黄牙鲷(Dentex hypselosomus)(n=5, 湛江近岸)等9种鲷鱼科鱼类进行黑棘鲷微卫星标记的跨物种扩增, 以检测开发的微卫星标记在其近缘种中的通用性。微卫星标记跨物种的PCR扩增体系、反应程序及电泳条件同1.4。扩增结果出现1条或1条以上的特异性等位基因条带的微卫星标记即为具有通用性位点。
1.6 数据分析各位点的遗传多样性参数, 如等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)等由Cervus 3.0.7软件计算(Kalinowski et al, 2007)。采用GenePop 4.3检测各位点的哈迪-温伯格平衡以及位点间的连锁不平衡现象(Rousset, 2008)。各位点的无效等位基因频率(Fua)(Brookfeld-1算法)和近交系数(Fis)分别由MicroChecker v.2.2.3和FSTAT 2.9.3软件分析(Goudet, 2001; Van Oosterhout et al, 2004)。
2 结果与分析经SLAF-seq简化基因组深度水平测序后, 获得黑棘鲷基因组DNA 3.47 M reads读长, 平均GC含量为41.24%, 其中大部分的碱基(75.4%)能满足测序质量要求(测序质量值Q≥30)。测序数据过滤后, 获得137358个SLAF标签, 标签长度选择范围为415—514bp, 平均测序深度为11.38×。采用微卫星识别软件MISA (http://pgrc.ipk-gatersleben.de/misa/misa.html/)对所得序列进行微卫星位点筛选, 共识别出38797个微卫星位点。其中单碱基重复(Mono-nucleotide)有16308个(占总位点的42.03%, 下同), 二碱基重复(Di-nucleotide)有16901个(43.56%), 三碱基重复(Tri-nucleotide)和四碱基重复(Tetra-nucleotide)分别有3526个(9.08%)和1717个(4.42%), 五碱基重复(Penta-nucleotide)和六碱基重复(Hexa-nucleotide)分别仅有303个(0.78%)和42个(0.11%)。
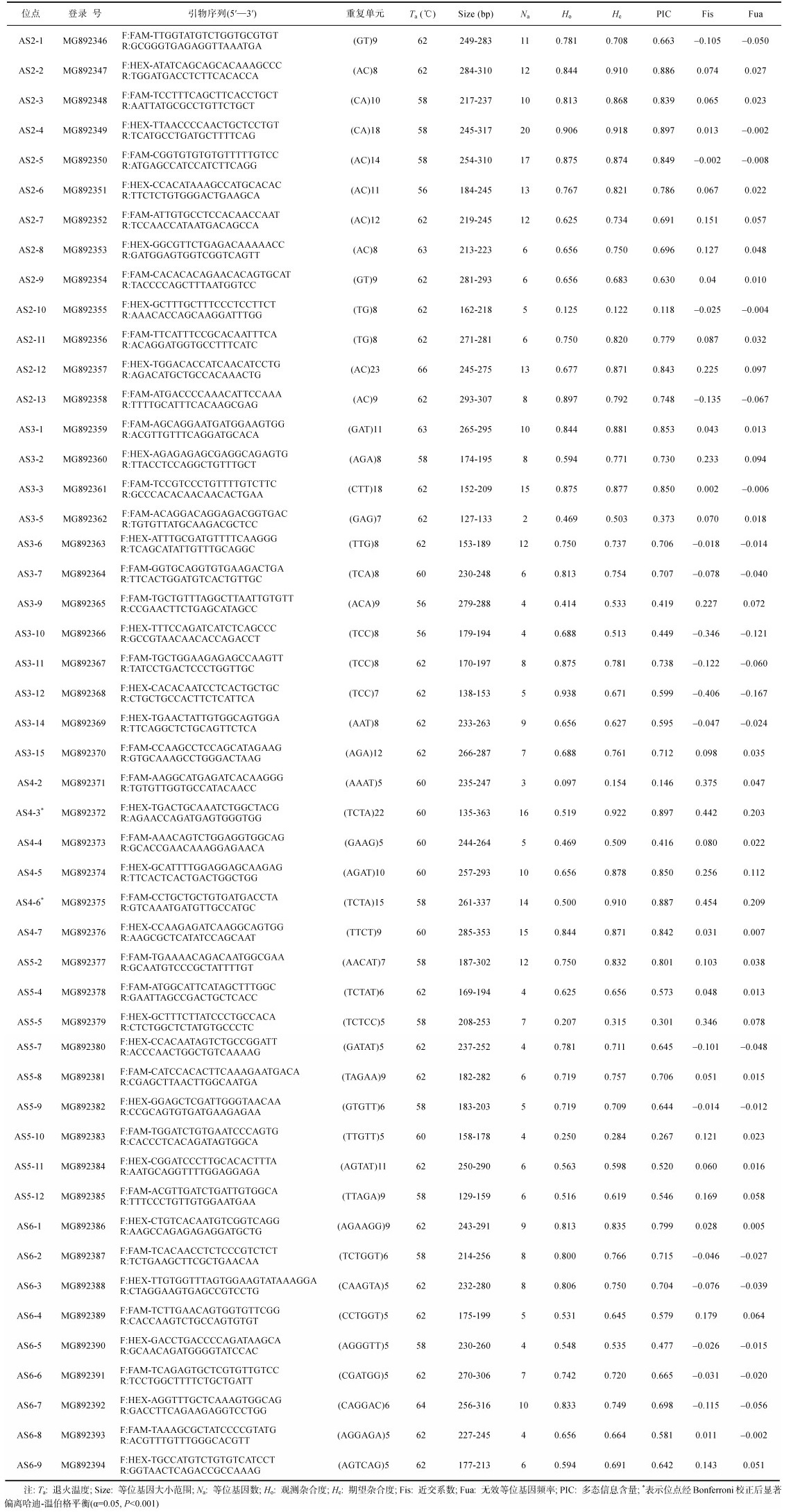
在随机挑选的157个二至六碱基重复微卫星位点中, 经4个黑棘鲷基因组DNA的PCR扩增检测, 筛选出57个(36.3%)位点具有多态性。这些多态性引物经5′端添加M13尾巴修饰后, 有49个位点在随后的PCR反应中能稳定扩增出清晰的等位基因条带, 包括13个二碱基、12个三碱基、6个四碱基、9个五碱基和9个六碱基重复位点, 其引物序列信息已上传至GenBank, 登录号为MG892346—MG892394。
49个微卫星位点在32个黑棘鲷基因组DNA中共扩增出407个等位基因, Na在2 (AS-5)和20 (AS-4)之间, 平均值为8.3(表 1)。Ho在0.097 (AS4-2)和0.938 (AS3-12)之间, He在0.122 (AS2-10)和0.922 (AS4-3)之间, 平均值分别为0.663和0.701。PIC分析显示, 49个位点的PIC平均值为0.6545, 除AS2-10、AS3-5、AS3-9、AS3-10、AS4-2、AS4-4、AS5-5、AS5-10、AS6-5等9个位点的PIC值小于0.5外, 其余40个位点的PIC值均大于0.5, 显示出较高的多态性。经Bonferroni校正后(校正P < 0.0011), 有47个位点均符合哈迪-温伯格平衡, 且各位点间未检测到连锁不平衡现象, 仅2个位点(AS4-3, AS4-6)偏离合哈迪-温伯格平衡。
跨物种扩增结果表明, 有43个黑棘鲷微卫星标记可在1种或1种以上鲷科鱼类中有效扩增(表 2)。其中AS3-5和AS3-12可在8种鲷科鱼类中扩增, AS3-11和AS5-2可在7种鲷科鱼类中扩增, AS3-7可在6种鲷科鱼类中扩增, AS2-4、AS3-8、AS4-5等3个位点可在5个物种中扩增, AS3-2、AS3-6、AS3-10、AS4-4、AS4-7、AS5-7等6个位点可在4个物种中扩增, AS2-1、AS2-2、AS2-3、AS2-9、AS2-10、AS2-11、AS2-13、AS3-1、AS3-14、AS3-15、AS4-2、AS5-5、AS5-8、AS5-9、AS5-12、AS6-2、AS6-4、AS6-8等18个位点在3个物种中扩增, AS2-12、AS3-3、AS3-9、AS4-3、AS4-6、AS5-4、AS5-10、AS6-1、AS6-6、AS6-7等10个位点在2个物种中扩增, AS6-5位点仅在1个物种中扩增。
| 位点 | 黄鳍棘鲷 (n=6) |
澳洲棘鲷 (n=2) |
太平洋棘鲷 (n=4) |
台湾棘鲷 (n=1) |
平鲷 (n=6) |
蓝点赤鲷 (n=2) |
真赤鲷 (n=6) |
二长棘犁齿鲷 (n=7) |
黄牙鲷 (n=5) |
| AS2-1 | +(9) | +(4) | +(7) | - | - | - | - | - | - |
| AS2-2 | +(10) | +(2) | +(6) | - | - | - | - | - | - |
| AS2-3 | +(12) | +(4) | +(8) | - | - | - | - | - | - |
| AS2-4 | - | +(2) | +(7) | - | +(8) | +(2) | +(6) | - | - |
| AS2-9 | +(7) | +(3) | +(5) | - | - | - | - | - | - |
| AS2-10 | +(6) | +(2) | +(1) | - | - | - | - | - | - |
| AS2-11 | +(7) | - | +(7) | - | +(5) | - | - | - | - |
| AS2-12 | +(10) | +(2) | - | - | - | - | - | - | - |
| AS2-13 | +(8) | +(2) | +(4) | - | - | - | - | - | - |
| AS3-1 | +(6) | +(3) | +(4) | - | - | - | - | - | - |
| AS3-2 | +(9) | +(3) | +(4) | - | - | - | - | - | +(7) |
| AS3-3 | - | +(3) | - | - | +(9) | - | - | - | - |
| AS3-5 | +(6) | +(2) | +(5) | - | +(7) | +(2) | +(10) | +(7) | +(7) |
| AS3-6 | +(9) | +(2) | +(4) | +(9) | - | - | - | - | |
| AS3-7 | +(9) | +(4) | - | - | - | +(4) | +(6) | +(14) | +(10) |
| AS3-8 | - | - | +(3) | - | +(3) | +(5) | +(5) | +(3) | - |
| AS3-9 | +(10) | +(2) | - | - | - | - | - | - | - |
| AS3-10 | +(8) | +(2) | +(4) | - | +(6) | - | - | - | - |
| AS3-11 | +(11) | +(2) | +(7) | - | +(6) | +(2) | - | +(7) | +(5) |
| AS3-12 | +(12) | +(4) | +(8) | - | +(12) | +(4) | +(12) | +(12) | +(10) |
| AS3-14 | +(7) | +(2) | +(5) | - | - | - | - | - | - |
| AS3-15 | +(7) | +(2) | +(6) | - | - | - | - | - | - |
| AS4-2 | +(11) | +(4) | +(8) | - | - | - | - | - | - |
| AS4-3 | +(9) | +(3) | - | - | - | - | - | - | - |
| AS4-4 | +(6) | +(4) | +(8) | - | +(11) | - | - | - | - |
| AS4-5 | +(6) | +(2) | +(4) | - | +(4) | - | +(6) | - | - |
| AS4-6 | +(6) | +(4) | - | - | - | - | - | - | - |
| AS4-7 | +(6) | +(3) | +(5) | - | +(6) | - | - | - | - |
| AS5-2 | +(7) | +(2) | +(7) | - | +(6) | +(5) | - | +(6) | +(4) |
| AS5-4 | +(11) | +(2) | - | - | - | - | - | - | - |
| AS5-5 | +(6) | +(2) | +(4) | - | - | - | - | - | - |
| AS5-7 | +(6) | +(3) | +(5) | - | +(6) | - | - | - | - |
| AS5-8 | +(10) | +(2) | +(6) | - | - | - | - | - | - |
| AS5-9 | +(4) | +(2) | +(4) | - | - | - | - | - | - |
| AS5-10 | +(6) | +(2) | - | - | - | - | - | - | - |
| AS5-12 | +(12) | +(4) | +(2) | - | - | - | - | - | - |
| AS6-1 | +(3) | +(2) | - | - | - | - | - | - | - |
| AS6-2 | +(6) | +(2) | +(8) | - | - | - | - | - | - |
| AS6-4 | +(9) | +(4) | +(7) | - | - | - | - | - | - |
| AS6-5 | - | - | +(4) | - | - | - | - | - | - |
| AS6-6 | +(8) | +(3) | - | - | - | - | - | - | - |
| AS6-7 | +(12) | - | +(4) | - | - | - | - | - | - |
| AS6-8 | +(6) | +(3) | +(4) | - | - | - | - | - | - |
| AL% | 90% | 90% | 76.7% | 0% | 32.6% | 16.2% | 13.9% | 13.9% | 13.9% |
| 注: n:样品数; +:可扩增; -:不可扩增; AL%:可扩增率; 括号内代表等位基因数 | |||||||||
在跨物种扩增成功率方面, 43个黑棘鲷微卫星标记在黄鳍棘鲷、澳洲棘鲷、太平洋棘鲷等3种棘鲷属鱼类中有较高的扩增成功率, 分别为90% (39个位点)、90% (39个位点)和67.7% (33个位点); 在平鲷中的扩增成功率次之, 为32.6% (14个位点); 而在蓝点赤鲷、真赤鲷、二长棘犁齿鲷和黄牙鲷的扩增成功率相对较低, 仅有13.9% (6个位点)至16.2% (7个位点)。值得一提的是, 49个标记在台湾棘鲷中的跨物种扩增均失败。在标记通用性方面, 有28个黑棘鲷微卫星标记(AS2-1, AS2-2, AS2-3, AS2-9, AS2-10, AS2-13, AS3-1, AS3-2, AS3-5, AS3-6, AS3-10, AS3-11, AS3-12, AS3-14, AS3-15, AS34-2, AS4-4, AS4-5, AS4-7, AS5-2, AS5-5, AS5-7, AS5-8, AS5-9, AS5-12, AS6-2, AS6-4, AS6-8)可在黄鳍棘鲷、澳洲棘鲷、太平洋棘鲷中成功扩增, 而只有2个标记(AS3-5和AS3-12)可在平鲷、蓝点赤鲷、真赤鲷、二长棘犁齿鲷及黄牙鲷中成功扩增。
3 讨论 3.1 SLAF-seq技术在黑棘鲷微卫星标记开发中的可行性Jeong等(2003, 2007)通过构建基因组酶切片段文库用探针从文库中筛选出微卫星位点, 两次仅开发8个二碱基重复的黑棘鲷微卫星标记; Yang等(2015)采用FIASCO法筛选出42个黑棘鲷的二碱基重复微卫星序列, 并开发出13个标记; 罗新惠(2010)、徐亘博(2010)、Mao等(2016)也基于FIASCO法分别从黑棘鲷微卫星富集文库中随机筛选出101个、60个、71个微卫星序列, 而后分别开发出30个、22个、8个二、三碱基重复的微卫星标记。本研究采用SLAF-seq技术识别出22489个二至六碱基重复的黑棘鲷微卫星位点, 其中短重复序列(二、三碱基)有20427个, 占总微卫星序列的90.8%;而长重复序列(四至六碱基)虽在比例上不占优势(9.2%), 但在数量上仍较可观, 有2062个。可见, 本研究所获得的微卫星序列不但数量庞大, 而且类型丰富, 可为黑棘鲷的微卫星标记开发提供充足的遗传物质来源和较多的选择余地, 位点筛选效率较之传统方法筛选微卫星序列提高了成百上千倍。在随机挑选的157个位点中最终成功开发出49个多态性的黑棘鲷微卫星标记, 包括5种不同碱基重复的位点, 一次性所开发的标记数量和类型也较之以往的开发方法更加丰富和多样化, 开发效率高且操作简便。
本研究基于SLAF-seq技术获得的24个黑棘鲷四至六碱基微卫星标记完全不同于已开发的81个二、三碱基重复微卫星标记(Jeong et al, 2003, 2007; 罗新惠, 2010; 徐亘博, 2010; Yang et al, 2015; Mao et al, 2016)。在PCR过程中, 这些长重复序列的微卫星新标记被认为比短重复序列标记更加稳定, 出现因影子带而干扰基因分型检测的概率更低(Restrepo et al, 2015)。长重复序列的微卫星位点被认为是更加精确、可靠的一类微卫星分子标记(Wu et al, 2016)。本研究的这些长重复序列的微卫星标记可为黑棘鲷相关遗传资源研究提供新的、高分辨力的分子标记和有力分析工具。此外, SLAF-seq技术还提供了传统开发方法难以获取的微卫星基因组序列特征, 而这些微卫星基因组序列信息可为更有效地筛选微卫星目标序列和高多态性标记以及引物设计提供数据基础(吕振明等, 2017), 为开发不同类型的微卫星标记提供了遗传物质来源保障。
3.2 黑棘鲷微卫星标记的多态性和哈迪-温伯格平衡偏离本研究的49个微卫星位点在黑棘鲷群体中共检测到407个等位基因, 位点的平均Na为8.3, 低于Jeong等(2003, 2007)和Yang等(2015)所报道的黑棘鲷二碱基重复微卫星位点的平均Na(分别为10.0—13.5和10.15)。这可能与本研究含有较多的三至六碱基重复位点(36个, 占总位点的69.7%)有关, 因为二碱基重复位点通常比多碱基重复位点具有更高的遗传变异(Castoe et al, 2012)。然而, 本研究的平均Na明显高于罗新惠(2010)和Mao等(2016)所报道的黑棘鲷二、三碱基重复位点的平均Na(分别为5.43和3.625)。根据Botstein等(1980)提出的以多态信息含量PIC来衡量基因变异程度的高低, 即分为高度(PIC > 0.5)、中度(0.25 < PIC < 0.5)、低度(PIC < 0.25)多态性基因位点。在本研究的49个微卫星标记中, 除9个标记的PIC值小于0.5外, 其余40个标记的PIC值均大于0.5。上述结果表明, 本研究的大部分微卫星位点具有高多态性, 所蕴含的遗传信息含量较为丰富, 在黑棘鲷种群遗传资源研究中具有较高的应用价值, 能够为黑棘鲷的遗传多样性评估与遗传结构分析提供有力的技术支持。
基因杂合度又称基因多样性, 是反映群体在多个基因上的遗传变异及群体遗传多样性的丰富度。杂合度越高, 表明该群体的遗传变异越复杂, 是衡量群体遗传变异的一个最适参数(Nei, 1978)。本研究的群体Ho为0.097—0.938(平均值为0.663), 低于Jeong等(2007)与Yang等(2015)分析的黑棘鲷群体平均Ho(分别为0.844—0.854和0.786), 但高于罗新惠(2010)和Mao等(2016)报道的黑棘鲷群体平均Ho(分别为0.549和0.629)以及DeWoody等(2000)统计32种鱼类微卫星标记的平均Ho(0.63), 表明所分析的黑棘鲷群体仍处于较高的遗传变异水平, 这可能与其是野生群体有关。研究表明, 养殖群体常因奠基者效应(Founder effects)、随机遗传漂变(Random genetic drift)、人工选育(Artificial breeding)和自然选择(Natural selection)等相互作用导致其遗传多样性下降(Mjlnerd et al, 1997; Norris et al, 1999), 这在斜带石斑鱼(Epinephelus coioides)和大黄鱼(Larimichthys crocea)的养殖群体中表现尤为明显(Wang et al, 2011, 2012)。在南海北部, 黑棘鲷种群长期遭受高强度的捕捞压力, 其资源急剧衰减(赵爽等, 2010; 施晓峰等, 2015)。然而, 所检测的黑棘鲷群体遗传多样性仍较为丰富, 意味着该群体具有较强的进化潜力和适应环境变化的能力(Koljonen et al, 2002; 蒙子宁等, 2003), 也提示该群体适用于补充黑棘鲷的繁育群体, 可为其人工选育提供所需的遗传变异(Wang et al, 2012)。
哈迪-温伯格遗传平衡(HWE)是指在理想状态下, 各等位基因的频率和基因型频率在代际遗传中是稳定不变的, 即保持着基因平衡状态。HWE检测是群体内基因频率和基因型频率是否保持平衡的重要检验标准(Schaid et al, 2006)。本研究显示, 经Bonferroni校正后有2个位点(AS4-3和AS4-6)显著偏离HWE, 显示出杂合子缺失现象。诸多因素如无效等位基因(Null alleles)、华伦德效应(Wahlund effect)、近亲杂交(Inbreeding)、人为干扰(Human disturbance)、自然选择(Natural selection)和种群退化(Population degradation)等皆有可能引起微卫星位点偏离HWE (Pemberton et al, 1995; Yu et al, 2002)。本研究的AS4-3和AS4-6位点均显示出较高的近交系数(分别为0.442和0.454)和无效等位基因频率(分别为0.203和0.209), 提示群体中存在的近交和无效等位基因频率是导致这两个位点偏离HWE的主要原因。由于本研究样品来自同一采样时间的同一海域, 可排除华伦德效应(样品中存在亚群体)导致检测群体偏离HWE。此外, 多年来的过度捕捞和近海环境变化无疑对黑棘鲷的资源衰退及种群生物学退化产生了显著的影响(赵爽等, 2010; 许思思, 2011), 同时近年来的大量增殖放流活动亦加大了不同地区、不同种群的黑棘鲷个体基因交流, 对黑棘鲷种群遗传特性的影响也不容忽视(施晓峰等, 2015)。因此, 还难以排除自然选择和种群退化这两个因素可能造成AS4-3和AS4-6位点偏离HWE。
Chapuis等(2007)提出根据无效等位基因频率可将微卫星标记划分为三类:低频(Fua < 0.05)、中频(0.05≤Fua < 0.2)及高频(Fua≥0.2)无效等位基因的标记。本研究的49个微卫星位点出现的无效等位基因频率为–0.167—0.209。其中, 低频率无效等位基因共有20个位点(占总位点的40.8%, 下同; Fua < 0.05), 这20个位点亦显示出高度多态性(PIC > 0.5), 除AS2-10外, 其余19个位点的观测杂合度也相对较高(0.449—0.897), 表明这类具有高多态性、高杂合度、低频无效等位基因的微卫星位点可作为黑棘鲷遗传资源研究的可靠分子标记和优先选用标记。中频无效等位基因共有27个位点(55%, 0.05≤Fua < 0.2), 该类型位点可能会造成群体遗传多样降低并导致群体间遗传距离和遗传分化指数过高(Chapuis et al, 2007), 因此, 这27个位点在后续的黑棘鲷群体遗传分析运用中需持谨慎态度。第三种类型为高频无效等位基因的位点, 包括AS4-3和AS4-6 (Fua≥0.2), 且这两个位点显著偏离HWE, 表明这两个位点不适用于黑棘鲷后续的遗传学研究。
3.3 黑棘鲷微卫星标记在鲷科鱼类中的通用性Primmer等(1996)提出微卫星DNA标记的跨物种扩增通用性反映了检测物种间的遗传分化程度, 二者呈负相关性。即微卫星标记跨物种扩增成功率随物种间的遗传距离增加而降低(Carreras-Carbonell et al, 2008; Wu et al, 2016; Zhang et al, 2016)。中国鲷科鱼类线粒体COI基因序列的系统进化研究表明, 棘鲷属先与平鲷属(Rhabdosargus)聚为一支(银灰体色种类), 而赤鲷属(Pagrus)、犁齿鲷属(Evynnis)、四长棘鲷属(Argyrops)及牙鲷属(Dentex)聚为另一支(红体色种类)(陈咏霞等, 2015)。在本研究中, 黑棘鲷微卫星标记在同属的黄鳍棘鲷、澳洲棘鲷和太平洋棘鲷中的跨物种扩增成功率较高(76.7%—90.0%), 在平鲷中次之(32.6%), 而在蓝点赤鲷、真赤鲷、二长棘犁齿鲷及黄牙鲷中的成功率较低(13.9%—16.2%)。本研究的微卫星标记跨物种扩增结果与线粒体COI基因序列分析结果相一致, 进一步证实了Primmer等(1996)的研究观点, 从核基因角度支持了中国鲷科鱼类可按体色分为两类群的系统进化研究结论(陈咏霞等, 2015)。
蓝点赤鲷是最近报道的一个中国鱼类新纪录, 目前仅见于北部湾北部海域(Chen et al, 2015); 澳洲棘鲷原记录仅分布于澳大利亚东岸, 20世纪九十年后才报道该物种在台湾和琉球群岛近岸有少量分布(沈世杰, 1993); 台湾棘鲷是仅分布在台湾南部海域的一个鲷科鱼类新种(Iwatsuki et al, 2006)。由于这三个物种的分布范围比较局限, 且分类鉴定存在困难, 导致本研究所能获取的这三个物种的个体数均少于微卫星标记跨物种扩增每个物种个体数需≥3的要求。虽然, 本研究的澳洲棘鲷和蓝点赤鲷的个体数较少(均为2个), 但其基因组DNA均可稳定扩增出目的条带, 同时本研究的跨物种扩增并未涉及微卫星标记的遗传多样性参数的估算。因此, 这两个物种有限个体的跨物种扩增结果的统计学争议或是误差并不突出, 其实验结果较为可信。值得提出的是, 开发标记在台湾棘鲷中未能检测出具有通用性的微卫星位点, 这很可能是因为本研究的台湾棘鲷仅有1个个体的基因组DNA质量不佳, 难以扩增出清晰的微卫星条带所致的。此外, 本研究有28个微卫星标记在棘鲷属(除台湾棘鲷)中具有较好的通用性, 可适用于该属鱼类的分子系统进化和种群遗传学研究。但只有3个标记可在蓝点赤鲷、真赤鲷、二长棘犁齿鲷及黄牙鲷中有效扩增, 还难以为这些物种提供数量充足且具有丰富遗传信息的分子标记。因此, 今后有必要单独开发这些物种的特有微卫星标记。
4 结论本研究基于SLAF-seq技术首次在黑棘鲷的基因组DNA中识别出数量庞大的微卫星位点, 并成功开发出49个多态性较高的黑棘鲷微卫星标记, 包括25个短重复序列微卫星位点和24个全新的长重复序列微卫星位点, 其中47个符合HWE的位点可作为今后黑棘鲷种质资源评估和群体遗传分析的有效分子标记和新的分析工具。这种快速、高效的开发方法也为鲷科其他鱼类的微卫星标记开发提供了可靠参考和经验借鉴。同时, 28个在棘鲷属中具有通用性的黑棘鲷微卫星标记亦可为该属鱼类的分子系统进化研究提供新的分子标记来源和分析角度。
万冬梅, 韩梅, 李成安, 等. 2016. 基于高通量测序的沼泽山雀简化基因组微卫星位点的开发. 辽宁大学学报(自然科学版), 43(3): 252-257 DOI:10.3969/j.issn.1000-5846.2016.03.012 |
吕振明, 侯龙, 龚理, 等. 2017. 基于de novo高通量测序的曼氏无针乌贼(Sepiella japonica)ESTs中微卫星位点筛选与特征分析. 海洋与湖沼, 48(4): 877-883 |
刘静, 吴仁协, 康斌, 等. 2016. 北部湾鱼类图鉴. 北京: 科学出版社
|
刘永新, 孙朝徽, 王玉芬, 等. 2015. 牙鲆家系亲权鉴定的微卫星DNA标记分析. 水产学报, 39(11): 1593-1603 |
许思思, 2011.人为影响下渤海渔业资源的衰退机制.青岛: 中国科学院研究生院(海洋研究所)博士学位论文, 149
|
孙波, 鲍毅新, 赵庆洋, 等. 2009. 微卫星位点获取方法的研究进展. 生态学杂志, 28(10): 2130-2137 |
李伟亚, 王伟继, 孔杰, 等. 2012. 中国对虾微卫星四重PCR技术的建立及其在模拟放流效果评估方面的应用. 海洋学报, 34(5): 213-220 |
杨慧荣, 江世贵. 2006. 用RAPD技术探讨5种鲷科鱼类的亲缘关系. 水产学报, 30(4): 469-474 |
沈世杰, 1993.台湾鱼类志.台北, 中国: 国立台湾大学动物学系, 364
|
沈新强, 周永东. 2007. 长江口、杭州湾海域渔业资源增殖放流与效果评估. 渔业现代化, 34(4): 54-57 DOI:10.3969/j.issn.1007-9580.2007.04.017 |
张浩冉, 梁镇邦, 吴仁协, 等. 2019. 利用SLAF-seq技术开发带鱼(Trichiurus japonicus)微卫星标记以及跨物种扩增检测. 基因组学与应用生物学, 38(2): 574-585 |
张浩冉, 梁镇邦, 吴仁协, 等. 2018. 基于SLAF-seq技术的沙带鱼(Lepturacanthus savala)微卫星标记开发以及在近缘种中的通用性. 基因组学与应用生物学, 37(8): 3331-3338 |
陈咏霞, 吴仁协, 梁娜, 等. 2015. 基于线粒体COI基因序列的中国鲷科鱼类系统进化关系. 海洋与湖沼, 46(3): 611-619 |
罗新惠, 2010.黑鲷(Acanthopagrus schlegeli)和半滑舌鳎(Cynoglossus semilaevis)微卫星标记的开发.上海: 上海海洋大学硕士学位论文, 9-27
|
赵爽, 章群, 乐小亮, 等. 2010. 中国近海5个黑鲷地理群体的遗传变异. 海洋科学, 34(2): 75-79 |
施晓峰, 苏永全, 王文成, 等. 2015. 基于mtDNA控制区序列的3个黑棘鲷群体遗传结构特性研究. 热带海洋学报, 34(1): 56-63 DOI:10.3969/j.issn.1009-5470.2015.01.008 |
徐亘博, 2010.半滑舌鳎等九种海水鱼微卫星分子标记的开发和应用.青岛:中国海洋大学硕士学位论文, 21-36
|
郭宝英, 谢从新, 熊冬梅. 2007. 微卫星DNA标记技术及其在鱼类中的应用. 水利渔业, 27(4): 5-9 DOI:10.3969/j.issn.1003-1278.2007.04.002 |
曹艳, 2016.基于线粒体控制区序列的中国沿海3种鲷科鱼类遗传多样性分析.广州: 暨南大学硕士学位论文
|
梁君, 王伟定, 林桂装, 等. 2010. 浙江舟山人工生境水域日本黄姑鱼和黑鲷的增殖放流效果及评估. 中国水产科学, 17(5): 1075-1084 |
鲁双庆, 刘臻, 肖调义. 2003. 微卫星标记在鱼类遗传育种研究中的应用. 水利渔业, 23(6): 9-11 DOI:10.3969/j.issn.1003-1278.2003.06.004 |
蒙子宁, 庄志猛, 金显仕, 等. 2003. 黄海和东海小黄鱼遗传多样性的RAPD分析. 生物多样性, 11(3): 197-203 DOI:10.3321/j.issn:1005-0094.2003.03.003 |
翟云, 吴仁协, 牛素芳, 等. 2018. 基于SLAF-seq技术开发蓝圆鲹微卫星标记及跨物种扩增检测. 应用海洋学学报, 37(3): 426-434 DOI:10.3969/J.ISSN.2095-4972.2018.03.016 |
Botstein D, White R L, Skolnick M et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 32(3): 314-331 |
Carreras-Carbonell J, Macpherson E, Pascual M, 2008. Utility of pairwise mtDNA genetic distances for predicting crossspecies microsatellite amplification and polymorphism success in fishes. Conservation Genetics, 9(1): 181-190 DOI:10.1007/s10592-007-9322-2 |
Castoe T A, Streicher J W, Meik J M et al, 2012. Thousands of microsatellite loci from the venomous coralsnake Micrurus fulvius and variability of select loci across populations and related species. Molecular Ecology Resources, 12(6): 1105-1113 DOI:10.1111/1755-0998.12000 |
Chapuis M P, Estoup A, 2007. Microsatellite null alleles and estimation of population differentiation. Molecular Biology and Evolution, 24(3): 621-631 DOI:10.1093/molbev/msl191 |
Chen Y X, Liu J, Wu R X, 2015. A new record of blue-spotted seabream Pagrus caeruleostictus from Chinese coastal waters documented from morphology and DNA barcoding. Chinese Journal of Oceanology and Limnology, 33(2): 500-505 DOI:10.1007/s00343-015-4059-0 |
DeWoody J A, Avise J C, 2000. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. Journal of Fish Biology, 56(3): 461-473 DOI:10.1111/jfb.2000.56.issue-3 |
Gonzalez E B, Murakami T, Teshima Y et al, 2009. Paternity testing of wild black rockfish Sebastes inermis (brownish type) from the Seto Inland Sea of Japan. Ichthyological Research, 56(1): 87-91 DOI:10.1007/s10228-008-0055-0 |
Goudet J, 2001. FSTAT: a program to estimate and test gene diversities and fi xation indices (Version 2.9.3.2). https: //www2.unil.ch/popgen/softwares/fstat.htm. Accessed on 2018-05-14
|
Grover A, Sharma P C, 2016. Development and use of molecular markers:past and present. Critical Reviews in Biotechnology, 36(2): 290-302 DOI:10.3109/07388551.2014.959891 |
Iwatsuki Y, Carpenter K, 2006. Acanthopagrus taiwanensis, a new sparid fish (Perciformes), with comparisons to Acanthopagrus berda (Forssk l, 1775) and other nominal species of Acanthopagrus. Zootaxa, 1202: 1-19
|
Jeong D S, Gonzalez E B, Morishima K et al, 2007. Parentage assignment of stocked black sea bream Acanthopagrus schlegelii in Hiroshima Bay using microsatellite DNA markers. Fisheries Science, 73(4): 823-830 DOI:10.1111/fis.2007.73.issue-4 |
Jeong D S, Umino T, Kuroda K et al, 2003. Genetic divergence and population structure of black sea bream Acanthopagrus schlegeli inferred from microsatellite analysis. Fisheries Science, 69(5): 896-902 DOI:10.1046/j.1444-2906.2003.00705.x |
Kalinowski S T, Taper M L, Marshall T C, 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 16(5): 1099-1106 DOI:10.1111/j.1365-294X.2007.03089.x |
Koljonen M L, T htinen J, S is M et al, 2002. Maintenance of genetic diversity of Atlantic salmon (Salmo salar) by captive breeding programmes and the geographic distribution of microsatellite variation. Aquaculture, 212(1-4): 69-92 DOI:10.1016/S0044-8486(01)00808-0 |
Liu Y G, Liu L X, Wu Z X et al, 2007. Isolation and characterization of polymorphic microsatellite loci in black sea bream (Acanthopagrus schlegeli) by cross-species amplification with six species of the Sparidae family. Aquatic Living Resources, 20(3): 257-262 DOI:10.1051/alr:2007038 |
Mao X Q, Li Z B, Yuan Y et al, 2016. Isolation and characterization of eight novel microsatellite markers in Acanthopagrus schlegelii. Genetics and Molecular Research, 15(2) |
Mjlnerd I B, Refseth U H, Karlsen E et al, 1997. Genetic differences between two wild and one farmed population of Atlantic salmon (Salmo salar) revealed by three classes of genetic markers. Hereditas, 127(3): 239-248 |
Nei M, 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 89(3): 583-590 |
Niu S F, Zhai Y, Wu R X et al, 2018. Isolation and characterization of 49 polymorphic microsatellite loci for Decapterus maruadsi using SLAF-seq, and cross-amplification to related species. Journal of Oceanology and Limnology, online published at https: //doi. org/10.1007/s00343-019-7299-6
|
Norris A T, Bradley D G, Cunningham E P, 1999. Microsatellite genetic variation between and within farmed and wild Atlantic salmon (Salmo salar) populations. Aquaculture, 180(3-4): 247-264 DOI:10.1016/S0044-8486(99)00212-4 |
Pemberton J M, Slate J, Bancroft D R et al, 1995. Nonamplifying alleles at microsatellite loci:a caution for parentage and population studies. Molecular Ecology, 4(2): 249-252 DOI:10.1111/j.1365-294X.1995.tb00214.x |
Primmer C R, M ller A P, Ellegren H, 1996. A wide-range survey of cross-species microsatellite amplification in birds. Molecular Ecology, 5(3): 365-378 DOI:10.1111/j.1365-294X.1996.tb00327.x |
Restrepo A, Páez V P, Vásquez A et al, 2015. Rapid microsatellite marker development in the endangered neotropical freshwater turtle Podocnemis lewyana (Testudines:Podocnemididae) using 454 sequencing. Biochemical Systematics and Ecology, 59: 220-225 DOI:10.1016/j.bse.2015.01.017 |
Rousset F, 2008. GENEPOP'007:a complete re-implementation of the GENEPOP software for Windows and Linux. Molecular Ecology Resources, 8(1): 103-106 DOI:10.1111/j.1471-8286.2007.01931.x |
Sambrook J, Russell D W, 2001. Molecular Cloning: A Laboratory Manual. 3rd ed. New York: Cold Spring Harbor Laboratory Press
|
Schaid D J, Batzler A J, Jenkins G D et al, 2006. Exact tests of Hardy-Weinberg equilibrium and homogeneity of disequilibrium across strata. American Journal of Human Genetics, 79(6): 1071-1080 DOI:10.1086/510257 |
Schuelke M, 2000. An economic method for the fluorescent labeling of PCR fragments. Nature Biotechnology, 18(2): 233-234 DOI:10.1038/72708 |
Sun X W, Liu D Y, Zhang X F et al, 2013. SLAF-seq:an efficient method of large-scale De Novo SNP discovery and genotyping using high-throughput sequencing. PLoS One, 8(3) |
Van Oosterhout C, Hutchinson W F, Wills D P M et al, 2004. MICRO-CHECKER:software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 4(3): 535-538 DOI:10.1111/men.2004.4.issue-3 |
Wang B S, Wang W J, Xiao G X et al, 2016. Genetic diversity analysis of spawner and recaptured populations of Chinese shrimp (Fenneropenaeus chinensis) during stock enhancement in the Bohai Bay based on an SSR marker. Acta Oceanologica Sinica, 35(8): 51-56 DOI:10.1007/s13131-016-0830-0 |
Wang L, Meng Z N, Liu X C et al, 2011. Genetic diversity and differentiation of the orange-spotted grouper (Epinephelus coioides) between and within cultured stocks and wild populations inferred from microsatellite DNA analysis. International Journal of Molecular Sciences, 12(7): 4378-4394
|
Wang L, Shi X F, Su Y Q et al, 2012. Loss of genetic diversity in the cultured stocks of the large yellow croaker, Larimichthys crocea, revealed by microsatellites. International Journal of Molecular Sciences, 13(5): 5584-5597
|
Wu R X, Zhang H R, Niu S F et al, 2016. Development of polymorphic microsatellites for Sillago sihama based on next-generation sequencing and transferability to Sillago japonica. Genetics and Molecular Research, 15(4): gmr15049046 |
Yang Y X, Cao X X, Feng R Y et al, 2015. Isolation and characterization of thirteen polymorphic microsatellite loci from black porgy (Acanthopagrus schlegeli). Journal of Genetics, 94(S1): 97-99 DOI:10.1007/s12041-014-0426-4 |
Yu H T, Lee Y J, Huang S W et al, 2002. Genetic analysis of the populations of Japanese anchovy (Engraulidae:Engraulis japonicus) using microsatellite DNA. Marine Biotechnology, 4(5): 471-479 DOI:10.1007/s10126-002-0035-8 |
Zhang H R, Niu S F, Wu R X et al, 2016. Development and characterization of 26 polymorphic microsatellite markers in Lateolabrax maculatus and cross-species amplification for the phylogenetically related taxa. Biochemical Systematics and Ecology, 66: 326-330 DOI:10.1016/j.bse.2016.05.008 |
 2019, Vol. 50
2019, Vol. 50