
中国海洋湖沼学会主办。
文章信息
- 张成, 祖露, 李清清, 成永旭, 吴旭干. 2019.
- ZHANG Cheng, ZU Lu, LI Qing-Qing, CHENG Yong-Xu, WU Xu-Gan. 2019.
- 基于脂肪酸生物标记法探讨中国沿海六群体绒螯蟹(Eriocheir sensu stricto)的种群遗传
- THE POPULATION INHERITANCE OF SIX MITTEN CRAB POPULATIONS (ERIOCHEIR SENSU STRICTO) IN COASTAL CHINA BASED ON PHOSPHOLIPID FATTY ACIDS BIOMARKERS
- 海洋与湖沼, 50(2): 388-397
- Oceanologia et Limnologia Sinica, 50(2): 388-397.
- http://dx.doi.org/10.11693/hyhz20180900212
-
文章历史
- 收稿日期:2018-09-04
- 收修改稿日期:2018-10-25
2. 上海海洋大学 水产动物遗传育种上海市协同创新中心 上海 201306;
3. 上海海洋大学 水产科学国家级实验教学示范中心 上海 201306
2. Shanghai Collaborative Innovation Center for Aquatic Animal Genetics and Breeding, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Centre for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China
绒螯蟹属(Eriocheir)隶属于节肢动物门(Arthropoda)、甲壳纲(Crustacea)、十足目(Decapoda)、方蟹科(Grapsidae), 主要包括中华绒螯蟹和日本绒螯蟹等, 其广泛分布于东亚地区, 特别是中国、韩国和日本的各大水系(Chu et al, 2003; Xu et al, 2012)。中国大陆地区主要分布于辽河、黄河、长江、瓯江、闽江和南流江等水系(Tang et al, 2003; Xu et al, 2012)。
中华绒螯蟹是中国重要的淡水养殖物种, 具有较高的营养价值, 2016年全国养殖产量高达81.21万吨(农业部渔业渔政管理局, 2017)。目前, 我国绒螯蟹的人工养殖主要集中于辽河、黄河和长江流域, 瓯江、闽江和南流江流域则很少养殖(王武等, 2013)。尽管我国绒螯蟹养殖产业巨大, 但也面临着近亲繁殖、种质混杂和养殖性能退化等一系列问题(Sui et al, 2011b; 刘青等, 2015)。系统研究我国沿海地区主要绒螯蟹种群的遗传多样性、养殖性能和营养品质, 对于绒螯蟹种质资源评价、开发利用和遗传育种均具有重要的理论和现实意义。
先前有关我国沿海地区绒螯蟹(Eriocheir sensu stricto)种群遗传的研究, 主要集中在形态学、同工酶、核基因组和线粒体DNA分子标记等方面(李晨虹等, 1999; 赵金良等, 1999; Chu et al, 2003; Tang et al, 2003; Xu et al, 2012; 刘青等, 2015; Zhang et al, 2017), 且不同学者对不同地理群体绒螯蟹(特别是中华绒螯蟹和日本绒螯蟹)的亲缘关系和命名存在较大分歧(Li et al, 1993; Guo et al, 1997; Chu et al, 2003; Tang et al, 2003; Wang et al, 2008; Sui et al, 2009; Xu et al, 2012)。脂肪酸是生物体内的一类重要营养物质和必需组成成分, 对甲壳动物的生长、发育、生殖和其他生理机能具有重要的作用(Sui et al, 2011a; Wu et al, 2011)。由于其具有结构多样性, 能够在一定程度上指示生物来源。水产动物肌肉中的脂肪酸组成通常具有一定的物种或群体保守性, 可以作为种质鉴定的一个重要指标(Jarvis et al, 1994; Dalsgaard et al, 2003), 目前已在微生物和植物种质鉴定中得到了广泛应用(Arslan et al, 2018; Santos et al, 2018), 但是在水产动物方面的研究及应用较少(Grahl-Nielsen et al, 2010; 董志国等, 2013)。迄今为止, 尚未见有关以脂肪酸生物标记评估绒螯蟹不同野生地理群体的遗传多样性和亲缘关系的研究报道, 这不利于绒螯蟹种质资源评价和开发利用。
鉴于此, 本研究采用脂肪酸生物标记法测定和比较了中国大陆6个主要水系野生绒螯蟹的肌肉脂肪酸组成, 筛选出基于脂肪酸生物标记判别6个野生种群绒螯蟹的方法, 并进一步评价了其遗传多样性和亲缘关系, 研究结果对不同水系绒螯蟹野生种质资源评价、开发利用和遗传选育等均具有重要的理论意义和应用前景。
1 材料与方法 1.1 样品采集本研究所使用的野生绒螯蟹样品于2017年10—12月间分别采自辽河(LH)、黄河(HH)、长江(YZ)、瓯江(OJ)、闽江(MJ)和南流江(NLJ)水系, 各个水系采集样品信息见表 1。样品采集方式为地笼抓捕或者人工捕捞, 活体样品迅速运输到上海海洋大学甲壳动物营养繁殖实验室。各水系随机抽取10只肢体健全的绒螯蟹活体解剖, 躯体经冷冻处理后, 精刮取出肌肉组织, 避免其他组织污染, 并保存于–40℃的冰箱中待脂肪酸分析。
| 采样地点 | 经度 | 纬度 | 水系 | 标号 | 样品数量 |
| 辽宁省盘锦市 | 122.70°E | 40.70°N | 辽河 | LH | 10 |
| 山东省东营市 | 118.52°E | 37.61°N | 黄河 | HH | 10 |
| 江苏省镇江市 | 119.27°E | 32.11°N | 长江 | YZ | 10 |
| 浙江省温州市 | 120.34°E | 28.06°N | 瓯江 | OJ | 10 |
| 福建省福州市 | 119.08°E | 26.08°N | 闽江 | MJ | 10 |
| 广西北海市 | 109.04°E | 21.39°N | 南流江 | NLJ | 10 |
根据Morrison和Smith的方法(Morrison et al, 1964), 使用14%三氟化硼-甲醇溶液(V:V)对脂肪酸样品甲酯化处理。使用Agilent 7890B-5977A气相色谱-质谱联用仪(GC-MS)分析脂肪酸数据, 其中色谱柱为Omegawax 320毛细管柱(30m×0.25mm×0.25μm; Supelco, Billefonte, PA, USA)。进样口温度保持在240℃, 辅助加热器保持在245℃。柱的最初温度为40℃, 以10℃/min的速率升温到170℃, 再以2℃/min的速率升至220℃, 并保持1min, 最后以2℃/min速率升至230℃, 直至脂肪酸指标全部出峰。通过比较保留时间与已知标准(Sigma-Aldrich Co., St. Louis, MO, USA), 再对图谱中的脂肪酸进行鉴定。脂肪酸组成表示为每种脂肪酸相对于总脂肪酸的百分比。
1.3 逐步判别分析基于含量大于0.3%的脂肪酸性状对6水系绒螯蟹群体进行逐步判别分析, 并建立判别方程式。通过判别方程式对6群体进行判别, 然后统计判别准确率, 计算公式如下:

其中, Ai为第i个水系中判别正确的绒螯蟹数量, Bi为第i个水系中实际判别的绒螯蟹数量, k表示水系的数量。
1.4 主成分分析基于所有含量大于0.3%的脂肪酸性状对6水系绒螯蟹群体进行主成分分析, 其中主成分分析以累计贡献率大于60%且特征值大于1为原则, 并根据各组贡献率最大的第一主成分和第二主成分绘制主成分散点图。
1.5 聚类分析根据含量大于0.3%的脂肪酸性状, 采用欧氏距离的未加权组内平均法, 对6水系绒螯蟹群体脂肪酸的平均值进行系统聚类分析, 并绘出聚类图。
1.6 数据处理本研究采用SPSS22.0软件对6水系绒螯蟹群体的脂肪酸性状进行分析, 数据结果均以平均值±标准误(X ± SE)表示。使用Levene’s法进行方差齐性检验, 若实验数据不满足齐性方差检验, 则对百分比数据进行平方根或者反正弦处理。使用ANOVA对实验数据进行方差分析。脂肪酸群体间差异进行单因子方差分析(以每种脂肪酸为变量), 应用邓肯氏法进行多重比较。对6水系绒螯蟹群体的脂肪酸进行聚类分析。
2 结果与分析 2.1 六水系绒螯蟹群体脂肪酸的组成差异、遗传变异与遗传多样性在6水系绒螯蟹群体的肌肉中均检测到26种脂肪酸, 含量排在前7位的脂肪酸有EPA (C20:5n3)、油酸(C18:1n9)、DHA(C22:6n3)、棕榈酸(C16:0)、花生四烯酸(C20:4n6)、亚油酸(C18:2n6)和硬脂酸(C18:0), 这7种脂肪酸的含量总和均在74%以上。其中MJ群体的EPA含量达16.3%, 高于其他5个群体, MJ与HH、YZ、OJ三群体间均无显著性差异, 但这4个群体与LH和NLJ群体均有显著性差异; C18:1n9和DHA的含量在YZ群体中均为最高, 分别为15.34%和13.25%, C18:1n9的含量在6群体之间均无显著性差异, 对于DHA, LH群体与其他5个群体之间均有显著性差异, 但YZ和MJ群体DHA含量与OJ、NLJ群体之间均无显著性差异。对于饱和脂肪酸(∑SFA), 含量最高的是HH群体, 为21.69%, HH、MJ群体∑SFA含量与其他5个群体均有显著性差异, 而LH、YZ、OJ和NLJ群体之间均无显著性差异。对于单不饱和脂肪酸(∑MUFA), 含量最高的是YZ群体, LH、NLJ群体∑MUFA含量与其他5个群体均有显著性差异, HH与MJ群体之间有显著性差异, 其他群体之间均无显著性差异; n-3/n-6的比值显示, LH群体与其他5个群体间均存在显著差异(表 2)。
| 脂肪酸 | LH n = 10 |
HH n = 10 |
YZ n = 10 |
OJ n = 10 |
MJ n = 10 |
NLJ n = 10 |
| C14:0 | 0.36 ± 0.03bc | 0.38 ± 0.02c | 0.38 ± 0.02bc | 0.31 ± 0.02ab | 0.27 ± 0.02a | 0.40 ± 0.02c |
| C15:0 | 0.30 ± 0.02bc | 0.39 ± 0.04d | 0.36 ± 0.02cd | 0.23 ± 0.01a | 0.25 ± 0.02ab | 0.23 ± 0.02a |
| C16:0 | 10.21 ± 0.14bc | 10.49 ± 0.34c | 10.58 ± 0.24c | 10.47 ± 0.29c | 9.55 ± 0.33ab | 8.98 ± 0.20a |
| C17:0 | 0.80 ± 0.03ab | 1.05 ± 0.07c | 0.91 ± 0.03bc | 0.76 ± 0.05a | 1.02 ± 0.07c | 1.67 ± 0.07d |
| C18:0 | 7.20 ± 0.07a | 8.45 ± 0.14c | 6.87 ± 0.11a | 7.63 ± 0.14b | 7.06 ± 0.15a | 7.88 ± 0.09b |
| C20:0 | 0.47 ± 0.01c | 0.50 ± 0.01cd | 0.33 ± 0.02ab | 0.37 ± 0.01b | 0.31 ± 0.02a | 0.53 ± 0.02d |
| C22:0 | 0.45 ± 0.03d | 0.43 ± 0.01cd | 0.26 ± 0.02a | 0.37 ± 0.02bc | 0.32 ± 0.03ab | 0.47 ± 0.03d |
| ∑SFA | 19.79 ± 0.15b | 21.69 ± 0.26c | 19.69 ± 0.24b | 20.15 ± 0.33b | 18.78 ± 0.44a | 20.18 ± 0.23b |
| C16:1n7 | 2.80 ± 0.25ab | 3.59 ± 0.26c | 3.01 ± 0.20bc | 2.25 ± 0.19a | 2.08 ± 0.14a | 3.56 ± 0.32c |
| C17:1n7 | 0.66 ± 0.05b | 0.83 ± 0.07b | 0.76 ± 0.05b | 0.48 ± 0.04a | 0.72 ± 0.06b | 1.57 ± 0.10c |
| C18:1n7 | 3.25 ± 0.12ab | 3.60 ± 0.11bc | 3.80 ± 0.130c | 3.38 ± 0.10bc | 3.56 ± 0.17bc | 2.92 ± 0.16a |
| C18:1n9 | 15.08 ± 0.44a | 15.01 ± 0.6a | 15.34 ± 0.46a | 14.61 ± 0.57a | 13.86 ± 0.91a | 14.68 ± 0.45a |
| C20:1n9 | 0.61 ± 0.02ab | 0.59 ± 0.05a | 0.97 ± 0.09c | 0.72 ± 0.04ab | 0.80 ± 0.05bc | 0.58 ± 0.05a |
| C22:1n9 | 0.32 ± 0.02d | 0.18 ± 0.01bc | 0.17 ± 0.02bc | 0.11 ± 0.01a | 0.19 ± 0.02c | 0.14 ± 0.01ab |
| ∑MUFA | 22.83 ± 0.63d | 23.87 ± 0.84c | 24.15 ± 0.50bc | 21.62 ± 0.73bc | 21.29 ± 0.93b | 23.54 ± 0.63a |
| C18:2n6 | 9.69 ± 0.47d | 8.23 ± 0.61c | 7.36 ± 0.40bc | 6.94 ± 0.33bc | 6.31 ± 0.40b | 3.34 ± 0.25a |
| C18:3n3 | 1.87 ± 0.13cd | 2.23 ± 0.18d | 1.64 ± 0.13c | 1.13 ± 0.10b | 0.60 ± 0.04a | 1.19 ± 0.06b |
| C18:3n6 | 0.20 ± 0.01c | 0.32 ± 0.02d | 0.13 ± 0.01b | 0.17 ± 0.01c | 0.12 ± 0.01b | 0.06 ± 0.00a |
| C20:2n6 | 1.99 ± 0.12c | 1.25 ± 0.10a | 1.32 ± 0.07a | 1.70 ± 0.13bc | 1.60 ± 0.15ab | 1.55 ± 0.11ab |
| C20:3n3 | 0.42 ± 0.04d | 0.36 ± 0.03cd | 0.38 ± 0.03d | 0.29 ± 0.02bc | 0.17 ± 0.01a | 0.27 ± 0.02b |
| C20:4n6 | 9.44 ± 0.57b | 6.63 ± 0.20a | 6.92 ± 0.59a | 8.63 ± 0.39b | 9.91 ± 0.43b | 9.34 ± 0.40b |
| C20:5n3 | 14.05 ± 0.39ab | 15.92 ± 0.45c | 15.53 ± 0.64bc | 15.8 ± 0.59c | 16.3 ± 0.64c | 13.13 ± 0.32a |
| C22:5n3 | 2.09 ± 0.12d | 1.23 ± 0.10b | 0.81 ± 0.05a | 1.73 ± 0.15cd | 2.10 ± 0.19d | 1.43 ± 0.09bc |
| C22:6n3 | 8.97 ± 0.78a | 10.26 ± 0.39b | 13.25 ± 0.77d | 11.84 ± 0.40bcd | 11.04 ± 0.45bc | 12.23 ± 0.36cd |
| ∑PUFA | 48.73 ± 0.78b | 46.42 ± 0.68b | 47.33 ± 0.68b | 48.23 ± 1.37b | 48.16 ± 1.08b | 42.55 ± 0.60a |
| ∑HUFA | 36.96 ± 0.59a | 35.65 ± 0.36a | 38.21 ± 0.85ab | 39.99 ± 1.24bc | 41.13 ± 0.93c | 37.95 ± 0.60ab |
| ∑n-3PUFA | 27.41 ± 0.93a | 30.00 ± 0.25bc | 31.6 ± 1.22c | 30.79 ± 0.81bc | 30.22 ± 0.91bc | 28.25 ± 0.61ab |
| ∑n-6PUFA | 21.31 ± 0.95c | 16.42 ± 0.77ab | 15.73 ± 0.86ab | 17.45 ± 0.64b | 17.94 ± 0.73b | 14.29 ± 0.58a |
| n-3/n-6 | 1.32 ± 0.10a | 1.83 ± 0.09b | 2.11 ± 0.20b | 1.78 ± 0.05b | 1.72 ± 0.10b | 2.03 ±0.13b |
| 注:含量小于0.3%的脂肪酸未在表中列出(下同)。同行数据上标中不含有相同字母表示差异显著(P < 0.05) | ||||||
对6水系绒螯蟹群体的脂肪酸参数进行分析, 由表 2得出: 6水系绒螯蟹群体脂肪酸种类均为26种, 其中以不饱和脂肪酸为主, 分别为C18:1n9、C18:2n6、C20:4n6、C22:5n3和C22:6n3; ∑SFA主要含有C16:0和C18:0两种。虽然脂肪酸种类在6水系绒螯蟹群体之间均相同, 但是脂肪酸含量在不同群体间亦不同, 其中LH、HH、YZ、OJ、MJ和NLJ群体的变异系数分别为: 2.99%—27.42%、5.05%—30.00%、5.20%— 29.84%、5.70%—29.76%、6.90%—29.72%和3.69%— 28.76%。就6群体的饱和脂肪酸而言, C16:0和C18:0两种脂肪酸的变异系数均在10%左右, 不饱和脂肪酸的变异系数均高于饱和脂肪酸。
由表 3可以得出6个群体肌肉中的脂肪酸性状Shannon-Weaver多样性指数。26个脂肪酸参数之间的遗传多样性指数差别较小, 其中平均遗传多样性指数为3.2981, 变化幅度是3.2732—3.3204, 遗传多样性指数最高的是∑SFA, 最低的是C22:1n9。就6水系绒螯蟹群体而言, 遗传多样性指数差异依然不大, 其中遗传多样性指数最高的是LH群体, 最低的是MJ群体。
| 脂肪酸 | LH | HH | YZ | OJ | MJ | NLJ | 平均值 |
| C14:0 | 3.2901 | 3.3027 | 3.2950 | 3.3043 | 3.2890 | 3.3031 | 3.2974 |
| C15:0 | 3.2969 | 3.2573 | 3.2981 | 3.2940 | 3.2827 | 3.2769 | 3.2843 |
| C16:0 | 3.3206 | 3.3144 | 3.3183 | 3.3162 | 3.3132 | 3.3185 | 3.3169 |
| C17:0 | 3.3116 | 3.2846 | 3.3153 | 3.2940 | 3.2832 | 3.3075 | 3.2994 |
| C18:0 | 3.3213 | 3.3200 | 3.3200 | 3.3196 | 3.3185 | 3.3209 | 3.3201 |
| C20:0 | 3.3164 | 3.3201 | 3.2952 | 3.3139 | 3.2782 | 3.3111 | 3.3058 |
| C22:0 | 3.2990 | 3.3142 | 3.2783 | 3.2924 | 3.2639 | 3.2954 | 3.2905 |
| ∑SFA | 3.3215 | 3.3209 | 3.3209 | 3.3200 | 3.3180 | 3.3210 | 3.3204 |
| C16:1n7 | 3.2668 | 3.2864 | 3.2883 | 3.2704 | 3.2869 | 3.2586 | 3.2762 |
| C17:1n7 | 3.2754 | 3.2673 | 3.2919 | 3.2565 | 3.2752 | 3.2914 | 3.2763 |
| C18:1n7 | 3.3114 | 3.3145 | 3.3133 | 3.3161 | 3.3067 | 3.3022 | 3.3107 |
| C18:1n9 | 3.3159 | 3.3109 | 3.3153 | 3.3108 | 3.2923 | 3.3153 | 3.3101 |
| C20:1n9 | 3.3147 | 3.2623 | 3.2566 | 3.3018 | 3.2877 | 3.2667 | 3.2816 |
| C22:1n9 | 3.2872 | 3.2761 | 3.2622 | 3.2738 | 3.2522 | 3.2876 | 3.2732 |
| ∑MUFA | 3.3164 | 3.3133 | 3.3188 | 3.3136 | 3.3085 | 3.3169 | 3.3146 |
| C18:2n6 | 3.3055 | 3.2822 | 3.3009 | 3.3049 | 3.2936 | 3.2838 | 3.2952 |
| C18:3n3 | 3.2883 | 3.2764 | 3.2772 | 3.2622 | 3.2817 | 3.3014 | 3.2812 |
| C18:3n6 | 3.2970 | 3.2776 | 3.2619 | 3.2915 | 3.2672 | 3.2943 | 3.2816 |
| C20:2n6 | 3.2932 | 3.2757 | 3.3012 | 3.2758 | 3.2541 | 3.2791 | 3.2799 |
| C20:3n3 | 3.2675 | 3.2541 | 3.2714 | 3.2821 | 3.2984 | 3.2813 | 3.2758 |
| C20:4n6 | 3.2932 | 3.3154 | 3.2684 | 3.3071 | 3.3084 | 3.3078 | 3.3001 |
| C20:5n3 | 3.3161 | 3.3160 | 3.3091 | 3.3116 | 3.3112 | 3.3176 | 3.3136 |
| C22:5n3 | 3.2989 | 3.2746 | 3.2877 | 3.2668 | 3.2658 | 3.2940 | 3.2813 |
| C22:6n3 | 3.2701 | 3.3114 | 3.2976 | 3.3135 | 3.3098 | 3.3156 | 3.3030 |
| ∑PUFA | 3.3201 | 3.3204 | 3.3204 | 3.3161 | 3.3183 | 3.3205 | 3.3193 |
| ∑HUFA | 3.3201 | 3.3212 | 3.3183 | 3.3149 | 3.3183 | 3.3201 | 3.3188 |
| ∑n-3PUFA | 3.3135 | 3.3214 | 3.3109 | 3.3169 | 3.3155 | 3.3186 | 3.3161 |
| ∑n-6PUFA | 3.3077 | 3.3063 | 3.2997 | 3.3118 | 3.3097 | 3.3095 | 3.3075 |
| n-3/n-6 | 3.2847 | 3.3047 | 3.2605 | 3.3169 | 3.2980 | 3.2937 | 3.2931 |
| Mean | 3.3020 | 3.2971 | 3.2969 | 3.2990 | 3.2932 | 3.3013 | 3.2981 |
为比较6水系绒螯蟹群体脂肪酸的差异, 找到符合种群鉴定的脂肪酸生物标记, 本实验使用逐步判别分析法对这6水系绒螯蟹群体的26种脂肪酸性状进行了分析, 逐步判别分析贡献比较大的脂肪酸性状共8种, 分别是C17:0、C18:0、C20:0、C22:1n9、C18:3n3、C18:3n6、C20:2n6和C22:5n3。对这8种贡献率较大的脂肪酸变量建立判别方程式, 判别公式如下: (其中这8种脂肪酸在公式中分别依次被X1—X8表示)

判别方程式建立后, 由这8个比例性状对6群体绒螯蟹进行判别分析, 并对测定数据进行预测分类, 结果显示:判别准确率P1的范围在90%—100%, P2在90.91%—100%, 这6个群体绒螯蟹的判别率均接近于100%, 并且综合判别准确率为98.33% (表 4)。故以上6个判别方程式可靠。
| 群体 | 样本数量 | 预测分类 | 判别准确率(%) | 综合判别准确率(%) | |||||||
| LH | HH | YZ | OJ | MJ | NLJ | P1 | P2 | ||||
| LH | 10 | 10 | 0 | 0 | 0 | 0 | 0 | 100 | 100 | 98.33 | |
| HH | 10 | 0 | 10 | 0 | 0 | 0 | 0 | 100 | 100 | ||
| YZ | 10 | 0 | 0 | 10 | 0 | 0 | 0 | 100 | 100 | ||
| OJ | 10 | 0 | 0 | 0 | 10 | 0 | 0 | 100 | 90.91 | ||
| MJ | 10 | 0 | 0 | 0 | 1 | 9 | 0 | 90 | 100 | ||
| NLJ | 10 | 0 | 0 | 0 | 0 | 0 | 10 | 100 | 100 | ||
基于26种脂肪酸参数创建6水系绒螯蟹群体脂肪酸的第一、二主成分散布图(PCA图)(见图 1)。图 1显示, 6水系绒螯蟹群体之间存在一定的差异, 且各群体主成分散布均形成了独立的区域, 但是群体之间也存在一定的重叠区域。其中, OJ与MJ群体重叠区域相对最大, 两者与NLJ群体重叠区域最小, HH与YZ的重叠面积大于其与LW重叠的面积。
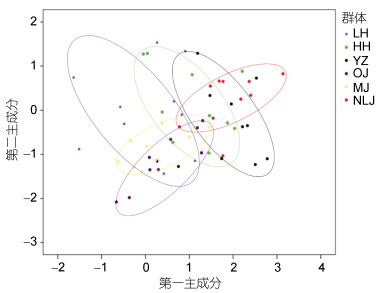 |
| 图 1 6水系绒螯蟹群体的第一和第二主成分散布图 Fig. 1 Scatter diagram for the first and the second principal components of Eriocheir s. s. populations from six river basins |
基于6水系绒螯蟹群体26种脂肪酸性状, 采用类平均法计算6群体之间的欧氏距离, 并构建聚类图。由图 2和表 5可知, OJ和MJ群体、HH和YZ群体分别两两相聚, 且OJ与MJ群体的欧氏距离最小, 为3.0352; HH与YZ群体的欧氏距离相对较大, 为5.3137;然后依次与LH、NLJ群体相聚; LH与NLJ群体的欧氏距离为12.1338。
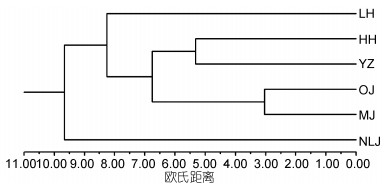 |
| 图 2 6水系绒螯蟹群体聚类分析图 Fig. 2 Diagram of cluster analysis of Eriocheir s. s. populations from six river basins 注: LH:辽河水系, HH:黄河水系, YZ:长江水系, OJ:瓯江水系, MJ:闽江水系, NLJ:南流江水系 |
| 种群 | LH | HH | YZ | OJ | MJ | NLJ |
| LH | 0.0000 | |||||
| HH | 7.8374 | 0.0000 | ||||
| YZ | 9.4440 | 5.3137 | 0.0000 | |||
| OJ | 7.6502 | 6.6356 | 4.7666 | 0.0000 | ||
| MJ | 8.1208 | 8.7152 | 6.9061 | 3.0352 | 0.0000 | |
| NLJ | 12.1338 | 8.8676 | 8.5425 | 9.1500 | 9.6286 | 0.0000 |
脂肪酸是脂类物质的重要组成部分, 对生物体的生长、繁殖、发育以及其它的生理机能有着重要的作用(Sui et al, 2011a; Wu et al, 2011)。组织中脂肪酸组成成分受内在因素(如系统发育、年龄、性别、生殖周期和遗传)和外部因素(如食物、温度、盐度和水深)的影响(Grahl-Nielsen et al, 2010)。许多学者认为:食物组成是决定组织中脂肪酸组成的主要因素之一, 但是在相同生长条件下, 不同物种或者地理群体的脂肪酸组成可能也具有一定的特异性, 可以作为种质鉴定的一个指标(Jarvis et al, 1994; Dalsgaard et al, 2003), 且已被广泛应用于分类学和生物标记(Shin et al, 2008; Imbs et al, 2009)。Neira等(2004)在相同条件下对不同地理群体的银鲑(Oncorhynchus kisutch)进行养殖, 发现银鲑肌肉中脂肪酸的组成成分依然不同。进一步研究表明:水生动物的肌肉脂肪酸组成可能与遗传因素相关(赵恒亮等, 2016; Grahl-Nielsen et al, 2010)。采用脂肪酸生物标记研究和鉴别不同地理种群的水生动物已经在三疣梭子蟹(Portunus trituberculatus)、八放珊瑚(Veretillum cynomorium)和鸟蛤(Cerastoderma edule)等物种上得到了应用(董志国等, 2013; Ricardo et al, 2015; Figueiredo et al, 2017)。本研究在辽河、黄河、长江、瓯江、闽江和南流江水系绒螯蟹群体的肌肉中均检测出26种脂肪酸, 其中C20:5n3、C18:1n9、C22:6n3、C16:0、C20:4n6、C18:2n6和C18:0为7种主要的脂肪酸, 这与先前对长江、黄河、辽河水系中华绒螯蟹幼蟹的研究结果完全一致(卢义等, 2016)。6群体绒螯蟹肌肉中脂肪酸的组成存在较大差异, 闽江群体肌肉中的EPA含量最高, 黄河和长江群体的ARA(20:4n6)含量较低; 对于DHA含量, 长江群体的含量最高, 辽河水系最低。造成脂肪酸含量差异的原因可能是: ①不同水系的环境因子, 比如盐度和水温等因素的不同均可能对脂肪酸含量造成影响(Chapelle, 1978; Wang et al, 2007); ②各水系绒螯蟹的饵料组成较为复杂, 不同的水域中包括水草、有机碎屑和贝类等底栖动物也可能不同, 这暗示着摄食不同的饵料可能对脂肪酸含量造成一定差异(Sui et al, 2011a); ③脂肪酸含量的差异可能由遗传因素起主导作用, 水生动物肌肉中的脂肪酸生物标记可以用于一些物种的种质鉴定和种群判别, 就是有力的佐证(Dalsgaard et al, 2003; 董志国等, 2013)。本研究筛选出8种脂肪酸建立判别方程, 可以用于判别不同地理种群绒螯蟹的来源, 综合判别准确率达98.33%。这对于野生绒螯蟹的种质资源和溯源具有一定的科学和经济价值。
遗传多样性一般是指种内个体之间或者一个群体内不同个体所携带遗传信息的总和(Atta-Krah et al, 2004)。先前研究, 绝大多数采用核基因组或者线粒体分子标记进行遗传多样性的分析。比如: Xu等(2012)基于核基因组图谱对东亚地区绒螯蟹群体差异、杂合程度和物种进化进行了分析, 结果表明: Okinawa地区的绒螯蟹遗传多样性指数低, 东亚地区其他群体的遗传多样性指数均较高。闫龙等(2015)基于线粒体D-loop区评价了辽河、黄河和长江水系中华绒螯蟹群体遗传多样性, 结果显示三水系绒螯蟹的遗传多样性指数均较高。不同水系绒螯蟹群体的脂肪酸性状类型越丰富, 遗传多样性水平越高。本研究结果中, 虽然不同群体间脂肪酸性状的变异水平存在较大差异, 但是6水系绒螯蟹群体的脂肪酸多样性指数均较高, 这与有关学者用微卫星技术对中华绒螯蟹所进行的研究结果一致(Ma et al, 2007; Chang et al, 2008)。出现此结果的原因可能是: ①绒螯蟹广泛分布于中国沿海各大水系中, 并且具有很强繁殖力和环境适应能力, 较大的有效群体造成了绒螯蟹遗传多样性水平较高(闫龙等, 2015); ②增殖放流、跨区域引种、人工养殖与逃逸等, 在一定程度上也提高了各水系绒螯蟹的遗传多样性(刘青等, 2015; 彭欣悦, 2016)。因此, 为了加强各水系绒螯蟹特有种质资源资源的保护, 防止不同流域的种质混杂, 亟需制定一份科学且有效的策略, 这样才能进一步开展生物多样性保护、优质野生种质资源的开发和良种选育等研究。
3.2 六大水系绒螯蟹群体亲缘关系聚类分析可以直观的表现出研究对象的差异, 并且可以根据不同生物群体的相似程度进行分类(Anandakrishnan et al, 2008)。主成分分析则是将原来变量重新组合成一组新的互相无关的几个综合变量, 同时根据实际需要从中取出几个较少的综合变量尽可能多反映原来变量信息的统计方法(Abdi et al, 2010)。本研究对6水系绒螯蟹群体26种脂肪酸性状进行聚类和主成分分析。结果显示:瓯江和闽江群体、黄河和长江群体分别先两两相聚, 再聚为一支, 然后依次与辽河、南流江群体相聚, 且瓯江和闽江群体之间的欧氏距离相对最小; PCA图中可看出, 瓯江和闽江群体重叠区域相对最大, 两者与南流江群体重叠区域相对最小, 黄河与长江群体的重叠面积大于与辽河群体的面积。本研究结果说明, 瓯江与闽江群体之间的脂肪酸组成和含量最为相似, 产生此结果的原因可能是:由于瓯江和闽江绒螯蟹群体的地理位置相对较近, 同时绒螯蟹具有很强的侵略性和运动能力(Herborg et al, 2003; Chen et al, 2014), 使两个水系绒螯蟹之间发生了遗传渗透, 或者人为因素造成两地绒螯蟹发生基因交流(成起萱, 2011), 致使瓯江和闽江绒螯蟹群体的亲缘关系相对最近。就辽河、黄河和长江群体的聚类结果可看出, 黄河和长江群体的脂肪酸组成和含量十分相似, 而两者与辽河群体的差异稍大。同样, 卢义等(2016)对长江、黄河、辽河水系中华绒螯蟹肌肉脂肪酸营养研究, 结果表明:无论是雌性还是雄性群体, 都是长江与黄河群体先聚为一支, 之后与辽河水系群体聚为一支; Zhang等(2017)基于线粒体Cytb基因标记对长江、黄河和辽河水系养殖与野生中华绒螯蟹群体的遗传多样性和群体结构进行了分析, 其结果表明:无论是养殖还是野生群体, 均是长江和黄河水系绒螯蟹群体先聚为一支, 然后才与辽河群体相聚。因此, 无论是从生化方面还是分子方面, 都是黄河与长江群体的亲缘关系相对最近。原因可能是: ①就辽河群体的地理位置和生存环境而言, 黄河和长江群体的地理位置相对较近, 生存环境也更为相似。相较于黄河和长江水系群体, 辽河水系群体是一个较为封闭的地理种群单位, 与其他水系群体遗传信息交流时可能存在较大的阻力; ②在黄河和长江流域具有很大的养殖绒螯蟹区域, 且长江水系中华绒螯蟹群体种质最为优良(李晨虹等, 1999), 一些养殖场盲目跨区域引种, 加大了黄河和长江水系绒螯蟹群体的基因交流。南流江群体的脂肪酸组成和含量与黄河、长江、瓯江和闽江群体的差异相对最大。从地理分布的角度来看, 南流江水系地理位置的纬度相对最低, 与其他5个水系相对较远, 且当地几乎没有人工养殖的绒螯蟹, 故南流江群体和其他群体之间没有人为因素的影响, 也没有基因交流的发生。因此, 南流江群体与其他群体之间的亲缘关系相对最远。
4 结论目前, 辽河、黄河和长江流域野生绒螯蟹群体的种质资源已有一定的开发和应用, 且经济效益显著, 而瓯江、闽江和南流江流域绒螯蟹群体的遗传信息尚未得到一个系统的评估和开发, 暗示绒螯蟹产业的发展依然存在巨大潜力。综上, 6大水系野生绒螯蟹群体之间遗传多样性指数均较高, 各水系间的亲缘关系明确, 且逐步判别分析贡献比较大的脂肪酸8种可以对绒螯蟹进行有效的种质鉴定, 为绒螯蟹的分类提供一定的理论依据。因此, 可根据营养品质对瓯江、闽江和南流江流域绒螯蟹群体进行优良经济性状的筛选, 以提高绒螯蟹群体的养殖性能。
王武, 王成辉, 马旭洲. 2013. 河蟹生态养殖. 北京: 中国农业出版社, 59-84
|
卢义, 吴旭干, 何杰, 等. 2016. 长江、黄河、辽河水系中华绒螯蟹野生扣蟹的形态学及生化组成. 中国水产科学, 23(2): 382-395 |
成起萱, 2011.绒螯蟹的分子遗传变异与进化.上海: 上海海洋大学硕士学位论文, 31-38
|
刘青, 刘皓, 吴旭干, 等. 2015. 长江、黄河和辽河水系中华绒螯蟹野生和养殖群体遗传变异的微卫星分析. 海洋与湖沼, 46(4): 958-968 |
闫龙, 宋娜, 王俊, 等. 2015. 基于线粒体控制区的中华绒螯蟹群体遗传多样性分析. 水生生物学报, 39(3): 615-620 |
农业部渔业渔政管理局. 2017. 2017年中国渔业统计年鉴. 北京: 中国农业出版社
|
李晨虹, 李思发. 1999. 中国大陆沿海六水系绒螯蟹(中华绒螯蟹和日本绒螯蟹)群体亲缘关系:形态判别分析. 水产学报, 23(4): 337-342 |
赵金良, 李思发. 1999. 中国大陆沿海六水系绒螯蟹(中华绒螯蟹和日本绒螯蟹)群体亲缘关系:生化遗传差异分析. 水产学报, 23(4): 331-336 |
赵恒亮, 吴旭干, 龙晓文, 等. 2016. 长江、黄河和辽河种群中华绒螯蟹雄体成蟹可食组织营养组成的比较. 中国水产科学, 23(5): 1117-1129 |
彭欣悦, 2016.长江口中华绒螯蟹亲蟹遗传多样性及其增殖放流效果评估的研究.上海: 上海海洋大学硕士学位论文, 24-29
|
董志国, 沈双烨, 李晓英, 等. 2013. 中国沿海三疣梭子蟹脂肪酸指纹标记的多元分析. 水产学报, 37(2): 192-200 |
Abdi H, Williams L J, 2010. Principal component analysis. Wiley Interdisciplinary Reviews:Computational Statistics, 2(4): 433-459 DOI:10.1002/wics.101 |
Anandakrishnan R, Onufriev A, 2008. Analysis of basic clustering algorithms for numerical estimation of statistical averages in biomolecules. Journal of Computational Biology, 15(2): 165-184 DOI:10.1089/cmb.2007.0144 |
Arslan Y, Hacioğlu B T, 2018. Seed fatty acid compositions and chemotaxonomy of wild safflower (Carthamus L. , Asteraceae) species in Turkey. Turkish Journal of Agriculture and Forestry, 42(1): 45-54 |
Atta-Krah K, Kindt R, Skilton J N et al, 2004. Managing biological and genetic diversity in tropical agroforestry. Agroforestry Systems, 61-62(1-3): 183-194 DOI:10.1023/B:AGFO.0000028998.32732.65 |
Chang Y M, Liang L Q, Ma H T et al, 2008. Microsatellite analysis of genetic diversity and population structure of Chinese mitten crab (Eriocheir sinensis). Journal of Genetics and Genomics, 35(3): 171-176 DOI:10.1016/S1673-8527(08)60023-5 |
Chapelle S, 1978. Influence of salinity on the lipid composition and the fatty-acid pattern of muscle and hepatopancreas of the Chinese crab Eriocheir sinensis. Archives Internationales de Physiologie et de Biochimie, 86(2): 393-401 DOI:10.3109/13813457809069914 |
Chen L H, Yang J L, Barnes M D, 2014. Effect of natural disturbance on migrations and population dynamics of a catadromous species, Eriocheir japonicus, along the Lo-Mei stream in northern Taiwan. Hydrobiologia, 726(1): 179-194 DOI:10.1007/s10750-013-1763-7 |
Chu K H, Ho H Y, Li C P et al, 2003. Molecular phylogenetics of the mitten crab species in Eriocheir, sensu lato (Brachyura:Grapsidae). Journal of Crustacean Biology, 23(3): 738-746 DOI:10.1651/C-2347 |
Dalsgaard J, John M S, Kattner G et al, 2003. Fatty acid trophic markers in the pelagic marine environment. Advances in Marine Biology, 46: 225-340 DOI:10.1016/S0065-2881(03)46005-7 |
Figueiredo C, Baptista M, Rosa I C et al, 2017. 3D chemoecology and chemotaxonomy of corals using fatty acid biomarkers:Latitude, longitude and depth. Biochemical Systematics and Ecology, 70: 35-42 DOI:10.1016/j.bse.2016.10.016 |
Grahl-Nielsen O, Jacobsen A, Christophersen G et al, 2010. Fatty acid composition in adductor muscle of juvenile scallops (Pecten maximus) from five Norwegian populations reared in the same environment. Biochemical Systematics Ecology, 38(4): 478-488 DOI:10.1016/j.bse.2010.04.010 |
Guo J Y, Ng N K, Dai A et al, 1997. The taxonomy of three commercially important species of mitten crabs of the genus Eriocheir de haan.. 1835 (Crustacea:Decapoda:Brachyura:Grapsidae). The Raffles Bulletin of Zoology, 45(2): 445-476 |
Herborg L M, Rushton S P, Clare A S et al, 2003. Spread of the Chinese mitten crab (Eriocheir sinensis H. Milne Edwards) in continental Europe:analysis of a historical data set. Hydrobiologia, 503(1-3): 21-28 |
Imbs A B, Demidkova D A, Dautova T N et al, 2009. Fatty acid biomarkers of symbionts and unusual inhibition of tetracosapolyenoic acid biosynthesis in corals (Octocorallia). Lipids, 44(4): 325-335 DOI:10.1007/s11745-008-3266-2 |
Jarvis B D W, Tighe S W, 1994. Rapid identification of Rhizobium species based on cellular fatty acid analysis. Plant and Soil, 161(1): 31-41 DOI:10.1007/BF02183083 |
Li G, Shen Q, Xu Z X, 1993. Morphometric and biochemical genetic variation of the mitten crab, Eriocheir, in southern China. Aquaculture, 111(1-4): 103-115 DOI:10.1016/0044-8486(93)90029-X |
Ma H T, Chang Y M, Yu D M et al, 2007. Microsatellite variations among four populations of Eriocheir sinensis. Zoological Research, 28(2): 126-133 |
Morrison W R, Smith L M, 1964. Preparation of fatty acid methyl esters and dimethylacetals from lipids with boron fluoride-methanol. Journal of Lipid Research, 5(4): 600-608 |
Neira R, Lhorente J P, Araneda C et al, 2004. Studies on carcass quality traits in two populations of Coho salmon (Oncorhynchus kisutch):phenotypic and genetic parameters. Aquaculture, 241(1-4): 117-131 DOI:10.1016/j.aquaculture.2004.08.009 |
Ricardo F, Pimentel T, Moreira A S P et al, 2015. Potential use of fatty acid profiles of the adductor muscle of cockles (Cerastoderma edule) for traceability of collection site. Scientific Reports, 5(120): 11125 |
Santos I C, Smuts J, Choi W S et al, 2018. Analysis of bacterial FAMEs using gas chromatography - vacuum ultraviolet spectroscopy for the identification and discrimination of bacteria. Talanta, 182: 536-543 DOI:10.1016/j.talanta.2018.01.074 |
Shin P K S, Yip K M, Xu W Z et al, 2008. Fatty acid as markers to demonstrating trophic relationships among diatoms, rotifers and green-lipped mussels. Journal of Experimental Marine Biology and Ecology, 357(1): 75-84 DOI:10.1016/j.jembe.2008.01.002 |
Sui L Y, Sun H X, Wu X G et al, 2011a. Effect of dietary HUFA on tissue fatty acid composition and reproductive performance of Chinese mitten crab Eriocheir sinensis (H. Milne-Edwards) broodstock. Aquaculture International, 19(2): 269-282 DOI:10.1007/s10499-010-9379-7 |
Sui L Y, Wille M, Cheng Y X et al, 2011b. Larviculture techniques of Chinese mitten crab Eriocheir sinensis. Aquaculture, 315(1-2): 16-19 DOI:10.1016/j.aquaculture.2010.06.021 |
Sui L Y, Zhang F M, Wang X M et al, 2009. Genetic diversity and population structure of the Chinese mitten crab Eriocheir sinensis in its native range. Marine Biology, 156(8): 1573-1583 DOI:10.1007/s00227-009-1193-2 |
Tang B P, Zhou K Y, Song D X et al, 2003. Molecular systematics of the Asian mitten crabs, genus Eriocheir (Crustacea:Brachyura). Molecular Phylogenetics and Evolution, 29(2): 309-316 DOI:10.1016/S1055-7903(03)00112-X |
Wang C H, Li C H, Li S F, 2008. Mitochondrial DNA-inferred population structure and demographic history of the mitten crab (Eriocheir sensu stricto) found along the coast of mainland China. Molecular Ecology, 17(15): 3515-3527 |
Wang G Z, Kong X H, Wang K J et al, 2007. Variation of specific proteins, mitochondria and fatty acid composition in gill of Scylla serrata (Crustacea, Decapoda) under low temperature adaptation. Journal of Experimental Marine Biology and Ecology, 352(1): 129-138 DOI:10.1016/j.jembe.2007.07.017 |
Wu X G, Wang Z K, Cheng Y X et al, 2011. Effects of dietary phospholipids and highly unsaturated fatty acids on the precocity, survival, growth and hepatic lipid composition of juvenile Chinese mitten crab, Eriocheir sinensis (H. Milne-Edwards). Aquaculture Research, 42(3): 457-468 DOI:10.1111/are.2011.42.issue-3 |
Xu J W, Chu K H, 2012. Genome scan of the mitten crab Eriocheir sensu stricto in East Asia:population differentiation, hybridization and adaptive speciation. Molecular Phylogenetics and Evolution, 64(1): 118-129 DOI:10.1016/j.ympev.2012.03.009 |
Zhang C, Li Q Q, Wu X G et al, 2017. Genetic diversity and genetic structure of farmed and wild Chinese mitten crab (Eriocheir sinensis) populations from three major basins by mitochondrial DNA COI and Cyt b gene sequences. Mitochondrial DNA Part A, 29(7): 1081-1089 |
 2019, Vol. 50
2019, Vol. 50


