
中国海洋湖沼学会主办。
文章信息
- 李芬, 陈绮萍, 何佩莹, 黄小琪, 陈忠凯, 周惠强, 钟东明, 舒琥. 2019.
- LI Fen, CHEN Qi-Ping, HE Pei-Ying, HUANG Xiao-Qi, CHEN Zhong-Kai, ZHOU Hui-Qiang, ZHONG Dong-Ming, SHU Hu. 2019.
- 北江大刺鳅(Mastacembelus armatus)的核型分析及线粒体Cyt b基因和D-loop的遗传多样性
- THE KARYOTYPE PATTERN AND GENETIC DIVERSITY OF MITOCHONDRIAL DNA Cyt b GENE AND D-LOOP SEQUENCE IN MASTACEMBELUS ARMTUS BEIJIANG RIVER, SOUTH CHINA
- 海洋与湖沼, 50(2): 449-454
- Oceanologia et Limnologia Sinica, 50(2): 449-454.
- http://dx.doi.org/10.11693/hyhz20181100275
-
文章历史
- 收稿日期:2018-11-16
- 收修改稿日期:2018-12-20
大刺鳅(Mastacembelus armatus)属合鳃目、刺鳅科、刺鳅属, 俗称辣锥、石锥等, 分布于非洲、东南亚国家及我国长江以南的各水系, 栖息于砾石底的江河溪流中, 常藏匿于石缝或洞穴中, 以小型无脊椎动物和部分植物为食(Nelson, 2006)。大刺鳅作为重要的经济鱼类, 其营养丰富(伍远安等, 2010)、味道鲜美, 深受消费者喜欢。因水质污染及过度捕捞, 大刺鳅野生资源遭受严重损害, 为保护该物种, 大刺鳅被广东、福建、贵州、湖南等地列入重点保护的野生水生动物。目前国内对大刺鳅的研究相对较少, 主要集中在大刺鳅的繁殖、营养、种群遗传及亲缘地理的研究(王方等, 2012;曾庆祥等, 2016;樊海平等, 2018), 本课题组在对大刺鳅野生群体的形态性状、遗传多样性以及大刺鳅微卫星标记开发和筛选方面进行了研究(杨华强等, 2016;舒琥等, 2017;房祖业等, 2018), 但对大刺鳅的遗传多样性的丰富度研究还较少, 对其染色体核型分析尚未见报道。
染色体是遗传物质的主要载体, 是研究物种演化、分类必不可少的重要手段。利用染色体组型分析比较, 对于探讨其系统演化、杂交育种、多倍体育种以及原位杂交、基因定位等研究具有重要作用(成美玲等, 2018)。
线粒体DNA(mtDNA)具核苷酸替代率高、进化快、核苷酸数少、母系遗传等特点, 在鱼类遗传多样性和系统发育的研究中应用广泛(Lynch et al, 1990;郭立等, 2011)。线粒体Cyt b和D-loop是研究应用比较广泛的序列, 可以从不同角度揭示鱼类种群遗传变异及多样性的特点, 适用于群体水平的多样性分析(于旭蓉等, 2011)。
本文从细胞学和线粒体DNA分子水平对北江野生大刺鳅的遗传多样性进行了分析, 探讨大刺鳅的种群遗传结构以及遗传多样性, 旨在为开展大刺鳅的种质资源的保护与开发利用提供科学依据。
1 材料与方法 1.1 实验材料实验用鱼33尾大刺鳅(Mastacembelus armatus)采自北江仁化、英德流域, 平均体质量为39.37g, 平均体长为18.90cm, 其中9尾(5雌4雄)用于核型分析, 暂养于实验室鱼缸内; 余下24尾, 取适量的背部肌肉和尾鳍置于2mL离心管中, 用无水乙醇固定, 于–20℃冰箱保存备用。
1.2 染色体标本制备及核型分析参照舒琥等(2010)等PHA注射法, 稍作改良进行大刺鳅的染色体标本制备实验。选取100个分散较好的来自不同个体的中期分裂相细胞, 利用(Olympus-BX51显微镜对其进行观察统计, 选取约10个较好的中期分裂相, 拍摄, 放大、测量每一条染色体参数并计算其臂比值和相对长度, 按照Levan等(1964)提出的标准进行配对和分类排列。
1.3 DNA提取、扩增及测序采用上海生物工程公司(上海生工)提供的DNA提取试剂盒, 按试剂盒的操作步骤进行总DNA的提取。用1%的琼脂糖凝胶电泳检测所提取DNA的质量和纯度, 保存于–20℃, 备用。PCR扩增引物由上海生工合成, Cyt b基因引物序列分别为MaCyt b-F: 5′-CCA CCGTTGTTATTCAACTACAAGA-3′和MaCyt b-R: 5′-GCTCTGGCGCTGAGCTATC-3′; D- loop引物序列分别为MaD-loopF: 5′-TTAACTTCC GCCCCTAACTCC-3′和MaD-loopR: 5'-GGGCCCAT TTTAACATCTTCAGTG-3′。PCR反应体系按照刘伟等(2016)所采用的体系进行。Cyt b基因扩增反应条件: 94℃预变性2min后, 再进行35个循环, 每一循环包括: 94℃变性45s, 58℃退火45s, 72℃延伸72s;最后72℃延伸10min。D-loop基因扩增94℃预变性2min, 其余反应条件参照吕凤义等(2008)。PCR产物用1%的琼脂糖凝胶电泳跑胶, 观察, 拍照, 送上海美吉生物医药科技有限公司进行序列检测。
1.4 数据处理测序成功后使用SeqMan II、ClustalX2.0软件对序列进行拼接和人工比对, 应用DNASP5.10和MEGA5.05软件对两个线粒体基因(Cyt b和D-loop)序列的多肽位点、遗传多样性参数(单倍型数(Number of Haplotypes, h)、单倍型多样性(Haplotype (gene) diversity, Hd)、核苷酸多样性(Nucleotide diversity, Pi)、平均核苷酸差异数(Average number of nucleotide differences, k)、种群大小变动、核酸组成和遗传距离等进行统计分析。
2 结果与分析 2.1 大刺鳅染色体核型分析选取100个分散充分的来自不同个体的中期分裂相进行观察统计, 结果见表 1。北江大刺鳅的中期分裂相染色体数目为48条, 占分裂相总数的50.0%, 由此可以判断大刺鳅的染色体众数为48。通过核型分析可知, 在大刺鳅的24对染色体中, 中着丝粒染色体(m)7对; 端着丝粒染色体(t)17对, 大刺鳅的染色体核型为2n=14m+34t, 臂数: NF=62 (表 2), 无异形性染色体, 次缢痕、随体等。北江大刺鳅的中期分裂相和染色体核型见图 1。
| 染色体数 | 细胞数 | 出现频率(%) |
| < 41 | 24 | 24.0 |
| 42—43 | 14 | 14.0 |
| 44—45 | 10 | 10.0 |
| 46—47 | 1 | 1.0 |
| 48 | 50 | 50.0 |
| > 49 | 1 | 1.0 |
| 编号 | 相对长度 | 臂比 | 类型 |
| 1 | 6.47±0.20 | 1.27±0.09 | m |
| 2 | 6.11±0.25 | 1.14±0.05 | m |
| 3 | 5.91±0.15 | 1.13±0.08 | m |
| 4 | 5.78±0.18 | 1.32±0.07 | m |
| 5 | 5.63±0.03 | 1.17±0.03 | m |
| 6 | 5.43±0.13 | 1.28±0.14 | m |
| 7 | 5.27±0.08 | 1.41±0.08 | m |
| 8 | 4.36±0.33 | ∞ | t |
| 9 | 4.13±0.00 | ∞ | t |
| 10 | 4.10±0.48 | ∞ | t |
| 11 | 3.90±0.28 | ∞ | t |
| 12 | 3.80±0.08 | ∞ | t |
| 13 | 3.59±0.18 | ∞ | t |
| 14 | 3.54±0.08 | ∞ | t |
| 15 | 3.52±0.31 | ∞ | t |
| 16 | 3.41±0.25 | ∞ | t |
| 17 | 3.41±0.10 | ∞ | t |
| 18 | 3.41±0.10 | ∞ | t |
| 19 | 3.41±0.05 | ∞ | t |
| 20 | 3.21±0.41 | ∞ | t |
| 21 | 3.13±0.03 | ∞ | t |
| 22 | 2.98±0.03 | ∞ | t |
| 23 | 2.80±0.31 | ∞ | t |
| 24 | 2.68±0.03 | ∞ | t |
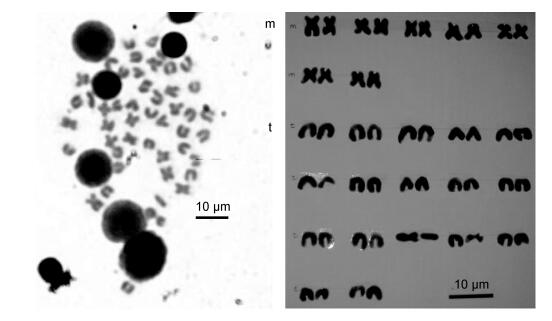 |
| 图 1 大刺鳅核型分析 Fig. 1 The karyotype of M. armatus |
Cyt b基因序列长度为1438bp, 出现4个单一信息位点、17个简约信息位点。A、T、C、G的平均含量分别为24.97%、28.46%、32.75%、13.82%, 其中A+T的含量(53.43%)比G+C的含量高(46.57%)(表 3), 符合动物Cyt b的特征, 并与其他鱼类的碱基组成比例相似(沈朕等, 2017)。
| 序列 | 碱基平均比率 | |||||
| A | T(U) | C | G | A+T | G+C | |
| Cyt b | 24.97% | 28.46% | 32.75% | 13.82% | 53.43% | 46.57% |
| D-loop | 31.90% | 35.25% | 19.64% | 13.21% | 67.15% | 32.85% |
D-loop基因序列长度为895bp, 出现6个单一信息位点、18个简约信息位点。A、T、C、G的平均含量分别为31.90%、35.25%、19.64%和13.21%, 其中A+T的含量(67.15%)显著高于G+C的含量(32.85%), 符合大多数脊椎动物线粒体DNA的特点。
2.2.2 遗传多样性分析大刺鳅遗传多样性的参数指标由DNASP5.10和MEGA5.05软件处理得出, 遗传多样性各个方面的体现由不同的线粒体基因(如Cyt b和D-loop)片段决定。
基于Cyt b基因, 北江大刺鳅种群平均核苷酸差异数(k)、单倍型多样性指数(Hd)和核苷酸多样性指数(Pi)分别为5.938、0.6560和00539(表 4)。
| 序列 | 单倍型数 | 单倍型多样性(Hd) | 核苷酸多样性(Pi) | 平均核苷酸差异数(k) |
| Cyt b | 8 | 0.656 | 0.00539 | 5.938 |
| D-loop | 7 | 0.541 | 0.00817 | 6.805 |
基于D-loop, 北江大刺鳅种群平均核苷酸差异数(k)、单倍型多样性指数(Hd)和核苷酸多样性指数(Pi)分别为6.805、0.541、0. 00817和(表 4)。
2.2.3 遗传距离分析将线粒体Cyt b和D-loop基因序列分别按照单倍型类型进行分组, 每个单倍型保留一个序列, 重新整理数据, 然后使用MEGA5.05软件进行分析, 经过数据处理后得到此两种基因序列中各单倍型间的遗传距离。
在Cyt b基因中, 实验所用24个个体的总平均遗传距离为0.005, 该8个单倍体间的遗传距离范围在0.000—0.018间, 单倍体间的遗传距离结果见表 5。
| 单倍型 | Hap1 | Hap2 | Hap3 | Hap4 | Hap5 | Hap6 | Hap7 | Hap8 |
| Hap1 | ||||||||
| Hap2 | 0.001 | |||||||
| Hap3 | 0.001 | 0.002 | ||||||
| Hap4 | 0.001 | 0.002 | 0.002 | |||||
| Hap5 | 0.013 | 0.012 | 0.014 | 0.014 | ||||
| Hap6 | 0.017 | 0.016 | 0.018 | 0.018 | 0.004 | |||
| Hap7 | 0.015 | 0.014 | 0.016 | 0.016 | 0.002 | 0.002 | ||
| Hap8 | 0.001 | 0.002 | 0.002 | 0.002 | 0.014 | 0.018 | 0.016 |
在D-loop基因中, 实验所用22个个体的总平均遗传距离为0.008, 该7个单倍体间的遗传距离范围在0.000—0.026间, 单倍体间的遗传距离结果见表 6。
| 单倍型 | Hap1 | Hap2 | Hap3 | Hap4 | Hap5 | Hap6 | Hap7 |
| Hap1 | |||||||
| Hap2 | 0.021 | ||||||
| Hap3 | 0.004 | 0.025 | |||||
| Hap4 | 0.020 | 0.006 | 0.023 | ||||
| Hap5 | 0.001 | 0.022 | 0.002 | 0.021 | |||
| Hap6 | 0.022 | 0.001 | 0.026 | 0.007 | 0.023 | ||
| Hap7 | 0.022 | 0.001 | 0.026 | 0.007 | 0.023 | 0.002 |
该群体的单倍体间的遗传距离未能达到划分亚种和种的范围(亚种范围是0.02—0.20, 种范围是0.05—0.30), 该划分依据由Shaklee等(1982)等提出。实验结果显示该群体的个体间基本没有发生遗传分化, 其遗传差异不显著。
2.2.4 种群的中性检验两个线粒体基因(Cyt b和D-loop)的Tajima’s D检验和Fu’s Fs检验结果由DNASP5.05软件处理数据后分析, 结果表明, 大刺鳅细胞色素b序列的Tajima’s D值和Fu’s Fs值都为正值, P值均不显著(P < 0.05表示显著), 由此说明该种群大小并未经历过明显种群扩张; D-Loop序列的Tajima’s D值和Fu’s Fs值也都为正值, 而且P值都不显著(P < 0.05表示显著), 由此说明该种群没有发生明显的扩张(表 7)。
| 序列名称 | Tajima’s D值 | P值 | Fu’s Fs值 | P值 |
| Cyt b | 0.20510 | P > 0.10 | 0.49034 | P > 0.10 |
| D-Loop | 0.12704 | P > 0.10 | 0.21762 | P > 0.10 |
染色体作为遗传物质的主要载体, 对其进行核型分析研究, 能够了解生物的遗传组成、变异规律以及生物的发育机理, 推测和鉴别种间杂交和多倍体育种, 为了解性别遗传的基本原理和基因组数、物种的进化起源以及种族关系的鉴别提供了重要的参考依据(吴仲庆, 1998)。
由本实验结果可知, 大刺鳅的二倍体染色体数目为2n=48, 核型为2n=14m+34t, 与武汉大学生物系细胞遗传实验室所做的研究结果2n=48t (生物系细胞遗传学实验室, 1985)一致, 但在染色体形态方面存在差异, 本文结果得出大刺鳅核型多数是端部着丝粒染色体, 少数是中部着丝粒染色体, 但均未出现异形性染色体及有特殊标志性特征的染色体。两者的差异可能是由于不同地理种群所表现出的染色体多态性, 前者样本采自33年前的广西, 本文样本采自广东北江流域的仁化、英德; 也可能是不同研究者的实验方法不同以及测量误差所致。在特定分类阶元中, 染色体数目多的(多倍体除外)具有较多端部着丝粒染色体的鱼类应是较原始的类型, 而染色体数目少的为较特化类型。例如核型公式为: 2n=48t, 可认为其是相对较原始的类型, 而本研究大刺鳅有7对中部着丝粒染色体, 臂数较多, 相对进化, 属于较为进化的类群。关于大刺鳅的分类地位, 国内学者(除本研究团队外)均沿用成庆泰等(1987)主编的中国鱼类系统检索中的分类结果, 将其归为鲈形目, 而Nelson(2006)根据线粒体DNA及分子标记等结果将大刺鳅归为合鳃目。虽然核型研究对鱼类分类问题的探讨至关重要, 但也存在其自身的局限, 本文的核型研究结果就不能为大刺鳅的分类归属提供直接证据。但从本课题组对大刺鳅的研究结果综合考虑认同Nelson(2006)的分类结果。
3.2 北江大刺鳅遗传多样性分析北江大刺鳅种群D-loop序列中A+T的含量高于Cyt b基因中A+T的含量, 这表明D-loop进化速度更快, 因为D-loop为非编码基因, 故D-loop所受自然选择压力小, 突变之后被保留下来的几率更大, 更适合于做种内遗传变异分析(Yang et al, 2002)。在北江大刺鳅种群Cyt b基因和D-loop序列中, G含量最低, 碱基A+T的含量高于G+C的含量, 。这在其他鱼类线粒体DNA碱基组成中也发现相似的特点(刘朋朋等, 2013;钟立强等, 2013)。
在评估种群遗传多样性时, 核苷酸多样性指数和平均遗传距离是两个非常重要的指标, 而核苷酸多样性指数是包含了各单倍型在群体中所占的比例, 因此核苷酸多样性指数较平均遗传距离在反映种群的遗传多样性情况时更为可靠和准确(姬南京等, 2014)。核苷酸多样性指数越高, 则遗传多态性越高, 反之亦然。北江大刺鳅的种群Cyt b基因的核苷酸多样性指数为0.00539, 故根据Grant等(1998)中遗传多样性高低的评价标准, 该种群的遗传多样性处于一般水平。大刺鳅Cyt b基因的单倍型数为8;单倍型多样性(Hd)是0.656, 处于中等水平; 核苷酸多样性(Pi)是0.00539, 处于较高水平:平均核苷酸差异数(k)为5.938, 平均核苷酸差异数越大, 说明遗传多样性越丰富; 大刺鳅D-loop基因的单倍型数为7;单倍型多样性(Hd)是0.541, 处于中等水平; 核苷酸多样性(Pi)是0.00817, 处于较高水平:平均核苷酸差异数(k)为6.805, 说明遗传多样性丰富。上述DNA多肽位点分析及多肽分析检验说明北江流域的大刺鳅遗传多样性拥有较高的丰富度。
根据中性检验分析的结果可知, 所检验的群体的种群数目较为稳定, 在总体上并未经历大规模的扩张。综上所述, 本实验的北江大刺鳅遗传多样性较丰富, 且彼此间差异不大, 每个个体间的亲缘关系较近。
4 结论影响大刺鳅遗传多样性水平的原因有很多种, 如水质环境变化、人为干预、过度捕捞、自身进化程度等等, 但是从目前来看, 主要是由于人为干预导致生态环境恶化所造成的。现在大刺鳅野生资源相对匮乏, 且其规模化全人工繁殖技术尚未成功, 故本实验得出的数据对进一步研究北江流域经济鱼类的遗传多样性具有重要参考价值, 并为大刺鳅的种群遗传多样性保护和种质资源的开发和利用提供了理论依据。
于旭蓉, 仇雪梅, 柳晓瑜, 等. 2011. 线粒体DNA多态性在海洋动物群体遗传结构研究中的应用. 生物技术通报, (10): 49-54 |
王方, 黄小林, 拉达等, 2012.中国南部地区大刺鳅种群遗传和亲缘地理研究.见: 中国鱼类学会.中国海洋湖沼学会鱼类学分会, 中国动物学会鱼类学分会2012年学术研讨会论文集.兰州: 中国海洋湖沼学会, 中国鱼类学会, 23
|
生物系细胞遗传学实验室. 1985. 四川广西两省59种鱼的染色体研究初报. 武汉大学学报(自然科学版), (1): 124-126 |
成庆泰, 郑葆珊. 1987. 中国鱼类系统检索. 北京: 科学出版社, 459
|
成美玲, 田永胜, 吴玉萍, 等. 2018. 云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交后代染色体核型分析. 渔业科学进展, 39(2): 67-75 |
吕凤义, 左艳玲, 范月明, 等. 2008. 4种鲿科鱼类基于线粒体DNA控制区序列的比较研究. 华南师范大学学报(自然科学版), (2): 105-110 |
伍远安, 梁志强, 李传武, 等. 2010. 两种刺鳅肌肉营养成分分析及评价. 营养学报, 32(5): 499-502 |
刘伟, 代应贵, 袁振兴, 等. 2016. 都柳江粗唇种群线粒体DNA Cyt b基因和D-loop序列组成及遗传多样性. 淡水渔业, 46(3): 10-15, 51 DOI:10.3969/j.issn.1000-6907.2016.03.002 |
刘朋朋, 钟立强, 潘建林, 等. 2013. 基于线粒体D-loop区分析黄颡鱼(Pelteobagrus fulvidraco)五个淡水湖泊群体的遗传多样性. 海洋与湖沼, 44(3): 728-733 |
杨华强, 李强, 舒琥, 等. 2016. 华南及邻近地区大刺鳅遗传多样性的ISSR分析. 水生生物学报, 40(1): 63-70 |
吴仲庆. 1998. 水产生物遗传育种学. 厦门: 厦门大学出版社, 7-2
|
沈朕, 关洪斌, 郑风荣, 等. 2017. 基于cytb和D-loop的4个大泷六线鱼群体遗传多样性分析. 海洋科学进展, 35(4): 524-534 DOI:10.3969/j.issn.1671-6647.2017.04.009 |
房祖业, 陈晓东, 吴咏诗, 等. 2018. 大刺鳅(Mastacembelus armatus)二、三、四碱基重复微卫星标记的筛选和特征分析. 海洋与湖沼, 49(1): 174-182 |
钟立强, 刘朋朋, 潘建林, 等. 2013. 长江中下游5个湖泊黄颡鱼(Pelteobagrus fulvidraco)种群线粒体细胞色素b基因的遗传变异分析. 湖泊科学, 25(2): 302-308 DOI:10.3969/j.issn.1003-5427.2013.02.019 |
郭立, 李隽, 王忠锁, 等. 2011. 基于四个线粒体基因片段的银鱼科鱼类系统发育. 水生生物学报, 35(3): 449-459 |
姬南京, 常亚青, 赵冲, 等. 2014. 3个地理群体仿刺参D-loop序列的变异及系统发生分析. 水产学报, 38(4): 477-482 |
舒琥, 江小璐, 杨华强, 等. 2017. 华南地区7个大刺鳅野生群体的形态差异分析. 广州大学学报(自然科学版), 16(3): 8-14 DOI:10.3969/j.issn.1671-4229.2017.03.002 |
舒琥, 蔡晓阅, 刘锋, 等. 2010. 鲀形目3种鱼的染色体组型分析. 动物学杂志, 45(2): 101-106 |
曾庆祥, 方园, 曾学平, 等. 2016. 大刺鳅的生物学特性与人工繁养殖技术. 中国水产, (3): 70-73 DOI:10.3969/j.issn.1002-6681.2016.03.030 |
樊海平, 邱曼丽, 钟全福, 等. 2018. 不同生长阶段野生和养殖大刺鳅营养成分的比较. 安徽农业科学, 46(8): 92-96 DOI:10.3969/j.issn.0517-6611.2018.08.027 |
Grant W A S, Bowen B W, 1998. Shallow population histories in deep evolutionary lineages of marine fishes:Insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 89(5): 415-426 DOI:10.1093/jhered/89.5.415 |
Levan A, Fredga K, Sandberg A A, 1964. Nomenclature for centromeric position on chromosomes. Hereditas, 52(2): 201-220 |
Lynch M, Crease T J, 1990. The analysis of population survey data on DNA sequence variation. Molecular Biology and Evolution, 7(4): 377-394 |
Nelson J S, 2006. Fishes of the World. 4th ed. New York: John Wiley and Sons, Inc.
|
Shaklee J B, Tamaru C S, Waples R S, 1982. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins. Pacific Science, 36(2): 141-157 |
Yang Y H, Kim K I, Cothran E G et al, 2002. Genetic diversity of Cheju Horses (Equus caballus) determined by using mitochondrial DNA D-loop polymorphism. Biochemical Genetics, 40(5-6): 175-186 |
 2019, Vol. 50
2019, Vol. 50


