中国海洋湖沼学会主办。
文章信息
- 王鹏斌, 戴鑫烽, 陆斗定. 2019.
- WANG Peng-Bin, DAI Xin-Feng, LU Dou-Ding. 2019.
- 米氏凯伦藻(Km02)共培养细菌群落的研究
- CO-CULTURED BACTERIAL COMMUNITY OF KARENIA MIKIMOTOI (KM02)
- 海洋与湖沼, 50(3): 644-651
- Oceanologia et Limnologia Sinica, 50(3): 644-651.
- http://dx.doi.org/10.11693/hyhz20180700179
文章历史
-
收稿日期:2018-07-23
收修改稿日期:2018-10-16
近年来, 在中国及周边国家乃至世界沿海有害赤潮的发生均呈现出频率逐渐增加、规模不断扩大的趋势, 危害人类的身体健康和生命安全, 同时也造成了巨大的经济损失(Lu et al, 2001; Dai et al, 2014; Park et al, 2014; Kim et al, 2018)。在我国东海, 长期以来都是由东海原甲藻(Prorocentrum donghaiense Lu)形成赤潮, 规模有时达数千平方公里, 实属世界罕见, 引起了我国政府、公众和科技界的广泛关注。自2002年5月4日在福鼎和霞浦东部海域出现米氏凯伦藻赤潮后, 东海海区米氏凯伦藻时有发生, 且频度与规模呈现增高增大的趋势, 有时米氏凯伦藻与东海原甲藻共同形成赤潮(许翠娅等, 2010; 刘志国等, 2014)。米氏凯伦藻在东海出现与暴发的时间在5—6月, 赤潮持续时间多数在12d之内, 但据许翠娅等(2010)报道, 在福建连江近岸海域也有过1起米氏凯伦藻的赤潮持续时间最长达35d。然而, 中国首次由米氏凯伦藻引发的赤潮时间可追溯到1980年的香港水域(香港渔农自然护理署, 2013)。后来, 张水浸等(1988)报道的1986年6月厦门西港区曾发生的一次裸甲藻赤潮, 被怀疑是米氏凯伦藻赤潮(刘志国等, 2014), 这可能是我国内地较早的关于米氏凯伦藻赤潮的报道。在广东珠江口的桂山岛、外伶仃岛和香港海域, 1998年从3月中旬至4月中旬, 发生了持续近30d的大面积米氏凯伦藻赤潮, 造成了大批养殖鱼、贝类死亡, 据统计粤港两地因此造成的经济损失达3.5亿元, 其中广东省近5000万元, 这同时也是国内明确记录的首次发生该种引发的赤潮, 也是目前我国该种造成经济损失最严重的一次(王朝晖等, 2001)。
作为有毒藻, 米氏凯伦藻能够产生的溶血毒素, 而溶血毒素是造成鱼类大量死亡的主要原因之一, 其成分以糖脂类、糖苷类和不饱和多脂肪酸类化合物为主, 也有少数蛋白质和肽类物质(刘桂英等, 2015)。由于溶血毒素能够造成鱼鳃小叶上皮细胞增生、邻近鳃叶粘连、上皮细胞脱落、鳃血管破裂、血细胞渗出等病理现象, 进而导致鱼类呼吸困难, 从而死亡(王朝晖等, 2001)。然而, 影响赤潮发生与毒性的因素多种多样, 大部分的研究集中于物理、化学与生理因素(曾江宁等, 2004; Heisler et al, 2008; Anderson et al, 2009; Park et al, 2014), 对于生态系统中生物因素(比如细菌等)对赤潮藻群落和产毒的影响与相互作用关注相对较少(Azam et al, 1983)。
近年来, 随着分子生物学和生态学技术的发展, 高通量测序技术的建立为研究复杂样品中细菌群落的物种的丰富度与多样性提供了强有力的技术支撑(Celikkol-Aydin et al, 2016; Guidi et al, 2018)。尽管目前已经有较多关于米氏凯伦藻的研究, 但对其共培养细菌菌群的物种及多样性背景信息尚知之甚少。研究赤潮原因种及其共生或寄生的细菌群落结构, 对研究细菌和赤潮藻的相互作用, 乃至对了解和预测赤潮的发生、变化趋势以及消亡都具有重要的意义(Croft et al, 2005; Bolch et al, 2011; Sarmento et al, 2012)。为此, 本文对我国沿海分离的一株米氏凯伦藻的共培养菌群进行了高通量测序分析, 并获得了该株系的共培养菌群的种类、相对丰度、多样性与进化位置等信息。研究结果可为后续开展藻菌关系、米氏凯伦藻赤潮暴发与终结, 产毒特性等生物机理的研究提供必要的研究方法与背景信息。
1 材料与方法 1.1 实验藻种与培养方法实验所用的藻种米氏凯伦藻(Karenia mikimotoi, Km02)分离自赤潮高发区珠海海域, 由暨南大学赤潮与水环境研究中心藻种室(吕颂辉教授课题组)提供。培养基采用外海天然海水, 按照Guillard等(1962)改良的不含硅酸盐的f/2配方配制。将对数期藻种接种于100mL培养液, 最终培养起始浓度为约100cells/mL。共进行三个平行培养。培养温度为20℃, 光照强度约为100μmol(m2·s), 光暗比为12h:12h。
1.2 样品总DNA的提取培养10d后, 米氏凯伦藻处于对数期, 对藻进行回收。取100mL微藻培养液抽滤于0.2μm GTTP滤膜上(Millipore filter membrane, Millipore Filter Corporation, Ireland)。将滤膜浸泡于盛有0.8mL细胞裂解液的2mL EP管。–20℃保存待DNA提取。
总DNA的提取参考Wang等(2017)提取海洋水体里环境DNA的方法。将EP管置于液氮中直到完全冻凝后放入65℃水浴锅, 进行冻融, 重复3次。加入8μL的10mg/mL蛋白酶K (10mg/mL溶解于TE缓冲液: 10mmol/L Tris-HCI, 1mmol/L EDTA; pH 8)。37℃水浴30min, 期间颠倒EP管3—4次使混匀。加80μL 20%的SDS溶液后, 65℃水浴2h, 期间颠倒EP管7—8次使混匀。振荡使混匀。加入等量的chloroform-isoamyalchol (24:1, V/V)后轻轻混匀。10000g离心5min, 用移液枪小心将上清液转移至新的2mL EP管, 不要扰动下层液体。如上清液不够清澈, 可重复加入chloroform- isoamyalchol (24:1, V/V)进行以上操作, 直到液体澄清。加入0.1倍等量体积的3mol/L的醋酸钠溶液后, 再加入0.6倍等量体积的异丙醇(isopropanol)。14000g离心30min, 小心倒去上清液。加入1mL 70%的4℃预冷乙醇洗涤, 振荡混匀。14000g离心15min, 小心倒去上清液。将EP管打开后倒置在吸水纸上, 使乙醇蒸发, 室温干燥15min以上。最后加入100μL灭菌的1×TE缓冲液, 振荡使DNA溶解混匀。
1.3 PCR扩增与高通量测序PCR扩增所使用细菌通用引物为Ⅴ3—Ⅴ4区338F—806R, 其中上游引物338F: 5′-ACTCCTACGG GAGGCAGCAG-3′; 下游引物806R: 5′-GGACTAC HVGGGTWTCTAAT- 3′, 进行PCR扩增。用20μL反应体系: 5×FastPfu Buffer (4μL)、2.5mmol/L dNTPs (2μL)、Forward Primer (5μmol/L) (0.4μL)、Reverse Primer (5μmol/L) (0.4μL)、FastPfu Polymerase (0.4μL)、Template DNA (10ng), 补ddH2O至20μL。PCR反应参数: 95℃预变性2min; 95℃变性30s, 55℃退火30s, 72℃延伸30s, 循环30次; 72℃终延伸5min, 10℃时停止。在进行胶回收, PCR产物定量, Miseq文库构建后, 通过Illumina MiSeq (Illumina Inc, San Diego, CA)平台进行高通量测序。
1.4 生物信息分析根据Miseq测序结果得到的PE reads, 根据overlap关系进行拼接, 并对序列质量进行质控和过滤。区分样本后, 对样本进行操作分类单元(Operational taxonomic units, OTU)聚类分析与物种分类学分析, 并通过OTU的多态性进行多样性指数分析, 以及对测序深度的检测与评估; 基于分类学分析结果, 在不同分类水平上进行细菌群落结构的统计与分析。
1.5 系统发生进化树在FastTree软件中(version 2.1.3 http://www.microbesonline.org/fasttree/)通过选择属水平上的分类信息对应序列, 根据最大似然法(approximately- maximum-likelihood phylogenetic trees)构建进化树, 并利用R语言将进化树图形化, 最终结果通过进化树与reads丰度组合图的形式呈现。
2 结果与分析 2.1 测序数据序列信息统计在培养10d后, 将藻液过膜后提取DNA完成后, 利用高通量测序技术分析了米氏凯伦藻(Km02)共培养菌群多样性。本次实验对米氏凯伦藻Km02株系的3个实验平行样品中的核糖体小亚基(16S)338F—806R区间进行扩增, 经过质控、拼接、去除barcode序列等处理, 本试验最终获得153647条有效序列(表 1), 单条序列平均长度样品平均长度442个碱基。
| 样本名 | 序列数 | 碱基数 | 平均长度(碱基) | 最短序列长度(碱基) | 最长序列长度(碱基) |
| Km_1 | 72826 | 32179710 | 442 | 279 | 508 |
| Km_3 | 45426 | 20096178 | 442 | 407 | 453 |
| Km_2 | 35395 | 15667399 | 443 | 421 | 451 |
| 总计 | 153647 | 67943287 | — | — | — |
米氏凯伦藻Km02株系的共培养菌群样品通过序列随机抽样法进行多样性稀释性曲线分析, 以抽到测序序列数与其代表OTU数目构建rarefaction曲线(图 1), 结果表明各个样品的高通量测序深度都很充分。样品多样性指数, 包括菌群多样性指数(Community diversity index) Shannon指数与Simpson指数, 菌群丰度指数(Community richness index) Ace指数与Chao指数, 及测序深度指数覆盖度(Coverage), 结果如表 2所示。通过多样性结果可看出3个实验平行效果好。其中, 米氏凯伦藻Km02株系共培养菌群有43个OTU。3个实验平行的平均Shannon指数为2.51, Simpson指数为0.12, Ace指数为43.98, Chao指数为43.50, 为此米氏凯伦藻Km02株系共培养细菌菌群的生物多样性较高(表 2)。测序深度指标覆盖度也几乎达到100%。
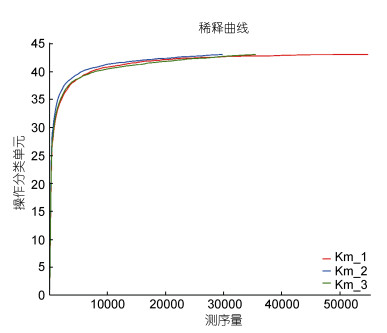 |
| 图 1 米氏凯伦藻(Km02)细菌菌群高通量测序结果稀释曲线 Fig. 1 Rarefaction curves of K. mikimotoi from the analysis of high-throughput sequencing 注: Km_1, Km_2, Km_3分别为米氏凯伦藻Km02株系的3个实验平行的样品 |
| 样本名 | 分类单元 (OTU) |
Shannon 指数 |
Simpson 指数 |
Ace 指数 |
Chao 指数 |
覆盖度 |
| Km_1 | 43 | 2.46 | 0.12 | 43.00 | 43.00 | 1.0000 |
| Km_2 | 43 | 2.56 | 0.11 | 44.40 | 44.00 | 0.9999 |
| Km_3 | 43 | 2.52 | 0.12 | 44.54 | 43.50 | 0.9999 |
| 平均 | 43 | 2.51 | 0.12 | 43.98 | 43.50 | 1.0000 |
操作分类单元(OTU)是系统发生学或群体遗传学研究中人为设置的分类单元标志, 以便进行比对分析(Blaxter et al, 2005; Ramanan et al, 2016)。表 3表示的是米氏凯伦藻(Km02)共培养菌群样品OTU代表性序列相似性在97%以上的序列数统计表, OTU聚类得到样品中各个OTU丰度及其在样品中序列数。序列数为米氏凯伦藻Km02株系43个OTU的3个平行实验样品结果的总和(表 3)。
| 操作分类单元OTU | 分类学地位 | 序列数* |
| OTU31 | Bacteria, Bacteroidetes, Bacteroidetes_Incertae_Sedis, Order_Ⅲ, Unknown_Family_Order_Ⅲ, Balneola | 24234 |
| OTU5 | Bacteria, Bacteroidetes, Bacteroidetes_Incertae_Sedis, Order_Ⅲ, Unknown_Family_Order_Ⅲ, Gracilimonas | 63 |
| OTU28 | Bacteria, Bacteroidetes, Cytophagia, Cytophagales, Flammeovirgaceae, Marinoscillum | 665 |
| OTU33 | Bacteria, Bacteroidetes, Cytophagia, Cytophagales, Flammeovirgaceae, Marinoscillum | 372 |
| OTU35 | Bacteria, Bacteroidetes, Cytophagia, Cytophagales, Flammeovirgaceae, Reichenbachiella | 6833 |
| OTU25 | Bacteria, Bacteroidetes, Cytophagia, Cytophagales, Flammeovirgaceae, Reichenbachiella | 241 |
| OTU37 | Bacteria, Bacteroidetes, Flavobacteriia, Flavobacteriales, Cryomorphaceae, norank_Cryomorphaceae | 43 |
| OTU36 | Bacteria, Bacteroidetes, Flavobacteriia, Flavobacteriales, Flavobacteriaceae, Mesoflavibacter | 6465 |
| OTU38 | Bacteria, Fibrobacteres, Chitinivibrionia | 1805 |
| OTU43 | Bacteria, Proteobacteria, AEGEAN-245 | 64 |
| OTU16 | Bacteria, Proteobacteria, Alphaproteobacteria, Caulobacterales, Hyphomonadaceae, Henriciella | 239 |
| OTU12 | Bacteria, Proteobacteria, Alphaproteobacteria, Kordiimonadales, Kordiimonadaceae, Kordiimonas | 30 |
| OTU22 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhizobiales, Phyllobacteriaceae, Mesorhizobium | 109 |
| OTU17 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhizobiales, Phyllobacteriaceae, Nitratireductor | 60 |
| OTU4 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhizobiales | 115 |
| OTU29 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodobacterales, Rhodobacteraceae, Labrenzia | 655 |
| OTU15 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodobacterales, Rhodobacteraceae, Roseovarius | 217 |
| OTU26 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodobacterales, Rhodobacteraceae, unclassified_Rhodobacteraceae | 15247 |
| OTU32 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodobacterales, Rhodobacteraceae unclassified_Rhodobacteraceae | 468 |
| OTU19 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodospirillales, Rhodospirillaceae, Magnetospira | 19 |
| OTU23 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodospirillales, Rhodospirillaceae, Nisaea | 2519 |
| OTU6 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodospirillales, Rhodospirillaceae | 11 |
| OTU1 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodospirillales, Rhodospirillaceae | 6 |
| OTU27 | Bacteria, Proteobacteria, Alphaproteobacteria, Rhodospirillales, Rhodospirillaceae, Thalassospira | 7454 |
| OTU18 | Bacteria, Proteobacteria, Alphaproteobacteria, Sphingomonadales, Erythrobacteraceae, Erythrobacter | 240 |
| OTU20 | Bacteria, Proteobacteria, Alphaproteobacteria | 96 |
| OTU14 | Bacteria, Proteobacteria, Betaproteobacteria, Burkholderiales, Burkholderiaceae, Limnobacter | 896 |
| OTU13 | Bacteria, Proteobacteria, Deltaproteobacteria, Myxococcales, Sandaracinaceae, Sandaracinus | 436 |
| OTU41 | Bacteria, Proteobacteria, Gammaproteobacteria, Alteromonadales, Alteromonadaceae, Alteromonas | 10613 |
| OTU10 | Bacteria, Proteobacteria, Gammaproteobacteria, Alteromonadales, Alteromonadaceae, Marinobacter | 2331 |
| OTU3 | Bacteria, Proteobacteria, Gammaproteobacteria, Alteromonadales, Alteromonadaceae, Marinobacter | 20674 |
| OTU40 | Bacteria, Proteobacteria, Gammaproteobacteria, Cellvibrionales, Porticoccaceae, Porticoccus | 652 |
| OTU39 | Bacteria, Proteobacteria, Gammaproteobacteria, Cellvibrionales, Porticoccaceae, SAR92_clade | 74 |
| OTU42 | Bacteria, Proteobacteria, Gammaproteobacteria, Cellvibrionales, Spongiibacteraceae, Spongiibacter | 412 |
| OTU9 | Bacteria, Proteobacteria, Gammaproteobacteria, Oceanospirillales, Alcanivoracaceae, Alcanivorax | 2726 |
| OTU7 | Bacteria, Proteobacteria, Gammaproteobacteria, Oceanospirillales, Oceanospirillaceae, Bermanella | 4 |
| OTU11 | Bacteria, Proteobacteria, Gammaproteobacteria, Oceanospirillales, Oceanospirillaceae, Pseudohongiella | 362 |
| OTU34 | Bacteria, Proteobacteria, Gammaproteobacteria, PYR10d3, norank_PYR10d3, norank_PYR10d3 | 412 |
| OTU8 | Bacteria, Proteobacteria, Gammaproteobacteria, Thiotrichales, Piscirickettsiaceae, Methylophaga | 9761 |
| OTU2 | Bacteria, Proteobacteria, Gammaproteobacteria, Thiotrichales, Thiotrichaceae, Thiothrix | 17 |
| OTU24 | Bacteria, Proteobacteria, Proteobacteria Incertae Sedis | 1508 |
| OTU21 | Bacteria | 249 |
| OTU30 | Bacteria | 113 |
| 注: *序列数为三个平行样品的总和 | ||
采用RDP classifier贝叶斯算法对高通量测序所获得的Km02株系共培养菌群结果, 以97%相似水平OTU代表序列进行分类学分析, 在不同的分类水平上统计分析样品微生物群落组成。结果表明, 米氏凯伦藻Km02株系含有的共培养菌群分布在4个门(Phylum), 11个纲(Class), 21个目(Order), 26个科(Family), 37个属(Genus), 40个种(Species), 此外含有3个OTU为未知门的种。从属水平比对结果来看(图 2), 其中优势菌属有8个(> 5%), 分别为Balneola (20.28%)、Marinobacter (19.25%)、Rhodobacteraceae科下的未知种属(13.15%)、Alteromonas (8.88%)、Methylophaga (8.17%)、Thalassospira (6.24%)、Reichenbachiella (5.92%)及Mesoflavibacter (5.41%)。结果表明米氏凯伦藻Km02株系含有生物多样性较高的共培养菌群, 且具有探寻未知海洋细菌的潜力, 如OTU20, OTU21, OTU26, OTU30等。
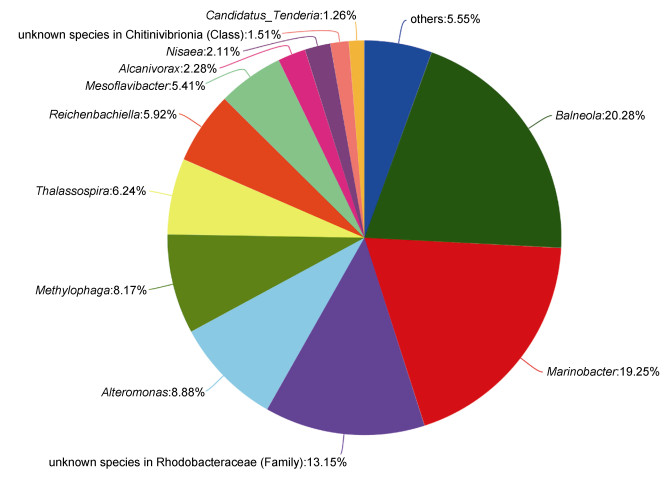 |
| 图 2 米氏凯伦藻(Km02)共培养细菌菌群属水平分布图 Fig. 2 Co-cultured bacterial community distribution of K. mikimotoi (Km02) at the genus level 注:小于0.01%的分类群被合并到其他(others) |
从分子进化的角度, 揭示米氏凯伦藻(Km02)共培养细菌菌群属分类在演化过程中的亲缘顺序关系。并对照分析了各优势属的测序read数量与标识各个纲下米氏凯伦藻(Km02)共培养细菌的所在位置。通过图 3我们可以看出, Alphaproteobacteria和Gammaproteobacteria两个纲的细菌不论在多样性还是相对丰度上都占有优势。其中, Gammaproteobacteria中的未定种属norank_ PYR10d3与Pseudohongiella属较接近, Alphaproteobacteria中的未定种属unclassified_ Rhodobacteraceae和norank_PS1_clade与Roseovarius有着较近的亲缘关系。其中, unclassified_ Rhodobacteraceae由于其在共培养群落中有着较大相对丰度与优势, 该OTU所代表的细菌不论是分类学还是生态意义, 都是今后进行米氏凯伦藻(Km02)共培养细菌分类培养实验中重点研究对象之一。
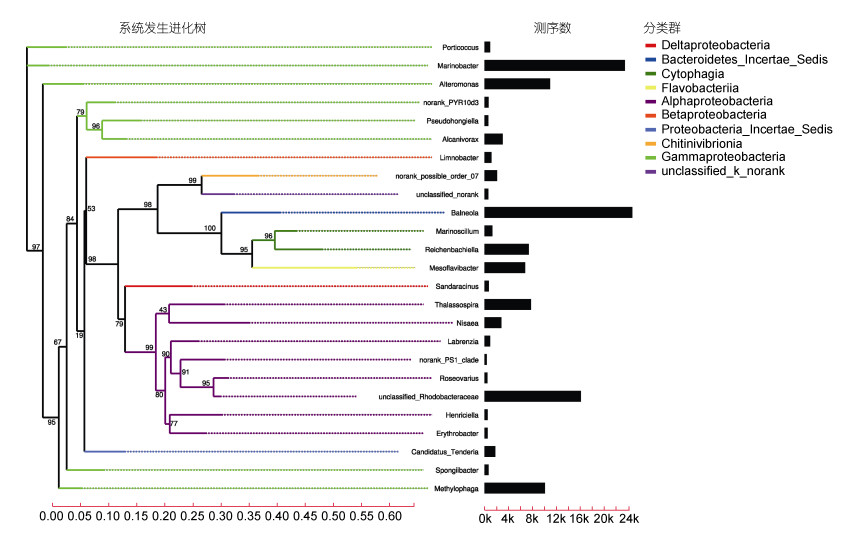 |
| 图 3 米氏凯伦藻Km02共培养细菌优势种的系统进化树及其优势度 Fig. 3 Phylogenetic tree of K. mikimotoi (Km02) co-cultured bacterial community and theirs reads number 注:左边为米氏凯伦藻Km02株共培养细菌相对丰度前25种的系统发生进化树, 进化树中每条树枝代表米氏凯伦藻Km02株的中的一种共培养细菌, 树枝长度为两个物种间的进化距离, 即共培养细菌的差异程度; 右边柱状图显示属于不同共培养细菌的Reads数量 |
尽管过去研究赤潮发生的机理主要集中在理化因素的影响, 而越来越多的证据表明仅凭借温度与营养盐等环境因素并不能解释大部分赤潮现象, 因此生物因素受到越来越多的关注, 近来藻际细菌被认为可能在藻类快速生长或赤潮生消过程中发挥着重要作用(Park et al, 2015, 2017)。有研究表明海洋藻际细菌群落和海洋中的游离细菌群落在系统发育上有着明显的区别, 说明两种不同生存环境下的细菌种群结构组成由不同的选择性压力所影响(Grossart et al, 2005; Rooney-Varga et al, 2005)。此外, 海洋微藻与其共生的细菌在长期自然进化的过程中, 形成复杂的共生体系, 藻菌之间不仅有能量与代谢产物的相互利用, 而且有遗传物质在不同物种之间进行基因水平转移(Horizontal gene transfer)甚至是基因融合(Gene fusion) (Hold et al, 2001; Green et al, 2004; Keeling et al, 2008)。高通量测序对微藻培养株共培养菌进行种群分析, 避免了利用传统的培养法所导致的一些海洋细菌特别是共生菌不可培养性的缺陷与技术瓶颈。为此, 藻际细菌的种群与其共生或寄生藻株有着息息相关的联系, 特别是在目前很多时候通过物理化学因素已经无法判断有毒有害赤潮发生的机制与产毒机制的背景下, 通过高通量测序技术等先进实验方法研究有害赤潮藻的藻际细菌显得尤为重要, 这也会为将来通过生物因子揭示对赤潮的暴发与产毒特性机理的研究提供了研究方向与方法。
4 结论本研究利用高通量测序技术, 首次阐明了我国米氏凯伦藻内共培养菌群的种类、丰度、多样性与种群进化关系。结果表明米氏凯伦藻Km02株含有生物多样性较高的共培养菌群, 且具有探寻未知海洋细菌的潜力, 并且为未来通过与不同米氏凯伦藻株的共培养菌群以及不同生长条件下培养的米氏凯伦藻所分析得到细菌群落奠定了基础, 未来通过深度挖掘细菌种群变动与藻株的生长促进与抑制等变化, 将对米氏凯伦藻赤潮的暴发与产毒的生物机制提供有力的证据。
王朝晖, 尹伊伟, 齐雨藻, 等. 2001. 珠海桂山岛米氏裸甲藻赤潮对鱼鳃损伤的病理学组织观察. 海洋学报, 23(1): 133-138 DOI:10.3321/j.issn:0253-4193.2001.01.018 |
刘志国, 王金辉, 蔡芃, 等. 2014. 米氏凯伦藻分布及其引发赤潮的发生规律研究. 国土与自然资源研究, (1): 38-41 DOI:10.3969/j.issn.1003-7853.2014.01.015 |
刘桂英, 葛坤, 宋伦, 等. 2015. 米氏凯伦藻的研究进展. 海洋科学, 39(9): 117-122 |
许翠娅, 黄美珍, 杜琦. 2010. 福建沿岸海域主要赤潮生物的生态学特征. 台湾海峡, 29(3): 434-441 DOI:10.3969/J.ISSN.1000-8160.2010.03.020 |
张水浸, 许昆灿, 陈其焕, 等. 1988. 厦门西港区一次赤潮的观测. 海洋学报, 10(5): 602-608 |
香港渔农自然护理署, 2013.香港有害海洋微藻
|
曾江宁, 曾淦宁, 黄韦艮, 等. 2004. 赤潮影响因素研究进展. 东海海洋, 22(2): 40-47 DOI:10.3969/j.issn.1001-909X.2004.02.007 |
Anderson D M, Burkholder J M, Cochlan W P et al, 2008. Harmful algal blooms and eutrophication: examining linkages from selected coastal regions of the United States. Harmful Algae, 8(1): 39-53 DOI:10.1016/j.hal.2008.08.017 |
Azam F, Fenchel T, Field J G et al, 1983. The ecological role of water-column microbes in the sea. Marine Ecology Progress Series, 10(3): 257-263 |
Blaxter M, Mann J, Chapman T et al, 2005. Defining operational taxonomic units using DNA barcode data. Philosophical Transactions of the Royal Society B: Biological Sciences, 360(1462): 1935-1943 DOI:10.1098/rstb.2005.1725 |
Bolch C J S, Subramanian T A, Green D H, 2011. The toxic dinoflagellate Gymnodinium catenatum (dinophyceae) requires marine bacteria for growth. Journal of Phycology, 47(5): 1009-1022 DOI:10.1111/j.1529-8817.2011.01043.x |
Celikkol-Aydin S, Gaylarde C C, Lee T et al, 2016. 16S rRNA gene profiling of planktonic and biofilm microbial populations in the Gulf of Guinea using Illumina NGS. Marine Environmental Research, 122: 105-112 DOI:10.1016/j.marenvres.2016.10.001 |
Croft M T, Lawrence A D, Raux-Deery E et al, 2005. Algae acquire vitamin B12 through a symbiotic relationship with bacteria. Nature, 438(7064): 90-93 DOI:10.1038/nature04056 |
Dai X F, Lu D D, Guan W B et al, 2014. Newly recorded Karlodinium veneficum dinoflagellate blooms in stratified water of the East China Sea. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 101: 237-243 DOI:10.1016/j.dsr2.2013.01.015 |
Green D H, Llewellyn L E, Negri A P et al, 2004. Phylogenetic and functional diversity of the cultivable bacterial community associated with the paralytic shellfish poisoning dinoflagellate Gymnodinium catenatum. FEMS Microbiology Ecology, 47(3): 345-357 DOI:10.1016/S0168-6496(03)00298-8 |
Grossart H P, Levold F, Allgaier M et al, 2005. Marine diatom species harbour distinct bacterial communities. Environmental Microbiology, 7(6): 860-873 DOI:10.1111/emi.2005.7.issue-6 |
Guidi F, Pezzolesi L, Vanucci S, 2018. Microbial dynamics during harmful dinoflagellate Ostreopsiscf.ovata growth: bacterial succession and viral abundance pattern. MicrobiologyOpen, 7(4): e00584 |
Guillard R R L, Ryther J H, 1962. Studies of marine planktonic diatoms: Ⅰ.Cyclotella nana Hustedt, and Detonula confervacea (Cleve) Gran. Canadian Journal of Microbiology, 8(2): 229-239 |
Heisler J, Glibert P M, Burkholder J M et al, 2008. Eutrophication and harmful algal blooms: a scientific consensus. Harmful Algae, 8(1): 3-13 DOI:10.1016/j.hal.2008.08.006 |
Hold G L, Smith E A, Rappé M S et al, 2001. Characterisation of bacterial communities associated with toxic and non-toxic dinoflagellates: Alexandrium spp.and Scrippsiella trochoidea. FEMS Microbiology Ecology, 37(2): 161-173 DOI:10.1111/fem.2001.37.issue-2 |
Keeling P J, Palmer J D, 2008. Horizontal gene transfer in eukaryotic evolution. Nature Reviews Genetics, 9(8): 605-618 DOI:10.1038/nrg2386 |
Kim J H, Wang P B, Park B S et al, 2018. Revealing the distinct habitat ranges and hybrid zone of genetic sub-populations within Pseudo-nitzschia pungens (Bacillariophyceae) in the West Pacific area. Harmful Algae, 73: 72-83 DOI:10.1016/j.hal.2018.01.007 |
Lu D D, Goebel J, 2001. Five red tide species in genus Prorocentrum including the description of Prorocentrum donghaiense Lu sp.nov. from the East China Sea. Chinese Journal of Oceanology and Limnology, 19(4): 337-344 DOI:10.1007/BF02850738 |
Park B S, Kim J H, Kim J H et al, 2015. Dynamics of bacterial community structure during blooms of Cochlodinium polykrikoides (Gymnodiniales, Dinophyceae) in Korean coastal waters. Harmful Algae, 48: 44-54 DOI:10.1016/j.hal.2015.07.004 |
Park B S, Wang P B, Kim J H et al, 2014. Resolving the intra-specific succession within Cochlodinium polykrikoides populations in southern Korean coastal waters via use of quantitative PCR assays. Harmful Algae, 37: 133-141 DOI:10.1016/j.hal.2014.04.019 |
Park J, Park B S, Wang P B et al, 2017. Phycospheric native bacteria Pelagibaca bermudensis and Stappia sp.ameliorate biomass productivity of Tetraselmis striata (KCTC1432BP) in co-cultivation system through mutualistic interaction. Frontiers in Plant Science, 8: 289 |
Ramanan R, Kim B H, Cho D H et al, 2016. Algae–bacteria interactions: evolution, ecology and emerging applications. Biotechnology Advances, 34(1): 14-29 DOI:10.1016/j.biotechadv.2015.12.003 |
Rooney-Varga J N, Giewat M W, Savin M C et al, 2005. Links between phytoplankton and bacterial community dynamics in a coastal marine environment. Microbial Ecology, 49(1): 163-175 DOI:10.1007/s00248-003-1057-0 |
Sarmento H, Gasol J M, 2012. Use of phytoplankton‐derived dissolved organic carbon by different types of bacterioplankton. Environmental Microbiology, 14(9): 2348-2360 DOI:10.1111/emi.2012.14.issue-9 |
Wang P B, Joo J H, Park B S et al, 2017. Relationship between dissolved organic carbon and bacterial community in the coastal waters of Incheon, Korea. Oceanological and Hydrobiological Studies, 46(1): 50-61 |
 2019, Vol. 50
2019, Vol. 50


