中国海洋湖沼学会主办。
文章信息
- 苏金净, 蔡伟聪, 李桂玲, 李健, 曾珺, 刘静雯. 2019.
- SU Jin-Jing, CAI Wei-Cong, LI Gui-Ling, LI Jian, ZENG Jun, LIU Jing-Wen. 2019.
- 浮游植物metacaspase的分布、结构及其功能特性
- DISTRIBUTION, STRUCTURE, AND FUNCTION OF PHYTOPLANKTON METACASPASES
- 海洋与湖沼, 50(5): 1059-1069
- Oceanologia et Limnologia Sinica, 50(5): 1059-1069.
- http://dx.doi.org/10.11693/hyhz20190300056
文章历史
-
收稿日期:2019-03-16
收修改稿日期:2019-05-20
2. 福建省食品微生物与酶工程重点实验室 厦门 361021
2. Fujian Provincial Key Laboratory of Food Microbiology and Enzyme Engineering, Xiamen 361021, China
精氨酸/赖氨酸特异性半胱氨酸蛋白酶(Metacaspases)广泛存在于高等植物、浮游植物、霉菌、真菌以及细菌中(Bidle et al, 2004), 它们均含有类似动物caspase酶类保守的结构域及包含组氨酸和半胱氨酸的催化位点(Read et al, 2013), 在非生物(如营养、光照、渗透压等)和生物(病毒感染、化感作用等)胁迫诱导的浮游植物程序性细胞死亡(programmed cell death, PCD)过程中起着关键作用(Bidle, 2016)。目前, 浮游植物中已确定的metacaspases有三种类型, 即Ⅰ型、Ⅱ型及新近发现的Ⅲ型metacaspases, 与Ⅰ型或Ⅱ型metacaspases蛋白酶的结构域不同, Ⅲ型metacaspases中p10和p20的结构域进行了重新排序, 使p10位于p20结构域的前端(Coll et al, 2010; Wang et al, 2012; Choi et al, 2013)。除此之外, 浮游植物中还存在一种类metacaspase蛋白酶(metacaspase-like proteases, MCP), 其与细菌的MCP类似, 暗示真核生物metacaspases蛋白酶可能起源于细菌(Choi et al, 2013)。Metacaspases参与众多胁迫诱导的浮游植物PCD过程, 如营养胁迫(Bidle et al, 2008; 郭莉莎等, 2012; Lu et al, 2017)、病毒感染(Bidle et al, 2007; Bidle, 2016; Liu et al, 2018)等。多数物种具有多个编码metacaspase的基因, 每个基因的功能也不尽相同, 它们除了调控细胞死亡, 在细胞信号转导、各种酶活性及蛋白质修饰过程中也发挥着作用(Bidle, 2015)。另外, metacaspase和经典caspase的生化特征以及它们之间在功能上的相互关系也是一个备受关注和讨论的焦点。与经典caspase相比, metacaspase虽然参与PCD过程, 也保留了类似caspase的组氨酸-半胱氨酸的催化活性位点, 但是它们具有不同的底物特异性、metacaspase的酶活不被caspases抑制剂所抑制、metacaspase和caspase对底物的切割位点不同等特点(Carmona-Gutierrez et al, 2010), 说明metacaspase和caspase的功能存在一定的差异。
浮游植物metacaspase的结构和功能的研究起步较晚, 浮游植物中普遍存在的metacaspase与PCD过程中活性caspase之间的相关性尚缺乏充分的证据。本文基于近年来浮游植物metacaspase与PCD过程相关性研究报道以及在线相关数据库信息, 采用生物信息学方法分析浮游植物metacaspases核酸及氨基酸序列特征、蛋白结构、系统进化关系及潜在的磷酸化位点等, 并对该蛋白进行基因本体(Gene Ontology, GO)功能注释及蛋白互作分析, 了解metacaspase与caspase之间在功能上的相关性特点。为进一步深入研究浮游植物metacaspase的功能提供参考。
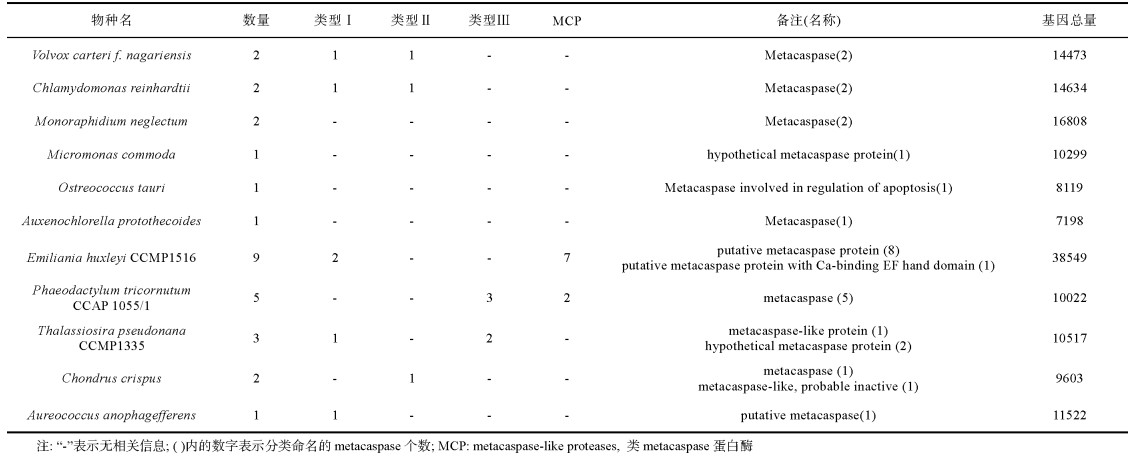
1 材料和方法 1.1 研究材料采用关键词“metacaspase”, 在NCBI数据库中搜索基因, 获得该数据库中所有metacaspases基因条目, 通过种属分类, 将所有属于浮游植物的metacaspases基因筛选出来并记录相关信息(基因登录号见附表 1)。
| 物种 | 缩写a | 基因ID | mRNA ID | 蛋白ID |
| Aureococcus anophagefferens | AaMC1 | 20221022 | XM_009042055.1 | XP_009040303.1 |
| Auxenochlorella protothecoides | ApMC1 | 23614527 | XM_011399004.1 | XP_011397306.1 |
| Chlamydomonas reinhardtii | CrMC1 | 5717424 | XM_001691774.1 | XP_001691826.1 |
| CrMC2 | 5722640 | XM_001696904.1 | XP_001696956.1 | |
| Chondrus crispus | CcMC1 | 17321622 | XM_005713836.1 | XP_005713893.1 |
| CcMC2 | 17324901 | XM_005717127.1 | XP_005717184.1 | |
| Emiliania huxleyi CCMP1516 | EhMC1 | 17284475 | XM_005791576.1 | XP_005791633.1 |
| EhMC2 | 17283241 | XM_005790343.1 | XP_005790400.1 | |
| EhMC3 | 17277722 | XM_005784825.1 | XP_005784882.1 | |
| EhMC4 | 17277488 | XM_005784588.1 | XP_005784645.1 | |
| EhMC5 | 17269785 | XM_005776613.1 | XP_005776670.1 | |
| EhMC6 | 17267081 | XM_005773908.1 | XP_005773965.1 | |
| EhMC7 | 17256866 | XM_005763016.1 | XP_005763073.1 | |
| EhMC8 | 17253171 | XM_005759676.1 | XP_005759733.1 | |
| EhMC9 | 17251088 | XM_005757309.1 | XP_005757366.1 | |
| Micromonas commoda | McMC1 | 8242492 | XM_002501537.1 | XP_002501583.1 |
| Monoraphidium neglectum | MnMC1 | 25726426 | XM_014051203.1 | XP_013906657.1 |
| MnMC2 | 25726427 | XM_014051204.1 | XP_013906658.1 | |
| Ostreococcus tauri | OtMC1 | 9831219 | XM_003083404.1 | XP_003083452.1 |
| Phaeodactylum tricornutum CCAP 1055/1 | PtMC1 | 7197248 | XM_002178072.1 | XP_002178108.1 |
| PtMC2 | 7200976 | XM_002180039.1 | XP_002180075.1 | |
| PtMC3 | 7203298 | XM_002182482.1 | XP_002182518.1 | |
| PtMC4 | 7203526 | XM_002182516.1 | XP_002182552.1 | |
| PtMC5 | 7203528 | XM_002182670.1 | XP_002182706.1 | |
| Thalassiosira pseudonana CCMP1335 | TpMC1 | 7449711 | XM_002290103.1 | XP_002290139.1 |
| TpMC2 | 7450918 | XM_002295318.1 | XP_002295354.1 | |
| TpMC3 | 7451860 | XM_002297194.1 | XP_002297230.1 | |
| Volvox carteri f. nagariensis | VcMC1 | 9618094 | XM_002946692.1 | XP_002946738.1 |
| VcMC2 | 9621258 | XM_002948025.1 | XP_002948071.1 | |
| a: MC, Metacaspase | ||||
针对NCBI数据库中所有浮游植物的metacaspases基因, 利用MEGA7软件进行多重序列比对, 选择邻接法(N-J)构建系统发育树。
1.2.2 Metacaspase的亚细胞定位选取海洋球石藻Emiliania huxleyi CCMP1516的9条metacaspases (EhMCs)蛋白序列, 利用TargetP (http://www.cbs.dtu.dk/services/TargetP/)分析其含有的各类信号肽序列。利用NLStradamus (http://www.moseslab.csb.utoronto.ca/NLStradamus/)和NetNES (http://www.cbs.dtu.dk/services/NetNES/)对9条EhMCs蛋白进行核定位信号和核输出信号预测。
1.2.3 Metacaspase空间结构分析以EhMC1为例, 在蛋白质数据库(protein data bank databases, PDB)中筛选与其序列匹配度最高的物种模板, 分别利用SWISS MODEL (https://swissmodel.expasy.org/)和Phyre2 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)构建蛋白质三级结构模型。
1.2.4 Metacaspase功能及蛋白质互作分析利用NCBI数据库收集所有浮游植物metacaspases蛋白的注释信息, 包括细胞成分(cellular component)、分子功能(molecular function)和生物过程(biological process)等, 为后续分析工作提供参考。基于String数据库(https://string-db.org/), 利用基因邻接(gene neighborhood)、基因融合(gene fusions)、共生关系(co-occurrence)、基因共表达(gene co-expression)、蛋白质互作实验(experimentally determined)、数据库记录(databases)以及文本挖掘(textmining)等方法分析与浮游植物metacaspases相互作用的蛋白。
1.2.5 Metacaspase蛋白质磷酸化位点预测蛋白磷酸化是最常见、最重要的一种翻译后修饰, 参与并调节机体的多种生命活动, 如信号转导、基因表达、细胞周期及细胞死亡等, 并对物种的遗传多样性具有重要贡献。利用Scansite在线工具(http://scansite3.mit.edu/#proteinScan)预测浮游植物metacaspases潜在的磷酸化修饰位点, 将有助于了解单细胞浮游植物PCD信号通路的调控方式及浮游植物多样性的进化轨迹。
2 结果与讨论 2.1 Metacaspase基因在浮游植物中的分布通过在NCBI数据库中对浮游植物的metacaspase基因进行统计, 确定浮游植物一共11个物种的29条metacaspases基因, 统计结果见表 1。与已完成基因组测序的其他物种相比, 浮游植物中metacaspase基因的数量分布并不均匀, 如海洋球石藻(E. huxleyi CCMP1516)中含有9条metacaspases基因, 而绿藻中的莱茵衣藻(Chlamydomonas reinhardtii)只含有2条, 同属于绿藻的金牛微球藻(Ostreococcus tauri)、原壳小球藻(Auxenochlorella protothecoides)及微胞藻(Micromonas commoda)则各只有1条。浮游植物metacaspases基因数在各物种间相差较大, 主要区别在于MCP数量的不同(表 1)。真核生物metacaspases起源于细菌两种类型的metacaspases, 即type Ⅰ (存在于β-, δ-变形杆菌门、放线菌和硝化螺旋菌中)和MCP (存在于α-变形杆菌门)(Choi et al, 2013)。进化过程中通过基因水平转移, 大多数浮游植物获得了MCP, 有些浮游植物MCP的p10结构域具有完整的底物识别序列, 但有些浮游植物MCP缺失了特异性识别底物的序列, 这种“冗余”蛋白逐渐丢失导致metacaspase数量减少(Choi et al, 2013)。
 |
利用MEGA7软件对浮游植物中29条及拟南芥(Arabidopsis thaliana)中1条metacaspases的氨基酸序列进行多重比对, metacaspases的活性位点用红色箭头标记(图 1)。
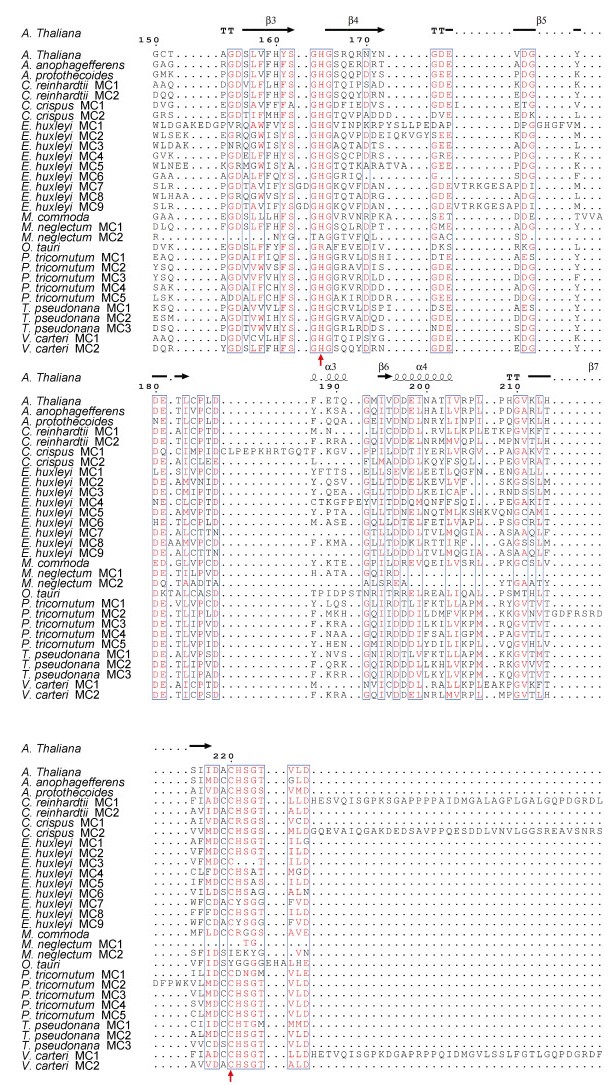 |
| 图 1 浮游植物与拟南芥的metacaspases氨基酸序列比对 Fig. 1 Multiple alignment of the deduced amino acid sequence of metacaspases in phytoplankton |
陆生植物metacaspase的活性位点一般是位于p20的164位组氨酸(His)和220位的半胱氨酸(Cys), 浮游植物与陆生植物拟南芥metacaspase 1位于第164位和第220位活性位点的氨基酸组成几乎完全相同, 表明浮游植物和陆生植物metacaspase基因存在一定的亲缘关系。单针藻(Monoraphidium neglectum) metacaspase 2和金牛微球藻metacaspase缺失活性位点的组氨酸和半胱氨酸, 在164位分别被丙氨酸(Ala)和精氨酸(Arg)所取代, 在220位则分别被异亮氨酸(Ile)和酪氨酸(Tyr)取代, 单针藻metacaspase 1在220位缺失半胱氨酸。组氨酸属于极性带正电荷的R基氨基酸, 丙氨酸属于脂肪族类非极性带R基氨基酸, 精氨酸属于极性带正电荷且有胍基的碱性氨基酸, 半胱氨酸是不带电荷的极性R基氨基酸, 异亮氨酸属于脂肪族类带R基氨基酸, 酪氨酸属于不带电荷的极性R基氨基酸, 可以看出取代的氨基酸在理化性质上与原来的氨基酸相比存在显著差异, 因此推测单针藻metacaspase 2和金牛微球藻metacaspase可能不参与PCD过程, 而可能具有其它功能。
系统发育树显示, 来自同一物种的不同metacaspase基因之间具有极强的亲缘关系, 如三角褐指藻(Phaeodactylum tricornutum)及海洋球石藻, 但莱茵衣藻(C. reinhardtii)虽然含有多条metacaspases基因, 这些metacaspases基因相互之间似乎并没有太多的亲缘关系(图 2)。已被鉴定的Ⅱ型莱茵衣藻metacaspase 1、团藻(Volvox carteri)metacaspase 1分别被分到同一个分枝, 说明相同类型的metacaspase尽管来自不同物种, 却有着极强的同源性。另外, 9条EhMCs被分成两类, 其中EhMC6独立为一类, 其他8条EhMCs聚为一类。
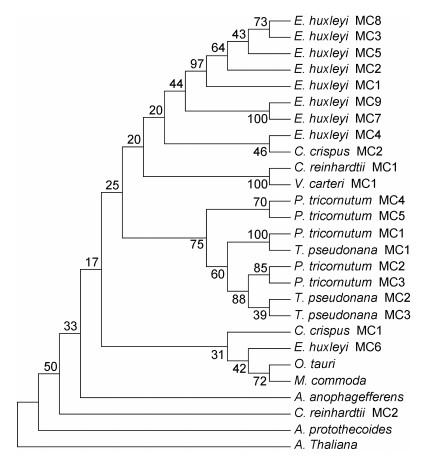 |
| 图 2 浮游植物Metacaspase系统发育树 Fig. 2 Phylogenetic tree of metacaspases in phytoplankton |
通过对9条EhMCs分泌信号肽和线粒体转运肽的预测分析, 发现它们氨基酸序列的SP值均低于0.5, 说明这些EhMCs的N端不具有分泌信号肽, 不会通过内膜系统分泌到细胞外, 可能为胞内蛋白。而EhMC5和EhMC6的mTP值均大于0.5, 极有可能进入线粒体中发挥作用。另外, EhMC8为具有核定位信号序列和核输出序列的核质蛋白。如在挪威云杉胚柄细胞退化诱导的PCD过程中, 一种Ⅱ型metacaspase从细胞质转运至细胞核, 促进染色体的降解, 与动物细胞中caspase-3, 6的作用类似(Suarez et al, 2004)。
2.4 Metacaspase蛋白质空间结构分析在PDB数据库中搜索到与EhMC2模板匹配度最高为33.05%, 是酿酒酵母(Saccharomyces cerevisiae)的metacaspase (Yca1), 其PDB数据库编号为: 4f6o.1。利用SWISS-MODEL对EhMC2进行同源建模并用Modeller软件对该模型进行评估。Modeller的平均评分小于−0.02时可信度较可靠。虽然EhMC2与模板的匹配度最高, 但模拟结果的评分值均大于−0.02, 可信度较低。因此, 我们进一步采用Phyre2分别对EhMC1和EhMC2进行精确的折叠识别建模, 并采用Modeller对模型结果进行评估, 结果显示该模型对EhMC1模拟结果如图 3a, 且平均评分值小于−0.02(图 3b), 可见在与模板蛋白同源性较低的情况下, 利用折叠识别建模的结果更加准确可靠。预测的EhMC1的三级结构显示, 具有6个α螺旋、8个β折叠, p20结构域中Cys192残基处于酶活性中心, 与His147构成水解二联体, 形成具有催化功能的水解酶折叠中心(图 3a)。实验结果显示, 酵母的活性Yca1具有同源二聚体特点, Yca1的p20结构域在Arg72和Lys86两处被切割成两个较小的36kDa片段并二聚化, 而钙离子能够特异性地增强Yca1的自剪切加工(Wong et al, 2012)。因此推测, 自剪切加工和二聚化可能也是EhMC1产生活性形式的关键步骤。
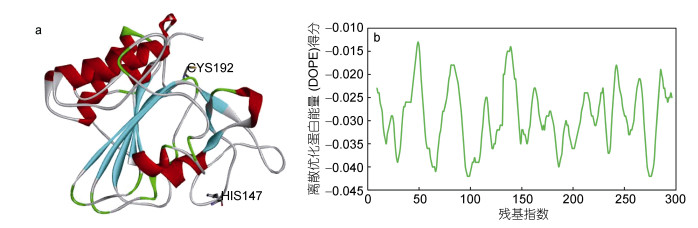 |
| 图 3 EhMC1蛋白三维结构预测及评价 Fig. 3 Three-dimensional structure and the evaluation of EhMC1 注: a.折叠识别建模三级结构; b.折叠识别建模评价 |
另外, 植物metacaspases中的某些氨基酸可以被S-亚硝基化, 从而导致metacaspase降解失活, 如拟南芥AtMC9的活性位点处的Cys-147在植物体内可被S-亚硝基化, 抑制其自我加工及降解底物活性(Belenghi et al, 2007)。S-亚硝基化是一氧化氮(NO)以共价键方式与蛋白质特定位置上的半胱氨酸(Cys)残基相连, 从而影响被修饰蛋白质的活性, 是一种翻译后基因表达调控方式。研究表明, 病毒感染诱导海洋球石藻细胞产生和释放NO并启动宿主细胞PCD过程(Rosenwasser et al, 2016)。病毒诱导球石藻PCD的精确机制尚不清楚, 不过由此我们可以推测S-亚硝基化这种翻译后修饰方式可能在病毒诱导的球石藻PCD过程中调控metacaspase的活性。
2.5 Metacaspase蛋白质功能分析Metacaspases与caspase家族半胱氨酸蛋白酶具有远缘关系, 且与植物和低等真核细胞中的PCD过程相关。Metacaspase是精氨酸/赖氨酸特异性半胱氨酸蛋白酶类, 其通过自剪切加工或二聚化激活(McLuskey et al, 2012)。在UniProt蛋白数据库中11个浮游植物物种的29个metacaspases都具有半胱氨酸蛋白酶活性, 并且在NCBI数据库的电子注释中发现EhMC4具有钙离子结合位点。许多生化证据表明, 高等植物Ⅰ型和Ⅱ型metacaspase的催化活性依赖于钙离子的存在(Du et al, 2011; Watanabe et al, 2011; Piszczek et al, 2012; Machado et al, 2013)。Choi等人(2013)通过结构预测分析显示, 隐藻(Cryptophyta, Guillardia theta)具有Ⅲ型metacaspase(GtMC2)(Choi et al, 2013), 随后的实验结果证实其具有蛋白内切酶活性, 并优先切割精氨酸残基, 且GtMC2的活性依赖于钙离子浓度(Klemenčič et al, 2018)。蓝藻(Microcystis aeruginosa)细胞具有缺乏p10结构域的MCP(MaOC1), 其依赖于钙离子并以二聚化形式激活, 即MaOC1通过切割Arg219残基后活化, 形成分子量为55kDa和25kDa的蛋白亚基(Klemenčič et al, 2015)。目前, 浮游植物的metacaspase相关实验数据较少, 基因组及蛋白注释也不完整, 它们在PCD过程中所处的位置尚不十分清晰。
利用String数据库(https://string-db.org/)分析推测与浮游植物metacaspase互作的蛋白质, 在中等条件下(score等于0.4)选取评分大于0.4的蛋白作为分析对象。结果发现, 莱茵衣藻、三角褐指藻及假微型海链藻(Thalassiosira pseudonana)具有蛋白互作相关数据, 且绝大多数互作蛋白都定位于胞质内或细胞膜上(表 2)。这些互作蛋白可能通过调控诸如依赖于钙离子的半胱氨酸蛋白酶、丝氨酸内肽酶及酰基转移酶的活性参与胁迫应答、蛋白水解、细胞PCD及自噬等生物学过程。
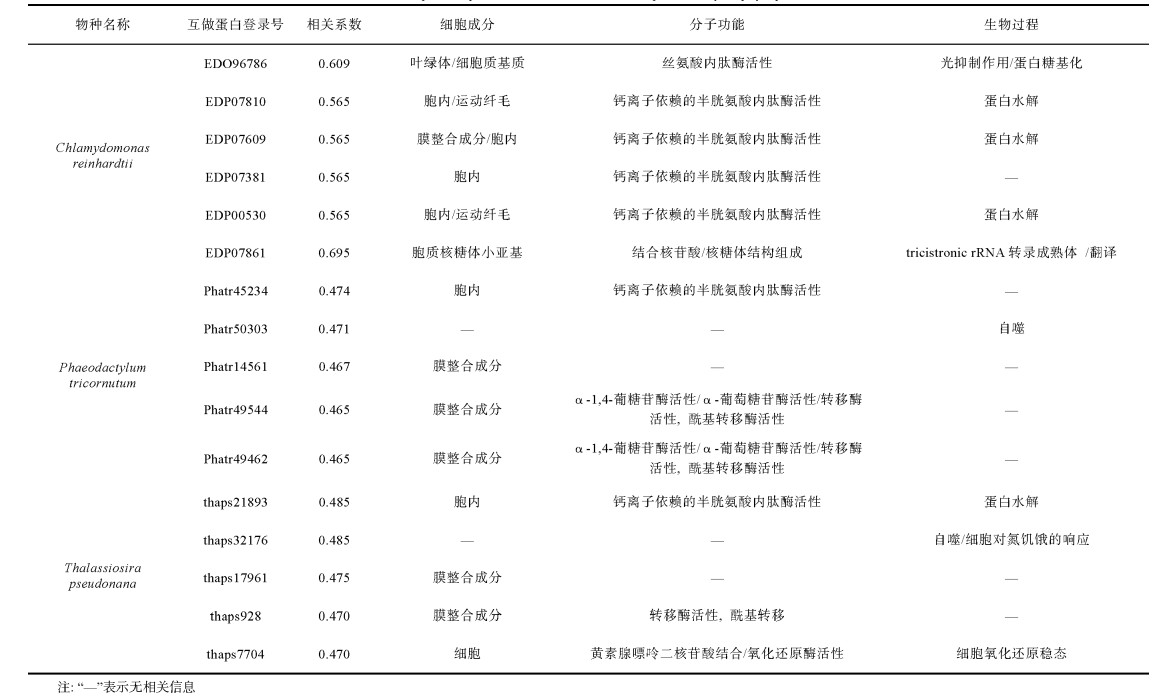 |
选择模序程序(Motif Scan)并设置“high”严谨度(百分位分数设置为0.1%), 对除EhMCs外20条metacaspases进行磷酸化修饰分析, 结果显示9个metacaspases可能被特定蛋白激酶磷酸化, 总共含有20个潜在的磷酸化位点(表 3), 且一个蛋白含有一个或多个磷酸化位点。如莱茵衣藻metacaspase (XP_ 001696956)有3个磷酸化结构域, 其中两个是Abl Kinase, 位点为Y73和Y131;另一个是PLK1 Kinase, 位点为T271;其余的物种也检测到磷酸化位点。
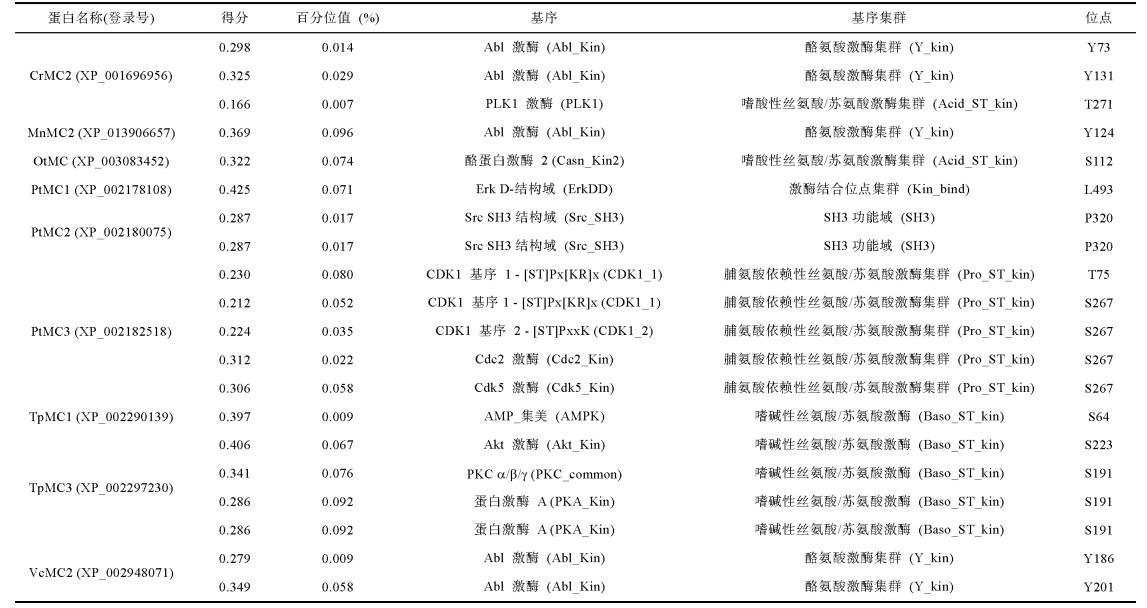 |
许多caspase的活性可以通过蛋白磷酸化直接调节(Dix et al, 2012)。Martins等人(1997)首次报道在依托泊苷(etoposide)诱导的细胞PCD过程中, HL-60细胞的胞浆中存在磷酸化形式的活性caspase, 并证实至少三种caspase由激酶磷酸化激活, 且caspase-3中含有多个潜在的磷酸化位点。有关浮游植物metacapase的磷酸化研究鲜有报道。Liu等(2018)发现海洋球石藻9条EhMCs的一级结构中均存在丝氨酸、苏氨酸和酪氨酸等多个潜在的磷酸化修饰位点, 并通过实验首次证实了病毒诱导球石藻PCD过程中伴随着磷酸化metacaspase的高表达(Liu et al, 2018)。另外, 海洋球石藻E. huxleiy磷脂酶C (PLC) (EhPLC)的一级结构中也存在多样性的磷酸化修饰, PLC作为一种重要的细胞信号分子, 是G蛋白信号转导过程中的重要调节因子之一, 在对环境胁迫应答及病原菌致病过程中发挥重要作用(郭强强等, 2018)。可见, 蛋白质的磷酸化与去磷酸化分子开关可能在调控浮游植物应答环境胁迫过程中起着关键作用。
3 结论本文利用生物信息学方法系统地分析了浮游植物PCD特征蛋白酶metacaspases的分布、结构特点及功能特性, 发现浮游植物该蛋白类型多样, 虽然它们的一级结构存在明显差异, 但活性位点的氨基酸基序具有高度保守性, 且蛋白三级结构与酵母的metacaspase高度相似, 暗示它们可能具有类似的功能。与浮游植物metacaspase相互作用的蛋白大部分具有依赖于钙离子的半胱氨酸蛋白酶、丝氨酸内肽酶及酰基转移酶的功能。浮游植物metacaspases存在多个潜在的磷酸化修饰位点, 暗示磷酸化修饰对该酶的活性功能具有一定的调控作用。
郭莉莎, 章军, 吴娟, 等. 2012. 黑暗限气条件下铜绿微囊藻细胞死亡的形态结构和生理生化变化. 微生物学报, 52(2): 228-235 |
郭强强, 蔡伟聪, 李桂玲, 等. 2018. 海洋球石藻(Emiliania huxleyi)磷脂酶C的结构特点及系统进化分析. 海洋与湖沼, 49(1): 106-116 |
Belenghi B, Romero-Puertas M C, Vercammen D et al, 2007. Metacaspase activity of Arabidopsis thaliana is regulated by S-nitrosylation of a critical cysteine residue. Journal of Biological Chemistry, 282(2): 1352-1358 DOI:10.1074/jbc.M608931200 |
Bidle K D, 2015. The molecular ecophysiology of programmed cell death in marine phytoplankton. Annual Review of Marine Science, 7: 341-375 DOI:10.1146/annurev-marine-010213-135014 |
Bidle K D, 2016. Programmed cell death in unicellular phytoplankton. Current Biology, 26(13): R594-R607 DOI:10.1016/j.cub.2016.05.056 |
Bidle K D, Bender S J, 2008. Iron starvation and culture age activate metacaspases and programmed cell death in the marine diatom Thalassiosira pseudonana. Eukaryotic Cell, 7(2): 223-236 DOI:10.1128/EC.00296-07 |
Bidle K D, Falkowski P G, 2004. Cell death in planktonic, photosynthetic microorganisms. Nature Reviews Microbiology, 2(8): 643-655 DOI:10.1038/nrmicro956 |
Bidle K D, Haramaty L, Barcelos E Ramos J et al, 2007. Viral activation and recruitment of metacaspases in the unicellular coccolithophore, Emiliania huxleyi. Proceedings of the National Academy of Sciences of the United States of America, 104(14): 6049-6054 DOI:10.1073/pnas.0701240104 |
Carmona-Gutierrez D, Fröehlich K U, Kroemer G et al, 2010. Metacaspases are caspases. Doubt no more. Cell Death and Differentiation, 17(3): 377-378 DOI:10.1038/cdd.2009.198 |
Choi C J, Berges J A, 2013. New types of metacaspases in phytoplankton reveal diverse origins of cell death proteases. Cell Death and Disease, 4(2) |
Coll N S, Vercammen D, Smidler A et al, 2010. Arabidopsis type Ⅰ metacaspases control cell death. Science, 330(6009): 1393-1397 DOI:10.1126/science.1194980 |
Dix M M, Simon G M, Wang C et al, 2012. Functional interplay between caspase cleavage and phosphorylation sculpts the apoptotic proteome. Cell, 150(2): 426-440 DOI:10.1016/j.cell.2012.05.040 |
Du F, Zhang Y, Ren H Y, 2011. The universal bundling activity of AtVLN4 in diffusely growing cells. Plant Signaling & Behavior, 6(9): 1290-1293 |
Klemenčič M, Funk C, 2018. Type Ⅲ metacaspases: calcium-dependent activity proposes new function for the p10 domain. New Phytologist, 218(3): 1179-1191 DOI:10.1111/nph.14660 |
Klemenčič M, Novinec M, Dolinar M, 2015. Orthocaspases are proteolytically active prokaryotic caspase homologues: the case of Microcystis aeruginosa. Molecular Microbiology, 98(1): 142-150 DOI:10.1111/mmi.13110 |
Liu J W, Cai W C, Fang X et al, 2018. Virus-induced apoptosis and phosphorylation form of metacaspase in the marine coccolithophorid Emiliania huxleyi. Archives of Microbiology, 200(3): 413-422 DOI:10.1007/s00203-017-1460-4 |
Lu Z Y, Sha J, Tian Y et al, 2017. Polyphenolic allelochemical pyrogallic acid induces caspase-3(like)-dependent programmed cell death in the cyanobacterium Microcystis aeruginosa. Algal Research, 21: 148-155 DOI:10.1016/j.algal.2016.11.007 |
Machado M F M, Marcondes M F, Juliano M A et al, 2013. Substrate specificity and the effect of calcium on Trypanosoma brucei metacaspase 2. The FEBS Journal, 280(11): 2608-2621 DOI:10.1111/febs.12248 |
Martins L M, Kottke T, Mesner P W et al, 1997. Activation of multiple interleukin-1β converting enzyme homologues in cytosol and nuclei of HL-60 cells during etoposide-induced apoptosis. Journal of Biological Chemistry, 272(11): 7421-7430 DOI:10.1074/jbc.272.11.7421 |
McLuskey K, Rudolf J, Proto W R et al, 2012. Crystal structure of a Trypanosoma brucei metacaspase. Proceedings of the National Academy of Sciences of the United States of America, 109(19): 7469-7474 DOI:10.1073/pnas.1200885109 |
Piszczek E, Dudkiewicz M, Mielecki M, 2012. Biochemical and bioinformatic characterization of Type Ⅱ metacaspase protein (TaeMCAⅡ) from wheat. Plant Molecular Biology Reporter, 30(6): 1338-1347 DOI:10.1007/s11105-012-0450-6 |
Read B A, Kegel J, Klute M J et al, 2013. Pan genome of the phytoplankton Emiliania underpins its global distribution. Nature, 499(7457): 209-213 DOI:10.1038/nature12221 |
Rosenwasser S, Ziv C, Van Creveld S G et al, 2016. Virocell metabolism: metabolic innovations during host-virus interactions in the ocean. Trends in Microbiology, 24(10): 821-832 DOI:10.1016/j.tim.2016.06.006 |
Suarez M F, Filonova L H, Smertenko A et al, 2004. Metacaspase-dependent programmed cell death is essential for plant embryogenesis. Current Biology, 14(9): R339-R340 DOI:10.1016/j.cub.2004.04.019 |
Wang X D, Wang X J, Feng H et al, 2012. TaMCA4, a novel wheat metacaspase gene functions in programmed cell death induced by the fungal pathogen Puccinia striiformis f. sp. tritici. Molecular Plant-Microbe Interactions, 25(6): 755-764 DOI:10.1094/MPMI-11-11-0283-R |
Watanabe N, Lam E, 2011. Calcium-dependent activation and autolysis of Arabidopsis metacaspase 2d. Journal of Biological Chemistry, 286(12): 10027-10040 DOI:10.1074/jbc.M110.194340 |
Wong A H H, Yan C Y, Shi Y G, 2012. Crystal structure of the yeast metacaspase Yca1. Journal of Biological Chemistry, 287(35): 29251-29259 DOI:10.1074/jbc.M112.381806 |
 2019, Vol. 50
2019, Vol. 50


