
中国海洋湖沼学会主办。
文章信息
- 陈治, 陈建威, 王晓艳, 高天翔, 刘岩. 2019.
- CHEN Zhi, CHEN Jian-Wei, WANG Xiao-Yan, GAO Tian-Xiang, LIU Yan. 2019.
- 舟山近海环境DNA保存方法的建立及优化
- ESTABLISHMENT AND OPTIMIZATION OF THE EDNA PRESERVATION METHOD FOR ZHOUSHAN COASTAL WATERS
- 海洋与湖沼, 50(5): 1098-1107
- Oceanologia et Limnologia Sinica, 50(5): 1098-1107.
- http://dx.doi.org/10.11693/hyhz20190200034
文章历史
-
收稿日期:2019-02-12
收修改稿日期:2019-05-23
2. 青岛华大基因研究院 青岛 266555;
3. 浙江海洋大学国家海洋设施养殖工程技术研究中心 舟山 316022;
4. 浙江海洋大学水产学院 舟山 316022;
5. 中国水产科学研究院南海水产研究所广州 510300
2. BGI-Qingdao, BGI-Shenzhen, Qingdao 266555, China;
3. National Engineering Research Center of Marine Facilities Aquaculture, Zhejiang Ocean University, Zhoushan 316022, China;
4. Fisheries College, Zhejiang Ocean University, Zhoushan 316022, China;
5. South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510300, China
生物多样性是维持生态平衡、促进人与自然和谐发展的重要成分, 是生态系统稳定的关键因素, 也是生态系统对环境变化响应与适应的重要指标(Myers et al, 2000)。准确掌握水生生物在水体环境中的生物量及其时空分布是开展水生生物多样性保护的基本前提和重要基础(Lodge et al, 2012)。传统的拖网、电鱼、探鱼仪、潜水观测等水生生物多样性调查方法具有调查费用高、所需时间久、靶生物捕获率低、物种鉴别困难及对调查对象及其所在生境破坏性大等缺点(马鸿娟等, 2016; 姜维等, 2016)。近年来, 环境DNA(environmental DNA, eDNA)技术开始被逐渐应用于水生生物多样性调查。eDNA是生物体释放于冰芯、土壤、空气、水体、底泥等环境中的游离的DNA总称(Fahner et al, 2016; Collins et al, 2018; Seymour et al, 2018)。该方法最早出现于20世纪80年代微生物生态学研究中(Ogram et al, 1987; Willerslev et al, 2003; Bohmann et al, 2014; Thomsen et al, 2015), 2008年开始被应用于水生生物研究领域(Ficetola et al, 2008)。
eDNA保存是eDNA分析的重要组成部分, 此步骤一般分酒精保存和冷冻保存两种(单秀娟等, 2018)。但目前尚无关于eDNA保存的统一标准。Takahara等(2012)对冷冻保存1—2d后的水样进行抽滤, 发现eDNA获得量与现场抽滤的效果相同。Minamoto等(2016)通过比较分析, 发现室温条件下eDNA降解速率较快, 而用酒精保存后eDNA在6d内不存在显著降解。同属冷冻保存, 一般认为超低温–80℃效果优于–20℃, 但目前已有的水生生物eDNA保存却极少使用–80℃冷冻保存(Deiner et al, 2015; 姜维等, 2016; Doğdu et al, 2016; Minamoto et al, 2016; Tsuji et al, 2016)。由此来看, eDNA的最佳保存方法, 可能与研究的对象、水域、目的、保存的灵便性等密切相关。舟山渔场是我国最大的近海渔场, 该海域泥沙含量高(张启龙等, 2007)。目前国内尚无该海域及类似高浊度水域水生生物eDNA保存方法的相关研究。本研究采用绝对定量(absolute quantification)和高通量测序方法(next-generation sequencing, NGS)分析了不同eDNA保存方法的差异, 以期探明舟山近海eDNA最佳保存方法, 为该海域开展基于eDNA技术的水生生物多样性调查提供参考。
1 材料与方法 1.1 实验材料定量检测对象为浙江省舟山市西轩岛乌贼繁育中心培育的曼氏无针乌贼(Sepiella japonica)群体, 胴长3.0—7.2cm, 体重24—127g。高通量测序对象为舟山渔场某海域天然海水。
1.2 冷冻、“酒精+常温”、“酒精+低温”三种保存方法初步比较实验开始之前统一使用浓度为0.1%的次氯酸钠溶液对水桶、量筒、烧杯、滤泵、镊子、剪刀等进行消毒处理。水样采集于浙江省舟山市海洋科学城(29°58′40.81″N, 122°12′57.47″E)附近海域。采样时间为2017年4月15日。取两只总重量为155.9g的曼氏无针乌贼(已死), 放入刚采集的10L海水水样中。冲洗乌贼身上的粘膜、墨汁等物质。玻璃棒轻搅1min后, 将乌贼用镊子取出。继续用玻璃棒轻搅1min, 使水样充分混合。将10L水样按1︰1000的比例加入浓度为10%的苯扎氯铵试剂(阳离子表面活性剂), 使水样中苯扎氯铵的终止浓度为0.01%。用直径为47mm、孔径为0.45μm的硝酸纤维滤膜抽取39个体积为100mL的水样。抽滤装置为不锈钢三联抽(Combisart 3-branch stainless steel manifold, GM-0.33A pump)。滤膜保存方式见表 1。
| 组别 | 滤膜数量 | 滤膜处理方式 |
| a | 12 | 锡箔纸包覆, –20℃冷冻保存 |
| b | 12 | 锡箔纸包覆, 加酒精后–20℃低温保存 |
| c | 12 | 锡箔纸包覆, 加酒精后常温保存 |
锡箔纸包覆时注意留一小部分滤膜与酒精接触。对最后3个水样及阴性对照立即进行eDNA提取。结果作为a、b、c三组的初始浓度(0d组)。其余36个滤膜则分别在保存7、15、30、60d后各取9个进行eDNA提取。eDNA提取方法参照姜维等(2016)、马竹欣(2016)。数字定量PCR(digital PCR, dPCR)扩增及后续数据处理参考Doi等(2015)。
1.3 酒精保存方法的优化针对1.2酒精保存(b常温组、c低温组)方法中出现的滤膜上富集物易渗漏等缺点, 本小节改进酒精保存方法。对渗漏的富集物二次过滤, 探讨二次过滤对eDNA获得量的影响。
采样地点、采样方式及防污染措施与1.2相同, 采样时间为2017年5月20日。取3只总重量为176.5g的曼氏无针乌贼, 按照1.2处理方式使水样中含有曼氏无针乌贼eDNA后, 抽滤63个体积为50mL的水样。滤膜保存方式见表 2。
| 组别 | 滤膜数量 | 滤膜处理方式 |
| a | 12 | 锡箔纸包覆, –20℃冷冻保存 |
| b-1 | 12 | 锡箔纸包覆, 加酒精后–20℃低温保存 |
| b-2 | 12 | 加酒精后–20℃低温保存 |
| c-1 | 12 | 锡箔纸包覆, 加酒精后常温保存 |
| c-2 | 12 | 加酒精后常温保存 |
对最后3个水样及阴性对照进行eDNA提取。结果作为五组样品的初始浓度(0d组)。其余60个滤膜则分别在保存7、15、30、60d后各取15个滤膜进行eDNA提取。eDNA提取时, b-1、c-1组不进行二次过滤; b-2、c-2组则用直径47mm、孔径0.22μm的滤膜进行二次过滤。过滤前先轻微晃动离心管, 并对离心管及滤膜用15mL蒸馏水反复润洗三次, 依次将全部液体加入新滤膜上进行过滤。eDNA提取方法及数字定量PCR扩增、后续数据分析参考1.2。
1.4 冷冻与“酒精+低温”两种保存方法高通量测序结果比较利用国家自然科学基金2018年东海春季共享航次和自然资源部第二海洋研究所2018年东海春季资源调查, 对浙江近海十个调查站位取两个表层海水平行样, 每个平行样体积为450mL。一组平行样–20℃冷冻保存(a组), 另一组平行样加酒精后–20℃冷冻保存(b组)。样品保存58、94d后从a、b两组各取5个水样, 并对b组样品进行二次过滤。在青岛华大基因研究院进行eDNA提取和鱼类12S rRNA metabarcoding测序分析。所用引物、建库流程及后续数据分析参照Miya等(2015)。
1.5 差异显著性分析运用SPSS13.0软件对不同样品eDNA分子获得量进行差异显著性分析。其中, 两组样本间比较采用T检验(student’s t test), 检验之前先使用F检验查看方差齐性。对于具有方差齐性的样本采用双样本等方差检验, 不具有方差齐性的样本采用双样本异方差检验; 多组样品间(三组及三组以上)比较采用单因素方差分析(one-way analysis of variance, one-way ANOVA)。显著差异阈值为P=0.05, 试验结果以“平均值±标准差”表示。
2 结果 2.1 三种eDNA保存方法初步比较单因子方差分析结果显示:随保存时间的增加, a、b、c三组曼氏无针乌贼分子拷贝数显著下降, 组内不同天数组之间存在极显著差异(a组: F=418.13, P < 0.001; b组: F=146.21, P < 0.001; c组: F=248.95, P < 0.001)。保存7、15、30、60d后eDNA获得量分别为初始浓度的0.59—0.95 (0.77±0.18)、0.57—0.69 (0.64±0.06)、0.21—0.45 (0.36±0.13)、0.05—0.38 (0.19± 0.17)倍。除保存时间外, 保存方法也显著(或极显著)影响eDNA获得量。保存7d、15d时, 冷冻保存组(a组)eDNA获得量最高, 是“酒精+低温”组(b组)和“酒精+常温”组(c组)的1.59(7d)、1.25倍(7d)和1.20(15d)、1.07倍(15d); 保存30d、60d时b组获得量最高, 分别为a组和c组的2.1(30d)、1.05倍(30d)和7.67(60d)、2.69倍(60d)。除初始组(0d)外, 其余不同天数组在三种保存方法间均存在显著或者极显著差异(0d: F=0, P=1; 7d: F=42.95, P=0.0062; 15d: F=27.46, P=0.0118; 30d: F=78.57, P=0.0026; 60d: F=216.61, P=0.0006)。三种保存方法在保存不同天数后其曼氏无针乌贼eDNA分子拷贝数(copies/μL)见图 1、表 3。
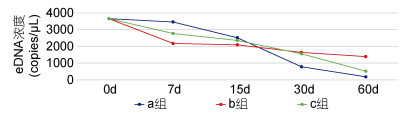 |
| 图 1 三种eDNA保存方法数字PCR定量检测结果 Fig. 1 Results of three eDNA preservation methods quantified by digital PCR |
| 滤膜保存天数(d) | 组别 | ||
| a组 | b组 | c组 | |
| 0 | 3501—3819(3653±160) | 3501—3819(3653±160) | 3501—3819(3653±160) |
| 7 | 3268—3631(3455±182) | 2090—2317(2172±126) | 2683—2825(2768±175) |
| 15 | 2386—2646(2515±130) | 1974—2274(2090±161) | 2269—2452(2353±292) |
| 30 | 686—899(779±109) | 1538—1726(1637±94) | 1442—1634(1556±101) |
| 60 | 149—211(182±31) | 1332—1445(1390±57) | 471—582(516±58) |
b-1、b-2除在0d组eDNA获得量无差异外, 其余不同天数组均存在显著或极显著差异(7d, P=0.0212; 15d, P=0.0071; 30d, P=0.0011; 60d, P= 0.0003)。b-2不同天数组eDNA浓度分别为b-1对应组的1.81、1.60、2.09、4.95倍。c-1、c-2虽然只在7d、30d时eDNA获得量有显著差异, 但c-2组的eDNA平均浓度一直高于c-1组(7d, P=0.0196; 15d, P=0.0732; 30d, P=0.0056)。c-2不同天数组eDNA浓度分别为c-1对应组的1.21、1.28、2.04倍(60d时c-1、c-2组浓度均为0)。
经优化后, b-2组无论是短期保存(15d以内)还是长期保存(15—60d), 其曼氏无针乌贼eDNA获得量均等于或显著高于冷冻保存组(a组)(图 2、表 4)。b-2不同天数组eDNA浓度分别为a组对应天数组的0.96、1.04、8.95倍(60d时a组eDNA浓度为0)(7d, P=0.6033; 15d, P=0.5298; 30d, P=0.0002; 60d, P=0.0001)。
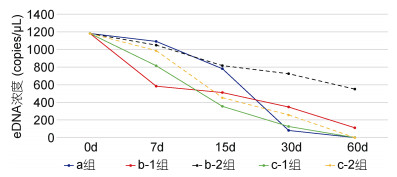 |
| 图 2 二次过滤后不同组数字PCR定量检测结果 Fig. 2 Results of quantitative detection by digital PCR for different groups after secondary filtration |
| 滤膜保存天数(d) | 组别 | ||||
| a组 | b-1组 | b-2组 | c-1组 | c-2组 | |
| 0 | 1067—1206(1182±105) | 1067—1206(1182±105) | 1067—1206(1182±105) | 1067—1206(1182±105) | 1067—1206(1182±105) |
| 7 | 1048—1148(1092±51) | 550—605(583±29) | 916—1123(1050±116) | 786—873(816±50) | 926—1048(987±61) |
| 15 | 731—846(782±59) | 467—556(511±45) | 750—884(817±67) | 331—378(355±24) | 386—519(453±67) |
| 30 | 69—101(81±17) | 329—374(347±24) | 688—778(725±47) | 93—164(126±36) | 239—282(257±22) |
| 60 | 0 | 97—132(111±19) | 514—588(550±37) | 0 | 0 |
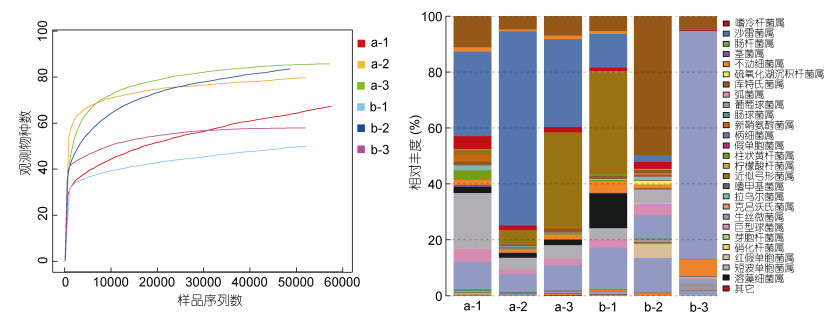
测序所得的observed species曲线后期呈平滑状态, 说明样品测序量足够, 可以用于后续数据分析(图 3, 左)。a、b两组所得的鱼类序列数分别为8442—32924 (24004±9527)、33455—58348 (50080±11404), OTU数目分别为21—40 (29.5±7.97)、24—45 (34.25±8.98)(表 5)。b组的鱼类eDNA序列数目显著高于a组, 是a组的1.08—6.91 (2.68±2.09)倍。a、b两组在Tag数目上接近极显著差异(P=0.0115)。b组5个站位的OTU数目均高于a组(图 3, 右; 表 5), 是a组的1.09—1.24 (1.15±0.06)倍, 但两组间不存在显著差别(P=0.4567)。此外, a、b两组在Shannon指数上存在显著差别(P=0.0221)。显示综合考虑种数和均匀度两方面因素时, 样品存在较大差异(表 5)。a、b两组共定义74个OTU, 其中有14种鱼类在OTU丰度上存在显著或者极显著差别。这14种鱼中, 除花鲈Lateolabrax maculatus为a组丰度高于b组外, 其余皆为b组高于a组(表 6)。样品种水平的物种profiling柱状图显示, a组相对丰度最高的五种鱼依次为角木叶鲽Pleuronichthys cornutus (16.52%)、黄鮟鱇Lophius litulon (13.12%)、小裸胸鳝Gymnothorax minor (9.7%)、柠檬斑鲆Pseudorhombus cinnamoneus (8.56%)、细条天竺鲷Jaydia lineata (8.43%), 但b组依次为小裸胸鳝Gymnothorax minor (13.75%)、小黄鱼Larimichthys pamoides (12.19%)、褐牙鲆Paralichthys olivaceus (8.18%)、黄鮟鱇Lophius litulon (7.69%)、叫姑鱼Johnius grypotus (6.56%)(图 4)。两组样品物种组成相对丰度并不相同, 这与Shannon指数上存在显著差别相对应。种水平的丰度热图结果显示, a、b两组聚类无规律, 10个样品间均未出现平行样间率先聚类的情况, 表明eDNA保存方法影响平行样间的相似性, 使平行样在物种组成和丰度上差异较大(图 5)。
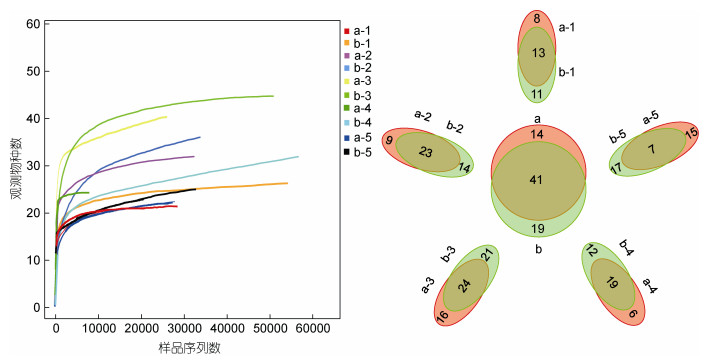 |
| 图 3 鱼类物种稀释曲线(左)及OTU信息(右) Fig. 3 Rarefaction curve of the observed fish species (left) and the OTU information (right) |
| 组别或P值 | Tag数 | OTU数 | Chao指数 | Ace指数 | Shannon指数 | Simpson指数 |
| a | 8442—32924(24004±9527) | 21—40(29.5±7.97) | 21—42.5(32.1±9.96) | 21.38—43.04(33.81±9.77) | 1.72—2.11(1.83±0.16) | 0.15—0.24(0.20±0.03) |
| b | 33455—58348(50080±11404) | 24—45(34.25±8.98) | 24.33—59.5(40.77±14.12) | 25—58.83(45.42±13.99) | 1.86—2.25(2.11±0.15) | 0.14—0.21(0.17±0.03) |
| P | 0.0115 | 0.4567 | 0.299 | 0.172 | 0.0221 | 0.2009 |
| 物种名称 | a组 | b组 | p | |||
| 绝对丰度 | 相对丰度 | 绝对丰度 | 相对丰度 | |||
| 前肛鳗 Dysomma anguillare |
0—126(28±54) | 0—0.71(0.16±0.31) | 0—8394(2095±3626) | 0—0.99(0.64±0.46) | 0.0496 | |
| 龙头鱼 Harpadon nehereus |
0—470(96±208) | 0—0.15(0.05±0.07) | 5—5150(2631±3648) | 0.85—0.99(0.95±0.07) | 0 | |
| 长蛇鲻 Saurida elongata |
0—70(14±31) | 0—0.12(0.02±0.05) | 0—501(139±219) | 0—1(0.38±0.52) | 0.0135 | |
| 日本鳀 Engraulis japonicus |
0—139(63±67) | 0—0.26(0.1±0.12) | 1—2154(873±913) | 0.74—1(0.90±0.12) | 0 | |
| 多棘腔吻鳕 Coelorinchus multispinulosus |
0—39(8±17) | 0—0.1(0.02±0.04) | 0—340(68±151) | 0—1(0.38±0.52) | 0.0048 | |
| 六丝钝尾虾虎鱼 Amblychaeturichthys hexanema |
0—144(31±63) | 0—0.08(0.02±0.03) | 0—5416(1724±2483) | 0—0.99(0.57±0.53) | 0.0039 | |
| 花鲈 Lateolabrax maculatus |
0—6333(1267±2831) | 0—1(0.4±0.54) | 0 | 0 | 0 | |
| 叫姑鱼 Johnius grypotus |
7—5632(1288±2436) | 0.01—0.92(0.23±0.38) | 63—11017(3066±4585) | 0.08—0.98(0.77±0.38) | 0.0405 | |
| 小黄鱼 Larimichthys polyactis |
0—304(99±124) | 0—0.41(0.09±0.18) | 0—22718(5699±9652) | 0—0.88(0.70±0.43) | 0.0323 | |
| 少鳞鱚 Sillago japonica |
0 | 0 | 0—245(50±108) | 0—1(0.4±0.55) | 0 | |
| 单指虎鲉 Minous monodactylus |
0 | 0 | 0—16(3±7) | 0—1(0.4±0.55) | 0 | |
| 短吻三线舌鳎 Cynoglossus abbreviatus |
0—6(2±4) | 0—0.09(0.02±0.04) | 0—19(4±8) | 0—1(0.38±0.52) | 0.0176 | |
| 镰鲳 Pampus echinogaster |
0—1312(422±604) | 0—0.38(0.1±0.17) | 1—5114(1767±2095) | 0.62—1(0.90±0.17) | 0 | |
| 燕魟未定种 Gymnura sp. |
0 | 0 | 0—8(2±8) | 0—1(0.4±0.55) | 0 | |
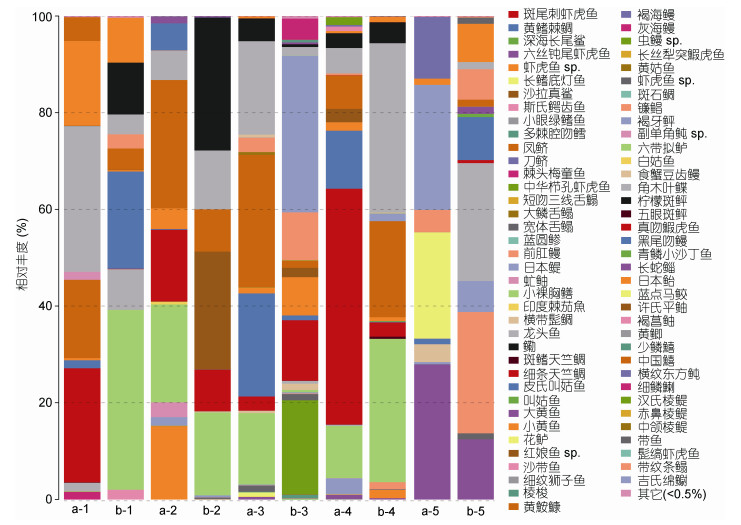 |
| 图 4 种水平的profiling柱状图 Fig. 4 The profiling histogram at species level |
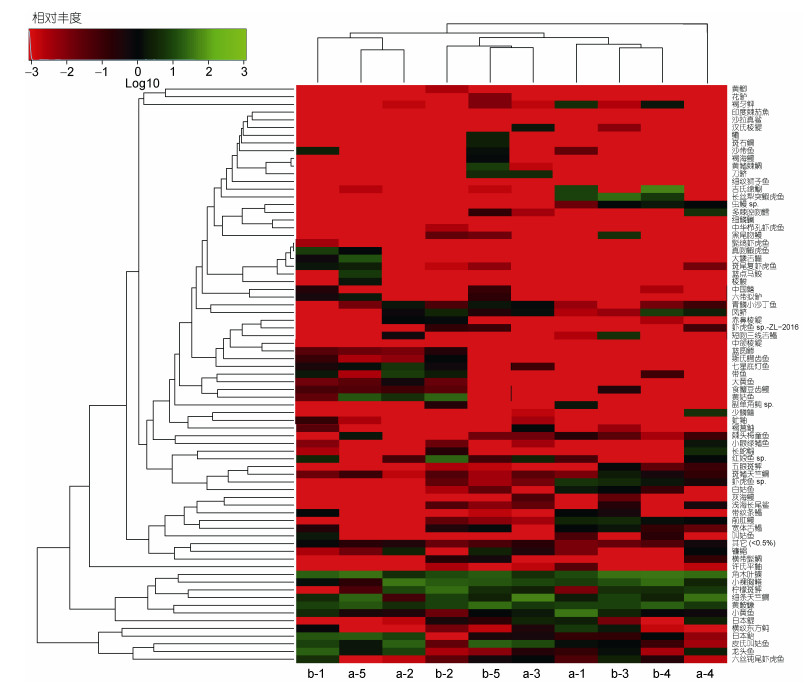 |
| 图 5 种水平的丰度热图 Fig. 5 Log-scaled percentage heat map at species level |
保存94d后的样品eDNA严重降解, 5个站位中有2个站位样品测序失败。剩余3个站位样品均未检测到鱼类信息, 高通量测序结果皆为微生物物种。测序所得的observed species指数曲线后期呈平滑状态(图 6, 左)。a、b两组的有效序列数分别为52571—58790 (56296±3582)、47563—52589 (50574±2895), OTU数分别为63—82 (73.2±9.71)、44—81 (59.2±20.81)。a组的序列数和OTU数略高于b组, 是b组的1.105—1.118 (1.114±0.007)和0.938—1.782 (1.384±0.424)。但两组之间并无显著区别(Tag number: P=0.0945; OTU number: P=0.2977) (表 7)。Chao指数和Simpson指数上存在显著差别(P=0.0319; P=0.023), 显示两组样品在物种组成上存在一定的差别。物种profiling图显示虽然a、b两组的第一优势菌群均为变形菌门(Proteobacteria; a: 67.18%, b: 62.45%), 但第二大优势菌群却分别为放线菌门(Actinobacteria; a: 17.04%, b:2.67%)和拟杆菌门(Bacteroidetes; a: 3.98%, b: 14.26%)(图 6, 右)。不同的eDNA保存方法也对微生物的物种组成产生一定影响。
 |
| 图 6 微生物物种稀释曲线(左)及属水平的profiling柱状图(右) Fig. 6 Rarefaction curve of observed micro-organisms species (left) and profiling histogram at genus level (right) |
| 组别或P值 | Tag number | OTU number | Ace指数 | Chao指数 | Shannon指数 | Simpson指数 |
| a | 52571— 58790(56296±3582) |
63— 82(73.2±9.71) |
64.33— 83.34(75.93±10.27) |
68— 105(89±19) |
1.81— 2.55(2.18±0.37) |
0.18— 0.31(0.23±0.07) |
| b | 47563— 52589(50574±2895) |
44— 81(59.2±20.81) |
46.95— 82.62(59.03±20.43) |
54— 95(68.67±22.85) |
0.89— 2.23(1.73±0.73) |
0.28— 0.67(0.44±0.20) |
| P | 0.0945 | 0.2977 | 0.2905 | 0.0319 | 0.1124 | 0.023 |
加入酒精保存的样品, 7d后其浓度约为初始浓度的69.03%—83.5% (76.1%±7.24%) (c、c-1、c-2组)。样品常温保存60d后, 曼氏无针乌贼eDNA分子拷贝数依然较高(c组: 471—582 (516±58))。在交通闭塞、取样困难、电力匮乏的野外, 蠕动泵抽滤后对滤膜进行“酒精+常温”保存不失为eDNA保存的优选(姜维等, 2016; 马竹欣, 2016)。低温条件下, 酒精对eDNA的保护效果依旧明显。样品保存30、60d时, 有酒精组(b、b-1、b-2组)曼氏无针乌贼eDNA分子获得量是无酒精组(a组)eDNA获得量的2.10—8.95(5.74±3.12)倍。并且保存时间越久, 差别倍数越高。高通量测序结果也表明, 有酒精组样品(b组)在样品总序列数和多个鱼种分序列数上均显著高于无酒精组(a组)。酒精对DNA有较好的保护作用, 因此Ficetola等(2008)、Thomsen等(2012a, b)、Minamoto等(2016)、姜维等(2016)、马竹欣(2016)等在野外采样时均曾使用酒精对eDNA进行保存。
酒精保存法有一定缺点, 主要表现在富集物容易渗漏、后续去酒精过程繁琐、若风干时间过长则容易导致eDNA降解。而无酒精保存组却不存在这些问题。Jo等以竹荚鱼(Trachurus japonicus)为定量检测对象, 发现温度显著影响eDNA降解速率, 建议使用低温对eDNA进行保存(Jo et al, 2019)。Yamanaka等(2017)的研究也表明高温加速eDNA降解。本研究中, 冷冻处理对样品短期保存(15d内)效果良好。在此时间范围内, a组的eDNA平均分子获得量高于b、b-1、c、c-1、c-2五组的分子获得量。样品的eDNA获得量是后五者的1.25—1.59倍(7d)和1.07—1.20倍(15d)。特别是7d组, 其eDNA浓度是初始组的92.39%—94.58%。采用冷冻保存法对滤膜进行短期保存, 不仅可以获得最高的eDNA获得量, 还可以避免酒精带来的繁琐操作。但是, 冷冻保存的eDNA样品在15d后有加速降解的趋势。保存30—60d时, 曼氏无针乌贼分子浓度最低值均来自a组, 仅为“酒精+常温”组(c、c-1、c-2)的0—64.29% (30.19%±26.11%)。因此不建议使用–20℃冷冻处理方式对滤膜进行长期保存。
3.2 二次过滤的优点目前, 有研究者在eDNA提取之前使用专门的唾液收集管对滤膜上的eDNA进行二次富集(离心), 以提高eDNA获得量(Takahara et al, 2013; Yamamoto et al, 2016; Minamoto et al, 2017)。但此类唾液收集管对离心机规格有要求, 且价格较贵。针对酒精保存法存在的问题, 本研究探索了二次过滤对eDNA获得量的影响。“2.2”、“2.3”的结果表明:二次过滤能够显著提高eDNA的获得量。
与冷冻保存法相比, 酒精保存法最大的劣势在于后期去酒精操作较为繁琐。但二次富集则可弥补这些缺陷。首先, 采用二次过滤法在取样阶段无需对滤膜进行锡箔纸包覆处理, 直接对滤膜浸泡即可。滤膜在浸泡过程中不易发生黏附。而锡箔纸包覆过的滤膜则易出现滤膜粘附成团块现象, 不利于后期展开风干。其次, 即使经过严格包覆处理, 样品也会出现eDNA富集物渗漏现象。二次过滤可将这部分eDNA重新收集。此操作效果类似Minamoto等(2017)用唾液收集管进行二次离心操作。最后, 离心管容积小、过滤时间短。二次过滤后用超纯水润洗滤泵、滤膜, 新滤膜上残留的酒精较少, 液体基本为水。可直接用于eDNA提取。既省去了酒精风干所浪费的时间, 也避免了风干带来的eDNA降解。
3.3 后续还需进一步优化的环节有研究表明滤膜孔径影响eDNA获得量(Deiner et al, 2015)。本研究使用孔径0.22μm的滤膜进行二次过滤。后续应探讨更小孔径滤膜的富集效果。保存94d的样品高通量测序后未能检测到鱼类信息。OTU经注释后皆为微生物。微生物的eDNA降解速率可能慢于大生物。使用更低的保存温度或许能延长鱼类eDNA的保存时间。同时, –20℃冷冻保存的曼氏无针乌贼eDNA样品在15d后有加速降解的趋势(见结果2.1、2.2部分), 这可能也是由未使用更低的冷冻温度造成(Strickler et al, 2015)。赵梦迪(2017)利用环境DNA分析冬季中国东黄海水域的鱼类多样性时使用–80℃对水样进行保存; Rodgers、Cowart等使用液氮对南、北极水样进行了超低温保存, 但三者的保存时间均未超过一个月, 且均未进行eDNA降解速率方面的研究(Cowart et al, 2018; Rodgers et al, 2018)。一些微生物eDNA在–80℃条件下可保存一年(赵裕栋等, 2012), 组织DNA在超低温条件下的保存时间则更久(庞峻峰等, 2001)。此类结果要优于本研究。但与微生物eDNA及组织DNA不同, 自然水体中大生物eDNA浓度低、降解快(Tsuji et al, 2016; Jo et al, 2019), 一般24h后即低于定量检测阈值。而在偏远地区(如山区或只能徒步采样的野外)往往无法携带复杂的超低温保存设备(如液氮罐等)。因此从样品保存的便捷性、紧迫性和可行性角度考虑, 目前已有的水生生物eDNA保存极少使用–80℃冷冻处理(Deiner et al, 2015; 姜维等, 2016; Doğdu et al, 2016; Minamoto et al, 2016; Tsuji et al, 2016)。已有的相关研究基本将实验温度设置在–20—45℃间, 并在此温度范围内建立和优化水生生物eDNA的保存方法(Deiner et al, 2015; Renshaw et al, 2015; Wegleitner et al, 2015; Minamoto et al, 2016; Yamanaka et al, 2017; Seymour et al, 2018)。本研究在实验之初亦未将超低温组考虑在内, 后续应探究更低温度下(如–80℃)水生生物eDNA的极限保存时间。
此外, 保存94d的样品a、b两组微生物总序列数并不存在显著差别。a组的Tag number反而稍微高于b组, 这与2.1、2.2、2.3的结果相矛盾。不同界(Kingdom)的eDNA最优保存方法可能并不一致。Deiner等(2015)的研究也表明, 对于原核生物, 建议使用沉淀法, 对于真核生物, 则建议使用抽滤法——两类生物的eDNA最优采集方法也不相同。后续应更加系统地比较这两类生物在eDNA保存方法上的差异。此外, b组鱼类Tag数值虽然显著高于a组, 但二者在OTU数上却不存在显著差别(P=0.4567)。eDNA metabarcoding技术灵敏度极高, 可有效检测低密度物种的存在。这可能是OTU number未能有效反映两种保存方法效果不同的原因。
4 结论本研究首次建立了舟山近海高浊度水样大生物eDNA最适保存方法, 建议使用“酒精+低温”法+“二次过滤”对相似水样进行eDNA长期保存。
马竹欣, 2016.利用环境DNA技术调查入侵种克氏原螯虾在元阳梯田的分布.昆明: 云南大学硕士学位论文 http://cdmd.cnki.com.cn/Article/CDMD-10673-1016228532.htm
|
马鸿娟, Kathryn S, 马利民, 等. 2016. 环境DNA及其在水生生态系统保护中的应用. 生态学杂志, 35(2): 516-523 |
张启龙, 王凡, 赵卫红, 等. 2007. 舟山渔场及其邻近海域水团的季节特征. 海洋学报, 29(5): 1-9 |
庞峻峰, 张亚平. 2001. 标本DNA研究进展. 动物学研究, 22(6): 490-496 DOI:10.3321/j.issn:0254-5853.2001.06.011 |
单秀娟, 李苗, 王伟继. 2018. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 39(3): 23-29 |
赵梦迪, 2017.利用环境DNA分析冬季中国东黄海水域的鱼类多样性.上海: 上海海洋大学硕士学位论文 http://cdmd.cnki.com.cn/Article/CDMD-10264-1017854350.htm
|
赵裕栋, 周俊, 何璟. 2012. 土壤微生物总DNA提取方法的优化. 微生物学报, 52(9): 1143-1150 |
姜维, 王启军, 邓捷, 等. 2016. 以川陕哲罗鲑为目标物种的水样环境DNA分析流程的优化. 应用生态学报, 27(7): 2372-2378 |
Bohmann K, Evans A, Gilbert M T P et al, 2014. Environmental DNA for wildlife biology and biodiversity monitoring. Trends in Ecology & Evolution, 29(6): 358-367 |
Collins R A, Wangensteen O S, O'Gorman E J et al, 2018. Persistence of environmental DNA in marine systems. Communications Biology, 1(1): 185 DOI:10.1038/s42003-018-0192-6 |
Cowart D A, Murphy K R, Cheng C H C, 2018. Metagenomic sequencing of environmental DNA reveals marine faunal assemblages from the West Antarctic Peninsula. Marine Genomics, 37: 148-160 DOI:10.1016/j.margen.2017.11.003 |
Deiner K, Walser J C, Machler E et al, 2015. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA. Biological Conservation, 183: 53-63 DOI:10.1016/j.biocon.2014.11.018 |
Doğdu S A, Turan C, 2016. Environmental DNA for detection of endangered grouper species (Epinephelus spp.). Natural and Engineering Sciences, 1(3): 42-48 DOI:10.28978/nesciences.286311 |
Doi H, Takahara T, Minamoto T et al, 2015. Droplet digital Polymerase Chain Reaction (PCR) outperforms real-time PCR in the detection of environmental DNA from an invasive fish species. Environmental Science & Technology, 49(9): 5601-5608 |
Fahner N A, Shokralla S, Baird D J et al, 2016. Large-scale monitoring of plants through environmental DNA metabarcoding of soil:recovery, resolution, and annotation of four DNA markers. PLoS One, 11(6): e0157505 DOI:10.1371/journal.pone.0157505 |
Ficetola G F, Miaud C, Pompanon F et al, 2008. Species detection using environmental DNA from water samples. Biology Letters, 4(4): 423-425 DOI:10.1098/rsbl.2008.0118 |
Jo T, Murakami H, Yamamoto S et al, 2019. Effect of water temperature and fish biomass on environmental DNA shedding, degradation, and size distribution. Ecology and Evolution, 9(3): 1135-1146 DOI:10.1002/ece3.4802 |
Lodge D M, Turner C R, Jerde C L et al, 2012. Conservation in a cup of water:estimating biodiversity and population abundance from environmental DNA. Molecular Ecology, 21(11): 2555-2558 DOI:10.1111/j.1365-294X.2012.05600.x |
Minamoto T, Naka T, Moji K et al, 2016. Techniques for the practical collection of environmental DNA:filter selection, preservation, and extraction. Limnology, 17(1): 23-32 DOI:10.1007/s10201-015-0457-4 |
Minamoto T, Fukuda M, Katsuhara K R et al, 2017. Environmental DNA reflects spatial and temporal jellyfish distribution. PLoS One, 12(2): e0173073 DOI:10.1371/journal.pone.0173073 |
Miya M, Sato Y, Fukunaga T et al, 2015. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes:detection of more than 230 subtropical marine species. Royal Society Open Science, 2(7): 150088 DOI:10.1098/rsos.150088 |
Myers N, Mittermeier R A, Mittermeier C G et al, 2000. Biodiversity hotspots for conservation priorities. Nature, 403(6772): 853-858 DOI:10.1038/35002501 |
Ogram A, Sayler G S, Barkay T, 1987. The extraction and purification of microbial DNA from sediments. Journal of Microbiological Methods, 7(2-3): 57-66 DOI:10.1016/0167-7012(87)90025-X |
Renshaw M A, Olds B P, Jerde C L et al, 2015. The room temperature preservation of filtered environmental DNA samples and assimilation into a phenol-chloroform-isoamyl alcohol DNA extraction. Molecular Ecology Resources, 15(1): 168-176 DOI:10.1111/1755-0998.12281 |
Rodgers T W, Olson J R, Klobucar S L et al, 2018. Quantitative PCR assays for detection of five arctic fish species:Lota lota, Cottus cognatus, Salvelinus alpinus, Salvelinus malma, and Thymallus arcticus from environmental DNA. Conservation Genetics Resources, 10(4): 859-865 DOI:10.1007/s12686-017-0883-1 |
Seymour M, Durance I, Cosby B J et al, 2018. Acidity promotes degradation of multi-species environmental DNA in lotic mesocosms. Communications Biology, 1(1): 4 DOI:10.1038/s42003-017-0005-3 |
Strickler K M, Fremier A K, Goldberg C S, 2015. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms. Biological Conservation, 183(9): 85-92 |
Takahara T, Minamoto T, Yamanaka H et al, 2012. Estimation of fish biomass using environmental DNA. PLoS One, 7(4): e35868 DOI:10.1371/journal.pone.0035868 |
Takahara T, Minamoto T, Doi H, 2013. Using environmental DNA to estimate the distribution of an invasive fish species in ponds. PLoS One, 8(2): e56584 DOI:10.1371/journal.pone.0056584 |
Thomsen P F, Kielgast J, Iversen L L et al, 2012a. Detection of a diverse marine fish fauna using environmental DNA from seawater samples. PLoS One, 7(8): e41732 DOI:10.1371/journal.pone.0041732 |
Thomsen P F, Kielgast J, Iversen L L et al, 2012b. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 21(11): 2565-2573 DOI:10.1111/j.1365-294X.2011.05418.x |
Thomsen P F, Willerslev E, 2015. Environmental DNA-an emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 183: 4-18 DOI:10.1016/j.biocon.2014.11.019 |
Tsuji S, Yamanaka H, Minamoto T, 2016. Effects of water pH and proteinase K treatment on the yield of environmental DNA from water samples. Limnology, 18(1): 1-7 |
Wegleitner B J, Jerde C L, Tucker A et al, 2015. Long duration, room temperature preservation of filtered eDNA samples. Conservation Genetics Resources, 7(4): 789-791 DOI:10.1007/s12686-015-0483-x |
Willerslev E, Hansen A J, Binladen J et al, 2003. Diverse plant and animal genetic records from holocene and pleistocene sediments. Science, 300(5620): 791-795 DOI:10.1126/science.1084114 |
Yamamoto S, Minami K, Fukaya K et al, 2016. Environmental DNA as a 'snapshot' of fish distribution:a case study of Japanese jack mackerel in Maizuru Bay, Sea of Japan. PLoS One, 11(3): e0149786 DOI:10.1371/journal.pone.0149786 |
Yamanaka H, Minamoto T, Matsuura J et al, 2017. A simple method for preserving environmental DNA in water samples at ambient temperature by addition of cationic surfactant. Limnology, 18(2): 233-241 DOI:10.1007/s10201-016-0508-5 |
 2019, Vol. 50
2019, Vol. 50


