
中国海洋湖沼学会主办。
文章信息
- 柴英辉, 高菲, 王金锋, 周文礼. 2019.
- CHAI Ying-Hui, GAO Fei, WANG Jin-Feng, ZHOU Wen-Li. 2019.
- 仿刺参(Apostichopus japonicus)肠道菌群的地域性差异与共性研究
- INTESTINAL MICROBIOTA IN APOSTICHOPUS JAPONICUS: REGIONAL DIFFERENCE AND COMMON FEATURE
- 海洋与湖沼, 50(5): 1127-1137
- Oceanologia et Limnologia Sinica, 50(5): 1127-1137.
- http://dx.doi.org/10.11693/hyhz20190200044
文章历史
-
收稿日期:2019-02-28
收修改稿日期:2019-05-31
2. 海南大学海洋学院 海口 570228;
3. 中国科学院北京生命科学研究院 北京 100101
2. College of Oceanology, Hainan University, Haikou 570228, China;
3. Beijing Institute of Life Sciences, Chinese Academy of Sciences, Beijing 100101, China
海参属于棘皮动物门海参纲, 是一类特殊的海洋无脊椎动物(Wang et al, 2018)。由于营养价值高、富含多种生物活性成分(Mashjoor et al, 2017), 海参在亚洲作为一种传统的保健食品已有上千年的食用历史(Zhu et al, 2018)。目前海参已成为世界范围内一种常见的、具有较高经济价值的海产品, 其水产养殖业也在不断扩大(Jo et al, 2017; Li et al, 2019)。仿刺参(Apostichopus japonicus Selenka)是海参中重要的养殖种类, 在中国、日本和韩国地区都有大规模培育, 在我国主要分布于辽宁、河北、山东和江苏沿海等水域(Wang et al, 2015)。在过去十几年的渔业发展中, 仿刺参已由传统的小规模食用养殖, 慢慢转变成区域、国家和国际的产业化格局(Conand, 2018)。
肠道菌群结构以及微生物互作被认为对维持宿主健康非常关键, 菌群稳态以及微生物群落与宿主之间的有益交流, 在人体和模式动物中的研究较为深入(Endesfelder et al, 2014; Coyte et al, 2015)。水生动物虽然不及人体和模式动物的研究深入, 但同样被发现有着复杂的肠道微生物群落(Ratana-Arporn et al, 2014)。水生动物肠道共生菌群与宿主之间也存在着密切的联系, 正常状态下物种组成和丰度相对稳定, 被认为是宿主的重要组成部分(O'Hara et al, 2006), 不仅决定了食物中复杂分子的分解情况(Fava et al, 2019), 还对宿主的发育、免疫和营养状态具有调节作用(Chi et al, 2014; Xiong et al, 2015; Zhu et al, 2016)。目前有许多研究尝试将益生菌应用于鱼虾养殖之中, 均取得了良好的效果(Chi et al, 2014; Lazado et al, 2014; Hai, 2015; Banerjee et al, 2017; Duan et al, 2017; Quiroz-Guzmán et al, 2018; Gupta et al, 2019)。仿刺参消化系统独特, 体腔中只有一根简单的肠, 具有移动缓慢、体积大、便于解剖、寿命长、分布广泛等特点, 是研究水生动物肠道微生物群进化和功能的最佳选择(Tunca et al, 2016; Yang et al, 2017)。有实验报道从仿刺参肠道分离出少量的细菌菌株, 并通过测序获得了基因组信息(Hong et al, 2017; Jo et al, 2017)。另有多项研究分别对中国、日本和韩国海域的仿刺参肠道微生物进行了高通量测序(Yamazaki et al, 2016; Kim et al, 2017; Bronstein et al, 2018), 为研究水生动物共生微生物提供了宝贵的数据, 但是仿刺参肠道微生物群落的地域差异和共性以及潜在的功能鲜有报道, 对仿刺参益生菌的研究也还处于实验起步阶段(Chen et al, 2018; Li et al, 2019)。
为了理解仿刺参肠道微生物群落的生态稳定性, 本研究通过对不同地域的仿刺参肠道、海水和沉积物微生物组进行生物信息学分析, 揭示仿刺参肠道微生物地域性差异和共性, 以及与水体环境之间的联系, 以期为仿刺参共生微生物及其与宿主健康的相关性研究提供基础性资料, 更好地推动水产养殖业和渔业的发展。
1 材料与方法 1.1 数据来源从NCBI-SRA数据库中, 对2000—2018年公开发表的16S rRNA高通量测序数据进行检索, 收集到中国仿刺参肠道样本数据42例, 日本仿刺参肠道样本数据20例, 韩国仿刺参肠道样本数据64例。作为比较同时下载了绿海胆(Lytechinus variegatus)肠道样本数据20例, 海水样本数据381例和沉积物样本数据94例(表 1)。
| 项目号 | 物种 | 地域 | 肠道 | 海水 | 沉积物 | 测序平台 | 16S rRNA | 坐标 | 采集时间 | 提交机构 |
| PRJNA430504 | 仿刺参 | 渤海 | 30 | - | - | Illumina | V4 | 未知 | 2017/3/20 | 烟台大学 |
| PRJNA337986 | 仿刺参 | 黄海 | 12 | - | 6 | Illumina | V3-V4 | 未知 | 2014/11 | 中国海洋大学水产学院 |
| PRJDB4366 | 仿刺参 | 日本 | 20 | 1 | - | 454 | V1-V2 | 42.13N,140E | 2017/8 | 北海道大学(Yamazaki et al, 2016) |
| PRJEB21905 | 仿剌参 | 韩国 | 64 | - | - | Illumina | V1-V3 | 37.89N, 127.74E | 2016/8-11 | 翰林大学(Kim et al, 2017) |
| PRJNA326427 | 绿海胆 | 美国 | 12 | 3 | - | Illumina | V4 | 29.8N, 85.36W | 2014/10 | 阿拉巴马大学伯明翰分校(Bronstein et al, 2018) |
| PRJNA291441 | 绿海胆 | 美国 | 8 | 1 | - | Illumina | V4 | 29.8N, 85.36W | 2013/4 | 阿拉巴马大学伯明翰分校 |
| PRJNA486858 | 海水 | 南海 | - | 30 | - | Illumina | V3-V4 | 22.61N, 114.56E | 2017/7/28 | 中国科学院南海海洋研究所 |
| PRJNA387529 | 海水 | 东海 | - | 67 | - | Illumina | V5-V6 | 26N, 120.56E | 2014/10/2 | 国立台湾大学 |
| PRJNA316898 | 海水 | 南海 | - | 25 | - | 454 | V4 | 20.76N, 119.8E | 2013/4 | 同济大学 |
| PRJDB3905 | 海水 | 东海 | - | 49 | - | 454 | V4-V5 | 29.87N, 121.89E | 2012/12/12 | 中国科学院土壤科学研究所 |
| PRJNA358780 | 海水 | 东海 | - | 8 | - | Illumina | V4-V5 | 30.2N, 122.69E | 2016/1/4 | 浙江海洋大学 |
| PRJNA427864 | 海水 | 渤海 | - | 12 | - | Illumina | V4-V5 | 37.75N, 120.106E | 2015/8/17 | 中国科学院烟台海岸带研究所 |
| PRJNA389159 | 海水 | 南海 | - | 24 | - | Illumina | V3-V4 | 未知 | 2014/6 | 山东大学 |
| PRJNA418051 | 海水 | 渤海 | - | 10 | - | Illumina | V4 | 39.36N, 119.45E | 2016/5/12 | 中国环境科学研究院 |
| PRJNA336443 | 海水 | 南海 | - | 23 | - | 454 | V1-V3 | 15.14N, 113E | 2012/10/9 | 中国海洋大学 |
| PRJDB4992 | 海水 | 东海 | - | 105 | - | Illumina | V4 | 29.54N, 121.76E | 2015/4/15 | 宁波大学 |
| PRJNA284317 | 海水 | 南海 | - | 12 | - | 454 | V4 | 22.34N,114.30E | 2013/8 | 香港科技大学 |
| PRJNA449229 | 海水 | 东海 | - | 11 | - | Illumina | V4-V5 | 28.14N, 127.12E | 2015/10 | 中国海洋大学 |
| PRJNA378681 | 沉积物 | 渤海 | - | - | 37 | 454 | V4 | 38.16N, 122.48E | 2012/5/14 | 中国科学院烟台海岸带研究所 |
| PRJNA214261 | 沉积物 | 渤海 | - | - | 32 | Illumina | V5-V6 | 未知 | 2011/5/1 | 大连海洋大学 |
| PRJNA328063 | 沉积物 | 南海 | - | - | 19 | Illumina | V4-V5 | 未知 | 2013/5/6 | 华东师范大学 |
使用FLASH软件及默认参数对16S rRNA双端读段进行对接获得长序列(Magoč et al, 2011)。去除带有N的对接序列, 剩余序列做切除引物处理, 并将序列转化成fasta格式以便于后续分析。运用Qiime软件对数据进行分析(Huang et al, 2014), 默认以97%的序列相似度聚类得到OTU(operational taxonomic unit), 即可操作分类单元, 并基于Greengenes数据库对OTU进行物种注释获得分类信息表(Quast et al, 2013)。
1.2.2 多样性分析统计分析和图形化过程使用R语言进行。运用CSS标准化OTU丰度, 使用vegan包计算Shannon多样性指数; 使用optparse、metagenomeSeq、biom包计算Bray-Curtis距离得到组间Beta多样性。
1.2.3 细菌共现性分析利用SparCC的方法, 基于所有仿刺参肠道样本的丰度信息计算菌属相关性, 阈值设置为P < 0.05、相关性系数大于0.5, 并使用Gephi软件绘制共存关系网络图(Wang et al, 2016)。
1.2.4 功能预测使用FAPROTAX软件(1.1版本)进行功能预测(Louca et al, 2016)。FAPROTAX软件包含一个基于已验证的可培养菌信息构建的物种分类与功能注释数据库, 将OTU分类表与该数据库进行关联, 以推测各物种携带的功能及其丰度, 得到微生物群落功能注释预测结果。
1.2.5 聚类分析使用ggplots和pheatmap包, 基于菌属丰度计算肠道、海水和沉积物样本Pearson系数后进行聚类, 分析微生物群落相似性(Gao et al, 2016); 基于预测的代谢功能对中国、日本和韩国样本进行聚类并绘制热图。
1.2.6 多组间比较使用plyr、scales和grid、VennDiagram包计算组间共有与差异菌属(Roux et al, 2016); 使用Galaxy平台进行LEfSe (linear discriminant analysis effect size)分析, 识别分组代表性菌属(Giardine et al, 2005); 使用Calypso平台进行菌属OddsRatio分析(Zakrzewski et al, 2017)。使用Wilcoxon秩和检验统计组间的差异显著程度。
2 结果 2.1 仿刺参肠道微生物与水环境的比较本研究在97%序列相似性的阈值下从所有样本中共识别出16383个OTUs。基于OTU数据计算各样本的Alpha多样性, 结果显示仿刺参肠道微生物多样性显著低于(P < 0.05)海水和沉积物中的微生物多样性(图 1a), 其中以沉积物中的多样性最高, 主要原因可能在于沉积物中不仅包含了水体开放环境的微生物, 还包括了其它水生动物的排泄物和水生植物沉积的微生物。在同样基于OTU数据的样本聚类结果中发现, 所有仿刺参肠道样本均与海水样本首先聚集(图 1b), 并且这些海水样本大多来自于渤海。鉴于许多沉积物样本同样取自渤海, 但并未与仿刺参肠道样本首先形成聚类关系, 提示仿刺参肠道菌群与海水较为相似, 而与沉积物差异相对较大。
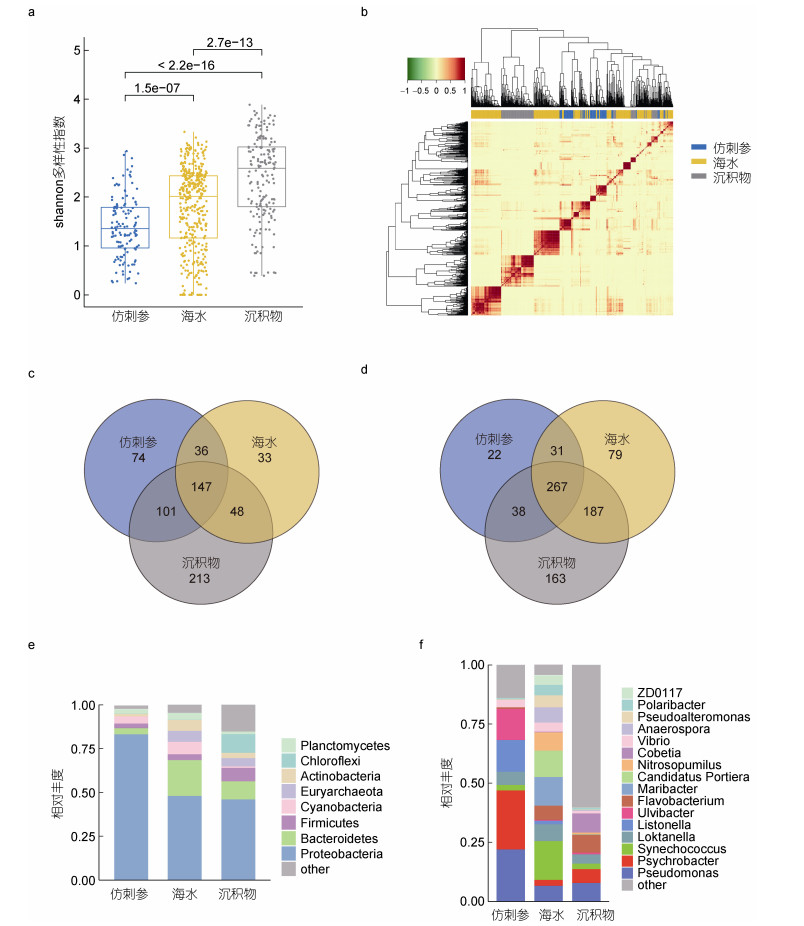 |
| 图 1 仿刺参肠道微生物与外界环境的联系 Fig. 1 Relationship between intestinal microorganisms and external environment of A. japonicus 注: a.仿刺参肠道、海水、沉积物菌群的Alpha多样性; b.仿刺参肠道、海水和沉积物菌群结构相关性, 基于细菌丰度计算Pearson系数后进行聚类; c.仿刺参肠道与黄海和渤海海水、沉积物共有和独有菌属; d.仿刺参肠道、海水和沉积物(渤海、黄海、东海和南海)样本中共有和独有菌属; e.仿刺参肠道、海水和沉积物优势细菌在门水平的比较; f.仿刺参肠道、海水和沉积物优势菌属的比较 |
对识别的OTU进行分类注释, 并从细菌属水平上将仿刺参肠道样品与其地理位置较近的黄海和渤海的海水及沉积物进行比较(图 1c), 发现仿刺参肠道微生物独有菌属远低于共有菌属数量(74 : 284), 且三种样本类型共有的比例较高。为进一步揭示仿刺参肠道微生物与海洋环境之间的关系, 添加了中国东海、南海水域的海水及沉积物样本, 仿刺参肠道独有菌属数量减少至22个(图 1d), 而与海水和沉积物三者共有菌属的数量由147个(图 1c)增加到四个海区的267个, 表明随着取样范围的扩大及识别菌属数量的增多, 其与水体环境间可能存在更加广泛的交流。
分别统计仿刺参肠道、海水和沉淀中相对丰度较高的在微生物类群(图 1e-f), 发现优势菌在三类样本中的组分十分相似, 主要是变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和蓝细菌门(Cyanobacteria)等。但与环境样本相比, 仿刺参肠道菌群中的变形细菌增多以及拟杆菌减少较为显著; 在属水平上假单胞菌属(Pseudomonas)、嗜冷杆菌属(Psychrobacter)和利斯顿氏菌属(Listonella)等的富集尤为明显(P < 0.05), 提示仿刺参在生长过程中会在体内对外界环境来源的微生物进行选择性的富集, 维持自身微生物的动态平衡并满足自身的生长需求。
2.2 仿刺参肠道微生物与绿海胆的异同为了更加深入地认识仿刺参肠道菌群特征, 本研究将其与其它棘皮动物绿海胆进行了比较, 结果发现仿刺参肠道微生物多样性显著低于(P=0.0024)绿海胆(图 2a)。两种棘皮动物的肠道微生物群落结构也明显不同, 主成分分析将它们分为了不同的两簇(图 2b)。在两种动物肠道菌群的分组中, 假单胞菌属、假交替单胞菌属(Pseudoalteromonas)、Octadecabacte、褐杆菌属(Phaeobacter)、陆丹氏菌属(Loktanella)和弧菌属(Vibrio)的贡献度较大。其中假单胞菌属在仿刺参与海水和沉积物样本的比较中, 同样显示出富集的效果, 提示高丰度的该菌或可作为仿刺参的特征菌属, 用于与环境样本及其它棘皮动物菌群进行区分。
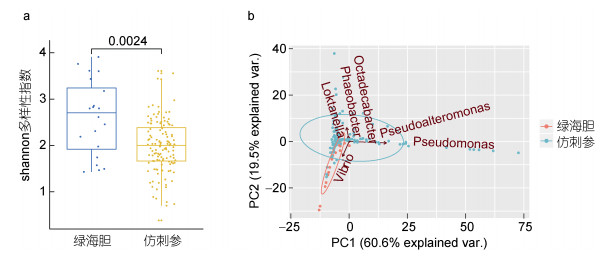 |
| 图 2 仿刺参与绿海胆肠道微生物的比较 Fig. 2 Comparison in intestinal microorganism between Apostichopus japonicus and L. variegatus 注: a.绿海胆与仿刺参肠道菌群的Alpha多样性; b.仿刺参与绿海胆肠道菌群的主成分分析, 基于两组中的优势分组菌属 |
对来自中国的42个、日本的20个和韩国的64个仿刺参肠道样本进行比较, 仿刺参肠道微生物Alpha多样性无显著差异(图 3a), 但是三个国家仿刺参肠道菌群结构各不相同(图 3b)。中国、日本和韩国仿刺参肠道微生物分别识别出299个、37个和139个菌属, 三个国家仿刺参肠道微生物共有菌属46个(图 3c)。尽管韩国来源的样本具有最大的样本量, 但来自中国的仿刺参肠道样本拥有最多的菌属数量, 表明样本量并非是决定肠道菌群中可识别微生物种类多少的根本因素, 而地理分布不同引起的水文、环境本底和海参摄食习性等因素的不同都可能是产生仿刺参肠道菌群地域间差异和聚簇效果的原因。
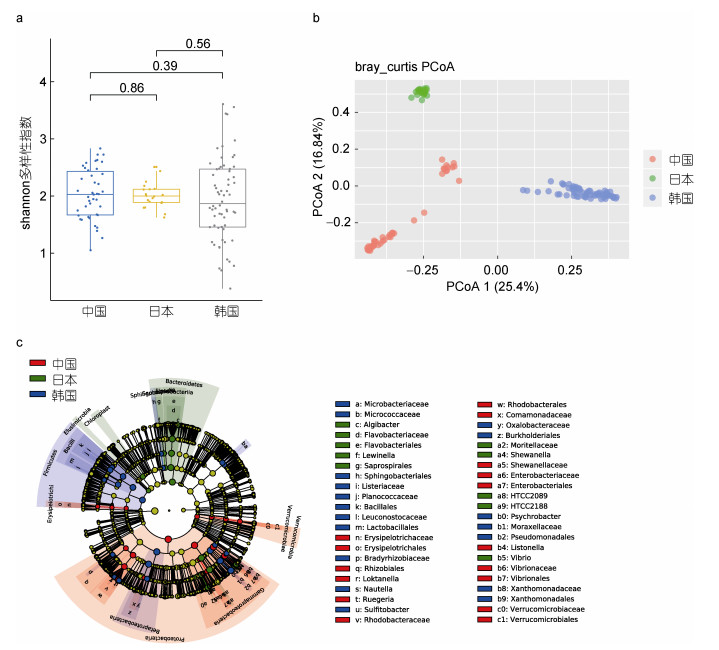 |
| 图 3 中国、日本和韩国仿刺参肠道微生物的比较 Fig. 3 Comparison in intestinal microorganism among Apostichopus japonicus from China, Japan, and Korea 注: a.中国、日本和韩国仿刺参肠道中微生物的Alpha多样性; b.中国、日本和韩国仿刺参肠道微生物基于BC距离的主坐标分析; c.中国、日本和韩国仿刺参肠道微生物的LEfSe分析 |
通过LEfSe分析得到三个国家仿刺参肠道显著差异的细菌, 同样体现出不同地域间的明显区别。其中中国的仿刺参肠道微生物特征菌为丹毒丝菌目(Erysipelotrichales)、根瘤菌目(Rhizobiales)、丹毒丝菌科(Erysipelotrichaceae)和陆丹氏菌属等; 日本的仿刺参肠道微生物为黄杆菌目(Flavobacteriales)、黄杆菌科(Flavobacteriaceae)和弧菌属等; 而韩国仿刺参肠道的特征微生物类别为鞘脂杆菌目(Sphingobacteriales)、微杆菌科(Microbacteriaceae)和微球菌科(Micrococcaceae)等(图 3d)。
2.4 中国不同地域仿刺参肠道微生物异同在本研究中, 中国仿刺参肠道样本来自黄海和渤海两个地域, 包含黄海仿刺参肠道样本12个, 渤海仿刺参肠道样本30个。黄海仿刺参肠道微生物多样性显著(P=0.00031)高于渤海(图 4a), 或与海区的开放程度有关。渤海和黄海仿刺参肠道菌群结构同样存在差异, 在主坐标分析(PCoA)中被分为彼此间隔的两组(图 4b)。对两海区样本进行比较, 渤海仿刺参肠道微生物主要富集利斯顿氏菌属、弓形菌属(Arcobacter)、盐单胞菌属(Halomonas)、Thalassomonas、弧菌属、发光菌属(Photobacterium)、Propionigenium、亮发菌属(Leucothrix)和Amaricoccus; 而黄海仿刺参肠道微生物主要富含Lutimonas、疣微菌属(Persicirhabdus)、鲁杰氏菌属(Ruegeria)、Robiginitalea、嗜碱海棍状菌属(Maritimibacter)、脱硫球菌属(Desulfococcus)、Donghicola、Dinoroseobacter、水栖菌属(Enhydrobacter)、聚球藻属(Synechococcus)和乳球菌属(Lactococcus)(图 4c)。
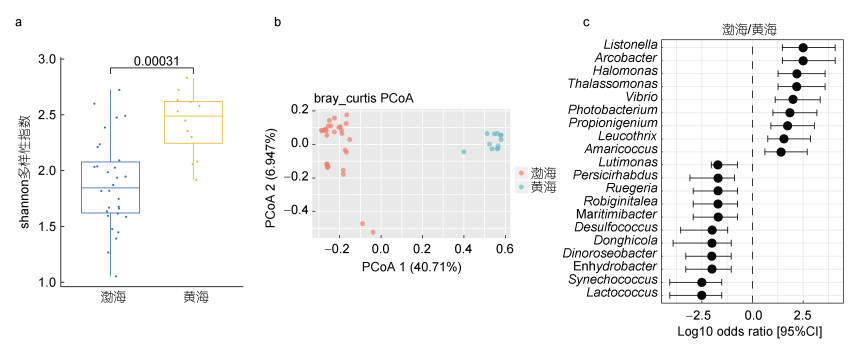 |
| 图 4 中国渤海与黄海仿刺参肠道菌群的异同 Fig. 4 Similarity and difference in intestinal flora between A. japonicus from the Bohai Sea and the Yellow Sea 注: a.渤海与黄海仿刺参肠道菌群的Alpha多样性; b.渤海与黄海仿刺参肠道菌群基于BC距离的主坐标分析; c.渤海与黄海仿刺参肠道菌群显著差异菌, 正值为渤海相对黄海显著富集, 负值为黄海相对渤海显著富集(P < 0.05) |
根据细菌分类列表对菌群代谢功能进行预测, 在三个国家仿刺参肠道微生物中, 发酵、化能异养和需氧化能异养功能均显著富集, 提示共生菌群在功能上存在共性, 并可能对宿主生理产生相同的作用。日本仿刺参肠道微生物光能自养功能较强, 而韩国仿刺参肠道微生物针对硫化合物呼吸作用、塑料降解作用和鱼类寄生虫相关的功能明显低于(P < 0.05)中国和日本(图 5a), 体现出不同国家仿刺参养殖群体在共生微生物功能上的区别。运用三个国家仿刺参肠道微生物中差异显著菌属进行相关性网络分析(图 5b), 结果同样发现假单胞菌属、嗜冷杆菌属、利斯顿氏菌属和弧菌属等细菌在相互作用网络中具有较高的连接度, 显示出这些细菌在仿刺参肠道菌群中可能发挥着非常重要的作用。其中假单胞菌属、乳球菌属与水产常见病原菌弧菌属呈强负相关, 提示可以通过增加两种菌属的数量实现抑制弧菌的效果, 利用生物方法控制水产疾病。
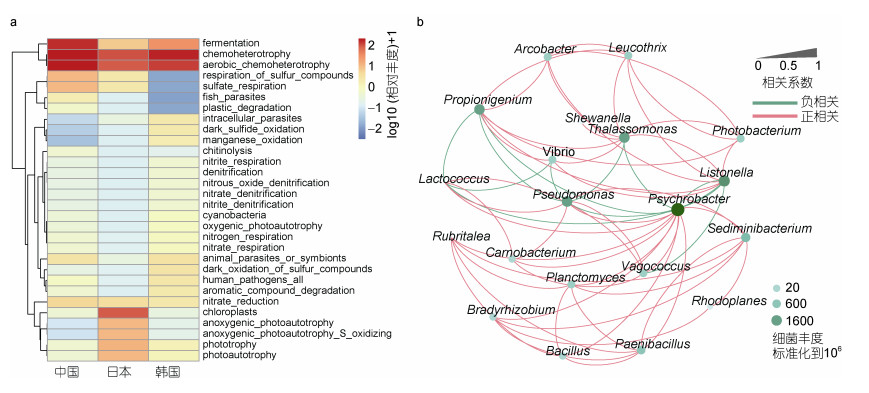 |
| 图 5 仿刺参肠道微生物相互关系与功能预测 Fig. 5 Correlation and function prediction of intestinal microorganisms in A. japonicus 注: a.三个国家仿刺参肠道微生物代谢功能预测, 将预测后的数据取log10后加1, 再进行聚; b.仿刺参肠道微生物的相互作用, 线的粗度表示相关系数的大小, 红色代表正相关, 绿色代表负相关, 点的大小表示菌的相对丰度(标准化至10万) |
近年来, 随着基因组测序技术的不断发展和数据的不断产生, 微生物组技术不仅被应用到致病菌的筛查, 还可通过组间共生菌群的差异分析, 寻找微生物与外界环境的相互作用, 以及揭示这些变化对宿主的生理、发育和免疫的影响(Chi et al, 2014; Xiong et al, 2015; Zhu et al, 2016), 积累数据为维护机体健康提供支撑。此前有研究表明肠道细菌群落与水生动物的健康直接相关(O'Hara et al, 2006)。另有对水产动物肠道微生物与生存环境的研究发现, 肠道与外界环境具有极高的菌群相似性(Zoqratt et al, 2018)。我们的研究表明, 仿刺参肠道与外界环境具有极高的共有菌比例, 特别是与海水菌群的相似度较高, 据此推测动物与水体之间存在着广泛的交流, 可能与养殖群体的健康密切相关。
本研究对三个国家的仿刺参进行分析, 显示出不同地域之间肠道菌群的差异。需要指出的是, 肠道微生物与物种生活环境戚戚相关, 不同环境会塑造不同的菌落结构(Kim et al, 2017); 由于日本样品来自于养殖群体, 所以不能排除该地域样本肠道菌群特征受到人工养殖因素影响的可能。对中国两个海区的仿刺参进行比较, 发现斯顿氏菌、弓形菌属、和弧菌属等在黄海仿刺参肠道菌群中显著高于渤海, 异养硝化菌属、疣微菌属和鲁杰氏菌属等在渤海中显著高于黄海, 进一步表明存在与海区分布相对应的特征菌群。嗜冷杆菌属在所有仿刺参肠道的富集与海参低温适应的习性可能存在一定的联系; 强化的发酵功能和化能异养功能可能是仿刺参肠道菌群的共性。这些结果为通过微生物组测序和菌群结构来判断海参地域和品系来源创造了可能, 由此也提示可以在物种迁移和养殖的过程中, 通过检测和分析地域水质和微生物组成, 最大程度避免仿刺参对养殖环境产生过度应激反应甚至发生养殖病害(Zhang et al, 2018)。除了了解仿刺参肠道微生物的组成及其与环境的交互外, 还应该识别和分离一些益生菌(Li et al, 2016; Li et al, 2016b; Zhang et al, 2016), 尝试通过补充某些特定的细菌来恢复有缺陷的功能, 或者提高宿主免疫力等(Li et al, 2016; Li et al, 2016a; Wang et al, 2017), 这些都是当前海参水产养殖病害防治方面亟待解决的问题(Li et al, 2019)。
在此前对海水的高通量测序中, 假单胞菌、乳球菌和弧菌等菌属均被发现广泛存在(肖怀东等, 2009; Saw et al, 2012)。在本研究中, 仿刺参与绿海胆肠道中也发现弧菌在菌群分组中发挥重要作用。值得注意的是, 弧菌在水产养殖过程中大多作为病原菌出现(Xue et al, 2017), 在虾类养殖中成为最大的隐患, 弧菌引起的细菌性传染病是造成水产养殖经济损失的主要原因(Torres et al, 2018)。本研究中呈现的假单胞菌和乳球菌在仿刺参肠道中富集, 且与弧菌存在强负相关性的结果, 或揭示了健康仿刺参维持肠道菌群稳定的方式, 也可为仿刺参水产养殖和健康管理提供参考, 即通过保持高丰度的特殊细菌的数量, 压制引起疾病的潜在病原菌的增长, 达到控制病害发生的目的。前人的研究中也发现海参肠道中存在大量的杆菌、乳酸菌和假单胞菌(Chi et al, 2014)。在水产养殖中, 这些属的大多数成员被当作益生菌, 用于对抗病原菌和刺激宿主免疫。本研究发现的显著富集菌属可能对仿刺参的生长具有一定的促进作用, 在后续研究中或可作为仿刺参养殖的益生菌加以发展。
4 结论我们在仿刺参肠道和周围环境中发现了许多常见细菌共存的现象, 提示它们之间可能存在密切的交互作用。此外, 本研究结果也表明了仿刺参与绿海胆的肠道菌群明显不同, 弧菌是区分仿刺参和绿海胆的特征性菌属; 而在仿刺参体内假单胞菌和乳酸菌与常见致病菌弧菌之间存在着强烈的负相关性, 提示前两者具有开发成为益生菌的潜力, 也可以通过特定细菌丰度的变化来判断仿刺参肠道健康状态, 将这些变化作为监测仿刺参感染的指标, 为改善养殖环境及促进动物健康生长提供支持。随着越来越多的测序数据公开, 整合分析将获得更多的线索和新的信息。虽然还需要进一步的研究来了解和阐明仿刺参共生微生物与宿主之间的关系, 但本研究为基于肠道共生菌群确定仿刺参地域分布以及病害防治创造了可能, 并为利用微生物组信息制定产品监测策略提供了有价值的参考。
肖怀东, 陈义光, 刘祝祥, 等. 2009. 湛江硇洲岛海葵相关可培养细菌系统发育多样性. 微生物学报, 49(2): 246-250 DOI:10.3321/j.issn:0001-6209.2009.02.017 |
Banerjee G, Ray A K, 2017. The advancement of probiotics research and its application in fish farming industries. Res Vet Sci, 115: 66-77 DOI:10.1016/j.rvsc.2017.01.016 |
Bronstein O, Kroh A, 2018. The first mitochondrial genome of the model echinoid Lytechinus variegatus and insights into Odontophoran phylogenetics. Genomics DOI:10.1016/j.ygeno.2018.04.008 |
Chen J H, Ren Y C, Li Y Q et al, 2018. Regulation of growth, intestinal microbiota, non-specific immune response and disease resistance of sea cucumber Apostichopus japonicus (Selenka) in biofloc systems. Fish Shellfish Immunol, 77: 175-186 DOI:10.1016/j.fsi.2018.03.053 |
Chi C, Liu J Y, Fei S Z et al, 2014. Effect of intestinal autochthonous probiotics isolated from the gut of sea cucumber (Apostichopus japonicus) on immune response and growth of A.japonicus. Fish Shellfish Immunol, 38(2): 367-373 DOI:10.1016/j.fsi.2014.04.001 |
Conand C, 2018. Tropical sea cucumber fisheries:changes during the last decade. Mar Pollut Bull, 133: 590-594 DOI:10.1016/j.marpolbul.2018.05.014 |
Coyte K Z, Schluter J, Foster K R, 2015. The ecology of the microbiome:networks, competition, and stability. Science, 350(6261): 663-666 DOI:10.1126/science.aad2602 |
Duan Y F, Zhang Y, Dong H B et al, 2017. Effect of the dietary probiotic Clostridium butyricum on growth, intestine antioxidant capacity and resistance to high temperature stress in kuruma shrimp Marsupenaeus japonicus. J Therm Biol, 66: 93-100 DOI:10.1016/j.jtherbio.2017.04.004 |
Endesfelder D, zu Castell W, Ardissone A et al, 2014. Compromised gut microbiota networks in children with anti-islet cell autoimmunity. Diabetes, 63(6): 2006-2014 DOI:10.2337/db13-1676 |
Fava F, Rizzetto L, Tuohy K M, 2019. Gut microbiota and health:connecting actors across the metabolic system. Proc Nutr Soc, 78(2): 177-188 DOI:10.1017/S0029665118002719 |
Gao Y, Wang J F, Zheng Y et al, 2016. Comprehensive identification of internal structure and alternative splicing events in circular RNAs. Nat Commun, 7: 12060 DOI:10.1038/ncomms12060 |
Giardine B, Riemer C, Hardison R C et al, 2005. Galaxy:a platform for interactive large-scale genome analysis. Genome Res, 15(10): 1451-1455 DOI:10.1101/gr.4086505 |
Gupta S, Fečkaninová A, Lokesh J et al, 2019. Lactobacillus dominate in the intestine of Atlantic salmon fed dietary probiotics. Front Microbiol, 9: 3247 DOI:10.3389/fmicb.2018.03247 |
Hai N V, 2015. The use of probiotics in aquaculture. J Appl Microbiol, 119(4): 917-935 DOI:10.1111/jam.12886 |
Hong H H, Choi H, Cheon S et al, 2017. Genome sequences of two Shewanella spp. isolated from the gut of the sea cucumber Apostichopus japonicus (Selenka, 1867). Genome Announc, 5(29): e00674-17 |
Huang S, Li R, Zeng X W et al, 2014. Predictive modeling of gingivitis severity and susceptibility via oral microbiota. ISME J, 8(9): 1768-1780 DOI:10.1038/ismej.2014.32 |
Jo J, Choi H, Lee S G et al, 2017. Draft genome sequences of Pseudoalteromonas tetraodonis CSB01KR and Pseudoalteromonas lipolytica CSB02KR, isolated from the gut of the sea cucumber Apostichopus japonicus. Genome Announc, 5(28): e00627-17 |
Kim T Y, Lee J J, Kim B S et al, 2017. Whole-body microbiota of sea cucumber (Apostichopus japonicus) from South Korea for improved seafood management. J Microbiol Biotechnol, 27(10): 1753-1762 DOI:10.4014/jmb.1707.07067 |
Lazado C C, Caipang C M A, 2014. Mucosal immunity and probiotics in fish. Fish Shellfish Immunol, 39(1): 78-89 DOI:10.1016/j.fsi.2014.04.015 |
Li L, Chen M Y, Storey K B, 2019. Metabolic response of longitudinal muscles to acute hypoxia in sea cucumber Apostichopus japonicus (Selenka):a metabolome integrated analysis. Comp Biochem Physiol Part D:Genom Proteomics, 29: 235-244 DOI:10.1016/j.cbd.2018.12.007 |
Li X Y, Jing K L, Wang X T et al, 2016a. Protective effects of chicken egg yolk antibody (IgY) against experimental Vibrio splendidus infection in the sea cucumber (Apostichopus japonicus). Fish Shellfish Immunol, 48: 105-111 DOI:10.1016/j.fsi.2015.11.024 |
Li Z, Li X Y, Zhang J C et al, 2016b. Use of phages to control Vibrio splendidus infection in the juvenile sea cucumber Apostichopus japonicus. Fish Shellfish Immunol, 54: 302-311 DOI:10.1016/j.fsi.2016.04.026 |
Li C, Ren Y C, Jiang S H et al, 2018. Effects of dietary supplementation of four strains of lactic acid bacteria on growth, immune-related response and genes expression of the juvenile sea cucumber Apostichopus japonicus Selenka. Fish Shellfish Immunol, 74: 69-75 DOI:10.1016/j.fsi.2017.12.037 |
Louca S, Parfrey L W, Doebeli M, 2016. Decoupling function and taxonomy in the global ocean microbiome. Science, 353(6305): 1272-1277 DOI:10.1126/science.aaf4507 |
Magoč T, Salzberg S L, 2011. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 27(21): 2957-2963 DOI:10.1093/bioinformatics/btr507 |
Mashjoor S, Yousefzadi M, 2017. Holothurians antifungal and antibacterial activity to human pathogens in the Persian Gulf. J Mycol Méd, 27(1): 46-56 DOI:10.1016/j.mycmed.2016.08.008 |
O'Hara A M, Shanahan F, 2006. The gut flora as a forgotten organ. EMBO Rep, 7(7): 688-693 DOI:10.1038/sj.embor.7400731 |
Quast C, Pruesse E, Yilmaz P et al, 2013. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools. Nucleic Acids Res, 41(D1): D590-D596 |
Quiroz-Guzmán E, Vazquez-Juárez R, Luna-González A et al, 2018. Administration of probiotics improves the brine shrimp production and prevents detrimental effects of pathogenic Vibrio species. Mar Biotechnol (NY), 20(4): 512-519 DOI:10.1007/s10126-018-9822-8 |
Ratana-Arporn P, Jommark N, 2014. Efficacy of neutral electrolyzed water for reducing pathogenic bacteria contaminating shrimp. J Food Prot, 77(12): 2176-2180 DOI:10.4315/0362-028X.JFP-14-161 |
Roux S, Brum J R, Dutilh B E et al, 2016. Ecogenomics and potential biogeochemical impacts of globally abundant ocean viruses. Nature, 537(7622): 689-693 DOI:10.1038/nature19366 |
Saw J H W, Yuryev A, Kanbe M et al, 2012. Complete genome sequencing and analysis of Saprospira grandis str. Lewin, a predatory marine bacterium. Stand Genomic Sci, 6(1): 84-93 |
Torres M, Reina J C, Fuentes-Monteverde J C et al, 2018. AHL-lactonase expression in three marine emerging pathogenic Vibrio spp. reduces virulence and mortality in brine shrimp (Artemia salina) and Manila clam (Venerupis philippinarum). PLoS One, 13(4): e0195176 DOI:10.1371/journal.pone.0195176 |
Tunca E, Aydın M, Şahin, 2016. Interactions and accumulation differences of metal(loid)s in three sea cucumber species collected from the Northern Mediterranean Sea. Environ Sci Pollut Res Int, 23(20): 21020-21031 DOI:10.1007/s11356-016-7288-7 |
Wang J F, Gao Y, Zhao F Q, 2016. Phage-bacteria interaction network in human oral microbiome. Environ Microbiol, 18(7): 2143-2158 DOI:10.1111/1462-2920.12923 |
Wang H D, Liu S K, Cui J et al, 2015. Identification and characterization of microRNAs from longitudinal muscle and respiratory tree in sea cucumber (Apostichopus japonicus) using high-throughput sequencing. PLoS One, 10(8): e0134899 DOI:10.1371/journal.pone.0134899 |
Wang X T, Sun Y X, Wang L L et al, 2017. Synbiotic dietary supplement affects growth, immune responses and intestinal microbiota of Apostichopus japonicus. Fish Shellfish Immunol, 68: 232-242 DOI:10.1016/j.fsi.2017.07.027 |
Wang H, Xue Z, Liu Z Q et al, 2018. A novel C-type lectin from the sea cucumber Apostichopus japonicus (AjCTL-2) with preferential binding of D-galactose. Fish Shellfish Immunol, 79: 218-227 DOI:10.1016/j.fsi.2018.05.021 |
Xiong J B, Wang K, Wu J F et al, 2015. Changes in intestinal bacterial communities are closely associated with shrimp disease severity. Appl Microbiol Biotechnol, 99(16): 6911-6919 DOI:10.1007/s00253-015-6632-z |
Xue S X, Xu W, Wei J L et al, 2017. Impact of environmental bacterial communities on fish health in marine recirculating aquaculture systems. Vet Microbiol, 203: 34-39 DOI:10.1016/j.vetmic.2017.01.034 |
Yamazaki Y, Meirelles P M, Mino S et al, 2016. Individual Apostichopus japonicus fecal microbiome reveals a link with polyhydroxybutyrate producers in host growth gaps. Sci Rep, 6: 21631 DOI:10.1038/srep21631 |
Yang G, Peng M, Tian X L et al, 2017. Molecular ecological network analysis reveals the effects of probiotics and florfenicol on intestinal microbiota homeostasis:an example of sea cucumber. Sci Rep, 7(1): 4778 DOI:10.1038/s41598-017-05312-1 |
Zakrzewski M, Proietti C, Ellis J J et al, 2017. Calypso:a user-friendly web-server for mining and visualizing microbiome-environment interactions. Bioinformatics, 33(5): 782-783 |
Zhang W W, Liang W K, Li C H, 2016. Inhibition of marine Vibrio sp. by pyoverdine from Pseudomonas aeruginosa PA1. J Hazard Mater, 302: 217-224 DOI:10.1016/j.jhazmat.2015.10.003 |
Zhang Z, Xing R L, Lv Z M et al, 2018. Analysis of gut microbiota revealed Lactococcus garviaeae could be an indicative of skin ulceration syndrome in farmed sea cucumber Apostichopus japonicus. Fish Shellfish Immunol, 80: 148-154 DOI:10.1016/j.fsi.2018.06.001 |
Zhu J Y, Dai W F, Qiu Q F et al, 2016. Contrasting ecological processes and functional compositions between intestinal bacterial community in healthy and diseased shrimp. Microb Ecol, 72(4): 975-985 DOI:10.1007/s00248-016-0831-8 |
Zhu Z J, Zhu B W, Sun Y J et al, 2018. Sulfated polysaccharide from sea cucumber modulates the gut microbiota and its metabolites in normal mice. Int J Biol Macromol, 120: 502-512 DOI:10.1016/j.ijbiomac.2018.08.098 |
Zoqratt M Z H M, Eng W W H, Thai B T et al, 2018. Microbiome analysis of Pacific white shrimp gut and rearing water from Malaysia and Vietnam:implications for aquaculture research and management. PeerJ, 6: e5826 DOI:10.7717/peerj.5826 |
 2019, Vol. 50
2019, Vol. 50


