
中国海洋湖沼学会主办。
文章信息
- 邓惠文, 刘峰, 李桂豪, 包卫洋, 邹松保, 郑鹏飞, 龚骏, 孙汝江. 2019.
- DENG Hui-Wen, LIU Feng, LI Gui-Hao, BAO Wei-Yang, ZOU Song-Bao, ZHENG Peng-Fei, GONG Jun, SUN Ru-Jiang. 2019.
- 黑鲷(Acanthopagrus schlegelii)肠道细菌群落对饲料中添加不同羽毛粉的响应
- RESPONSE OF GUT BACTERIAL COMMUNITY IN BLACK SEA BREAM TO DIFFERENT FEATHER MEAL IN FEED
- 海洋与湖沼, 50(6): 1309-1317
- Oceanologia et Limnologia Sinica, 50(6): 1309-1317.
- http://dx.doi.org/10.11693/hyhz20190400072
文章历史
-
收稿日期:2019-04-12
收修改稿日期:2019-07-08
2. 南方海洋科学与工程广东省实验室 珠海 519000;
3. 中国农业大学烟台研究院 烟台 264670;
4. 大连海洋大学水产与生命学院 大连 116023;
5. 中国科学院烟台海岸带研究所 烟台 264003
2. Southern Laboratory of Ocean Science and Engineering(Guangdong, Zhuhai), Zhuhai 519000, China;
3. Yantai Research Institute, China Agricultural University, Yantai 264670, China;
4. College of Fisheries and Life Science, Dalian Ocean University, Dalian 116023, China;
5. Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai 264003, China
鱼类肠道内的细菌群落, 尤其是定植的固有菌群, 是鱼体不可缺少的重要组成部分, 它和宿主鱼体之间相互作用形成了一个整体的、复杂的系统, 鱼类肠道菌群在提高消化、营养物质的吸收效率和提高宿主的免疫防御功能方面具有重要作用(Austin, 2006; Round et al, 2009; Nayak, 2010; Wei et al, 2010)。在鱼类肠道中, 有相当一部分的细菌来自于周围的水环境、土壤或沉积物环境及其所食用的饵料中(Nayak, 2010)。同一种鱼类的肠道菌群结构和多样性特征还会因其生长阶段、水体环境、摄食等因素的不同而发生改变(McDonald et al, 2012; Wong et al, 2012; Bolnick et al, 2014; Miyake et al, 2015; 刘增新, 2017)。已有的研究也多次证实不同饲料组分可能导致肠道微生物群落组成的变化(Ingle, 2013; 钟雷等, 2014; Apper et al, 2016; Huyben, 2017)。
近年来, 有关植物蛋白源(例如:玉米蛋白粉、棉粕、发酵豆粕、脱脂豆粕、生物加工后的豆粕、商业大豆浓缩蛋白等)(彭翔等, 2012; 王赛等, 2012; 杨彬彬等, 2015; Michl et al, 2017)、动物蛋白源(例如:桡足动物粉、啤酒酵母、脱脂蚕蛹、羽毛粉等动物蛋白源)(王赛等, 2012; 付文忠, 2014; 刘峰等, 2017)替代鱼粉在水产养殖的应用与评价研究中不断涌现, 其中, 羽毛粉通常由禽畜羽毛和抽绒剩余下的羽毛梗加工而成, 是一种氨基酸种类比较齐全、含维生素B12的具有较高营养价值的饲料蛋白源(王裕玉等, 2013)。据统计, 我国年废弃羽毛近150万吨(陈盈霖等, 2017), 在水产动物饲料中添加适量的羽毛粉和氨基酸, 不仅可以减少鱼粉用量, 降低饲料成本, 提高经济效益, 而且能提高资源利用率, 减少环境污染(王裕玉等, 2013; 郭爱红, 2014)。羽毛粉的化学结构中较多的二硫键使其物理与化学性质相对稳定, 动物自身分泌的酶类基本上无法将其水解, 因此, 必须对羽毛粉进行高温、高压、酸、碱或酶的加工处理, 变成短肽或游离氨基酸, 然后通过动物肠道中的微生物对其降解以提高羽毛粉的使用效率(周凡等, 2012)。除了通过研究鱼类的生长指标和血清生化指标来评价饲料替代品对鱼体生理、生化的影响, 鱼类肠道微生物多样性及群落结构的研究也能为水产饲料研究与应用提供一个新的视角, 如评价鱼类新型配合饲料所取得的效果、潜在益生型菌株的添加效果等(王纯, 2017)。然而, 目前对饲料添加物对鱼类肠道微生物多样性及群落结构影响的研究还相对较少(钟雷等, 2014; Huyben, 2017; Michl et al, 2017), 羽毛粉添加饲料对鱼类肠道微生物的影响研究还未见报道。
黑鲷(black sea bream, Acanthopagrus schlegelii)属于硬骨鱼纲、鲈形目、鲷科、鲷属, 广泛分布于我国东南沿海、韩国、日本及东南亚等地, 因其具有广盐广温、杂食性强、生长快、抗病力强等多种生物学特性, 黑鲷已经成为了我国网箱养殖的重要经济鱼类品种(周凡, 2011; 刘峰等, 2017)。但由于黑鲷市场需求量过高, 野生黑鲷资源日益衰减、捕获量下降, 因此积极发展黑鲷的人工养殖成为了必然趋势, 研制高效低成本的环境友好型黑鲷配合饲料已成为当务之急。本研究将以黑鲷为实验对象, 利用16S rRNA基因测序技术, 首次从肠道细菌群落结构和多样性的角度出发, 探究饲料中羽毛粉的有无及不同加工方式羽毛粉的差异对黑鲷肠道细菌产生的影响; 此外, 结合刘峰等(2017)的研究结果, 进一步联系了黑鲷肠道细菌群落特点与生长及血液生化指标的潜在相关关系, 以期为海水养殖鱼类肠道益生菌的鉴定与筛选、鱼类饲料添加剂的开发等提供一份参考资料。
1 材料与方法 1.1 样品采集本研究中选用的实验动物样本同刘峰等(2017)。黑鲷(Acanthopagrus schlegelii)由山东省烟台开发区天源水产有限公司提供, 在中国农业大学烟台研究院的室内循环水养殖系统中饲养12周后, 随机捞取黑鲷个体解剖获取粪便。按所投喂饲料的来源与处理方式, 将实验对象分为6个处理组, 根据是否添加羽毛粉将6个处理组又分对照组(NF)和实验组(F); 对照组包括2种无任何羽毛粉添加的鱼粉组(FN)和商业组(FS), 实验组包括4种以不同方式处理羽毛粉后添加到饲料中的生羽毛粉组(FO)、水解羽毛粉组(FH)、酸解羽毛粉组(FA)、膨化羽毛粉组(FP)。添加羽毛粉的比例均为5%, 其中生羽毛粉组添加的羽毛粉未经任何加工处理; 经高温高压条件下蒸汽水解成块状蛋白质凝胶, 经烘干、粉碎后制成水解羽毛粉; 利用酸性物质使羽毛角蛋白的二硫键断裂后的羽毛粉称为酸解羽毛粉; 在膨化机高温高压和高剪切作用下, 角质蛋白牢固空间结构已被破坏的称为膨化羽毛粉。每个处理组含有4个养殖水桶(重复), 每桶含黑鲷30尾。对每个处理组解剖3—8尾, 剪开肠道, 获取并合并尚未排出的肠道内容物, 达到每组至少0.50g的内容物。
1.2 黑鲷粪便细菌总DNA的提取、扩增和测序黑鲷粪便样品的总DNA采用快速核酸提取试剂盒(FastDNA® Spin Kit for Soil, MP Biomedicals)提取, DNA的浓度和纯度用微量紫外分光光度计进行测定并记录。提取获得的总DNA于–80℃冰箱保存备用。然后利用原核生物通用引物515F (5′-GTGCCAGC MGCCGCGG)和806R (5′-GGACTACHVGGGTWTC TAAT) (Caporaso et al, 2012)对16S rDNA的V4区进行扩增; 使用Illumina MiSeq PE250进行高通量测序(北京诺禾致源)。
1.3 数据处理与统计分析用QIIME v.1.8.0(Caporaso et al, 2010)对MiSeq下机数据进行序列拼接、样品标记条纹码的提取、割库、嵌合体的去除, 用MOTHUR软件包v.1.34.4(Schloss et al, 2009)去除其引物、同聚体, 然后得到高质量的序列, 再用UCLUST v.1.2.2以97%的相似性水平划分操作分类单元(OTUs), 去除只含有1条序列的OTU, 然后去除其中的古菌、叶绿体和线粒体16S rRNA基因序列, 选取剩余的OTUs代表序列与数据库Greengenes(版本gg_13_8)进行比对, 从而进行物种分类。
在OTU表基础上, 根据最小样品序列数设定重采样序列最大值为3320, 计算各个处理组中细菌群落阿尔法多样性指数和各主要细菌类群在群落中所占的相对丰度, 阿尔法多样性指数包括丰富度(OTU richness)、Chao1指数(Chao1 index)、辛普森指数(Simpson index)、香农指数(Shannon index); 构建基于Bray-Curtis的群落距离矩阵, 群落距离的可视化通过R软件中的非计量多维尺度分析(non-metric multidimensional scaling, NMDS)来进行, 并利用相似性分析(Analysis of Similarity, ANOSIM)检验样品组间细菌群落结构差异是否显著(α=0.050)。利用SPSS 24.0软件, 采用曼-惠特尼检验(Mann-Whitney U test)比较对照组和实验组主要菌群的群落阿尔法多样性指数和主要种群的相对丰度的平均数; 采用克鲁斯卡尔-沃利斯检验(Kruskal-Wallis test)比较鱼粉组、生羽毛粉组和水解羽毛粉组的群落阿尔法多样性指数和主要种群的相对丰度的平均数(α=0.050)。
有关黑鲷生长的参数和血液生化指标均已在前文中发表(刘峰等, 2017)。
2 结果 2.1 黑鲷肠道细菌的阿尔法多样性利用MiSeq高通量测序平台对24个样品中的17个样品成功测序:鱼粉组(4个重复)、商业组(2个重复)、生羽毛粉组(3个重复)、水解羽毛粉组(4个重复)、酸解羽毛粉组(2个重复)和膨化羽毛粉组(2个重复), 共得到原始序列1009063条。经过质量控制后得到569148条高质量序列, 去除古菌、叶绿体和线粒体序列后得到560509条序列(表 1)。
| 样品 | 原始序列 | 修整序列 | 去古菌、线粒体和叶绿体后的剩余序列 |
| 鱼粉组样品1(FN1) | 55203 | 35425 | 35181 |
| 鱼粉组样品2(FN2) | 57659 | 35823 | 33142 |
| 鱼粉组样品3(FN3) | 62892 | 31738 | 30261 |
| 鱼粉组样品4(FN4) | 60998 | 32124 | 31517 |
| 商业饲料组样品2(FS2) | 62906 | 3380 | 3320 |
| 商业饲料组样品4(FS4) | 59339 | 10001 | 9856 |
| 生羽毛粉组样品1(FO1) | 70280 | 45747 | 45558 |
| 生羽毛粉组样品2(FO2) | 56470 | 36647 | 36424 |
| 生羽毛粉组样品4(FO4) | 64511 | 45184 | 45097 |
| 水解羽毛粉组样品1(FH1) | 58713 | 39551 | 39319 |
| 水解羽毛粉组样品2(FH2) | 65416 | 39395 | 38926 |
| 水解羽毛粉组样品3(FH3) | 48456 | 32773 | 32601 |
| 水解羽毛粉组样品4(FH4) | 53843 | 36641 | 36488 |
| 酸解羽毛粉组样品3(FA3) | 66556 | 44481 | 44306 |
| 酸解羽毛粉组样品4(FA4) | 44633 | 26479 | 25573 |
| 膨化羽毛粉组样品2(FP2) | 56115 | 33765 | 33538 |
| 膨化羽毛粉组样品4(FP4) | 65073 | 39994 | 39670 |
总体来看, 羽毛粉实验组中黑鲷肠道细菌的多样性明显低于对照组(图 1)。实验组丰富度平均值与标准差为285±174, 丰富度均值比对照组低了约22%。同样地, 除了Chao1指数外(P=0.216), 实验组的辛普森指数和香农指数均显著地低于对照组(P < 0.05)。就鱼粉、生羽毛粉、水解羽毛粉这三个组的比较来看:鱼粉组的肠道细菌多样性略高于水解羽毛粉组, 生羽毛粉组多样性最低, 但统计结果表明这三个组细菌多样性的差异都没有达到显著性水平(P > 0.05, 图 1)。
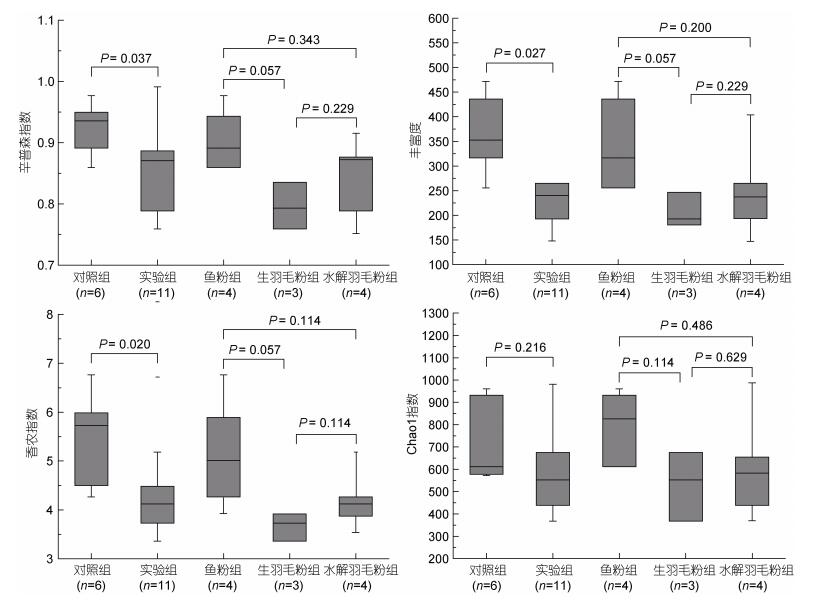 |
| 图 1 箱线图示实验组(添加羽毛粉)与对照组之间, 以及鱼粉组、生羽毛粉组和水解羽毛粉组之间黑鲷肠道细菌多样性指数的比较 Fig. 1 Comparisons of α-diversity estimators of gut bacteria between the feather-amended treatments and the control, and among the three treatments, the fish meal, raw feather and hydrolyzed feather |
NMDS排序图显示, 实验组与对照组黑鲷肠道细菌群落结构有一定的区别, 但其中三个鱼粉组样品(FN1-3)与实验组样品混杂在一起(图 2)。ANOSIM检验结果也显示:实验组黑鲷肠道细菌群落结构与对照组的差异不显著(R=0.121, P=0.126)。在NMDS排序图中, 酸解羽毛粉组的两个样品(FA3、FA4)位于实验组样品的外缘(具有较为不同的群落结构)(图 2); 当将这两个酸解羽毛粉样品剔除后, 再次比较实验组与对照组的细菌群落结构时, 结果显示差异显著(R=0.264, P=0.033), 即使用添加有5%生羽毛粉、水解羽毛粉、膨化羽毛粉的饲料喂养黑鲷12周后, 发现黑鲷肠道细菌群落结构发生了显著性变化。
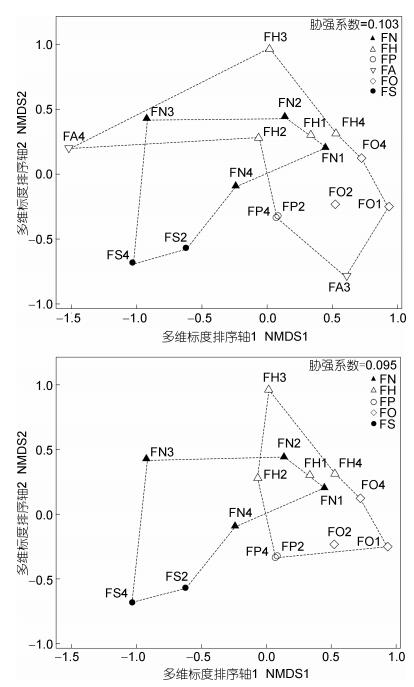 |
| 图 2 黑鲷肠道细菌群落结构非度量多维定标图 Fig. 2 Differences in the intestinal bacterial community structure of the black sea bream revealed by non-metric multidimensional scaling 注:虚线连接的空心标志为添加有5%生羽毛粉(FO)、水解羽毛粉(FH)、膨化羽毛粉(FP)、酸解羽毛粉(FA)的饲料喂养黑鲷对应的肠道细菌群落, 虚线连接的实心标志为鱼粉(FN)与商业饲料(FS)喂养后对应的肠道细菌群落 |
在17个黑鲷肠道样品共检测到37个细菌门(图 3), 主要类群为厚壁菌门(Firmicutes; 57.3%)、变形菌门(Proteobacteria; 36.3%)、放线菌门(Actinobacteria; 2.4%)。其中, 相对丰度最高的厚壁菌主要包括52.1%的芽胞杆菌(Bacilli)和5.1%的梭菌(Clostridia), 变形菌则主要包括26.3%的γ-变形菌(Gammaproteobacteria)和5.6%的α-变形菌(Alphaproteobacteria)。
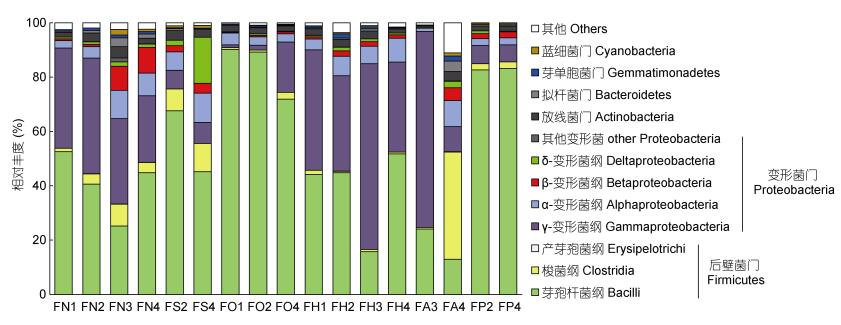 |
| 图 3 以添加不同羽毛粉的饲料(FO、FH、FA、FP)与无羽毛粉添加的饲料(FN、FS)喂养后黑鲷肠道细菌主要类群的组成及分布 Fig. 3 Community composition and distributions of major bacterial taxa in the gut of black sea breams fed with feather-amended or non-feather meals |
与对照组相比, 处理组中细菌群落中梭菌纲(平均4.7% vs. 5.9%)、δ-变形菌纲(0.8% vs. 3.9%)、拟杆菌纲(0.4% vs. 0.8%)、草酸杆菌科(0.8% vs. 3.2%)和青枯菌属(Ralstonia, 0.7% vs. 3.0%)的相对丰度显著降低(表 2)。而在实验组中, 添加不同类型的羽毛粉也导致了肠道细菌群落的结构变化:相对于水解羽毛粉组, 生羽毛粉组中的变形菌门(11.5% vs. 54.2%)、γ-变形菌纲(7.1% vs. 45.2%)、海洋螺菌目(4.7% vs. 25.2%)、盐单胞菌科(4.7% vs. 25.2%)和盐单胞菌属(4.7% vs. 25.2%)的相对丰度都显著降低。在比较生羽毛粉组与鱼粉组的细菌群落时, 生羽毛粉组芽孢杆菌(14.0% vs. 4.5%)的相对丰度显著升高, 而拟杆菌(0.1% vs. 1.6%)、草酸杆菌(0.2% vs. 3.6%)和青枯菌属(0.2% vs. 3.3%)的相对丰度显著降低(表 2)。
| 分类 | 组别 | |||||
| 对照组(n=6) | 实验组(n=11) | 鱼粉组(n=4)FN | 生羽毛粉组(n=3)FO | 水解羽毛粉组(n=4)FH | P | |
| Firmicutes | 51.80±14.22 | 60.26±26.44 | 44.91±8.93 | 85.04±9.72 | 40.01±15.95 | 0.042 |
| Clostridia | 5.87±3.45a | 4.74±11.51b | 4.19±2.77 | 1.30±1.03 | 0.91±0.46 | 0.068 |
| Bacilli | 45.92±14.08 | 55.50±29.58 | 40.68±11.68 | 83.74±10.29 | 39.10±15.96 | 0.049 |
| Lactobacillales | 36.32±12.78 | 47.26±25.57 | 36.07±11.11 | 69.76±7.46 | 34.31±15.45 | 0.050 |
| Leuconostocaceae | 19.95±8.76 | 28.74±16.43 | 20.33±9.73 | 44.43±6.75 | 20.68±10.47 | 0.050 |
| Weissella | 14.97±6.55 | 21.97±13.86 | 15.16±7.37 | 40.02±5.54 | 15.66±8.76 | 0.050 |
| Lactobacillaceae | 15.09±4.74 | 17.92±9.57 | 14.65±2.12 | 24.97±0.83 | 12.80±5.49 | 0.049 |
| Lactobacillus | 7.58±2.80 | 7.75±4.53 | 7.66±2.64 | 7.70±0.21 | 6.08±2.10 | 0.462 |
| Bacillales | 9.51±8.16 | 8.12±4.75 | 4.49±0.84a | 13.98±3.15b | 4.77±0.56ab | 0.041 |
| Bacillaceae | 5.49±6.82 | 2.60±1.54 | 1.47±0.57 | 3.73±0.84 | 1.52±0.29 | 0.045 |
| Bacillus | 1.99±1.67 | 2.00±1.29 | 1.07±0.55 | 3.33±0.82 | 1.02±0.29 | 0.050 |
| Planococcaceae | 0.45±0.48 | 0.75±0.80 | 0.14±0.05 | 1.95±0.21 | 0.16±0.10 | 0.049 |
| Proteobacteria | 40.55±12.17 | 34.00±25.92 | 46.55±5.32ab | 11.46±9.61a | 54.16±15.77b | 0.041 |
| Gammaproteobacteria | 24.95±14.89 | 26.97±25.86 | 33.75±7.75ab | 7.13±9.88a | 45.22±16.19b | 0.035 |
| Oceanospirillales | 4.96±5.97 | 10.58±13.42 | 7.21±6.26ab | 4.68±7.79a | 25.19±10.09b | 0.024 |
| Halomonadaceae | 4.73±6.10 | 10.53±13.45 | 6.91±6.57ab | 4.66±7.80a | 25.17±10.10b | 0.024 |
| Halomonas | 4.73±6.10 | 10.52±13.45 | 6.90±6.57ab | 4.66±7.81a | 25.16±10.11b | 0.024 |
| Alphaproteobacteria | 7.17±3.24 | 4.78±2.78 | 6.39±3.51 | 3.50±0.73 | 6.57±2.01 | 0.342 |
| Betaproteobacteria | 4.24±3.86 | 1.47±1.31 | 4.88±4.79 | 0.47±0.26 | 1.45±0.52 | 0.080 |
| Burkholderiales | 3.98±3.69 | 1.15±1.11 | 4.48±4.62 | 0.33±0.24 | 0.95±0.26 | 0.184 |
| Oxalobacteraceae | 3.23±3.05a | 0.80±0.92b | 3.64±3.83a | 0.19±0.08b | 0.55±0.28ab | 0.041 |
| Ralstonia | 2.96±2.74a | 0.73±0.82b | 3.28±3.45a | 0.18±0.08b | 0.51±0.27ab | 0.041 |
| Deltaproteobacteria | 3.92±6.40a | 0.76±0.65b | 1.13±0.39 | 0.35±0.20 | 0.88±0.37 | 0.051 |
| Actinobacteria | 2.90±0.90 | 2.14±0.79 | 2.73±1.08 | 2.19±0.26 | 2.38±0.63 | 0.700 |
| Actinobacteria | 2.30±0.86 | 1.61±0.49 | 2.01±0.86 | 1.91±0.29 | 1.69±0.34 | 0.719 |
| Bacteroidetes | 1.21±1.08 | 0.64±1.07 | 1.56±1.20a | 0.12±0.06b | 0.45±0.31ab | 0.041 |
| Bacteroidia | 0.81±1.03a | 0.36±0.96b | 1.15±1.15 | 0.04±0.06 | 0.03±0.04 | 0.030 |
| 注:加粗字体表示显著性差异(P < 0.05) | ||||||
将厚壁菌与变形菌的相对丰度的比值(F/P)作为一个群落结构特征进行组间比较后发现:实验组F/P比值平均为4.93, 似乎高于对照组(平均为1.60), 但两组之间的差异并不显著(P=0.462)。相对于鱼粉组和水解羽毛粉组, 生羽毛粉组F/P比值最高(平均为7.42), 高于鱼粉组(0.97)与水解羽毛粉组(0.74)且均非常接近显著性水平(P=0.057)。
3 讨论 3.1 饲料中添加羽毛粉导致黑鲷肠道细菌阿尔法多样性降低我们的比较结果表明:使用添加5%羽毛粉的喂养黑鲷12周后, 黑鲷肠道细菌多样性呈现明显的下降趋势。该现象与前人对植物蛋白添加后导致鱼类肠道多样性下降的结果非常类似。Michl等(2017)用较高比例的植物蛋白源替代鱼粉投喂虹鳟(Oncorhynchus mykiss)后发现, 虹鳟肠道细菌群落的丰富度、辛普森指数、香农指数和Chao1指数均显著降低。鱼类肠道细菌多样性的降低这一现象可能与饲料中所含的氨基酸种类、含量及比例有关:实验组中, 未经处理的生羽毛粉富含角蛋白, 它几乎无法被鱼类消化酶分解, 所以其消化利用率最低, 而不同处理方法在生羽毛粉的基础上, 将角蛋白处理成了动物可消化吸收的可溶性蛋白(陈盈霖等, 2017), 一定程度上提高了羽毛粉的消化利用率。但即便如此, 实验组的营养物质消化利用率依然低于对照组, 因为鱼粉比羽毛粉具有更高的脂肪酸含量和氨基酸种类, 是水产饲料中不可完全替代的更优质的蛋白源(李弋等, 2017)。因此我们认为羽毛粉的替代会导致可利用的营养物质减少, 营养物质的利用难度增加, 所以实验组细菌群落的丰富度、辛普森指数和香农指数发生了显著下降。
3.2 黑鲷肠道细菌群落组成前人研究发现鱼类肠道菌群中厚壁菌门与变形菌门相对丰度的比值上升与添加饲料种类和鱼体营养状态有关, 例如以含30%大豆蛋白的饲料投喂虹鳟(Desai et al, 2012)及饥饿处理大鲵(许超, 2017)后会产生这种变化。此外还有研究发现水解5min后的生羽毛粉, 其脂质消化率可低至40%(Bureau et al, 1999)。由此我们推测以生羽毛粉替代5%鱼粉蛋白时, 由于生羽毛粉本身消化利用率低, 所以生羽毛粉组黑鲷所能利用的营养物质无法满足其所需, 营养状态较差, 因此导致了生羽毛粉组的厚壁菌门与变形菌门相对丰度的比值高于鱼粉组和水解羽毛粉组。
芽孢杆菌属(Bacillus)被认为对角蛋白酶的产生起着重要作用, 是细菌中主要的产角蛋白酶类群(侯玉煌等, 2018; 文冰洁, 2018)。其相对丰度在生羽毛粉组、鱼粉组和水解羽毛粉组中具有显著性差异且在生羽毛粉组中相对丰度最高, 这表明它在生羽毛粉组富含角蛋白的情况下能够优先利用角蛋白促进自身生长。
乳酸菌被认为是潜在益生菌, 而乳酸杆菌目在生羽毛粉组中的相对丰度却远大于水解组和鱼粉组。这一现象与Desai等(2012)发现一致, 他们发现添加不同的植物蛋白源会导致虹鳟远端肠道中潜在有益类群的相对丰度在不利于虹鳟的生长的情况下出现升高; 另外, Ringø(1993)研究发现饲料中多不饱和脂肪酸可能会抑制乳酸菌的生长, 由此推测生羽毛粉可能含有更多的多不饱和脂肪酸。
3.3 黑鲷肠道细菌在各分类水平上的群落分布与生产性能及血液生化指标的相关关系与水解羽毛粉组相比, 鱼粉组盐单胞菌属(Halomonas, 6.9% vs. 25.2%)和回线属(Treponema)的相对丰度显著降低(P=0.029), 鱼粉组对应的谷草转氨酶(225 vs. 407 U/L)和谷丙转氨酶活性(15 vs. 26 U/L)也发生显著降低(刘峰等, 2017), 而鱼粉组的紫单胞菌科(Porphyromonadaceae, 0.1% vs. 0.0%)相对丰度显著上升(P=0.029)且鱼粉组对应的特定生长率、饲料系数、肥满度、总胆固醇、总蛋白质、溶菌酶、超氧化物歧化酶7项指标也发生了显著升高(刘峰等, 2017)。紫单胞菌科属于拟杆菌目(Bacteroidales), 前人在探索人类肠道菌群作用时发现拟杆菌在帮助宿主分解营养物质、提高营养利用率及维持肠道微生态平衡等方面具有着重要作用(Bäckhed et al, 2004; Sears, 2005), 因此我们推测鱼粉组黑鲷更好的生产性能与其肠道内紫单胞菌科相对丰度的显著提高直接相关。另一方面, 已知高温高压水解法易造成热敏氨基酸(半胱氨酸、赖氨酸等)的损失(王裕玉等, 2013; 刘峰等, 2017), 基于周凡(2011)的研究, 当饲料中赖氨酸含量从2.08%上升到3.25%时, 黑鲷的特定生长率, 血清总蛋白和血清谷草转氨酶和谷丙转氨酶活性变化与刘峰等(2017)实验结果吻合, 由此推测紫单胞菌科细菌可能与赖氨酸等热敏性氨基酸的产生过程有关, 但总胆固醇及超氧化物歧化酶在两组间的变化无法用赖氨酸的差异解释。
实验结果显示水解羽毛粉组中盐单胞菌属的相对丰度显著高于鱼粉组。已知盐单胞菌属是一类能在绝对盐度为0—32%条件下生长的耐盐好氧细菌(朱凤玲等, 2011), 由此我们推测水解羽毛粉黑鲷肠道环境可能比鱼粉组具有更高的盐度。
4 结论以羽毛粉替代饲料中5%的鱼粉后, 实验组黑鲷肠道细菌群落的丰富度、辛普森指数和香农指数发生了显著下降, 实验组肠道细菌的群落结构(不考虑酸解处理的情况下)也产生了显著变化(P < 0.033), 这种变化体现在处理组的拟杆菌纲、梭菌纲、δ-变形菌纲、草酸杆菌科和青枯菌属的相对丰度发生了显著降低。
王纯, 2017.芽孢杆菌V4和胶红酵母对鲑鳟鱼生长免疫及肠道菌群影响研究.青岛: 中国科学院大学(中国科学院海洋研究所)博士学位论文
|
王赛, 陈刚, 张健东, 等. 2012. 不同蛋白质源部分替代鱼粉对褐点石斑鱼幼鱼生长性能、体组成以及血清生化指标的影响. 动物营养学报, 24(1): 160-167 DOI:10.3969/j.issn.1006-267x.2012.01.023 |
王裕玉, 于世亮, 石野, 等. 2013. 羽毛粉在水产动物饲料中的应用. 中国饲料, (15): 39-43 DOI:10.3969/j.issn.1004-3314.2013.15.011 |
文冰洁, 2018.羽毛降解菌株的分离鉴定及产酶研究.西安: 西北农林科技大学硕士学位论文
|
付文忠, 2014.黑鲷幼鱼饲料中桡足动物粉替代鱼粉的研究.杭州: 浙江大学硕士学位论文
|
朱凤玲, 曲凌云, 洪旭光, 等. 2011. 盐单胞菌属一新种的分离与鉴定. 海洋科学进展, 29(2): 221-228 DOI:10.3969/j.issn.1671-6647.2011.02.012 |
刘峰, 申旭红, 李婠婠, 等. 2017. 不同工艺羽毛粉对黑鲷生长性能及血液生化指标的影响. 饲料工业, 38(24): 20-25 |
刘增新, 2017.牙鲆(Paralichthys olivaceus)仔稚幼鱼肠道菌群结构变化及定植规律研究.上海: 上海海洋大学硕士学位论文
|
许超, 2017.养殖大鲵肠道菌群结构及肠道细菌药敏性研究.重庆: 西南大学硕士学位论文
|
李弋, 黄文文, 周歧存. 2017. 不同干燥方式对鱼粉营养价值及新鲜度的影响. 宁波大学学报(理工版), 30(3): 1-5 |
杨彬彬, 华颖, 肖金星, 等. 2015. 棉粕替代部分鱼粉对黑鲷幼鱼消化酶活性及肠道组织结构的影响. 扬州大学学报(农业与生命科学版), 36(4): 45-51 |
陈盈霖, 李佳凝, 刘洋, 等. 2017. 羽毛粉加工及应用的研究进展. 黑龙江畜牧兽医, (10): 78-82 |
周凡, 2011.饲料赖氨酸和精氨酸对黑鲷幼鱼生长影响及其拮抗作用机理研究.杭州: 浙江大学博士学位论文
|
周凡, 何丰, 王亚琴. 2012. 真鲷饲料营养研究进展. 养殖与饲料, (2): 41-46 DOI:10.3969/j.issn.1671-427X.2012.02.029 |
钟雷, 吉红, 夏耘, 等. 2014. 脱脂蚕蛹替代日粮中鱼粉对建鲤肠道菌群的影响. 中国水产科学, 21(3): 531-540 |
侯玉煌, 丁宏标. 2018. 角蛋白酶研究进展及其在饲料工业中的应用. 中国畜牧杂志, 54(1): 13-18 |
郭爱红. 2014. 羽毛粉营养价值及其在畜禽生产中的应用. 广东饲料, 23(7): 30-32 |
彭翔, 宋文新, 周凡, 等. 2012. 发酵豆粕替代鱼粉对黑鲷胃肠道和血清指标的影响. 江苏农业学报, 28(5): 1096-1103 |
Apper E, Weissman D, Respondek F et al, 2016. Hydrolysed wheat gluten as part of a diet based on animal and plant proteins supports good growth performance of Asian seabass (Lates calcarifer), without impairing intestinal morphology or microbiota. Aquaculture, 453: 40-48 DOI:10.1016/j.aquaculture.2015.11.018 |
Austin B, 2006. The bacterial microflora of fish, revised. The Scientific World Journal, 6: 931-945 DOI:10.1100/tsw.2006.181 |
Bäckhed F, Ding H, Wang T et al, 2004. The gut microbiota as an environmental factor that regulates fat storage. Proceedings of the National Academy of Sciences of the United States of America, 101(44): 15718-15723 DOI:10.1073/pnas.0407076101 |
Bolnick D I, Snowberg L K, Hirsch P E et al, 2014. Individuals' diet diversity influences gut microbial diversity in two freshwater fish (threespine stickleback and Eurasian perch). Ecology Letters, 17(8): 979-987 DOI:10.1111/ele.12301 |
Bureau D P, Harris A M, Cho C Y, 1999. Apparent digestibility of rendered animal protein ingredients for rainbow trout (Oncorhynchus mykiss). Aquaculture, 180(3-4): 345-358 DOI:10.1016/S0044-8486(99)00210-0 |
Caporaso J G, Kuczynski J, Stombaugh J et al, 2010. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 7(5): 335-336 DOI:10.1038/nmeth.f.303 |
Caporaso J G, Lauber C L, Walters W A et al, 2012. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. The ISME Journal, 6(8): 1621-1624 DOI:10.1038/ismej.2012.8 |
Desai A R, Links M G, Collins S A et al, 2012. Effects of plant-based diets on the distal gut microbiome of rainbow trout (Oncorhynchus mykiss). Aquaculture, 350-353: 134-142 DOI:10.1016/j.aquaculture.2012.04.005 |
Huyben D, 2017. Effects of feeding yeasts on blood physiology and gut microbiota of rainbow trout. Uppsala: Doctor Dissertation of Swedish University of Agricultural Sciences
|
Ingle E, 2013. Biodiversity for the protein sources utilized in feeds for intensive fish culture. Como, Varese: Doctor Dissertation of University of Insubria
|
McDonald R, Schreier H J, Watts J E M, 2012. Phylogenetic analysis of microbial communities in different regions of the gastrointestinal tract in Panaque nigrolineatus, a wood-eating fish. PLoS One, 7(10): e48018 DOI:10.1371/journal.pone.0048018 |
Michl S C, Ratten J M, Beyer M et al, 2017. The malleable gut microbiome of juvenile rainbow trout (Oncorhynchus mykiss):Diet-dependent shifts of bacterial community structures. PLoS One, 12(5): e0177735 DOI:10.1371/journal.pone.0177735 |
Miyake S, Ngugi D K, Stingl U, 2015. Diet strongly influences the gut microbiota of surgeonfishes. Molecular Ecology, 24(3): 656-672 DOI:10.1111/mec.13050 |
Nayak S K, 2010. Role of gastrointestinal microbiota in fish. Aquaculture Research, 41(11): 1553-1573 DOI:10.1111/j.1365-2109.2010.02546.x |
Ringø E, 1993. Does dietary linoleic acid affect intestinal microflora in Arctic charr, Salvelinus alpinus (L.)?. Aquaculture Research, 24(1): 133-135 DOI:10.1111/j.1365-2109.1993.tb00837.x |
Round J L, Mazmanian S K, 2009. The gut microbiota shapes intestinal immune responses during health and disease. Nature Reviews Immunology, 9(5): 313-323 DOI:10.1038/nri2515 |
Schloss P D, Westcott S L, Ryabin T et al, 2009. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 75(23): 7537-7541 DOI:10.1128/AEM.01541-09 |
Sears C L, 2005. A dynamic partnership:celebrating our gut flora. Anaerobe, 11(5): 247-251 DOI:10.1016/j.anaerobe.2005.05.001 |
Wei H, Dong L, Wang T T et al, 2010. Structural shifts of gut microbiota as surrogate endpoints for monitoring host health changes induced by carcinogen exposure. FEMS Microbiology Ecology, 73(3): 577-586 |
Wong S, Rawls J F, 2012. Intestinal microbiota composition in fishes is influenced by host ecology and environment. Molecular Ecology, 21(13): 3100-3102 DOI:10.1111/j.1365-294X.2012.05646.x |
 2019, Vol. 50
2019, Vol. 50


