中国海洋湖沼学会主办。
文章信息
- 樊博琳, 潘丽霞, 王忠良, 黎源, 简纪常, 王蓓. 2020.
- FAN Bo-Lin, PAN Li-Xia, WANG Zhong-Liang, LI Yuan, JIAN Ji-Chang, WANG Bei. 2020.
- 罗非鱼源无乳链球菌(Streptococcus agalactiae)新型AI-2信号分子受体RbsB蛋白结晶生长研究
- CRYSTAL GROWTH OF NOVEL AI-2 SIGNALING MOLECULE RECEPTOR RbsB PROTEIN FROM STREPTOCOCCUS AGALACTIAE
- 海洋与湖沼, 51(1): 132-140
- Oceanologia et Limnologia Sinica, 51(1): 132-140.
- http://dx.doi.org/10.11693/hyhz20190500103
文章历史
-
收稿日期:2019-05-31
收修改稿日期:2019-07-22
2. 广西科学院 南宁 536000;
3. 中国科学院实验海洋生物学重点实验室 中国科学院海洋研究所 青岛 266071;
4. 海洋生物学与生物技术功能实验室 青岛海洋科学与技术试点国家实验室 青岛 266071
2. Guangxi academy of Science, Nanning 536000, China;
3. Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China;
4. Laboratory for Marine Biology and Biotechnology, National Laboratory for Marine Science and Technology, Qingdao 266071, China
无乳链球菌(Streptococcus agalactiae)是一类致病性革兰氏阳性细菌, 又被称为B群链球菌(group B Streptococcus, GBS)。能引起人类败血症、脑膜炎等疾病及多种动物链球菌病(Pereira et al, 2010), 也是近年引起罗非鱼链球菌病的主要病原菌。无乳链球菌诱发的罗非鱼发病率和死亡率极高, 发病率一般达20%—30%, 发病鱼死亡率达95%以上, 严重影响了我国罗非鱼养殖业的经济效益(卢迈新等, 2010)。
AI-2信号分子受体(呋喃酰硼酸二酯受体, Furanosyl borate diester receptor)在细菌识别和感知周围环境变化, 并做出适应性反应(细菌密度感应系统, Quor-um sensing, QS)的过程中发挥着重要的作用(Bassler et al, 2006; 马艳平等, 2013)。迄今为止, 研究者们已从遗传学、生物化学和生物信息学三个层面鉴定出了两种AI-2特异性表面结合受体(Chen et al, 2002; Miller et al, 2004), 分别为LuxP蛋白和LsrB蛋白(Pereira et al, 2013)。然而, 研究发现并不是所有细菌均存在以上两类受体蛋白。那么在其他种类的细菌中, 由谁来担当AI-2信号分子受体呢?近年来, 科学家们展开了一系列研究, Armbruster等(2010)在分析伴放线放线杆菌(Actinobacillus actinomycetemcomitan)和流感嗜血杆菌(Haemophilusin fluenzae)全基因组序列时, 发现了一种被称作核糖结合蛋白B(Ribose binding protein B, RbsB)的可溶性细胞周质蛋白家族成员, 它们与大肠杆菌(Escherichia coli) LsrB蛋白的核酸和氨基酸序列同源性分别达到了86%和76%, 且与LsrB蛋白一样, 具有由L-S-G-G-Q氨基酸残基组成的高亲和力蛋白特征性模Walker C(Armbruster et al, 2010)。Armbruster及其团队通过体外人工补充AI-2分子, 构建luxS和rbsB基因缺失株, 获得了流感嗜血杆菌生物膜形成表型的正负反馈调控的实验证据。综上所述, 推测RbsB可能是一种新型的细菌AI-2信号分子受体, 并在AI-2介导的QS系统中单独或与LsrB和LuxP经典受体共同发挥作用(Armbruster et al, 2009, 2010, 2011)。
研究AI-2信号分子介导的细菌密度感应系统对于了解无乳链球菌毒力因子调控、生物膜结构稳定性及抗药性产生等生物学效应具有重要的意义, 也是寻找新型药物靶标的重要理论基础(马艳平等, 2013)。本研究以期获得无乳链球菌RbsB具有AI-2信号受体功能的可靠证据, 并开展对RbsB的AI-2分子受体功能分析及作用机制研究, 旨在为开发罗非鱼无乳链球菌病的新型抗菌候选药物提供科学依据。
1 材料与方法 1.1 材料罗非鱼源无乳链球菌强毒株(S. agalactiae)ZQ 0910(中国微生物菌种保藏中心保藏编号为CGMCC 7.264)由本实验室分离保存; 大肠杆菌表达菌株BL 21(DE3)与pGEX-6P-1载体均为本实验室保存; 限制性核酸内切酶BamHI和XhoI, 克隆载体pMD18-T, ExTaq DNA聚合酶, T4 Ligase购买自宝生物工程有限公司(大连); 质粒提取试剂盒、凝胶回收试剂盒和PierceTM HRV 3C Protease Solution Kit购买自赛默飞世尔科技(Thermo Fisher Scientific)公司; 引物、氨苄青霉素(Ampicillin)、异丙基硫代-β-D-半乳糖苷(IPTG)买自生工生物工程有限公司(广州); 考马斯亮蓝快速染液购买自全式金生物技术有限公司(北京), NeXtal Tubes JCSG Core Suite Ⅰ, Ⅱ, Ⅲ, Ⅳ为德国QIAGEN产品。
1.2 方法 1.2.1 提取细菌基因组DNA及扩增RbsB基因提取无乳链球菌基因组DNA, 根据GenBank上已登录的RbsB基因序列(GenBank Accession: AP018935.1)设计特异性引物RbsBF/RbsBR, 引入酶切位点BamHI和XhoI, 引物由生工生物工程(广州)有限公司合成。
RbsBF: 5′-GGATCCATGAAATTTGGAAAAAAACTT-3′
RbsBR: 5′-AAGTTTTTTTCCAAATTTCATCTCGAG-3′
反应条件为: 94℃预变性5min, 94℃ 30s, 60℃ 30s, 72℃ 90s, 共30个循环, 72℃再延伸10min。用1%琼脂糖凝胶电泳检测PCR产物后再切胶回收, 连接入pMD18-T载体, 菌落PCR鉴定后将阳性克隆送生工生物工程股份有限公司(广州)测序。
1.2.2 构建重组质粒载体及诱导表达融合蛋白PCR扩增产物经纯化试剂盒纯化回收后用BamHI和XhoI限制性内切酶进行双酶切, 对目的条带进行切胶回收后定向插入经BamHI和XhoI内切酶双酶切的pGEX-6P-1质粒载体中, 构建重组质粒pGEX-6p- 1-RbsB转化至大肠杆菌BL21(DE3)感受态细胞中, 涂布于Amp+的LB琼脂平板, 37℃条件下培养12h, 从平板挑取单菌落于Amp+的LB液体培养基中, 37℃、220r/min培养1h, 提取重组质粒送至测序(广州生工)测序正确后, 进行菌落PCR鉴定和双酶切检测, 检测正确后接种于新鲜的Amp+的LB液体培养基(比例1︰100)中, 37℃继续震荡培养, 至OD600值达到0.6—0.8, 加IPTG(终浓度为0.2mmol/L)进行诱导表达。
1.2.3 重组融合蛋白表达条件的优化将鉴定正确的表达菌接种到2mL Amp+的LB液体培养基中, 加入IPTG进行诱导(终浓度为0.2mmol/L), 选择16℃、30℃、37℃三个温度条件下220r/min振荡培养4h后离心收集菌体沉淀。经超声波破碎离心后分别取样品上清液和菌体沉淀(溶于尿素中)并进行3个相同条件的重复组, 用SDS-PAGE电泳分析目的蛋白表达的最佳条件。
1.2.4 RbsB蛋白纯化及鉴定大量表达pGEX- 6P-1-RbsB重组融合蛋白, 离心收集菌体沉淀后用Equilibration Buffer (50mmol/L Tris-HCl, 150mmol/L NaCl, 10% glycerol pH 8.0)溶液混悬, 超声波破碎并高速离心制备菌体上清。取3mL谷胱甘肽琼脂糖树脂(Glutathione Sepharose 4B)加入到5mL层析柱中, 层析柱与DH电脑恒流泵和HUXI紫外检测仪连接。使用50mL GST Equilibration Buffer平衡层析柱30min, 加入制备好的菌体上清, 使菌体上清与谷胱甘肽琼脂糖树脂静置结合30min, 使用GST Elution Buffer (50mmol/L Tris-HCl, 150mmol/L NaCl, 15mmol/L reduced glutathione pH 8.0)洗脱液进行洗脱, 根据紫外显示仪数字变化(波长为A280nm)收集洗脱液, 收集原始菌体上清液, 流穿液, 洗脱液进行SDS-PAGE电泳。
1.2.5 切除融合表达蛋白GST标签使用超滤浓缩管(Milipore)(10kDa)浓缩收集到的蛋白洗脱液后, 用PierceTM HRV 3C Protease Solution Kit试剂盒进行切除GST标签, 纯化后的重组蛋白2900μL中加入HRV 3C Protease 100μL, 4℃冷藏过夜。用AKTATM purifier全自动层析仪根据分子量大小不同分离GST标签与Rbsb蛋白, 收集洗脱峰对应样品, 用SDS- PAGE进行检测并采用蛋白定量试剂盒进行定量检测。
1.2.6 RbsB基因序列分析通过使用DNAMAN程序获得RbsB推导氨基酸序列; 使用ExPASy (http://expasy.org/tools/)网址进行RbsB蛋白质理化性质分析; 使用Softberry程序对氨基酸功能位点进行分析; 使用signalIP4.0预测信号肽位置及剪切位点; 使用InterProScan程序(http://www.ebi.ac.uk/Tools/pfa/iprscan/)进行结构域预测; 使用(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)网址进行蛋白二级结构预测, 并通过圆二色谱实验对预测结果进行验证(实验在持续氮气流的作用下, 对样品的圆二色谱变化进行测定, 扫描波长范围在200—260nm之间, 比色皿的光程为0.5cm, 扫描速度为50nm/min, 进行3次重复扫描); 使用SWISS-MODEL在线软件(http://www.expasy.org/)对RbsB蛋白3D三级结构进行预测。
1.2.7 RbsB蛋白的结晶条件筛选将RbsB蛋白分别浓缩到10mg/mL、20mg/mL和40mg/mL, 利用NeXtal Tubes JCSG Core Suite (Ⅰ, Ⅱ, Ⅲ, Ⅳ)蛋白结晶条件筛选试剂盒, 共384个结晶条件。对RbsB蛋白通过坐滴气相扩散法进行结晶条件的初步筛选, 将RbsB蛋白溶液与池液在晶体板的样品槽内1︰1等比例混合, 用封膜密封, 置于16℃恒温室中, 每3天通过显微镜观察蛋白液滴内晶体的生长情况。
2 结果 2.1 扩增RbsB基因片段以及构建可溶性表达载体以无乳链球菌基因组DNA为模板, 用带酶切位点的引物扩增后, 获得一大小约为969bp的目的条带(图 1), 与预期相符。将测序正确的重组质粒用限制性核酸内切酶BamHI和XhoI进行双酶切, 图 2中泳道片段大小分别约为5000bp和969bp, 测序结果表明重组质粒构建成功。
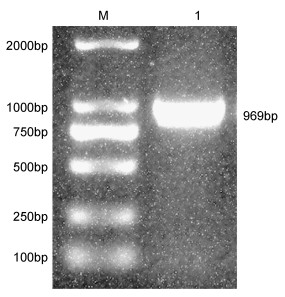 |
| 图 1 无乳链球菌RbsB基因的克隆 Fig. 1 Cloning of RbsB gene from S. agalactiae 注: M: DL2000DNA分子标准; 1: RbsB PCR扩增产物 |
 |
| 图 2 重组质粒pGEX-6p-1-RbsB的酶切鉴定 Fig. 2 The result of pGEX-6p-1-RbsB digested by BamHI and XhoI 注: M: DL10000 DNA分子标准; 1:酶切后产物 |
从图 3中可以看出, 在保证其他实验条件相同情况下, 不同温度诱导融合蛋白的表达量和可溶性分析表明37℃条件下RbsB全菌蛋白表达量高于16℃和30℃的表达量; 在沉淀中的融合蛋白表达量显著低于在上清中的表达量。进行3个重复组结果均一致, 故该融合蛋白最佳诱导温度是37℃, 并选择在上清中进行纯化。
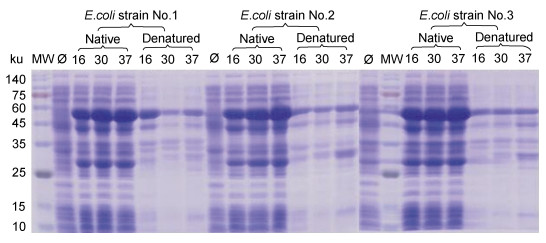 |
| 图 3 不同温度诱导表达的SDS-PAGE分析 Fig. 3 SDS-PAGE analysis of expression of the Rbsb fusion protein at different temperatures 注: MW:蛋白分子量标准; Ø:未进行诱导细菌表达(阴性对照) |
将pGEX-6P-1-RbsB质粒转化入大肠杆菌BL21 (DE3)感受态细胞中, 经优化条件(0.2mmol/L IPTG, 4h, 37℃)诱导后, 根据紫外显示仪数字变化在第二个峰值处(波长为A280nm)收集洗脱液(图 4)。分别取上清液、流穿液、洗脱液进行SDS-PAGE电泳鉴定。结果表明, 经过纯化后可以得到含量较高的融合蛋白(10. 464mg/mL), 且在59ku处有一条明显蛋白条带(图 5), 与预期相符。
 |
| 图 4 紫外吸收图谱 Fig. 4 The ultraviolet absorption spectrum |
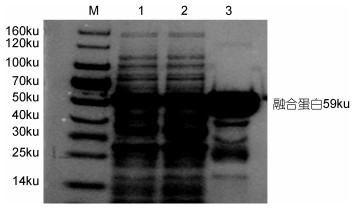 |
| 图 5 重组蛋白GST-RbsB纯化后的SDS-PAGE分析 Fig. 5 SDS-PAGE analysis of purified GST-RbsB 注: M:蛋白分子量标准; 1:上清; 2:流穿液; 3:洗脱液 |
将收集到的洗脱液浓缩至3mL, 用试剂盒进行切除GST标签工作, 经谷胱甘肽琼脂糖树脂(Glutathione Sepharose 4B)纯化后收集3管洗脱液, 分别与HRV 3C Protease过夜反应, SDS-PAGE电泳结果表明, 本研究成功将GST标签从融合蛋白成除, 分别得到GST标签蛋白(26ku)和RbsB蛋白(33ku)。
2.5 分子筛分离RbsB蛋白将收集到的3管融合蛋白与HRV 3C Protease过夜反应上清浓缩至2mL, 使用AKTATM purifier全自动层析仪, 根据分子量大小不同彻底分离GST-tag与RbsB蛋白, 根据全自动层析仪电脑操作页面数据变化显示, 选取峰值处10管进行SDS-PAGE鉴定。结果显示第1—6管为RbsB蛋白(33ku), 第7—10管为GST-tag(26ku)与融合蛋白(59ku)混合液, 分子量大小正确(图 7)。
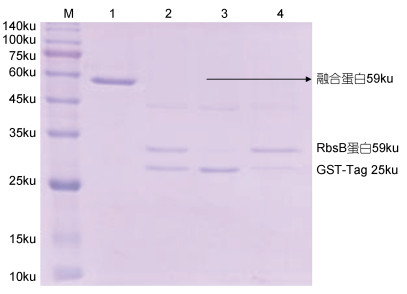 |
| 图 6 GST-Rbsb融合蛋白纯化及切除GST标签 Fig. 6 Purity of GST-Rbsb fusion protein excision of GST-tag 注: M:蛋白分子量标准; 1:融合蛋白; 2—4:融合蛋白与HRV 3C Protease过夜反应上清 |
 |
| 图 7 使用AKTATM purifier切除GST标签后RbsB蛋白的SDS-PAGE分析 Fig. 7 SDS-PAGE analysis of single RbsB protein using AKTATM purifier 注: M:蛋白分子量标准; 1—6: RbsB蛋白; 7—10: GST标签与融合蛋白 |
RbsB基因(GenBank Accession: AP018935.1)全长969bp, 可编码322个氨基酸, ProtParam分析RbsB推导氨基酸序列, RbsB蛋白理论分子量33.9ku, 等电点为9.41, 其中丙氨酸(Ala)含量最高为13.4%, 赖氨酸(Lys)其次为11.8%。其中带正电荷的氨基酸有42个, 带负电荷的氨基酸有31个。SoftBerry-Psite预测该序列功能位点, 发现该序列有N-糖基化位点4个, cAMP-和cGMP依赖性蛋白激酶磷酸化位点1个, 蛋白激酶C磷酸化位点5个, 酪蛋白激酶Ⅱ磷酸化位点2个, N-豆蔻酰化位点8个, 酰胺化位点3个, 原核膜脂蛋白脂质附着部位1个, 微生物C末端靶向信号7个。signalIP4.0预测RbsB存在信号肽, 1—24为信号肽区域, 其中信号肽剪切位点在24—25氨基酸之间。
2.7 RbsB蛋白二级结构预测与验证分析RbsB的二级结构, 从RbsB蛋白二级结构预测图(图 9)中可以看出RbsB蛋白的二级结构有: α-螺旋(46.27%)、无规卷曲(38.15%)、β-折叠(15.22%)。蛋白质二级结构的变化情况可以用圆二色谱实验结果来说明, 已知α-螺旋构象的CD谱在222nm、208nm处呈负峰, 在190nm附近有一正峰。结果显示所测二级结构具有明显的α螺旋结构且所占比重最高, 与预测结果一致。
 |
图 8 RbsB基因核苷酸序列及推导的氨基酸序列和功能位点 Fig. 8 Open reading frame and deduced amino acid sequences of RbsB gene 注: “_” N-糖基化位点; “  ” cAMP-和cGMP依赖性蛋白激酶磷酸化位点; “ ” cAMP-和cGMP依赖性蛋白激酶磷酸化位点; “  ”蛋白激酶C磷酸化位点; “ ”蛋白激酶C磷酸化位点; “  ”酪蛋白激酶Ⅱ磷酸化位点; “…”N-豆蔻酰化位点; “ ”酪蛋白激酶Ⅱ磷酸化位点; “…”N-豆蔻酰化位点; “  ”酰胺化位点; “ ”酰胺化位点; “  ”原核膜脂蛋白脂质附着部位; “ ”原核膜脂蛋白脂质附着部位; “  ”微生物C末端靶向信号; “*”终止子 ”微生物C末端靶向信号; “*”终止子 |
 |
| 图 9 无乳链球菌RbsB蛋白二级结构预测 Fig. 9 Predicted secondary structure of RbsB protein 注: “c”; 无规卷曲; “e”; β-延伸; “h”; α-螺旋 |
用InterProScan程序分析预测结构域得在氨基酸50—300间有一结构域, 为周质结合蛋白区域。采用在线SWISS-MODEL程序对RbsB蛋白同源建模, 建模结果如图 11所示, 其中三维图是以2joy.1.A为模版, 分别利用无乳链球菌ZQ0910菌株RbsB和大肠杆菌核糖结合蛋白A氨基酸序列49—318区域构建的三维结构, 相似性达到47.78%。
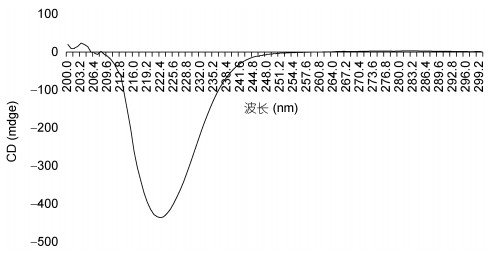 |
| 图 10 圆二色谱测RbsB蛋白二级结构 Fig. 10 Determination of secondary structure of RbsB protein by circular dichroism |
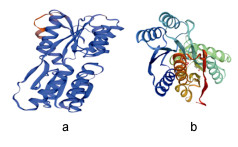 |
| 图 11 无乳链球菌RbsB蛋白(a)与大肠杆菌周质糖结合蛋白A(b)蛋白三维结构预测图 Fig. 11 Three-dimensional structure prediction 注: a. Streptococcus agalactiae RbsB protein; b. Escherichia coli Periplasmic sugar-binding protein A protein |
通过蛋白质结晶条件筛选试剂盒NeXtal Tubes JCSG Core (Ⅰ, Ⅱ, Ⅲ, Ⅳ)共计384个条件对RbsB蛋白进行了结晶条件的初步筛选。如图 12所示, 在NeXtal Tubes JCSG Core Suite Ⅲ (0.2mol/L di-Potassium hydrogen phosphate, 20% (W/V) PEG3350)和NeXtal Tubes JCSG Core Suite Ⅳ (1.5mol/L ammonium sulfate, 25% (V/V) Glycerol)条件下有晶体长出, 该晶体呈棱柱状较为规整, 对应于图 12中a、b, 提示有希望进一步优化获得可衍射的晶体。
 |
| 图 12 RbsB蛋白结晶体 Fig. 12 The crystallization of RbsB protein |
尼罗罗非鱼(Oreochromis niloticus)不但肉质鲜嫩, 而且具有生长快、环境适应力强等优点, 受到了国内外养殖户的青睐。但是近年随着养殖规模的不断扩大, 养殖环境的持续恶化, 链球菌病的大规模暴发, 严重影响了我国国内尼罗罗非鱼养殖业的发展(张新艳等, 2008; 黄锦炉, 2012)。罗非鱼链球菌病(Streptococcosis)具有高温好发、传染性高和死亡率高的特点, 其从属于细菌性疾病。在世界范围内其病原主要为无乳链球菌(S. agalactiae)和海豚链球菌(S. iniae), 无乳链球菌(S. agalactiae)是我国南方地区链球菌病的主要病原菌(王蓓等, 2013)。研究AI-2信号分子介导的细菌密度感应系统对于了解无乳链球菌毒力因子调控、生物膜结构稳定性及抗药性产生等生物学效应具有重要意义。本研究将对探索RbsB分子作为“第3类”AI-2信号分子受体提供直接证据。
获得晶体的前提条件是得到纯度好、浓度高的可溶性蛋白, 因此在表达载体和表达菌株的选择上, 我们采用了可溶表达性更高的pGEX-6P-1载体, 并采用大肠杆菌BL21作为表达菌株。pGEX-6P-1表达载体自带GST融合标签, 具有一定的溶解度在大肠杆菌中, 同时还参与了体系中二硫键还原等过程, 为后续蛋白质的纯化与鉴定提供了方便(Maina et al, 1988; Sharrocks, 1994)。重组蛋白用大肠杆菌BL21(DE3)表达有两种形式; 一种表达形式是以包涵体出现, 另外一种是可溶性表达, 即重组蛋白存在于上清中。经过SDS-PAGE实验验证3组重复试验均显示在上清中表达量明显高于在包涵体中的表达量, 故实验选择在上清中表达利于后续蛋白纯化工作, 获得有活性的RbsB蛋白。
重组融合蛋白的表达水平不仅与宿主和表达载体有关, 也和诱导条件有着密切的关系(Sharrocks, 1994)。其中一个重要的影响条件是诱导温度, 在诱导过程中温度会影响氧的溶解度快慢、并对酶的反应速率造成加快或减慢, 进而影响蛋白的活性。本实验通过固定其他因子, 对单个因子温度进行16℃、30℃、37℃三个条件进行诱导, 经SDS-PAGE验证37℃表达量最佳。为了降低诱导剂IPTG对大肠杆菌代谢的毒性作用, 用终浓度为0.2mmol/L的IPTG诱导蛋白表达, 诱导时间为4h, 用Glutathione Sepharose 4B亲和树脂纯化重组蛋白, 结果测得在上清中取得了10.464mg/mL较高的重组蛋白表达量。使用PierceTM HRV 3C Protease切掉GST-tag然后经AKTATM purifier全自动层析仪按分子量大小将Rbsb蛋白与GST-tag分离开。SDS-PAGE检测表观彻底将RbsB蛋白与GST-tag分离开, 成功获取到纯度很高的RbsB蛋白。
生物信息学是研究基因组学和蛋白质组学的重要工具, 是一门检索、储存并分析生物信息的科学, 它将信息技术应用到了生命科学领域。本研究对RbsB蛋白进行了一系列基因序列分析包括; 蛋白质理化性质分析; 氨基酸功能位点进行分析; 预测信号肽位置及剪切位点; 预测结构域; 进行蛋白二级结构和蛋白3D三级结构预测等工作。通过氨基酸功能位点分析可知蛋白激酶C磷酸化位点5个, N-豆蔻酰化位点8个以及微生物C末端靶向信号7个(数量最多)蛋白激酶C能作用于细胞核中的转录因子, 能激活细胞质中的靶酶参与生化反应的调控, 参与细胞生长和分化相关的基因表达的调控(周小东等, 2013); 而N-豆蔻酰化位点和微生物C末端靶向信号位点也在信号转导中起着重要作用, 更为验证了RbsB可能是一种新型的细菌AI-2信号分子受体, 并在AI-2介导的QS系统中单独或与LsrB和LuxP经典受体共同发挥作用这一推测。通过二级结构预测和圆二色谱实验发现α螺旋结构所占比重最高, 而α螺旋结构是蛋白质构象中最稳定的一种结构, 不论是长链还是短链, 也不论α螺旋相互作用是强还是弱, 一旦螺旋结构形成, 在拉伸过程中都能保持α螺旋结构的稳定性, 这无疑为后续蛋白晶体优化实验提供了保证。
20世纪70年代初期, 中国科学工作者测定了亚洲地区第一个蛋白质晶体结构-猪胰岛素三方二锌晶体结构, 成为中国结构生物学历史发展的起点, 进入新世纪后该学科更是展现出快速发展姿态(王大成, 2014)。本研究利用蛋白质结晶试剂盒NeXtal Tubes JCSG Core (Ⅰ, Ⅱ, Ⅲ, Ⅳ)共计384个条件筛选出了两个可形成柱状晶体的条件, 从结晶筛选条件和晶体外观可判断出这些晶体应为蛋白晶体, 说明RbsB蛋白具备可结晶性。柱状蛋白晶体相比较针状蛋白晶体来说, 其通常衍射能力较强, 可以满足后期收集较高分辨率数据的需要。对于单颗晶体, 我们也经常可以对采用多点收集以及螺旋收集(helical collection)的方法来获得更好的数据质量与分辨率(闫创业, 2014)。为了能够得到良好的衍射数据, 我们拟在后续实验中, 以上述筛选到的结晶条件为基础, 继续优化结晶条件, 以提升获得可用于衍射数据收集的高质量RbsB晶体的概率, 关于RbsB蛋白晶体, 我们将从功能位点进行分析并辅以细菌表型的研究从而确认其受体功能, 旨在为寻找新型药物靶标的重要理论基础。
4 结论本研究成功克隆RbsB基因, 该基因全长969碱基, 编码322个氨基酸, 二级结构中α螺旋结构所占比重最高, RbsB蛋白理论分子量33.9ku, 筛选到RbsB蛋白的初始结晶条件为: 0.2mol/L di-Potassium hydrogen phosphate, 20% (W/V) PEG3350; 1.5mol/L ammonium sulfate, 25% (V/V) Glycerol), 获得RbsB蛋白结晶体。本研究结果可为无乳链球菌核糖结合蛋白(RbsB)的功能解析提供实验及理论基础, 将从功能位点进行分析并辅以细菌表型的研究从而确认其受体功能, 旨在为寻找新型药物靶标的重要理论基础。
马艳平, 李嘉彬, 郝乐, 等. 2013. 罗非鱼源无乳链球菌S-核糖基高半胱氨酸酶基因(luxS)的克隆及其推导蛋白的三维结构预测. 南方水产科学, 9(1): 43-47 DOI:10.3969/j.issn.2095-0780.2013.01.007 |
王蓓, 简纪常, 鲁义善, 等. 2013. 罗非鱼源无乳链球菌ZQ0910转录调控因子rovS基因的克隆及表达研究. 水产学报, 37(4): 584-592 |
王大成. 2014. 结构生物学研究在中国. 生物化学与生物物理进展, 41(10): 944-971 |
卢迈新, 黎炯, 叶星, 等. 2010. 广东与海南养殖罗非鱼无乳链球菌的分离、鉴定与特性分析. 微生物学通报, 37(5): 766-774 |
闫创业, 2014.蛋白质晶体学中的相位解析优化以及三维模型构建的研究.北京: 清华大学博士学位论文
|
张新艳, 樊海平, 钟全福, 等. 2008. 罗非鱼无乳链球菌的分离、鉴定及致病性研究. 水产学报, 32(5): 772-779 |
周小东, 沈富兵. 2013. MAP30蛋白结构功能的生物信息学研究. 广西植物, 33(4): 560-563, 542 DOI:10.3969/j.issn.1000-3142.2013.04.024 |
黄锦炉, 2012.罗非鱼无乳链球菌病病原学、病理学及cpsE基因的原核表达研究.雅安: 四川农业大学博士学位论文
|
Armbruster C E, Hong W Z, Pang B et al, 2009. LuxS promotes biofilm maturation and persistence of nontypeable Haemophilus influenzae in vivo via modulation of lipooligosaccharides on the bacterial surface. Infection and Immunity, 77(9): 4081-4091 DOI:10.1128/IAI.00320-09 |
Armbruster C E, Hong W Z, Pang B et al, 2010. Indirect pathogenicity of Haemophilus influenzae and Moraxella catarrhalis in polymicrobial otitis media occurs via interspecies quorum signaling. mBio, 1(3): e00102-10 |
Armbruster C E, Pang B, Murrah K et al, 2011. RbsB (NTHI_0632) mediates quorum signal uptake in nontypeable Haemophilus influenzae strain 86-028NP. Molecular Microbiology, 82(4): 836-850 DOI:10.1111/j.1365-2958.2011.07831.x |
Bassler B L, Losick R, 2006. Bacterially speaking. Cell, 125(2): 237-246 DOI:10.1016/j.cell.2006.04.001 |
Chen X, Schauder S, Potier N et al, 2002. Structural identifycation of a bacterial quorum-sensing signal containing boron. Nature, 415(6871): 545-549 DOI:10.1038/415545a |
Maina C V, Riggs P D, Grandea Ⅲ A G et al, 1988. An Escherichia coli vector to express and purify foreign proteins by fusion to and separation from maltose-binding protein. Gene, 74(2): 365-373 DOI:10.1016/0378-1119(88)90170-9 |
Miller S T, Xavier K B, Campagna S R et al, 2004. Salmonella typhimurium recognizes a chemically distinct form of the bacterial quorum-sensing signal AI-2. Molecular Cell, 15(5): 677-687 DOI:10.1016/j.molcel.2004.07.020 |
Pereira C S, Thompson J A, Xavier K B, 2013. AI-2-mediated sig nalling in bacteria. FEMS Microbiology Reviews, 37(2): 156-181 DOI:10.1111/j.1574-6976.2012.00345.x |
Pereira U P, Mian G F, Oliveira I C M et al, 2010. Genotyping of Streptococcus agalactiae strains isolated from fish, human and cattle and their virulence potential in Nile tilapia. Veterinary Microbiology, 140(1-2): 186-192 DOI:10.1016/j.vetmic.2009.07.025 |
Sharrocks A D, 1994. A T7 expression vector for producing N- and C-terminal fusion proteins with glutathione S-transferase. Gene, 138(1-2): 105-108 DOI:10.1016/0378-1119(94)90789-7 |
 2020, Vol. 51
2020, Vol. 51


