
中国海洋湖沼学会主办。
文章信息
- 盛鹏程, 周冬仁, 韩新荣, 胡晓波, 叶雪平, 徐磊, 周聃, 吴琦芳, 郝贵杰. 2020.
- SHENG Peng-Cheng, ZHOU Dong-Ren, HAN Xin-Rong, HU Xiao-Bo, YE Xue-Ping, XU Lei, ZHOU Dan, WU Qi-Fang, HAO Gui-Jie. 2020.
- 不同饲养方式对乌鳢(Ophiocephalus argus)肠道微生物群落结构差异及种类多样性的影响
- EFFECTS OF DIFFERENT FEEDING MATTERS ON INTESTINAL MICROBIAL STRUCTURE DIVERSITY OF OPHIOCEPHALUS ARGUS
- 海洋与湖沼, 51(1): 148-155
- Oceanologia et Limnologia Sinica, 51(1): 148-155.
- http://dx.doi.org/10.11693/hyhz20190800160
文章历史
-
收稿日期:2019-08-28
收修改稿日期:2019-10-13
2. 桐乡市水产工作站 桐乡 314500
2. Tongxiang Fishery Workstation, Tongxiang 314500, China
乌鳢(Ophiocephalus argus), 又名黑鱼、乌鱼、生鱼、财鱼、蛇鱼、火头鱼等, 隶属于鳢科、鳢属。除新疆、西藏外, 乌鳢在我国各大水系广泛分布。乌鳢营养丰富、骨剌少、蛋白含量高, 且具有去瘀生新、滋补调养、健脾利水的医疗功效, 是集食用与药补于一身的名贵鱼类。据不完全统计, 目前乌鳢的养殖面积5764ha, 总产量达到51788t, 总产值为10.38亿元。
微生物大量地存在于肠道中, 将其作为栖息地。肠道不仅有复杂的微生态系统, 而且其作为宿主生命活动的重要组成部分, 对宿主的生长发育也具有重要的影响(刘增新等, 2017)。稳定的肠道微生物区系对多种宿主功能都有影响, 如肠道J肠道微生物的组成及其比例。目前, 对水产养殖动物肠道微生物多样性进行的研究很多, 如黄颡鱼(Pelteobagrus fulvidraco)、草鱼(Cten opharyngodon idellus)、对黑虎虾(P. monodon)和太平洋蓝虾(Litopenaeus stylirostris) (Wu et al, 2010; Rungrassamee et al, 2014; Li et al, 2015; Cardona et al, 2016), 但对乌鳢肠道微生物的研究鲜有报道。
目前不少养殖户为了降低养殖成本, 在乌鳢的养殖过程中投喂畜禽内脏, 但投喂畜禽内脏对乌鳢的影响尚未明确。本试验以乌鳢为研究对象, 将冰鲜鱼、畜禽内脏两种投喂方式应用于乌鳢的养殖, 对乌鳢消化道的微生物开展相关研究, 评价不同的养殖方式对乌鳢肠道中微生物菌群种类的影响, 为评价乌鳢的产品质量, 发展健康绿色生态养殖提供参考。为探究饲粮因素对乌鳢肠道的菌群组成与宿主间特异性关系等提供基础数据。
1 材料与方法 1.1 动物试验试验场地选址于浙江省嘉兴市某渔场, 选择健康、规格基本一致的乌鳢(Ophiocephalus argus)随机分为试验W1组、W2组。每组2个重复, 分别饲喂于4个池塘中, W1组投喂冰鲜鱼, W2组投喂畜禽内脏。以乌鳢体重4%左右投喂饲粮。每日7:00与17:00各投喂1次, 共投喂为12周。
1.2 样品采集乌鳢养殖试验结束后进行样品采集, 每组取6尾鱼, 无菌操作台中将其肠道迅速分离, 内容物取出并混合后, 于液氮中速冻, 最后置于–80℃超低温冰箱保存备用。
1.3 总DNA的提取从–80℃超低温冰箱取出两组样品, 在无菌条件下用电动组织研磨器充分研磨, 参照粪便基因组DNA提取试剂盒说明步骤提取总DNA。将提取的DNA取2μL利用电泳仪(120V, 30min)检测, 选取具有单一条带的DNA作为模板, 扩增其16S rRNA V4—V5区, 扩增引物采用515F和907R。PCR采用20μL扩增体系; 4μL 5×Fast Pfu Buffer, 2μL 2.5mmol/L dNTPs, 0.4μL上游引物, 0.4μL下游引物, 0.4μL Fast Pfu Polymerase, 10ng Template DNA, 加灭菌水补至20μL。扩增条件; 95℃预变性2min; 25个循环, 95℃ 30s, 55℃ 30s, 72℃ 45s; 72℃延伸10min。
1.4 文库构建和测序PCR产物采用2%琼脂糖凝胶电泳验证, 目的条带胶采用QIA quick GelEx-traction Kit试剂盒(Qiagen, 德国)回收纯化, 将纯化产物送至上海美吉生物有限公司, 测序平台为Illumina MiSeq。DNA样品送至上海生工进行Illumina Hiseq高通量测序分析, 样品编号分别为; W1-1、W1-2; W2-1、W2-2。
1.5 生物信息学分析首先将得到的数据进行处理, 然后进行OTU聚类分析, 采用Alpha多样性分析、衡量样本物种的多样性。对序列进行物种分类, 并对每个样本和每个物种单元分类进行序列丰度的计算, 构建样本和物种分类单元序列的丰度矩阵, 分析物种的分类。基于OTU聚类分析及物种分类的共有进行多维度分析。绘制所有群落分布间的进化树图, 同时绘制OTU聚类结果中丰度较高的OTU进化树图, 并标注出其所属的群落分布信息, 对所涉及的物种进行分类树构建。使用Beta多样性分析, 比较多(组)样本之间的差别度量。最后进行菌群差异分析和功能预测分析。
2 结果与分析 2.1 序列数与OTU数量统计 2.1.1 样本数据统计高通量测序的最原始的测序序列数、经过处理和质控后得到的最终序列以及序列平均长度(表 1), 由表可知, 每个样品所得条带序列均在15000条以上, 共得到序列242609条。
| 组别 | 样本 | Barcode | 原始reads数目 | 原始序列平均长度 | QC后剩余reads数目 | QC后序列平均长度 |
| W1 | W1-1 | CTTGTA | 49092 | 455.23 | 47853 | 415.75 |
| W1 | W1-2 | GTTTCG | 56582 | 455.83 | 55013 | 416.22 |
| W2 | W2-1 | GTCGGA | 40793 | 453.9 | 39833 | 414.88 |
| W2 | W2-2 | AATATC | 42362 | 459.86 | 41284 | 420.49 |
去除嵌合体及非特异性扩增序列后, 得到数据见表 2。由表 2可知, 去除嵌合体及非特异性扩增序列后W1-1、W1-2、W2-1、W2-2的序列数分别为47800、54448、38546、40050。
| 组别 | 样本名 | 处理前序列总数 | 比对到细胞器组织序列数目 | 非靶区序列数目 | 嵌合体数目 | 处理后剩余序列 |
| W1 | W1-1 | 47853 | 7 | 0 | 46 | 47800 |
| W1 | W1-2 | 55013 | 11 | 0 | 554 | 54448 |
| W2 | W2-1 | 39833 | 758 | 0 | 529 | 38546 |
| W2 | W2-2 | 41284 | 1127 | 4 | 103 | 40050 |
VENN图用来统计样本中共有和独有OTU的数目, 直观展现出环境样品OTU数目组成的相似性及重叠情况等。优化序列在Silva数据库进行相似性比对, OTU序列分类(97%相似水平)是采用RDP classifier贝叶斯算法进行的, W1-1组肠道细菌获得OTU 1072个, W1-2组肠道细菌获得OTU 1559个, W2-1组肠道细菌获得OTU2385个, W2-2组肠道细菌获得OTU 1729个, W1组和W2组共同含有OTU 19个(图 1)。
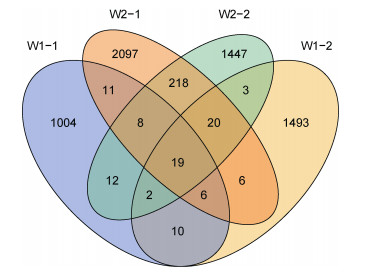 |
| 图 1 OTU分布Venn图 Fig. 1 Venn diagram of OTU distribution |
样本聚类树图(图 2)通过树枝结构直观反映出多个样品间的相似性和差异关系。根据beta多样性距离矩阵进行层次聚类(Hierarchical clustering)分析, 再使用非加权组平均法UPGMA (Unweighted pair group method with arithmetic mean)算法进行树状结构构建, 从而获得树状关系形式进行可视化分析。
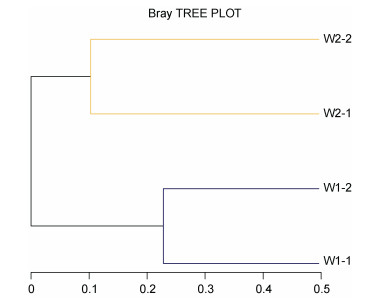 |
| 图 2 基于OTU样本的聚类树图 Fig. 2 Bray tree plot based on OUT |
各样品的OTU数目、Shannon指数、ACE指数、Chao1指数及样品覆盖率如表 3所示, 由表 3可知, 4组样品的覆盖率均在96%以上, 较好地反映了样本菌群情况, W1样品中ACE和Chao1指数最大, 说明饲喂冰鲜鱼乌鳢肠道中菌群总数最多, W2样品中OTU数目最多且Shannon指数最高, 说明饲喂畜禽内脏乌鳢肠道中菌群丰度最高, 多样性最好。
| 样本名称 | Seq数目 | OTU数目 | Shannon指数 | ACE指数 | Chao1指数 | Coverage覆盖率 | Simpson多样性指数 |
| W1-1 | 47800 | 1072 | 1.24 | 67267.24 | 28263.37 | 0.98 | 0.42 |
| W1-2 | 54448 | 1559 | 1.40 | 253288.39 | 73616.56 | 0.97 | 0.39 |
| W2-1 | 38546 | 2385 | 3.78 | 16508.72 | 8561.10 | 0.96 | 0.12 |
| W2-2 | 40050 | 1729 | 4.18 | 18076.75 | 12253.11 | 0.97 | 0.04 |
通过MiSeq测序平台对饲喂冰鲜鱼和畜禽内脏乌鳢肠道细菌的16S rDNA基因V3—V4区进行微生物多样性的检测分析, 获得W1组和W2组平均细菌总优化序列51124和39298条。采用Mothur软件绘制稀释曲线(Shannon rarefaction plot)。曲线慢慢走向平稳, 即表明测序数据量合理可靠(图 3)。
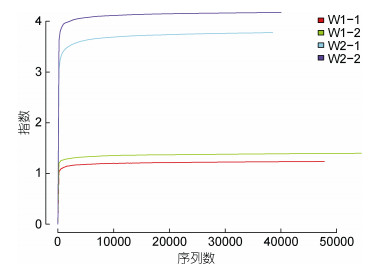 |
| 图 3 Alpha指数稀疏曲线图 Fig. 3 Alpha exponential sparse curve |
见图 4。不同颜色的点代表不同的样本, 若空间距离越近, 则相似度越高。从图 4中均可以看出, W1冰鲜鱼饲喂乌鳢肠道中菌群差异比较小, W2饲喂畜禽内脏乌鳢肠道中菌群相似度较高。
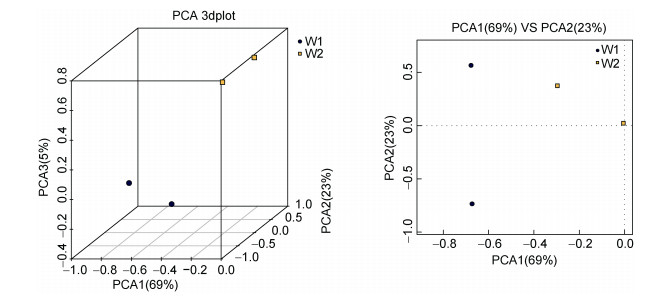 |
| 图 4 基于OTU的PCA图 Fig. 4 PCA based on OTU |
样本间菌群丰度差异分析(图 5)显示, W1组饲喂冰鲜鱼乌鳢肠道中未分类菌群丰度较小, W2组畜禽内脏饲喂乌鳢肠道中未分类菌群丰度较大; W1组畜禽内脏饲喂乌鳢肠道中邻单胞菌属(Plesiomonas)占极大比例, W2组邻单胞菌属(Plesiomonas)丰度显著小于W1组。W2组未分类菌群丰度差异不明显, W1组中邻单胞菌属(Plesiomonas)丰度差异明显。
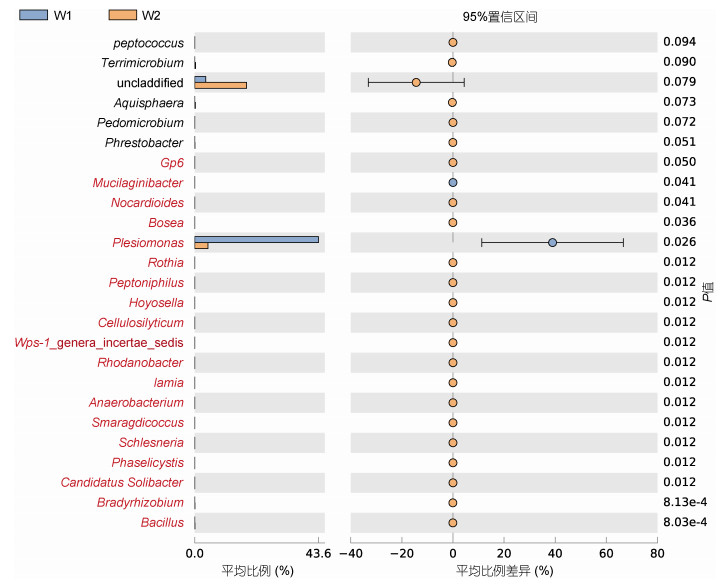 |
| 图 5 差异比较的误差线图 Fig. 5 Error line diagram for difference comparisons |
通过属分类水平上的统计分析, W1组颜色偏蓝, W2组颜色偏红, 说明饲喂畜禽内脏乌鳢肠道中菌群丰度高于饲喂冰鲜鱼乌鳢组(图 6, 图 7); 通过种属分类水平上的统计分析, 确定饲喂冰鲜鱼乌鳢组肠道种属种类较少, 其中优势种属为邻单胞菌属(Plesiomonas)、狭义梭菌属(Clostridium sensu stricto)、鲸蜡菌属(Cetobacterium), 其他及未分类细菌; 饲喂畜禽内脏乌鳢组肠道种属种类比较丰富, 其中优势种属为狭义梭菌属(Clostridium sensu stricto)、邻单胞菌属(Plesiomonas)、Spartobacteria、甲基副球菌属(Methyloparacoccus)、杆菌属(Alsobacter)、Saccharibacteria、分支杆菌属(Mycobacterium)、其他及未分类细菌。
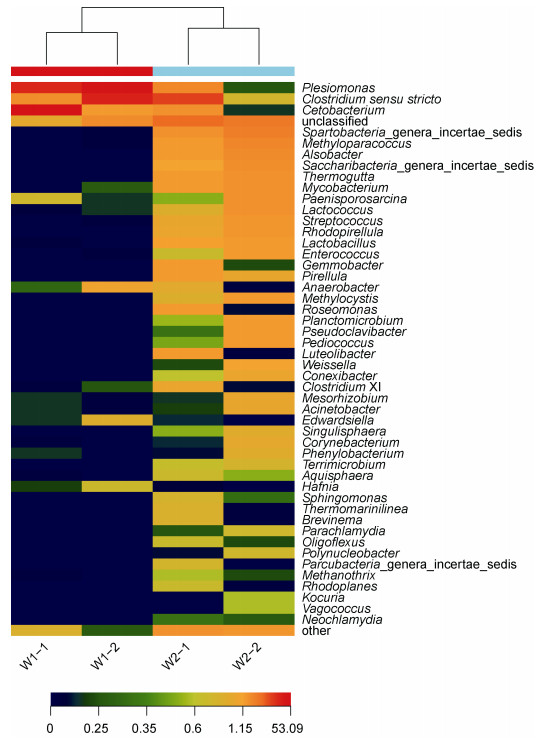 |
| 图 6 属分类水平多样本热图 Fig. 6 Microbial community heatmap at the genus level |
 |
| 图 7 属分类水平所有样本群落结构分布图 Fig. 7 Distribution of microbial community at the genus level |
目前很多养殖户为了降低养殖成本, 在乌鳢的养殖过程中投喂畜禽内脏, 投喂畜禽内脏对乌鳢的影响尚未明确。此外, 鱼类是典型的水生脊椎动物, 物种数目繁多, 其消化道中同样栖居着种类繁多的微生物, 发挥着重要的生理功能(王纯, 2017), 肠道中丰富的微生物已被证明对宿主具有举足轻重的影响(Benjamino et al, 2018)。水产动物肠道微生物存在于宿主肠道中特定的位置, 并且在宿主体内平衡、代谢、肠道发育、神经发育以及进化等方面发挥重要的作用(邢孟欣, 2013)。为了揭示一定时间内, 冰鲜鱼、畜禽内脏两种不同饲粮的组成是如何影响乌鳢肠道的菌群组成, 课题组设计了本试验。
上述研究结果表明, 投饲冰鲜鱼饲料和畜禽内脏饲料的乌鳢肠道细菌的优势菌群种类和数量方面相似度不高, 差异显著, 而且饲喂畜禽内脏饲料乌鳢肠道细菌的群落种类多样性、OTU丰富度和物种总数均高于饲喂冰鲜鱼乌鳢。饲喂畜禽内脏饲料乌鳢肠道细菌的优势类群为狭义梭菌属(Clostridium sensu stricto)、邻单胞菌属(Plesiomonas)、Spartobacteria、甲基副球菌属(methyloparacoccus)、杆菌属(Alsobacter)、Saccharibacteria、分支杆菌属(Mycobacterium), 而饲喂冰鲜鱼饲料乌鳢肠道细菌的优势类群为邻单胞菌属(Plesiomonas)、狭义梭菌属(Clostridiumsensustricto)、鲸蜡菌属(Cetobacterium)。饲喂冰鲜鱼饲料乌鳢和饲喂畜禽内脏饲料乌鳢肠道细菌有2种相同的优势类群, 分别为狭义梭菌属(Clostridium sensu stricto)和邻单胞菌属(Plesiomonas), 狭义梭菌属在低蛋白质的饲粮生长育肥猪粪便中有显著降低(王晶等, 2018), Fan等(2017)研究表明, 饲粮蛋白质水平的降低, 猪回肠中狭义梭菌属的数量明显降低, 分析原因认为是饲粮蛋白质水平的降低导致发酵底物氮源的缺乏而引起的。狭义梭菌属(Clostridium sensu stricto)同时是饲喂冰鲜鱼饲料乌鳢和饲喂畜禽内脏饲料乌鳢肠道的优势菌群, 这也与投喂饲粮有关, 冰鲜鱼和畜禽内脏均为高蛋白质饲粮。De La Rosa(2011)通过454焦磷酸测序技术发现邻单胞菌属是罗非鱼肠道中的优势属之一, 此外, 吴振聪等(2019)利用PCR-DGGE和Illumina测序技术研究了罗非鱼(Oreochromis niloticus)肠道菌群结构, 发现优势菌属为鲸杆菌属(Cetobacterium)、Neochlamydia、邻单胞菌属(Plesiomonas)、不动杆菌属(Acinetobacter)和分支杆菌属(Mycobacterium)等6个菌属和一些未知属。邻单胞菌属中的常见菌是一些条件致病菌, 如食用未煮熟的贝类会导致Plesiomonas shigelloides感染, 引起腹泻(Holmberg et al, 1984, 1986; Niedziela et al, 2002)。但Larsen等(2014)对斑点叉尾
本研究表明W1组饲喂冰鲜鱼饲料和W2组饲喂畜禽内脏饲料的乌鳢肠道细菌的优势菌群种类和数量方面相似度不高, 差异显著, 且W2组乌鳢肠道细菌群落种类的多样性、OTU的丰富度和物种的总数均高于W1乌鳢肠道; W1组肠道有益菌OTU丰富度大于W2组, 且W2组还存在一些潜在致病菌, 从乌鳢肠道微生物角度研究, 冰鲜鱼较畜禽内脏更适合作为乌鳢的饲粮。
王纯, 2017.芽孢杆菌V4和胶红酵母对鲑鳟鱼生长免疫及肠道菌群影响研究.青岛: 中国科学院大学(中国科学院海洋研究所)博士学位论文, 196 http://cdmd.cnki.com.cn/Article/CDMD-80068-1017099318.htm
|
王晶, 王四新, 刘辉, 等. 2018. 低蛋白质水平饲粮对生长育肥猪生长性能及肠道菌群的影响. 动物营养学报, 30(12): 4864-4873 DOI:10.3969/j.issn.1006-267x.2018.12.014 |
邢孟欣, 2013.养殖大菱鲆肠道微生物多样性及功能分析.青岛: 中国科学院研究生院(海洋研究所)博士学位论文, 129 http://cdmd.cnki.com.cn/Article/CDMD-80068-1013300040.htm
|
刘增新, 柳学周, 史宝, 等. 2017. 牙鲆(Paralichthys olivaceus)仔稚幼鱼肠道菌群结构比较分析. 渔业科学进展, 38(1): 111-119 |
吴振聪, 张莎莎, 杨转, 等. 2019. 投喂鲍内脏多糖对罗非鱼肠道菌群结构的影响. 中国水产科学, 26(3): 546-558 |
饶刘瑜, 李学梅, 李星浩, 等. 2018. 转基因鲤鱼与对照鲤肠道微生物群落差异研究. 水生生物学报, 42(2): 349-355 |
Bao Z H, Sato Y, Fujimura R et al, 2014. Alsobacter metallidurans gen. nov., sp. nov., a thallium-tolerant soil bacterium in the order Rhizobiales. International Journal of Systematic and Evolutionary Microbiology, 64(3): 775-780 |
Benjamino J, Lincoln S, Srivastava R et al, 2018. Low-abundant bacteria drive compositional changes in the gut microbiota after dietary alteration. Microbiome, 6: 86 DOI:10.1186/s40168-018-0469-5 |
Cardona E, Gueguen Y, Magré K et al, 2016. Bacterial community characterization of water and intestine of the shrimp Litopenaeus stylirostris in a biofloc system. BMC Microbiology, 16(1): 157 DOI:10.1186/s12866-016-0770-z |
De La Rosa M G N, 2011. Preliminary evaluation of the bacterial populations associated with the intestinal tract of tilapia (Oreochromis niloticus) exposed to essential oils of oregano in the diet. Bogotá: National University of Colombia Faculty of Sciences
|
Eissa A E, Zaki M M, Saeid S, 2011. Epidemic mortalities in the dusky grouper, Epinephelus marginatus (Lowe, 1834) in Egyptian coastal waters. In: Proceedings of the 4th Global Fisheries and Aquaculture Research Conference. Giza, Egyp: The Egyptian International Center for Agriculture, 111-122 https://www.mendeley.com/catalogue/epidemic-mortalities-dusky-grouper-epinephelus-marginatus-lowe-1834-egyptian-coastal-waters/
|
Fan P X, Liu P, Song P X et al, 2017. Moderate dietary protein restriction alters the composition of gut microbiota and improves ileal barrier function in adult pig model. Scientific Reports, 7: 43412 DOI:10.1038/srep43412 |
Hoefman S, van der Ha D, Iguchi H et al, 2014. Methyloparacoccus murrellii gen. nov., sp. nov., a methanotroph isolated from pond water. International Journal of Systematic and Evolutionary Microbiology, 64(6): 2100-2107 |
Holmberg S D, Farmer III J J, 1984. Aeromonas hydrophila and Plesiomonas shigelloides as causes of intestinal infections. Reviews of Infectious Diseases, 6(5): 633-639 DOI:10.1093/clinids/6.5.633 |
Holmberg S D, Wachsmuth I K, Hickman-Brenner F W et al, 1986. Plesiomonas enteric infections in the United States. Annals of Internal Medicine, 105(5): 690-694 DOI:10.7326/0003-4819-105-5-690 |
Lara-Flores M, Aguirre-Guzman G, Balan-Zetina S B et al, 2014. Identification of Mycobacterium agent isolated from tissues of Nile tilapia (Oreochromis niloticus). Turkish Journal of Fisheries and Aquatic Sciences, 14(1/2): 575-580 |
Larsen A M, Mohammed H H, Arias C R, 2014. Characterization of the gut microbiota of three commercially valuable warmwater fish species. Journal of Applied Microbiology, 116(6): 1396-1404 DOI:10.1111/jam.12475 |
Li T T, Long M, Gatesoupe F J et al, 2015. Comparative analysis of the intestinal bacterial communities in different species of carp by pyrosequencing. Microbial Ecology, 69(1): 25-36 DOI:10.1007/s00248-014-0480-8 |
Niedziela T, Lukasiewicz J, Jachymek W et al, 2002. Core oligosaccharides of Plesiomonas shigelloides O54:H2 (Strain CNCTC 113/92): structural and serological analysis of the lipopolysaccharide core region, the O-Antigen biological repeating unit, and the linkage between them. Journal of Biological Chemistry, 277(14): 11653-11663 DOI:10.1074/jbc.M111885200 |
Ravel J, Blaser M J, Braun J et al, 2014. Human microbiome science: vision for the future, Bethesda, MD, July 24 to 26.2013. Microbiome, 2(1): 16 DOI:10.1186/2049-2618-2-16 |
Rungrassamee W, Klanchui A, Maibunkaew S et al, 2014. Characterization of intestinal bacteria in wild and domesticated adult black tiger shrimp (Penaeus monodon). PLoS One, 9(3): e91853 DOI:10.1371/journal.pone.0091853 |
Tsuchiya C, Sakata T, Sugita H, 2008. Novel ecological niche of Cetobacterium somerae, an anaerobic bacterium in the intestinal tracts of freshwater fish. Letters in Applied Microbiology, 46(1): 43-48 |
Wu S G, Gao T H, Zheng Y Z et al, 2010. Microbial diversity of intestinal contents and mucus in yellow catfish (Pelteobagrus fulvidraco). Aquaculture, 303(1-4): 1-7 DOI:10.1016/j.aquaculture.2009.12.025 |
Xu Z J, Knight R, 2015. Dietary effects on human gut microbiome diversity. British Journal of Nutrition, 113(Suppl): S1-S5 |
 2020, Vol. 51
2020, Vol. 51


