
中国海洋湖沼学会主办。
文章信息
- 董忠典, 黎学友, 廖健, 张宁, 郭昱嵩, 王中铎. 2020.
- DONG Zhong-Dian, LI Xue-You, LIAO Jian, ZHANG Ning, GUO Yu-Song, WANG Zhong-Duo. 2020.
- 雌、雄弓背青鳉(Oryzias curvinotus)肝脏转录组比较分析
- COMPARATIVE TRANSCRIPTOME ANALYSIS OF MALE AND FEMALE LIVER OF ORYZIAS CURVINOTUS
- 海洋与湖沼, 51(5): 1203-1213
- Oceanologia et Limnologia Sinica, 51(5): 1203-1213.
- http://dx.doi.org/10.11693/hyhz20191200274
文章历史
-
收稿日期:2019-12-24
收修改稿日期:2020-03-01
肝脏是脊椎动物最重要的器官之一, 在物质能量代谢、激素合成及免疫反应等生理过程中具有重要意义(van der Oost et al, 2003; Tom et al, 2004; Lai et al, 2015; Zhang et al, 2017)。在鱼类中, 雌性肝脏还是卵黄蛋白原(vitellogenins, vtgs)、透明带蛋白前体(choriogenins, chgs)、雌激素受体(estrogen receptor, er)和细胞色素P450(cytochrome P450s, cyp450s)等基因的主要表达器官, 这些基因对环境中雌激素物质较为敏感, 当水体环境中存在雌激素类物质时, 可以诱导雄鱼肝脏表达这些基因(Lee et al, 2002; Chen et al, 2008; Lai et al, 2015)。
弓背青鳉(Oryzias curvinotus)是一种广泛分布于我国南海沿岸水域的小型鱼类, 为红树林水域的常见物种(Hayakawa et al, 2015; Wang et al, 2017)。弓背青鳉具有体型小、生长快、性成熟时间短和胚胎发育透明的特点, 可以用于实验室饲养繁殖; 其对盐度有较强的适应能力, 具备开发成海洋环境毒理及盐度适应研究新模式种的潜力(Hayakawa et al, 2015)。同时, 弓背青鳉的性染色体为XX/XY型, 具有性别标记, 是研究海洋鱼类性别决定和分化机制的潜在模型(Matsuda et al, 2003; 董忠典等, 2018)。同属的日本青鳉(Oryzias latipes)和海水青鳉(Oryzias melastigma)已被广泛应用于淡水和海水环境检测研究(Metcalfe et al, 2000; Chen et al, 2008; Hong et al, 2015; Cong et al, 2017; Abdel-moneim et al, 2018)。然而, 缺乏基因组和转录组信息, 在很大程度上限制了弓背青鳉资源的开发和利用。本研究采用RNA-Seq技术分别对雌、雄弓背青鳉肝脏进行了转录组测序、基因功能注释和表达谱分析, 以期为弓背青鳉基因资源的开发提供基础数据。
1 材料与方法 1.1 弓背青鳉的采集及总肝脏RNA提取本研究所用性成熟弓背青鳉(平均体长29.25mm, 平均体质量0.27g)采集自湛江雷州半岛近岸的高桥国家级红树林保护区(21°36′24"N, 109°47′8"E), 实验室暂养30d, 温度25±1℃, 盐度15, 光暗周期14h:10h。解剖通过性腺类型确定弓背青鳉生理性别, 雌雄各3尾取肝脏分别进行总RNA提取, 使用DNase I(宝生物, 大连)去除基因组污染。使用1%琼脂糖凝胶电泳分析RNA完整性, Nanodrop检测RNA的纯度(OD260/OD280比值), Qubit检测总RNA浓度, RNA满足进行下一步的文库构建的要求。对3尾雌性肝脏总RNA, 分别取等量(1.0μg)混合为一个RNA样品池(FL), 用同样的方法获得雄性肝脏RNA样品池(ML), 对FL和ML各取1.5μg总RNA, 分别进行cDNA文库构建和转录组测序。
1.2 文库构建及测序使用带Oligo(dT)的磁珠富集真核生物mRNA, 加入fragmentation buffer将mRNA打断成短片段, 以mRNA为模板, 用六碱基随机引物合成第一链cDNA, 然后加入缓冲液、dNTPs和DNA聚合酶和RNase H合成第二链cDNA, 再用AMPure XP beads纯化双链cDNA。纯化的双链cDNA先进行末端修复、加A尾并连接测序接头, 再用AMPure XP beads选择150—200bp片段。随后进行PCR扩增, 并用AMPure XP beads纯化PCR产物, 得到最终的文库。文库构建完成后, 用Qubit2.0进行初步定量并稀释至1.5ng/μL, 用Agilent 2100确认文库插入片段大小, 最后用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度 > 2nmol/L), 以保证文库质量。库检合格后, 进行Illumina HiSeq测序, cDNA文库的制备和测序由诺禾致源生物信息科技有限公司(天津)完成。
1.3 转录组测序质量控制和组装为了保证转录组分析质量, 对原始测序数据(raw reads)过滤, 去除带接头的reads、N(N表示无法确定碱基信息)的比例大于0.1%的reads、低质量(质量值Qphred < =20的碱基数占整个reads的50%以上)的reads, 得到高质量测序数据(clean reads)。采用Trinity软件对clean reads进行拼接(Grabherr et al, 2011)。
1.4 基因功能注释为获得全面的基因功能信息, 使用7大数据库对Trinity拼接获得的unigenes进行功能注释。使用在线BLAST程序对unigenes在Nr (http://www.ncbi.nlm.nih.gov)期望值e-value < e–5、Nt (http://www.ncbi.nlm.nih.gov) e-value < e–5、Swiss-Prot (http://www.expasy.ch/sprot) e-value < e–5、KOG (http://www.ncbi.nlm.nih.gov) e-value < e–3、KO (https://www.kegg.jp/kegg/ko.html) e-value < e–10和GO (http://www.geneontology.org/) e-value < e–6进行比对注释。通过HMMER3.0 (e-value < 0.01)程序对unigenes在Pfam数据库中进行蛋白家族注释(Eddy, 2011)。
1.5 基因表达水平和差异分析将Trinity拼接得到的转录组作为参考序列, 通过RSEM对每个样本的基因表达水平进行估计:将Clean data比对到组装好的参考序列上, 根据比对结果得到每个基因的Readcount数目, 采用TMM对read count数据进行标准化处理, 再用DEGseq进行差异分析, 筛选阈值为qvalue < 0.005且|log2(foldchange)| > 1 (Wang et al, 2010)。
1.6 差异基因GO和KEGG富集分析使用R软件中的GOseq对弓背青鳉雌雄肝脏转录组差异基因进行显著性富集分析(Young et al, 2010)。使用KOBAS软件检测差异基因在KEGG通路中富集情况(Mao et al, 2005)。
1.7 实时荧光定量PCR(RT-qPCR)验证利用RT-qPCR技术, 弓背青鳉雌雄肝脏转录组中选择12个基因进行定量验证(引物见表 1)。RT-qPCR反应体系15μL, 包含7.5μL 2×Power Green qPCR Mix (东盛生物, 广州), 0.6μL上下游引物(10μmol/L), 1.5μL cDNA, 4.8μL ddH2O。每个样品技术重复3次, 反应置于Roche LightCycler 96(罗氏, 瑞士)上运行。反应程序如下: 95℃ 3min; 95℃ 15s, 60℃ 15s, 72℃ 30s (采集荧光), 40个循环; 熔解曲线分析检测PCR产物的特异性。gapdh和adp作为内参基因, 使用SPSS17.0采用2–ΔΔCt方法对目的基因表达水平进行统计(Livak et al, 2001)。
| Gene ID | 基因 | 引物序列 |
| Cluster-16672.13788 | p4502k1 | F: AAGGGCTCTCCCATTCATTCTC R: CAAAGACTGGCAAACATCGTCC |
| Cluster-16672.14045 | camp | F: GATTTAGAGGAAGGTCCAACGG R: TGGTAGTCAACAACAGCAGTCAC |
| Cluster-16672.12590 | endoplasmin | F: AGCCGTGAAGTGGATGTGAG R: AAGACCGTATGGGACTGGGAAC |
| Cluster-16672.15532 | sult2b | F: AAAGGGAAAGACAAGCCAGC R: AATGAAACTGGAGAGCGAGC |
| Cluster-16672.14794 | utp | F: TGATGCTAACTGACCCGTGC R: TGGGACATAGGATAAAGGTGACG |
| Cluster-16672.13077 | gapdh | F: TTGTGATGGGTGTCAACCAC R: CTGTGATGGCGTGAACTGTG |
| Cluster-16672.13170 | adp | F: GCGATTACATCATCATCACACG R: AAAGAGCGGACCAAACTGAG |
| Cluster-16672.13129 | chgl | F: TCCTCTCTGGGTTCCATTCTC R: GCTTGACGGTAGCCTCTATGTG |
| Cluster-16672.13099 | chgh | F: CAACTCATCACAGTCGCTTCC R: CATCCGTTCTAATGGCTACAGC |
| Cluster-16672.13123 | vtg2 | F: CCCTGGCATCTCTACAAGTGTTC R: TTTGAATCCTCGCTGATAACG |
| Cluster-16672.13069 | vtg1 | F: TTGGCATCAGAAGAGCGTTC R: CAACATAAACAACAACCTGGTATCC |
| Cluster-16672.13027 | chghm | F: AGTCACCTCGTGTGGCACTG R: CTGTCCCTGGTAATGGCTCC |
| Cluster-16672.13113 | vtglike | F: TTTGAGGGATTGACTGGTCTG R: TTGAAGGTTCTGGCATCACC |
| Cluster-16672.12461 | p45027c | F: TGTTCACACACACTGCCTTGAG R: TCAGCCTCGGACAATAGACG |
采用MISA (http://pgrc.ipk-gatersleben.de/misa/misa.html)对弓背青鳉肝脏转录组拼接获得的unigenes进行微卫星标记(SSR)检测, 设置各重复单元类型的最少重复次数为: 1—10、2—6、3—5、4—5、5—5、6—5。以1—10为例, 该设置表示单核苷酸重复类型至少重复10次才被算为微卫星。
2 结果 2.1 测序结果与组装本研究分别构建了雌、雄弓背青鳉肝脏cDNA文库, 并进行了转录组测序。测序结果经过质控后, 分别获得80095044和87043984条Clean reads。经Trinity组装, 共得到49912个unigenes, 其中N50长度为2394bp, 其中长度大于1000bp的unigenes共计25423个(图 1)。
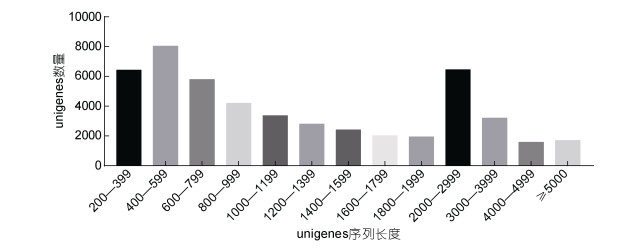 |
| 图 1 unigenes序列长度分布图 Fig. 1 The distribution of sequence length of the unigenes |
通过BLAST比对, 分别在NR、NT、SwissProt、PFAM、KOG数据库中注释到32651、47988、19005、25587和19005个unigenes。与NR数据库比对发现有32649个unigenes与390个物种基因序列高度同源, 其中77.6%(25349个)的unigenes和日本青鳉基因序列同源, 其次有3.6%的unigenes和深裂眶锯雀鲷(Stegastes partitus)同源(图 2)。为进一步了解弓背青鳉雌、雄肝脏表达基因集的功能, 将拼接的unigenes在GO和KO数据库中进行了比对分析。25630个unigenes被归类到GO三个大类(生物学过程、分子功能和细胞成分)55个功能分类中(图 3)。生物学过程包括25个功能分类, 参与“细胞过程”(15094)和“代谢过程”(12821)的基因最多; 细胞成分包含20个功能分类, “细胞”(8419)和“细胞部分”(8419)基因最多; 分子功能包含10个功能分类, “结合”(16155)和“催化活性”(10114)基因最多。对基因做KO注释后, 可根据它们参与的KEGG代谢通路进行分类。共有18887个unigenes在KO数据库中获得注释, 富集到232个KEGG信号通路中, 其中“PI3K-Akt”信号通路含有最多(657)的unigenes, 其次是“内吞”(615)、“斑黏连”(521)和“肌动蛋白骨架”的调节通路(508)。
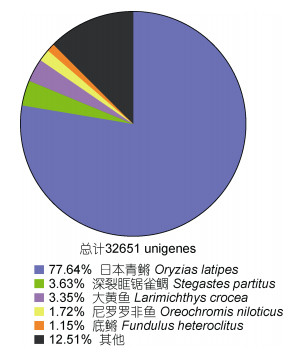 |
| 图 2 弓背青鳉unigenes同源物种分布 Fig. 2 Distribution of homologous species of O. curvinotus unigenes |
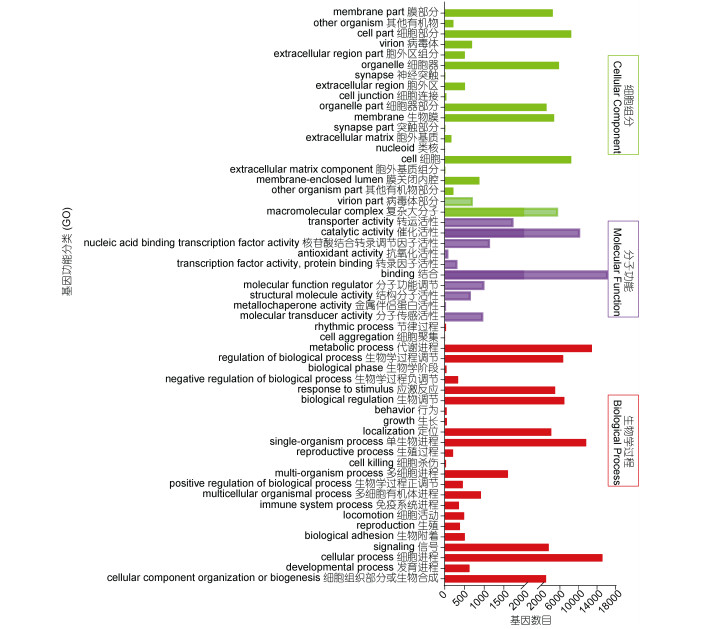 |
| 图 3 弓背青鳉unigenes GO分类图 Fig. 3 Gene ontology (GO) assignment of assembled unigenes of O. curvinotus 注:纵坐标为GO三个大类的下一层级的GO term, 横坐标为注释到该term下的基因个数 |
通过DEGseq对弓背青鳉雌、雄肝脏转录组基因进行差异表达分析(差异基因筛选条件为: qvalue < 0.005 & |log2(foldchange)| > 1), 结果如图 4所示, 有207个unigenes在雌鱼肝脏中上调, 364个unigenes在雄鱼肝脏上调。对差异基因(DEGs)进行GO和KEGG富集分析, GO富集结果显示雌性肝脏高表达基因主要参与蛋白质合成过程, 雄性高表达基因主要参与免疫及氧化还原反应(图 5)。KEGG分析结果显示DEGs富集到244个KEGG通路中, 涉及核糖体、PPAR信号通路、卵巢类固醇激素生成、类固醇生物合成、不饱和脂肪酸合成和内质网蛋白质加工等通路(图 6)。
 |
| 图 4 雌、雄弓背青鳉肝脏基因差异表达分析火山图 Fig. 4 Volcano plot of differences in gene expression between female liver (FL) and male liver (ML) 注:横坐标代表基因在FL/ML不同样品中表达倍数变化; 纵坐标代表基因表达量变化的统计学显著程度。图中的散点代表各个基因, 蓝色圆点表示无显著性差异的基因, 红色圆点表示FL有显著性差异的上调基因, 绿色圆点表示ML有显著性差异的下调基因 |
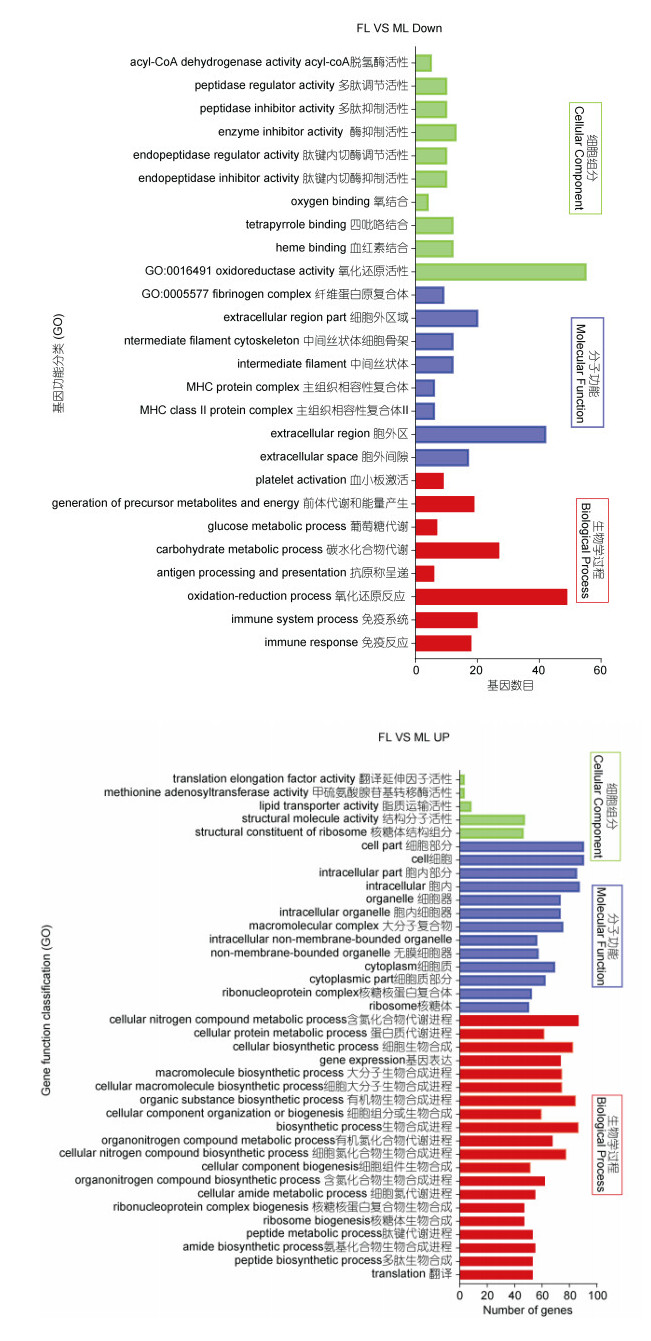 |
| 图 5 弓背青鳉雌雄肝脏差异基因GO富集 Fig. 5 GO assignment of differentially expressed genes (DEGs) of O. curvinotus 注:纵坐标为GO三个大类的下一层级的GO term, 横坐标为注释到该term下的基因个数 |
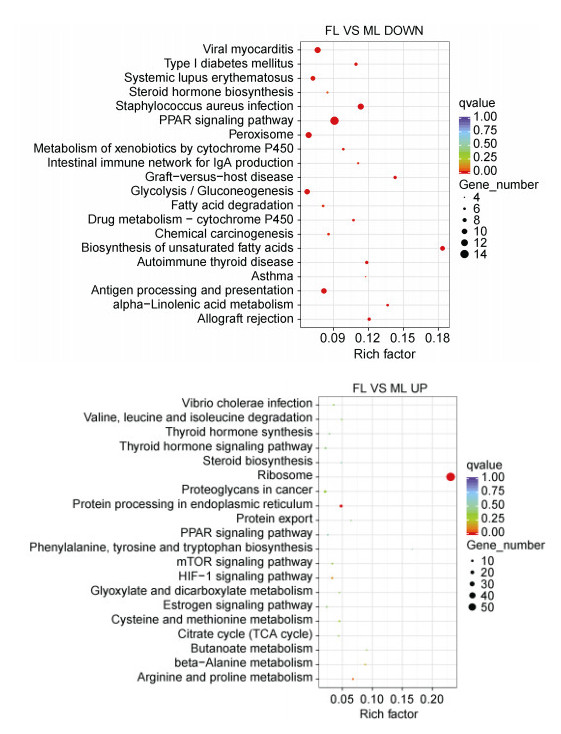 |
| 图 6 弓背青鳉雌雄肝脏差异基因KEGG富集 Fig. 6 KEGG assignment of differentially expressed genes of O. curvinotus 注:纵轴表示pathway名称, 横轴表示pathway对应的Rich factor, qvalue的大小用点的颜色来表示, qvalue越小则颜色越接近红色, 每个pathway下包含的差异基因的多少用点的大小来表示 |
本研究选择了在雌、雄弓背青鳉肝脏RNA-Seq分析中的12个差异表达基因, 通过RT-qPCR技术对RNA-Seq结果进行验证。所选的验证基因包括环腺苷三磷酸依赖的转录因子camp, 内质网基因endoplasmin, UDP-葡萄糖醛酸转移酶基因utp, 磺基转移酶家族2B基因sult2b及部分检测环境雌激素物质的标记基因如卵黄蛋白原基因vtg1、vtg2、vtglike, 透明带蛋白前体基因chgl、chgh、chghm, 细胞色素P450基因p4502k1、p45027c。结果表明所选基因的表达模式与RNA-Seq分析相一致, 说明RNA-Seq分析结果可信(见图 7)。
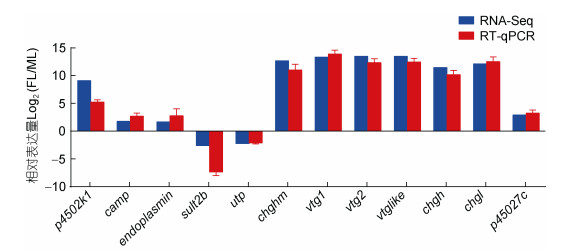 |
| 图 7 RT-qPCR检测RNA-Seq结果 Fig. 7 Validation of RNA-Seq data using RT-qPCR 注: RNA-Seq结果为log2(雌性read count /雄性read count), RT-qPCR为log2(FL/ML)gapdh和adp作为内参基因, 每种性别选3个生物学重复(n=3) |
利用MISA软件对弓背青鳉进行SSR标记鉴定, 用于遗传多样性研究。有18326条unigenes序列含有SSR标记, 共计28195个, 其中6373条unigenes包含一个以上的SSR。各种类型的SSR标记出现的频率不同(表 2), 单核苷酸重复SSR出现的频率最高, 占总SSR的73.17%, 其次是三核苷酸重复SSR, 占总SSR的19%。另外, 不同重复序列的相对丰度差异很大。A/T在单核苷酸SSR中最常见, 在双核苷酸SSR中, AG/CT最为常见。AGG/CCT和AAAC/GTTT是三、四核苷酸SSR中最常见的元件。
| 统计类别 | 数目 |
| 序列总数 | 49912 |
| 碱基总数(bp) | 76849041 |
| SSR位点总数 | 28195 |
| 含有SSR位点序列 | 18326 |
| 含有1个以上SSR位点的序列 | 6373 |
| 单核苷酸重复 | 20631 |
| 二核苷酸重复 | 2901 |
| 三核苷酸重复 | 4282 |
| 四核苷酸重复 | 349 |
| 五核苷酸重复 | 27 |
| 六核苷酸重复 | 5 |
为了阐明弓背青鳉肝脏表达基因的功能和涉及的生物学过程, 本研究分别构建了性成熟雌、雄弓背青鳉肝脏cDNA文库, 并在Illumina平台进行了RNA-Seq测序, 分别获得了80095044和87043984条Clean reads。N50长度是评价RNA-Seq组装质量的重要参数, 本研究弓背青鳉肝脏转录组N50长度为2394bp, 高于多个已发表的水生动物肝脏转录组, 包括海水青鳉(2162bp) (Lai et al, 2015), 七鳃鳗(lampetra japonica) (1447bp) (李庆伟等, 2018), 脂鲤(Pterygoplichthys anisitsi) (1571bp) (Parente et al, 2017), 低于海鲈(Dicentrarchus labrax) (3257bp) (Magnanou et al, 2014)。对NR数据库比对, 发现有77.64% (32649条unigenes中的25349个)的可比对序列与日本青鳉基因同源(图 2), 可能是因为弓背青鳉和日本青鳉均属于青鳉属, 亲缘关系密切(Wang et al, 2017)。
肝脏在鱼类生长和物质能量代谢中起着重要的作用。在弓背青鳉肝脏转录组中, “细胞过程”、“细胞”和“结合”分别在GO三大功能类别中富集(图 3)。这与其他水生动物的肝脏RNA-Seq结果类似, 说明属于这几类功能的基因在物种间保守(Magnanou et al, 2014; Chen et al, 2016; 张燕萍等, 2018)。KEGG的注释结果表明, 弓背青鳉肝脏转录组中unigenes主要参与了信号转导(3130)、内分泌系统(1536)及免疫系统(1491)的生物学过程。结果可能提示了肝脏在弓背青鳉生命过程中的主要功能, 为了解肝脏的功能演变奠定了基础。
3.2 弓背青鳉免疫相关基因肝脏是鱼类的重要免疫器官, 通过KEGG分析, 从弓背青鳉肝脏转录组中筛选出了多个可能参与免疫相关防御途径的unigenes(表 3): 373个unigenes参与了血小板活化, 272个unigenes参与了白细胞跨内皮迁移, 245个unigenes参与了T细胞受体信号通路, 136个unigenes参与了补体和凝血级联, 188个unigenes参与了自然杀伤细胞介导的细胞毒性, 163个unigenes参与了Toll-样受体信号通路等过程。在七鳃鳗的肝脏转录组中也有相似的结果(李庆伟等, 2018)。
| 通路编号 | KEGG通路名称 | 基因数目 |
| ko04612 | 抗原处理及呈递 | 123 |
| ko04662 | B细胞受体信号通路 | 179 |
| ko04062 | 趋化因子信号通路 | 353 |
| ko04610 | 补体和凝集系统 | 136 |
| ko04623 | 胞质DNA检验通路 | 63 |
| ko04664 | Fc epsilon RI信号通路 | 157 |
| ko04666 | Fc gamma R信号调理吞噬 | 241 |
| ko04640 | 造血 | 79 |
| ko04672 | IgA生产的肠道免疫网络 | 45 |
| ko04670 | 白细胞跨内皮迁移 | 272 |
| ko04621 | NOD样受体信号通路 | 93 |
| ko04650 | 自然杀伤细胞介导细胞毒性 | 188 |
| ko04611 | 血小板活性 | 373 |
| ko04622 | RIG-样受体信号通路 | 109 |
| ko04660 | T细胞受体信号通路 | 245 |
| ko04620 | Toll-样受体信号通路 | 163 |
血小板上具有toll样受体, 在机体的先天免疫应答中起着关键作用。当病原菌与血小板结合时, 血小板活化同时分泌抗菌肽, 引起机体免疫反应(Cox et al, 2011)。白细胞跨内皮迁移在先天性和适应性免疫系统应答中起重要作用(Muller, 2011)。当机体受损时, 就会发生炎症反应, 它涉及血液中预先形成的可溶性元素迅速而短暂地运送到受伤部位, 随后是较长时间的白细胞运送, 经内皮细胞迁移可能是炎症反应的不可逆点(Jimenez et al, 2010; Muller, 2011)。补体和凝集通路在机体固有免疫防御中具有重要意义(Bajic et al, 2016), 在小瓜虫感染的大黄鱼(Larimichthys crocea)早期免疫中发挥重要作用; 另外, 补体和凝集通路在鳗弧菌感染后半滑舌鳎的免疫反应中也发挥了重要作用(Zhang et al, 2015; Yin et al, 2016)。综上, 我们推测弓背青鳉的肝脏可能在抵御病原体入侵的免疫防御中发挥重要作用。
3.3 环境内分泌干扰物的生物标志物鱼类肝脏常被用作检测环境中雌激素物质的靶器官, 通常用于检测环境雌激素物质的标记基因在雌鱼肝脏中大量表达, 如chgs、vtg、cyp450s和er (Bucheli et al, 1995; Arukwe et al, 2001; Lee et al, 2002; Chen et al, 2008)等。Chgs基因编码放射带蛋白(ZPs)的前体, ZPs占据了鱼卵壳的大部分, 由ZP-1、2和ZP-3两个主要亚基群组成(Hamazaki et al, 1989; Murata et al, 1991)。在性成熟雌鱼中, 雌激素诱导Chgs在肝脏中合成, 随血液流动整合到放射带中。当雄鱼被环境雌激素物质刺激时, 肝脏也产生Chgs。在日本青鳉中, 双酚A、壬基酚和乙炔雌二醇均可以诱导Chgl和Chgh的表达, 表达量的增加与雌激素剂量呈正相关(Lee et al, 2002)。Chen等(2008)研究表明海水青鳉chgl和chgh基因对环境雌激素物质也有相似的响应。卵黄蛋白原是硬骨鱼类卵黄蛋白前体, 虽然Vtgs在成熟的雌性体内自然存在, 而在雄性体内则不存在, 但雌性激素或环境雌激素分泌物可以诱导雄性产生Vtgs。因此, 雄性肝脏中的vtgs转录产物, 可用于指示检测对象过去或当前暴露于雌激素或具有雌激素效应的环境干扰物。本研究在弓背青鳉肝脏转录产物中发现了多种环境雌激素的生物标志物, 这些基因在性成熟雌性肝脏中的表达水平明显高于雄性肝脏, 可用于将弓背青鳉开发成广盐性环境监测模式物种。
3.4 SSR标记SSRs是一种重要的分子标记, 可作为群体遗传学研究的资源。本研究, 从18326个unigenes中共获得28195个SSRs。在已鉴定的SSRs中, 单核苷酸重复基序最为丰富, 重复核苷酸较多的SSRs较少(表 2), 这与斑石鲷(Oplegnathus punctatus)(Du et al, 2017)、花蟹(Charybdis feriatus)(Zhang et al, 2017)及多鳞鱚(Sillago sihama)(Tian et al, 2019)的SSRs分析相一致。本研究获得的弓背青鳉SSR标记, 为遗传多样性研究提供了数据, 也有助于进一步绘制弓背青鳉遗传连锁图谱和开发弓背青鳉遗传资源。
4 结论本研究首次获得了弓背青鳉雌雄肝脏转录组。共获得49912个unigenes, 48391个unigenes被定位到主要数据库, 丰富了弓背青鳉的功能基因资源。通过功能分析推测肝脏可能在弓背青鳉抵御病原体入侵的免疫防御中发挥作用, 同时鉴定到了多个环境雌激素生物标志基因(vtgs, chgs, er, cyp450等), 为将其开发成环境监测物种提供了基础。此外, 从18326个含有SSR序列的unigenes中获得了28195个SSR, 为弓背青鳉遗传多样性的研究提供数据。
李庆伟, 华一杉, 冯少姝, 等. 2018. 基于RNA-Seq技术的日本七鳃鳗(Lampetra japonica)肝脏转录组从头组装及分析. 辽宁师范大学学报(自然科学版), 41(1): 68-78 |
张燕萍, 章海鑫, 崔璀, 等. 2018. 基于RNA-seq的黄尾鲴肝脏转录组测序与分析. 水生态学杂志, 39(6): 87-94 |
董忠典, 龙水生, 黄承勤, 等. 2018. 一种快速鉴定弓背青鳉遗传性别的方法. 广东海洋大学学报, 38(3): 25-29 |
Abdel-moneim A, Mahapatra C T, Sepúlveda M S, 2018. In vivo visual reporter system for estrogenic contaminant exposure using transgenic see-through Japanese medaka Oryzias latipes. Chemosphere, 201: 251-253 DOI:10.1016/j.chemosphere.2018.02.184 |
Arukwe A, Kullman S W, Hinton1 D E, 2001. Differential biomarker gene and protein expressions in nonylphenol and estradiol-17β treated juvenile rainbow trout (Oncorhynchus mykiss). Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology, 129(1): 1-10 |
Bajic G, Degn S E, Thiel S et al, 2016. Complement activation, regulation, and molecular basis for complement-related diseases. The EMBO Journal, 34(22): 2735-2757 |
Bucheli T D, Fent K, 1995. Induction of cytochrome P450 as a biomarker for environmental contamination in aquatic ecosystems. Critical Reviews in Environmental Science and Technology, 25(3): 201-268 DOI:10.1080/10643389509388479 |
Chen X P, Li V W T, Yu R M K et al, 2008. Choriogenin mRNA as a sensitive molecular biomarker for estrogenic chemicals in developing brackish medaka (Oryzias melastigma). Ecotoxicology and Environmental Safety, 71(1): 200-208 |
Chen Q L, Luo Z, Huang C et al, 2016. De novo characterization of the liver transcriptome of javelin goby Synechogobius hasta and analysis of its transcriptomic profile following waterborne copper exposure. Fish Physiology and Biochemistry, 42(3): 979-994 DOI:10.1007/s10695-015-0190-2 |
Cong Y, Jin F, Wang J Y et al, 2017. The embryotoxicity of ZnO nanoparticles to marine medaka, Oryzias melastigma. Aquatic Toxicology, 185: 11-18 DOI:10.1016/j.aquatox.2017.01.006 |
Cox D, Kerrigan S W, Watson S P, 2011. Platelets and the innate immune system:mechanisms of bacterial-induced platelet activation. Journal of Thrombosis and Haemostasis, 9(6): 1097-1107 DOI:10.1111/j.1538-7836.2011.04264.x |
Du X X, Wang B, Liu X M et al, 2017. Comparative transcriptome analysis of ovary and testis reveals potential sex-related genes and pathways in spotted knifejaw Oplegnathus punctatus. Gene, 637: 203-210 DOI:10.1016/j.gene.2017.09.055 |
Eddy S R, 2011. Accelerated profile HMM searches. PLoS Computational Biology, 7(10): e1002195 DOI:10.1371/journal.pcbi.1002195 |
Grabherr M G, Haas B J, Yassour M et al, 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nature Biotechnology, 29(7): 644-652 DOI:10.1038/nbt.1883 |
Hamazaki T S, Nagahama Y, Iuchi I et al, 1989. A glycoprotein from the liver constitutes the inner layer of the egg envelope (zona pellucida interna) of the fish, Oryzias latipes. Developmental Biology, 133(1): 101-110 DOI:10.1016/0012-1606(89)90301-1 |
Hayakawa H, Le Q D, Kinoshita M et al, 2015. Genetic similarity of the Hainan medaka populations collected from hyper-and hypo-osmotic environments in northern Vietnam. Ocean Science Journal, 50(2): 231-235 DOI:10.1007/s12601-015-0020-6 |
Hong H Z, Shen R, Liu W X et al, 2015. Developmental toxicity of three hexabromocyclododecane diastereoisomers in embryos of the marine medaka Oryzias melastigma. Marine Pollution Bulletin, 101(1): 110-118 |
Jimenez J J, Jy W, Mauro L M et al, 2010. Transendothelial migration of leukocytes is promoted by plasma from a subgroup of immune thrombocytopenic purpura patients with small-vessel ischemic brain disease. American Journal of Hematology, 83(3): 206-211 |
Lai K P, Li J W, Wang S Y et al, 2015. Tissue-specific transcriptome assemblies of the marine medaka Oryzias melastigma and comparative analysis with the freshwater medaka Oryzias latipes. BMC Genomics, 16(1): 135 DOI:10.1186/s12864-015-1325-7 |
Lee C, Jin G N, Lee K C et al, 2002. Choriogenin mRNA induction in male medaka, Oryzias latipes as a biomarker of endocrine disruption. Aquatic Toxicology, 61(3-4): 233-241 DOI:10.1016/S0166-445X(02)00060-7 |
Livak K J, Schmittgen T D, 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 25(4): 402-408 |
Magnanou E, Klopp C, Noirot C et al, 2014. Generation and characterization of the sea bass Dicentrarchus labrax brain and liver transcriptomes. Gene, 544(1): 56-66 DOI:10.1016/j.gene.2014.04.032 |
Mao X Z, Cai T, Olyarchuk J G et al, 2005. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary. Bioinformatics, 21(19): 3787-3793 DOI:10.1093/bioinformatics/bti430 |
Matsuda M, Sato T, Toyazaki Y et al, 2003. Oryzias curvinotus has DMY, a gene that is required for male development in the medaka, O. latipes. Zoological Science, 20(2): 159-161 |
Metcalfe T L, Metcalfe C D, Kiparissis Y et al, 2000. Gonadal development and endocrine responses in Japanese medaka (Oryzias latipes) exposed to o, p'-DDT in water or through maternal transfer. Environmental Toxicology and Chemistry, 19(7): 1893-1900 DOI:10.1002/etc.5620190725 |
Muller W A, 2011. Mechanisms of leukocyte transendothelial migration. Annual Review of Pathology:Mechanisms of Disease, 6: 323-344 DOI:10.1146/annurev-pathol-011110-130224 |
Murata K, Hamazaki T S, Iuchi I et al, 1991. Spawning female-specific egg envelope glycoprotein-like substances in Oryzias latipes. Development, Growth and Differentiation, 33(6): 553-562 DOI:10.1111/j.1440-169X.1991.00553.x |
Parente T E, Moreira D A, Magalhães M G P et al, 2017. The liver transcriptome of suckermouth armoured catfish (Pterygoplichthys anisitsi, Loricariidae):Identification of expansions in defensome gene families. Marine Pollution Bulletin, 115(1-2): 352-361 DOI:10.1016/j.marpolbul.2016.12.012 |
Tian C X, Li Z Y, Dong Z D et al, 2019. Transcriptome analysis of male and female mature gonads of silver sillago (Sillago sihama). Genes, 10(2): E129 |
Tom M, Chen N, Segev M et al, 2004. Quantifying fish metallothionein transcript by real time PCR for its utilization as an environmental biomarker. Marine Pollution Bulletin, 48(7-8): 705-710 DOI:10.1016/j.marpolbul.2003.10.013 |
van der Oost R, Beyer J, Vermeulen N P E, 2003. Fish bioaccumulation and biomarkers in environmental risk assessment:a review. Environmental Toxicology and Pharmacology, 13(2): 57-149 DOI:10.1016/S1382-6689(02)00126-6 |
Wang L K, Feng Z X, Wang X et al, 2010. DEGseq:an R package for identifying differentially expressed genes from RNA-seq data. Bioinformatics, 26(1): 136-138 DOI:10.1093/bioinformatics/btp612 |
Wang Z D, Long S S, Liao J et al, 2017. Complete mitogenome of Hainan medaka Oryzias curvinotus (Teleostei:Beloniformes) and transcriptional differences between male and female liver. Mitochondrial DNA Part B, 2(1): 157-158 DOI:10.1080/23802359.2017.1303340 |
Yin F, Gao Q X, Tang B J et al, 2016. Transcriptome and analysis on the complement and coagulation cascades pathway of large yellow croaker (Larimichthys crocea) to ciliate ectoparasite Cryptocaryon irritans infection. Fish & Shellfish Immunology, 50: 127-141 |
Young M D, Wakefield M J, Smyth G K et al, 2010. Gene ontology analysis for RNA-seq:accounting for selection bias. Genome Biology, 11(2): R14 |
Zhang X, Wang S L, Chen S L et al, 2015. Transcriptome analysis revealed changes of multiple genes involved in immunity in Cynoglossus semilaevis during Vibrio anguillarum infection. Fish & Shellfish Immunology, 43(1): 209-218 |
Zhang X Y, Wen H S, Wang H L et al, 2017. RNA-Seq analysis of salinity stress-responsive transcriptome in the liver of spotted sea bass (Lateolabrax maculatus). PLoS One, 12(3): e0173238 DOI:10.1371/journal.pone.0173238 |
 2020, Vol. 51
2020, Vol. 51


