中国海洋湖沼学会主办。
文章信息
- 吴海燕, 陈佳琦, 董晨帆, 王松, 崔海栋, 谭志军. 2020.
- WU Hai-Yan, CHEN Jia-Qi, DONG Chen-Fan, WANG Song, CUI Hai-Dong, TAN Zhi-Jun. 2020.
- UHPLC-LTQ-Orbitrap-MS非定向筛查贝类中氮杂螺环酸毒素及其代谢产物
- UNTRAGETED SCREENING OF AZASPIRACIDS AND ITS METABOLITES IN SHELLFISH AND TOXIN PRODUCING ALGAE BY UHPLC-LTQ-ORBITRAP
- 海洋与湖沼, 51(6): 1432-1439
- Oceanologia et Limnologia Sinica, 51(6): 1432-1439.
- http://dx.doi.org/10.11693/hyhz20200300057
文章历史
-
收稿日期:2020-03-06
收修改稿日期:2020-04-23
2. 上海海洋大学食品学院 上海 201306;
3. 青岛卫辽医用生物材料有限公司 青岛 266071
2. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China;
3. Qingdao Value & Value Medical Biomaterial Co., Ltd, Qingdao 266071, China
氮杂螺环酸毒素(azaspiracids, AZAs)是一类具有独特聚醚类螺环结构(图 1)的脂溶性贝类毒素, 具有毒性强、残留高且代谢慢等显著特点, 因此成为国际研究热点和管控重点(EFSA, 2008; Furey et al, 2010; Hess et al, 2016; Pelin et al, 2018)。AZAs主要由环胺藻(Azadinium spp.)产生, 通过食物链传递到海洋贝类中, 并经贝类的生物转化作用产生多种代谢产物, 从而对消费者带来食用安全风险(Hess et al, 2016)。一般来说, 环胺藻产生的AZAs组分主要为AZA1、AZA2和AZA3中的一种或两种(Rossi et al, 2017; Krock et al, 2019), 也是毒性最强的三种组分(Pelin et al, 2018)。最新研究确定产毒藻原生AZA多达26种(Tillmann et al, 2017; Krock et al, 2019), 且经过贝类代谢(Jauffrais et al, 2013; Ji et al, 2018)后可形成超过50余种的代谢产物(Miles et al, 2018), 而且部分代谢产物如AZA17和AZA19的毒性较高(Krock et al, 2015)。贝类中AZAs的组成及其毒性强度总和决定了产品食用安全性, 因此采用高通量AZA及其代谢产物筛查方法进行检测与安全性评估尤为重要。
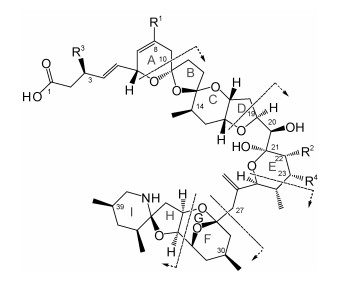 |
| 图 1 Azaspiracids毒素的化学结构 Fig. 1 The chemical structure of azaspiracids |
AZAs的检测方法包括液相色谱法、液相色谱串联质谱法、酶联免疫检测方法(Samdal et al, 2015; Leonardo et al, 2017)等。由于毒素标准品的匮乏, 仅能分析鉴别AZA1—3三种组分, 而多达40余种的代谢产物尚缺少标准物质(吴海燕等, 2016)。同时, 贝类制品的基质较为复杂, 目前仅通过前处理过程增加如QuECHERS技术(韩深等, 2013)、固相萃取柱等净化方法, 虽可显著降低贝类基质干扰, 但在代谢物非定向筛查和鉴定中, 仍存在不同程度的基质干扰效应, 给定性鉴别来带一定困难。近年来, 线性离子阱、静电场轨道阱以及飞行时间高分辨质谱(韩深等, 2014)在多毒素筛查、代谢产物的发掘和结构鉴定等方面均有广泛应用。其中, Rehmann等(2008)使用超高效液相色谱串联质谱法确定了AZA12-32的结构; 基于高分辨串联质谱与核磁共振的鉴别技术也同时应用于新型毒素37-epi-1鉴定(Rehmann et al, 2008; Kilcoyne et al, 2014)、AZA7—9 (Kilcoyne et al, 2015)、AZA26、AZA33、AZA34和AZA37 (Kilcoyne et al, 2018)等新型AZAs代谢产物结构鉴定。但目前的检测方法较为单一, 主要是限量3种毒素定量方法和单一化合物结构鉴定方法, 缺乏系统的非定向筛查与确证方法。
本研究利用固相萃取净化前处理, 结合超高效液相色谱-线性离子阱/静电场轨道阱组合式高分辨质谱整合全扫描二级质谱模式Full MS/dd MS2、目标离子扫描二级质谱模式Target-SIM/dd MS2、平行反应监测模式PRM模式等多种数据采集、挖掘策略快速分析方法, 分析贝类样品中的AZAs及其代谢产物, 解析质谱裂解规律及特征碎片离子组成, 为研究贝类中AZAs的代谢机制提供了快速、直观和准确的参考方法。
1 材料与方法 1.1 材料与试剂甲醇、乙腈(HPLC级, 美国Merk公司); 乙酸铵(HPLC级, 美国Sigma Aldrich公司); 超纯水(18.2MΩ·cm); Strata-X固相萃取柱(200mg/6mL, 美国Waters公司), 其他未作特殊说明的试剂均为分析纯。AZA1 [CRM-AZA1, (1.24±0.07)g/mL], AZA2 [CRM- AZA2, (1.28±0.05)g/mL], AZA3 [CRM-AZA3, (1.04± 0.04)g/mL]以及AZA阳性基质样品, 其各组分含量分别为AZA1 (1.16μg/g), AZA2 (0.273μg/g)和AZA3 (0.211μg/g), 购自加拿大国家研究理事会海洋生物科学研究所。
腹孔环胺藻(Azadinium poporum)为我国海域分布的AZA产毒藻(Krock et al, 2014), 具有独特的AZA组成(Krock et al, 2019), 由国家海洋局第三海洋研究所提供。实验室以f/2培养液单种培养, 温度(20±1)℃, 光照6000lx, 光暗比12h︰12h。经检测该藻主要产生AZA2, 单细胞产度能力一般为(7.05± 0.52)fg/cell。
栉孔扇贝(Chlamys Farreri)产自青岛市胶南灵山湾养殖海域的2龄贝(67mm±8mm)进行腹孔环胺藻(AZA2产毒藻)暴露试验(吴海燕等, 2017), 分别取3、7和22d内脏团样品, 均质后冷藏于-18℃, 作为AZA代谢实验样本。
1.2 仪器与设备超高效液相色谱-线性离子阱/静电场轨道阱组合式高分辨质谱联用仪(UHPLC-LTQ-Orbitrap-MS):美国赛默飞世尔公司产品, 配有加热喷雾离子源(HESI)、Xcalibur 2.1化学工作站以及Compound discovery 2.1数据比对分析软件; Dionex UltiMate 3000超高效液相色谱系统:美国赛默飞世尔公司产品, 配有变色龙色谱工作站。Himac CR 22GⅡ高速离心机(日本Hitachi公司); N-EVAP 112氮吹仪(美国Organomation公司); Milli-Q超纯水仪(美国Millipore公司); 57250-U固相萃取装置(美国Supelco公司)。
1.3 仪器检测方法 1.3.1 色谱条件色谱柱: Kinetex XB-C18 (100mm×2.1mm, 2.6μm); 柱温: 40℃; 流速: 0.30mL/min; 进样量: 5μL; 流动相: A为0.15% (v/v)甲酸水溶液, B为乙腈; 洗脱梯度: 0—7.0min, 20%— 90% B; 90% B持续3min; 10.0—10.1min, 90%—20% B; 20% B持续1min。
1.3.2 质谱条件加热电喷雾离子源(HESI), 正离子模式; 喷雾电压: 3.5kV; 毛细管温度320℃; 加热器温度: 50℃; 鞘气: 40arb; 辅助气: 5arb;
扫描模式: Full MS/dd MS2与Target-SIM/dd MS2:采集范围m/z 250—1500;一级全扫描(Full MS)分辨率为70000FWHM, C-trap最大容量(AGC target): 5×105, Maximum IT时间: 100ms; 数据依赖二级子离子扫描(dd-MS2)分辨率为17500FWHM, C-trap最大容量(AGC target): 1×105, C-trap最大注入时间: 50ms。碰撞能量CE为30、50、70。
平行反应监测模式PRM模式:采集范围m/z 250—1500; C-trap最大容量(AGC target): 2×105, Maximum IT时间: 100ms; 碰撞能量CE为30、50、70。
1.4 样品制备方法称取(2.00±0.02)g样品, 分别用3 mL甲醇提取3次, 离心后合并上清液, 于40℃氮吹至约1mL, 加入3mL超纯水待净化。依次用1mL甲醇、1mL 30% (v/v)甲醇水溶液活化Strata-X固相萃取柱, 然后加入提取液, 再用1mL 20% (v/v)甲醇水溶液淋洗, 最后用1mL 0.3% (v/v)氨水甲醇溶液洗脱, 收集洗脱液, 用甲醇定容至1mL过0.22μm滤膜, 上机分析。
1.5 数据采集与处理本研究运用UHPLC-LTQ-Orbitrap MS技术整合多种数据采集、挖掘策略, 对复杂贝类基质中AZAs及其代谢产物筛查与鉴别。首先, 比较全扫描二级质谱模式Full MS/dd MS2、目标离子扫描二级质谱模式Target-SIM/dd MS2、平行反应监测模式PRM模式采集方法, 确定标准品与基质样品中的裂解规律和特征碎片; 应用Full MS/dd MS2筛查与PRM目标鉴定, 结合精确分子质量和文献信息, 共鉴别出20种具有AZAs母核结构的化合物; 选择不同质量段碎片离子, 通过PRM的子离子扫描模式, 设置最大分子质量误差为5×10–6, C、H、O和N原子数目的范围分别为20—50、40—80、5—15和1, 实现了未知代谢产物筛查的功能, 通过高能碰撞(HCD)采集方法, 对疑似AZAs代谢产物进行精细区分和结构鉴定。对确证后的AZA代谢物采用半定量方法进行数据分析。
2 结果与分析 2.1 标准品与基质样品中AZAs的质谱裂解规律在LTQ-Orbitrap MS HESI源电离正离子检测模式下(分辨率70000), AZA1、AZA2和AZA3的准分子离子峰为[M+H]+。典型的AZAs的二级碎片离子特征碎片主要包括: [M+H]+、[M+H-nH2O]+、[M+H-H2O- C9H10O2R1R3]+, 特征离子包括m/z 672.4106 (AZA-3 m/z 658.3954)、m/z 462.3214 (AZA-3 m/z 448.3050)和m/z 362.2690等(图 3)。AZAs分子电离产生的最小结构离子碎片为发生RDA (Retro-Diels-Alder)反应的A环断裂。除此以外, 其结构中不同碎片均产生脱H2O, 在高能诱导裂解HCD下, 多采用分子离子的脱水峰(18u)作为特征离子, 也可作为定性的判定依据。在HCD裂解下, MS2图谱中产生了A、D、E、G环等碎片离子。其中m/z 362.2707的特征碎片结构式为C22H36NO3+(Δ-1.03×10–6)是目前已鉴定结构AZA化合物特有碎片离子(除AZA36和AZA39外)。
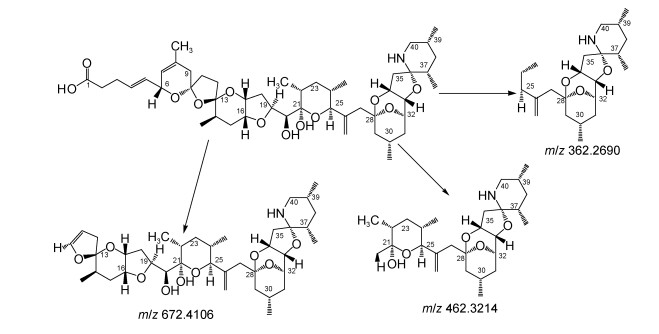 |
| 图 3 PRM模式下AZA2质谱裂解途径 Fig. 3 Fragmentation pathways of AZA2 in PRM mode |
由于贝类的基质效应造成的干扰, 使在实际检测过程中造成假阳性现象。研究应用多种数据采集、挖掘策略, 包括Full MS/dd MS2、PRM和Target- SIM/dd MS2三种鉴定模式, 比较了基质溶液中AZAs的质谱裂解规律。其中Full MS/dd MS2能获得部分主要裂解碎片, 但特异性差。通过建立已知结构的AZAs化合物Inclusion条件, 采用PRM和Target- SIM/dd MS2检测模式, 在四级杆过滤掉大量干扰离子提高了灵敏度的同时, 显著提高二级高分辨质谱选择性, 灵敏度更佳。比较而言, PRM所获得离子碎片信息更为丰富, 且响应提高1个数量级, 贝类基质干扰得到有效排除。因此, 采用目标筛查PRM AZA基质标准物质在不同扫描模式下AZA2的特征碎片(图 2)。
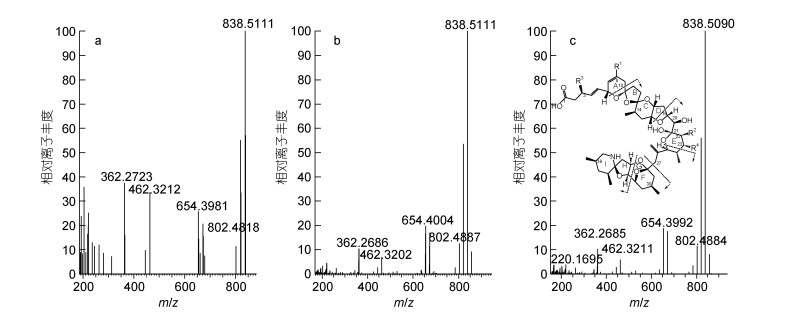 |
| 图 2 不同扫描模式下基质标准物质中AZA2质谱图 Fig. 2 Mass spectrometry of AZA2 in matrix reference materials by different scanning modes 注: a: Full MS/dd MS2模式; b: Target-SIM/dd MS2模式; c: PRM模式 |
通过甲醇提取, 固相萃取柱净化前处理过程, 针对强残留能力的脂溶性AZAs代谢物进行分离提取。运用LTQ-Orbitrap MS仪PRM数据采集方法, 比较代谢物的精确分子质量、保留时间以及AZAs二级碎片离子碎裂规律与特征碎片对代谢样品进行快速筛查和鉴定。共鉴别出了20种AZAs代谢产物, 结果见表 1 (化合物1—20)。AZAs代谢产物类型包括分子结构中的C3和C23的羟基化, C8和C22甲基化及羧基化代谢产物(Rehmann et al, 2008)。结合色谱保留行为和文献报道发现, 经过贝类的代谢作用, 与母体化合物(AZA2)相比, 多种代谢产物的极性增加(Krock et al, 2015), 在反相色谱柱上的保留时间提前, 从而在代谢过程中更易于排出体外。
| 峰 | 保留时间(min) | 理论值(m/z) | 实测值(m/z) | 分子式 | 误差/10–6 | MS/MS裂解碎片 | 鉴定 |
| 1 | 4.71 | 886.4947 | 886.4965 | C48 H72 O14 N | 1.95 | 824.4954, 702.3827, 658.3954, 516.3296, 392.2453 | AZA19 |
| 2 | 6.34 | 716.4732 | 716.4724 | C41 H66 O9 N | 1.02 | 698.4632, 462.3223, 362.2685 | AZA33 |
| 3 | 5.01 | 872.5155 | 872.5163 | C48 H74 O13 N | 0.92 | 818.4786, 806.4824, 658.3945, 546.3477, 378.2639 | AZA11 |
| 4 | 5.64 | 872.5155 | 872.5135 | C48 H73 O14 N | –2.29 | 765.5430, 729.5336, 462.3128, 443.2963, 361.2560 | AZA12 |
| 5 | 3.92 | 842.5049 | 842.4960 | C47 H71 O12 N | –1.06 | 613.3621, 602.2907, 585.3664, 483.2120, 451.2728, 338.1908, 311.1248 | AZA6 |
| 6 | 5.02 | 872.4791 | 872.5138 | C48 H74 O13 N | –1.92 | 824.4947, 818.4838, 810.2746, 806.4875, 658.3924, 598.6357, 478.3164, 378.2626 | AZA17 |
| 7 | 4.21 | 902.4897 | 902.4889 | C48 H72 O15 N | –0.43 | 840.4916, 674.3911, 606.4189, 553.7598, 543.0434, 428.0240, 373.1351, 341.5772, 336.7645 | AZA45 |
| 8 | 5.50 | 858.4998 | 858.5062 | C47 H71 O13 N | 0.62 | 751.5260, 733.5148, 570.3542, 362.2709, 316.3268 | AZA9 |
| 9 | 6.18 | 858.4998 | 858.5115 | C47 H71 O13 N | 1.36 | 838.5074, 674.4147, 656.4048, 464.3278, 364.2744 | AZA10 |
| 10 | 6.00 | 842.5049 | 842.5054 | C47 H72 O12 N | 0.63 | 810.4794, 806.4847, 792.4653, 530.3488, 462.3207, 362.2687 | AZA1 |
| 11 | 6.20 | 856.5206 | 856.5215 | C48 H74 O12 N | 1.09 | 824.496, 820.5001, 806.4761, 672.4109, 640.3850, 636.3896, 462.3221, 362.2695 | AZA2 |
| 12 | 5.69 | 828.4893 | 828.4892 | C46 H70 O12 N | –0.07 | 658.3946, 516.3336, 448.3053, 362.2686, 262.1802 | AZA3 |
| 13 | 5.12 | 844.4842 | 844.4858 | C48 H76 O12 N | 1.89 | 826.4727, 674.3874, 446.2893, 390.2651, 362.2693, 262.1802 | AZA4 |
| 14 | 5.27 | 844.4842 | 844.4861 | C48 H76 O12 N | 2.25 | 808.4643, 790.4537, 674.3892, 446.2902, 362.2692 | – |
| 15 | 5.89 | 844.4842 | 844.4860 | C48 H76 O12 N | 2.13 | 808.4904, 660.4013, 642.3908, 362.2688 | AZA5 |
| 16 | 6.00 | 844.4842 | 844.4863 | C48H76 O12 N | 1.30 | 824.5034, 812.4886, 674.4167, 464.3264, 364.2745 | – |
| 17 | 4.97 | 888.4740 | 888.4734 | C48 H74 O14 N | –0.67 | 812.4788, 808.4914, 674.4158, 464.3238, 364.2760 | AZA21 |
| 18 | 4.70 | 888.4740 | 888.4727 | C48 H74 O14 N | –1.46 | 840.4868, 834.4713, 826.5006, 704.3915, 660.4006 | – |
| 19 | 5.17 | 860.4791 | 860.5025 | C47 H74 O13 N | 2.72 | 842.4968, 824.4865, 806.4733, 690.4220, 672.4015, 462.3112, 372.2230 | AZA13 |
| 20 | 5.38 | 860.4791 | 860.5015 | C47 H74 O13 N | 2.62 | 824.4865, 812.4835, 810.4695, 690.4120, 479.3187, 462.3112 | AZA13 |
| AZAs种类 | 毒素含量(μg/kg) | ||
| 3d | 7d | 22d | |
| AZA2 | 166±16.5 | 210±19.2 | 89.7±5.96 |
| AZA12 | 11.8±2.65 | 36.2±2.20 | 35.2±2.59 |
| AZA19 | 5.15±0.65 | 30.7±1.05 | 42.6±1.95 |
| AZA6 | 2.05±0.25 | 1.68±0.15 | 6.48±0.26 |
| AZA9 | 2.56±0.18 | 9.45±0.68 | 15.6±0.96 |
| AZA10 | 2.32±0.36 | 12.8±1.24 | 19.8±0.16 |
| AZA11 | 5.46±0.22 | 5.61±0.68 | 6.54±0.67 |
| AZA23 | 2.65±0.15 | 7.62±0.19 | 6.55±1.26 |
| 峰14 | - | - | 4.89±0.68 |
| 峰16 | - | - | 9.54±0.97 |
| 峰18 | 0.65±0.11 | - | 1.94±0.12 |
| 注:用AZA2标准品进行相对定量 | |||
在代谢产物样品中, 检测到目标m/z 858.4998通道, 保留时间分别为5.51和6.18min的两种成分, 其高分辨质谱中显示的精确分子离子峰分别为m/z 858.5062 (Δ0.62×10–6)和m/z 858.5115 (Δ1.36×10–6), 与理论分子量比对质量精度均小于3×10–6, 初步判定为同分异构体AZA9和AZA10 (吴海燕等, 2018), 为AZA2在贝类基质的代谢中间过程的上游代谢产物。其中, AZA9(RT 5.51min)在R1位发生羟基化反应(图 4), 而AZA10在R4位发生羟基化反应, 因此其[M+H-H2O-C9H10O2R1R3]+碎片不同, 分别为m/z 658.3991和m/z 674.4143, 差值为一个O(16u)原子。该碎裂模式与AZA9和AZA10结构匹配。
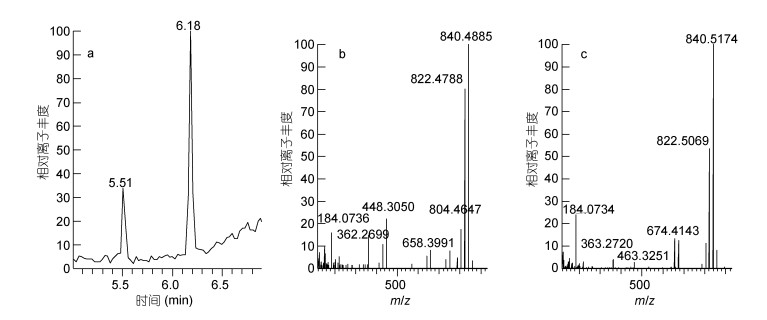 |
| 图 4 m/z 858.4998提取离子流图和二级质谱图 Fig. 4 Extracted ion chromatogram and MS/MS spectra of m/z 858.4998 注: a:提取离子流图; b: RT 5.51化合物二级质谱图; c: RT 6.18化合物二级质谱图 |
已知AZA19为AZA2 R3位发生羧基化产物, 其分子离子丰度为m/z 886.4965 (Δ1.95×10–6), 其A环断裂碎片为658.3951 (Δ0.26×10–6); AZA19的R2位发生去羰基化反应后的AZA17, 其A环断裂碎片为670.3940 (Δ-1.45×10–6)(图 5)。通过上述裂解规律分析860.4791通道, 分别在5.17和5.38min有两个同分异构体化合物, 精确分子离子峰分别为m/z 858.5062 (Δ0.62×10–6)和m/z 858.5115 (Δ1.36×10–6), 其碎片2的m/z分别为C44H54O4N 660.3935 (Δ-1.82×10–6)和C45H46O5 672.4135 (Δ-2.61×10–6)丢失C (12u)碎片, 判定该化合物为AZA13同分异构体, 为R1位发生羧基化反应。
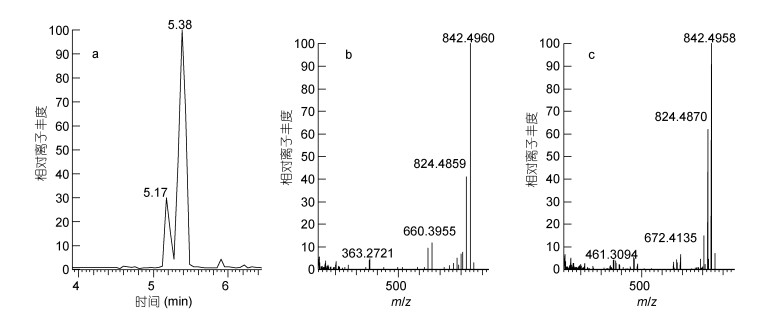 |
| 图 5 m/z 860.4791提取离子流图和二级质谱图 Fig. 5 Extracted ion chromatogram and MS/MS spectra of m/z 860.4791 注: a:提取离子流图; b: RT 5.17化合物二级质谱图; c: RT 5.38化合物二级质谱图 |
m/z 716.4727通道中, 保留时间为6.34min的化学成分, 其结构中有丰富的去H2O (18u)碎片, 且m/z 362.2689碎片与AZAs的C22H36NO3+ (Δ- 0.18×10–6), m/z 462.3224碎片与AZAs的C27H44NO5+ (Δ2.10×10–6)符合AZA的基本的碎裂模式, 将该化合物鉴别为AZA33, 其多级质谱图及裂解规律示于图 6。
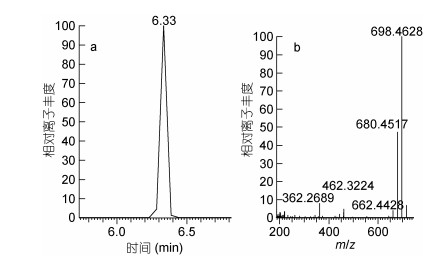 |
| 图 6 m/z 716.4727提取离子流图和二级质谱图 Fig. 6 Extracted ion chromatogram and MS/MS spectra of m/z 716.4727 注: a:提取离子流图; b: RT 6.33化合物二级质谱图 |
应用Full MS/dd MS2和PRM结合的方式, 对AZAs代谢实验解毒器官——内脏团样品(O’Driscoll et al, 2014)进行代谢产物筛查鉴定分析。共鉴别11种AZA代谢产物, 主要成分包括AZA2、AZA6、AZA9、AZA10、AZA11、AZA12、AZA19、AZA23以及3种新型AZAs成分。对3种新型AZAs成分查阅文献比对裂解方式, m/z 844.4842初步鉴定为AZA4和AZA5的同分异构体, 888.4740为AZA21的同分异构体。通过该检测方法获得检测结果与液相色谱-四极杆/线性离子阱复合质谱(吴海燕等, 2016)比较, 灵敏度更高, 定性更为精准。AZA12和AZA19为主要的代谢产物, 在3d代谢样品中含量比例分别为5.9%和2.6%, 主要以AZA2形式存在(含量为84.0%)。在7d代谢样品中, 共检测到9种代谢产物组分, AZA2含量显著降低, 约为66.9%, AZA12和AZA19含量升高, 含量分别为11.5%和9.8%。至代谢22d样品中, 检出全部11种代谢产物。其中部分代谢产物如AZA9, AZA10, AZA19均随代谢过程持续升高, 是AZA2在贝类代谢过程中的末端产物。因此, 应用本方法对促进AZAs的代谢进程具有重要的作用。
综上可见, 脂溶性AZAs在贝类基质体内, 以多种代谢物形式进行代谢转化, 代谢产物极性增加, 当残留能力较强, 可长时间残留在基质样品中。由于目前多种AZAs代谢产物的毒性当量因子未知, 因此整体增加贝类对消费者食用安全的风险性, 需加以重视。
3 结论本研究采用UHPLC-LTQ-Orbitrap-MS高分辨质谱技术建立了多种数据采集模式、挖掘策略用于AZAs及其代谢物质的精准筛查。分析比较标准品和基体标准物质, 共获得20种AZAs系列化合物二级碎片信息, 初步推测了3种新型AZAs代谢产物的结构。应用该方法分析AZA2在贝类中的代谢轮廓, 共检测出11种AZAs, 其中AZA12和AZA19为主要的代谢上游产物。该方法能够实现复杂基质中的AZAs的精准鉴别, 可为AZA的代谢机理研究提供参考。
吴海燕, 李清云, 邴晓菲, 等. 2017. 氮杂螺环酸毒素在栉孔扇贝体内的代谢规律. 中国水产科学, 24(6): 1298-1306 |
吴海燕, 郭萌萌, 郑关超, 等. 2018. 氮杂螺环酸毒素的生态分布、蓄积代谢与检测技术监控研究进展. 中国渔业质量与标准, 8(1): 7-15 |
吴海燕, 郭萌萌, 赵春霞, 等. 2016. 液相色谱-串联质谱法筛查原多甲藻酸毒素及其代谢产物. 色谱, 34(4): 401-406 |
韩深, 王珮玥, 刘萤, 等. 2013. QuEChERS净化技术结合超高效液相色谱-串联质谱法筛查食用贝类中的3种原多甲藻酸贝类毒素. 色谱, 31(10): 939-945 |
韩深, 刘鑫, 李建辉, 等. 2014. 超高压液相色谱-高分辨质谱快速筛查和确证食用贝类中多种原多甲藻酸贝类毒素. 食品科学, 35(4): 116-121 |
EF SA, 2008. Marine biotoxins in shellfish-azaspiracid group-scientific opinion of the panel on contaminants in the food chain. EFSA Journal, 6(10): 723 |
Furey A, O'Doherty S, O'Callaghan K et al, 2010. Azaspiracid poisoning (AZP) toxins in shellfish:Toxicological and health considerations. Toxicon, 56(2): 173-190 DOI:10.1016/j.toxicon.2009.09.009 |
Hess P, Twiner M J, Kilcoyne J et al, 2016. Azaspiracid toxins: toxicological profile. In: Gopalakrishnakone P, Haddad Jr V, Kem W R et al eds. Marine and Freshwater Toxins. Dordrecht: Springer, 169-191
|
Jauffrais T, Kilcoyne J, Herrenknecht C et al, 2013. Dissolved azaspiracids are absorbed and metabolized by blue mussels (Mytilus edulis). Toxicon, 65: 81-89 DOI:10.1016/j.toxicon.2013.01.010 |
Ji Y, Qiu J B, Xie T et al, 2018. Accumulation and transformation of azaspiracids in scallops (Chlamys farreri) and mussels (Mytilus galloprovincialis) fed with Azadinium poporum, and response of antioxidant enzymes. Toxicon, 143: 20-28 DOI:10.1016/j.toxicon.2017.12.040 |
Kilcoyne J, McCarron P, Hess P et al, 2015. Effects of heating on proportions of azaspiracids 1-10 in mussels (Mytilus edulis) and identification of carboxylated precursors for azaspiracids 5, 10, 13, and 15. Journal of Agricultural and Food Chemistry, 63(51): 10980-10987 DOI:10.1021/acs.jafc.5b04609 |
Kilcoyne J, McCarron P, Twiner M J et al, 2014. Epimers of azaspiracids:isolation, structural elucidation, relative LC-MS response, and in vitro toxicity of 37-epi-azaspiracid-1. Chemical Research in Toxicology, 27(4): 587-600 DOI:10.1021/tx400434b |
Kilcoyne J, McCarron P, Twiner M J et al, 2018. Identification of 21, 22-dehydroazaspiracids in mussels (Mytilus edulis) and in vitro toxicity of azaspiracid-26. Journal of Natural Products, 81(4): 885-893 DOI:10.1021/acs.jnatprod.7b00973 |
Krock B, Tillmann U, Potvin É et al, 2015. Structure elucidation and in vitro toxicity of new azaspiracids isolated from the marine dinoflagellate Azadinium poporum. Marine Drugs, 13(11): 6687-6702 DOI:10.3390/md13116687 |
Krock B, Tillmann U, Tebben J et al, 2019. Two novel azaspiracids from Azadinium poporum, and a comprehensive compilation of azaspiracids produced by Amphidomataceae, (Dinophyceae). Harmful Algae, 82: 1-8 DOI:10.1016/j.hal.2018.12.005 |
Krock B, Tillmann U, Witt M et al, 2014. Azaspiracid variability of Azadinium poporum (Dinophyceae) from the China Sea. Harmful Algae, 36: 22-28 DOI:10.1016/j.hal.2014.04.012 |
Leonardo S, Rambla-Alegre M, Samdal I A et al, 2017. Immunorecognition magnetic supports for the development of an electrochemical immunoassay for azaspiracid detection in mussels. Biosensors and Bioelectronics, 92: 200-206 DOI:10.1016/j.bios.2017.02.015 |
Miles C O, Kilcoyne J, McCarron P et al, 2018. Selective extraction and purification of azaspiracids from blue mussels (Mytilus edulis) using boric acid gel. Journal of Agricultural and Food Chemistry, 66(11): 2962-2969 DOI:10.1021/acs.jafc.8b00346 |
O'Driscoll D, ŠkrabákováZ, James K J, 2014. Confirmation of extensive natural distribution of azaspiracids in the tissue compartments of mussels (Mytilus edulis). Toxicon, 92: 123-128 DOI:10.1016/j.toxicon.2014.10.012 |
Pelin M, Kilcoyne J, Nulty C et al, 2018. Toxic equivalency factors (TEFs) after acute oral exposure of azaspiracid 1, -2 and -3 in mice. Toxicology Letters, 282: 136-146 DOI:10.1016/j.toxlet.2017.10.016 |
Rehmann N, Hess P, Quilliam M A, 2008. Discovery of new analogs of the marine biotoxin azaspiracid in blue mussels (Mytilus edulis) by ultra-performance liquid chromatography/tandem mass spectrometry. Rapid Communications in Mass Spectrometry, 22(4): 549-558 DOI:10.1002/rcm.3385 |
Rossi R, Dell'Aversano C, Krock B et al, 2017. Mediterranean Azadinium dexteroporum (Dinophyceae) produces six novel azaspiracids and azaspiracid-35:a structural study by a multi-platform mass spectrometry approach. Analytical and Bioanalytical Chemistry, 409(4): 1121-1134 DOI:10.1007/s00216-016-0037-4 |
Samdal I A, Løvberg K E, Briggs L R et al, 2015. Development of an ELISA for the detection of azaspiracids. Journal of Agricultural and Food Chemistry, 63(35): 7855-7861 DOI:10.1021/acs.jafc.5b02513 |
Tillmann U, Trefault N, Krock B et al, 2017. Identification of Azadinium poporum (Dinophyceae) in the Southeast Pacific:morphology, molecular phylogeny, and azaspiracid profile characterization. Journal of Plankton Research, 39(2): 350-367 |
 2020, Vol. 51
2020, Vol. 51


