中国海洋湖沼学会主办。
文章信息
- 王龙升, 李浩然, 郭东晖. 2020.
- WANG Long-Sheng, LI Hao-Ran, GUO Dong-Hui. 2020.
- 中国东南沿海地区球状伪镖水蚤种群遗传多样性及遗传结构研究
- POPULATION GENETIC DIVERISTY AND GENETIC STRUCTURE OF PSEUDODIAPTOMUS FORBESI FROM THE COASTAL AREAS OF SOUTHEAST CHINA
- 海洋与湖沼, 51(6): 1463-1471
- Oceanologia et Limnologia Sinica, 51(6): 1463-1471.
- http://dx.doi.org/10.11693/hyhz20200300056
文章历史
-
收稿日期:2020-03-05
收修改稿日期:2020-06-09
2. 海洋生物多样性与全球变化研究中心 厦门 361102;
3. 厦门市海湾生态保护与修复重点实验室 厦门 361102;
4. 福建省海陆界面生态环境重点实验室 厦门 361102
2. Marine Biodiversity and Global Change Research Center, Xiamen 361102, China;
3. Xiamen City Key Laboratory of Urban Sea Ecological Conservation and Restoration, Xiamen 361102, China;
4. Fujian Provincial Key Laboratory for Coastal Ecology and Environmental Studies, Xiamen University, Xiamen 361102, China
遗传多样性作为生物多样性的重要组成部分, 是物种多样性和生态系统多样性的基础。物种的进化潜力及其对环境变化的适应能力取决于物种种内的遗传多样性或变异性(施立明, 1990)。开展种群遗传多样性及遗传结构的研究, 对于了解种群数量变动规律和补充机制、揭示种群间的遗传差异与进化关系具有重要的意义。
球状伪镖水蚤(Pseudodiaptomus forbesi)隶属于节肢动物门(Arthropoda)、甲壳动物亚门(Crustacea)、六肢幼虫纲(Hexanauplia)、桡足亚纲(Copepoda)、哲水蚤目(Calanoida)、伪镖水蚤科(Pseudodiaptomidae)、伪镖水蚤属(Pseudodiaptomus)。其主要生活于淡水和咸淡水, 广泛分布于我国东南地区沿海及内陆的湖泊、池塘和江河中(沈嘉瑞等, 1979), 在湖泊、河口等水体的生态系统中扮演着重要的角色。因此, 对其种群遗传多样性的研究具有十分重要的意义。
线粒体细胞色素氧化还原酶Ⅰ(mtCOⅠ)基因是动物线粒体基因组中发现的一种较为保守的蛋白质编码基因(Brown, 1985), 其作为后生动物种群遗传和进化研究中最常用的DNA条形码之一(Shao et al, 2007; Bucklin et al, 2011), 广泛应用于水母类、桡足类等浮游生物及鱼类等游泳生物的研究(Bernatchez et al, 1992; Makino et al, 2010; 程方平等, 2012)。mtCOⅠ基因在伪镖水蚤研究中也有较多的应用, 可作为伪镖水蚤形态学分类的辅助手段, 为新种的发现提供分子生物学方面支持(Sakaguchi et al, 2010; Soh et al, 2012; 王博文等, 2019a, b); 也能在伪镖水蚤类群划分和系统发育的研究中, 提供分子系统学上的证据(Eyun et al, 2007; 王博文, 2019)。目前利用COⅠ基因对伪镖水蚤的研究主要聚焦于种类鉴定及系统发育学, 在种群遗传学上的研究工作较少。本研究基于mtCOⅠ序列, 对我国东南沿海5个地区的球状伪镖水蚤群体进行种群遗传学研究, 从遗传多样性、遗传结构和历史动态方面对其进行群体遗传分析, 以期更好地了解其地理分化格局和群体动态。
1 材料与方法 1.1 样品采集与保存2017年4月—2018年10月, 采用浮游生物网在中国东南沿海地区(无锡、上海、漳州、广州、顺德)5个采样点进行浮游动物样品的采集(表 1), 现场以无水乙醇固定、保存。实验室内对浮游动物样品进行鉴定并挑出球状伪镖水蚤单独存放于无水乙醇中, 4℃低温保存用于后续分析。
| 群体 | 采样地点 | 经度 | 纬度 | 采样日期(年.月.日) |
| 无锡 | 无锡蠡湖 | 120°14′E | 31°31′N | 2018.09.28 |
| 上海 | 上海奉贤区 | 121°32′E | 30°49′N | 2018.09.29 |
| 漳州 | 漳州九龙江 | 117°47′E | 24°29′N | 2017.04.26 |
| 广州 | 广州珠江 | 113°18′E | 23°07′N | 2017.11.23 |
| 顺德 | 顺德顺峰山公园 | 113°17′E | 22°49′N | 2018.10.31 |
使用微量基因组DNA试剂盒(北京全式金生物技术有限公司, EasyPure Micro Genomic DNA Kit)提取单只球状伪镖水蚤的基因组DNA。采用通用引物进行mtCOⅠ基因片段的扩增(Folmer et al, 1994), 引物分别为LCO-1490(5’-GGTCAACAAATCATAAAG ATATTGG-3’)和HCO-2198(5’-TAAACTTCAGGGTG ACCAAAAAATCA-3’)。PCR扩增体系总体积50μL, 其中基因组DNA模板5μL, 正反向引物各0.6μL (50μmol/L), Taq DNA聚合酶(TaKaRa Premix Taq)25μL, 纯水18.8μL。PCR反应的程序为: 94℃预变性4min; 94℃变性1min, 40℃退火1min, 72℃延伸1min, 循环35次; 最后72℃充分延伸10min。PCR产物经电泳检测后, 交由生工生物工程(上海)股份有限公司以Sanger法进行正反向测序。
1.3 数据处理与分析测序所获得的原始序列首先与对应的峰图进行对比以确保数据的准确性, 对比后的mtCOⅠ序列在软件BioEdit v7.2.5(Hall, 1999)及其所附带Clustal W中进行正反向拼接及多重比对, 删除两端不对齐的序列后获得580bp的mtCOⅠ序列。剪切后的COⅠ序列使用Arlequin 3.5(Excoffier et al, 2010)计算碱基组成、转换/颠换、单倍型多样性(h)、核苷酸多样性(π)等遗传多样性参数, 并通过软件的分子变异分析(Analysis of Molecular Variance, AMOVA)来评估群体间的遗传变异、遗传分化系数(Fst)、基因流(Nm)及其显著性。MEGA 5.0计算Kimura 2-Parameter遗传距离。通过Tajima’s D(Tajima, 1989)和Fu’Fs(Fu, 1997)中性检验(Neutrality tests)和核苷酸不配对分布(Mismatch distribution analysis)检测球状伪镖水蚤种群历史动态。以中华异水蚤(Acartiella sinensis GenBank登录号: MT800776)作为外群, 使用PhyML v3.0基于AIC准则下的HKY+I模型通过最大似然法(Maximum Likelihood, ML)构建系统进化树(Guindon et al, 2010), 各分支的置信度经过重复抽样分析(bootstrap test)1000次检验; 基于贝叶斯法以MrBayes 3.27构建进化树(Ronquist et al, 2012), 采用马尔可夫蒙特卡洛分析, 使用4条马尔科夫链(1条热链, 3条冷链), 运行1000000代, 每100代抽样一次。各分支可信度通过后验概率(Posterior probability, PP)检验, 当PP > 95%时该分支可视为有效。通过Haploviewer软件构建COⅠ单倍型网络图(Salzburger et al, 2011)。
2 结果 2.1 mtCOⅠ序列特征及遗传多样性本研究共获得5个采样群体135条580bp的mtCOⅠ序列(GenBank登录号: MT776177—MT 776311), 所有序列中, 碱基A+T的平均含量为63.4%, 明显高于G+C的含量(36.6%), 显示出较强的AT偏好性, 5个群体间的COⅠ基因序列碱基组成无明显差异。共检测到49个核苷酸变异位点, 占分析总位点数的8.4%, 包括8个单核苷酸变异位点, 41个多态简约信息位点, 无插入和缺失位点; 转换数(Ts)共有43次, 明显高于颠换数(Tv)(9次), Ts/Tv=4.78, 表明mtDNA序列未达到突变饱和状态, 适合遗传变异研究。
135条序列共检测到38个单倍型, 其中单倍型H1、H2和H26出现次数较多, 共包含60只个体, 占总样本数的44.4%;未出现5个采样群体共享的单倍型, 且漳州群体与其他4个群体之间均无共享单倍型(表 2)。遗传多样性参数显示, 5个群体的球状伪镖水蚤单倍型多样性指数在0.6285—0.9214之间, 核苷酸多样性指数在0.00766—0.02010之间, 其中上海群体遗传多样性最高(h=0.9046±0.0366; π=0.02010± 0.01048), 其他四个群体遗传多样性差别不大, 同样也处于较高水平, 表明各个球状伪镖水蚤群体均具有较丰富的遗传多样性(表 3)。
| 单倍型 | 群体 | 个体数合计 | ||||
| 无锡 | 上海 | 漳州 | 广州 | 顺德 | ||
| H1 | 5 | 15 | 4 | 24 | ||
| H2 | 1 | 8 | 9 | 18 | ||
| H3 | 1 | 1 | ||||
| H4 | 1 | 4 | 4 | 9 | ||
| H5 | 1 | 1 | 2 | |||
| H6 | 1 | 1 | 2 | |||
| H7 | 1 | 1 | 2 | |||
| H8 | 1 | 1 | 2 | |||
| H9 | 6 | 6 | ||||
| H10 | 1 | 1 | ||||
| H11 | 3 | 3 | ||||
| H12 | 1 | 1 | ||||
| H13 | 2 | 2 | ||||
| H14 | 2 | 2 | ||||
| H15 | 4 | 4 | ||||
| H16 | 5 | 5 | ||||
| H17 | 3 | 3 | ||||
| H18 | 1 | 1 | ||||
| H19 | 1 | 1 | ||||
| H20 | 1 | 1 | ||||
| H21 | 1 | 1 | ||||
| H22 | 1 | 1 | ||||
| H23 | 1 | 1 | ||||
| H24 | 1 | 1 | 2 | |||
| H25 | 1 | 1 | ||||
| H26 | 14 | 4 | 18 | |||
| H27 | 3 | 1 | 4 | |||
| H28 | 1 | 1 | 2 | |||
| H29 | 1 | 1 | ||||
| H30 | 1 | 1 | ||||
| H31 | 6 | 6 | ||||
| H32 | 1 | 1 | ||||
| H33 | 1 | 1 | ||||
| H34 | 1 | 1 | ||||
| H35 | 1 | 1 | ||||
| H36 | 1 | 1 | ||||
| H37 | 1 | 1 | ||||
| H38 | 1 | 1 | ||||
| 单倍型数 | 8 | 14 | 14 | 8 | 8 | 38 |
| 群体 | 个体数 | 单倍型数 | 单倍型多样性(h) | 核苷酸变异位点 | 转换/颠换 | 核苷酸多样性(π) |
| 无锡 | 23 | 8 | 0.6285 ± 0.1115 | 30 | 27/3 | 0.00915 ± 0.00511 |
| 上海 | 26 | 14 | 0.9046 ± 0.0366 | 33 | 31/2 | 0.02010 ± 0.01048 |
| 漳州 | 32 | 14 | 0.9214 ± 0.0240 | 27 | 22/5 | 0.00973 ± 0.00533 |
| 广州 | 32 | 8 | 0.7198 ± 0.0628 | 14 | 13/3 | 0.00766 ± 0.00431 |
| 顺德 | 22 | 8 | 0.7922 ± 0.0673 | 22 | 21/2 | 0.00890 ± 0.00500 |
| 总计 | 135 | 38 | 0.9244 ± 0.0115 | 49 | 43/9 | 0.01819 ± 0.00924 |
对5个采样群体间的遗传分化系数(Fst)和基因流(Nm)进行检测, 结果显示(表 4), 无锡与上海群体之间产生中度遗传分化(Fst=0.20863, P < 0.01; Nm=0.94829);广州与顺德群体之间遗传分化不显著, 基因交流频繁(Fst=0.04972, P > 0.05; Nm=4.77816);其他各群体之间均存在显著遗传分化(Fst: 0.26611—0.69248, P < 0.01; Nm: 0.11102—0.68946)。基于Kimura 2-parameter计算的群体内及群体间遗传距离显示, 上海群体的群体内遗传距离为0.021, 其他群体的群体内遗传距离在0.008—0.010之间; 广州与顺德群体间遗传距离(0.009)为各群体间最小, 与其二者间未产生显著遗传分化和较高的基因流相对应, 无锡与上海群体间遗传距离为0.019, 其他各群体间遗传距离在0.015—0.031之间。
| 群体 | 无锡 | 上海 | 漳州 | 广州 | 顺德 |
| 无锡 | — | 0.94829 | 0.12120 | 0.11102 | 0.12982 |
| 上海 | 0.20863* | — | 0.43768 | 0.55239 | 0.68946 |
| 漳州 | 0.67349* | 0.36354* | — | 0.38009 | 0.39081 |
| 广州 | 0.69248* | 0.31157* | 0.39677* | — | 4.77816 |
| 顺德 | 0.65820* | 0.26611* | 0.39013* | 0.04972 | — |
| 注:上三角为基因流Nm, 下三角为遗传分化系数Fst, *表示差异极显著(P < 0.01) | |||||
根据上述结果, 将广州与顺德群体合并为珠江种群, 无锡与上海群体合并为长江种群, 漳州群体即为九龙江种群。再次进行种群遗传结构分析, Fst和Nm显示(表 5): 3个球状伪镖水蚤种群相互间均存在显著遗传分化(Fst: 0.38675—0.48758, P < 0.01; Nm绝对值: 0.26274—0.39641)。对3个种群进行分子方差分析(AMOVA), 结果显示(表 6): 54.82%的遗传变异出现在种群内, 45.18%种群间总的遗传分化系数Fst为0.45183, 差异显著(P < 0.01), 支持3个球状伪镖水蚤种群间存在一定程度的遗传分化。基于Kimura 2-parameter计算的种群内及种群间遗传距离显示, 种群间遗传距离(0.015—0.028)高于种群内遗传距离(0.009—0.018)。上述遗传结构分析结果显示, 合并后的长江、九龙江和珠江3个球状伪镖水蚤种群之间已经产生了显著遗传分化。
| 种群 | 长江 | 九龙江 | 珠江 |
| 长江 | 0.26274 | 0.29801 | |
| 九龙江 | 0.48758* | 0.39641 | |
| 珠江 | 0.45620* | 0.38675* | |
| 注:上三角为基因流Nm, 下三角为遗传分化系数Fst, *表示差异极显著(P < 0.01) | |||
| 变异来源 | 自由度d.f. | 平方和 | 方差组分 | 变异百分率(%) | 遗传分化系数Fst |
| 种群间 | 2 | 255.205 | 2.82131 | 45.18 | 0.45183 (P < 0.01) |
| 种群内 | 132 | 451.825 | 3.42292 | 54.82 | |
| 合计 | 134 | 707.03 | 6.24423 |
以中华异水蚤(Acartiella sinensis)作为外群, 基于贝叶斯法和最大似然法构建球状伪镖水蚤135只个体的系统进化树, 两种方法构建的进化树基本一致。基于贝叶斯法构建的系统进化树(图 1)显示:球状伪镖水蚤均以较高支持率形成多个进化分支。其中, 漳州群体形成2个独立的分支(ZZ), 广州、顺德群体与少数上海群体个体合并为一支(GZ/SD/SH), 另外广州与顺德群体形成1个独立分支(GD/SD), 无锡与上海群体也单独形成一分支(WX/SH)。单倍型网络图(图 2)与贝叶斯进化树(图 1)结果基本一致, 漳州群体形成2个独立支系, 无锡与上海群体于网络图右侧形成一独立支系, 广州与顺德群体聚集于网络图中部, 并与少量上海群体共享部分单倍型(表 2, 图 2), 各支系间未出现共有的祖先单倍型。系统进化树和单倍型网络图结果均表明, 5个采样群体聚为多个支系, 群体遗传结构与Fst和Nm所得到的结果基本一致。
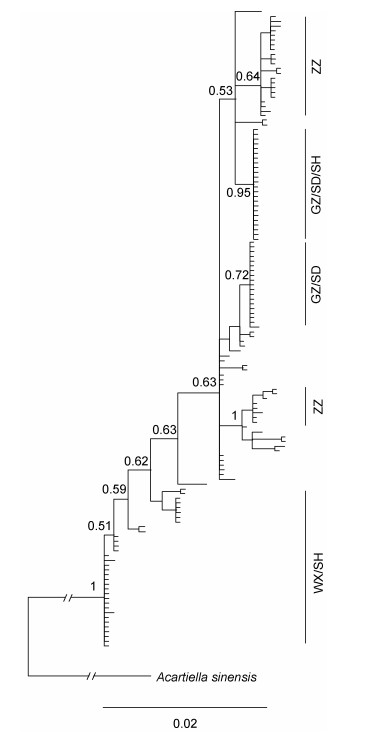 |
| 图 1 基于贝叶斯法构建的球状伪镖水蚤系统进化树 Fig. 1 The phylogenetic tree of P. forbesi based on BI method 注: GZ.广州群体; SD.顺德群体; SH.上海群体; WX.无锡群体; ZZ.漳州群体 |
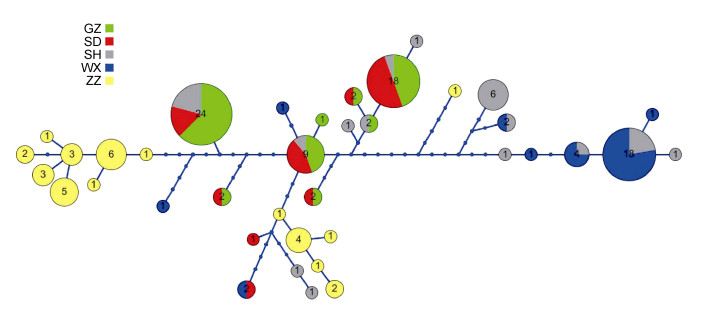 |
| 图 2 球状伪镖水蚤COⅠ单倍型网络图 Fig. 2 Haplotype network of P. forbesi based on COⅠ 注: GZ.广州群体; SD.顺德群体; SH.上海群体; WX.无锡群体; ZZ.漳州群体 |
中性检验结果如表 7所示, 3个种群中性检验结果均不显著(P > 0.1), 表明3个种群在过去并未出现过种群扩张。核苷酸不配对分析结果与中性检验结果吻合, 3个种群的观测曲线均呈现出多峰曲线分布, 与预期曲线相背离(图 3), 表明3个种群均未经历种群扩张, 种群大小维持稳定。
| 种群 | Tajima’D | Fu’s Fs |
| 长江 | 0.61954(P > 0.1) | –0.28719(P > 0.1) |
| 九龙江 | –0.55957(P > 0.1) | –1.62624(P > 0.1) |
| 珠江 | 0.02186(P > 0.1) | 1.91827(P > 0.1) |
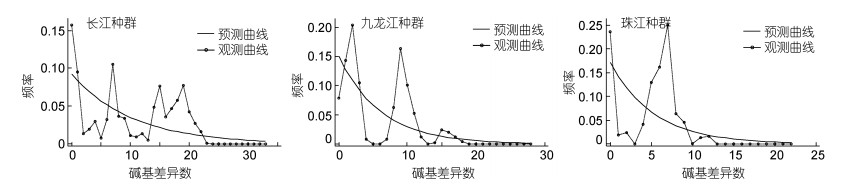 |
| 图 3 球状伪镖水蚤核苷酸不配对分布 Fig. 3 The nucleotide mismatch distribution of P. forbesi |
本研究中, 球状伪镖水蚤mtCOⅠ序列的碱基组成显示出较强的AT偏好性(63.4%), 与其他桡足类的mtCOⅠ基因序列碱基组成特点相同(Huang et al, 2014; 王兴霞等, 2018; 王博文等, 2019a, b)。在部分鱼类、双壳类和介形类中, COⅠ碱基组成同样也显示出较强的AT偏好性(沈玉帮等, 2011; 刘连为等, 2013; Xu et al, 2019)。AT碱基的占比与线粒体DNA进化地位的高低呈正相关(Folmer et al, 1994), 球状伪镖水蚤的A+T碱基占比高达63.4%, 表明其具有较明显的进化优势, 这与本研究球状伪镖水蚤高度遗传分化的结果相一致。五个采样群体的单倍型多样性指数在0.6285—0.9214之间, 核苷酸多样性指数π在0.00766—0.02010之间, 各群体遗传多样性均处于较高水平(表 3)。从生物学和生态学的角度来说, 维持自然种群较高的遗传多样性需要有大的有效种群、环境异质性以及适于群体快速增长的生活环境作为基础; 从历史进化的角度看, 种群的高遗传多样性特征通常是由一个大而稳定的有效种群经过长时间进化所产生(Nei, 1987)。球状伪镖水蚤在中国东南沿海地区的湖泊、池塘及河流中分布很广, 表明其对环境有较强的适应能力, 同时广阔的生境使其面临较小的自然选择压力。这些合适的环境条件及自身因素可能是球状伪镖水蚤各群体维持较高的遗传多样性的主要原因。
3.2 球状伪镖水蚤种群遗传结构及历史动态分析遗传分化系数Fst是用于反映种群间遗传分化大小的重要参数, 其值在0到1之间, 数值越大表明种群间的遗传分化越显著。当Fst > 0.25时, 表明不同种群间存在非常高的遗传分化; 当Fst值处于0.15—0.25之间, 表示种群间出现了中度分化; 当Fst值处于0.05—0.15之间, 表示两种群间的分化程度较低; 当Fst < 0.05时, 表示两种群间没有发生遗传分化(Wright, 1978)。
球状伪镖水蚤5个采样群体遗传分化系数Fst值在0.04972—0.69248之间(表 4), 广州与顺德群体之间遗传分化程度较低, 无锡与上海群体之间产生中度遗传分化, 其他各群体之间均存在显著遗传差异。将球状伪镖水蚤5个地区个体分为长江、九龙江和珠江种群后, 各种群之间的遗传分化指数Fst在0.38675—0.48758之间(表 5), 且差异均极显著; 表 6的AMOVA分析结果显示:种群间总的Fst值为0.45183(P < 0.01), 种群间和种群内变异所占的百分比相差不大, 表明长江、九龙江和珠江3个种群之间遗传分化程度很高。类似的种群遗传学研究在淡水桡足类中镖水蚤(Sinodiaptomus valkanovi)、咸淡水桡足类近缘真宽水蚤(Eurytemora affinis)、海水桡足类剑乳点水蚤(Pleuromamma xiphias)和虎斑猛水蚤(Tigriopus californicus)中均有报道(Edmands, 2001; Winkler et al, 2008; Makino et al, 2010; Goetze, 2011)。其中淡水桡足类和咸淡水桡足类相对于海水生活的桡足类更容易形成种群遗传分化, 其原因可能是淡水和半咸淡水之间比较容易形成地理阻隔, 且咸淡水的环境条件多变, 导致生活在不同地区的同一桡足类比较容易产生显著的遗传分化; 海水的连通性较强, 地理距离较大的地区之间也能有较强的基因交流, 因此较难产生遗传差异。在以往鱼类和双壳类的种群遗传学研究中也有类似的结果, 如鲇(Silurus asotus)、乌鳢(Channa argus)和松江鲈(Trachidermus fasciatus)的种群在不同水系或河流之间各自形成显著遗传差异(高天翔等, 2013; 董新培等, 2014; 周伟等, 2017; 徐丹丹等, 2017); 而位于同一湖泊或河流的洪泽湖河蚬(Corbicula fluminea)、松江鲈和墨脱裂腹鱼(Schizothorax molesworthi)的种群之间均未产生显著遗传分化(高天翔等, 2013; 李大命等, 2015; 俞丹等, 2019)。
本研究中无锡与上海的采样点均地处长江水系, 相距约130km; 广州与顺德采样点距离约为40km, 同属珠江水系; 漳州采样点则位于九龙江水系。九龙江水系与长江水系及珠江水系各采样点间的直线距离分别为800km和500km左右, 长江与珠江水系采样点之间的直线距离约为1200km。同一水系内采样点的地理距离相近, 水体间有一定连通性, 因此群体之间基因交流相对频繁, 遗传分化不显著。广州与顺德之间的地理距离较近, 且水体连通性大于无锡与上海, 这可能是无锡与上海之间种群遗传分化大于前二者的原因。长江、九龙江和珠江3个水系之间相距较远, 连通性差; 且球状伪镖水蚤属于低盐种, 无法在高盐度的海水中生活, 海水的盐度阻隔及水系间的空间距离使得各种群之间产生地理隔离, 导致基因交流的匮乏, 各个种群经过长期的分化形成了显著的遗传结构。
单倍型网络图和系统进化树显示, 长江、九龙江及珠江3个种群均形成了独立的分支(图 1, 图 2), 与3个种群之间形成显著遗传分化的结果(表 5, 表 6)相一致。无锡群体与上海群体不仅形成独立的分支, 还分别与珠江种群共享单倍型, 其中上海群体还与珠江种群共同形成独立分支, 使得类群的划分更为复杂。上海群体与多个其他群体形成共同的分支和单倍型, 可能与其具有最高的遗传多样性和群体内遗传距离有关。在本研究的基础上, 若进一步扩大采样范围, 采用隐种划分的方式来划分类群(Belyaeva et al, 2009; Kordbacheh et al, 2017), 则能更深入地探讨球状伪镖水蚤隐种存在的可能性。
种群历史动态通过Tajima’s D和Fu’Fs的中性检验以及核苷酸不配对分布来检测。中性检验的统计值如果是负值并且显著性差异, 说明种群扩张引起了偏离中性的进化(Tajima, 1989; Fu, 1997);核苷酸不配对分布图谱呈现单峰分布, 说明种群可能经历了扩张, 反之, 如呈现多峰分布, 说明种群十分稳定。本研究中, 中性检验以及核苷酸不配对分布均显示(表 7及图 3), 球状伪镖水蚤种群保持相对稳定, 未经历过种群扩张事件。单倍型网络图也没有显示出与种群扩张相对应的星状演化关系(Winkler et al, 2008; Huang et al, 2014; 刘硕博等, 2019), 而是分化成为多个明显的支系, 也表明种群未经历过种群扩张事件, 符合种群历史动态结果。
4 结论(1) 基于DNA条形码(mtCOⅠ)序列, 中国东南沿海地区5个球状伪镖水蚤采样群体均具有较高的遗传多样性。
(2) 除广州与顺德群体、上海与无锡群体以外, 其他各采样群体间均存在显著遗传分化; 各群体间无共同的祖先单倍型。
(3) 5个采样群体可整合为长江、九龙江和珠江3个种群, 种群间差异明显, 地理隔离可能是造成种群分化的主要因素。中性检验表明, 各种群均尚未经历过扩张事件。
王兴霞, 徐磊, 王亮根, 等. 2018. 基于COⅠ基因序列的长腹剑水蚤系统进化关系. 海洋学报, 40(6): 92-103 |
王博文, 2019.中国东南9种伪镖水蚤的形态学与系统发育学研究.厦门: 厦门大学硕士学位论文, 72-85
|
王博文, 郭东晖. 2019a. 厦门湾伪镖水蚤科一新种——郑氏伪镖水蚤. 厦门大学学报(自然科学版), 58(3): 375-381 |
王博文, 郭东晖. 2019b. 九龙江口伪镖水蚤科一新种——沈氏伪镖水蚤. 海洋学报, 41(6): 93-102 |
刘连为, 许强华, 陈新军, 等. 2013. 基于线粒体DNA分子标记的东太平洋茎柔鱼群体遗传多样性比较分析. 水产学报, 37(11): 1618-1624 |
刘硕博, 唐祖蓉, 申望, 等. 2019. 基于F型mtDNA D-Loop的厚壳贻贝(Mytilus coruscus)群体遗传多样性研究. 海洋与湖沼, 50(2): 355-364 |
李大命, 张彤晴, 唐晟凯, 等. 2015. 基于线粒体COⅠ序列的洪泽湖河蚬(Corbicula fluminea)遗传多样性和种群结构分析. 海洋与湖沼, 46(6): 1339-1346 |
沈玉帮, 张俊彬, 冯冰冰, 等. 2011. 基于线粒体COⅠ序列分析对紫贻贝群体遗传多样性的研究分析. 海洋通报, 30(4): 435-440 |
沈嘉瑞, 戴爱云, 张崇洲. 1979. 中国动物志-节肢动物门甲壳纲淡水桡足类. 北京: 科学出版社, 69
|
周伟, 高天翔, 王俊, 等. 2017. 乌鳢群体遗传多样性和遗传结构分析. 水产学报, 41(10): 1521-1532 |
俞丹, 张智, 张健, 等. 2019. 基于Cyt b基因的雅鲁藏布江下游墨脱江段及察隅河墨脱裂腹鱼的遗传多样性及种群历史动态分析. 水生生物学报, 43(5): 923-929 |
施立明. 1990. 遗传多样性及其保存. 生物科学信息, 2(4): 158-164 |
徐丹丹, 黄燕, 曾庆, 等. 2017. 基于mtDNACytb基因序列的我国不同水系野生鲇种群遗传多样性与种群历史分析. 水产学报, 41(10): 1489-1499 |
高天翔, 毕潇潇, 赵林林, 等. 2013. 基于线粒体Cytb基因全序列的松江鲈群体遗传结构分析. 水生生物学报, 37(2): 199-207 |
董新培, 穆淑梅, 周楠, 等. 2014. 不同地理群体乌鳢线粒体DNA控制区结构分析及遗传多样性. 水产学报, 38(9): 1277-1285 |
程方平, 王敏晓, 王彦涛, 等. 2012. 中国北方习见水母类的DNA条形码分析. 海洋与湖沼, 43(3): 451-459 |
Belyaeva M, Taylor D J, 2009. Cryptic species within the Chydorus sphaericus species complex (Crustacea:Cladocera) revealed by molecular markers and sexual stage morphology. Molecular Phylogenetics and Evolution, 50(3): 534-546 DOI:10.1016/j.ympev.2008.11.007 |
Bernatchez L, Guyomard R, Bonhomme F, 1992. DNA sequence variation of the mitochondrial control region among geographically and morphologically remote European brown trout Saltno trutta populations. Molecular Ecology, 1(3): 161-173 DOI:10.1111/j.1365-294X.1992.tb00172.x |
Brown W M, 1985. The mitochondrial genome of animals. In: MacIntyre R H ed. Molecular Evolutionary Genetics. New York: Plenum Press, 95-130
|
Bucklin A, Steinke D, Blanco-Bercial L, 2011. DNA barcoding of marine Metazoa. Annual Review of Marine Science, 3: 471-508 DOI:10.1146/annurev-marine-120308-080950 |
Edmands S, 2001. Phylogeography of the intertidal copepod Tigriopus californicus reveals substantially reduced population differentiation at northern latitudes. Molecular Ecology, 10(7): 1743-1750 DOI:10.1046/j.0962-1083.2001.01306.x |
Excoffier L, Lischer H E L, 2010. Arlequin suite ver.3.5:a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10(3): 564-567 DOI:10.1111/j.1755-0998.2010.02847.x |
Eyun S I, Lee Y H, Suh H L et al, 2007. Genetic identification and molecular phylogeny of Pseudodiaptomus species (Calanoida, Pseudodiaptomidae) in Korean waters. Zoological Science, 24(3): 265-271 DOI:10.2108/zsj.24.265 |
Folmer O, Black M, Hoeh W et al, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology, 3(5): 294-299 |
Fu Y X, 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics, 147(2): 915-925 |
Goetze E, 2011. Population differentiation in the open sea:Insights from the pelagic copepod Pleuromamma xiphias. Integrative and Comparative Biology, 51(4): 580-597 DOI:10.1093/icb/icr104 |
Guindon S, Dufayard J F, Lefort V et al, 2010. New algorithms and methods to estimate maximum-likelihood phylogenies:assessing the performance of PhyML 3.0. Systematic Biology, 59(3): 307-321 DOI:10.1093/sysbio/syq010 |
Hall T A, 1999. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 41(41): 95-98 |
Huang Y S, Liu G X, Cheng X F, 2014. Molecular phylogeography and population genetic structure of the planktonic copepod Calanus sinicus Brodsky in the coastal waters of China. Acta Oceanologica Sinica, 33(10): 74-84 DOI:10.1007/s13131-014-0542-2 |
Kordbacheh A, Garbalena G, Walsh E J, 2017. Population structure and cryptic species in the cosmopolitan rotifer Euchlanis dilatata. Zoological Journal of the Linnean Society, 181(4): 757-777 DOI:10.1093/zoolinnean/zlx027 |
Makino W, Knox M A, Duggan I C, 2010. Invasion, genetic variation and species identity of the calanoid copepod Sinodiaptomus valkanovi. Freshwater Biology, 55(2): 375-386 DOI:10.1111/j.1365-2427.2009.02287.x |
Nei M, 1987. Molecular Evolutionary Genetics. New York: Columbia University Press, 185
|
Ronquist F, Teslenko M, van der Mark P et al, 2012. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 61(3): 539-542 DOI:10.1093/sysbio/sys029 |
Sakaguchi S O, Ueda H, 2010. A new species of Pseudodiaptomus (Copepoda:Calanoida) from Japan, with notes on the closely related P. inopinus Burckhardt. 1913 from Kyushu Island. Zootaxa, 2623(1): 52-68 |
Salzburger W, Ewing G B, Von Haeseler A, 2011. The performance of phylogenetic algorithms in estimating haplotype genealogies with migration. Molecular Ecology, 20(9): 1952-1963 DOI:10.1111/j.1365-294X.2011.05066.x |
Shao R, Barker S C, 2007. Mitochondrial genomes of parasitic arthropods:Implications for studies of population genetics and evolution. Parasitology, 134(2): 153-167 DOI:10.1017/S0031182006001429 |
Soh H Y, Kwon S W, Lee W et al, 2012. A new Pseudodiaptomus (Copepoda, Calanoida) from Korea supported by molecular data. Zootaxa, 3368(1): 229-244 DOI:10.11646/zootaxa.3368.1.11 |
Tajima F, 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics, 123(3): 585-595 |
Winkler G, Dodson J J, Lee C E, 2008. Heterogeneity within the native range:population genetic analyses of sympatric invasive and noninvasive clades of the freshwater invading copepod Eurytemora affinis. Molecular Ecology, 17(1): 415-430 DOI:10.1111/j.1365-294X.2007.03480.x |
Wright S, 1978. Evolution and the Genetics of Populations. Chicago: University of Chicago Press, 79-506
|
Xu L, Li H, Wang L G et al, 2019. Genetic structure and haplotype pattern of marine planktonic ostracod (Porroecia spinirostris) from South China Sea based on mitochondrial COⅠ Gene. Ocean Science Journal, 54(1): 107-116 DOI:10.1007/s12601-018-0057-4 |
 2020, Vol. 51
2020, Vol. 51


