中国海洋湖沼学会主办。
文章信息
- 丛岩懿, 谢玉素, 张留所. 2020.
- CONG Yan-Yi, XIE Yu-Su, ZHANG Liu-Suo. 2020.
- 海洋线虫Litoditis marina酸性pH胁迫响应的转录组分析
- TRANSCRIPTOME ANALYSIS OF THE RESPONSE OF MARINE NEMATODE LITODITIS MARINA TO ACIDIC STRESS
- 海洋与湖沼, 51(6): 1472-1482
- Oceanologia et Limnologia Sinica, 51(6): 1472-1482.
- http://dx.doi.org/10.11693/hyhz20200300070
文章历史
-
收稿日期:2020-03-12
收修改稿日期:2020-04-18
2. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室 青岛 266237;
3. 中国科学院海洋大科学研究中心 青岛 266071;
4. 中国科学院大学 北京 100049
2. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
3. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
海洋生物作为海洋生态系统的重要组成, 需要适应其生存环境的非生物因素, pH就是常见非生物因素之一(隋永年, 1986)。自工业革命以来, 由于人类大量的开采使用煤、石油和天然气等化石燃料, 并砍伐了大量树木植被, 大气中的二氧化碳含量水平逐年上升。海洋吸收大气中的二氧化碳, 导致二氧化碳分压升高, 海水pH降低, 造成海洋酸化(Havenhand et al, 2019)。化工生产, 矿物燃料的燃烧, 交通运输等产生的酸性气体也会通过大气形成雨、雾、雪等向海洋输送, 使得近海和远海的表层海水的pH值降低(石强等, 2011)。污染物及工业废水的排放, 例如基于形成水合物策略的海水淡化技术中排放的酸性污水等, 也会导致近岸海水pH降低(Montgomery et al, 2009)。
海水酸化的直接后果是对海洋生物的生长发育、代谢、钙化、行为等过程产生影响。当水体从pH 8.3降低到6.9时, 凡纳滨对虾的肠道形态结构遭到破坏, 消化和代谢相关的酶的活性被诱导改变(Duan et al, 2019)。低pH条件下, 极地鳕鱼的游泳能力下降(Kunz et al, 2018)。当海水pH从8.1下降到7.4时, 珍珠贝Pinctada fucata壳的生长受到明显抑制, 壳基质蛋白基因表达显著下调(Liu et al, 2017)。
海洋线虫Litoditis marina, 曾用名(Rhabditis marina, Pellioditis marina), 隶属于线虫动物门(Nematoda), 色矛纲(Chromadorea), 小杆目(Rhabditida), 小杆科(Rhabditidae), Litoditis属; 与生物医学模式生物秀丽线虫属于同一个科。L. marina为雌雄异体, 繁殖上限温度为25℃, 在9—25℃范围内, 该物种的发育速度会随着温度的升高而加快(Moens et al, 2000)。在20℃左右, 盐度为20左右的条件下L. marina产生的子代数量最多(Moens et al, 2000)。目前关于海洋线虫L. marina在不同pH条件下的生长发育及其调控机制尚未报道(曹绪文等, 2018; Xie et al, 2020)。
酸性pH胁迫普遍存在并且会给生物体带来一系列负面影响, 因此研究生物体响应环境酸性pH胁迫的生物学机制意义重大。由于海洋两性繁殖无脊椎动物中还没有真正意义的模式生物, 所以目前关于海洋无脊椎动物应对海水酸性pH胁迫的分子机制的研究还不够深入。作者所在团队经过3年努力, 已经构建了成熟的海洋线虫模式体系(Xie et al, 2020)。本研究以海洋线虫为研究对象, 通过线虫响应不同pH环境的表型实验与转录组分析, 以期为探究无脊椎动物应答酸性pH胁迫的分子机制提供参考。
1 材料与方法 1.1 品系的获得海洋线虫Litoditis marina初始样本采集于山东青岛汇泉湾潮间带, 相较于常规的秀丽线虫NGM培养基(pH为6.33), L. marina在海水NGM培养基(pH为5.92)的生长与繁殖更好。实验室也尝试通过添加Tris-HCl和KOH等试剂调节海水NGM培养基的pH至7.7(汇泉湾海水的pH为7.7左右), 但L. marina的生长情况并不是很好。经过很多的摸索和尝试, 最终选择了海水NGM培养基(pH为5.92)作为L. marina的最佳培养基。本研究中使用的线虫是经过2年实验室驯化培养后的L. marina品系, 20℃条件下培养于接种大肠杆菌E. coli OP50的海水NGM培养板。
1.2 海洋线虫的同步化在已接种OP50的海水培养板上培养海洋线虫, 至平板上有大量虫卵分布时, 收集虫卵至15mL离心管中, 3600g, 离心2min。弃上清后加入灭菌水清洗2次。在室温下用碱性次氯酸盐溶液处理虫卵30s, 1300g, 离心1min。用灭菌水清洗卵沉淀2次, 1300g, 离心1min去上清后再加入海水(Da Silva, 2005)。用玻璃管转移卵液至空的培养基平板上(有OP50, 1滴卵液/板), 20℃孵化过夜, 孵育20h后得到大量同步化的海洋线虫L1幼虫。
1.3 海洋线虫表型实验鉴于目前实验室海洋线虫最佳培养基的pH为5.92, 本研究以pH 5.92为对照组, pH 5.33、pH 4.33和pH 3.33为酸性胁迫组。配制特定pH的海洋线虫培养基小板(1.7g Agar, 0.25g Peptone, 97.5mL海水, 0.1mL 1mol/L MgSO4, 0.1mL 1mol/L CaCl2, 0.1mL 5mg/mL胆固醇乙醇溶液, 通过适量50%盐酸溶液调节pH, 加入2.5mL 1mol/L K2HPO4缓冲溶液使其保持一个稳定的pH), 每个pH小板接种10μL大肠杆菌OP50。待菌液干燥后, 每个pH小板转入70只同步化的L1幼虫, 观察它们的生长发育情况, 记录每个平板上观察到的最早出现海洋线虫成体的时间。每个pH条件进行3个重复。
1.4 RNA-seq建库准备及RNA-seq分析通过1.2的方法得到大量的同步化的L1海洋线虫幼虫后, 用M9溶液将L1幼虫从培养基上洗脱下来, 转入15mL离心管中, 1300g, 离心2min后弃上清。将收集的L1幼虫转移到已接种OP50的直径9cm的pH平板上, 每个pH平板上放约60000只线虫。20℃孵育3h后用M9溶液分别洗脱在不同pH环境下的L1幼虫, 将洗脱液转入15mL离心管中, 3600g离心2min去上清, 再加入M9溶液离心去上清, 如此反复, 洗涤三次, 去除大部分残留细菌。然后将L1线虫样品转移到1.5mL的离心管中, 2000g离心5min, 除去多余的上清。样品立即在液氮中冷冻5min, –80℃保存。每个pH条件实验(pH 4.33, pH 5.33和pH 5.92)进行三次重复。
将样本用液氮迅速研磨后, 通过Trizol法提取RNA。通过琼脂糖凝胶电泳和Agilent 2100 Bioanalyzer分析样品RNA完整性及是否存在DNA污染; 通过Qubit2.0 Fluorometer对RNA浓度进行精确定量。使用Illumina的NEBNext® UltraTM RNA Library Prep Kit试剂盒进行文库构建。把不同文库按照有效浓度及目标下机数据量的需求pooling后进行Illumina测序, 并产生150bp配对末端读数, 测序片段被高通量测序仪测得的图像数据经CASAVA碱基识别转化为序列数据。通过HISAT2 v2.0.5构建参考基因组的索引, 将配对末端clean reads与参考基因组比对(Kim et al, 2015, 2019; Pertea et al, 2016)。参考基因组为实验室测得的海洋线虫基因组。featureCounts v1.5.0-p3被用来计算映射到每个基因的读数(Liao et al, 2014)。根据基因的长度计算每个基因的FPKM, 并计算映射到该基因的读数。基于负二项分布, 通过R程序包DESeq2(1.16.1)进行两个比较组合之间的差异表达分析(Love et al, 2014)。使用Benjamini和Hochberg法来调整所得P值以控制错误发现率。通过DESeq2筛选出校正后的P值小于0.05(P-adjusted < 0.05)的基因被分配为差异表达基因。显著差异表达的筛选标准为校正后的P值< 0.05以及|log2foldchange| > 1。使用R程序包clusterProfiler实现差异表达基因的GO富集分析和KEGG通路分析, 以校正后的P值小于0.05作为为显著性富集的阈值(Yu et al, 2012)。
2 结果与分析 2.1 海洋线虫L. marina在不同酸性pH条件下的生长发育比较为了探究在不同酸性pH条件下海洋线虫L. marina生长发育的速度, 我们进行了表型实验。将同步化的70只海洋线虫L1幼虫转移到不同pH平板上, 每个pH条件三次重复, 记录平板上出现成体的时间和成体数量。结果显示:在pH 5.92条件下, 海洋线虫L. marina在第4天出现成体, 成体率约为36%, 后续可以产卵繁殖(图 1); 在pH 5.33条件下, 第4天出现成体, 成体率约为18%, 也可以产卵繁殖(图 1); 在pH 4.33条件下, 第4天线虫不能发育至成体, 只能长至L4期幼体, 最终在第6天死亡。在pH 3.33条件下, L1幼虫不能存活, 2h内全部死亡。
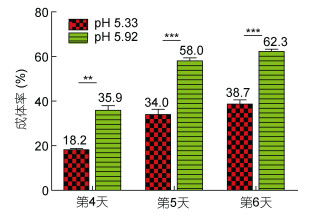 |
| 图 1 海洋线虫在不同pH值的成体率 Fig. 1 The adult rate of L. marina under different pH values 注:误差条代表 3次重复实验的标准误差(*P < 0.05, **P < 0.01, ***P < 0.001) |
我们通过高通量RNA测序(RNA-seq)来识别和量化海洋线虫L. marina在不同pH条件下的差异表达基因。以pH 5.92为对照组, pH 5.33, pH 4.33分别作为处理组, 错误发现率FDR < 5%和|log2FoldChange| > 1作为差异基因筛选阈值。图 2是差异表达基因的聚类热图。各比较组合中上调基因和下调基因的数量如表 1所示。
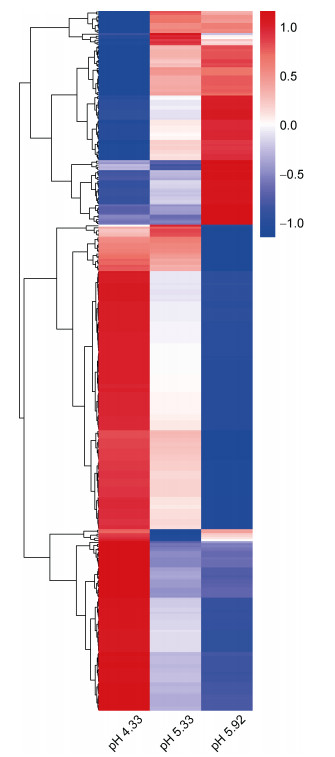 |
| 图 2 海洋线虫在不同pH值下差异表达基因的聚类热图 Fig. 2 Heatmap of differentially expressed genes of L. marina exposed to different pH values 注:横坐标为样品名, 纵坐标为差异基因FPKM归一化后的数值, 红色表示上调, 蓝色表示下调 |
| 各pH组的比较 | 总的差异基因数 | 上调基因数 | 下调基因数 |
| 4.33 vs 5.92 | 321 | 231 | 90 |
| 5.33 vs 5.92 | 61 | 47 | 14 |
| 4.33 vs 5.33 | 84 | 60 | 24 |
通过KEGG分析, 我们发现当pH下降到4.33时, 海洋线虫L. marina脂肪酸β-氧化通路涉及的关键酶(Watts et al, 2017)基因表达显著上调(图 3, 图 4a, 图 4b)。在秀丽线虫中, 对应AMP-binding enzyme注释的绝大多数为酰基辅酶A合成酶家族基因(acs, acyl-CoA Synthetase), 因此在海洋线虫L. marina中的这7个注释为AMP-binding enzyme的基因极有可能就是表达酰基辅酶A合成酶的基因(图 4a)。酰基辅酶A合成酶可以催化脂肪酸与辅酶A形成硫酯键, 形成脂酰-CoA, 这是脂肪酸β-氧化的起始步骤(许雪梅等, 2009; Ellis et al, 2010)。此外, 随着pH的下降, 我们还发现了3个与不饱和脂肪酸合成相关的基因的表达量显著升高(图 3, 图 4c), 其中EVM0000114属于ELO家族, 秀丽线虫的脂肪酸延长酶基因elo参与了不饱和脂肪酸延长的主要步骤, 因此海洋线虫EVM0000114基因很有可能也是编码脂肪酸延长酶的基因。
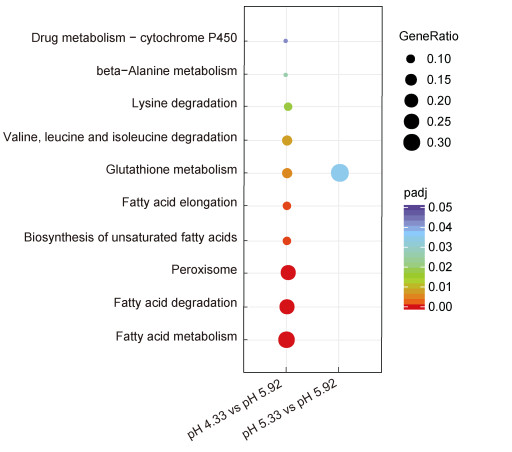 |
| 图 3 各比较组合中上调基因的KEGG富集情况 Fig. 3 KEGG enrichment of up-regulated genes in each combination 注:由于下调基因数量较少, 所以下调基因并未被KEGG富集到任何通路中 |
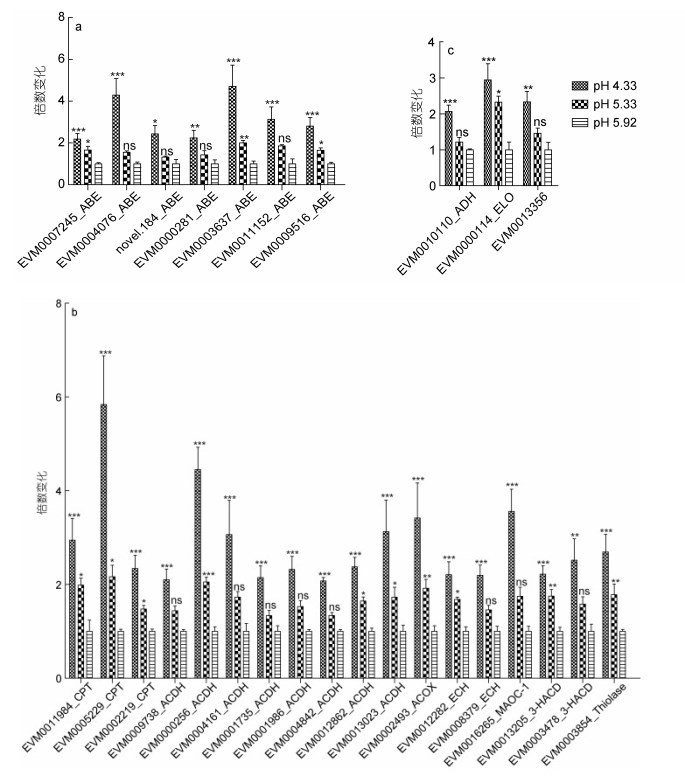 |
| 图 4 海洋线虫脂肪酸代谢相关基因的表达情况 Fig. 4 Expression of the genes related to fatty acid metabolism in L. marina 注: a.酰基辅酶A合成酶基因的转录水平; ABE. AMP-binding enzyme, AMP结合酶; b.脂肪酸β-氧化相关基因的转录水平; CPT. carnitine palmitoyl transferase, 肉碱棕榈酰转移酶; ACDH. acyl-CoA dehydrogenase, 酰基辅酶A脱氢酶; ACOX. acyl-CoA oxidase, 酰基辅酶A氧化酶; ECH. enoyl CoA hydratase, 烯酰辅酶A水合酶; 3-HACD. 3-hydroxyacyl CoA dehydrogenase, 3-羟酰辅酶A脱氢酶; Thiolase.硫解酶; c.海洋线虫不饱和脂肪酸合成相关基因的转录水平。ADH. Zinc-binding dehydrogenase, 锌结合的脱氢酶; ELO. fatty acid elongase, 脂肪酸延长酶。倍数变化表示处理组(pH 4.33, pH 5.33)与对照组(pH 5.92)的基因表达量的比值(处理组FPKM值/对照组FPKM值)。误差条代表 3次重复实验的标准误差。*P < 0.05, **P < 0.01, ***P < 0.001 |
当pH下降到4.33时, 海洋线虫L. marina表皮合成相关的2个虾红素(Astacin)基因nas和6个补丁家族基因(Patched family)ptr表达显著上调(图 5a, 5b)。但是, 表皮胶原基因col类基因的表达没有发生显著变化(图 5c)。
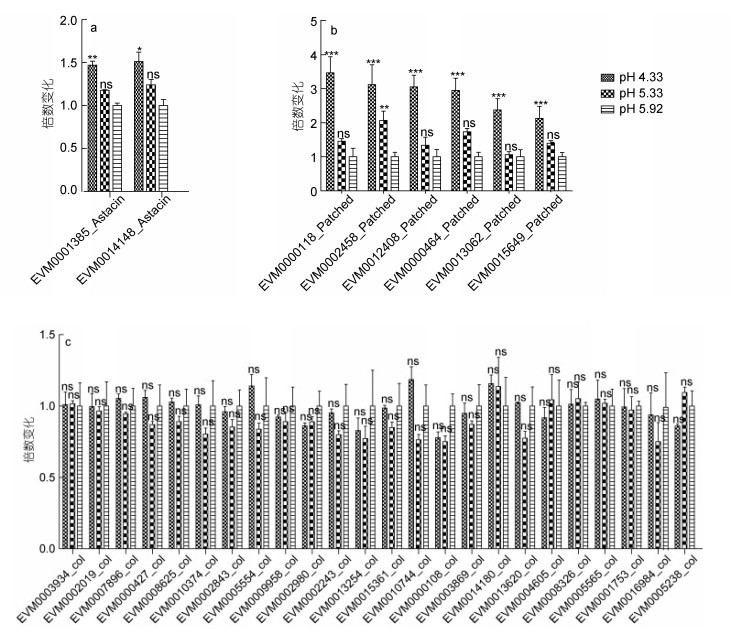 |
| 图 5 海洋线虫表皮相关基因的表达情况 Fig. 5 Expression of cuticle related genes in L. marina 注: a.线虫虾红素样金属蛋白酶基因nas转录水平; b.补丁蛋白相关基因ptr转录水平; c.胶原基因col的转录水平。倍数变化表示处理组(pH 4.33, pH 5.33)与对照组(pH 5.92)的基因表达量的比值(处理组FPKM值/对照组FPKM值)。误差条代表 3次重复实验的标准误差。*P < 0.05, **P < 0.01, ***P < 0.001 |
在pH 4.33的条件下, KEGG pathway分析结果显示, 海洋线虫L. marina中细胞色素P450通路得到显著富集; 细胞色素P450氧化酶、黄素结合的单加氧酶、谷胱甘肽-S-转移酶、ABC转运蛋白等基因的表达量都显著升高(图 3, 图 6)。而在pH 5.33条件下, 细胞色素P450通路相关基因中仅EVM0009829、EVM0007061、EVM0012458、EVM0004308、EVM0011078表达上调。此外, 海洋线虫L. marina核激素受体基因nhr(nuclear hormone receptor)也在pH下降到4.33时表达量显著升高(图 6)。
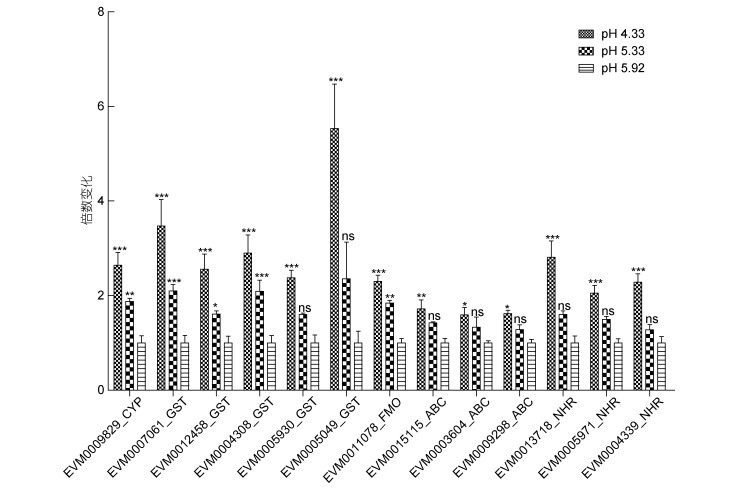 |
| 图 6 海洋线虫细胞色素P450通路相关基因及核激素受体基因的表达情况 Fig. 6 Expression of drug-metabolism cytochrome P450 pathway genes and nuclear hormone receptors genes in L. marina 注: CYP. cytochrome P450 oxidase, 细胞色素P450氧化酶; FMO. Flavin-binding monooxygenase, 黄素结合的单加氧酶; GST. glutathione-S-transferase, 谷胱甘肽-S-转移酶; ABC. ATP-binding cassette transporters, ABC转运蛋白; NHR. nuclear hormone receptors, 核激素受体。倍数变化表示处理组(pH 4.33, pH 5.33)与对照组(pH 5.92)的基因表达量的比值(处理组FPKM值/对照组FPKM值)。误差条代表 3次重复实验的标准误差。*P < 0.05, **P < 0.01, ***P < 0.001 |
在pH 4.33条件下, 海洋线虫L. marina中3个注释为HSP70和1个注释为HSP90的基因, 以及5个凝集素C基因的表达量显著升高(图 7a, 7b)。但是, 在pH 5.33条件下, 这些基因的表达量并没有发生显著变化。
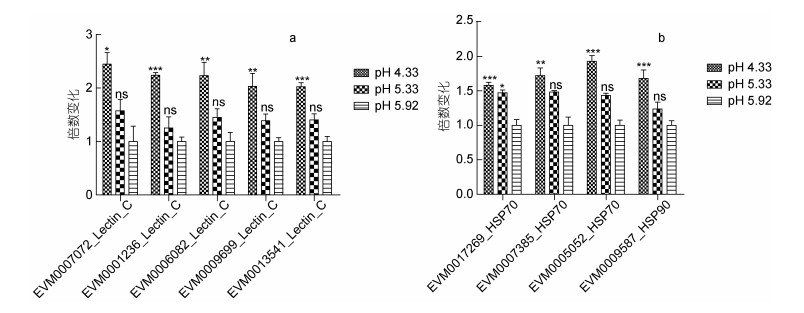 |
| 图 7 海洋线虫凝集素C基因(a)和热休克蛋白基因(b)的表达情况 Fig. 7 Expression of lectin C genes (a) and hsp genes (b) in L. marina 注:倍数变化表示处理组(pH 4.33, pH 5.33)与对照组(pH 5.92)的基因表达量的比值(处理组FPKM值/对照组FPKM值)。误差条代表 3次重复实验的标准误差。*P < 0.05, **P < 0.01, ***P < 0.001 |
从分析结果中发现当pH下降到4.33时, 25个脂肪酸β-氧化通路基因表达显著上调(图 4a, 图 4b), 这些基因数目占鉴定出的所有上调基因的11%。脂肪酸β-氧化过程是一个高度的放能过程, 每一轮β-氧化会产生一个NADH, 1个FADH2和1个乙酰-CoA(Houten et al, 2016), 每个乙酰-CoA经过氧化可产生10个ATP(Ashrafi, 2007; Braeckman et al, 2009; Akram, 2014)。氧化的脂质底物可能是在海洋线虫L. marina生命早期阶段满足发育所需能量的主要来源, 脂肪酸β-氧化通路基因表达上调, 反映了海洋线虫L. marina动员能量储备来应对环境pH的下降。
pH下降导致的脂肪酸分解代谢增加在不少文献中都有所报道。Hall等(2008)的研究发现, 当pH从7下降到4, 秀丽线虫3个脂肪酸β-氧化通路基因表达量都显著升高, 包括酰基辅酶A合成酶(acs-2, F28F8.2), 酰基辅酶A氧化酶(acox-1.5, F08A8.4)和3-羟酰辅酶A脱氢酶(hacd-1, R09B5.6)基因。该研究认为pH下降影响了钠氢交换体NHX-2耦合H+浓度的营养物质的摄入, 导致线虫只能通过脂肪酸的分解来提供一种替代的能量来源(Hall et al, 2008)。与海洋线虫L. marina相比, 秀丽线虫脂肪酸β-氧化通路上调的基因数量少很多(Cong et al, 2020), 可见脂肪酸β-氧化虽然参与了秀丽线虫对酸性胁迫的响应, 但可能并不发挥主要作用。紫海胆在海水pH下降时, 脂肪酸氧化途径中的多个基因表达上调(Evans et al, 2017), 该研究认为酸性胁迫作用于代谢基因网络, 使ATP转向维持细胞内环境的酸碱平衡, 以此来增强生物体对海水酸化的耐受性(Evans et al, 2017)。
3.2 细胞色素P450通路基因参与海洋线虫对酸性pH胁迫的响应细胞色素P450通路的物质代谢过程主要分为2个阶段。第一阶段:亲脂性物质被细胞色素P450单氧化酶(CYP)等修饰成亲电或亲核物质。第二阶段:经过第一阶段反应产生的亲水性化合物在谷胱甘肽转移酶等的催化作用下与极性配体结合。最后由ABC转运蛋白把这些性状改变的化合物运输到细胞外(Barnes, 1960; Dieterich et al, 2008; Croom, 2012)。当pH从8.0降到7.3时, 长牡蛎Cytochrome P450 1A5和Glutathione S-transferase Ω-1表达量显著升高(Timmins-Schiffman et al, 2014)。在本研究中, 当pH下降到4.33时, 10个物质代谢细胞色素P450通路基因表达显著上调(图 3, 图 6)。我们在秀丽线虫中也发现, 当pH从6.33(秀丽线虫正常培养基的pH)下降到3.13时, 32个细胞色素P450通路基因表达上调(Cong et al, 2020)。秀丽线虫在pH 3.13的条件下依然可以产卵繁殖, 而海洋线虫L. marina在pH 4.33的条件下不能长成成体, 海洋线虫表现得更不耐酸; 我们推测造成这种差异的原因是海洋线虫L. marina基因组中的细胞色素P450通路基因与陆生自由生活线虫相比, 发生了显著缩减(Xie et al, 2020)。物质代谢细胞色素P450通路基因的启动表达很可能是为了保护生物体免受更酸的环境产生的有毒物质的侵害, 或清除因更酸的环境导致生理状态紊乱而积累在其体内的有毒物质。
核激素受体NHRs是参与多种生理过程的重要转录调控因子, 研究表明NHRs可以调节P450通路中Ⅰ期和Ⅱ期解毒基因cyp和gst、ugt等的表达(Fisher et al, 2006), 它在调节解毒酶功能方面具有广泛的底物特异性, 在对内源及外源有毒物质的代谢过程中发挥着关键作用(Hoffmann et al, 2015), 有毒物质很可能通过配体间接或直接与核激素受体结合, 然后激活外源物质解毒相关基因的表达(Lindblom et al, 2006; Hoffmann et al, 2015)。
鉴于细胞色素P450通路基因可能在线虫应对极端酸性环境胁迫时可能发挥的重要作用, 并且该通路基因数量众多, 功能复杂, 本实验室在今后的研究中会着重开展此通路基因的筛选与鉴定, 通过大规模突变体筛选和CRISPR基因编辑技术探究决定线虫响应和适应酸性环境胁迫的关键基因, 以期为生物能够在不断变化的pH环境中生存的分子机制的理解提供参考, 也可能会为提高评估和监测生态修复的后果提供新的解决方案。
3.3 表皮相关基因、凝集素C基因及热休克蛋白基因共同参与海洋线虫对酸性pH胁迫的响应在秀丽线虫中, nas(Nematode Astacin)编码一种类似虾红素的金属蛋白酶(WormBase, 2019), 它与表皮合成密切相关(Novelli et al, 2004)。注释为Patched family的基因在秀丽线虫中绝大多数都是ptr(patched-related protein)基因, 这些基因参与了线虫的表皮更替过程; ptr基因的破坏会导致秀丽线虫的蜕皮障碍(Zugasti et al, 2005)。在本研究中, nas和ptr基因的表达上调说明表皮相关基因参与了海洋线虫L. marina对酸性pH胁迫的响应。我们前期的研究结果显示, 当pH下降到4.33时, 秀丽线虫中有大量表皮胶原基因col表达上调(Cong et al, 2020); 但海洋线虫L. marina的col基因表达却没有发生显著变化(图 5c), 推测可能是由于两种线虫的表皮结构成分不同, 所以对酸性pH胁迫的响应不同。
C型凝集素是一种Ca2+依赖的糖结合蛋白(Drickamer, 1989)。当海水pH从8.1下降到7.7时, 翼足类动物Clio pyramidata的C型凝集素家族基因表达量也显著上调(Maas et al, 2015), 特别的是, 这些C型凝集素家族基因的功能可能与生物的矿化作用相关。此外, C型凝集素在维持机体稳态、免疫防御以及免疫监视等生理过程中也发挥着重要作用。当海水pH从8.1下降到7.7时, 紫贻贝Mytilus chilensis的C型凝集素基因的表达量也显著升高, 该研究认为海水酸化可能在紫贻贝感染致病菌Vibrio anguillarum的早期阶段触发特定的免疫相关基因, 促进抗菌肽和模式识别受体的转录(Castillo et al, 2017)。我们鉴定到的这5个上调的C型凝集素基因在海洋线虫对酸性pH胁迫的响应中究竟是发挥何种功能, 有待于进一步研究。
热休克蛋白作为一种常见的细胞应激的指示蛋白, 在许多刺激下如热休克、缺氧、重金属、寒冷、渗透、盐、紫外线、氧化应激和病原体感染等都会被诱导表达(Morimoto, 1998; Swindell et al, 2007)。Urbarova等(2019)的研究发现, 海葵Anemonia viridis在低pH环境条件下, 应激相关转录本的数量急剧增加, 其中非常显著的就是热休克蛋白Hsp70和Hsp90的上调。本研究显示, 热休克蛋白家族基因也参与了海洋线虫L. marina对酸性pH胁迫的响应, 它们发挥的具体功能有待于进一步研究。
4 结论我们的研究结果表明, 当pH从5.92下降到4.33时, 海洋线虫L. marina通过大量脂肪酸β-氧化通路基因上调来响应酸化胁迫, 细胞色素P450通路基因、凝集素C基因、表皮相关基因(ptr和nas)和HSP70家族基因等共同参与响应过程。这些基因的表达变化与表型实验的结果相吻合, 当pH从5.92下降到5.33时, 海洋线虫L. marina生长发育的速度明显减慢, 但依然可以产卵繁殖, 上述这些基因的表达并没有发生显著变化(只有其中几个基因表达上调); 而当pH下降到4.33时, 海洋线虫L. marina则不能长成成体, 上述这些基因表达显著上调。我们在海洋线虫L. marina中发现的应答酸化胁迫的调控模式, 为理解生物体能够在低pH环境中生存的分子机制提供了参考, 同时为筛选和识别生物体响应和适应酸化胁迫的关键基因奠定了基础。
石强, 杨东方. 2011. 渤海夏季海水pH值年际时空变化. 中国环境科学, 31(S1): 58-68 |
许雪梅, 王大勇. 2009. 秀丽线虫脂肪积累调控的生理与分子机制. 生理科学进展, 40(2): 129-134 |
隋永年. 1986. 海水pH及其影响因素. 山东海洋学院学报, S1: 146-159, 207-208 |
曹绪文, 张留所. 2018. CRISPR/Cas9基因编辑技术在线虫中的应用进展和展望. 中国科学:生命科学, 48(5): 513-520 |
Akram M, 2014. Citric acid cycle and role of its intermediates in metabolism. Cell Biochemistry and Biophysics, 68(3): 475-478 DOI:10.1007/s12013-013-9750-1 |
Ashrafi K, 2007. Obesity and the regulation of fat metabolism. WormBook:the online review of C. elegans biology, 9: 1-20 DOI:10.1895/wormbook.1.130.1 |
Barnes J M, 1960. Detoxication mechanisms. The metabolism and detoxication of drugs, toxic substances and other organic compounds. British Journal of Industrial Medicine, 17(3): 244 |
Braeckman B P, Houthoofd K, Vanfleteren J R, 2009. Intermediary metabolism. In: WormBook: the Online Review of C. Elegans Biology, 1-24, doi: 10.1895/wormbook.1.146.1
|
Castillo N, Saavedra L M, Vargas C A et al, 2017. Ocean acidification and pathogen exposure modulate the immune response of the edible mussel Mytilus chilensis. Fish & Shellfish Immunology, 70: 149-155 |
Cong Y Y, Yang H W, Zhang P C et al, 2020. Transcriptome analysis of the nematode Caenorhabditis elegans in acidic stress environments. bioRxiv DOI:10.1101/2020.03.06.980102 |
Croom E, 2012. Chapter three-metabolism of xenobiotics of human environments. Progress in Molecular Biology and Translational Science, 112: 31-88 DOI:10.1016/B978-0-12-415813-9.00003-9 |
Da Silva A P, 2005. Pristionchus pacificus genetic protocols. In: WormBook: the Online Review of C. Elegans Biology, 1-8, doi: 10.1895/wormbook.1.114.1
|
Dieterich C, Clifton S W, Schuster L N et al, 2008. The Pristionchus pacificus genome provides a unique perspective on nematode lifestyle and parasitism. Nature Genetics, 40(10): 1193-1198 DOI:10.1038/ng.227 |
Drickamer K, 1989. Demonstration of carbohydrate-recognition activity in diverse proteins which share a common primary structure motif. Biochemical Society Transactions, 17(1): 13-15 |
Duan Y F, Wang Y, Liu Q S et al, 2019. Changes in the intestine barrier function of Litopenaeus vannamei in response to pH stress. Fish & Shellfish Immunology, 88: 142-149 |
Ellis J M, Frahm J L, Li L O et al, 2010. Acyl-coenzyme a synthetases in metabolic control. Current Opinion in Lipidology, 21(3): 212-217 DOI:10.1097/MOL.0b013e32833884bb |
Evans T G, Pespeni M H, Hofmann G E et al, 2017. Transcriptomic responses to seawater acidification among sea urchin populations inhabiting a natural pH mosaic. Molecular Ecology, 26(8): 2257-2275 DOI:10.1111/mec.14038 |
Fisher A L, Lithgow G J, 2006. The nuclear hormone receptor DAF-12 has opposing effects on caenorhabditis elegans lifespan and regulates genes repressed in multiple long-lived worms. Aging Cell, 5(2): 127-138 DOI:10.1111/j.1474-9726.2006.00203.x |
Hall R A, Vullo D, Innocenti A et al, 2008. External pH influences the transcriptional profile of the carbonic anhydrase, CAH-4B in Caenorhabditis elegans. Molecular and Biochemical Parasitology, 161(2): 140-149 |
Havenhand J N, Filipsson H L, Niiranen S et al, 2019. Ecological and functional consequences of coastal ocean acidification:perspectives from the baltic-skagerrak system. Ambio, 48(8): 831-854 DOI:10.1007/s13280-018-1110-3 |
Hoffmann J M, Partridge L, 2015. Nuclear hormone receptors:roles of xenobiotic detoxification and sterol homeostasis in healthy aging. Critical Reviews in Biochemistry and Molecular Biology, 50(5): 380-392 DOI:10.3109/10409238.2015.1067186 |
Houten S M, Violante S, Ventura F V et al, 2016. The biochemistry and physiology of mitochondrial fatty acid β-oxidation and its genetic disorders. Annual Review of Physiology, 78(1): 23-44 DOI:10.1146/annurev-physiol-021115-105045 |
Kim D, Langmead B, Salzberg S L, 2015. HISAT:a fast spliced aligner with low memory requirements. Nature Methods, 12(4): 357-360 DOI:10.1038/nmeth.3317 |
Kim D, Paggi J M, Park C et al, 2019. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype. Nature Biotechnology, 37(8): 907-915 DOI:10.1038/s41587-019-0201-4 |
Kunz K L, Claireaux G, Pörtner H O et al, 2018. Aerobic capacities and swimming performance of polar cod (Boreogadus saida) under ocean acidification and warming conditions. The Journal of Experimental Biology, 221(21): jeb184473 DOI:10.1242/jeb.184473 |
Liao Y, Smyth G K, Shi W, 2014. Featurecounts:an efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics, 30(7): 923-930 |
Lindblom T H, Dodd A K, 2006. Xenobiotic detoxification in the nematode Caenorhabditis elegans. Journal of Experimental Zoology Part A:Comparative Experimental Biology, 305A(9): 720-730 DOI:10.1002/jez.a.324 |
Liu W G, Yu Z H, Huang X D et al, 2017. Effect of ocean acidification on growth, calcification, and gene expression in the pearl oyster, Pinctada fucata. Marine Environmental Research, 130: 174-180 DOI:10.1016/j.marenvres.2017.07.013 |
Love M I, Huber W, Anders S, 2014. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology, 15(12): 550 DOI:10.1186/s13059-014-0550-8 |
Maas A E, Lawson G L, Tarrant A M, 2015. Transcriptome-wide analysis of the response of the thecosome pteropod Clio pyramidata to short-term CO2 exposure. Comparative Biochemistry and Physiology Part D:Genomics and Proteomics, 16: 1-9 DOI:10.1016/j.cbd.2015.06.002 |
Moens T, Vincx M, 2000. Temperature and salinity constraints on the life cycle of two brackish-water nematode species. Journal of Experimental Marine Biology and Ecology, 243(1): 115-135 DOI:10.1016/S0022-0981(99)00113-6 |
Montgomery M T, Boyd T J, Osburn C L et al, 2009. Desalination technology waste streams:effect of pH and salinity on metabolism of marine microbial assemblages. Desalination, 249(2): 861-864 DOI:10.1016/j.desal.2009.03.018 |
Morimoto R I, 1998. Regulation of the heat shock transcriptional response:cross talk between a family of heat shock factors, molecular chaperones, and negative regulators. Genes & Development, 12(24): 3788-3796 |
Novelli J, Ahmed S, Hodgkin J, 2004. Gene interactions in Caenorhabditis elegans define DPY-31 as a candidate procollagen C-proteinase and SQT-3/ROL-4 as its predicted major target. Genetics, 168(3): 1259-1273 |
Pertea M, Kim D, Pertea G M et al, 2016. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and ballgown. Nature Protocols, 11(9): 1650-1667 DOI:10.1038/nprot.2016.095 |
Swindell W R, Huebner M, Weber A P, 2007. Transcriptional profiling of arabidopsis heat shock proteins and transcription factors reveals extensive overlap between heat and non-heat stress response pathways. BMC Genomics, 8(1): 125 DOI:10.1186/1471-2164-8-125 |
Timmins-Schiffman E, Coffey W D, Hua W et al, 2014. Shotgun proteomics reveals physiological response to ocean acidification in Crassostrea gigas. BMC Genomics, 15(1): 951 DOI:10.1186/1471-2164-15-951 |
Urbarova I, Forêt S, Dahl M et al, 2019. Correction:ocean acidification at a coastal CO2 vent induces expression of stress-related transcripts and transposable elements in the sea anemone Anemonia viridis. PLoS One, 14(6): e0218009 DOI:10.1371/journal.pone.0218009 |
Watts J L, Ristow M, 2017. Lipid and carbohydrate metabolism in Caenorhabditis elegans. Genetics, 207(2): 413-446 |
WormBase, 2019. Explore Worm Biology. https: //wormbase.org
|
Xie Y S, Zhang P C, Xue B N et al, 2020. Establishment of a marine nematode model for animal functional genomics, environmental adaptation and developmental evolution. bioRxiv DOI:10.1101/2020.03.06.980219 |
Yu G C, Wang L G, Han Y Y et al, 2012. Clusterprofiler:an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology, 284-287
|
Zugasti O, Rajan J, Kuwabara P E, 2005. The function and expansion of the patched-and hedgehog-related homologs in C. elegans. Genome Research, 15(10): 1402-1410 DOI:10.1101/gr.3935405 |
 2020, Vol. 51
2020, Vol. 51


