中国海洋湖沼学会主办。
文章信息
- 隋明益, 杨祖晶, 于海涛, 彭程, 邢强, 黄晓婷, 胡景杰, 包振民. 2021.
- SUI Ming-Yi, YANG Zu-Jing, YU Hai-Tao, PENG Cheng, XING Qiang, HUANG Xiao-Ting, HU Jing-Jie, BAO Zhen-Min. 2021.
- 栉孔扇贝(Chlamys farreri)过氧化物酶体增殖物激活受体基因的鉴定与表达分析
- IDENTIFICATION AND EXPRESSION PROFILING OF THE PPAR GENE IN CHLAMYS FARRERI
- 海洋与湖沼, 52(1): 153-161
- Oceanologia et Limnologia Sinica, 52(1): 153-161.
- http://dx.doi.org/10.11693/hyhz20200300093
文章历史
-
收稿日期:2020-03-25
收修改稿日期:2020-05-07
2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266237;
3. 中国海洋大学三亚海洋研究院热带海洋生物种质资源开发与种业工程实验室 三亚 572000
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
3. Laboratory of Tropical Marine Germplasm Resources and Breeding Engineering, SANYA Oceanographic Institution of the Ocean University of CHINA(SOI-OUC), Sanya 572000, China
过氧化物酶体(peroxisome, P)是单膜包裹的细胞器, 在真核生物细胞中普遍存在, 参与众多代谢途径如长链脂肪酸的β氧化, 过氧化氢的解毒还有醚磷脂和胆汁酸的合成等(Honsho et al, 2016)。过氧化物酶体能够在结构各异的化学物质作用下发生增殖, 形成过氧化物酶体增殖物(peroxisome proliferator, PP)。过氧化物酶体增殖物激活受体(peroxisome proliferator-activated receptors, PPARs)是一类配体激活的核转录因子家族, 属于核激素受体超家族, 能够被内源性脂肪酸或者外源性过氧化物酶体增殖物作为配体激活(Glass et al, 2006; 陈亮, 2011)。1990年, PPARs首先在啮齿动物中发现(Tyagi et al, 2011)。脊椎动物的PPARs包含3种亚型: PPARα, PPARβ/δ和PPARγ, 它们均包含五个功能域, 其中N端的A、B结构域为激活功能域1(activation function 1, AF1), 不同亚型的A、B区域在结构上高度可变, 并不保守, 它的激活并不依赖于配体的存在, 主要是通过磷酸化的作用来调节配体和受体之间的亲和力用以调节PPARs的活性; C区域为高度保守的DNA结合域(DNA-binding domain, DBD), 由两个锌指结构折叠成球状, 保守性较高, 通过与类视黄醇X受体(retinoid X receptor, RXR)组成异源二聚体, 然后与靶基因启动子中的过氧化物酶体增殖物反应元件(PPAR response elements, PPREs)结合, 进而调节靶基因的转录; D区域为铰链区, 连接DNA结合区域和配体结合区域; E/F区域位于C末端, 为配体结合区(ligand binding domain, LBD), 通过识别结合配体, 在信号转导过程中发挥重要作用, 该区域氨基酸序列的差异使各PPAR亚型分别对不同配体产生亲和力(Desvergne et al, 1999; Glass et al, 2006; 宁丽军, 2016; Bervejillo et al, 2019)。PPARs的靶基因在脂肪代谢与能量平衡、细胞分化、炎症反应中具有重要作用(Glass et al, 2006; Menendez-Gutierrez et al, 2012; Wang et al, 2019)。脊椎动物的PPARα和PPARβ能够激活脂肪分解代谢相关的酶, 参与线粒体的β-氧化反应, 而PPARγ控制脂肪的积累并且能够调节脂肪形成(Dreyer et al, 1992; 宁丽军等, 2019; 赵亭亭等, 2020)。临床研究表明, PPARs在许多生理与病理过程中发挥关键作用, 特别是与葡萄糖和脂质代谢、炎症反应相关的疾病(Glass et al, 2006)。另外, PPARs活化后能够改善许多中枢神经退行性疾病如阿尔兹海默症、帕金森病、多发性硬化病、肌萎缩侧索硬化症以及中枢神经系统急慢性损伤的病理过程(施丹莉等, 2018)。
栉孔扇贝(Chlamys farreri)主要分布于我国北部、韩国和日本沿海, 是我国传统的经济养殖贝类(Waller, 1992)。近年来随着我国扇贝养殖规模的不断扩大, 养殖密度过高、环境污染加剧等导致养殖个体生长缓慢、产品品质下降、病害加剧等问题, 对产业造成了严重的打击(王运涛等, 1999; Xiao et al, 2005; 李成林等, 2011; 杨彩霞等, 2013; 孙瑜等, 2014)。扇贝中含有较为丰富的脂肪酸, 尤其是高度不饱和脂肪酸含量丰富, 是决定扇贝产品品质的重要因素之一(Dong, 2001; 楼乔明等, 2012; Xu et al, 2015; Wright et al, 2018)。过氧化物酶体增殖物激活受体PPARs家族在脊椎动物脂肪代谢中发挥重要的调节功能, 但扇贝中是否存在着与脊椎动物类似的PPARs调控通路仍未可知, 扇贝脂质代谢的调控机制仍鲜有研究。本研究利用生物信息学方法鉴定栉孔扇贝PPARs基因, 预测其编码蛋白的结构与功能, 并探究其时空表达模式, 研究结果对于揭示贝类脂质代谢调控机理以及优良扇贝品种的培育提供参考。
1 材料与方法 1.1 研究材料通过NCBI(National Center for Biotechnology Information)网站(https://www.ncbi.nlm.nih.gov/guide/proteins/)查找并下载多物种PPARs蛋白序列, 获得包括脊椎动物, 如人(Homo sapiens)、小鼠(Mus musculus)、原鸡(Gallus gallus)、斑马鱼(Danio rerio)、爪蟾(Xenopus laevis), 和无脊椎动物, 如仿刺参(Apostichopus japonicus)、紫海胆(Strongylocentrotus purpuratus)、美洲牡蛎(Crassostrea virginica)等(表 1)。
| 基因 | 种类 | 序列号 |
| HsPPAR alpha | 人 Homo sapiens | NP_001001928.1 |
| MmPPAR alpha | 小鼠 Mus musculus | NP_001106889.1 |
| GgPPAR alpha | 原鸡 Gallus gallus | NP_001001464.1 |
| DrPPAR alpha | 斑马鱼 Danio rerio | NP_001154805.1 |
| XlPPAR alpha | 非洲爪蟾 Xenopus laevis | NP_001083282.1 |
| HsPPAR delta | 人 Homo sapiens | BAH02282.1 |
| MmPPAR delta | 小鼠 Mus musculus | NP_035275.1 |
| GgPPAR delta | 原鸡 Gallus gallus | NP_990059.1 |
| DrPPAR delta | 斑马鱼 Danio rerio | NP_571543.1 |
| XlPPAR delta | 非洲爪蟾 Xenopus laevis | NP_001081310.1 |
| HsPPAR gamma | 人 Homo sapiens | AAA80314.2 |
| MmPPAR gamma | 小鼠 Mus musculus | AAA19971.1 |
| GgPPAR gamma | 原鸡 Gallus gallus | NP_001001460.1 |
| DrPPAR gamma | 斑马鱼 Danio rerio | NP_571542.1 |
| XlPPAR gamma | 非洲爪蟾 Xenopus laevis | NP_001081312.1 |
| DmE75B | 果蝇 Drosophila melanogaster | AAN11687.1 |
| Cenhr-23 | 秀丽隐杆线虫 Caenorhabditis elegans | NP_001020976.1 |
| SpPPAR gamma | 紫海胆 Strongylocentrotus purpuratus | XP_784429.4 |
| SpPPAR alpha | 紫海胆 Strongylocentrotus purpuratus | XP_030842204.1 |
| ApPPAR gamma | 长棘海星 Acanthaster planci | XP_022112223.1 |
| ApPPAR delta | 长棘海星 Acanthaster planci | XP_022086594.1 |
| AjPPAR alpha | 仿刺参 Apostichopus japonicus | PIK48830.1 |
| MyPPAR delta | 虾夷扇贝 Mizuhopecton yessoensis | XP_021371614.1 |
| ScPPAR | 缢蛏 Sinonovacula constricta | QGX48190.1 |
| CvPPAR delta | 美洲牡蛎 Crassostrea virginica | XP_022301760.1 |
| CgPPAR delta | 长牡蛎 Crassostrea gigas | XP_011420515.1 |
利用实验室的SSH(Secure Shell Client)平台, 将获得的同源序列导入服务器, 命令为blastall-p blastp-i Z.txt-d CF.pep.fa- o hsp90zCF -e 1e-5 -v 40 -b 20 -a 5 -m 8初步筛选获得备选的栉孔扇贝PPARs基因蛋白序列。通过NCBI数据库进行反向BLAST比对(https://blast.ncbi.nlm.nih.gov/Blast.cgi), SMART(http://smart.embl.de/)和NCBI Conserved Domain (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi?RID=HA6KDEF8015&mode=all)网站进行同源性和蛋白质结构域分析, 确定栉孔扇贝的PPAR基因。
1.2.2 栉孔扇贝PPAR基因与蛋白结构分析利用生物信息学的方法分析栉孔扇贝PPAR基因结构、预测蛋白质结构和功能, 具体包括:利用基因组文库的注释信息获得PPARs的基因结构。通过ProtParam程序(http://web.expasy.org/protparam/)获得蛋白质分子量MW及等电点pI信息。通过Geneious4.8.4 (http://www.geneious.com/)预测蛋白质的二级结构。利用Protein Homology/analogY Recognition Engine V 2.0 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?)程序预测蛋白质三级结构信息, 数据导入VMD1.9.3软件构建三维结构图。利用德泰生物信息学在线网站预测有无信号肽(http://www.detaibio.com/tools/signal-peptide.html)和跨膜结构域(http://www.detaibio.com/tools/transmembrane.html)。
1.2.3 PPARs家族序列比对与系统发生分析多序列比对采用线上工具Clustal Omega (https://www.ebi.ac.uk/Tools/msa/clustalo/)。利用MEGA7.0软件对PPARs蛋白序列基于相邻连接法(Neighbor-Joining, NJ)构建系统发生树, Bootstrap值设置为1000。DBD和LBD序列比对图使用GENEDOC软件绘制。
1.2.4 栉孔扇贝PPAR基因时空表达分析利用实验室已发表的栉孔扇贝转录组数据(Li et al, 2017), 获得在胚胎幼虫发育时期包括受精卵, 2-8细胞, 囊胚期, 原肠期, 担轮幼虫, D型幼虫, 壳顶幼虫前期、中期和后期, 眼点幼虫和稚贝时期; 成体组织包括外套膜、眼、鳃丝、横纹肌、平滑肌、足、肝胰腺、肾脏、血细胞和性腺的CfPPAR-like基因表达量, 表达水平由RPKM(Reads Per Kilobase per Million mapped read)表示, 利用GraphPad Prism v8.0.2.263软件绘制柱状图。
2 结果 2.1 栉孔扇贝中PPAR基因鉴定与蛋白序列特征从栉孔扇贝基因组和转录组中鉴定出1个PPAR基因, 命名为CfPPAR-like, 表 2统计CfPPAR-like基因的序列特征。CfPPAR-like基因全长1604 bp, 包含9个外显子和8个内含子(图 1), 编码486个氨基酸, 开放阅读框长度为1572, 理论分子量MW为54305.3 Da, 等电点pI为8.3。栉孔扇贝PPAR蛋白质二级结构如图 2所示, 包含21个α螺旋, 29个β折叠, 33个卷曲和36个转角。预测的蛋白质三级结构显示(图 3), CfPPAR-like蛋白包含PPARs家族5个功能域, DBD功能域和LBD功能域分别位于两个明显不同的区域。利用德泰生物信息学在线工具预测结果显示栉孔扇贝PPAR蛋白不含跨膜区和信号肽, 属于膜内蛋白。
| 特征 | 数据 |
| 总长度(bp) | 1604 |
| 5'端长度(bp) | 17 |
| 3'端长度(bp) | 15 |
| 开放式阅读框长度(bp) | 1572 |
| 氨基酸长度(aa) | 486 |
| 分子量MW (Da) | 54305.3 |
| 等电点pI | 8.3 |
| 外显子数 | 9 |
| 内含子数 | 8 |
| α螺旋数 | 21 |
| β折叠数 | 29 |
| 卷曲数 | 33 |
| 转角数 | 36 |
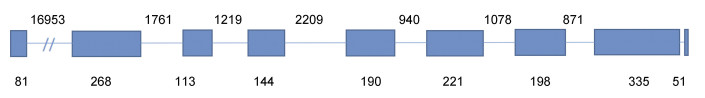 |
| 图 1 CfPPAR-like基因结构 Fig. 1 Structure of gene CfPPAR-like 注:图中蓝色框代表外显子; 蓝色细线代表内含子; 数字代表内含子和外显子的长度 |
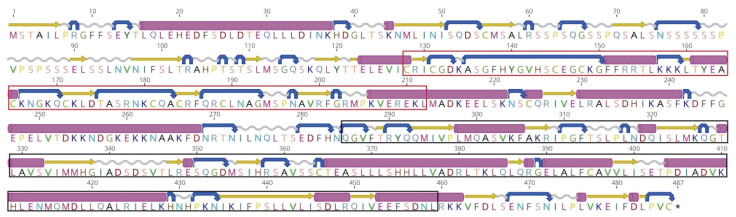 |
| 图 2 CfPPAR-like蛋白二级结构 Fig. 2 The secondary structures of CfPPAR-like protein 注:粉色部分代表α螺旋; 黄色部分代表β折叠; 灰色部分代表卷曲; 蓝色部分代表转角; 红色框标记部分为DBD结构域; 黑色框标记部分为LBD结构域 |
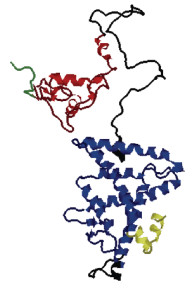 |
| 图 3 CfPPAR-like蛋白质三级结构预测图 Fig. 3 Prediction of the tertiary structure of CfPPAR-like protein 注:绿色部分为A/B区; 红色部分为DBD结构域; 黑色部分为铰链区; 蓝色部分为LBD结构域; 黄色部分为F区 |
利用Clustal Omega对CfPPAR-like蛋白与在NCBI数据库中筛查到的其他物种的PPARs蛋白序列进行多序列比对, DBD区域和LBD区域序列比对结果如图 4所示, 可以看出, DBD结构域非常保守, 种间氨基酸相似度范围在49.40%-96.43%, 而LBD结构域变异程度较大, 氨基酸相似度范围在14.66%-96.04%。
 |
| 图 4 栉孔扇贝CfPPAR与其他物种PPARs DBD (A)和LBD(B)结构域的序列比对结果 Fig. 4 Alignment of DBD (A) and LBD (B) domains of CfPPAR-like with their homologs in other species. 注: Hs:人H. apiens; Mm:小鼠M. musculus; Gg:鸡G.gallus; Dr:斑马鱼D.rerio; Xl:非洲爪蟾X.laevis; Dm:果蝇D.melanogaster; Ce:秀丽隐杆线虫C.elegans; Sp:紫海胆S.purpuratus; Ap:长棘海星A.planci; Aj:仿刺参A.japonicus; Cv:美洲牡蛎C.virginica; Cg:长牡蛎C.gigas; Sc:缢蛏S.constricta; My:虾夷扇贝M.yessonsis |
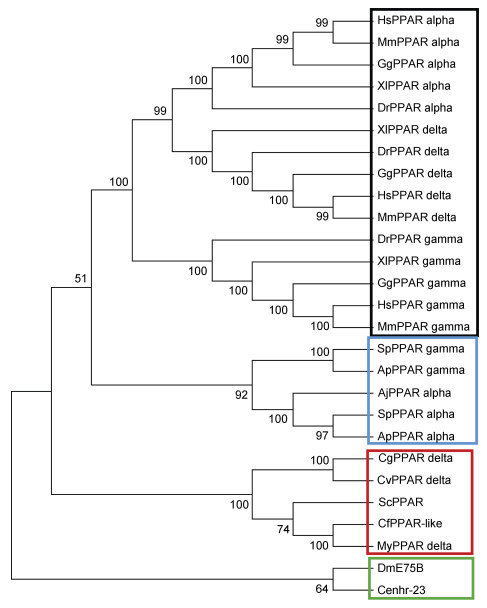
利用邻近相位法(NJ法)对CfPPAR-like蛋白和其他物种PPARs蛋白序列构建系统发生树(图 5), PPARs家族成员主要聚为三个亚支, 脊椎动物的PPARs家族的三种亚型分别聚类后聚为单独亚支, 三种亚型上, PPARα和PPARβ两种亚型同源性较高先聚在一起, 后与PPARγ聚在一起。无脊椎动物中, 棘皮动物聚为一支, 软体动物聚为一支, 果蝇与线虫的PPAR基因同源物E75B和nhr-23聚在进化树的最外端。
CfPPAR-like在栉孔扇贝胚胎和幼虫发育时期的表达主要经历了三个阶段(图 6), 首先在受精卵至原肠期表达量先升高后下降, 在2-8细胞期表达量达到最高水平, 此时期细胞分裂旺盛, 表明CfPPAR-like参与扇贝胚胎发育早期的细胞分裂; 第二阶段为原肠期至壳顶幼虫时期, 在D型幼虫表达量再次升高, 暗示其可能在扇贝贝壳的形成过程中发挥作用; 最后一个阶段包含眼点幼虫和稚贝时期, CfPPAR-like在这两个时期保持较高的表达水平, 表明CfPPAR-like参与扇贝幼虫附着变态发育为稚贝的这一重要过程。
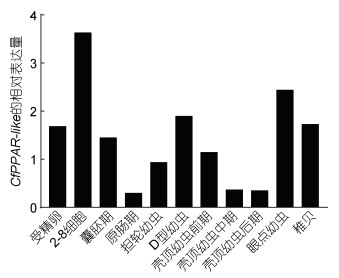 |
| 图 6 CfPPAR-like在栉孔扇贝各个发育时期中的表达量 Fig. 6 Expression levels of CfPPAR-like during developmental stages |
CfPPAR-like在栉孔扇贝成体各组织中均有表达(图 7)。肾脏组织中表达量最高, 其次是雄性性腺、消化腺和雌性性腺, 眼、鳃丝、外套膜、平滑肌和足中有少量表达, 横纹肌和血细胞中微量表达。
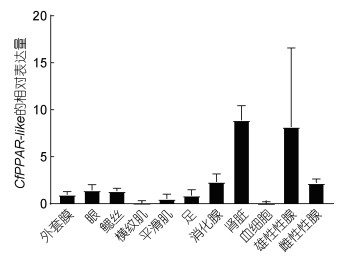 |
| 图 7 CfPPAR-like在栉孔扇贝不同组织中的表达量 Fig. 7 Expression levels of CfPPAR-like in different tissues |
众多研究都表明PPARs是调节机体脂质代谢的重要枢纽, 在调控畜禽机体肝脏脂质代谢、鱼类脂肪酸β-氧化等方面具有重要作用(宁丽军, 2016; 王朝阳等, 2018; 宁丽军等, 2019)。目前, 对无脊椎动物PPARs的研究相对较少。扇贝作为我国的重要经济贝类, 不饱和脂肪酸是干贝品质的重要因素, 探究PPARs对扇贝脂肪代谢的调控机制具有重要意义。本研究利用已发表的栉孔扇贝基因组与转录组数据, 获得栉孔扇贝CfPPAR-like基因, 并分析其基因与蛋白结构信息, 同时分析了栉孔扇贝CfPPAR-like基因在胚胎幼虫各发育时期和成体各组织中的表达模式, 以期为贝类脂质代谢机制研究提供参考。
相比脊椎动物PPARs家族具有PPARα, PPARβ/δ和PPARγ三种亚型, 栉孔扇贝基因组中仅鉴定出一个PPAR基因。已有线虫和果蝇核激素受体家族的研究指出, 线虫和果蝇等蜕皮动物中并没有PPAR基因, 仅存在PPAR基因的同源物E75B和nhr-23(Maglich et al, 2001; Hong et al, 2010)。通过对栉孔扇贝和其他物种的PPAR结构域DBD和LBD的蛋白多序列比对发现, 相较于LBD的高变异性, 所有物种DBD保守性较高。PPAR结构域在脊椎动物与无脊椎动物之间差异较大, 如DBD的第23-24、36、58-59、79、91-92和94-95位上共10个氨基酸位点在脊椎动物中完全保守, 而在无脊椎动物中存在变异, 而LBD序列中104-290这段序列上脊椎动物之间的相似性更高, 在无脊椎动物中存在较高变异度。系统发生树的结果表明三种脊椎动物的PPARs亚型分别聚类后再与无脊椎动物的PPAR聚类, 而果蝇和线虫的PPAR同源物E75B和nhr-23位于进化树的最外端, 支持PPAR所属的核激素受体超家族经历两次演化, 第一次是后生动物出现早期产生了核激素受体各种家族(如PPAR、RAR等), 第二次在脊椎动物进化早期产生了各种亚型(如alpha, delta和gamma等)(Escriva et al, 2000)。
从时空表达上看, CfPPAR-like基因在胚胎幼虫的发育时期主要分为三个阶段, 在2-8细胞期、D型幼虫、眼点幼虫和稚贝均保持较高的表达水平, 这些时期均为扇贝幼虫发育的关键时期, 暗示CfPPAR-like在扇贝早期细胞分裂、贝壳形成、幼虫附着变态等方面发挥重要作用。在牛、人和小鼠胚胎中均发现PPARs不同亚型的表达, 其中PPARδ和PPARγ通过对脂质、碳水化合物和氨基酸代谢的影响, 促进胚胎着床和胎盘发育, 所产生的代谢物可作为胎儿能量代谢、细胞膜合成和信号分子的底物(Bogacka et al, 2015)。从组织表达上来看, CfPPAR-like基因在所有扇贝组织中普遍表达, 在肾脏、性腺和消化腺中表达量较高, 表明CfPPAR-like同时在扇贝的脂肪代谢和生殖等过程中发挥重要功能。已有研究表明PPAR基因的表达具有一定的物种特异性, 例如人、小鼠等哺乳动物的PPARs基因主要在脂肪细胞中高表达, 草鱼、大黄鱼PPARs基因主要在肝脏中大量表达, 团头鲂PPARα基因主要在肌肉、肝脏中高表达, 而虹鳟的PPARα主要在脂肪组织中表达(Meng et al, 2005; 孔路军等, 2006; 周旋等, 2009; 陈亮, 2011; 高俊等, 2016; 赵亭亭等, 2020), 这些高表达的PPARs基因在动物脂类代谢和脂肪酸β氧化等过程中具有重要作用。另外, 所有PPAR亚型均在不同物种(啮齿动物、绵羊、奶牛、猪、兔子、水牛和人类)的卵巢中表达, 主要发挥包括卵巢组织的重塑、类固醇的生成等功能, 其中PPARγ相较于其他类型作用研究更为广泛(Bogacka et al, 2015)。团头鲂(Megalobrama amblycephala)PPARα在精巢和卵巢中都有表达, 而且在精巢中的表达量比卵巢更高, 暗示其参与了生殖细胞的成熟过程(Zhao et al, 2011)。
4 结论本研究获得了栉孔扇贝PPAR基因CfPPAR-like全长ORF序列, 进化分析和蛋白分析结果表明CfPPAR-like包含保守结构域DBD和LBD, 多序列比对结果表明DBD保守性较高, 而LBD具有较高的多样性, 进化树结果支持PPAR所属的核激素受体超家族在脊椎动物进化早期出现了各亚型这一假说。栉孔扇贝CfPPAR-like主要在2-8细胞、D型幼虫、眼点幼虫和稚贝时期高表达, 成体组织中性腺、消化腺和肾脏中表达量较高。研究结果对揭示PPAR基因在贝类脂肪代谢中的调控机制以及优良扇贝品种的选育提供参考。
王运涛, 相建海. 1999. 栉孔扇贝大规模死亡的原因探讨. 海洋与湖沼, 30(6): 770-774 DOI:10.3321/j.issn:0029-814X.1999.06.029 |
王朝阳, 蓝立明, 白丁平, 等. 2018. 畜禽PPARs基因研究进展. 生物技术通报, 34(12): 32-40 |
孔路军, 王爱国, 傅金恋, 等. 2006. 猪PPARs基因组织表达特性的研究. 中国畜牧杂志, 42(19): 1-4 DOI:10.3969/j.issn.0258-7033.2006.19.001 |
宁丽军, 2016.尼罗罗非鱼过氧化物体增殖激活受体(PPARα)激活及其降脂机制研究.上海: 华东师范大学博士学位论文
|
宁丽军, 李加敏, 孙胜香, 等. 2019. 鱼类脂肪酸β-氧化研究进展. 水产学报, 43(1): 128-142 |
孙瑜, 慕永通. 2014. 中国扇贝产量、产值在世界所处的地位. 中国渔业经济, 32(4): 100-106 DOI:10.3969/j.issn.1009-590X.2014.04.015 |
李成林, 宋爱环, 胡炜, 等. 2011. 山东省扇贝养殖产业现状分析与发展对策. 海洋科学, 35(3): 92-98 |
杨彩霞, 李赟, 王崇明, 等. 2013. 两个海区栉孔扇贝感染AVNV的比较分析. 水产学报, 37(10): 1579-1584 |
陈亮, 2011.草鱼PPAR基因的克隆、组成型表达及与诱导剂作用的研究.广州: 暨南大学硕士学位论文
|
周旋, 2009.大黄鱼部分脂肪代谢相关基因的克隆及其表达分析.厦门: 集美大学硕士学位论文
|
赵亭亭, 杨双双, 马爱军, 等. 2020. 大菱鲆脂质代谢相关基因PPARs的组织表达及其对高温胁迫的响应. 水产学报, 44(4): 515-522 |
施丹莉, 王若冰, 柳林. 2018. 过氧化物酶体增殖物激活受体在视神经保护中的作用. 上海交通大学学报(医学版), 38(4): 477-480 DOI:10.3969/j.issn.1674-8115.2018.04.024 |
高俊, 徐钢春, 杜富宽, 等. 2016. 鱼类PPARs基因组织表达及功能研究进展. 长江大学学报(自然科学版), 13(33): 43-46 DOI:10.3969/j.issn.1673-1409(s).2016.33.010 |
楼乔明, 王玉明, 徐杰, 等. 2012. 栉孔扇贝和海湾扇贝脂质及其脂肪酸组成分析. 中国食品学报, 12(11): 198-203 |
Bervejillo M L, Ferreira A M, 2019. Understanding peroxisome proliferator-activated receptors: from the structure to the regulatory actions on metabolism. In: Trostchansky A, Rubbo H eds. Bioactive Lipids in Health and Disease. Cham: Springer, 39-57
|
Bogacka I, Kurzynska A, Bogacki M et al, 2015. Peroxisome proliferator-activated receptors in the regulation of female reproductive functions. Folia Histochemica et Cytobiologica, 53(3): 189-200 DOI:10.5603/fhc.a2015.0023 |
Desvergne B, Wahli W, 1999. Peroxisome proliferator-activated receptors:nuclear control of metabolism. Endocrine Reviews, 20(5): 649-688 |
Dong F M, 2001. The nutritional value of shellfish. http://citeseerx.ist.psu.edu/viewdoc/download;jsessionid=4AEC67776A7588AAC4F6C5A1E0B8C30E?doi=10.1.1.169.7751&rep=rep1&type=pdf
|
Dreyer C, Krey G, Keller H et al, 1992. Control of the peroxisomal β-oxidation pathway by a novel family of nuclear hormone receptors. Cell, 68(5): 879-887 DOI:10.1016/0092-8674(92)90031-7 |
Escriva H, Delaunay F, Laudet V, 2000. Ligand binding and nuclear receptor evolution. Bioessays, 22(8): 717-727 DOI:10.1002/1521-1878(200008)22:8<717::AID-BIES5>3.0.CO;2-I |
Glass C K, Ogawa S, 2006. Combinatorial roles of nuclear receptors in inflammation and immunity. Nature Review Immunology, 6(1): 44-55 DOI:10.1038/nri1748 |
Hong J W, Park K W, 2010. Further understanding of fat biology:lessons from a fat fly. Experimental & Molecular Medicine, 42(1): 12-20 |
Honsho M, Yamashita S I, Fujiki Y, 2016. Peroxisome homeostasis:mechanisms of division and selective degradation of peroxisomes in mammals. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research, 1863(5): 984-991 DOI:10.1016/j.bbamcr.2015.09.032 |
Li Y L, Sun X Q, Hu X L et al, 2017. Scallop genome reveals molecular adaptations to semi-sessile life and neurotoxins. Nature Communications, 8(1): 1721 DOI:10.1038/s41467-017-01927-0 |
Maglich J M, Sluder A, Guan X et al, 2001. Comparison of complete nuclear receptor sets from the human, Caenorhabditis elegans and Drosophila genomes. Genome Biology, 2(8): research0029 |
Menendez-Gutierrez M P, Roszer T, Ricote M, 2012. Biology and therapeutic applications of peroxisome proliferator-activated receptors. Current Topics in Medicinal Chemistry, 12(6): 548-584 DOI:10.2174/156802612799436669 |
Meng H, Li H, Zhao J G et al, 2005. Differential expression of peroxisome proliferator-activated receptors alpha and gamma gene in various chicken tissues. Domestic Animal Endocrinology, 28(1): 105-110 DOI:10.1016/j.domaniend.2004.05.003 |
Tyagi S, Gupta P, Saini A S et al, 2011. The peroxisome proliferator-activated receptor:a family of nuclear receptors role in various diseases. Journal of Advanced Pharmaceutical Technology & Research, 2(4): 236-240 |
Waller T R, 1992. Evolutionary relationships among commercial scallops (Mollusca: Bivalvia: Pectinidae). In: Shumway S ed. Scallops: Biology, Ecology and Aquaculture. Amsterdam: Elsevier, 1-74
|
Wang Y X, Yu Y P, Wang Q et al, 2019. PPAR-δ of orange-spotted grouper exerts antiviral activity against fish virus and regulates interferon signaling and inflammatory factors. Fish & Shellfish Immunology, 94: 38-49 |
Wright A C, Fan Y, Baker G L, 2018. Nutritional value and food safety of bivalve molluscan shellfish. Journal of Shellfish Research, 37(4): 695-708 DOI:10.2983/035.037.0403 |
Xiao J, Ford S E, Yang H S et al, 2005. Studies on mass summer mortality of cultured Zhikong scallops (Chlamys farreri Jones et Preston) in China. Aquaculture, 250(3-4): 602-615 DOI:10.1016/j.aquaculture.2005.05.002 |
Xu Q, Gao F, Wang H et al, 2015. Quality indices as potential markers indicating the origin of cultured scallop (Argopecten irradians) in the North China Sea. Journal of Shellfish Research, 34(3): 743-750 DOI:10.2983/035.034.0303 |
Zhao Y H, Gul Y, Li S et al, 2011. Cloning, identification and accurate normalization expression analysis of PPARα gene by GeNorm in Megalobrama amblycephala.. Fish & Shellfish Immunology, 31(3): 462-468 |
 2021, Vol. 52
2021, Vol. 52