中国海洋湖沼学会主办。
文章信息
- 李斯远, 何治江, 吕泓玥, 汤秋晗, 廖智, 王健鑫, 严小军, 张晓林. 2021.
- LI Si-Yuan, HE Zhi-Jiang, LYU Hong-Yue, TANG Qiu-Han, LIAO Zhi, WANG Jian-Xin, YAN Xiao-Jun, ZHANG Xiao-Lin. 2021.
- 厚壳贻贝(Mytilus coruscus)养殖海域与天然生长海域的微生物群落比较研究
- COMPARATIVE STUDY ON MICROBIAL COMMUNITY IN MUSSEL MYTILUS CORUSCUS BODY AND SEAWATER OF ITS NATURAL AND CULTURAL SEA AREA IN ZHOUSHAN, ZHEJIANG
- 海洋与湖沼, 52(1): 196-205
- Oceanologia et Limnologia Sinica, 52(1): 196-205.
- http://dx.doi.org/10.11693/hyhz20200700217
文章历史
-
收稿日期:2020-07-22
收修改稿日期:2020-09-11
2. 浙江海洋大学 国家海洋设施养殖工程技术研究中心 舟山 316022
2. National Engineering Research Center of Marine Facilities Aquaculture, Zhejiang Ocean University, Zhoushan 316022, China
厚壳贻贝Mytilus coruscus属软体动物门Mollusca、双壳纲Bivalvia、异柱目Anisomyaria、贻贝科Mytilidae、贻贝属Mytilus(叶莹莹等, 2011), 俗称淡菜, 为温水性种, 主要分布在我国黄海、渤海和东海沿岸, 以浙江沿海资源量最大。厚壳贻贝具有肉质鲜美、营养丰富、蛋白质含量高、生长繁殖快、抗病能力强、易于人工养殖等诸多优点, 是浙江舟山海域具有重要经济价值的养殖贝类之一(徐晴晴等, 2018)。目前厚壳贻贝主要养殖区域为浙江嵊泗海域和东极海域。厚壳贻贝在养殖过程中, 对于海域环境中的藻类和微生物多样性具有较强的依赖性; 一方面, 藻类是贻贝的主要食物来源(朱雨瑞等, 2010), 另一方面, 贻贝在养殖过程中对海域微生物群落具有特异的富集作用(陈瑜等, 2016), 但目前尚不清楚微生物群落对贻贝的养殖具有哪些影响。
但近年来, 随着厚壳贻贝的养殖区域的不断扩展和养殖密度逐渐增加, 其养殖过程显现种种问题, 例如贻贝个体的肥满度下降, 生长速度下降, 贻贝逃苗和病害也时有发生(林永添, 2007)。有研究显示, 微生物群落变化能够影响贻贝的附着和防病(杨娜, 2018; 梁箫等, 2020)。国内外已有针对不同贝类生长区海域浮游群落的调查研究(陈雷等, 2013; 尹洁慧, 2013; 金雷等, 2015; Fogel et al, 2015), 但其主要焦点集中于海域中浮游植物的种类和丰度对贝类摄食以及营养价值的影响。关于海域微生物群落与贻贝生长过程以及生理生化的影响尚未见报道。厚壳贻贝作为我国东部海域最重要的养殖贝类之一, 其生存环境的微生物群落调查以及与其体内微生物群落的对比分析将对贻贝养殖具有重要研究意义。为此, 本文将针对厚壳贻贝生长海域的微生物群落结构特征及其体内微生物组成开展研究, 以期为厚壳贻贝的健康、生态养殖提供重要的理论依据。
本研究主要以厚壳贻贝为对象, 首先将调查嵊泗县枸杞岛和东极镇庙子湖岛厚壳贻贝养殖区和野生区海域的微生物群落的结构特征, 并与厚壳贻贝体内分离到的微生物组成情况相结合进行比较分析, 以探明厚壳贻贝生长海域的微生物群落分布特征以及养殖和野生生长海域的微生物组成差异。本论文的研究结果一方面能够为厚壳贻贝的人工养殖和野生驯化的海域选择提供强有力的参考, 另一方面可以维持海域微生物群落的动态平衡, 促进贝类养殖产业的健康、可持续发展。
1 材料与方法 1.1 厚壳贻贝生存海域的海水微生物群落分析 1.1.1 实验材料于2019年5—6月, 选择嵊泗县枸杞岛(30°42′N, 122°46′E)厚壳贻贝养殖区海域5、10 m水深和厚壳贻贝野生生长区海域5、10 m水深; 东极镇庙子湖岛(30°11′N, 122°41′E)厚壳贻贝养殖海域5、10 m水深和厚壳贻贝野生生长区海域5、10 m水深, 共计8个点位, 具体采样位置见图 1。在调查期间, 每个点位采集3个平行海水样本, 送往实验室经静置沉淀并过滤后进行16S rRNA扩增子测序。
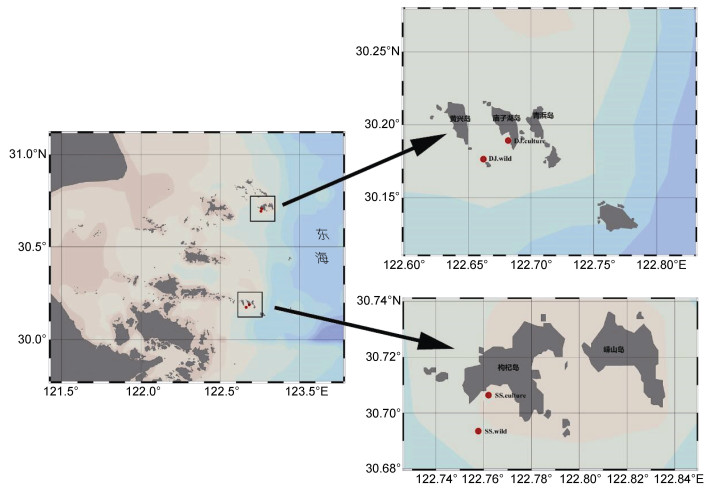 |
| 图 1 枸杞岛(右下)和东极岛(右上)厚壳贻贝养殖和野生生长海域采样站位图 Fig. 1 Location of the sampling points for M. coruscus in natural and cultural regions in Gouqi Island (lower right) and Dongji Island (upper right) |
取质量合格的基因组DNA样品30 ng及对应的融合引物配置PCR反应体系, 设置PCR反应参数进行PCR扩增, 使用Agencourt AMPure XP磁珠对PCR扩增产物进行纯化, 并溶于Elution Buffer, 贴上标签, 完成建库。使用Agilent2100 Bioanalyzer对文库的片段范围及浓度进行检测。检测合格的文库根据插入片段大小, 选择HiSeq平台进行测序。然后对所有样品的全部序列进行聚类, 以97%的相似度将序列聚类成OUT, 然后对OTU的代表序列进行物种注释, 确定16S rRNA序列对应的微生物名称(赵立君等, 2019)。
1.1.3 生物信息学分析下机数据过滤, 剩余高质量的Clean data用于后期分析(Magoc et al, 2011); 通过reads之间的overlap关系将reads拼接成Tags; 将Tags聚类成OTU并与数据库比对、物种注释; 基于OTU和注释结果进行样品物种复杂度分析, 组间物种差异分析等。再利用UCHIME(v 4.2.40)将PCR扩增产生的嵌合体从OTU代表序列中去除, 使用usearch_global方法将所有Tags比对回OTU代表序列(Wang et al, 2007), 对每个样品的OTU丰度、多样性指数等进行分析, 同时对物种注释在门分类水平上进行群落结构的统计分析。在以上分析的基础上, 进行基于OTU的主成分分析(PCA)。
1.2 厚壳贻贝体内组织微生物分离与鉴定 1.2.1 培养基制备第一类分离选用2216E普通海水培养基。为还原贻贝组织的营养成分, 尽可能分离到贻贝体内全部种类的微生物, 选取足量的厚壳贻贝全组织进行匀浆并过滤, 120℃、21 min高温高压处理, 加入适量琼脂制备成厚壳贻贝肉汤培养基, 用于第二类分离。
1.2.2 微生物分离与筛选选取来自同一生长环境的厚壳贻贝10只, 解剖取其鳃、性腺、消化腺、外套膜、足并用无菌海水轻轻冲洗去除组织表面杂质, 使用两种不同方式分离微生物: (1)第一类分离针对贻贝组织表面附着的微生物, 用无菌棉签分别刮取不同组织的表面, 将棉签伸至于放有玻璃珠的三角瓶中并加入少量无菌海水, 常温振荡培养1 h。(2)第二类次分离针对可能在贻贝体内生长的微生物, 分别将上述5类组织匀浆后, 加入放有玻璃珠的三角瓶中, 常温振荡培养1 h。两次分离都根据选用的不同组织分成5组, 每组3次重复。
取上述适量处理过的培养液接种到对应的培养基中, 28℃过夜培养, 分别挑取不同细胞形态、大小、颜色的菌落至海水LB培养基上, 反复纯化三次。
1.2.3 微生物鉴定使用试剂盒(TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit)分别提取筛选菌落的基因组DNA, 选用通用引物27F (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-TACGGTTACCTTGTTACGACTT-3′)PCR扩增16S rDNA, PCR产物送至生工生物工程(上海)有限公司进行进一步的分析和鉴定。
2 结果与分析 2.1 海水样品测序结果如表 1、表 2所示, 两个海域的所有样品优化后序列占优化前序列的80%以上, 因此可以很好地用于后续的进一步分析。
| 样品名称 | 优化前序列数 | 优化后序列数 | 序列优化百分比 |
| culture.10m | 74974 | 60785 | 81.07% |
| culture.5m | 74917 | 61905 | 82.63% |
| wild.10m | 74949 | 60756 | 81.06% |
| wild.5m | 74912 | 62122 | 82.93% |
| 样品名称 | 优化前序列数 | 优化后序列数 | 序列优化百分比 |
| culture.10m | 74937 | 61514 | 82.09% |
| culture.5m | 74919 | 64847 | 86.56% |
| wild.10m | 74902 | 61326 | 81.87% |
| wild.5m | 74906 | 62122 | 82.93% |
通过多样性指数可以对两个海域海水样品中微生物多样性进行分析。从表 3可得, 东极海域海水样品有效序列共计245568条, 其中养殖区海域10 m水深样品包含60785条, OTU数674个; 养殖区海域5 m水深样品包含61905条, OTU数616个; 野生生长区海域10 m水深样品包含60756条, OTU数635个; 野生生长区海域5 m水深样品包含62122条, OTU数535个。
| 样品名称 | 有效序列 | OTU数 | Sobs | Chao | Ace | Shannon | Simpson | Coverage |
| culture.10m | 60785 | 674 | 674 | 754.70 | 758.30 | 4.35 | 0.0267 | 0.9988 |
| culture.5m | 61905 | 616 | 616 | 681.58 | 680.68 | 4.34 | 0.0254 | 0.9988 |
| wild.10m | 60756 | 635 | 635 | 638.10 | 701.45 | 4.43 | 0.0217 | 0.9988 |
| wild.5m | 62122 | 535 | 535 | 656.92 | 658.79 | 4.04 | 0.0386 | 0.9982 |
从表 4可得, 嵊泗海域海水样品有效序列共计249809条, 其中养殖区海域10 m水深样品包含61514条, OTU数592个; 养殖区海域5 m水深样品包含64847条, OTU数584个; 野生生长区海域10 m水深样品包含61326条, OTU数623个; 野生生长区海域5 m水深样品包含62122条, OTU数570个。
| 样品名称 | 有效序列 | OTU数 | Sobs | Chao | Ace | Shannon | Simpson | Coverage |
| culture.10m | 61514 | 592 | 592 | 631.30 | 648.59 | 4.32 | 0.0274 | 0.9986 |
| culture.5m | 64847 | 584 | 584 | 622.14 | 649.05 | 4.25 | 0.0324 | 0.9986 |
| wild.10m | 61326 | 623 | 623 | 648.71 | 657.24 | 4.36 | 0.0322 | 0.9989 |
| wild.5m | 62122 | 570 | 570 | 612.50 | 625.54 | 4.18 | 0.0348 | 0.9987 |
综上可以得, 东极海域养殖区微生物(OTU为616—674)丰富度大于野生生长区微生物(OTU为535—635), 10 m水深微生物(OTU为635—674)丰富度大于5 m水深微生物(OTU为535—616);嵊泗海域野生生长区区微生物(OTU为535—674)丰富度大于养殖区区微生物(OTU为584—592), 10 m水深微生物(OTU为592—623)丰富度大于5 m水深微生物(OTU为570—584)。嵊泗海域总体微生物(OTU为570—623)丰富度大于东极海域(OTU为535—674)。
本文通过包括Sobs指数、Chao指数、ACE指数、Shannon指数、Simpson指数以及Good- coverage指数的Alpha多样性来分析海水样品中微生物多样性差异(Schloss et al, 2009)。Sobs指数、Chao指数和ACE指数反映样品中群落的丰富度, 而Shannon指数以及Simpson指数反映群落的多样性, Good-coverage反映了样品文库的覆盖率。其中, Sobs指数、Chao指数和ACE指数和Shannon指数越大, Simpson指数越小, 说明样品中的物种越丰富; Good-coverage数值越高, 则表明样品中序列没有被测出来的概率越低, 该值反映了测序结果是否代表样品的真实情况, 所有样品的Good-coverage指数均在0.998以上, 因此本次测序结果代表了样本中微生物的真实情况。
由此可得, 东极海域不同采样点微生物丰富度及多样性由大到小分别为culture.10m > wild.10m > culture.5m> wild.5m;嵊泗海域不同采样点微生物丰富度及多样性由大到小分别为wild.10m > culture.10m > culture.5m > wild.5m。
Core-Pan OTU以花瓣图展示所有样本共有和特有的OTU, 分析数据与维恩图相同, 但相比于维恩图只展示5组(或5个样品)以内的数据, 花瓣图可以展示任意组(或样品), 因此可以尽可能多的展示组间OTU的异同。
从图 2可知, 东极海域样品微生物OTU数为440—473, 共有OTU达到436, 说明东极海域总体上微生物组成相似度较高, 养殖区海域10 m水深特有OTU最高, 达到37;野生生长区5 m水深特有OTU最低, 仅达到4。从图 3可知, 嵊泗海域样品微生物OTU数为455—473, 共有OTU达到439, 说明嵊泗海域总体上微生物组成相似度也较高, 野生生长区海域10m水深特有OTU最高, 达到34;养殖区海域特有OTU均最低, 仅达到16。
 |
| 图 2 东极海域样品OTU分布Core-Pan图 Fig. 2 The Core-pan diagram of OTU distribution in the Dongji sea area |
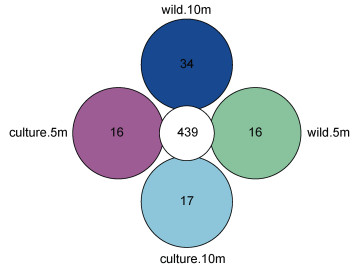 |
| 图 3 嵊泗海域样品OTU分布Core-Pan图 Fig. 3 The Core-pan diagram of OTU distribution in the Shengsi sea area |
物种柱状图可以直观地展示各样本物种组成及比例, 反映样本间物种的变化情况。通过与数据库进行比对, 对OTU进行物种分类, 本文选取在门分类水平上(物种丰富度均大于0.5%)对各样品所作的物种丰度柱状图。
从图 4可知, 东极海域主要包含变形菌门Proteobacteria、拟杆菌门Bacteroidetes、广古菌门Euryarchaeota、放线菌门Actinobacteria、泉古菌门Crenarchaeota等在内的30个门类, 其中变形菌门是四个采样点的最大优势门类, 其在野生生长区10 m水深、野生生长区5 m水深、养殖区10 m水深、养殖区5 m水深的百分比分别为37.58%、28.53%、36.26%、37.13%, 拟杆菌门(19.72%—26.41%)、放线菌门(9.23%—20.86%)是东极海域第二、第三优势细菌门, 这三大优势细菌门类占据东极海域细菌比例的60%以上。
 |
| 图 4 东极海域微生物门分类水平物种丰度柱状图 Fig. 4 The phylum abundance in the Dongji sea area |
此外, 东极养殖区海域和野生生长区海域微生物门类无明显差异, 但可明显得知野生生长区海域各类优势细菌门所占比例大于养殖区海域各类优势细菌门所占比例。
从图 4可知, 东极海域主要包含变形菌门Proteobacteria、拟杆菌门Bacteroidetes、广古菌门Euryarchaeota、放线菌门Actinobacteria、泉古菌门Crenarchaeota等在内的27个门类, 其中变形菌门是四个采样点的最大优势门类, 其在野生生长区10 m水深, 野生生长区5 m水深, 养殖区10 m水深, 养殖区5 m水深的百分比分别为51.48%、54.66%、61.88%、60.46%, 拟杆菌门(17.48%—19.72%), 放线菌门(9.08%—11.99%)是嵊泗海域第二、第三优势细菌门, 这三大优势细菌门类占据东极海域细菌比例的80%以上。
同样, 嵊泗养殖区海域和野生生长区海域微生物门类无明显差异, 但可明显得知野生生长区海域各类优势细菌门所占比例大于养殖区海域各类优势细菌门所占比例。
综合图 4、图 5可知, 东极海域总体微生物丰富度要大于嵊泗海域, 但是在同样优势菌门的条件下, 嵊泗海域各类优势菌门所占的微生物总比例远高于东极海域的各类优势菌门。
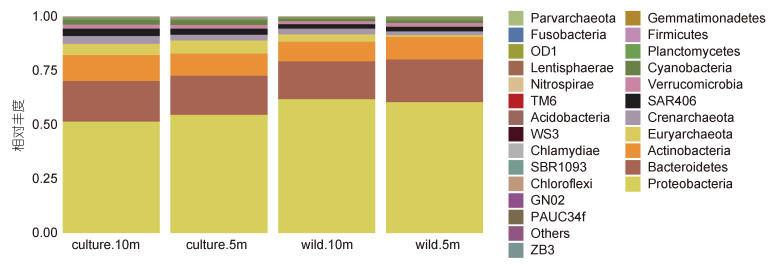 |
| 图 5 嵊泗海域微生物门分类水平物种丰度柱状图 Fig. 5 The phylum abundance in the Shengsi sea area |
从图 6可知, 东极海域主要包含厌氧氨氧化菌属Candidatus Portiera (5.57%—8.03%), 海洋氨氧化古菌属Nitrosopumilus (5.27%—6.94%), Fluviicola (4.21%—6.34%), 蓝细菌聚球藻属Synechococcus (1.82%—3.09%), 弧菌属Vibrio (0.33%—2.50%)等在内的9个属类。从表 5可知, 东极海域厚壳贻贝养殖区和野生生长区属分类水平微生物丰度之间的比较无明显规律。
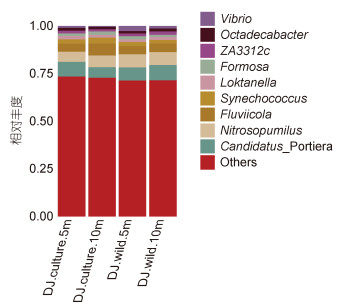 |
| 图 6 东极海域微生物属分类水平物种丰度柱状图 Fig. 6 The genus abundance in the Dongji sea area |
| Taxon分类 | wild.10m | wild.5m | culture.10m | culture.5m |
| Candidatus_Portiera | 0.077605504 | 0.055680757 | 0.067911491 | 0.080268153 |
| Nitrosopumilus | 0.052686154 | 0.060896301 | 0.069392120 | 0.067845893 |
| Fluviicola | 0.042102838 | 0.063407488 | 0.042905322 | 0.044875212 |
| Synechococcus | 0.022977155 | 0.030874730 | 0.021107181 | 0.018140699 |
| Loktanella | 0.018105208 | 0.015051029 | 0.013868553 | 0.016444552 |
| ZA3312c | 0.014698137 | 0.011396929 | 0.014526610 | 0.017381472 |
| Vibrio | 0.008460070 | 0.003332153 | 0.025055524 | 0.011873031 |
从图 7可知, 嵊泗海域主要包含十八杆菌属Octadecabacter (10.80%—15.04%), 假交替单胞菌属Pseudoalteromonas (2.41%—8.77%), 厌氧氨氧化菌属Candidatus_Portiera (3.57%—6.69%), ZA3312c(1.80%—3.21%), 沉积物陆丹氏菌属Loktanella (3.26%—4.29%), 弧菌属Vibrio (0.21%—6.36%)等在内的13个属类。
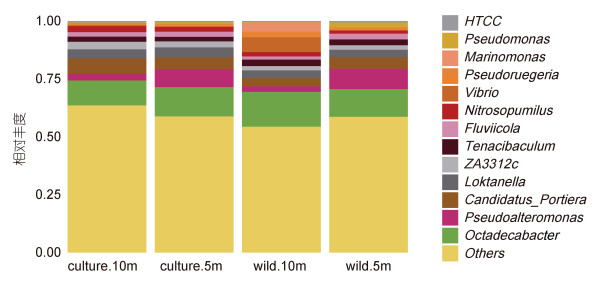 |
| 图 7 嵊泗海域微生物属分类水平物种丰度柱状图 Fig. 7 The genus abundance in the Shengsi sea area |
从表 6可知, 嵊泗海域厚壳贻贝养殖区和野生生长区属分类水平微生物丰度之间可观察到明显的规律, 总体上养殖区海域各类优势属微生物丰度大于野生生长区海域。
| Taxon分类 | wild.10m | wild.5m | culture.10m | culture.5m |
| Octadecabacter | 0.150388529 | 0.120236865 | 0.107963996 | 0.126734490 |
| Candidatus_Portiera | 0.035422831 | 0.049300662 | 0.066888432 | 0.051930073 |
| Loktanella | 0.032935592 | 0.032568970 | 0.039053582 | 0.042899456 |
| ZA3312c | 0.018028416 | 0.019769612 | 0.032074487 | 0.026077718 |
| Pseudoalteromonas | 0.024124590 | 0.087667895 | 0.028748002 | 0.076848781 |
| Nitrosopumilus | 0.018841239 | 0.012830200 | 0.025551968 | 0.019880236 |
| Vibrio | 0.063611536 | 0.002143507 | 0.004565763 | 0.002865330 |
本文采用主成分(PCA)分析, 通过分析不同样品在图上的分散和聚集的情况, 根据离散程度可以了解不同样品的组成是否具有相似性(Jolliffe, 1986; Avershina et al, 2013)。从图 8可知:轴1 (PC1)贡献值为72.72%, 轴2 (PC2)贡献值为13.13%, 总贡献值为85.85%。从整体上来看, 东极和嵊泗海域之间的微生物群落存在明显差异, 两个海域的野生生长区5 m和10 m之间的微生物群落相似度明显低于养殖区5 m和10 m的微生物群落相似度, 而这种差异在嵊泗海域尤为明显。
 |
| 图 8 各海域样品PCA分析图 Fig. 8 The PCA analysis diagram of sea area samples |
由表 7可知, 从厚壳贻贝的鳃、性腺、足、消化腺中均分离出微生物, 主要包括:黏着杆菌属Tenacibaculum sp., 蜡样芽孢杆菌属Bacillus cereus, 红色杆菌属Erythrobacter sp., 鲁杰氏菌属Ruegeria sp., 弧菌属Vibrio sp., 巨型弧菌属Vibrio gigantis, 假交替单胞菌属Pseudoalteromonas这7类菌属。
| 分离方式 | 分离组织 | 菌属名 | 相似度(%) | 覆盖度(%) |
| 第一类 | 鳃、消化腺 | 弧菌 Vibrio sp. | 99.79 | 99 |
| 鳃 | 蜡样芽孢杆菌 Bacillus cereus | 99.86 | 97 | |
| 第二类 | 鳃、性腺 | 红色杆菌 Erythrobacter sp. | 99.71 | 99 |
| 足、消化腺 | 鲁杰氏菌 Ruegeria sp. | 99.71 | 99 | |
| 鳃、性腺 | 弧菌 Vibrio sp. | 100.00 | 99 | |
| 消化腺 | 巨型弧菌 Vibrio gigantis | 99.93 | 100 | |
| 鳃、性腺 | 蜡样芽孢杆菌 Bacillus cereus | 99.66 | 99 | |
| 鳃 | 假交替单胞菌 Pseudoalteromonas | 99.58 | 98 |
结合上文图 7可发现, 在厚壳贻贝生长海域水体中检测分析得到的假交替单胞菌属 Pseudoalteromonas, 黏着杆菌属Tenacibaculum sp.以及弧菌属Vibrio sp.均在厚壳贻贝体内组织中检测到, 且这三类菌属都出现于厚壳贻贝的鳃组织中。
3 讨论 3.1 厚壳贻贝养殖环境及其体内微生物群落多样性分析嵊泗海域总体微生物丰富度大于东极海域。嵊泗海域不同采样点微生物丰富度由大到小分别为wild.10m > culture.10m > culture.5m > wild.5m, 其中嵊泗海域野生生长区10 m水深微生物丰富度最高, 野生生长区5 m水深微生物丰富度最低; 东极海域不同采样点微生物丰富度由大到小分别为culture.10m > wild.10m > culture.5m > wild.5m, 东极海域养殖区10 m水深微生物丰富度最高, 野生生长区10 m水深微生物丰富度最低。
从属分类水平来看, 东极海域的优势微生物群落主要为厌氧氨氧化菌属Candidatus_Portiera、海洋氨氧化古菌属Nitrosopumilus、Fluviicola、蓝细菌聚球藻属Synechococcus以及弧菌属Vibrio; 嵊泗海域的优势微生物群落主要为十八杆菌属Octadecabacter、假交替单胞菌属Pseudoalteromonas, 厌氧氨氧化菌属Candidatus_Portiera、ZA3312c、沉积物陆丹氏菌属Loktanella以及弧菌属Vibrio。
总体上, 东极海域4个取样点的微生物群落相似度比嵊泗海域高。由于受到厚壳贻贝长期选择性摄食的影响, 东极和嵊泗两个海域的野生生长区5m和10m之间的微生物群落相似度明显低于养殖区5m和10m的微生物群落相似度, 且这种差异在嵊泗海域尤为明显。这可能是受嵊泗近海海域厚壳贻贝的大面积养殖的影响。从已有的文献中可知(王芳等, 1998; 刘绪博, 2011; 周曦杰, 2014), 包括贻贝在内的许多双壳贝类有着选择性滤食的习性, 即这些贝类会优先选择摄食水体中的一类浮游生物, 因而导致该区域水体中浮游生物种类相对单一, 相似度较高。相比于嵊泗海域, 东极海域4个取样点的微生物群落相似度较高, 而东极近海海域厚壳贻贝的养殖规模远不如嵊泗海域大, 因此近海微生物群落受厚壳贻贝的摄食影响较小, 和野生生长区微生物群落相比, 相似度就更高。
Vezzulli等人对位于意大利利古里亚海的地中海贻贝养殖场的水体微生物群落进行了分析, 发现弧菌属和假交替单胞菌属是该海域的主要优势微生物(Vezzulli et al, 2018), 这与本文嵊泗海域的主要优势微生物结果相一致。而东极海域也发现假交替单胞菌这一种优势微生物, 同时也间接印证了东极海域的微生物丰富度不如嵊泗海域。
在厚壳贻贝生长海域以及厚壳贻贝体内组织都鉴定到了假交替单胞菌属Pseudoalteromonas、黏着杆菌属Tenacibaculum sp.以及弧菌属Vibrio sp.这三类微生物, 初步推测厚壳贻贝和这些自然水域中存在的微生物产生了某种共生关系, 有报道说海洋双壳贝类会将某些环境中的细菌甚至是致病菌富集在鳃上, 以此到达帮助宿主摄食消化、阻止其他病原体侵入的作用, 这也是贻贝不易染病的原因之一(Yu et al, 2019)。相似的研究在紫贻贝中也有报道, 通过对土耳其伊兹密尔沿海生长的紫贻贝体内微生物进行宏基因组分析, 结果发现紫贻贝中也存在弧菌、变形杆菌、摩根氏菌等在内的34种菌属(Bozcal et al, 2020)。
3.2 贻贝及其共生微生物的独特适应性无论是厚壳贻贝生长海域, 还是其体内均分离鉴定到大量弧菌。而之前的报道显示在紫贻贝体内也大量存在弧菌(Bozcal et al, 2020)。其中弧菌是水产养殖中常见的致病菌, 在紫贻贝和厚壳贻贝体内都存在大量的弧菌, 说明贻贝对弧菌具有特殊的适应性, 其特殊的适应机制和病原识别模式还有待于进一步研究。目前关于弧菌和宿主的共生已有一些研究, 例如有报道显示在海洋鱼类中, 弧菌会在进入宿主肠道系统后, 通过其Ⅵ型分泌系统分泌一些效应分子, 刺激宿主肠道产生收缩使得宿主肠道内原有的常驻菌群被迫排出, 最终弧菌在宿主肠道微生物群落中占据主导地位, 然而在这一肠道微生物群落更替变化中似乎并不会对宿主的健康产生损害, 弧菌本身似乎也不受肠道运动增强的影响(Logan et al, 2018); 而Vezzulli等人认为贻贝体内的弧菌与其宿主经历了一段较为漫长的共同进化史(Vezzulli et al, 2018); Septer等人的研究表明, 夏威夷鱿鱼在孵化后的数个小时内就会被费氏弧菌所感染, 这种感染将逐渐演变成一种共生, 并且持续至宿主的一生, 而这种共生关系和夏威夷鱿鱼的发光器官有直接的联系(Septer, 2019)。
4 结论总的来说, 目前对于海洋双壳贝类共生菌的报道尚比较匮乏, 其识别微生物的机理及共生机制还尚不完全明确, 本文从厚壳贻贝体内分离得到的微生物是否和厚壳贻贝发生了共生关系, 亦或是对厚壳贻贝的某些生理功能产生影响等问题, 还需进一步探索。
王芳, 董双林, 范瑞青, 等. 1998. 四种滤食性贝类滤食器官鳃的扫描电镜观察. 青岛海洋大学学报, 28(2): 240-244 |
尹洁慧, 2013.獐子岛海域浮游动物群落时空变化特征及其与环境因子之间的关系.青岛: 中国科学院研究生院(海洋研究所)博士学位论文
|
叶莹莹, 徐梅英, 吴常文. 2011. 几种环境因子对厚壳贻贝浮游幼虫生长与存活的影响. 浙江海洋学院学报(自然科学版), 30(4): 292-296 DOI:10.3969/j.issn.1008-830X.2011.04.003 |
朱雨瑞, 徐继林, 严小军. 2010. 5种微藻对4种滩涂贝类稚贝生长的影响. 海洋学研究, 28(3): 60-66 DOI:10.3969/j.issn.1001-909X.2010.03.008 |
刘绪博, 2011.双壳贝类滤食对浮游植物——沉水植物种间关系的影响.武汉: 华中农业大学硕士学位论文
|
杨娜, 2018.厚壳贻贝肠道细菌群落组成及肠道细菌对稚贝附着的作用研究.上海: 上海海洋大学硕士学位论文
|
陈瑜, 金雷, 朱敬萍, 等. 2016. 双壳贝类微生物富集能力及环境水体对其影响分析. 山东化工, 45(17): 13-14, 17 DOI:10.3969/j.issn.1008-021X.2016.17.006 |
陈雷, 许传才, 刘海映, 等. 2013. 葫芦岛贝类养殖区浮游生物的群落结构和时空分布. 大连海洋大学学报, 28(2): 199-205 DOI:10.3969/j.issn.2095-1388.2013.02.017 |
林永添. 2007. 紫贻贝(Mytilus edulis Linnaeu)大量死亡原因的初步研究. 现代渔业信息, 22(3): 26-28 DOI:10.3969/j.issn.1004-8340.2007.03.009 |
金雷, 陈雪昌, 陈瑜, 等. 2015. 乐清湾滩涂养殖贝类微生物富集及海水净化研究. 安徽农业科学, 43(32): 80-81, 84 DOI:10.3969/j.issn.0517-6611.2015.32.027 |
周曦杰, 2014.枸杞岛典型生境螺贝类代表种——角蝾螺、紫贻贝摄食生态初步研究.上海: 上海海洋大学硕士学位论文
|
赵立君, 刘云根, 王妍, 等. 2019. 砷污染湖滨湿地底泥微生物群落结构及多样性. 中国环境科学, 39(9): 3933-3940 DOI:10.3969/j.issn.1000-6923.2019.09.040 |
徐晴晴, 赵晟. 2018. 海水盐度对厚壳贻贝呼吸和钙化的影响. 浙江海洋大学学报(自然科学版), 37(5): 400-404 DOI:10.3969/j.issn.1008-830X.2018.05.004 |
梁箫, 童欢, 彭莉华, 等. 2020. 纤维素对海洋细菌生物被膜形成及厚壳贻贝幼虫附着变态的调控. 大连海洋大学学报, 35(1): 75-82 |
Avershina E, Frisli T, Rudi K, 2013. De novo semi-alignment of 16S rRNA gene sequences for deep phylogenetic characterization of next generation sequencing data. Microbes and Environments, 28(2): 211-216 DOI:10.1264/jsme2.ME12157 |
Bozcal E, Dagdeviren M, 2020. Bacterial metagenome analysis of Mytilus galloprovincialis collected from Istanbul and Izmir coastal stations of Turkey. Environmental Monitoring and Assessment, 192(3): 186 DOI:10.1007/s10661-020-8129-1 |
Fogel J, Kelly T, Charnigo R et al, 2015. Predictors of the response to oral 9-THC in regular cannabis users:focus on sex differences. Drug and Alcohol Dependence, 146: e279-e280 |
Jolliffe I T, 1986. Principal component analysis. Journal of Marketing Research, 87(100): 513 |
Logan S L, Thomas J, Yan J Y et al, 2018. The Vibrio cholerae type Ⅵ secretion system can modulate host intestinal mechanics to displace gut bacterial symbionts. Proceedings of the National Academy of Sciences of the United States of America, 115(16): E3779-E3787 DOI:10.1073/pnas.1720133115 |
Magoc T, Salzberg S L, 2011. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 27(21): 2957-2963 DOI:10.1093/bioinformatics/btr507 |
Schloss PD, Westcott SL, Ryabin T et al, 2009. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 75(23): 7537-7541 DOI:10.1128/AEM.01541-09 |
Septer A N, 2019. The Vibrio-squid symbiosis as a model for studying interbacterial competition. mSystems, 4(3): e00108-19 |
Vezzulli L, Stagnaro L, Grande C et al, 2018. Comparative 16S rDNA gene-based microbiota profiles of the Pacific oyster (Crassostrea gigas) and the Mediterranean mussel (Mytilus galloprovincialis) from a shellfish farm (Ligurian Sea, Italy). Microbial Ecology, 75(2): 495-504 DOI:10.1007/s00248-017-1051-6 |
Wang Q, Garrity G M, Tiedje J M et al, 2007. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 73(16): 5261-5267 DOI:10.1128/AEM.00062-07 |
Yu J J, Wang M X, Liu B Z et al, 2019. Gill symbionts of the cold-seep mussel Bathymodiolus platifrons:composition, environmental dependency and immune control. Fish & Shellfish Immunology, 86: 246-252 |
 2021, Vol. 52
2021, Vol. 52


