中国海洋湖沼学会主办。
文章信息
- 陈楠生. 2021.
- CHEN Nan-Sheng. 2021.
- 海洋生物多样性中心形成与演变机制的比较基因组研究进展
- ADVANCES IN COMPARATIVE GENOMICS ANALYSIS OF MECHANISMS UNDERLYING THE FORMATION AND EVOLUTOIN OF MARINE BIODIVERSITY
- 海洋与湖沼, 52(2): 274-286
- Oceanologia et Limnologia Sinica, 52(2): 274-286.
- http://dx.doi.org/10.11693/hyhz20201200344
文章历史
-
收稿日期:2020-12-29
收修改稿日期:2021-01-23
2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室 青岛 266237;
3. 中国科学院海洋大科学研究中心 青岛 266071
2. Functional Laboratory for Marine Ecology and Environmental Science, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
3. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China
创始于19世纪中叶的海洋系统地理学(phylogeography)和海洋生物地理学(biogeography)研究, 囊括了基于物种形态的物种分类、物种地理分布研究和物种遗传关系的探索, 揭示了全球海域基于物种组成划分的若干生物地理区(biogeographic provinces)(Bowen et al, 2016)。针对不同生物地理区域的代表性物种和物种优势度的比较与分析, 发现每一个生物地理区都可能是某些物种的起源中心(center of origin), 并且新物种倾向于往起源中心以外扩散, 而生物地理区只有跟踪物种的起源中心才能够得到清楚解释(Bowen et al, 2016)。20世纪70年代DNA测序技术取得突破性进展, 促使生物地理区研究实现了一个质的飞跃, 从根本上改变了我们对生物多样性中心的形成与演变规律的认知, 进一步加强了对生物地理学的认识(Avise et al, 1987)。这个认识转变的一个重要标志是人们对生物多样性的认识从物种水平, 深入到了种下水平, 即遗传多样性(genetic diversity)(Bowen et al, 2016)。
研究表明, 在过去约50 Ma间, 海洋生物多样性中心发生了三次重大迁移, 从始新世(Eocene)时期的西南欧洲、西北非洲、阿拉伯半岛、巴基斯坦和西印度迁移到晚始新世(Late Eocene)时期的阿拉伯中东地区; 随着阿拉伯生物多样性中心的消失, 新的生物多样性中心于20 Ma前的早中新世(Early Miocene)再次迁移, 到达现在的印太交汇区(Indo-Pacific Convergence Region), 几乎跨越了半个地球; 迁移的时间和地点与主要地球板块事件高度吻合, 显示地球板块事件在生物多样性中心迁移过程中的重要性(Renema et al, 2008)。现在的海洋生物多样性中心位于印太交汇区, 包括位于菲律宾、印度尼西亚和新几内亚之间的珊瑚大三角(the Coral Triangle), 及其周围的特有物种热点海域(endemism hotspots)(Miller et al, 2018)。这些特有物种热点海域包括位于印太交汇区边缘的夏威夷和红海。在过去约两千万年间, 珊瑚大三角一直具有稳定的珊瑚礁, 而这些稳定的珊瑚礁则是形成和传播新物种的关键(Pellissier et al, 2014)。
本文从生态、遗传和分子三个层面综述海洋生物多样性中心形成与演变的机理, 重点介绍基于比较基因组分析方法研究生物多样性中心形成与演变的研究进展, 并对存在的问题和研究方向进行展望。
1 印太交汇区及其生物多样性概述生物多样性(biodiversity)既包括物种多样性, 也包括物种内的多样性, 即遗传多样性。许多物种在同一地域群体(sympatric population)内显示出很高的遗传多样性。印太交汇生物多样性中心最显著的特征是其丰富的珊瑚(Bellwood et al, 2009; DeVantier et al, 2020)、岛礁鱼类(Cowman et al, 2017; Miller et al, 2018; Barth et al, 2019; Pinheiro et al, 2019; Limmon et al, 2020)和贝类。除此之外, 印太交汇区的藻类(Jorde et al, 2018)和海胆也显示出很高的生物多样性(Moore et al, 2019)。有关生物多样性的研究进展和研究成果受研究方法的影响很大, 基于形态的物种分类虽然揭示了印太交汇区显著的生物多样性, 但是同时可能也严重低估了生物多样性。分子研究手段的引入不仅揭示了更高的生物多样性, 而且揭示了更加复杂的动态分布, 以及不同生境之间的连通性(Torres et al, 2018)。分子研究方法的广泛使用揭示了不同海域珊瑚的生物多样性, 包括我国南海深海珊瑚(Deltocyathus magnificus)(Liu et al, 2020b)、我国台湾Pocillopora属珊瑚(Palmas et al, 2018)、我国南海和新加坡海域Micromussa amakusensis珊瑚(Ng et al, 2019)、日本海域珊瑚(Liu et al, 2020a)、印度尼西亚珊瑚(Wijayanti et al, 2018)和澳大利亚珊瑚(Underwood et al, 2018)。
生物多样性分子分析的一个突破性进展是隐种(cryptic species)的发现(Pfenninger et al, 2007)。几乎所有珊瑚物种都存在隐种(Warner et al, 2015; Rosser et al, 2017; Kang et al, 2020; Wepfer et al, 2020)。对珊瑚隐种的准确分析和判断深化了遗传多样性研究(Sheets et al, 2018)。分子分析方法的引入也提高了大家对物种的分类水平和对物种边界的认识, 推动了初级物种假说(Primary Species Hypothesis, PSH)和次级物种假说(Secondary Species Hypothesis, SSH)的提出(Pante et al, 2015)(图 1)。印太交汇区Pocillopora属具有很高的生物多样性和大量隐种, 不仅体现了初级物种假说和次级物种假说的思想, 也反映了相应研究思路的重要性(Gélin et al, 2017)。
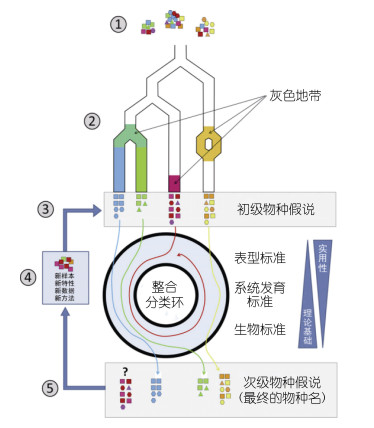 |
| 图 1 整合分类环: 物种假设包括初级物种假设和次级物种假设(改自Pante et al, 2015) Fig. 1 Integrative taxonomy loop: The species hypotheses includes primary species hypothesis and secondary species hypothesis (modified from Pante et al, 2015) 注: ①群体采样; ②采集到的样本可能具有较大的分化; ③基于形态和分子依据的初级物种假设; ④对初级物种假设进行评估; ⑤由初级物种假设到次级物种假设 |
研究表明, 不仅珊瑚本身具有很高的生物多样性, 珊瑚的共生微生物也具有很高的生物多样性(Glasl et al, 2020)。比如印太交汇区虫黄藻具有很高的遗传多样性(Wham et al, 2016), 大堡礁珊瑚的共生微生物也具有显著的多样性和适应性(van Oppen et al, 2018)。
印太交汇区鱼类生物多样性尤其高(Cowman et al, 2017; Miller et al, 2018), 其中珊瑚大三角海域迄今得到描述的海洋鱼类物种就有4350种(Limmon et al, 2020), 以大型岛礁鱼类为主, 有相当大一部分的小型岛礁鱼类(cryptobenthic reef fishes)尺寸小于50 mm(Brandl et al, 2018)。分子标记分析表明, 还有更多的鱼类物种有待进一步鉴定和发现(Cowman, 2014), 因此鱼类多样性可能是被严重低估的。的确, 自2010年起印太交汇区岛礁鱼类平均每年都有51.3个新种的报导(Allen, 2015)。珊瑚岛礁鱼类与珊瑚的进化具有一定的相互依赖性和相似性(Bellwood et al, 2017)。在古新世(Paleocene)和始新世(Eocene), 即66—34 Ma, 绝大多数的现代鱼类和珊瑚类物种已经形成, 功能形态性状也已经相当丰富。与此相比, 在渐新世(Oligocene)和第三纪中新世(Miocene), 即34—5.3 Ma, 鱼类和珊瑚类物种显示出快速分化。在此期间, 岛礁面积增加, 生境扩张, 鱼类丰度也有增加, 现代珊瑚岛礁获得了很多功能性性状。很多物种形成于最近的5.3 Ma, 即上新世-更新世(Plio-Pleistocene), 其中鱼类物种的形成时间为5.3 Ma, 珊瑚为1.9 Ma (Bellwood et al, 2017)。值得一提的是, 这些不同时期的鱼类和珊瑚性状的区别仅仅局限于颜色或微小形态方面, 物种之间的差异不显著。
印太交汇区腹足类具有很高的生物多样性, 主要驱动因素是较高的新物种形成速度(Frey et al, 2008)。腹足类新物种形成机制也逐步得到揭示。比如最近研究表明, 适应性辐射促进了腹足类生物多样性的提升(Layton et al, 2018)。近年来, 多个海域报道了海蛞蝓的生物多样性, 包括新加坡海域(Chan et al, 2019)、澳大利亚和塔斯曼海海域(Nimbs et al, 2018), 以及印度尼西亚海域海(Eisenbarth et al, 2018; Undap et al, 2019; Papu et al, 2020)。另外, 我国海南岛砗磲也具有较高的多样性, 包括很多隐种(Liu et al, 2020a)。
2 海洋生物多样性中心形成与演变的生态机理生态学的一个核心目标是揭示地球上特异性分布格局(Mora et al, 2003)。进化论先驱者达尔文(Charles Darwin)和华莱士(Alfred Russel Wallace)的工作为生物多样性中心形成与演变奠定了基本框架。在过去70年间, 尽管针对物种形成的地理位置和驱动生物多样性中心形成的过程尚未达成共识(Cowman, 2014), 大量基于形态学特征的分类研究仍产生了多个有关印太交汇区生物多样性中心的形成和演变机理的假说。从生态层面分析, 印太交汇区生物多样性中心的形成是物种类型、生态、环境、地质与水动力等多因素、综合驱动下的结果, 可能受到了多个相互独立的机理驱动(Bellwood et al, 2009)。因此, 生物多样性中心(center of biological diversity)并不等同于新物种形成中心(center of origin)。海水鱼类的多样性在赤道附近高于两极, 以印太交汇区珊瑚大三角的多样性水平最高; 而海水鱼类新物种形成的速度在两极却高于赤道附近海域(图 2), 其与海水鱼类物种的多样性以及海水温度成反比(Rabosky et al, 2018)。
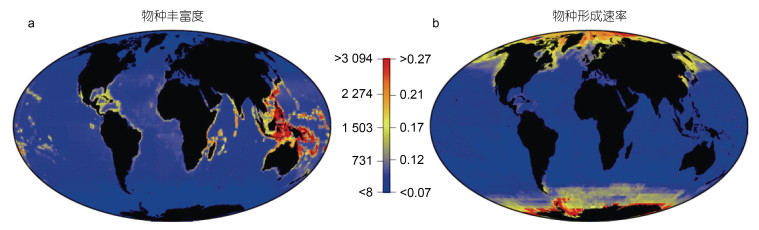 |
| 图 2 海水鱼的生物多样性与物种形成速度的维度梯度(改自Rabosky et al, 2018) Fig. 2 Latitudinal distribution patterns of species diversity and speciation rate in marine fishes (modified from Rabosky et al, 2018) 注: a. 海水鱼的多样性分布格局; b. 海水鱼新物种形成速度 |
新物种形成中心只是生物多样性中心形成的机制之一, 而印太交汇区被认为是多种不同机制共同作用的结果, 显示了多个机制的叠加效应。比如, 菲律宾近岸海域鱼类被认为是鱼类生物多样性中心的中心(center of the center), 而所有这些假说的共同作用可能驱动了这个中心的形成(Carpenter et al, 2005)。目前, 不同生态机理对印太交汇区生物多样性中心形成的相对贡献并不清楚, 而比较基因组学分析可以追溯不同机理的相对贡献。比如, 通过分子标记cox2-3序列分析印太交汇区软粒藻属(Portieria)红藻物种多样性的驱动力和生物地理学过程中发现, 物种形成中心假说(center of origin hypothesis)和物种汇聚假说(center of accumulation hypothesis)两种机制的共同作用, 而非单个机制的独立作用, 解释了软粒藻属物种的生物多样性分布格局(Leliaert et al, 2018)。通过比较分析cox1、16S rDNA和28S rDNA等通用分子标记基因的多样性, Eilertsen等(2015)揭示了深海水手螺(Scaphander)的物种分化和新物种起源机理。通过比较分析通用分子标记cox1、组蛋白基因H3、线粒体基因16S rDNA、核糖体基因18S rDNA和28S rDNA基因, Rodriguez-Flores等(2020)揭示了深海铠甲蟹(squat lobster)的生殖隔离以及与浅海铠甲蟹之间的连通性。
针对全球海洋生物多样性中心形成的理论探究, 在生态层面的主要假说主要包括物种形成中心假说、物种汇聚中心假说、物种重叠中心假说(center of overlap hypothesis)、物种保存中心假说(center of survival hypothesis), 以及物种扩散中心(center of expansion hypothesis)假说(图 3)。
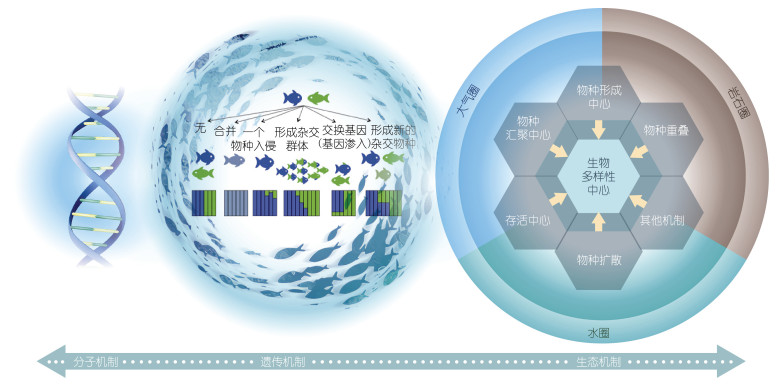 |
| 图 3 生物多样性中心形成与演变的生态、遗传和分子机制(改自Runemark et al, 2019) Fig. 3 Schematic illustration of ecological, genetic, and molecular mechanisms underlying the formation and evolution of the Biodiversity Center in the Indo-Pacific Convergence Region (modified from Runemark et al, 2019) |
新物种形成中心假说的核心思想是新物种在新物种形成中心不断形成并逐步扩散到周边的其他海域(Briggs, 2003)。根据这个模型, 高特异性生境中的强烈竞争导致具有更高适应性的新物种的形成(Bowen et al, 2013)。印太交汇区新物种形成中心形成的岛礁鱼类物种逐步出现在周边的群岛(Mora et al, 2003)。印太交汇区新物种形成始于20—12 Ma以前的中新世, 生物多样性输出始于7 Ma前, 一直持续到现在, 覆盖了上新世(Pliocene)、更新世(Pleistocene)和全新世(Holocene)(Bowen et al, 2013)。新物种形成中心假说的直接推论是位于新物种形成中心的物种比较年轻, 而位于新物种形成中心周边的物种比较古老(Bowen et al, 2013; Huang et al, 2018)。物种形成中心通常也是物种扩散中心, 从生物多样性中心扩散出去的物种, 可能在获得新的变异以后返回到生物多样性中心, 进一步丰富了生物多样性中心。
物种汇聚中心假说认为新物种在生物多样性中心的周边群岛等海域形成, 逐步随着海流汇聚到生物多样性中心(Budd et al, 2010)。研究表明珊瑚大三角中的造礁珊瑚生物绝大多数来源于其他海域, 通过物种扩张到达珊瑚大三角进行累积, 表明地理范围移动(geographic range shifts)是珊瑚大三角造礁珊瑚生物多样性中心形成的主要原因(Huang et al, 2018)。
物种重叠中心假说首先由美国科学家Woodland (1983)于1983年提出。比如, 蓝子鱼科(Siganidae)鱼类有印度洋和西太平洋两个特有物种分布热点(areas of endemism)。Woodland认为篮子鱼科在东印度群岛(East Indies)海域的高物种多样性缘于印度洋和西太平洋这两个特有物种分布热点的重叠。
物种保存中心假设, 认为印太交汇区的物种灭亡速度低于周围海域(Barber et al, 2005)。比如, 珊瑚礁海猪鱼(Halichoeres)在印太交汇区的生物多样性很高。通过对cox1、16S rDNA和12S rDNA三个分子标记线粒体基因分析表明, 印太交汇区与新世界(New World)的海猪鱼显示出很大的特异性。两个不同海域的海猪鱼相距21.2—18.1 Ma年, 表明二者都是特提斯海(Tethys)的后代。这个分析表明印太交汇区鱼类的生物多样性可能以物种保存中心机制为主, 但是也不能排除具有多机制起源。
随着基于分子标记和基因组信息的生物多样性研究的深入, 有关生物多样性中心形成与演变的认识也将逐步深入。最近, Hu等(2021)利用三个分子标记对我国陆地野生生物的比较研究揭示了它们的地理分布格局, 发现它们在我国南部和西南部具有更高的多样性, 并探讨了驱动分布格局的生态因子, 发现包括降雨和气温在内的气候因子起重要作用。类似规模的针对印太交汇区海域生物多样性的研究将揭示高分辨率的生物多样性格局及其动态变化过程。
3 生物多样性起源与演变的遗传机理研究整体来讲, 生态机制为生物多样性中心的形成与演变机制提供了一个平台。生态机制可以说是生物多样性中心形成的第一驱动力。生物多样性中心初步形成以后, 物种间或物种内杂交(hybridization)驱使生物多样性发生下一个层次的变化。杂交在物种演化和多样性形成过程中起关键作用(Marques et al, 2019)。新物种起源就是杂交可育自然种群与其他类似种群产生生殖隔离的过程(Marques et al, 2019)。杂交可以把种群中已经存在很长时间的古老的突变组合起来形成新物种, 并适应新的环境, 这些驱动新物种形成突变的过程经常比物种分离时间更加古老, 而这些古老突变的组合可以驱动新物种的快速形成(Marques et al, 2019)。由杂交形成新物种的机制也被称为杂交起源机制(hybrid speciation)(Mallet, 2007)。根据迈尔(Ernst Mayr)和杜布赞斯基(Theodosius Dobzhansky)的观点, 生殖隔离(reproductive barrier)是逐步形成的, 起因是在位于不同地理位置的种群的互作基因中累积了突变, 即贝特森–杜步赞斯基–穆勒不相容(BDMIs)。因此, 物种起源是在种群生殖隔离条件下产生的(Kulmuni et al, 2020), 生殖隔离使不同种群在邻近地理区域中维持其基因型和表型的特异性(Seehausen et al, 2014)。
适应性辐射(adaptive radiation)是生态和进化共同作用的结果(Gillespie et al, 2020)。适应性辐射的基础是突变, 但是由于突变过程比较慢, 因此通过适应性辐射形成新物种的速度也比较慢(Wagner et al, 2012; Stroud et al, 2016)。适应性辐射包括三个必需要素(Gillespie et al, 2020): 第一个必需要素是生态机会(ecological opportunity), 生态机会可能以多种形式出现, 比如一个物种密度低或者没有被充分利用的生态环境, 某种新颖性状使得物种可以获得一个独特的与环境互作的能力, 或者导致该优势物种的灭亡; 第二个必需要素是时间, 适应性辐射在物种获得生态机会的初期往往比较快, 随着生境的逐步饱和, 适应性辐射的速度会逐步慢下来; 适应性辐射的第三个必需要素是适应性响应(adaptive response), 在进化过程中, 辐射可以是适应性的, 也可以是非适应性(nonadaptive)的, 即表现出很弱或者没有对生态位的适应性。
通过基因渗入(introgression)形成新物种的时间往往比较漫长(Hedrick, 2013; Poelstra et al, 2018)。基因渗入促进同域性物种形成(sympatric speciation) (Bolnick et al, 2007)。根据物种组成, 印太交汇区可以划分为至少6个生物地理区(biogeographic provinces)(He et al, 2019)。生物地理区的缝合区(suture zones)形成杂交带(hybrid zone), 进化关系较近, 还没有形成完全生殖隔离的物种可能发生“第二次接触”(secondary contact), 导致物种间杂交(Mallet, 2007)。物种间杂交可能导致多种不同结果, 包括: (1) 杂交后代可能具有生殖隔离; (2) 可能导致基因渗入, 并逆转物种分化; (3) 产生新物种和辐射事件(He et al, 2019)。印太交汇区存在两个公认的缝合区, 位于东印度洋的圣诞和科科斯/基林岛(Christmas and Cocos/Keeling Islands), 以及位于西印度洋的索科特拉群岛(Socotra Archipelago)。这些缝合区是印度洋、太平洋以及红海的交汇区, 是研究杂交和基因渗入如何引起新物种形成的一个理想海域(He et al, 2019)。
在岛礁生态系统中, 鹿角珊瑚属内的不同物种之间存在广泛杂交(即网状进化, reticulate evolution), 并导致基因渗入和多倍体的产生, 形成大量的共配群(syngameon)(Mao, 2020)(图 4)。鹿角珊瑚是珊瑚中分布最为广泛的一个属, 且具有很多物种, 广泛分布在印太交汇区和加勒比海, 是研究物种起源与演化的理想研究对象(van Oppen et al, 2001)。其中有些物种分布范围比较广, 另外一些物种分布范围则比较窄, 有70余物种的分布区域重叠。通过对5个鹿角珊瑚物种的全基因组比较, 发现了一个基因渗入事件, 以及多个独立的基因流事件(Mao et al, 2018)。除珊瑚外, 在鱼类(Harrison et al, 2017; He et al, 2019)和硅藻(De Decker et al, 2018)中也发现了杂交和基因渗入现象。
如果说生态机制作为生物多样性中心形成的第一驱动力, 为生物多样性的形成与演变提供了平台, 遗传机制则可以被认为是生物多样性中心的形成与演变的第二驱动力, 为遗传多样性的形成与演变提供了可能性。在此基础上, 分子机制可以被认为是生物多样性中心形成与演变的最关键的一步。在比较基因组技术被用于研究物种形成和演化的过程中, 逐步形成了一门新兴学科——物种起源基因组学(speciation genomics)(Marques et al, 2019)。理解物种起源的关键是寻找触发物种形成的突变和基因, 即物种起源基因(speciation gene), 并跟踪这些基因的形成时间, 由此推测物种起源时间(Ravinet et al, 2017)。1953年沃森(James Watson)和克里克(Francis Crick)提出的DNA双螺旋模型(DNA double helix model)奠定了分子生物学的基础, 近20年来, 随着DNA测序通量技术的迅速提升, 成本急剧下降, 测序质量和长度逐步提升, 比较基因组技术得以被广泛应用于解析物种起源的分子机制。因此, 研究生物多样性中心形成与演变已经逐步成为一个活跃的研究领域。
4.1 新基因、新变异、新剪切、新调控基因的变化, 包括新基因的形成、基因变异、基因的剪切变化和转录调控变异都可以驱动物种演化。比如, 达尔文雀(Darwin's finches)物种之间的一个显著差异是它们的喙形态。通过比较分析具有不同喙形态的物种发现基因ALX1变异控制喙形态(Lamichhaney et al, 2015)。这个基因编码一个包括同源异型域(homeodomain)的蛋白质, 在面颅间质(craniofacial mesenchyme)、第一鳃弧(first branchial arch)和肢芽(limb bud)发育过程中起关键作用(Uz et al, 2010); 另一个则包括与环境相互作用的基因如免疫相关基因在进化过程中起关键作用。
非洲慈鲷的演化受感觉驱动力(sensory drive)的影响, 即感觉驱动力可以导致颜色多样性, 并且导致新物种形成(Seehausen et al, 2008)。在非洲慈鲷物种中总共发现了8个视觉色素基因, 但是在每一个个体中仅有4个视觉色素基因。这些基因具有很高的变异, 特别是长波长敏感性视蛋白基因LWS。非洲维多利亚湖雌性慈鲷对雄性颜色具有很强的选择, 这种选择导致了新物种的形成(Seehausen et al, 2008), 而这类新物种形成不依赖地理隔离。比如, 维多利亚湖的两种慈鲷鱼Pundamilia pundamilia和Pundamilia nyererei的地理分布完全相同, 但它们的颜色表型完全不同, P. pundamilia的雄性具有蓝灰色体色, 而P. nyererei的雄性则是腹部为深红色, 身体其他部位为黄色。最终, 雄性的不同体色促成了生殖隔离。基因LWS的变异位点影响了雄性体色, 并且突变方式与个体在水体中的分布密切相关。
三刺鱼(threespine stickleback, Gasterosteus aculeatus)是一种著名的用于研究进化的模式动物(Nelson et al, 2018)。典型的三刺鱼生活在海洋环境, 具有用于自卫的长刺。而生活在淡水中的三刺鱼由海水物种进化而来, 他们的刺却比较短, 这些性状的变化可能通过基因的突变实现。其中, 有一些是通过基因转录调控元件的变化实现, 比如转录因子基因Pitx1的顺式调控元件突变导致了腹刺消失(Shapiro et al, 2004)。另外一些则是通过基因的剪切变异实现, 比如转录因子基因MSX2A的一个剪切变异导致了背刺缩短(Howes et al, 2017)。
4.2 染色体反转事件(chromosomal inversion)和超级基因(supergene)染色体反转事件可以降低染色体重组(Kirkpatrick, 2010), 从而阻止等位基因的互换, 增加物种的适应性, 促进生物多样性增加(Mérot, 2020)。具体机理包括如下几个方面(Faria et al, 2019): 首先, 染色体反转可以促进累积更多突变, 从而加大生殖隔离位点(barrier locus)的范围; 其次, 阻止物种之间的二次接触, 促进累积更多的突变, 形成更强的生殖隔离; 最后, 保护局部适应性基因结构, 即保护有利于生殖隔离的等位基因组合。
比较基因组分析发现染色体反转广泛存在。比如三刺鱼基因组中存在大量的染色体反转事件(Jones et al, 2012)。单核苷酸多样性(SNP)分析发现潮间带岩石玉黍螺(Littorina saxatilis)不同株系的基因组中存在多个反转(Westram et al, 2018; Faria et al, 2019; Kess et al, 2019)。最近, 通过群体遗传学分析发现了刀鲚(Coilia nasus)对环境的适应机制, 染色体倒位是刀鲚适应与分化的关键分子机制(Zong et al, 2020)。
通过构建全基因组, 可以系统挖掘染色体反转事件。不过早期的基因组组装的contig偏短, 往往只能够鉴定出小尺度反转事件, 而不能够用于确定大尺度染色体反转事件。大西洋鲱鱼(Clupea harengus)是全球最丰富的脊椎动物, 也是一个用于研究物种适应的模式动物(Pettersson et al, 2019)。利用最新的PacBio三代测序技术和Hi-C分析技术, 结合遗传图谱构建了大西洋鲱鱼高质量参考基因组, 发现共有16条染色体, 全长726 Mb。针对1170个大西洋鲱鱼个体的45000个SNP的连锁不平衡(LD)分析发现, 其第12号染色体上有一个7.8 Mb长的反转, 这个反转对大西洋鲱鱼的生态适应性起关键作用, 也是区分不同种群的重要遗传标记(Pettersson et al, 2019)。适应性研究表明, 这个7.8 Mb的反转是一个超级基因(Kirkpatrick, 2010; Pettersson et al, 2019)。超级基因是染色体上两个或多个基因形成的基因簇, 也被称为“共适应基因复合体(co-adapted gene complexes)”(Schwander et al, 2014)。大西洋鲱鱼的这个超级基因包括225个蛋白编码基因(Pettersson et al, 2019)。除此之外, 位于反转序列附近但是位于反转以外的10个基因可能也起到了关键作用(Pettersson et al, 2019)。
染色体反转可能对性染色体起源至关重要(Kirkpatrick, 2010)(图 5)。性别对抗性选择(sex-antagonistic selection)指不同的等位基因具有性别特异性, 并且这个选择倾向于降低性别决定基因的重组。在进化过程中, 包括性别决定基因的染色体反转会逐步扩张。
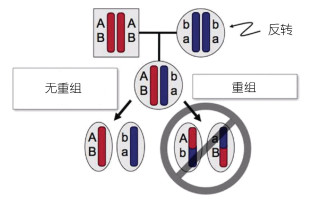 |
| 图 5 杂合染色体反转抑制重组(改自Kirkpatrick, 2010) Fig. 5 Heterozygous inversion suppresses recombination (modified from Kirkpatrick, 2010) |
除了染色体反转, 其他类型的基因组结构变异也可以在物种起源过程中起重要作用。随着基因组技术的普遍应用, 更多的基因组结构变异在物种起源过程中的作用将会得到解释(Mérot et al, 2020)。
4.3 染色体核型变异染色体数目的变异强烈影响生殖隔离, 导致染色体数目变化的机制很多, 包括染色体融合(fusion)和染色体分裂(fission)(de Vos et al, 2020)。研究表明染色体融合或分裂导致的染色体核型变异虽然并不常见, 但是存在(Salzburger, 2018)。比如非洲鲫鱼的染色体数目绝大多数是22 (2n = 44), 美洲鲫鱼的染色体数目绝大多数是24 (2n = 48), 但是, 在新物种形成过程中, 染色体数目发生了变化, 染色体数目从16 (2n = 32)到30 (2n = 60)均有出现(Poletto et al, 2010)。
4.4 多倍体在进化和物种形成过程中, 基因组复制是一个很普遍的现象, 包括同源多倍体(autopolyploid)或异源多倍体(allopolyploid)(Van de Peer et al, 2017)。比较基因组分析发现了大量以前忽视的多倍体现象, 说明基因组复制在物种形成过程中的重要性远超预期(Schumer et al, 2018)。鹿角珊瑚属包括150个物种, 是当今世界上物种最为丰富的一个造礁珊瑚属, 分布范围在31°N和31°S之间的印太交汇区, 其多样性在印太交汇区中部达到最高(Wallace et al, 2006)。尽管诸多证据表明鹿角珊瑚生物多样性缘于基因组多倍化(polyploidization)(Renema et al, 2016), 但比较基因组分析首次证明了基因组加倍驱动了鹿角珊瑚(staghorn coral)的生物多样性形成和分化(Mao et al, 2019; Mao, 2020)。通过对6个鹿角珊瑚物种的比较分析发现, 这些物种的共同祖先在31 Ma前有一个基因组复制(whole genome duplication, WGD)事件, 并且这个复制时间几乎与世界范围内珊瑚物种的大灭亡同时发生(Mao et al, 2019)。基因组复制形成的基因中, 参与基因调节(比如胁迫反应)的基因显著富集。
异源多倍体现象在高等植物中比较常见, 而在动物中少见。鲤鱼(Cyprinus carpio)是一个异源四倍体(allotetraploid), 比较基因组分析发现它的二倍体祖先物种的分化大约于23 Ma年前完成, 而异源四倍体新物种的形成大约于12.4 Ma前完成(Xu et al, 2014, 2019)。鲤鱼基因组分成两个亚基因组(subgenome), 各有25条染色体, 具有很好的共线性关系。与异源多倍体植物基因组相比, 鲤鱼的亚基因组之间的相似性很高, 没有出现亚基因组优势化(subgenome dominance)现象。金鱼(Carassius sp.)也是一个异源四倍体, 与鲤鱼具有相同的祖先, 于大约14 Ma前形成(Chen et al, 2019)。金鱼基因组中的两条亚基因组也具有很高的相似性, 与鲤鱼的亚基因组具有很高的共线性(Chen et al, 2019)。
藻类的异源多倍体现象比较少见。比较基因组分析发现, 产油硅藻Fistulifera solaris是一个异源多倍体, 并且具有很高的杂合度, 表明它是由两个物种杂交而形成的新物种(Tanaka et al, 2015)。由于相关硅藻的基因组信息缺乏, 目前尚没有它的祖先信息。利用基因组技术开展更多的藻类基因组研究将有助于解析藻类生物多样性形成与演变的规律与机制(Caron et al, 2019)。
4.5 其他分子机制除了上述机制, 还有很多分子机制可以驱动新物种的形成。这些分子机制包括基因家族的扩张与收缩, 基因复制与新功能基因(neo-functionization)的形成, 水平基因转移(horizontal gene transfer)与基因岛的形成(Ravinet et al, 2017; Wolf et al, 2017), 基因的转录调控机制的改变, microRNA的形成与分化等。在非洲鲫鱼新物种的共同祖先中, 基因复制速率比其他物种高4—5倍, 表明基因复制速率与物种多样性形成具有正相关关系(Salzburger, 2018)。
5 结论与展望地球上的生物多样性处于永恒的动态变化过程中, 新生物不断涌现, 现生生物也不断灭亡。海洋生物的分布格局受海洋环流的影响, 而海洋环流又同步受地球构造板块的影响, 从而形成特定的海洋生物多样性中心。在过去50 Ma期间, 全球范围内海洋生物多样性中心经历了三次重大迁移, 印太交汇区是一个最为突出的生物多样性中心, 近10 Ma暖池的形成推动了该海域生物多样性中心的形成。通过对印太交汇区生物多样性中心形成与演变的研究, 不仅可以帮助我们理解生物多样性形成与演变的机制, 也可以使我们更加有效地保护生物多样性, 科学地利用生物资源。
海洋生物多样性研究可以归纳为三个层次或三个相互交叉的阶段。第一层次的研究以物种分类和系统学分析为主, 统计不同海域的代表性物种的物种数, 这个阶段的研究以形态学分析为主, 积累了大量样本和数据, 获得了生物多样性生态学方面的认知, 提出了多个有关生物多样性形成的假设, 包括物种形成中心假设、物种汇聚假设、物种重叠假设以及物种保存中心假设。第二个层次的研究是遗传层次的研究, 解析了物种形成的遗传机理, 包括种间和种内杂交以及适应性辐射。由于DNA测序技术的进步, 生物多样性研究进入第三个层次, 即分子层次。比较基因组分析方法是分析生物多样性形成和演变的有效方法, 解释了新物种形成的重要分子机制, 包括新基因的形成、染色体的变化、基因组复制以及超级基因的形成等。随着DNA和基因组测序技术的日趋成熟, 分析成本逐步降低, 生物信息学分析技术不断提升, 生物多样性中心的形成与演变研究正在进入黄金时代, 而重要物种的生物多样性中心的起源和演化机制也必将得以逐步解析。同时, 这些进步也会促进对印太交汇区生物资源的认识、保护和合理开发。
Allen G R, 2015. Review of Indo-Pacific coral reef fish systematics: 1980 to 2014. Ichthyological Research, 62(1): 2-8 DOI:10.1007/s10228-014-0411-1 |
Avise J C, Arnold J, Ball R M et al, 1987. Intraspecific phylogeography: the mitochondrial DNA bridge between population genetics and systematics. Annual Review of Ecology and Systematics, 18: 489-522 DOI:10.1146/annurev.es.18.110187.002421 |
Barber P H, Bellwood D R, 2005. Biodiversity hotspots: evolutionary origins of biodiversity in wrasses (Halichoeres: Labridae) in the Indo-Pacific and new world tropics. Molecular Phylogenetics and Evolution, 35(1): 235-253 DOI:10.1016/j.ympev.2004.10.004 |
Barth J M I, Villegas-Ríos D, Freitas C et al, 2019. Disentangling structural genomic and behavioural barriers in a sea of connectivity. Molecular Ecology, 28(6): 1394-1411 DOI:10.1111/mec.15010 |
Bellwood D R, Goatley C H R, Bellwood O, 2017. The evolution of fishes and corals on reefs: form, function and interdependence. Biological Reviews, 92(2): 878-901 DOI:10.1111/brv.12259 |
Bellwood D R, Meyer C P, 2009. Searching for heat in a marine biodiversity hotspot. Journal of Biogeography, 36(4): 569-576 DOI:10.1111/j.1365-2699.2008.02029.x |
Bolnick D I, Fitzpatrick B M, 2007. Sympatric speciation: models and empirical evidence. Annual Review of Ecology, Evolution, and Systematics, 38: 459-487 DOI:10.1146/annurev.ecolsys.38.091206.095804 |
Bowen B W, Gaither M R, DiBattista J D et al, 2016. Comparative phylogeography of the ocean planet. Proceedings of the National Academy of Sciences of the United States of America, 113(29): 7962-7969 DOI:10.1073/pnas.1602404113 |
Bowen B W, Rocha L A, Toonen R J et al, 2013. The origins of tropical marine biodiversity. Trends in Ecology & Evolution, 28(6): 359-366 |
Brandl S J, Goatley C H R, Bellwood D R et al, 2018. The hidden half: ecology and evolution of cryptobenthic fishes on coral reefs. Biological Reviews, 93(4): 1846-1873 DOI:10.1111/brv.12423 |
Briggs J C, 2003. Marine centres of origin as evolutionary engines. Journal of Biogeography, 30(1): 1-18 DOI:10.1046/j.1365-2699.2003.00810.x |
Budd A F, Pandolfi J M, 2010. Evolutionary novelty is concentrated at the edge of coral species distributions. Science, 328(5985): 1558-1561 DOI:10.1126/science.1188947 |
Caron D A, Hu S K, 2019. Are we overestimating protistan diversity in nature?. Trends in Microbiology, 27(3): 197-205 DOI:10.1016/j.tim.2018.10.009 |
Carpenter K E, Springer V G, 2005. The center of the center of marine shore fish biodiversity: the Philippine Islands. Environmental Biology of Fishes, 72(4): 467-480 DOI:10.1007/s10641-004-3154-4 |
Chan I Z W, Chang J J M, Huang D W et al, 2019. Colour pattern measurements successfully differentiate two cryptic Onchidiidae Rafinesque, 1815 species. Marine Biodiversity, 49(4): 1743-1750 DOI:10.1007/s12526-019-00940-4 |
Chen Z L, Omori Y, Koren S et al, 2019. De novo assembly of the goldfish (Carassius auratus) genome and the evolution of genes after whole-genome duplication. Science Advances, 5(6): eaav0547 DOI:10.1126/sciadv.aav0547 |
Cowman P F, 2014. Historical factors that have shaped the evolution of tropical reef fishes: a review of phylogenies, biogeography, and remaining questions. Frontiers in Genetics, 5: 394 |
Cowman P F, Parravicini V, Kulbicki M et al, 2017. The biogeography of tropical reef fishes: endemism and provinciality through time. Biological Reviews, 92(4): 2112-2130 DOI:10.1111/brv.12323 |
De Decker S, Vanormelingen P, Pinseel E et al, 2018. Incomplete reproductive isolation between genetically distinct sympatric clades of the pennate model diatom Seminavis robusta. Protist, 169(4): 569-583 DOI:10.1016/j.protis.2018.05.003 |
De Palmas S, Soto D, Denis V et al, 2018. Molecular assessment of Pocillopora verrucosa (Scleractinia; Pocilloporidae) distribution along a depth gradient in Ludao, Taiwan. PeerJ, 6: e5797 DOI:10.7717/peerj.5797 |
De Vos J M, Augustijnen H, Bätscher L et al, 2020. Speciation through chromosomal fusion and fission in Lepidoptera. Philosophical Transactions of the Royal Society B: Biological Sciences, 375(1806): 20190539 DOI:10.1098/rstb.2019.0539 |
DeVantier L, Turak E, Szava-Kovats R, 2020. Species richness and abundance of reef-building corals in the Indo-West Pacific: the local-regional relation revisited. Frontiers in Marine Science, 7: 487 DOI:10.3389/fmars.2020.00487 |
Eilertsen M H, Malaquias M A E, 2015. Speciation in the dark: diversification and biogeography of the deep-sea gastropod genus Scaphander in the Atlantic Ocean. Journal of Biogeography, 42(5): 843-855 DOI:10.1111/jbi.12471 |
Eisenbarth J H, Undap N, Papu A et al, 2018. Marine heterobranchia (Gastropoda, Mollusca) in Bunaken National Park, North Sulawesi, Indonesia-a follow-up diversity study. Diversity, 10(4): 127 DOI:10.3390/d10040127 |
Faria R, Chaube P, Morales H E et al, 2019. Multiple chromosomal rearrangements in a hybrid zone between Littorina saxatilis ecotypes. Molecular Ecology, 28(6): 1375-1393 DOI:10.1111/mec.14972 |
Frey M A, Vermeij G J, 2008. Molecular phylogenies and historical biogeography of a circumtropical group of gastropods (Genus: Nerita): implications for regional diversity patterns in the marine tropics. Molecular Phylogenetics and Evolution, 48(3): 1067-1086 DOI:10.1016/j.ympev.2008.05.009 |
Gélin P, Postaire B, Fauvelot C et al, 2017. Reevaluating species number, distribution and endemism of the coral genus Pocillopora Lamarck, 1816 using species delimitation methods and microsatellites. Molecular Phylogenetics and Evolution, 109: 430-446 DOI:10.1016/j.ympev.2017.01.018 |
Gillespie R G, Bennett G M, De Meester L et al, 2020. Comparing adaptive radiations across space, Time, and Taxa. Journal of Heredity, 111(1): 1-20 DOI:10.1093/jhered/esz064 |
Glasl B, Robbins S, Frade P R et al, 2020. Comparative genome-centric analysis reveals seasonal variation in the function of coral reef microbiomes. The ISME Journal, 14(6): 1435-1450 DOI:10.1038/s41396-020-0622-6 |
Harrison H B, Berumen M L, Saenz-Agudelo P et al, 2017. Widespread hybridization and bidirectional introgression in sympatric species of coral reef fish. Molecular Ecology, 26(20): 5692-5704 DOI:10.1111/mec.14279 |
He S, Robitzch V, Hobbs J P A et al, 2019. An examination of introgression and incomplete lineage sorting among three closely related species of chocolate-dipped damselfish (genus: Chromis). Ecology and Evolution, 9(9): 5468-5478 DOI:10.1002/ece3.5142 |
Hedrick P W, 2013. Adaptive introgression in animals: examples and comparison to new mutation and standing variation as sources of adaptive variation. Molecular Ecology, 22(18): 4606-4618 DOI:10.1111/mec.12415 |
Howes T R, Summers B R, Kingsley D M, 2017. Dorsal spine evolution in threespine sticklebacks via a splicing change in MSX2A. BMC Biology, 15(1): 115 DOI:10.1186/s12915-017-0456-5 |
Hu Y B, Fan H Z, Chen Y H et al, 2021. Spatial patterns and conservation of genetic and phylogenetic diversity of wildlife in China. Science Advances, 7(4): eabd5725 DOI:10.1126/sciadv.abd5725 |
Huang D W, Goldberg E E, Chou L M et al, 2018. The origin and evolution of coral species richness in a marine biodiversity hotspot. Evolution, 72(2): 288-302 DOI:10.1111/evo.13402 |
Jones F C, Grabherr M G, Chan Y F et al, 2012. The genomic basis of adaptive evolution in threespine sticklebacks. Nature, 484(7392): 55-61 DOI:10.1038/nature10944 |
Jorde P E, Andersson A, Ryman N et al, 2018. Are we underestimating the occurrence of sympatric populations?. Molecular Ecology, 27(20): 4011-4025 DOI:10.1111/mec.14846 |
Kang J H, Jang J E, Kim J H et al, 2020. The origin of the subtropical coral Alveopora japonica (Scleractinia: Acroporidae) in high-latitude environments. Frontiers in Ecology and Evolution, 8: 12 DOI:10.3389/fevo.2020.00012 |
Kess T, Boulding E G, 2019. Genome-wide association analyses reveal polygenic genomic architecture underlying divergent shell morphology in Spanish Littorina saxatilis ecotypes. Ecology and Evolution, 9(17): 9427-9441 DOI:10.1002/ece3.5378 |
Kirkpatrick M, 2010. How and why chromosome inversions evolve. PLoS Biology, 8(9): e1000501 DOI:10.1371/journal.pbio.1000501 |
Kulmuni J, Butlin R K, Lucek K et al, 2020. Towards the completion of speciation: the evolution of reproductive isolation beyond the first barriers. Philosophical Transactions of the Royal Society B: Biological Sciences, 375(1806): 20190528 DOI:10.1098/rstb.2019.0528 |
Lamichhaney S, Berglund J, Almén M S et al, 2015. Evolution of Darwin's finches and their beaks revealed by genome sequencing. Nature, 518(7539): 371-375 DOI:10.1038/nature14181 |
Layton K K S, Gosliner T M, Wilson N G, 2018. Flexible colour patterns obscure identification and mimicry in Indo-Pacific Chromodoris nudibranchs (Gastropoda: Chromodorididae). Molecular Phylogenetics and Evolution, 124: 27-36 DOI:10.1016/j.ympev.2018.02.008 |
Leliaert F, Payo D A, Gurgel C F D et al, 2018. Patterns and drivers of species diversity in the Indo-Pacific red seaweed Portieria. Journal of Biogeography, 45(10): 2299-2313 DOI:10.1111/jbi.13410 |
Limmon G, Delrieu-Trottin E, Patikawa J et al, 2020. Assessing species diversity of Coral Triangle artisanal fisheries: a DNA barcode reference library for the shore fishes retailed at Ambon harbor (Indonesia). Ecology and Evolution, 10(7): 3356-3366 DOI:10.1002/ece3.6128 |
Liu J, Cui D, Wang H et al, 2020a. Extensive cryptic diversity of giant clams (Cardiidae: Tridacninae) revealed by DNA-sequence-based species delimitation approaches with new data from Hainan Island, South China Sea. Journal of Molluscan Studies, 86(1): 56-63 DOI:10.1093/mollus/eyz033 |
Liu S Y V, Hsin Y C, Cheng Y R, 2020b. Using particle tracking and genetic approaches to infer population connectivity in the deep-sea scleractinian coral Deltocyathus magnificus in the South China sea. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 161: 103297 DOI:10.1016/j.dsr.2020.103297 |
Mallet J, 2007. Hybrid speciation. Nature, 446(7133): 279-283 DOI:10.1038/nature05706 |
Mao Y F, 2020. Genomic insights into hybridization of reef corals. Coral Reefs, 39(1): 61-67 DOI:10.1007/s00338-019-01869-7 |
Mao Y F, Economo E P, Satoh N, 2018. The roles of introgression and climate change in the rise to dominance of Acropora corals. Current Biology, 28(21): 3373-3382. e5 DOI:10.1016/j.cub.2018.08.061 |
Mao Y F, Satoh N, 2019. A likely ancient genome duplication in the speciose reef-building coral genus, Acropora. iScience, 13: 20-32 DOI:10.1016/j.isci.2019.02.001 |
Marques D A, Meier J I, Seehausen O, 2019. A combinatorial view on speciation and adaptive radiation. Trends in Ecology & Evolution, 34(6): 531-544 |
Mérot C, 2020. Making the most of population genomic data to understand the importance of chromosomal inversions for adaptation and speciation. Molecular Ecology, 29(14): 2513-2516 DOI:10.1111/mec.15500 |
Mérot C, Oomen R A, Tigano A et al, 2020. A roadmap for understanding the evolutionary significance of structural genomic variation. Trends in Ecology & Evolution, 35(7): 561-572 |
Miller E C, Hayashi K T, Song D Y et al, 2018. Explaining the ocean's richest biodiversity hotspot and global patterns of fish diversity. Proceedings of the Royal Society B: Biological Sciences, 285(1888): 20181314 DOI:10.1098/rspb.2018.1314 |
Moore A M, Tassakka A C M, Ambo-Rappe R et al, 2019. Unexpected discovery of Diadema clarki in the Coral Triangle. Marine Biodiversity, 49(5): 2381-2399 DOI:10.1007/s12526-019-00978-4 |
Mora C, Chittaro P M, Sale P F et al, 2003. Patterns and processes in reef fish diversity. Nature, 421(6926): 933-936 DOI:10.1038/nature01393 |
Nelson T C, Cresko W A, 2018. Ancient genomic variation underlies repeated ecological adaptation in young stickleback populations. Evolution Letters, 2(1): 9-21 DOI:10.1002/evl3.37 |
Ng C S L, Jain S S, Nguyen N T H et al, 2019. New genus and species record of reef coral Micromussa amakusensis in the southern South China Sea. Marine Biodiversity Records, 12(1): 17 DOI:10.1186/s41200-019-0176-3 |
Nimbs M J, Smith S D A, 2018. Beyond capricornia: tropical sea slugs (gastropoda, heterobranchia) extend their distributions into the Tasman Sea. Diversity, 10(3): 99 DOI:10.3390/d10030099 |
Pante E, Puillandre N, Viricel A et al, 2015. Species are hypotheses: avoid connectivity assessments based on pillars of sand. Molecular Ecology, 24(3): 525-544 DOI:10.1111/mec.13048 |
Papu A, Undap N, Martinez N A et al, 2020. First study on marine heterobranchia (Gastropoda, Mollusca) in Bangka Archipelago, North Sulawesi, Indonesia. Diversity, 12(2): 52 DOI:10.3390/d12020052 |
Pellissier L, Leprieur F, Parravicini V et al, 2014. Quaternary coral reef refugia preserved fish diversity. Science, 344(6187): 1016-1019 DOI:10.1126/science.1249853 |
Pettersson M E, Rochus C M, Han F et al, 2019. A chromosome-level assembly of the Atlantic herring-detection of a supergene and other signals of selection. Genome Research, 29: 1919-1928 DOI:10.1101/gr.253435.119 |
Pfenninger M, Schwenk K, 2007. Cryptic animal species are homogeneously distributed among taxa and biogeographical regions. BMC Evolutionary Biology, 7(1): 121 DOI:10.1186/1471-2148-7-121 |
Poelstra J W, Richards E J, Martin C H, 2018. Speciation in sympatry with ongoing secondary gene flow and a potential olfactory trigger in a radiation of Cameroon cichlids. Molecular Ecology, (21): 4270-4288 |
Pinheiro H T, Shepherd B, Castillo C et al, 2019. Deep reef fishes in the world's epicenter of marine biodiversity. Coral Reefs, 38(5): 985-995 DOI:10.1007/s00338-019-01825-5 |
Poletto A B, Ferreira I A, Cabral-de-Mello D C et al, 2010. Chromosome differentiation patterns during cichlid fish evolution. BMC Genetics, 11(1): 50 DOI:10.1186/1471-2156-11-50 |
Rabosky D L, Chang J, Title P O et al, 2018. An inverse latitudinal gradient in speciation rate for marine fishes. Nature, 559(7714): 392-395 DOI:10.1038/s41586-018-0273-1 |
Ravinet M, Faria R, Butlin R K et al, 2017. Interpreting the genomic landscape of speciation: a road map for finding barriers to gene flow. Journal of Evolutionary Biology, 30(8): 1450-1477 DOI:10.1111/jeb.13047 |
Renema W, Bellwood D R, Braga J C et al, 2008. Hopping hotspots: global shifts in marine biodiversity. Science, 321(5889): 654-657 DOI:10.1126/science.1155674 |
Renema W, Pandolfi J M, Kiessling W et al, 2016. Are coral reefs victims of their own past success?. Science Advances, 2(4): e1500850 DOI:10.1126/sciadv.1500850 |
Rodríguez-Flores P C, Buckley D, Macpherson E et al, 2020. Deep-sea squat lobster biogeography (Munidopsidae: Leiogalathea) unveils Tethyan vicariance and evolutionary patterns shared by shallow-water relatives. Zoologica Scripta, 49(3): 340-356 DOI:10.1111/zsc.12414 |
Rosser N L, Thomas L, Stankowski S et al, 2017. Phylogenomics provides new insight into evolutionary relationships and genealogical discordance in the reef-building coral genus Acropora. Proceedings of the Royal Society B: Biological Sciences, 284(1846): 20162182 DOI:10.1098/rspb.2016.2182 |
Runemark A, Vallejo-Marin M, Meier J I, 2019. Eukaryote hybrid genomes. PLoS Genetics, 15(11): e1008404 DOI:10.1371/journal.pgen.1008404 |
Salzburger W, 2018. Understanding explosive diversification through cichlid fish genomics. Nature Reviews Genetics, 19(11): 705-717 DOI:10.1038/s41576-018-0043-9 |
Schumer M, Rosenthal G G, Andolfatto P, 2018. What do we mean when we talk about hybrid speciation?. Heredity, 120(4): 379-382 DOI:10.1038/s41437-017-0036-z |
Schwander T, Libbrecht R, Keller L, 2014. Supergenes and complex phenotypes. Current Biology, 24(7): R288-R294 DOI:10.1016/j.cub.2014.01.056 |
Seehausen O, Butlin R K, Keller I et al, 2014. Genomics and the origin of species. Nature Reviews Genetics, 15(3): 176-192 DOI:10.1038/nrg3644 |
Seehausen O, Terai Y, Magalhaes I S et al, 2008. Speciation through sensory drive in cichlid fish. Nature, 455(7213): 620-626 DOI:10.1038/nature07285 |
Shapiro M D, Marks M E, Peichel C L et al, 2004. Genetic and developmental basis of evolutionary pelvic reduction in threespine sticklebacks. Nature, 428(6984): 717-723 DOI:10.1038/nature02415 |
Sheets E A, Warner P A, Palumbi S R, 2018. Accurate population genetic measurements require cryptic species identification in corals. Coral Reefs, 37(2): 549-563 DOI:10.1007/s00338-018-1679-9 |
Stroud J T, Losos J B, 2016. Ecological opportunity and adaptive radiation. Annual Review of Ecology, Evolution, and Systematics, 47: 507-532 DOI:10.1146/annurev-ecolsys-121415-032254 |
Tanaka T, Maeda Y, Veluchamy A et al, 2015. Oil accumulation by the oleaginous diatom Fistulifera solaris as revealed by the genome and transcriptome. The Plant Cell, 27(1): 162-176 DOI:10.1105/tpc.114.135194 |
Torres A F, Ravago-Gotanco R, 2018. Rarity of the "common" coral Pocillopora damicornis in the western Philippine archipelago. Coral Reefs, 37(4): 1209-1216 DOI:10.1007/s00338-018-1729-3 |
Undap N, Papu A, Schillo D et al, 2019. First survey of Heterobranch Sea Slugs (Mollusca, Gastropoda) from the Island Sangihe, North Sulawesi, Indonesia. Diversity, 11(9): 170 DOI:10.3390/d11090170 |
Underwood J N, Richards Z T, Miller K J et al, 2018. Genetic signatures through space, time and multiple disturbances in a ubiquitous brooding coral. Molecular Ecology, 27(7): 1586-1602 DOI:10.1111/mec.14559 |
Uz E, Alanay Y, Aktas D et al, 2010. Disruption of ALX1 causes extreme microphthalmia and severe facial clefting: expanding the spectrum of autosomal-recessive ALX-related frontonasal dysplasia. The American Journal of Human Genetics, 86(5): 789-796 DOI:10.1016/j.ajhg.2010.04.002 |
Van de Peer Y, Mizrachi E, Marchal K, 2017. The evolutionary significance of polyploidy. Nature Reviews Genetics, 18(7): 411-424 DOI:10.1038/nrg.2017.26 |
van Oppen M J H, Bongaerts P, Frade P et al, 2018. Adaptation to reef habitats through selection on the coral animal and its associated microbiome. Molecular Ecology, 27(14): 2956-2971 DOI:10.1111/mec.14763 |
van Oppen M J H, McDonald B J, Willis B et al, 2001. The evolutionary history of the coral genus acropora (Scleractinia, Cnidaria) based on a mitochondrial and a nuclear marker: reticulation, incomplete lineage sorting, or morphological convergence?. Molecular Biology and Evolution, 18(7): 1315-1329 DOI:10.1093/oxfordjournals.molbev.a003916 |
Wagner C E, Harmon L J, Seehausen O, 2012. Ecological opportunity and sexual selection together predict adaptive radiation. Nature, 487(7407): 366-369 DOI:10.1038/nature11144 |
Wallace C C, Rosen B R, 2006. Diverse staghorn corals (Acropora) in high-latitude Eocene assemblages: implications for the evolution of modern diversity patterns of reef corals. Proceedings of the Royal Society B: Biological Sciences, 273(1589): 975-982 DOI:10.1098/rspb.2005.3307 |
Warner P A, van Oppen M J H, Willis B L, 2015. Unexpected cryptic species diversity in the widespread coral Seriatopora hystrix masks spatial-genetic patterns of connectivity. Molecular Ecology, 24(12): 2993-3008 DOI:10.1111/mec.13225 |
Wepfer P H, Nakajima Y, Sutthacheep M et al, 2020. Evolutionary biogeography of the reef-building coral genus Galaxea across the Indo-Pacific ocean. Molecular Phylogenetics and Evolution, 151: 106905 DOI:10.1016/j.ympev.2020.106905 |
Westram A M, Rafajlović M, Chaube P et al, 2018. Clines on the seashore: the genomic architecture underlying rapid divergence in the face of gene flow. Evolution Letters, 2(4): 297-309 DOI:10.1002/evl3.74 |
Wham D C, LaJeunesse T C, 2016. Symbiodinium population genetics: testing for species boundaries and analysing samples with mixed genotypes. Molecular Ecology, 25(12): 2699-2712 DOI:10.1111/mec.13623 |
Wijayanti D P, Indrayanti E, Nuryadi H, 2018. Molecular identification and genetic diversity of Acropora hyacinthus from Boo and Deer Island, Raja Ampat, West Papua. IOP Conference Series: Earth and Environmental Science, 116: 012065 DOI:10.1088/1755-1315/116/1/012065 |
Wolf J B W, Ellegren H, 2017. Making sense of genomic islands of differentiation in light of speciation. Nature Reviews Genetics, 18(2): 87-100 DOI:10.1038/nrg.2016.133 |
Woodland D J, 1983. Zoogeography of the Siganidae (Pisces): an interpretation of distribution and richness patterns. Bulletin of Marine Science, 33(3): 713-717 |
Xu P, Xu J, Liu G J et al, 2019. The allotetraploid origin and asymmetrical genome evolution of the common carp Cyprinus carpio. Nature Communications, 10(1): 4625 DOI:10.1038/s41467-019-12644-1 |
Xu P, Zhang X F, Wang X M et al, 2014. Genome sequence and genetic diversity of the common carp, Cyprinus carpio. Nature Genetics, 46(11): 1212-1219 DOI:10.1038/ng.3098 |
Zong S B, Li Y L, Liu J X, 2020. Genomic architecture of rapid parallel adaptation to fresh water in a wild fish. Molecular Biology and Evolution DOI:10.1093/molbev/msaa290 |
 2021, Vol. 52
2021, Vol. 52


