中国海洋湖沼学会主办。
文章信息
- 王璐, 产久林, 李倩, 张琳琳. 2021.
- WANG Lu, CHAN Jiu-Lin, LI Qian, ZHANG Lin-Lin. 2021.
- 海洋贝类Toll样受体及其接头蛋白MyD88的分子进化研究
- EVOLUTIONARY HISTORY OF THE TOLL-LIKE RECEPTOR AND MYD88 IN MOLLUSCS
- 海洋与湖沼, 52(4): 936-946
- Oceanologia et Limnologia Sinica, 52(4): 936-946.
- http://dx.doi.org/10.11693/hyhz20210200041
文章历史
-
收稿日期:2021-02-07
收修改稿日期:2021-04-13
2. 中国科学院海洋大科学研究中心 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室 青岛 266237;
4. 中国科学院大学 北京 100049
2. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China;
3. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
地球上的生物面临复杂的选择压力, 比如温度、盐度、含氧量等环境因素, 以及病毒、细菌和寄生虫等病原因素, 这些压力也成为宿主适应环境并进化的主要驱动力(Howard et al, 1991)。其中, 免疫系统是生物在长期进化过程中形成的, 生物体内一系列的生物学结构和过程组成的病原防御系统, 可以分为天然免疫和适应性免疫。天然免疫普遍存在于各种生物体内, 是一种快速、广泛的免疫反应, 通过模式识别受体(pattern recognition receptors, PRRs)识别微生物, 而适应性免疫是一种复杂、专一的免疫反应, 可以通过形成记忆细胞产生持久的免疫应答。大多数无脊椎动物缺乏基于经典抗体和记忆细胞的适应性免疫(Litman et al, 2005), 仅依靠天然免疫进行机体防御。随着研究的深入, 研究者们发现无脊椎动物基因组中存在丰富的功能性免疫分子, 它们可能以不同于脊椎动物的机制赋予无脊椎动物免疫“特异性” (章跃陵等, 2005)。
免疫识别是宿主识别异己成分、启动免疫反应的第一步, 在无脊椎动物先天免疫应答过程中起着至关重要的作用。PRRs识别并结合目标病原体相关分子模式(pathogen-associated molecular patterns, PAMPs)后, 免疫反应被启动。Toll样受体(Toll-like receptor, TLR)是经典的天然免疫模式识别受体, 是天然免疫系统中一类重要的蛋白, 最早在果蝇胚胎发育的相关研究中被发现, 随后被证实在抗感染免疫方面发挥重要的作用, 从而为一系列天然免疫识别分子的发现打下基础(Lemaitre et al, 1997)。TLR属于Ⅰ型跨膜蛋白, 胞外具LRR结构域能够识别多种病原, 胞内则以TIR结构域招募不同的受体分子并与之作用激活下游信号通路。TLR信号通路通过一系列分子作用将免疫信号向下传递最终诱导发生细胞免疫反应, 释放各种细胞因子和干扰素来消除病原微生物。MyD88分子是TLR一类重要的接头蛋白(Akira et al, 2001), 该蛋白含有TIR结构域, 能够与TLR的TIR结构域相互作用, 在TLR介导的天然免疫信号通路中起着至关重要的作用。当TLR接收到外界刺激时, 通过信号级联放大激活NF-κB等下游信号分子参与天然免疫过程(Medzhitov et al, 1998)。
海洋贝类暴露在复杂多变的海水环境中, 强大的免疫防御系统对其适应环境具有重要的生态学意义。近年来, 大量的研究表明TLR信号通路在生物免疫防御中具有重要的作用(Akira et al, 2001; Coscia et al, 2011)。已有研究发现, 海洋贝类中TLR基因存在明显的扩张现象(Goodson et al, 2005; Zheng et al, 2005; Zhang et al, 2014), 但目前研究多聚焦于单个基因的分子免疫或单个物种的基因家族特点研究, 而对于整个海洋贝类类群中天然免疫信号通路的分子演化研究较少。本文聚焦于海洋贝类中TLR通路的关键组分、模式识别受体TLR及其接头分子MyD88, 欲探讨这两个基因家族的分子进化历程和在不同病原诱导下的基因表达模式, 这有助于扩展我们对TLR信号通路的认识, 为深入了解海洋无脊椎动物天然免疫多样性和特异性提供基础。
1 材料与方法 1.1 基因组下载与TLR基因鉴定基于NCBI (https://www.ncbi.nlm.nih.gov/)数据库获得32个物种基因组数据, 这32个物种是物种演化过程中的代表性物种, 包括栉水母(Mnemiopsis leidyi)、堡礁海绵(Amphimedon queenslandica)、新星海葵(Nematostella vectensis)、水螅(Hydra vulgaris)、三带黑豹蠕虫(Hofstenia miamia)、海豆芽(Lingula anatina)、水蛭(Helobdella robusta)、小头虫(Capitella teleta)、石鳖(Acanthopleura granulata)、中国真蛸(Octopus sinensis)、加州海兔(Aplysia californica)、福寿螺(Pomacea canaliculata)、红鲍螺(Haliotis rufescens)、猫头鹰帽贝(Lottia gigantea)、魁蚶(Scapharca broughtonii)、欧洲扇贝(Pecten maximus)、紫扇贝(Argopecten purpuratus)、栉孔扇贝(Chlamys farreri)、虾夷扇贝(Mizuhopecten yessoensis)、沼蛤(Limnoperna fortunei)、马氏珠母贝(Pinctada fucata)、美洲牡蛎(Crassostrea virginica)、长牡蛎(Crassostrea gigas)、悉尼岩牡蛎(Saccostrea glomerata)、秀丽隐杆线虫(Caenorhabditis elegans)、黑腹果蝇(Drosophila melanogaster)、西方蜜蜂(Apis mellifera)、海星(Acanthaster planci)、紫色球海胆(Strongylocentrotus purpuratus)、斑马鱼(Danio rerio)、小鼠(Mus musculus)和人(Homo sapiens), 并分别在每个物种的基因组中进行TLR信号通路基因的鉴定。从Pfam蛋白质数据库(http://pfam.xfam.org/)分别下载TIR结构域(PF01582)、LRR结构域(PF13855)、Death结构域(PF00531)、SAM结构域(PF00536)、Arm结构域(PF00514)、TPR结构域(PF13176)、EGF结构域(PF00008)、IG结构域(PF13895), 然后通过基于隐马尔可夫模型的hmmsearch对各物种基因组中可能的TLR、MyD88、SARM、ARM-TIR、IG-TIR、TIR-TPR、EGF-TIR基因进行检索, 获得TLR信号通路基因序列。利用已获得的基因序列作为种子序列, 利用Tblastn的方法在全基因组范围内进一步寻找未鉴定出的基因。TLR由LRR和TIR结构域组成, MyD88由Death结构域和TIR结构域组成, SARM由SAM结构域和TIR结构域组成, ARM-TIR由Arm结构域和TIR结构域组成, IG-TIR由IG结构域和TIR结构域组成, TIR-TPR由TPR结构域和TIR结构域组成, EGF-TIR由EGF结构域和TIR结构域组成。通过SMART (http://smart.embl-heidelberg.de/)和InterProScan (https://www.ebi.ac.uk/interpro/search-/sequence/)对已鉴定的TLR通路基因的蛋白结构域进行分析和确认, 结构域对比结果evalue > 1e-5时认为具有可信度。正确的TLR通路蛋白应该由完整结构域组成, 且具有较高的序列保守度。对于编码结构域不完整的TLR和MyD88基因, 我们首先利用GeneWise (Ewan et al, 2004)在基因组层面对基因进行矫正, 如果矫正不到完整的基因结构则不计入本文的研究范畴。
1.2 系统发育分析获取各物种TLR蛋白全长中保守的TIR结构域, 通过ClustalW (Thompson et al, 1994)对TIR蛋白序列进行对比分析, 并利用trimAl (Capella- Gutiérrez et al, 2009)进行对比分析结果的优化, 最后基于两种不同算法对TIR进行建树, 并从中选择支持率最高的分子进化树模型。第一种方法是利用IQ-TREE (Nguyen et al, 2015)基于最大似然法(Maximum Likelihood Method, ML)对基因进行系统发育分析, IQ-TREE可以自动测试和选择最佳替代模型; 第二种方法是利用MEGA7 (Kumar et al, 2016)基于邻接法(Neighbor Joining Method, NJ)对基因进行系统发育分析, 尝试各种模型后, 根据树形的可靠程度选择最适模型进行建树, 本文中使用的模型为p-distance。构建的进化树都使用自展法(Bootstrap)检测置信度, 重复数为1 000。系统发育树用ITOL (https://itol.embl.de/index.shtml) (Letunic et al, 2016)进行可视化。
1.3 基因表达分析利用长牡蛎在不同条件下的转录组表达数据来研究海洋贝类TLR和MyD88的分子进化。第一套转录组数据是长牡蛎在不同发育阶段、不同成体器官组织和不同胁迫处理条件下的转录组表达数据(NCBI登录号: GSE31012), 这些转录组数据主要来自8个器官(外套膜、鳃、闭壳肌、消化腺、血细胞、唇瓣、雌性和雄性性腺)、38个发育阶段和各种非生物胁迫, 包括5、10、15、25、30、35 ℃的梯度温度胁迫, 5、10、15、20、25、40的梯度盐度胁迫, 饥饿、缺氧的干露胁迫和锌、镉、铜、汞、铅的重金属胁迫; 第二套转录组数据是长牡蛎在不同致病弧菌(鳗弧菌Vibrio anguillarum、塔氏弧菌Vibrio tubiashii、河口弧菌Vibrio aestuarianus、溶藻弧菌Vibrio alginolyticus、滕黄微球菌Micrococcus luteus)和四种弧菌等量混合物(鳗弧菌、塔氏弧菌、河口弧菌、溶藻弧菌)、LPS(Lipopolysaccharide)、牡蛎疱疹病毒(Ostreid Herpevirus-1, OsHV-1)胁迫下的样品表达数据(NCBI登录号: SRP019967)。
RPKM计算。获得RNA-seq的原始数据后, 用Topha将所有测序读段通过序列映像定位到参考基因组上, 这是后续处理和分析的基础(Trapnell et al, 2009)。RPKM (reads per kilo bases per million reads)是每百万读段中来自于某基因每千碱基长度的读段数。
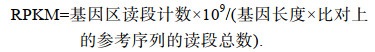 (1)
(1)差异基因筛选。差异表达基因的识别参考Chen等(2010)的文章进行。FDR值(False discovery rate)作为p值的检测指标评价某基因在两个转录组中是否有表达差异, 本研究中, FDR阈值设定为0.01。筛选在牡蛎病毒OsHV-1、LPS、鳗弧菌V. anguillarum、塔氏弧菌V. tubiashii、河口弧菌V. aestuarianus、溶藻弧菌V. alginolyticus、滕黄微球菌M. luteus、四种弧菌混合物、非生物因素(温度、盐度、干露、重金属)胁迫下的差异表达基因。差异基因的选取主要包含两个要求: (1) 要求差异基因在一半以上处理组中有相同的上调或下调表达趋势(和对照组比较)并且FDR均小于0.01; (2) 有些基因虽然有差异表达, 但其在不同条件下的表达量都很低(对照组和处理组RPKM均 < 1), 这种基因不算作差异表达基因。
通过以上方法我们得到在不同胁迫下差异表达以及在特定时期、器官特异性表达的TLR和MyD88基因。将系统进化分析与基因表达分析相结合, 探究海洋贝类TLR和其接头蛋白的分子进化。
2 结果与分析 2.1 海洋贝类TLR信号通路关键分子的扩张为了在物种演化的背景下探讨TLR信号通路的进化模式, 我们基于NCBI公共数据库获得29个无脊椎动物以及其他3个脊椎动物(斑马鱼、小鼠、人)的基因组, 并从中鉴定TLR信号通路成员(图 1)。通过分析, 我们发现在不同的物种中, TLR基因及其下游接头分子(MyD88、SARM、ARM-TIR、IG-TIR、TIR-TPR、EGF-TIR)的数量存在着较大的差异。在较原始的栉水母和多孔动物中没有鉴定到含有经典结构域的TLR基因, 只鉴定出了MyD88基因; 刺胞动物新星海葵中鉴定出TLR基因和下游的MyD88、ARM-TIR、IG-TIR基因, 但数量较少; 在两侧对称的扁形动物H. miamia中只鉴定到SARM、ARM-TIR两个接头分子而没有发现经典结构域的TLR基因。然而, 在冠轮动物中TLR通路基因存在明显的扩增现象, 包括TLR基因及其下游的接头分子。在美洲牡蛎、长牡蛎、马氏珠母贝中分别发现130、96、83个TLR基因, 在虾夷扇贝、栉孔扇贝、长牡蛎中分别发现23、13、10个MyD88基因, 说明TLR信号通路的扩张现象普遍存在于海洋贝类中。无脊椎动物中另一个较明显的TLR扩张现象发生在海胆中, 紫海胆(S. purpuratus)中存在222个TLR基因, 但TLR信号通路下游的接头分子并不存在扩张现象。
 |
| 图 1 物种树及对应TLR通路基因数目 Fig. 1 Species tree and the number of TLR pathway genes 注: Mle: 栉水母; Aqu: 堡礁海绵; Nve: 新星海葵; Hvu: 水螅; Hmi: 三带黑豹蠕虫; Lin: 海豆芽; Hro: 水蛭; Cte: 小头虫; Aca: 石鳖; Osi: 中国真蛸; Apl: 加州海兔; Pom: 福寿螺; Hru: 红鲍螺; Lot: 猫头鹰帽贝; Sbr: 魁蚶; Pma: 欧洲扇贝; Apu: 紫扇贝; Cfa: 栉孔扇贝; Mye: 虾夷扇贝; Lfo: 沼蛤; Pfu: 马氏珠母贝; Cvi: 美洲牡蛎; Cgi: 长牡蛎; Sgl: 悉尼岩牡蛎; Cel: 秀丽隐杆线虫; Dme: 黑腹果蝇; Ame: 西方蜜蜂; Ap: 棘冠海星; Spu: 紫色球海胆; Dre: 斑马鱼; Mmu: 小鼠; Hsa: 人 |
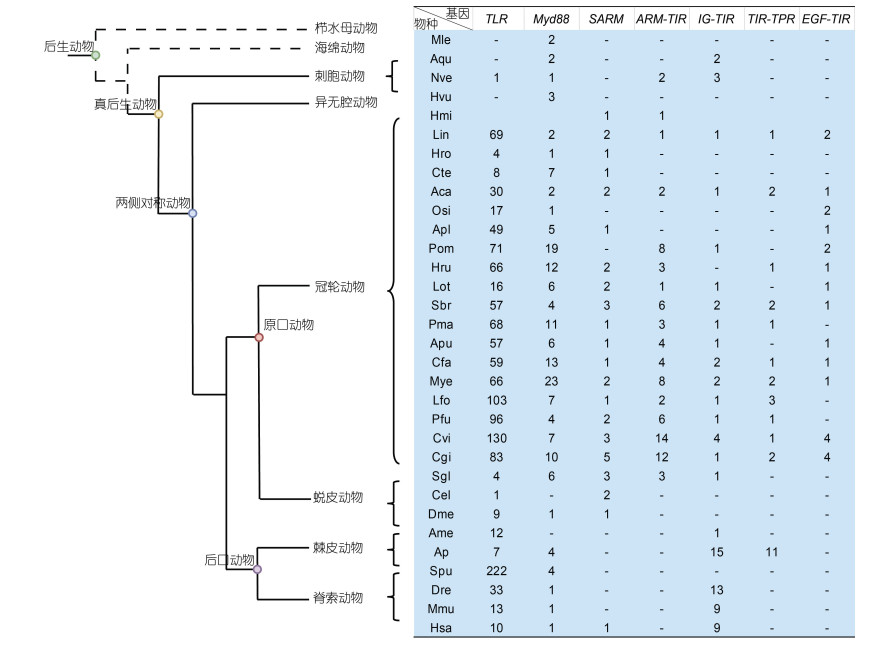
软体动物TLR基因可以根据结构域类型分为五类: V (Vertebrate-type TLR, 脊椎动物类), P (Protostome-like TLR, 原口动物型, 具有LRRCT和LRRNT胞外结构域), sP (不含LRRCT-LRRNT胞外结构域的短原口动物类)、sPP (含LRRCT-LRRNT胞外结构域的短原口动物类)和Ls (具LRRCT特异性胞外结构域) (Zhang et al, 2015)。为了更好地了解软体动物TLR基因家族的整体结构和系统演化, 将9种软体动物(多板纲: 颗粒棘石鳖A. granulata; 头足纲: 中国真蛸O. sinensis; 腹足纲: 海蜗牛A. californica、福寿螺P. canaliculata、猫头鹰帽贝L. gigantea; 双壳类: 栉孔扇贝C. farreri、马氏珠母贝P. fucata、美洲牡蛎C. virginica、长牡蛎C. gigas)和2种外群动物(腕足动物海豆芽L. anatina、环节动物小头虫C. teleta)中鉴定得到的TLR基因基于TIR结构域序列构建最大似然树(图 2)。从图 2可以看到, 软体动物中双壳类和腹足类TLRs发生明显的扩张现象, 双壳类TLRs的扩张可以大体分为五支, 且大多是谱系特异性扩张, 与腹足类TLRs呈现相对独立的关系。根据腹足纲和双壳类的TLRs结构域注释我们发现这两类软体动物的TLR扩张大多为sP类型, 在504条TLRs中76.2%为sP-TLRs, 这种sP型主导且相对独立发生的扩张证实了牡蛎TLR基因的特异性扩张, 可能在宿主防御中发挥重要功能。
 |
|
图 2 软体动物TLR基因的系统发育树(基于最大似然法) Fig. 2 Phylogenetic tree (constructed with the maximum likelihood method) of TLRs in mollusks
注: 基因ID不同底色代表相应物种, 色带与分支颜色代表软体动物结构域分类 注: Lin: 海豆芽; Cte: 小头虫; Aca: 石鳖; Osi: 中国真蛸; Apl: 加州海兔; Pom: 福寿螺; Lot: 猫头鹰帽贝; Cfa: 栉孔扇贝; Pfu: 马氏珠母贝; Cvi: 美洲牡蛎; Cgi: 长牡蛎 |
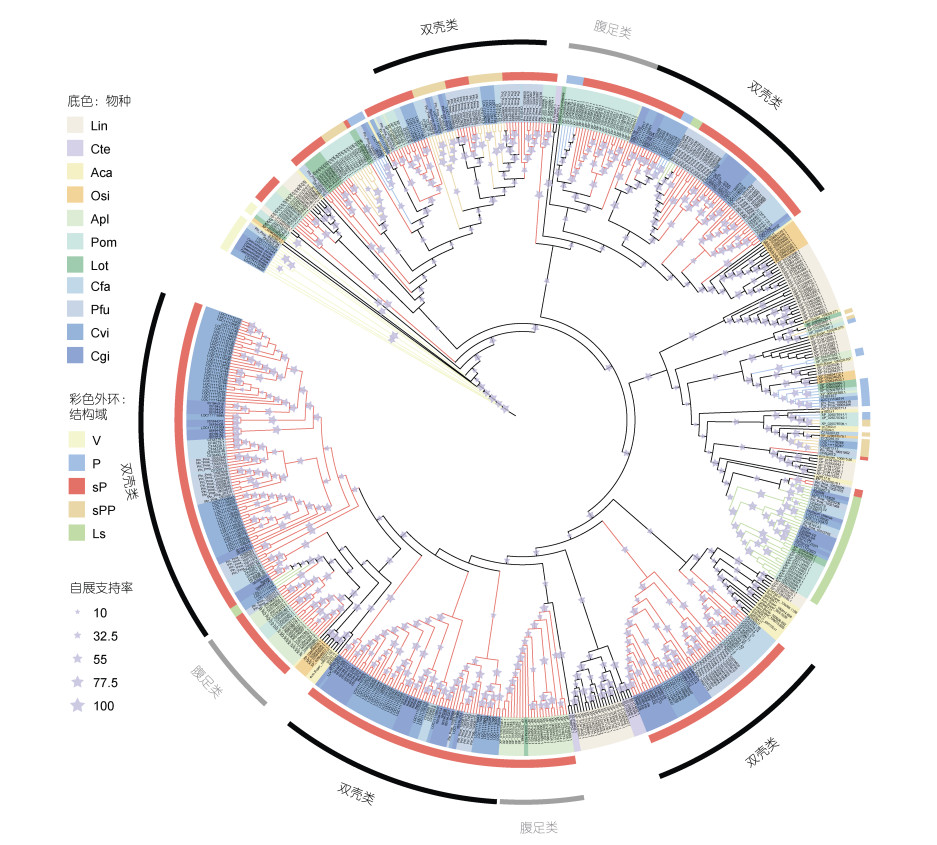
通过分析, 我们也在软体动物的接头分子MyD88中检测到显著的扩张现象。MyD88整个结构由3个功能结构域组成, 包括C端的TIR结构域、中间域及N端的Death结构域, 是软体动物TLR受体信号向下游传导的核心分子。为了丰富对TLR信号通路的了解, 本文将选取的9种软体动物及2种外群动物鉴定得到的MyD88基因通过TIR结构域序列构建最大似然树(图 3)。图 3的结果表明, 腹足纲与双壳类MyD88s的扩张是相对独立的, 其中腹足纲扩张的MyD88s可以分为三支, 腹足类-1与腹足类-3两支谱系特异性扩张, 腹足类-2这一支与海豆芽、双壳贝类聚在一起, 可能是协同演化的结果。而双壳类扩张的MyD88s可以主要分为两支, 这两支都属于谱系特异性扩张。在软体动物双壳类、腹足纲中MyD88s的谱系特异性扩张现象也进一步证明了软体动物TLR信号通路的特异性。
 |
| 图 3 软体动物MyD88基因的系统发育树(基于最大似然法) Fig. 3 Phylogenetic tree (constructed with the maximum likelihood method) of MyD88s in mollusks. 注: Lin: 海豆芽; Cte: 小头虫; Aca: 石鳖; Osi: 中国真蛸; Apl: 加州海兔; Pom: 福寿螺; Lot: 猫头鹰帽贝; Cfa: 栉孔扇贝; Pfu: 马氏珠母贝; Cvi: 美洲牡蛎; Cgi: 长牡蛎 |
潮间带地区的温度、盐度和含氧量等在每日间的变化极大, 而双壳类长牡蛎营潮间带生活, 受到众多的环境选择压力, 是研究无脊椎动物TLR信号通路的绝佳对象, 我们通过NJ法构建长牡蛎TLRs系统发育树, 并利用各种长牡蛎转录组数据探究TLRs的演化与功能(图 4)。结果显示, 长牡蛎基因组中存在83个TLR基因, 其中包含5个V型、2个P型、63个sP型、2个sPP型和8个Ls型TLR, 其中sP类型的TLRs根据进化树可以分为两大支, 且扩张最广。
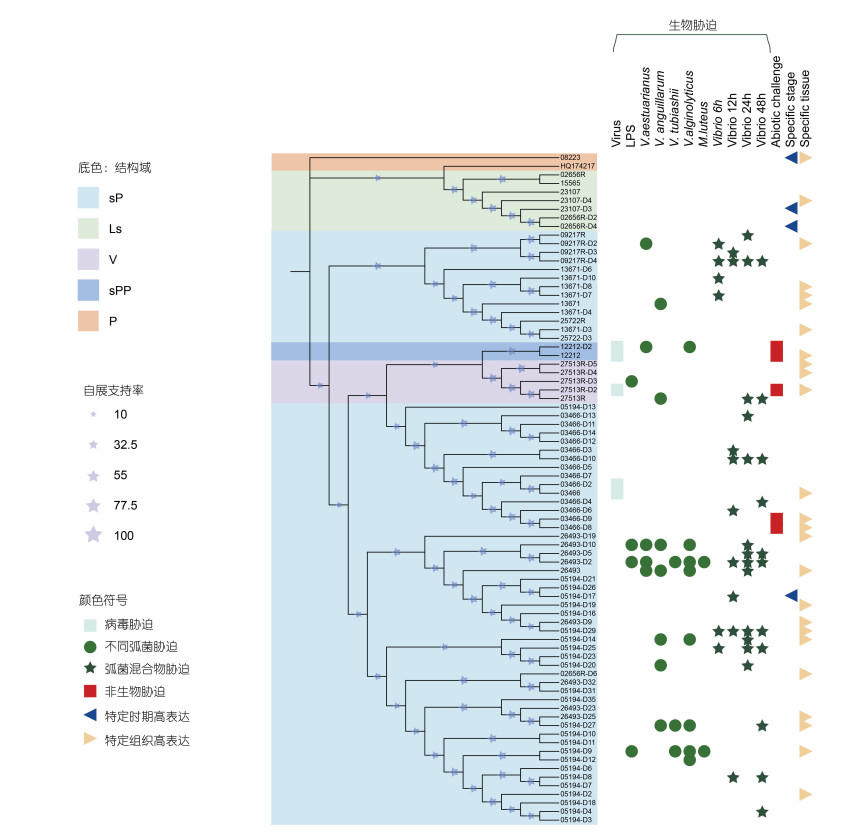 |
| 图 4 长牡蛎TLRs的邻接进化树及其在生物、非生物胁迫环境和发育过程中的表达分化 Fig. 4 The neighbor-joining tree of TLRs in Crassostrea gigas and their expression divergence under biotic, abiotic challenge and during development. 注: 不同颜色符号分别代表在不同条件下的显著上调基因, 绿色-生物胁迫, 红色-非生物胁迫, 蓝色-特定时期高表达, 橘色-特定组织高表达 |
对不同胁迫处理下长牡蛎的转录组数据进行筛选和差异表达分析, 我们获得大量的TLR差异表达基因-CgTLRs。共有21个CgTLRs在生物和非生物胁迫下显著下调表达, 46个CgTLRs在生物和非生物胁迫下显著上调表达。我们重点关注在胁迫条件下上调表达的基因, 如图 4所示, 在牡蛎病毒OsHV-1、LPS、弧菌、革兰氏阳性菌(藤黄微球杆菌)的胁迫刺激下, 分别有4、6、33、4个CgTLRs显著上调表达; 在非生物因素(温度、盐度和干露)胁迫下, 5个CgTLRs显著上调表达。通过分析发现, 有41个CgTLRs只在生物胁迫下特异性高表达, 2个CgTLRs只在非生物胁迫下特异性高表达, 不同的CgTLRs在响应外界胁迫时具有一定的特异性。除此之外有4个CgTLRs在特定的发育时期表达, 27个CgTLRs存在组织特异性表达, 尤其倾向于在唇瓣、鳃、血液高表达。
2.5 长牡蛎MyD88s在胁迫环境以及早期发育时期的基因表达模式对长牡蛎MyD88s进行系统发育与表达分析(图 5), 长牡蛎中共鉴定出10个CgMyD88s, 其中6个具有典型的Death-TIR结构域组合, 4个仅具有TIR结构域。7个串联重复的CgMyD88s分布在三个scaffold, 在系统发育树上分别聚类。
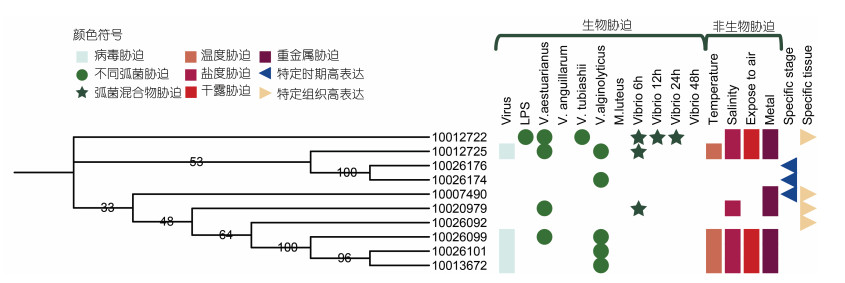 |
| 图 5 长牡蛎MyD88s的邻接进化树及其在生物、非生物胁迫环境和发育过程中的表达分化 Fig. 5 The neighbor-joining tree of MyD88s in Crassostrea gigas and their expression divergence under biotic, abiotic challenge and during development. 注: 不同颜色符号分别代表在不同条件下的显著上调基因, 绿色-生物胁迫, 红色-非生物胁迫, 蓝色-特定时期高表达, 橘色-特定组织高表达 |
共有8个CgMyD88s在生物和非生物胁迫下显著下调表达, 7个CgMyD88s在生物和非生物胁迫下显著上调表达。如图 5所示, 在OsHV-1病毒、LPS、弧菌、革兰氏阳性菌的胁迫刺激下, 分别有4、1、7、1个CgMyD88s显著上调表达; 而在温度、盐度、干露、重金属的胁迫刺激下, 分别有4、6、5、7个CgMyD88s显著上调表达。大部分CgMyD88s对生物和非生物胁迫敏感, 7个CgMyD88s在生物因素和非生物因素刺激下都上调表达。而ID为OYG_ 10026174_10008756的Cgmyd88只在弧菌诱导下上调表达但对非生物刺激不敏感; OYG_10007490_10008864在鳃、消化腺受到重金属胁迫时上调表达但对生物胁迫没有响应。此外有3个CgMyD88s在特定的发育时期高表达, 其中2个CgMyD88s在D时期特异高表达, 1个CgMyD88在T时期特异高表达; 4个CgMyD88s存在组织特异性表达, 尤其倾向于在血液高表达。
3 讨论 3.1 TLR通路在海洋贝类中的显著扩张TLR介导的天然免疫信号通路是对外来病原体的响应机制, 主要的功能就是识别病原体, 通过一系列分子作用将免疫信号向下传递最终诱导发生细胞炎症反应, 释放各种细胞因子和干扰素来消除病原微生物。有关TLRs的研究几乎涵盖动物界的所有门类, 从简单的海绵动物到复杂的哺乳动物都有TLR及其同系物的相关报道, 因此认为其是最古老的模式识别受体之一(Coscia et al, 2011)。虽然目前在所有海绵动物门类中都没有发现含经典LRR结构域和TIR结构域的TLR基因, 但已经发现TIR蛋白到NF-kB的MyD88依赖下游信号存在于海绵动物中(Brennan et al, 2018)。前期的研究表明, 在海洋贝类基因组中天然免疫相关基因家族均存在不同程度的扩张现象(Imamura et al, 2002; Goodson et al, 2005; Zheng et al, 2005; Wang et al, 2011; Zhang et al, 2014), 进而增强其对病原体的天然免疫功能(Zhang et al, 2012; Zhang et al, 2015)。例如, 在长牡蛎基因组中注释得到了83个TLR序列, 其中19个TLRs在病毒和细菌的刺激下差异表达(Zhang et al, 2015)。然而, TLR信号通路在海洋贝类中的系统演化关系并没有得到很好的梳理。因此, 本研究以海洋贝类为主要研究对象, 基于基因组和转录组数据, 结合生物信息学手段, 研究海洋贝类TLR和其接头蛋白MyD88s的分子进化, 有助于扩展我们对TLR信号通路的认识, 为深入了解海洋贝类天然免疫多样性和特异性提供基础。
本研究基于生物演化过程中32个代表性物种的基因组序列, 对TLR信号通路中相关基因进行了筛选和鉴定。其中, 在美洲牡蛎、长牡蛎、马氏珠母贝中分别发现130、96、83个TLR基因, 表明TLR信号通路的扩张现象普遍存在于海洋贝类中, 并进一步证实了TLR信号通路在海洋贝类中的显著扩张事件(图 1)。此外, 在本研究中我们还发现TLR通路其他相关基因在海洋贝类中同样存在扩张现象(图 1)。MyD88作为TLR通路中关键的接头分子, 在大多数海洋贝类中均能鉴定到其扩张现象, 其中在虾夷扇贝、栉孔扇贝、长牡蛎中分别发现23、13、10个MyD88基因, 这揭示了MyD88依赖型TLR信号通路在海洋贝类天然免疫系统中的保守性、重要性和关键性, 同时提示海洋贝类基因组在进化中积累了相对较多的防御系统基因资源以适应复杂多变的环境(Zhang et al, 2015; Guo et al, 2016)。
3.2 海洋贝类TLRs和MyD88s扩张起源在长期进化过程中, 海洋贝类已进化出独特的免疫防御体系, 如通过免疫基因的大量扩张等策略来适应环境。前期的研究表明, 串联复制等多种机制是基因家族扩张的主要途径, 包括脊椎动物的MHC和Ig基因、海胆的TLRs基因、文昌鱼的TNFs基因和淡水蜗牛的FREPs基因在内的基因家族扩张事件均已被证实与基因的串联重复相关(Hanington et al, 2010)。
在本文中, 我们发现在海洋贝类基因组中TLR通路相关基因存在显著的基因扩张现象, 然而其扩张的起源和机制仍不清楚。因此, 我们以海洋贝类代表种长牡蛎为研究对象, 通过对其TLRs和MyD88s的系统进化分析, 试图阐明TLR通路相关基因在海洋贝类中的扩张起源和机制。结果显示CgTLRs和CgMyD88s的扩张均表现为谱系特异性, 并且CgTLRs的扩张大多来源于sP-TLRs的串联重复事件(图 2, 3), 揭示了这些免疫分子是通过基因串联复制和谱系特异性扩张途径完成适应性进化的。此外, 结合长牡蛎转录数据分析, 发现不同环境因子胁迫下长牡蛎差异表达的TLRs和MyD88s大多为串联重复基因并且这些差异表达基因偏向于在基因组多拷贝并呈现高度分化, 结合软体动物系统发育分析推测其多为长牡蛎特异的新基因。这可能暗示了在海洋贝类祖先物种中, 应激调控基因(例如TLRs等免疫防御基因)受到更大的环境选择压力, 并发生多次复制事件, 以提供更多的基因资源帮助海洋贝类适应生存环境。
此外, 在研究中我们还发现在紫海胆中TLR基因显著扩张, 但下游只有3个MyD88基因, 暗示着紫海胆增加了天然免疫识别的特异性, 但在信号传导通路上仍是保守的。另一方面, 这种演化模式与部分贝类中的TLR与MyD88协同扩张并不一致, 也再次揭示海胆和贝类中TLR的扩张可能是独立演化而来。
3.3 TLRs和MyD88s功能演化研究TLR作为天然免疫受体, 能够识别病原微生物的PAMPs, 并通过其TIR结构域招募下游信号分子或接头蛋白, 从而激活机体的免疫防御等(Kirschning et al, 2001)。前期研究表明, TLR蛋白在结构功能上是比较保守的, 但是他们在所识别的PAMPs上却存在着较大的差异(Oshiumi et al, 2008)。本研究中, 我们通过对不同环境因子胁迫下长牡蛎的转录组数据进行分析, 获得大量的差异表达CgTLRs, 初步表明TLR基因家族在参与海洋贝类机体防御方面发挥重要作用。此外, TLRs被证明在识别PAMPs的进化方面相对保守, 识别的PAMPs从病毒、细菌、真菌到寄生虫, 并且表现出较强的目标特异性(Underhill et al, 1999; Campos et al, 2001; Hoebe et al, 2003; Meier et al, 2003)。
在本文中, 基于不同环境胁迫下长牡蛎的转录组数据, 我们检测到33个CgTLRs特异性识别生物因素胁迫, 2个CgTLRs特异性识别非生物胁迫, 表现为不同种类的TLRs应对不同类型的外界胁迫(图 4)。有趣的是, 我们发现CgTLRs具有组织表达特异性, 大部分CgTLRs在免疫组织中高表达, 比如唇瓣、鳃、血液等(图 4), 这与之前对栉孔扇贝TLR的表达研究相一致(Qiu et al, 2007)。结合软体动物TLR基因系统发育分析(图 2), 我们还发现这些免疫相关组织特异性高表达的CgTLRs多为长牡蛎特异的新基因。另外, 前期对蚯蚓(Eisenia andrei) TLR的研究中发现P-TLR能参与调控蚯蚓的发育过程(Škanta et al, 2013), 而相似类型的P-TLR与Ls-TLR也被发现存在于CgTLRs中(图 4), 并且仅在长牡蛎特定的发育时期特异性高表达, 表明在长牡蛎中参与调节发育的TLR基因起源相对古老。这也揭示了TLRs在长牡蛎中发生功能分化, 参与机体发育相关的TLRs起源相对古老, 而参与免疫调节相关的TLRs多属于特异扩张的新基因。
MyD88是TLR信号通路中重要的接头蛋白, 通过信号级联放大激活NF-κB信号通路参与天然免疫过程(Medzhitov et al, 1998), 并在海洋贝类TLR信号通路及天然免疫中发挥至关重要的作用。前人的研究发现, 厚壳贻贝经沙氏弧菌感染后, MyD88基因表达量在免疫相关的特异性组织中急剧上升(梁箫等, 2019), 类似的结论在魁蚶中也得到验证(黄永欢, 2016)。在本研究中, 对不同环境因子胁迫下长牡蛎转录组中MyD88s的表达分析进一步证实其免疫功能的特异性, 如图 5所示, CgMyD88基因(OYG_10026174_10008756)仅在溶藻弧菌诱导下特异性上调表达, 而CgMyD88基因(OYG_10007490_10008864)仅在重金属胁迫下特异性上调表达。同时, 我们还发现处在同一串联重复簇上的CgMyD88s可能对不同的环境胁迫表现为差异性的响应模式, 揭示了CgMyD88免疫功能的明显分化。另外, 与CgTLRs类似, CgMyD88s除了应对外界胁迫, 也参与调节长牡蛎的发育过程, 并且倾向于在血淋巴中高表达, 而血淋巴是长牡蛎中重要的免疫场所, 暗示了其在海洋贝类免疫反应中发挥着重要作用(Matozzo et al, 2007)。
4 结论本文探究Toll样受体及其接头蛋白MyD88s在海洋贝类中的分子和功能演化机制, 揭示了TLR通路相关基因在海豆芽、福寿螺、长牡蛎、马氏珠母贝等贝类中存在显著扩张现象, 并且在腹足类和双壳类中均是谱系特异性的扩张。此外, 结合海洋贝类代表种长牡蛎的胁迫转录组数据, 本文进一步探讨了海洋贝类代表物种长牡蛎中TLR及MyD88表达特异性分化的分子进化过程, 结果表明发育时期差异表达的TLR基因起源相对古老, 而海洋双壳贝类特异性扩张的TLR基因能够被病原感染激活, 且对不同的病原感染表达模式具有特异性。海洋贝类通过串联重复和谱系特异性扩张赋予海洋贝类天然免疫系统特异性和复杂性, 这些结果不仅丰富了我们对海洋贝类TLR信号通路功能的理解, 也为后期深入探索无脊椎动物免疫系统提供理论基础。然而, 本文主要以长牡蛎作为海洋贝类的代表进行研究, 还需更多的海洋贝类数据支持来丰富海洋贝类TLR通路的分子进化研究。
致谢 本文获得中国科学院海洋研究所海洋大数据中心的支持, 谨致谢忱。
黄永欢, 2016. 魁蚶MyD88、CAT和GST的克隆及功能分析. 上海: 上海海洋大学硕士学位论文, 1-62
|
章跃陵, 王三英, 彭宣宪. 2005. 无脊椎动物适应性免疫的研究进展. 水产科学, 24(8): 43-45 DOI:10.3969/j.issn.1003-1111.2005.08.016 |
梁箫, 刘钰珠, 陈珂, 等. 2019. 厚壳贻贝MyD88-4基因的生物学特性及其对沙氏弧菌的免疫应答. 水产学报, 43(11): 2347-2358 |
Akira S, Takeda K, Kaisho T, 2001. Toll-like receptors: critical proteins linking innate and acquired immunity. Nature Immunology, 2(8): 675-680 DOI:10.1038/90609 |
Birney E, Clamp M, Durbin R, 2004. GeneWise and genomewise. Genome Research, 14(5): 988-995 DOI:10.1101/gr.1865504 |
Brennan J J, Gilmore Thomas D, 2018. Evolutionary Origins of Toll-like Receptor Signaling. Molecular biology and evolution, 35(7): 1576-1587 DOI:10.1093/molbev/msy050 |
Campos M A S, Almeida I C, Takeuchi O et al, 2001. Activation of Toll-like receptor-2 by glycosylphosphatidylinositol anchors from a protozoan parasite. The Journal of Immunology, 167(1): 416-423 DOI:10.4049/jimmunol.167.1.416 |
Capella-Gutiérrez S, Silla-Martínez J M, Gabaldón T, 2009. TrimAl: a tool for automated alignment trimming in large-scale phylogenetic analyses. Bioinformatics, 25(15): 1972-1973 DOI:10.1093/bioinformatics/btp348 |
Chen S, Yang P C, Jiang F et al, 2010. De novo analysis of transcriptome dynamics in the migratory locust during the development of phase traits. PLoS One, 5(12): e15633 DOI:10.1371/journal.pone.0015633 |
Coscia M R, Giacomelli S, Oreste U, 2011. Toll-like receptors: an overview from invertebrates to vertebrates. Invertebrate Survival Journal, 2(8): 210-226 |
Goodson M S, Kojadinovic M, Troll J V et al, 2005. Identifying components of the NF-κB pathway in the beneficial Euprymna scolopes-Vibrio fischeri light organ symbiosis. Applied and Environmental Microbiology, 71(11): 6934-6946 DOI:10.1128/AEM.71.11.6934-6946.2005 |
Guo X M, Ford S E, 2016. Infectious diseases of marine molluscs and host responses as revealed by genomic tools. Philosophical Transactions of the Royal Society B: Biological Sciences, 371(1689): 20150206 DOI:10.1098/rstb.2015.0206 |
Hanington P C, Forys M A, Dragoo J W et al, 2010. Role for a somatically diversified lectin in resistance of an invertebrate to parasite infection. Proceedings of the National Academy of Sciences of the United States of America, 107(49): 21087-21092 DOI:10.1073/pnas.1011242107 |
Hoebe K, Du X, Georgel P et al, 2003. Identification of Lps2 as a key transducer of MyD88-independent TIR signalling. Nature, 424(6950): 743-748 DOI:10.1038/nature01889 |
Howard J C, 1991. Disease and evolution. Nature, 352(6336): 565-567 DOI:10.1038/352565a0 |
Imamura M, Yamakawa M, 2002. Molecular cloning and expression of a Toll receptor gene homologue from the silkworm, Bombyx mori. Biochimica et Biophysica Acta (BBA)-Gene Structure and Expression, 1576(3): 246-254 DOI:10.1016/S0167-4781(02)00336-6 |
Kirschning C J, Bauer S, 2001. Toll-like receptors: cellular signal transducers for exogenous molecular patterns causing immune responses. International Journal of Medical Microbiology, 291(4): 251-260 DOI:10.1078/1438-4221-00128 |
Kumar S, Stecher G, Tamura K, 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology & Evolution, 33(7): 1870-1874 |
Lemaitre B, Reichhart J M, Hoffmann J A, 1997. Drosophila host defense: differential induction of antimicrobial peptide genes after infection by various classes of microorganisms. Proceedings of the National Academy of Sciences of the United States of America, 94(26): 14614-14619 DOI:10.1073/pnas.94.26.14614 |
Letunic I, Bork P, 2016. Interactive tree of life (iTOL) v3:an online tool for the display and annotation of phylogenetic and other trees. Nucleic Acids Research, 44(W1): W242-W245 DOI:10.1093/nar/gkw290 |
Litman G W, Cannon J P, Dishaw L J, 2005. Reconstructing immune phylogeny: new perspectives. Nature Reviews Immunology, 5(11): 866-879 |
Matozzo V, Rova G, Marin M G, 2007. Haemocytes of the cockle Cerastoderma glaucum: morphological characterisation and involvement in immune responses. Fish & Shellfish Immunology, 23(4): 732-746 |
Medzhitov R, Preston-Hurlburt P, Kopp E et al, 1998. MyD88 is an adaptor protein in the hToll/IL-1 receptor family signaling pathways. Molecular Cell, 2(2): 253-258 DOI:10.1016/S1097-2765(00)80136-7 |
Meier A, Kirschning C J, Nikolaus T et al, 2003. Toll-like receptor (TLR) 2 and TLR4 are essential for Aspergillus-induced activation of murine macrophages. Cellular Microbiology, 8(5): 561-570 |
Nguyen L T, Schmidt H S, Von Haeseler A et al, 2015. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution, 32(1): 268-274 DOI:10.1093/molbev/msu300 |
Oshiumi H, Matsuo A, Matsumoto M et al, 2008. Pan-vertebrate Toll-like receptors during evolution. Current Genomics, 9(7): 488-493 DOI:10.2174/138920208786241234 |
Qiu L M, Song L S, Xu W et al, 2007. Molecular cloning and expression of a Toll receptor gene homologue from Zhikong Scallop, Chlamys farreri. Fish & Shellfish Immunology, 22(5): 451-466 |
Škanta F, Roubalová R, Dvořák J et al, 2013. Molecular cloning and expression of TLR in the Eisenia andrei earthworm. Developmental & Comparative Immunology, 41(4): 694-702 |
Thompson J D, Higgins D G, Gibson T J, 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 22(22): 4673-4680 DOI:10.1093/nar/22.22.4673 |
Trapnell C, Pachter L, Salzberg S L, 2009. TopHat: discovering splice junctions with RNA-Seq. Bioinformatics, 25(9): 1105-1111 DOI:10.1093/bioinformatics/btp120 |
Underhill D M, Ozinsky A, Hajjar A M et al, 1999. The Toll-like receptor 2 is recruited to macrophage phagosomes and discriminates between pathogens. Nature, 401(6755): 811-815 DOI:10.1038/44605 |
Wang M Q, Yang J L, Zhou Z et al, 2011. A primitive Toll-like receptor signaling pathway in mollusk Zhikong scallop Chlamys farreri. Developmental & Comparative Immunology, 35(4): 511-520 |
Zhang G F, Fang X D, Guo X M et al, 2012. The oyster genome reveals stress adaptation and complexity of shell formation. Nature, 490(7418): 49-54 DOI:10.1038/nature11413 |
Zhang L L, Li L, Guo X M et al, 2015. Massive expansion and functional divergence of innate immune genes in a protostome. Scientific Reports, 5(1): 8693 DOI:10.1038/srep08693 |
Zhang L L, Li L, Zhu Y B et al, 2014. Transcriptome analysis reveals a rich gene set related to innate immunity in the eastern oyster (Crassostrea virginica). Marine Biotechnology, 16(1): 17-33 DOI:10.1007/s10126-013-9526-z |
Zheng L, Zhang L, Lin H et al, 2005. Toll-like receptors in invertebrate innate immunity. Invertebrate Survival Journal, 2(2): 105-113 |
 2021, Vol. 52
2021, Vol. 52


