
中国海洋湖沼学会主办。
文章信息
- 金晶磊, 周前进, 邵鑫斌, 王瑶华, 陈炯, 闫茂仓, 苗亮, 唐标, 张严峻. 2021.
- JIN Jing-Lei, ZHOU Qian-Jin, SHAO Xin-Bin, WANG Yao-Hua, CHEN Jiong, YAN Mao-Cang, MIAO Liang, TANG Biao, ZHANG Yan-Jun. 2021.
- 东海温州水域4类常见耐药基因的流行情况研究
- INVESTIGATION ON RESISTANCE GENES AGAINST FOUR TYPES OF COMMON ANTIBIOTICS IN WENZHOU WATERS OF THE EAST CHINA SEA
- 海洋与湖沼, 52(4): 947-959
- Oceanologia et Limnologia Sinica, 52(4): 947-959.
- http://dx.doi.org/10.11693/hyhz20210200040
文章历史
-
收稿日期:2021-02-06
收修改稿日期:2021-03-31
2. 宁波大学海洋学院 宁波 315832;
3. 浙江省海洋水产养殖研究所 浙江省近岸水域生物资源开发与保护重点实验室 温州 325005;
4. 浙江省农业科学院 农产品质量标准研究所 杭州 310021;
5. 浙江省疾病预防控制中心 杭州 310051
2. School of Marine Science, Ningbo University, Ningbo 315832, China;
3. Zhejiang Key Laboratory of Exploitation and Preservation of Coastal Bio-resource, Zhejiang Mariculture Research Institute, Wenzhou 325005, China;
4. Institute of Quality and Standard for Agro-Products, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China;
5. Zhejiang Provincial Center for Disease Control and Prevention, Hangzhou 310051, China
由于抗生素的常年使用, 未代谢的抗生素在环境中积累和转化, 对环境中的微生物进行持续不断的选择, 造成耐药基因与耐药菌的流行, 对公共卫生与生态环境造成了巨大的威胁(Arias et al, 2009; Kennedy, 2013; Willyard, 2017)。海洋作为地球上耐药基因与耐药菌的主要储存库之一, 不但自身拥有丰富的耐药基因与耐药菌, 其他来源的耐药菌与耐药基因还可能通过雨水冲刷、潮汐、污水排放等方式进入海洋环境(Chen et al, 2013; Hatosy et al, 2015)。近海水域作为人类活动的主要区域之一, 来自于畜牧与水产养殖、临床医疗、工业生产活动的抗生素残留、耐药菌、耐药基因等污染物可通过一系列方式传入其中, 增加近海水域中耐药基因与耐药菌的种类与丰度, 对海洋生态环境造成危害。此外, 耐药基因还可能通过菌种之间的遗传物质传递从而传入海洋致病菌中, 增加了海洋耐药病原菌病害发生与流行的可能性(Normark et al, 2002)。
温州海域面积约11 000 km2, 海岸线长355 km, 拥有436个岛屿, 是我国重要的海水养殖区, 做好温州近海水域耐药基因的监控与评估, 对温州海水养殖环境及公共卫生健康均有重要的意义。针对温州地区陆地环境中耐药基因的流行情况, 有学者进行过一定的调查分析。严春等(2016)从医院重症监护室中分离到携带有SHV、TEM、CTX-M-9等超广谱β-内酰胺酶基因的肺炎克雷伯菌; Liao等(2020)从病患体内分离的大肠杆菌中检测到多粘菌素耐药基因MCR-1; Lu等(2017)则通过荧光定量PCR方法从猪粪中检测到磺胺类、四环素类、喹诺酮类抗生素的耐药基因及多种β-内酰胺酶基因。同时, 也有个别学者对温州海洋水体环境和海产品中耐药基因的分布情况做过调查。Chen等(2017)调查发现温州海水养殖水域有着较高的磺胺类抗生素残留, 利用荧光定量PCR方法检测到较高丰度Sul2基因污染; 吴越等(2020)对温州水产品上分离的哈维氏弧菌进行耐药分析, 从中检测到了磺胺类抗生素耐药基因Sul1和Sul2, 四环素耐药基因Tet34、Tet35及Tet36, 喹诺酮类耐药基因QnrD等, 且多数菌株出现多重耐药现象。本研究通过抗性平板法于温州近海水域海水中分别筛选了磺胺类、四环素类、喹诺酮类抗生素抗性的可培养耐药菌分离株, 通过PCR方法检测耐药菌对应的耐药基因携带情况, 在含有目标耐药基因的耐药菌分离株中进一步测试多粘菌素耐药基因的污染情况, 综合分析四类抗生素耐药基因在温州近海水域的分布; 利用16S rDNA测序, 鉴定耐药菌分离株的种属特性, 分析耐药基因在不同种属细菌中的分布; 同时, 通过微量二倍稀释法测定耐药菌对受试抗生素的最小抑菌浓度(Minimal Inhibit Concentration, MIC), 分析其多重耐药现象, 研究将为评估温州近海水域的细菌耐药性风险, 规范抗生素的使用提供科学依据。
1 材料与方法 1.1 样品采集于2019—2020年冬、春两季, 沿东海温州近海水域, 选择25个采样点进行海水样品的采集(表 1)。每个采样地点以采水器采集表层海水样品(采样深度: 水面下15 cm) 1 500 mL置于2.5 L的无菌聚乙烯扁方桶中。采集的海水样品在16—20 ℃下运输并贮存, 用于后续的实验室分析。
| 样品采集地点 | 温州市区 | 乐清市 | 平阳县 | 苍南县 | 小计 | |
| 龙湾区 | 洞头区 | |||||
| 采样点数量 | 1 | 5 | 12 | 2 | 5 | 25 |
每份样品, 分别取500 mL海水样品, 经过0.22 μm的无菌混合纤维素滤膜(生工生物工程(上海)股份有限公司, 中国)过滤以收集细菌。在无菌条件下将滤膜剪碎, 放置于10 mL的无菌磷酸缓冲盐溶液(Phosphate Buffer Saline, PBS)中, 经涡旋振荡器充分震荡后, 吸取50 μL分别涂布于含有不同种类抗生素(磺胺类: 4 μg/mL甲氧苄啶; 四环素类: 16 μg/mL四环素; 喹诺酮类: 1 μg/mL环丙沙星)的2216E琼脂(青岛海博生物技术有限公司, 中国)平板上, 并置于28 ℃下培养16 h。根据菌落的形态、颜色、大小等形态学特征于每个平板上差异挑选一定数量的单菌落分别接种至2216E液体培养基(青岛海博生物技术有限公司, 中国)中过夜培养。由于某些细菌生长较为缓慢, 此后每隔12 h进行菌株的补筛。耐药分离株使用终浓度25%的甘油保存于–80 ℃用于后续实验。
1.3 目标耐药基因与16S rDNA的分子鉴定针对上述3类抗生素, 我们分别选择了在海洋环境中较为普遍的耐药基因, 对收集到的耐药分离株进行耐药基因的鉴定(表 2), 然后对耐药基因阳性的耐药分离株进行其他两类抗生素耐药基因的鉴定。在前期实验中, 无法利用抗性平板从多粘菌素耐药的分离株鉴定到耐药基因MCR, 而文献报道携带MCR基因的多重耐药质粒的水平传播是引起MCR基因流行的主要原因(Sun et al, 2018), 因此, 我们对磺胺类、四环素类和喹诺酮类耐药基因阳性的耐药分离株进行MCR基因的测定, 以期能够获得多粘菌素类抗生素耐药基因的流行情况。耐药基因阳性的耐药分离株通过16S rDNA序列测定进一步确定其种属, 引物信息见表 2。鉴定过程的操作简述如下: 将步骤1.2中冻存的耐药分离株复苏后, 使用煮沸法粗提细菌的基因组DNA。利用PCR方法, 按照文献提供的方法克隆耐药分离株的对应耐药基因片段(Lane, 1991; Aminov et al, 2001; Ng et al, 2001; Park et al, 2006; Hoa et al, 2008; Rutgersson et al, 2014; Rebelo et al, 2018; Borowiak et al, 2020)。PCR产物经过1.5%的琼脂糖凝胶电泳检测后送至上海生工生物工程有限公司测序, 测序结果上传至NCBI的BLAST检索系统进行同源性对比分析。对含有目标耐药基因的耐药分离株, 按照文献方法扩增16S rDNA基因序列, PCR产物经上述方法鉴定后送至上海生工生物工程有限公司进行测序, 结果同样上传至NCBI的BLAST检索系统进行同源性对比分析。
| 引物名称 | 引物序列(5′—3′) | 靶标基因 | 产物长度(bp) | 退火温度(℃) | 耐药类型 | 参考文献 |
| sul1-F | CGGCGTGGGCTACCTGAACG | Sul1 | 433 | 55 | 磺胺类抗生素 | Hoa et al, 2008 |
| sul1-R | GCCGATCGCGTGAAGTTCCG | |||||
| sul2-F | GCGCTCAAGGCAGATGGCATT | Sul2 | 293 | |||
| sul2-R | GCGTTTGATACCGGCACCCGT | |||||
| sul3-F | TCAAAGCAAAATGATATGAGC | Sul3 | 787 | |||
| sul3-R | TTTCAAGGCATCTGATAAAGAC | |||||
| tet(A)-F | GCTACATCCTGCTTGCCTTC | TetA | 210 | 四环素类抗生素 | Aminov et al, 2001; Ng et al, 2001 | |
| tet(A)-R | CATAGATCGCCGTGAAGAGG | |||||
| tet(B)-F | TTGGTTAGGGGCAAGTTTTG | TetB | 659 | |||
| tet(B)-R | GTAATGGGCCAATAACACCG | |||||
| tet(C)-F | CTTGAGAGCCTTCAACCCAG | TetC | 418 | |||
| tet(C)-R | ATGGTCGTCATCTACCTGCC | |||||
| tet(D)-F | AAACCATTACGGCATTCTGC | TetD | 787 | |||
| tet(D)-R | GACCGGATACACCATCCATC | |||||
| tet(E)-F | AAACCACATCCTCCATACGC | TetE | 278 | |||
| tet(E)-R | AAATAGGCCACAACCGTCAG | |||||
| tet(G)-F | GCTCGGTGGTATCTCTGCTC | TetG | 468 | |||
| tet(G)-R | AGCAACAGAATCGGGAACAC | |||||
| tet(L)-F | TCGTTAGCGTGCTGTCATTC | TetL | 844 | |||
| tet(L)-R | GTATCCCACCAATGTAGCCG | |||||
| tet(M)a-F | GTGGACAAAGGTACAACGAG | TetMa | 406 | |||
| tet(M)a-R | CGGTAAAGTTCGTCACACAC | |||||
| tet(W)-F | GAGAGCCTGCTATATGCCAGC | TetW | 168 | 64 | ||
| tet(W)-R | GGGCGTATCCACAATGTTAAC | |||||
| qnrA-F | TGCCAACTGCTTTGGCATAGA | QnrA | 153 | 60 | 喹诺酮类抗生素 | Park et al, 2006; Rutgersson et al, 2014 |
| qnrA-R | TGGCCACTCAAGTTGGTATAGG | |||||
| qnrB-F | CTTCACACATTGCGATCTGAC | QnrB | 100 | |||
| qnrB-R | CAACGATGCCTGGTAGTTGT | |||||
| qnrC-F | TGCAGACCTACGAGATGCTT | QnrC | 95 | |||
| qnrC-R | CGCATTCTCTCAATTCAAGG | |||||
| qnrD-F | AAGTGCGAACTGTGGGAAA | QnrD | 112 | |||
| qnrD-R | CAGCCAAAGACCAATCAAAC | |||||
| qnrS-F | GATATCGAAGGCTGCCACTT | QnrS | 115 | |||
| qnrS-R | CACGGAACTCTATACCGTAGCA | |||||
| qnrVC-F | GCTCAAACCTCCGAGATACAC | QnrVC | 99 | |||
| qnrVC-R | AAGCATCTCGAAGATCAGCAT | |||||
| aac(6')-Ib-cr-F | TTGCGATGCTCTATGAGTGGCTA | aac(6')-Ib-cr | 482 | 55 | ||
| aac(6')-Ib-cr-R | CTCGAATGCCTGGCGTGTTT | |||||
| MCR-1-F | AGTCCGTTTGTTCTTGTGGC | MCR-1 | 320 | 58 | 多粘菌素类抗生素 | Borowiak et al, 2020; Rebelo et al, 2018 |
| MCR-1-R | AGATCCTTGGTCTCGGCTTG | |||||
| MCR-2-F | CAAGTGTGTTGGTCGCAGTT | MCR-2 | 715 | |||
| MCR-2-R | TCTAGCCCGACAAGCATACC | |||||
| MCR-3-F | AAATAAAAATTGTTCCGCTTATG | MCR-3 | 929 | |||
| MCR-3-R | AATGGAGATCCCCGTTTTT | |||||
| MCR-4-F | TCACTTTCATCACTGCGTTG | MCR-4 | 1 116 | |||
| MCR-4-R | TTGGTCCATGACTACCAATG | |||||
| MCR-5-F | ATGCGGTTGTCTGCATTTATC | MCR-5 | 1 644 | |||
| MCR-5-R | TCATTGTGGTTGTCCTTTTCTG | |||||
| MCR-6-F | AGCTATGTCAATCCCGTGAT | MCR-6 | 252 | 55 | ||
| MCR-6-R | ATTGGCTAGGTTGTCAATC | |||||
| MCR-7-F | GCCCTTCTTTTCGTTGTT | MCR-7 | 551 | |||
| MCR-7-R | GGTTGGTCTCTTTCTCGT | |||||
| MCR-8-F | TCAACAATTCTACAAAGCGTG | MCR-8 | 856 | |||
| MCR-8-R | AATGCTGCGCGAATGAAG | |||||
| MCR-9-F | TTCCCTTTGTTCTGGTTG | MCR-9 | 1 011 | |||
| MCR-9-R | GCAGGTAATAAGTCGGTC | |||||
| 16S-27F | AGAGTTTGATCCTGGCTCAG | 16S rDNA | 约1 400 bp | 52 | Lane, 1991 | |
| 16S-1492R | GGTTACCTTGTTACGACTT |
对含有目标耐药基因的耐药分离株进行药物敏感性实验。药物敏感性实验采用微量二倍稀释法, 参照美国临床和实验室标准协会(Clinical and Laboratory Standards Institute, CLSI)的标准判断耐药分离株的药物敏感性, 以大肠杆菌(Escherichia coli) ATCC25922作为质控菌株。在选择目标耐药基因所对应的抗生素时, 磺胺类抗生素选用甲氧苄啶, 四环素类抗生素选用四环素, 喹诺酮类抗生素选用环丙沙星, 多粘菌素类抗生素选用多粘菌素E。同时, 在96孔板上设置无药无菌的阴性对照与无药有菌的阳性对照。于每个孔中加入上述稀释至1×104 cfu/mL的菌液5 μL, 置于28 ℃培养箱中静置16—24 h后, 取出96孔板置于酶标仪上测定A600的吸光度值(optical density, OD)。在阳性对照生长良好, 阴性对照未有无污染时, 以OD值判断能够抑制细菌生长的最低药物浓度作为该菌株的最小抑菌浓度。同时根据文献对于多重耐药菌(multidrug-resistant organism, MDRO)的定义, 对含有目标耐药基因的耐药分离株进行多重耐药分析, 统计其多重耐药率(Magiorakos et al, 2012)。
2 实验结果 2.1 耐药分离株的分离鉴定如表 3所示, 从25个采样点的海水样品中共分离纯化获得耐药分离株1 605株, 通过分子鉴定, 共计51株耐药分离株检测出目标耐药基因, 占总耐药分离株数量的3.18%。51株耐药分离株中, 从平阳县邻近水域分离29株, 占该地区耐药分离株总数的19.33%。其中23株检出Sul1, 检出率为15.33%; 21株检出TetE, 检出率为14.00%; 1株检出MCR-3, 检出率为0.67%; 4株检出aac(6')-Ib-cr, 6株检出QnrS, 检出率为6.67%。苍南县邻近水域分离20株, 占该地区耐药分离株总数的5.33%。其中10株检出Sul1, 检出率为2.6%; 15株检出TetE, 检出率为4.00%; 1株检出MCR-3, 检出率为0.27%; 7株检出aac(6')-Ib-cr, 检出率为1.87%。乐清市邻近水域分离2株, 占该地区耐药分离株总数的0.28%, 2株均检测出Sul2, 检出率为0.28%。
| 项目 | 温州市区 | 乐清市 | 平阳县 | 苍南县 | 总计 | |
| 龙湾区 | 洞头区 | |||||
| 合计 | 60 | 300 | 720 | 150 | 375 | 1 605 |
| 含有耐药基因的耐药分离株数目 | 2 | 29 | 20 | 51 | ||
| 占比(%) | 0.28 | 19.33 | 5.33 | 3.18 | ||
| 磺胺类耐药基因 | Sul2 (2) | Sul1 (23) | Sul1 (10) | 35 | ||
| 四环素类耐药基因 | TetE (21) | TetE (15) | 36 | |||
| 多粘菌素类耐药基因 | MCR-3 (1) | MCR-3 (1) | 2 | |||
| 喹诺酮类耐药基因 | aac(6')-Ib-cr (4), QnrS (6) | aac(6')-Ib-cr (7) | 17 | |||
利用平板法, 51株含有目标耐药基因的耐药分离株中, 磺胺类耐药基因检测到Sul1和Sul2, 其中Sul1检出33株, 占比68.63%, Sul2检出2株, 占比3.92%; 四环素类耐药基因检测到TetE, 共计36株, 占比70.56%; 多粘菌素类耐药基因检测到MCR-3, 共2株, 占比3.92%; 喹诺酮类耐药基因检测出aac(6')-Ib-cr和QnrS, 其中aac(6')-Ib-cr检出10株, 占比19.61%, QnrS检出6株, 占比11.76%。具体如表 4所示。
| 含有目标耐药基因的耐药分离株种属 | 分离株数目 | 磺胺类耐药基因 | 四环素类耐药基因 | 多粘菌素耐药基因 | 喹诺酮类耐药基因 | ||||||
| 株数 | 占比(%) | 株数 | 占比(%) | 株数 | 占比(%) | 株数 | 占比(%) | ||||
| 维氏气单胞菌 (Aeromonas veronii) |
1 | Sul1 (1) | 1.96 | TetE (1) | 1.96 | MCR-3 (1) | 1.96 | aac(6')-Ib-cr(1) | 1.96 | ||
| 豚鼠气单胞菌 (Aeromonas caviae) |
9 | Sul1 (9) | 17.65 | TetE (6) | 11.76 | MCR-3 (1) | 1.96 | aac(6')-Ib-cr(2) | 3.92 | ||
| 嗜水气单胞菌 (Aeromonas hydrophila) |
2 | Sul1 (2) | 3.92 | aac(6')-Ib-cr(1) | |||||||
| 中间气单胞菌 (Aeromonas media) |
10 | Sul1 (7) | 13.73 | TetE (9) | 17.65 | aac(6')-Ib-cr(3), QnrS(1) | 7.84 | ||||
| 杀鲑气单胞菌 (Aeromonas salmonicida) |
1 | Sul1 (1) | 1.96 | TetE (1) | 1.96 | aac(6')-Ib-cr(1) | 1.96 | ||||
| 不确定种气单胞菌属 (Aeromonas sp.) |
20 | Sul1 (8) | 15.69 | TetE (16) | 31.37 | aac(6')-Ib-cr(3), QnrS(5) | 15.69 | ||||
| 大肠杆菌(Escherichia coli) | 1 | Sul1 (1) | 1.96 | ||||||||
| 希瓦氏菌属(Shewanella sp.) | 2 | Sul1 (2) | 3.92 | TetE (2) | 3.92 | ||||||
| 弗氏柠檬酸杆菌 (Citrobacter freundii) |
2 | Sul1 (2) | 3.92 | ||||||||
| Thalassobius sp. | 1 | Sul2 (1) | 1.96 | ||||||||
| 大西洋美女神菌 (Amphritea atlantica) |
1 | Sul2 (1) | 1.96 | ||||||||
| 类芽孢杆菌属 (Paenibacillus sp.) |
1 | TetE (1) | 1.96 | ||||||||
| 总计 | 51 | Sul1 (33), Sul2 (2) | 68.63 | TetE (36) | 70.56 | MCR-3 (2) | 3.92 | aac(6')-Ib-cr(11), QnrS(6) | 31.37 | ||
51株含有目标耐药基因的耐药分离株, 通过16S rDNA序列分析鉴定为7个不同属, 分别为气单胞菌属(Aeromonas sp.)、埃希氏菌属(Escherichia sp.)、希瓦氏菌属(Shewanella sp.)、枸橼酸杆菌属(Citrobacter sp.)、Thalassobius sp.、Amphritea sp.、类芽孢杆菌属(Paenibacillus sp.)。其中, 气单胞菌属共计43株, 占比84.32%, 包括维氏气单胞菌1株, 占比1.96%, 豚鼠气单胞菌9株, 占比17.65%, 嗜水气单胞菌2株, 占比3.92%, 中间气单胞菌10株, 占比19.61%, 杀鲑气单胞菌1株, 占比1.96%, 不确定种的气单胞菌属细菌20株, 占比39.22%; 希瓦氏菌属2株, 占比3.92%; 埃希氏菌属1株, 为大肠杆菌, 占比1.96%; 枸橼酸杆菌属2株, 为弗氏柠檬酸杆菌, 占比3.92%; Thalassobius sp.和类芽孢杆菌属各有1株, 占比为3.92%; Amphritea sp.属的大西洋美女神菌1株, 占比1.96%。耐药基因主要分布于气单胞菌属细菌中(图 1, 表 4)。此外, 对51株耐药分离株进一步分析发现2株中间气单胞菌, 1株杀鲑气单胞菌和2株其他不确定种的气单胞菌中同时检出Sul1、TetE与aac(6')-Ib-cr, 1株维氏气单胞菌和1株豚鼠气单胞菌中同时检出Sul1、TetE、MCR-3与aac(6')-Ib-cr (图 1, 表 4)。具体如表 4所示。从区域上归类, 可以发现上述51株耐药分离株中, 苍南县邻近水域20株, 平阳县邻近水域29株, 乐清市邻近水域2株, 温州其他县区邻近水域未检出目标耐药基因阳性的耐药分离株(表 5)。
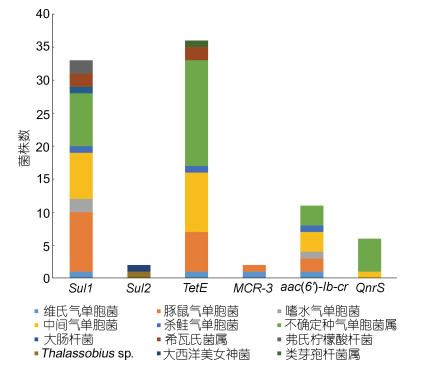 |
| 图 1 51株耐药分离株耐药基因的分布 Fig. 1 Distribution of target antibiotic resistance genes in 51 antibiotic resistant isolates 注: 横坐标代表不同种类的耐药基因, 纵坐标代表含有该耐药基因的耐药分离株数, 柱状图中不同颜色的色块表示不同种属的耐药分离株, 高度代表耐药分离株数目 |
| 含有目标耐药基因的耐药分离株种属 | 乐清市 | 平阳县 | 苍南县 | 小计 | |||||||
| 株数 | 占比(%) | 株数 | 占比(%) | 株数 | 占比(%) | 株数 | 占比(%) | ||||
| 维氏气单胞菌(Aeromonas veronii) | 1 | 3.45 | 1 | 1.96 | |||||||
| 豚鼠气单胞菌(Aeromonas caviae) | 7 | 24.14 | 2 | 10.00 | 9 | 17.65 | |||||
| 嗜水气单胞菌(Aeromonas hydrophila) | 1 | 3.45 | 1 | 5.00 | 2 | 3.92 | |||||
| 中间气单胞菌(Aeromonas media) | 7 | 24.14 | 3 | 15.00 | 10 | 19.61 | |||||
| 杀鲑气单胞菌(Aeromonas salmonicida) | 1 | 5.00 | 1 | 1.96 | |||||||
| 不确定种气单胞菌属(Aeromonas sp.) | 9 | 31.03 | 11 | 55.00 | 20 | 39.22 | |||||
| 大肠杆菌(Escherichia coli) | 1 | 3.45 | 1 | 1.96 | |||||||
| 希瓦氏菌属(Shewanella sp.) | 1 | 3.45 | 1 | 5.00 | 2 | 3.92 | |||||
| 弗氏柠檬酸杆菌(Citrobacter freundii) | 2 | 6.90 | 2 | 3.92 | |||||||
| Thalassobius sp. | 1 | 50 | 1 | 1.96 | |||||||
| 大西洋美女神菌(Amphritea atlantica) | 1 | 50 | 1 | 1.96 | |||||||
| 类芽孢杆菌属(Paenibacillus sp.) | 1 | 5.00 | 1 | 1.96 | |||||||
| 合计 | 2 | 100.00 | 29 | 100.00 | 20 | 100.00 | 51 | 100.00 | |||
尽管在使用相应浓度的抗生素测试时, Thalassobius sp.分离株和大西洋美女神菌仍旧表现出耐药性, 但是由于在CLSI的标准中没有这2株分离株的耐药标准, 故相关数据计算时, 未列入其中。49株含有目标耐药基因的耐药分离株对4种抗生素的药物敏感性试验结果如图 2和表 6所示: 仅对1种抗生素耐药的分离株有2株, 占比4.08%, 对2种抗生素耐药的分离株有8株, 占比16.33%, 对3种抗生素耐药的分离株有16株, 占比32.65%, 对4种抗生素耐药的分离株有23株, 占比46.94%。其中, 苍南县邻近水域有10株, 占本地区分离总数的50.00%, 平阳县邻近水域有13株, 占本地区分离株总数的48.82% (表 6)。49株耐药分离株均对四环素耐药, 41株耐药分离株对多粘菌素E耐药, 占比83.67%; 34株耐药分离株对环丙沙星耐药, 占比69.39%; 34株耐药分离株对甲氧苄啶耐药, 占比69.39%。多数条件致病菌(如豚鼠气单胞菌、维氏气单胞菌、中间气单胞菌、杀鲑气单胞菌、嗜水气单胞菌、大肠杆菌等)对3类以上抗生素具有较高的MIC值(环丙沙星MIC > 1 μg/mL; 多粘菌素E的MIC > 8 μg/mL; 四环素MIC > 16 μg/mL; 甲氧苄啶MIC > 64 μg/mL) (图 3), 不同来源的耐药分离株在四种抗生素的MIC分布上未见显著差别(图 4)。
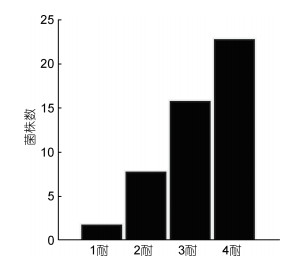 |
| 图 2 含有目标基因的耐药分离株的多重耐药分析结果 Fig. 2 Analysis of multi-resistant of antibiotic resistant isolates containing target antibiotic resistance genes 注: 1耐: 对一种抗生素有耐药性; 2耐: 对两种抗生素有耐药性; 3耐: 对三种抗生素有耐药性; 4耐: 对四种抗生素有耐药性 |
| 编号 | 来源地 | MIC (μg/mL) | |||
| 环丙沙星 | 多粘菌素E | 四环素 | 甲氧苄啶 | ||
| Y-1 | 苍南县 | 0.500 | 16.000 | 64.000 | 128.000 |
| Y-2 | 0.500 | 16.000 | 128.000 | 128.000 | |
| Y-3 | 0.500 | 8.000 | 64.000 | 256.000 | |
| Y-4 | 4.000 | 8.000 | 128.000 | 128.000 | |
| Y-5 | 4.000 | 16.000 | 128.000 | 64.000 | |
| Y-6 | 0.500 | 16.000 | 128.000 | 128.000 | |
| Y-7 | 0.500 | 16.000 | 32.000 | 16.000 | |
| Y-8 | 1.000 | 8.000 | 64.000 | < 1.000 | |
| Y-9 | 0.250 | 4.000 | 128.000 | 64.000 | |
| Y-10 | 2.000 | 4.000 | 64.000 | 128.000 | |
| Y-11 | 8.000 | 64.000 | 64.000 | 64.000 | |
| Y-12 | 64.000 | 16.000 | 64.000 | 128.000 | |
| Y-13 | 16.000 | 16.000 | 64.000 | 256.000 | |
| Y-14 | 64.000 | 8.000 | 64.000 | 128.000 | |
| Y-15 | 8.000 | 16.000 | 128.000 | 256.000 | |
| Y-16 | 2.000 | 16.000 | 32.000 | 256.000 | |
| Y-17 | 4.000 | 16.000 | 128.000 | 128.000 | |
| Y-18 | 4.000 | 16.000 | 64.000 | > 512.000 | |
| Y-19 | 8.000 | 16.000 | 256.000 | 256.000 | |
| Y-20 | 2.000 | 8.000 | 128.000 | 128.000 | |
| Q-1 | 平阳县 | < 0.125 | 32.000 | 32.000 | 64.000 |
| Q-2 | 1.000 | 16.000 | 128.000 | 128.000 | |
| Q-3 | 2.000 | 4.000 | 64.000 | 128.000 | |
| Q-4 | 64.000 | 4.000 | 32.000 | 64.000 | |
| Q-5 | 2.000 | 32.000 | 64.000 | 512.000 | |
| Q-6 | 1.000 | 8.000 | 32.000 | 64.000 | |
| Q-7 | 4.000 | 2.000 | 128.000 | 256.000 | |
| Q-8 | 2.000 | 2.000 | 32.000 | < 1.000 | |
| Q-9 | 0.250 | 16.000 | 128.000 | 128.000 | |
| Q-10 | < 0.125 | 8.000 | 128.000 | 64.000 | |
| Q-11 | 2.000 | 8.000 | 128.000 | > 512.000 | |
| Q-12 | 2.000 | 4.000 | 64.000 | 256.000 | |
| Q-13 | 32.000 | 8.000 | 64.000 | 256.000 | |
| Q-14 | 64.000 | 16.000 | 32.000 | 256.000 | |
| Q-15 | 1.000 | 2.000 | 32.000 | 64.000 | |
| Q-16 | 4.000 | 16.000 | 128.000 | 256.000 | |
| Q-17 | 16.000 | 16.000 | 128.000 | 128.000 | |
| Q-18 | 16.000 | 8.000 | 32.000 | 256.000 | |
| Q-19 | > 64.000 | 16.000 | 32.000 | 64.000 | |
| Q-20 | 0.250 | 16.000 | 16.000 | 256.000 | |
| Q-21 | 4.000 | 16.000 | 128.000 | 256.000 | |
| Q-22 | 2.000 | 16.000 | 128.000 | 256.000 | |
| Q-23 | 16.000 | 16.000 | 128.000 | 128.000 | |
| Q-24 | > 64.000 | 16.000 | 128.000 | 256.000 | |
| Q-25 | > 64.000 | 32.000 | 128.000 | 64.000 | |
| Q-26 | > 64.000 | 16.000 | 128.000 | 256.000 | |
| Q-27 | 8.000 | 16.000 | 512.000 | 128.000 | |
| X-1 | 0.500 | 16.000 | 32.000 | < 1.000 | |
| X-2 | 2.000 | 32.000 | 64.000 | 16.000 | |
| 100-10 | 乐清市 | 64.000 | 128.000 | 128.000 | 256.000 |
| 95-10 | 0.500 | 64.000 | 32.000 | 128.000 | |
| 占比 | 69.39% | 83.67% | 100.00% | 69.39% | |
| 注: 统计时, 排除了1株Thalassobius与1株大西洋美女神菌(Amphritea atlantica) | |||||
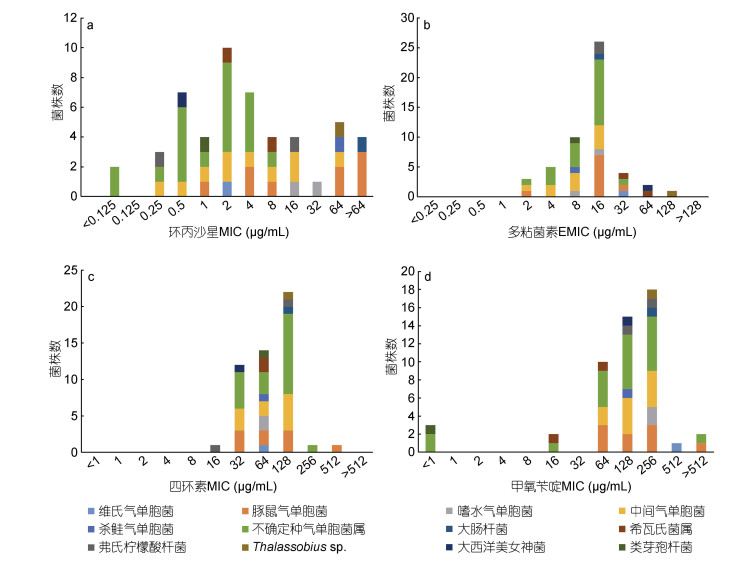 |
| 图 3 51株耐药分离株的四种抗生素的MIC分布 Fig. 3 MIC distribution of the 51 antibiotic resistant isolates 注: 51株不同种属的耐药分离株对: 环丙沙星(a)、多粘菌素E(b)、四环素(c)、甲氧苄啶(d)的MIC分布。横坐标代表耐药分离株对该种抗生素的MIC值, 纵坐标代表耐药分离株的数目, 柱状图中不同颜色的色块表示不同种属的耐药分离株, 其高度代表耐药分离株数目 |
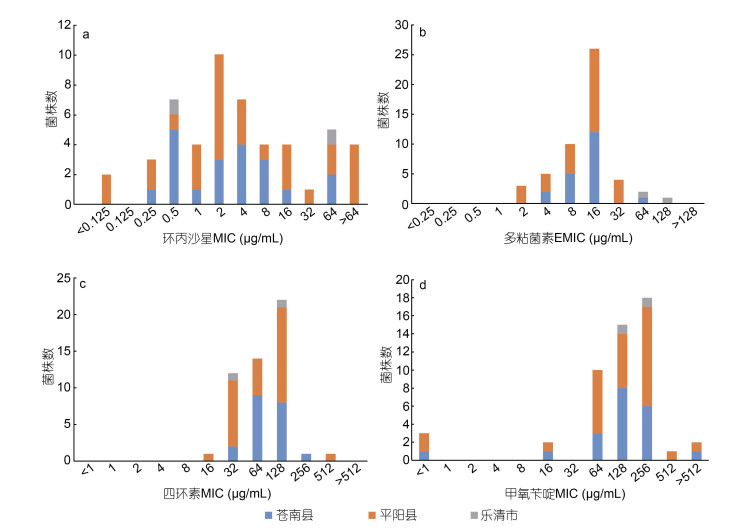 |
| 图 4 不同采样地点的耐药分离株的四种抗生素MIC分布差异分析 Fig. 4 MIC distribution of the antibiotic resistant isolates isolated from different sampling locations 注: 来自苍南县、平阳县及乐清市的耐药分离株对: 环丙沙星(a)、多粘菌素E(b)、四环素(b)、甲氧苄啶(d)的MIC分布差异。横坐标代表耐药分离株对该种抗生素的MIC值, 纵坐标代表耐药分离株的数目, 柱状图中不同颜色的色块表示不同来源的耐药分离株, 其高度代表耐药分离株数目 |
耐药基因和耐药菌能够经由多种途径进入近海水体, 扩散传播, 给海洋生态环境和公共卫生带来巨大威胁(Normark et al, 2002; Chen et al, 2013)。本文初步分析了东海温州近海水域可培养细菌中磺胺类、四环素类、喹诺酮类和多粘菌素类耐药基因的流行情况。研究通过抗性平板法筛选了耐药分离株1605株, 获得含目标耐药基因的51株, 分属于气单胞菌属等7个属, 分别携带有耐药基因Sul1、Sul2、TetE、MCR-3、aac(6')-Ib-cr以及QnrS等, 其中39株存在多重耐药现象; 此外, 研究还分离获得了携带多粘菌素耐药基因MCR-3的条件致病菌维氏气单胞菌和豚鼠气单胞菌各1株, 表明多粘菌素的耐药基因在海洋水体中有流行。
上述51株含目标耐药基因的耐药菌集中发现于平阳县和苍南县邻近水域(分别是29、20株), 乐清市邻近水域有少量发现(2株), 表明尽管温州地区近海水域整体耐药基因流行水平低(含有目标耐药基因的耐药菌占比仅为3.18%), 但相对于温州其他海域, 平阳县与苍南县邻近海域耐药基因流行情况要更为突出。据温州市统计局的资料, 平阳县海水养殖规模很小, 畜牧业不发达, 其近海水域与西湾风景区相邻, 景区较高的人流量与景区内部小而分散的社区医院可能是其耐药基因与耐药菌的重要来源; 苍南县是温州海水与畜牧养殖的主要县市之一, 本研究苍南邻近水域的采样点位于河口附近, 上游的养殖排放物容易在此沉积, 一定规模的养殖和特殊的地理环境可能是苍南县采样点耐药基因流行的重要原因。
已有研究发现, 海水环境中存在多种四环素类耐药基因(包括TetA、TetB、TetC、TetD、TetE、TetM等), 耐药基因的分布差异与地理环境及细菌耐药性的进化有关(Kim et al, 2004; Balassiano et al, 2007; Nikolakopoulou et al, 2008)。国内海水样品与水产品源细菌中检出的四环素耐药基因类型多为TetA、TetB等(赵敏等, 2014; 王钥等, 2015), 国外发现从食品中分离的气单胞菌和养殖海水与底泥中能大量检出TetE (Balassiano et al, 2007; Tamminen et al, 2011)。本研究从其他1605株耐药分离株中检测出含有TetE的耐药分离株36株, 耐药基因检出率上低于国内已有报道(王钥等, 2015; 吴越等, 2020)。耐药基因Sul1、Sul2和Sul3是介导磺胺类抗生素耐药的主要原因之一(Hoa et al, 2008)其中, Sul1与Sul2是国内养殖海水与水产品中主要流行的磺胺类耐药基因, Sul1的流行与耐药基因在细菌间的转移有关(马辰婕等, 2017; 汪涛等, 2017; 吴越等, 2020)。本研究中检测到的磺胺类耐药基因以Sul1为主(平阳县23株, 苍南县10株), 有少量的Sul2 (乐清市2株), 未检测到Sul3基因, 在耐药基因类型上与已有报道一致, 但是耐药基因的检出率低于已有报道(黄志坚等, 2012; 吴越等, 2020)。上述结果表明温州近海水域中磺胺类与四环素类耐药基因流行情况并不严重, 但集中出现在平阳和苍南两个近海区域。喹诺酮类抗生素的耐药情况在不同种属与不同来源的细菌中差异较大。本研究从平阳县临近水域检测到含有喹诺酮类耐药基因的耐药分离株11株, 苍南县临近水域7株, 低于已有报道, 可能与不同的药物使用策略有关(Zhao et al, 2012; 张舒婷等, 2019; 赵姝等, 2019)。喹诺酮类耐药基因QnrS与aac(6')-Ib-cr均能够介导较低水平的喹诺酮类药物耐药, 但是携带QnrS会提高细菌染色体自发突变的频率, 从而导致高水平的喹诺酮类耐药细菌的产生(Martínez et al, 1998), 而aac(6')-Ib-cr同时具有氨基糖苷类与喹诺酮类耐药活性, 增加了细菌在抗菌药物选择下的适应能力, 说明在药物使用策略上要减少喹诺酮类药物的使用并且避免与氨基糖苷类药物的联用, 防止高水平喹诺酮类药物耐药菌的出现和aac(6')-Ib-cr基因的流行。另外, 多重耐药质粒和携带多种耐药基因的整合子是导致细菌多重耐药的主要原因(马辰婕等, 2017; Sun et al, 2018)。本研究于平阳和苍南邻近水域筛选出至少含有3类耐药基因的多重耐药分离株39株(占比达79.59%), 暗示该区域的多种耐药基因可能集中在整合子或质粒等载体上传播, 提升细菌对多种抗生素的耐药能力。
水产养殖中通常很少使用多粘菌素类药物, 部分资料表明水产中通常只有弧菌具有较高的多粘菌素耐药性(刘开放等, 2017; 张西萌等, 2020)。质粒介导的可移动的多粘菌素耐药基因MCR-1至MCR-10等是目前认为介导多粘菌素耐药的主要因素之一, 使得多粘菌素的使用受到极大限制(Sun et al, 2018; Borowiak et al, 2020)。海洋水体中MCR系列基因的报道相对较少, MCR-3与MCR-5在气单胞菌中有检测到(Eichhorn et al, 2018; Ma et al, 2018)。本研究在平阳邻近水域检出含有MCR-3的维氏气单胞菌, 在苍南邻近水域检出含有MCR-3的豚鼠气单胞菌, 尽管与国外水产动物分离的气单胞菌结论一致(Eichhorn et al, 2018), 整体检出率也较低, 但MCR-3的来源是值得关注的问题。有研究认为水体环境是MCR-3的主要传播途径, 气单胞菌属是其重要的储存库(Eichhorn et al, 2018)。因此, 本研究在温州近海水域的两种气单胞菌中检出MCR-3, 可能是外源多粘菌素残留导致的定向选择结果。
我国近海海域或者水产养殖动物中分离的耐药菌一般以弧菌属与气单胞菌属为主(黄志坚等, 2012; Ye et al, 2013; Dong et al, 2019)。本研究发现温州近海水域分离的耐药菌以气单胞菌为主, 占比达到84.31%, 未发现弧菌属细菌, 表明气单胞菌属细菌可能具有高效的耐药基因转移方式, 成为该地区耐药基因传播的重要途径。在气单胞菌属细菌中, 海洋水体和水产养殖中常以维氏气单胞菌和嗜水气单胞菌为主, 畜禽养殖中多为豚鼠气单胞菌(黄志坚等, 2012; 邓玉婷等, 2019; 刘小芳等, 2021)。本研究鉴定的23株条件致病性气单胞菌属细菌中, 包括中间气单胞菌10株、豚鼠气单胞菌9株、嗜水气单胞菌2株、杀鲑气单胞菌和维氏气单胞菌各1株。其中, 豚鼠气单胞菌检出的数量较多, 与先前的报道有所差异(黄志坚等, 2012; 邓玉婷等, 2019), 可能是外源性细菌输入的结果。气单胞菌普遍存在于近海水域和水生动物体内, 可以作为温州近海水域耐药基因流行情况监测的指标微生物, 为该地区抗生素的合理使用提供科学参考。
4 结论本研究调查分析了2019—2020年期间东海温州近海水域磺胺类、四环素类、喹诺酮类以及多粘菌素类耐药基因和耐药菌的分布情况。对筛选获得的51株耐药基因阳性的耐药分离株分析发现, 对四类抗生素产生耐药的比例较高, 其中对四环素类抗生素的耐药比例最高, 达100%。同时, 尽管含耐药基因的耐药分离株检出率较低, 但同一耐药基因的细菌种类丰富, 但又以气单胞菌属细菌为主, 占比84.31%。多重耐药现象严重, 含3种及以上耐药基因的耐药菌占比76.47%。另外, 获得了多粘菌素耐药基因MCR-3阳性的条件致病菌维氏气单胞菌和豚鼠气单胞菌各1株。上述系列研究结果将为评估温州近海水域的细菌耐药性风险, 规范抗生素的使用提供科学依据。
马辰婕, 吴小梅, 林茂, 等. 2017. 水产养殖环境耐药细菌中复合1型整合子的流行特征. 微生物学通报, 44(9): 2089-2095 |
王玥, 苏洁, 明红霞, 等. 2015. 浴场海水中大肠杆菌耐药性及其耐药基因研究. 海洋学报, 37(12): 123-128 DOI:10.3969/j.issn.0253-4193.2015.12.013 |
邓玉婷, 谭爱萍, 张瑞泉, 等. 2019. 广东主要水产养殖地区气单胞菌耐药状况的调查分析. 南方农业学报, 50(11): 2375-2383 DOI:10.3969/j.issn.2095-1191.2019.11.01 |
刘小芳, 任燕, 张德锋, 等. 2021. 鱼源气单胞菌的毒力基因检测、分型及致病力. 水产学报, 45(3): 462-471 |
刘开放, 步君, 章翔, 等. 2017. 华南地区海水养殖水域中哈维氏弧菌的抗药性分析. 海南大学学报自然科学版, 35(2): 131-139 |
严春, 张颖, 何卫, 等. 2016. NICU患儿肺炎克雷伯菌耐药基因携带和同源性分析. 中国卫生检验杂志, 26(18): 2637-2640, 2645 |
汪涛, 杨再福, 陈勇航, 等. 2017. 磺胺类抗性基因的产生及演变研究进展. 环境污染与防治, 39(11): 1251-1255 |
吴越, 郑伊诺, 陆荣茂, 等. 2020. 温州地区水产品中哈维氏弧菌的耐药分析. 水产科学, 39(6): 837-843 |
张西萌, 付溥博, 魏海燕, 等. 2020. 北京进出口水产品中259株霍乱弧菌分离株的耐药性研究. 食品安全质量检测学报, 11(14): 4901-4906 |
张舒婷, 陈赛赛. 2019. 青岛海水浴场多重耐药菌的分布特征研究. 生物技术通报, 35(4): 57-63 |
赵姝, 李健, 马立才, 等. 2019. 海水养殖动物源弧菌喹诺酮类药物耐药表型与基因型分析. 海洋渔业, 41(4): 463-471 DOI:10.3969/j.issn.1004-2490.2019.04.008 |
赵敏, 汪开毓, 王均, 等. 2014. 斑点叉尾鮰源维氏气单胞菌对四环素类抗生素的耐药性及耐药基因的检测. 水生生物学报, 38(2): 386-392 |
黄志坚, 陈旭凌, 路晓峰, 等. 2012. 水产养殖生物和养殖环境细菌鉴定及抗生素抗性基因检测. 中山大学学报(自然科学版), 51(6): 92-96 |
Aminov R I, Garrigues-Jeanjean N, Mackie R I, 2001. Molecular ecology of tetracycline resistance: development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins. Applied and Environmental Microbiology, 67(1): 22-32 DOI:10.1128/AEM.67.1.22-32.2001 |
Arias C A, Murray B E, 2009. Antibiotic-resistant bugs in the 21st century-a clinical super-challenge. The New England Journal of Medicine, 360(5): 439-443 DOI:10.1056/NEJMp0804651 |
Balassiano I T, De Freire Bastos M D C, Madureira D J et al, 2007. The involvement of tetA and tetE tetracycline resistance genes in plasmid and chromosomal resistance of Aeromonas in Brazilian strains. Memorias Do Instituto Oswaldo Cruz, 102(7): 861-866 DOI:10.1590/S0074-02762007005000121 |
Borowiak M, Baumann B, Fischer J et al, 2020. Development of a Novel mcr-6 to mcr-9 Multiplex PCR and Assessment of mcr-1 to mcr-9 Occurrence in Colistin-Resistant Salmonella enterica Isolates from Environment, Feed, Animals and Food (2011-2018) in Germany. Frontiers in Microbiology, 11: 80 DOI:10.3389/fmicb.2020.00080 |
Chen B W, Yang Y, Liang X M et al, 2013. Metagenomic profiles of antibiotic resistance genes (ARGs) between human impacted estuary and deep ocean sediments. Environmental Science & Technology, 47(22): 12753-12760 |
Chen C Q, Zheng L, Zhou J L et al, 2017. Persistence and risk of antibiotic residues and antibiotic resistance genes in major mariculture sites in Southeast China. Science of the Total Environment, 580: 1175-1184 DOI:10.1016/j.scitotenv.2016.12.075 |
Dong P Y, Cui Q J, Fang T T et al, 2019. Occurrence of antibiotic resistance genes and bacterial pathogens in water and sediment in urban recreational water. Journal of Environmental Sciences, 77: 65-74 DOI:10.1016/j.jes.2018.06.011 |
Eichhorn I, Feudi C, Wang Y et al, 2018. Dentification of novel variants of the colistin resistance gene mcr-3 in Aeromonas spp. from the national resistance monitoring programme GERM-Vet and from diagnostic submissions. Journal of Antimicrobial Chemotherapy, 73(5): 1217-1221 |
Hatosy S M, Martiny A C, 2015. The ocean as a global reservoir of antibiotic resistance genes. Applied and Environmental Microbiology, 81(21): 7593-7599 DOI:10.1128/AEM.00736-15 |
Hoa P T P, Nonaka L, Viet P H et al, 2008. Detection of the sul1, sul2, and sul3 genes in sulfonamide-resistant bacteria from wastewater and shrimp ponds of north Vietnam. Science of the Total Environment, 405(1/3): 377-384 |
Kennedy D, 2013. Time to deal with antibiotics. Science, 342(6160): 777 DOI:10.1126/science.1248056 |
Kim S R, Nonaka L, Suzuki S, 2004. Occurrence of tetracycline resistance genes tet(M) and tet(S) in bacteria from marine aquaculture sites. FEMS Microbiology Letters, 237(1): 147-156 DOI:10.1111/j.1574-6968.2004.tb09690.x |
Lane D J, 1991. 16S/23S rRNA sequencing. In: Stackebrandt E, Goodfellow M eds. Nucleic Acid Techniques in Bacterial Systematics. New York, USA: Wiley, 115-147
|
Liao W, Lin J, Jia H et al, 2020. Resistance and heteroresistance to colistin in Escherichia coli isolates from Wenzhou, China. Infection and Drug Resistance, 13: 3551-3561 DOI:10.2147/IDR.S273784 |
Lu X M, Li W F, Li C B, 2017. Characterization and quantification of antibiotic resistance genes in manure of piglets and adult pigs fed on different diets. Environmental Pollution, 229: 102-110 DOI:10.1016/j.envpol.2017.05.080 |
Ma S Z, Sun C T, Hulth A et al, 2018. Mobile colistin resistance gene mcr-5 in porcine Aeromonas hydrophila. Journal of Antimicrobial Chemotherapy, 73(7): 1777-1780 DOI:10.1093/jac/dky110 |
Magiorakos A P, Srinivasan A, Carey R B et al, 2012. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clinical Microbiology and Infection, 18(3): 268-281 DOI:10.1111/j.1469-0691.2011.03570.x |
Martínez-Martínez L, Pascual A, Jacoby G A, 1998. Quinolone resistance from a transferable plasmid. The Lancet, 351(9105): 797-799 DOI:10.1016/S0140-6736(97)07322-4 |
Ng L K, Martin I, Alfa M et al, 2001. Multiplex PCR for the detection of tetracycline resistant genes. Molecular and Cellular Probes, 15(4): 209-215 DOI:10.1006/mcpr.2001.0363 |
Nikolakopoulou T L, Giannoutsou E P, Karabatsou A A et al, 2008. Prevalence of tetracycline resistance genes in Greek seawater habitats. The Journal of Microbiology, 46(6): 633-640 DOI:10.1007/s12275-008-0080-8 |
Normark B H, Normark S, 2002. Evolution and spread of antibiotic resistance. Journal of Internal Medicine, 252(2): 91-106 DOI:10.1046/j.1365-2796.2002.01026.x |
Park C H, Robicsek A, Jacoby G A et al, 2006. Prevalence in the United States of aac(6')-Ib-cr encoding a ciprofloxacin-modifying enzyme. Antimicrobial Agents and Chemotherapy, 50(11): 3953-3955 DOI:10.1128/AAC.00915-06 |
Rebelo A R, Bortolaia V, Kjeldgaard J S et al, 2018. Multiplex PCR for detection of plasmid-mediated colistin resistance determinants, mcr-1, mcr-2, mcr-3, mcr-4 and mcr-5 for surveillance purposes. Eurosurveillance, 23(6): 17-00672 |
Rutgersson C, Fick J, Marathe N et al, 2014. Fluoroquinolones and qnr genes in sediment, water, soil, and human fecal flora in an environment polluted by manufacturing discharges. Environmental Science & Technology, 48(14): 7825-7832 |
Sun J, Zhang H M, Liu Y H et al, 2018. Towards understanding MCR-like colistin resistance. Trends in Microbiology, 26(9): 794-808 DOI:10.1016/j.tim.2018.02.006 |
Tamminen M, Karkman A, Lõhmus A et al, 2011. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure. Environmental Science & Technology, 45(2): 386-391 |
Willyard C, 2017. The drug-resistant bacteria that pose the greatest health threats. Nature, 543(7643): 15 DOI:10.1038/nature.2017.21550 |
Ye L, Lu Z, Li X H et al, 2013. Antibiotic-resistant bacteria associated with retail aquaculture products from Guangzhou, China. Journal of Food Protection, 76(2): 295-301 DOI:10.4315/0362-028X.JFP-12-288 |
Zhao J Y, Dang H Y, 2012. Coastal seawater bacteria harbor a large reservoir of plasmid-mediated quinolone resistance determinants in Jiaozhou Bay, China. Microbial Ecology, 64(1): 187-199 DOI:10.1007/s00248-012-0008-z |
 2021, Vol. 52
2021, Vol. 52


