中国海洋湖沼学会主办。
文章信息
- 何渊, 杨娟娟, 沈颂东. 2021.
- HE Yuan, YANG Juan-Juan, SHEN Song-Dong. 2021.
- 浒苔光照和盐度胁迫响应基因UpMYB44的克隆与表达分析
- CLONING AND EXPRESSION OF THE UPMYB44 GENE IN RESPONSE TO LIGHT AND SALINITY STRESS IN ULVA PROLIFERA
- 海洋与湖沼, 52(5): 1191-1200
- Oceanologia et Limnologia Sinica, 52(5): 1191-1200.
- http://dx.doi.org/10.11693/hyhz20210200038
文章历史
-
收稿日期:2021-02-06
收修改稿日期:2021-06-02
非生物胁迫严重影响了植物的生长和发育, 面对不利的环境, 植物可以通过引发一系列的生理生化反应去适应这种不良影响(Huang et al, 2012)。MYB转录因子可以作为植物响应非生物胁迫的重要调控因子, 已被广泛研究(Ambawat et al, 2013), MYB (V-myb avian myeloblastosis viral oncogene homolog)转录因子是植物中最大的转录因子家族之一, 存在于真核生物中, 广泛参与植物细胞分化、信号转导和次级代谢调控等多种生命活动, 特别在植物的转录调节中发挥着重要的作用(Mikami et al, 2013)。根据MYB蛋白含有的不同数量的结构域, MYB基因可以分为1R、R2R3、3R、4R这四种类型(杜静静等, 2019)。尤其是R2R3-MYB类转录因子, 该类转录因子含有2个MYB结构域, 参与调节植物在面对环境胁迫时的初级和次级代谢活动(Chen et al, 2019)。植物中最早发现MYB基因的是玉米体内的C-MYB, 该基因参与玉米的花青素合成过程(Marocco et al, 1989), 在后续的研究中又陆续在多种植物中发现了MYB基因, 在棉花中发现了219个基因(Baldoni et al, 2015), 在水稻中鉴定出183个MYB基因(Chen et al, 2006), GmMYB010参与了大豆抗虫的过程(陆捡花等, 2017)。
在环境胁迫响应方面, 对MYB基因也进行了相应的研究。比如在刚毛柽柳(Tamarix hispida)中对MYB基因家族在环境胁迫下的表达进行了差异分析(Zhang et al, 2018), TaMYB参与了拟南芥(Arabidopsis thaliana)对干旱环境胁迫的响应过程(Zhao et al, 2018), 植物中大部分的R2R3-MYB能够响应植物的盐胁迫从而提高其耐盐能力(张遵强等, 1998)。在诸多的环境因素中, 光照和盐度是影响藻类生命活动的重要环境因子(He et al, 2017)。在光照方面, 高光照条件下会影响杜氏盐藻和雨生红球藻类胡萝卜素合成途径中关键基因的表达水平(Pick et al, 2019), 光照强度同样对浒苔的生长状态有关键的影响(Li et al, 2012); 在盐度方面, 高盐度的培养会促进杜氏盐藻类胡萝卜素的合成(Shang et al, 2018), 盐度同样会影响浒苔的氮元素富集从而影响其增殖(Zheng et al, 2019)。
浒苔(Ulva prolifera)是一种常见的大型海洋绿藻, 具有单层细胞围绕成的管状结构, 广泛分布于低潮区的滩涂、礁石和底泥之中(Fan et al, 2014)。浒苔具有细丝状易于漂浮的外观, 多样的繁殖方式以及能够抵御多种环境因子胁迫等生物学特征可以帮助浒苔承受多种环境压力, 例如高盐、干旱和强光照条件, 且可以在较广的温度范围内存活, 是一种生命力很强的海藻(Xiao et al, 2016)。从2007年起, 我国的黄海海域每年都会暴发以浒苔为主的“绿潮”灾害, 据统计每年“绿潮”灾害所引发的生物量达到了数百万吨(Liu et al, 2015), 给沿海地区造成了巨大的经济损失和严重社会影响(Zhang et al, 2017; 王宗灵等, 2018; Zhao et al, 2019a)。相对于MYB家族在高等植物中的生长发育、生理代谢、生物胁迫与非生物胁迫功能研究的大量研究成果, 人们对MYB家族转录因子在浒苔中的结构和功能研究较少。浒苔能够引发“绿潮”灾害和浒苔具有抵御夏季海面的极端环境的能力密切相关, 研究表明MYB家族的转录因子具有应答外界环境胁迫的能力(Dubos et al, 2010), 因此我们推测MYB类转录因子在浒苔夏季抵御海面严酷的环境胁迫大量增殖的过程中起到了一定的作用。
本研究依据前期已经完成的浒苔光照和盐度胁迫下的转录组测序结果, 筛选出具有表达水平显著变化的MYB类基因MYB44, 将其命名为UpMYB44, 进一步对其进行序列扩增、生物信息学分析、实时荧光定量PCR分析等实验初步探究了该基因的表达模式和分子功能, 证明其在非生物胁迫例如盐度和光照的响应过程中发挥了重要的调控作用, 为今后深入研究UpMYB44基因的功能奠定了坚实的基础。
1 材料与方法 1.1 实验材料于2020年7月在山东省青岛市市南区栈桥潮间带(37.46°N, 121.71°E)采集浒苔配子体样品, 放入容器中带回苏州大学藻类实验室。用无菌水清洗藻体, 除去表面的泥沙和其他杂物, 放置于光照培养箱培养。培养条件为f/2培养基, 温度为20 ℃, 光照强度为90 μmol photons/(m2·s), 盐度为24, 光周期为: L: D=12 h : 12 h。每3 d换一次过滤海水, 预培养7 d。
1.2 光照和盐度胁迫处理用三种不同光照培养浒苔5 d, 18 μmol photons/(m2·s)设置为低光照组(l), 90 μmol photons/(m2·s)设置为中光照组(m), 216 μmol photons/(m2·s)设置为高光照组(h); 用三种不同盐度海水培养浒苔5 d, 低盐度组(L)设置为12, 中盐度组(M)设置为24, 高盐度组(H)设置为40, 培养密度为20 g/L, 温度为20 ℃, 光周期为: L: D=12 h : 12 h, 1 d换一次过滤海水, 以上处理的每种样品均设置3个生物学重复。
1.3 RNA提取和cDNA合成参照(He et al, 2018)的方法使用Takara的总RNA提取试剂盒对三种不同光照和盐度处理的浒苔进行总RNA的提取, 同时通过Thermo Fisher NanoDrop 2000和琼脂糖凝胶电泳对所提取的RNA的浓度和纯度进行检测, 使用Takara Reverse Transcription kit将2 μg检测合格的RNA合成为cDNA第1链。
1.4 UpMYB44基因的cDNA全长克隆根据实验室获得的浒苔光照和盐度胁迫下的转录组数据(GenBank: SRX4552126, SRX5502767)设计一对特异性引物UpMYB44F/R, UpMYB44F: ATGACTTCCGAGGGGATGGAG, UpMYB44R: GACGAGCTGCTCGCGGTTAAA。以之前获得的cDNA为模板, 使用2×HieffTM PCR Master Mix (Yeasen) 扩增UpMYB44的ORF全长序列, 50 μL PCR反应体系如下: 2×HieffTM PCR Master Mix 25 μL, 上游引物和下游引物(10 μmol/L)各2 μL, cDNA模板2 μL, 加19 μL的ddH2O补至50 μL。PCR反应程序如下: 94 ℃预变性5 min, 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸1 min, 30个循环, 72 ℃延伸10 min。取上述PCR产物10 μL进行1%琼脂糖凝胶电泳, 使用Gel Extraction Kit (康为世纪)切取符合目的基因片段大小的条带进行回收。使用Hieff Clone® Zero TOPO- Blunt Cloning Kit (Yeasen)进行目的片段平末端扩增, 将构建好的质粒接种于LB固体培养基上过夜培养, 第二天挑取阳性克隆测序, 获得UpMYB44基因ORF区全长序列。
1.5 UpMYB44生物信息学分析使用DNAMAN软件对UpMYB44进行氨基酸保守序列分析; 利用Expays (https://web.Expasy.Org/protparam/)在线软件分析编码蛋白的大小、氨基酸数量和等电点; 使用NCBI的CDD数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对蛋白结构域进行预测; 利用ProtScale工具(http://web.Expasy.org/protscale/)对UpMYB44蛋白进行亲水性/疏水性分析; 采用ExPaSy-SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线软件预测蛋白质的二级结构; 利用SWISS-MODEL (https://swissmodel.expasy.org/interactive) 对UpMYB44蛋白3D结构进行分析。
1.6 表达载体的构建及转化参照Al-Muhanna等(2018)的方法采用T4连接酶将UpMYB44目的片段与pET-32a载体(反应体系包含0.1 pmol目的片段, 0.03 pmol载体片段, 3 U T4 DNA连接酶, 1×T4 DNA连接酶缓冲液)在16 ℃反应12 h。采用热激法将连接产物转入大肠杆菌Trans-T1感受态细胞。用双酶切进行(Nde I, Xba I)鉴定, 将阳性质粒送去苏州金唯智公司进行测序鉴定。
1.7 UpMYB44的蛋白表达参照Baneyx(1999)的方法将pET-32a+ UpMYB44(+)1 μL加入100 μL BL21(DE3)感受态细菌中, 置冰上20 min, 37 ℃, 220 r/min振摇1 h, 离心后全部涂布于含50 μg/mL Amp的LB平板, 37 ℃倒置培养过夜。挑取转化平板上的单克隆接种在含50 μg/mL Amp的3 mL LB液体培养基的培养管中, 在220 r/min, 37 ℃, 4 h摇床里进行培养, 当分光光度计测量值显示其OD600(600 nm处的吸光值)≈ 0.6—0.8时, 滴入浓度为1.0 mmol/L IPTG, 37 ℃ 220 r/min经过振摇4 h诱导蛋白表达后取2 mL, 然后14 000 r/min离心, 弃去上清液, 采用PBS来重悬沉淀, 加2×十二烷基硫酸钠(sodium dodecyl sulfate loading buffer, SDS)上样缓冲液混合均匀之后, 沸水煮沸10 min。然后进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)超声破碎后收集上清, 用0.45 μm滤膜过滤, 用3倍体积的平衡液[ 25 mmol /L Tris(pH值8.0), 300 mmol/L NaCl, 10 mmol/L咪唑] 平衡His纯化基质, 将过滤后的上清与His纯化基质于50 mL离心管中4 ℃混匀1 h。用洗涤缓冲液[25 mmol /L Tris (pH值8.0), 300 mmol/L NaCl, 30 mmol/L咪唑] 冲洗4次, 用洗脱缓冲液[25 mmol/L Tris (pH值8.0), 300 mmol/L NaCl, 250 mmol/L咪唑]洗脱3次, SDS- PAGE电泳检测融合蛋白纯化效果。
1.8 UpMYB44亚细胞定位参照Xiong等(2019)的方法设计亚细胞定位引物YFP-UpMYB44F/R, YFP-UpMYB44F: AAGAGA CAGGATCCGAATTCATGACTTCCGAGGGGATGGA G, YFP-UpMYB44R: ACCTCCGACCGGTGCACTA GTGACGAGCTGCTCGCGGTTAAA。将酶切位点BamH I和Sac I加入目的基因ORF两端, 将测序成功的阳性质粒T-blunt-UpMYB44为模板进行扩增和切胶回收。使用BamH I和Sac I对质粒pC131-YFP进行双酶切, 产物纯化。将目的基因片段和载体pC131-YFP进行连接转入大肠杆菌DH5α感受态细胞, 获得重组质粒pC131-UpMYB44-YFP。将空载质粒pC131-YFP和重组质粒pC131-UpMYB44-YFP分别转入农杆菌GV3101菌株中, 将阳性克隆放入含有50 μg/mL卡那霉素和35 μg/mL利福平的LB培养基中进行培养, 放置于28 ℃摇床中以250 r/min速度培养过夜, 收集并重悬菌体, 当菌体浓度的OD600值在0.5—1.0之间, 静置3 h后注射入烟草叶片中, 注射后的烟草放置于28 ℃培养箱中培养2 d。用激光共聚焦显微镜观察侵染的烟草叶片, 进行UpMYB44蛋白亚细胞观察。
1.9 实时荧光定量PCR参照He等(2019)的方法设计实时荧光定量PCR并以不同光照和盐度处理的浒苔cDNA为模板进行实时荧光定量PCR实验, 荧光定量PCR采用Hieff® qRT-PCR SYBR Green Master Mix (Yeasen)试剂, 使用ASA-4800 Real-Time PCR仪器(百源基因)进行反应, 以18S rDNA为内参基因, 具体引物序列如表 1所示, 上述实验重复3次。结果分析采用2-ΔΔCT法进行相对定量, 采用软件Origin 8.0对表达结果进行单因素方差统计(ANOVA)。数据使用SPSS 21.0进行统计分析, P < 0.05设定为显著性差异, P < 0.01设定为极显著性差异。
| 基因名称 | 引物名称 | 序列信息 |
| UpMYB44 | qUpMYB44F | ATAACCAGCTTTGCCCCGACATCA |
| qUpMYB44R | GGGAGCGGGACAGGGATGTTGA | |
| 18S rRNA | 18SF | ACCACATCCAAGGAAGGCAGCAG |
| 18SR | TGCGTCCCACCCGAAATCCAAC |
参照Cai等(2016)的方法制备Y2H Gold酵母感受态细胞, 将UpCPP5作为诱饵构建PGBKT7-CPP5重组质粒进行毒性检测和自激活检测: 于100 ℃沸水煮carrier DNA 5 min, 立即置于冰上2 min, 如此重复一次, 取1.5 mL无菌EP管, 配制转化体系, 50 μL感受态细胞+5 μL carrier DNA+100 ηg诱饵质粒+100 ηg猎物空载, 加入500 μL PEG/LiAc (8 mL 50% PEG+1 mL LiAc+1 mL TE), 30 ℃水浴30 min, 加入20 μL DMSO, 42 ℃水浴15 min, 800 ×g离心1 min, 去上清, 加入800 μL YPDA重悬, 30 ℃, 150 r/min, 振荡培养1.5 h, 800 ×g离心5 min, 弃上清, 加入1 mL 0.9% NaCl重悬, 取150 μL涂平板, 30 ℃倒置培养4 d, 观察菌落直径及颜色; 构建PGADT7- MYB44重组质粒作为猎物进行诱饵和猎物的共转化, 依次从自激活、共转化实验组和共转化阴阳性对照组涂布的平板DDO上挑单菌落, 分别接种于5 mL DDO液体培养基, 30 ℃ 250 r/min培养16 h, 取300 μL菌液于50 mL DDO中, 30 ℃ 250 r/min培养8—12 h至OD600值为0.4—0.6; 各取10 μL菌液点种到平板DDO、TDO/3AT (15 mmol/L)、QDO上, 30 ℃培养箱培养3 d, 观察菌斑生长情况, 若涂布在平板DDO上长菌则共转化成功, 在平板TDO/3AT (15 mmol/L)上长菌而在QDO上不长菌为阳性结果, 在平板TDO/3AT (15mmol/L)上不长菌为阴性结果。
2 结果 2.1 UpMYB44 ORF区全长序列的克隆与序列分析利用RT-PCR以浒苔cDNA为模板, PCR扩增相应片段, 测序得到UpMYB44的ORF区全长序列, 序列全长为1 437 bp, 共编码478个氨基酸。序列上传至Genbank数据库, 登录号为MW174238。
2.2 UpMYB44生物信息学分析结果通过NCBI与其他物种的MYB蛋白进行BLASTn比对, 结果显示UpMYB44蛋白与另一种绿藻小球藻的MYB44蛋白有较高的同源性, 氨基酸的一致性为50.00%, 与高等植物番木瓜、荷花、木槿、牵牛花、柿树和向日葵的氨基酸相似率也在45.00%—50.00%之间, 用DNAMAN将UpMYB44氨基酸序列与上述物种进行比对, 发现了较为保守的序列区域(图 1), 推测与这些MYB类转录因子有相似的生物学功能。利用Expays在线网站预测结果显示UpMYB44蛋白含有478个氨基酸, 分子量为52.49 kDa, 等电点为6.15; 利用CDD数据库分析显示UpMYB44含有两个SANT结构域, 是典型的R2R3-MYB(图 2); 利用ProtScale工具对UpMYB44蛋白进行亲水性/疏水性分析, 结果显示UpMYB44为亲水性蛋白; 在SOPMA在线网站预测蛋白二级结构, UpMYB44蛋白二级结构中含有42.68%的α-螺旋结构、8.16%的延伸连结构、9.62%的β-转角结构、39.54%的无规则卷曲结构(图 3)。利用Swiss-model网站对UpMYB44蛋白进行3D结构预测, 发现与其蛋白二级结构结果类似, 具有较多的α-螺旋结构和无规则卷曲结构(图 4)。
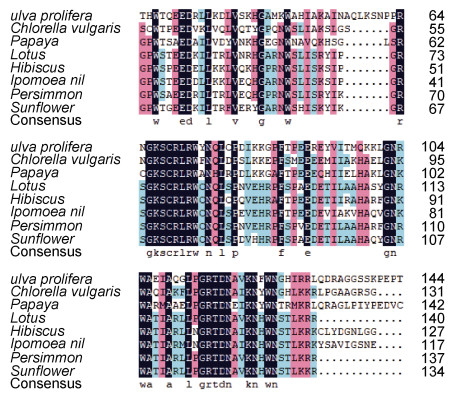 |
| 图 1 UpMYB44与其他物种MYB类转录因子氨基酸序列的多重比对 Fig. 1 Multiple alignment of the amino acid sequence of UpMYB44 and MYB transcription factors from other species 注: 浒苔Ulva prolifera (Genbank序列号: MW174238);小球藻Chlorella vulgaris (PRW44975.1); 番木瓜Papaya (XP_021900987.1); 荷花Lotus (XP_010268609.1); 木槿Hibiscus (KAE8672586.1); 牵牛花Ipomoea nil (XP_019168759.1); 柿树Persimmon (XP_011030579.1); 向日葵Sunflower (XP_021983625.1) |
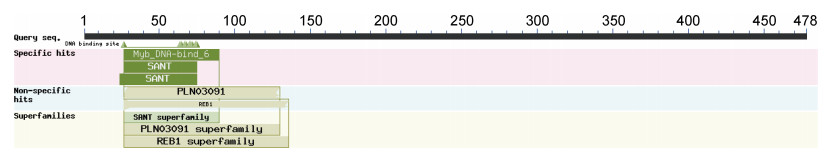 |
| 图 2 UpMYB44蛋白保守结构域预测 Fig. 2 Prediction of conservative domain of UpMYB44 protein |
 |
| 图 3 UpMYB44二级结构预测 Fig. 3 Prediction of secondary structure of UpMYB44 注: 红色: 延伸链结构; 紫色: 无规则卷曲结构; 蓝色: α-螺旋结构; 绿色: β-转角结构 |
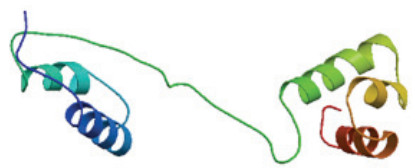 |
| 图 4 UpMYB44蛋白三维结构预测模型 Fig. 4 Predicted 3D structure model of UpMYB44 protein |
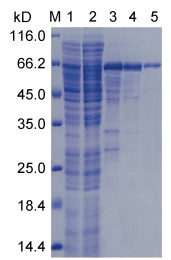
结果表明, UpMYB44融合蛋白主要以可溶性蛋白的形式存在于上清中, 但是在包涵体沉淀中也有一定量的表达(图 5)。选择上清中可溶性蛋白进行后续蛋白纯化实验, 通过4次洗涤和3次洗脱, 获得了大小约为65 kD (含标签)的纯度较好的UpMYB44融合蛋白(图 6)。
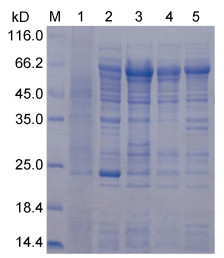 |
| 图 5 UpMYB44蛋白表达鉴定SDS-PAGE分析 Fig. 5 Identification of UpMYB44 protein expression by SDS-PAGE analysis 注: M: 蛋白质分子质量标准(14.4—116 kD); 1: pET-32a诱导; 2: 诱导前; 3: 诱导后; 4: 诱导破碎后上清; 5: 诱导破碎后沉淀 |
 |
| 图 6 UpMYB44蛋白纯化SDS-PAGE分析 Fig. 6 Purification of UpMYB44 protein by SDS-PAGE analysis 注: M: 蛋白质分子质量标准(14.4—116 kD); 1: 破碎后处理样品; 2: 流穿; 3, 4: 30 mmol/L咪唑洗脱; 5: 250 mmol/L咪唑洗脱 |
在对照组空载pC131-YFP中, 黄色荧光在细胞膜、细胞核和细胞质中都有分布, 而融合表达载体pC131-UpMYB44-YFP的细胞中, 黄色荧光仅在细胞核中被发现, 而在细胞膜和细胞质中都没有荧光发现, 并且黄色荧光和核定位的marker蛋白的红色荧光发生重合。结果显示UpMYB44蛋白仅定位在细胞核上, 这一点符合MYB类转录因子的特点(图 7)。
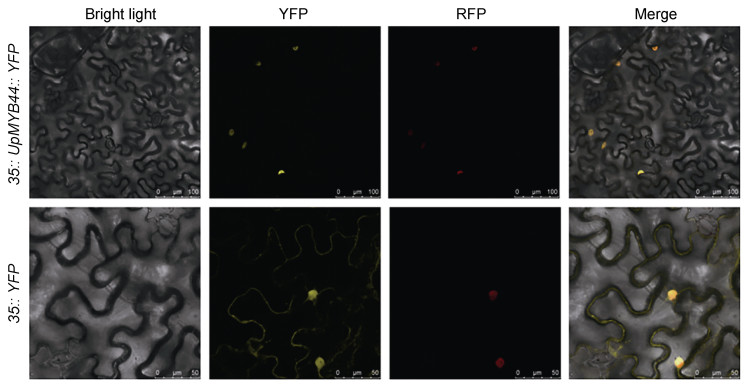 |
| 图 7 UpMYB44蛋白的亚细胞定位 Fig. 7 Subcellular localization of the UpMYB44 protein 注: 35:: UpMYB44:: YFP: 融合表达载体pC131-UpMYB44-YFP; 35:: YFP: 对照组空载pC131-YFP在烟草叶片中转入了融合表达载体(UpMYB44- YFP)和核标记NLS-RFP (NLS, 核定位序列)。图中依次显示了明场(Bright light)、黄色荧光蛋白(YFP)、红色荧光蛋白(RFP)和合并(Merge)的图像 |
UpMYB44在三种不同光照和盐度处理下的表达量发生了显著变化, 在光照组中, 18 μmol photons/(m2·s)处理的低光组中UpMYB44的表达量最高, 随着光照强度的增加, UpMYB44的表达量逐渐降低(图 8), UpMYB44的表达量在216 μmol photons/(m2·s)处理的高光组中最低; 在盐度组中, 随着盐度的增加, UpMYB44的表达量逐渐升高, UpMYB44的表达量在盐度40处理的高盐组中最高, 在盐度12处理的低盐组中最低(图 9)。
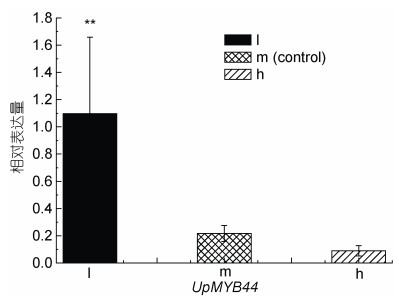 |
| 图 8 UpMYB44在三种光照强度条件下的表达分析 Fig. 8 Expression analysis of UpMYB44 under three lights stresses 注: l: 低光照; m: 中光照; h: 高光照 |
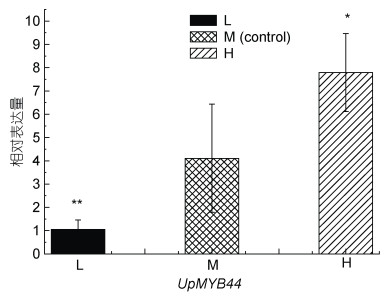 |
| 图 9 UpMYB44在三种盐度强度条件下的表达分析 Fig. 9 Expression analysis of UpMYB44 under three salinities stresses 注: L: 低盐度; M: 中盐度; H: 高盐度 |
MYB类转录因子通常需要与其他蛋白互作进行发挥其功能。利用STRING在线网站预测UpMYB44的互作蛋白, 根据生物信息学预测结果发现UpMYB44和另一种调控植物生长发育和响应非生物胁迫中起重要作用的转录因子UpCPP存在互作关系, 以缺失蛋白UpCPP5为诱饵, 通过酵母双杂交点对点验证, 结果表明重组诱饵质粒PGBKT7-CPP5对酵母细胞无毒性且3-氨基-1, 2, 4三唑(3AT, 15mmol/L)能抑制激活Y2H Gold酵母报告基因, 酵母双杂交的结果显示, 将猎物质粒PGADT7-MYB44与诱饵质粒PGBKT7-CPP5共转化酵母细胞: 对照结果符合预期, 说明该系统可用于双杂交验证; 实验组涂布DDO平板能生长说明诱饵和猎物共转化成功, 涂布TDO/3AT (15mmol/L)板能生长, QDO平板不长, 说明激活报告基因HIS3的表达, 说明UpMYB44蛋白和UpCPP5蛋白之间存在相互作用(图 10)。
 |
| 图 10 UpMYB44与UpCPP5互作蛋白验证 Fig. 10 Verification of UpMYB44 interacting with UpCPP5 注: 1: Y2H [PGBKT7-CPP+ PGADT7]; 2: Y2H [PGBKT7-CPP + PGADT7-MYB44]; +: Y2H [pGBKT7-53 + pGADT7-T]; –: Y2H [pGBKT7-lam + pGADT7-T] |
研究发现在植物中MYB类转录因子参与了多种生物和非生物胁迫的响应过程(Lea et al, 2007)。超表达玉米中的ZmMYB3R可以增加其对干旱和盐度的耐受性(Yin et al, 2017), 过量表达烟草的NtMYB15可以促进其适应低温胁迫(Wu et al, 2019), 在模式生物拟南芥中超表达AtMYB74同样具有增强其盐耐受性的功能(Xu et al, 2015), 而且MYB类转录因子常常需要与其他蛋白互作来发挥功能(Kaspar et al, 1999)。
本研究以受关注度较高的引发“绿潮”灾害的浒苔为研究对象, 初步分析了MYB类家族成员UpMYB44的生物学功能。结果表明UpMYB44有两个MYB结构域, 属于典型的R2R3-MYB转录因子, 研究表明该类转录因子参与了棉花和月季对高盐胁迫的响应(Zhao et al, 2019b, 包颖等, 2020)。盐度和光照是影响藻类正常生长和生理生化指标的重要的两个环境因子(Arai et al, 1991), 特别是对浒苔而言, 盐度和光照的改变会引起其氧化应激反应(Luo et al, 2011), 以及影响其体内的总类胡萝卜素含量(He et al, 2020)。在本研究中, UpMYB44在高盐条件下的表达量发生了显著提高, 说明该转录因子参与了浒苔的对高盐胁迫的响应过程, 可能对提高浒苔适应恶劣的外界环境的能力有一定帮助, 最终有利于其快速增殖。R2R3-MYB转录因子也会对光照的改变作出响应, 在早期的研究中发现红色紫苏中存在一种光诱导的Myb-p1基因, 其在花青素的形成过程中起到了决定性作用(Gong et al, 1999), 在马铃薯中发现的StR2R3-MYB1就是一种光响应MYB类转录因子, 基于对光处理后基因表达特性分析的结果表明该转录因子可能受到光信号和环境胁迫的诱导(秦玉芝等, 2015)。在浒苔中UpMYB44的表达量同样受到了不同光照强度的影响, 且随着光照的逐渐增强其表达量呈下降趋势, 这和一般应激反应的表现有差异, 推测夏季浒苔在漂浮状态下的高光照强度胁迫会影响其正常的生长状态, 导致部分基因的表达出现下调, 说明过高的光强对其增殖反而不利。UpMYB44基因在浒苔受到光照和盐度胁迫时会发生响应, 对夏季抵御海面严酷的环境胁迫起到一定作用。对MYB类转录因子互作模式的研究也已经开展, 比如在苹果中MdMYB23蛋白通过结合MdBT2蛋白来抑制原花色素的积累(An et al, 2018), 通过鉴定与AtMAPK3P相互作用的MYB转录因子成员, 增强了我们对拟南芥中MAPK相互作用蛋白网络的认识, 也证实了蛋白质与蛋白质的相互作用对于生物过程至关重要(Giri et al, 2014)。
在本研究中, 通过酵母双杂交系统证明了UpMYB44蛋白和一种CPP蛋白具有互作, 通过和Genbank中的序列进行比对, 我们将其命名为UpCPP5。CPP蛋白又称为tesmin/TSO1-like蛋白, 在植物中广泛分布, 该家族在植物生长发育和细胞分裂控制中起重要作用(Yang et al, 2008), TSO1参与了拟南芥花发育的细胞分裂过程(Liu et al, 1997), 如果该基因轻度的突变会影响拟南芥胚珠发育, 而强突变对所有花卉组织都有影响(Andersen et al, 2007), 因此, 猜测这两类转录因子的相互作用影响了浒苔的细胞分裂过程, 这个研究方向正好和目前浒苔如何引发“绿潮”灾害的研究热点相互关联。本实验的研究材料浒苔是一种能够快速生长的灾害绿藻, 只要外界条件合适, 其生长速率相当惊人(Hiraoka et al, 2008), 每年夏季由于其暴发性生长对我国沿海城市造成了巨大的环境污染(Yu et al, 2017; He et al, 2019), 对近海的生态系统也造成了严重的破坏(Qu et al, 2020)。根据之前浒苔基因组的研究, 我们推测浒苔的快速生长和细胞的快速分裂密切相关, 但是浒苔增殖最终重要的是营养增殖, 因此后续可以通过深入研究浒苔中MYB家族和CPP家族的互作是否会影响浒苔的生长发育和细胞分裂, 并结合浒苔营养增殖方面的研究为进一步了解环境胁迫调控网络提供依据, 更重要的是为研究浒苔的快速繁殖机制提供了全新的思路。
4 结论本研究在本实验室已有的研究基础上, 克隆了1个R2R3-MYB转录因子基因——UpMYB44的ORF区全长序列, 其氨基酸序列与GenBank中已有的小球藻MYB蛋白具有相似性, 我们对其进行了生物学功能验证, 在异于浒苔最适环境的光照和盐度的培养条件下, UpMYB44的表达量会发生显著变化, 证明其参与了浒苔响应光照和盐度压力的过程, 可能在盐度和光照压力应答中具有关键作用。实验证明UpMYB44与CPP类转录因子UpCPP5存在较强的互作, 推测二者的结合可能参与了浒苔的生长发育和细胞分裂过程, 但其具体的作用机制还需要进一步研究。
王宗灵, 傅明珠, 肖洁, 等. 2018. 黄海浒苔绿潮研究进展. 海洋学报, 40(2): 1-13 |
包颖, 李泽卿, 魏琳燕, 等. 2020. 月季MYB转录因子基因RcWER-like的克隆及表达分析. 核农学报, 34(6): 1144-1151 |
杜静静, 田岳, 冯昊, 等. 2019. 陆地棉基因GhMYB52的克隆及特征分析. 棉花学报, 31(6): 505-514 |
张遵强, 何希诚, 胡超宗, 等. 1998. 紫竹鞭段繁殖研究. 竹子研究汇刊, 17(2): 40-43 |
陆捡花, 李祥, 赵为, 等. 2017. 大豆MYB转录因子GmMYB010与其拟南芥同源基因的功能差异. 中国油料作物学报, 39(3): 281-293 |
秦玉芝, 邢铮, 潘妃, 等. 2015. 马铃薯光响应StR2R3-MYB1基因的克隆与表达分析. 湖南农业大学学报(自然科学版), 41(4): 378-384 |
Al-Muhanna S G, Al-Muhanna S A, 2018. Construction and transformation of recombinant pET28a expression vector in BL21 (DE3) cells with basic bioinformatics analysis. Biochemical and Cellular Archives, 18(1): 147-151 |
Ambawat S, Sharma P, Yadav N R et al, 2013. MYB transcription factor genes as regulators for plant responses: an overview. Physiology and Molecular Biology of Plants, 19(3): 307-321 DOI:10.1007/s12298-013-0179-1 |
An J P, Li R, Qu F J et al, 2018. R2R3-MYB transcription factor MdMYB23 is involved in the cold tolerance and proanthocyanidin accumulation in apple. The Plant Journal, 96(3): 562-577 DOI:10.1111/tpj.14050 |
Andersen S U, Algreen-Petersen R G, Hoedl M et al, 2007. The conserved cysteine-rich domain of a tesmin/TSO1-like protein binds zinc in vitro and TSO1 is required for both male and female fertility in Arabidopsis thaliana. Journal of Experimental Botany, 58(13): 3657-3670 DOI:10.1093/jxb/erm215 |
Arai A, Miura A, 1991. Effects of salinity and light intensity on the growth of brown algae, Sargassum ringgoldianum. Aquaculture Science, 39(3): 315-319 |
Baldoni E, Genga A, Cominelli E, 2015. Plant MYB transcription factors: their role in drought response mechanisms. International Journal of Molecular Sciences, 16(7): 15811-15851 DOI:10.3390/ijms160715811 |
Baneyx F, 1999. Recombinant protein expression in Escherichia coli. Current Opinion in Biotechnology, 10(5): 411-421 DOI:10.1016/S0958-1669(99)00003-8 |
Cai Y F, Pan L Q, Miao J J et al, 2016. Identification of interacting proteins with aryl hydrocarbon receptor in scallop Chlamys farreri by yeast two hybrid screening. Ecotoxicology and Environmental Safety, 133: 381-389 DOI:10.1016/j.ecoenv.2016.07.013 |
Chen Y H, Yang X Y, He K et al, 2006. The MYB transcription factor superfamily of Arabidopsis: expression analysis and phylogenetic comparison with the rice MYB family. Plant Molecular Biology, 60(1): 107-124 DOI:10.1007/s11103-005-2910-y |
Chen C, Zhang K X, Khurshid M et al, 2019. MYB transcription repressors regulate plant secondary metabolism. Critical Reviews in Plant Sciences, 38(3): 159-170 DOI:10.1080/07352689.2019.1632542 |
Dubos C, Stracke R, Grotewold E et al, 2010. MYB transcription factors in Arabidopsis. Trends in Plant Science, 15(10): 573-581 DOI:10.1016/j.tplants.2010.06.005 |
Fan X, Xu D, Wang Y T et al, 2014. The effect of nutrient concentrations, nutrient ratios and temperature on photosynthesis and nutrient uptake by Ulva prolifera: implications for the explosion in green tides. Journal of Applied Phycology, 26(1): 537-544 DOI:10.1007/s10811-013-0054-z |
Giri P, Kumar A, Taj G, 2014. In silico-prediction of downstream MYB interacting partners of MAPK3 in Arabidopsis. Bioinformation, 10(12): 721-725 DOI:10.6026/97320630010721 |
Gong Z Z, Yamazaki M, Saito K, 1999. A light-inducible Myb-like gene that is specifically expressed in red Perilla frutescens and presumably acts as a determining factor of the anthocyanin forma. Molecular and General Genetics MGG, 262(1): 65-72 |
He Y, Ao Y, Yin Y et al, 2019. Comparative transcriptome analysis between floating and attached Ulva prolifera in studying green tides in the Yellow Sea. Algal Research, 44: 101712 DOI:10.1016/j.algal.2019.101712 |
He Y, Ma Y F, Du Y et al, 2018. Differential gene expression for carotenoid biosynthesis in a green alga Ulva prolifera based on transcriptome analysis. BMC Genomics, 19(1): 916 DOI:10.1186/s12864-018-5337-y |
He Y, Yan Z H, Du Y et al, 2017. Molecular cloning and expression analysis of two key genes, HDS and HDR, in the MEP pathway in Pyropia haitanensis. Scientific Reports, 7(1): 17499 DOI:10.1038/s41598-017-17521-9 |
He Y, Ye Y T, Shen S D, 2020. Effects of light and salinity on carotenoid biosynthesis in Ulva prolifera. Acta Oceanologica Sinica, 39(10): 50-57 DOI:10.1007/s13131-020-1577-1 |
Hiraoka M, Oka N, 2008. Tank cultivation of Ulva prolifera in deep seawater using a new "Germling cluster" method. Journal of Applied Phycology, 20(1): 97-102 DOI:10.1007/s10811-007-9186-3 |
Huang G T, Ma S L, Bai L P et al, 2012. Signal transduction during cold, salt, and drought stresses in plants. Molecular Biology Reports, 39(2): 969-987 DOI:10.1007/s11033-011-0823-1 |
Kaspar P, Dvoráková M, Králová J et al, 1999. Myb-interacting protein, ATBF1, represses transcriptional activity of Myb oncoprotein. Journal of Biological Chemistry, 274(20): 14422-14428 DOI:10.1074/jbc.274.20.14422 |
Lea U S, Slimestad R, Smedvig P et al, 2007. Nitrogen deficiency enhances expression of specific MYB and bHLH transcription factors and accumulation of end products in the flavonoid pathway. Planta, 225(5): 1245-1253 DOI:10.1007/s00425-006-0414-x |
Li Y X, Zhang X W, Xu D et al, 2012. Differential gene expression in Ulva prolifera under low light and low temperature conditions. Current Genetics, 58(4): 235-244 DOI:10.1007/s00294-012-0380-8 |
Liu X Q, Li Y, Wang Z L et al, 2015. Cruise observation of Ulva prolifera bloom in the southern Yellow Sea, China. Estuarine, Coastal and Shelf Science, 163: 17-22 DOI:10.1016/j.ecss.2014.09.014 |
Liu Z C, Running M P, Meyerowitz E M, 1997. TSO1 functions in cell division during Arabidopsis flower development. Development, 124(3): 665-672 DOI:10.1242/dev.124.3.665 |
Luo M B, Liu F, 2011. Salinity-induced oxidative stress and regulation of antioxidant defense system in the marine macroalga Ulva prolifera. Journal of Experimental Marine Biology and Ecology, 409(1-2): 223-228 DOI:10.1016/j.jembe.2011.08.023 |
Marocco A, Wissenbach M, Becker D et al, 1989. Multiple genes are transcribed in Hordeum vulgare and Zea mays that carry the DNA binding domain of the myb oncoproteins. Molecular and General Genetics MGG, 216(2-3): 183-187 DOI:10.1007/BF00334354 |
Mikami K, Hosokawa M, 2013. Biosynthetic pathway and health benefits of fucoxanthin, an algae-specific xanthophyll in brown seaweeds. International Journal of Molecular Sciences, 14(7): 13763-13781 DOI:10.3390/ijms140713763 |
Pick U, Zarka A, Boussiba S et al, 2019. A hypothesis about the origin of carotenoid lipid droplets in the green algae Dunaliella and Haematococcus. Planta, 249(1): 31-47 DOI:10.1007/s00425-018-3050-3 |
Qu T F, Zhao X Y, Hao Y et al, 2020. Ecological effects of Ulva prolifera green tide on bacterial community structure in Qingdao offshore environment. Chemosphere, 244: 125477 DOI:10.1016/j.chemosphere.2019.125477 |
Shang C H, Wang W, Zhu S N et al, 2018. The responses of two genes encoding phytoene synthase (Psy) and phytoene desaturase (Pds) to nitrogen limitation and salinity up-shock with special emphasis on carotenogenesis in Dunaliella parva. Algal Research, 32: 1-10 DOI:10.1016/j.algal.2018.03.002 |
Wu J D, Jiang Y L, Liang Y N et al, 2019. Expression of the maize MYB transcription factor ZmMYB3R enhances drought and salt stress tolerance in transgenic plants. Plant Physiology and Biochemistry, 137: 179-188 DOI:10.1016/j.plaphy.2019.02.010 |
Xiao J, Zhang X H, Gao C L et al, 2016. Effect of temperature, salinity and irradiance on growth and photosynthesis of Ulva prolifera. Acta Oceanologica Sinica, 35(10): 114-121 DOI:10.1007/s13131-016-0891-0 |
Xiong C, Luo D, Lin A H et al, 2019. A tomato B-box protein SlBBX20 modulates carotenoid biosynthesis by directly activating PHYTOENE SYNTHASE 1, and is targeted for 26S proteasome-mediated degradation. New Phytologist, 221(1): 279-294 DOI:10.1111/nph.15373 |
Xu R, Wang Y H, Zheng H et al, 2015. Salt-induced transcription factor MYB74 is regulated by the RNA-directed DNA methylation pathway in Arabidopsis. Journal of Experimental Botany, 66(19): 5997-6008 DOI:10.1093/jxb/erv312 |
Yang Z F, Gu S L, Wang X F et al, 2008. Molecular evolution of the CPP-like gene family in plants: insights from comparative genomics of Arabidopsis and Rice. Journal of Molecular Evolution, 67(3): 266-277 DOI:10.1007/s00239-008-9143-z |
Yin X M, Cui Y C, Wang M L et al, 2017. Overexpression of a novel MYB-related transcription factor, OsMYBR1, confers improved drought tolerance and decreased ABA sensitivity in rice. Biochemical and Biophysical Research Communications, 490(4): 1355-1361 DOI:10.1016/j.bbrc.2017.07.029 |
Yu D F, Gai Z G, Kong X F et al, 2017. Monitoring Ulva prolifera in the Yellow Sea and East China Sea derived from multi-source remote sensing images. In: Proceedings of SPIE 10605, LIDAR Imaging Detection and Target Recognition 2017. Changchun: SPIE, 1060535
|
Zhang C, Lu J, Wu J et al, 2017. Removal of phenanthrene from coastal waters by green tide algae Ulva prolifera. Science of the Total Environment, 609: 1322-1328 DOI:10.1016/j.scitotenv.2017.07.187 |
Zhang T Q, Zhao Y L, Wang Y C et al, 2018. Comprehensive analysis of MYB gene family and their expressions under abiotic stresses and hormone treatments in Tamarix hispida. Frontiers in Plant Science, 9: 1303-1315 DOI:10.3389/fpls.2018.01303 |
Zhao Y, Cheng X Y, Liu X D et al, 2018. The wheat MYB transcription factor TaMYB31 is involved in drought stress responses in Arabidopsis. Frontiers in Plant Science, 9: 1426 DOI:10.3389/fpls.2018.01426 |
Zhao X Y, Tang X X, Hu S X et al, 2019a. Photosynthetic response of floating Ulva prolifera to diurnal changes of in-situ environments on the sea surface. Journal of Oceanology and Limnology, 37(2): 589-599 DOI:10.1007/s00343-019-8081-5 |
Zhao Y Y, Yang Z E, Ding Y P et al, 2019b. Over-expression of an R2R3 MYB Gene, GhMYB73, increases tolerance to salt stress in transgenic Arabidopsis. Plant Science, 286: 28-36 DOI:10.1016/j.plantsci.2019.05.021 |
Zheng M S, Lin J J, Zhou S D et al, 2019. Salinity mediates the effects of nitrogen enrichment on the growth, photosynthesis, and biochemical composition of Ulva prolifera. Environmental Science and Pollution Research, 26(19): 19982-19990 DOI:10.1007/s11356-019-05364-y |
 2021, Vol. 52
2021, Vol. 52


