
中国海洋湖沼学会主办。
文章信息
- 王静, 王毅超, 王洪淑, 刘峰, 陈楠生. 2021.
- WANG Jing, WANG Yi-Chao, WANG Hong-Shu, LIU Feng, CHEN Nan-Sheng. 2021.
- 我国新记录绿潮物种Ulva laetevirens的比较叶绿体基因组学研究
- COMPARATIVE CHLOROPLAST GENOME ANALYSIS OF NEW-GREEN TIDE CAUSATIVE SPECIES IN CHINA: ULVA LAETEVIRENS
- 海洋与湖沼, 52(5): 1201-1213
- Oceanologia et Limnologia Sinica, 52(5): 1201-1213.
- http://dx.doi.org/10.11693/hyhz20210200045
文章历史
-
收稿日期:2021-02-09
收修改稿日期:2021-03-04
2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室 青岛 266237;
3. 中国科学院海洋大科学研究中心 青岛 266071;
4. 中国科学院大学 北京 100049
2. Functional Laboratory for Marine Ecology and Environmental science, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
3. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
绿潮(green tide)在全球范围内频繁暴发, 欧洲、北美、澳洲和亚洲等海域均有报道。欧洲北亚得里亚海(Runca et al, 1996)、波罗的海(Bäck et al, 2000)、德国瓦登海(Kolbe et al, 1995)等, 美国罗得岛州(Guidone et al, 2013)、华盛顿州(Nelson et al, 2003), 亚洲中国(Liu et al, 2013)、日本(Shimada et al, 2003)、菲律宾(Largo et al, 2004)等国的海域, 均发生过大规模的绿潮事件。近些年, 全球范围内绿潮呈现出常态化的暴发趋势, 对经济和社会活动造成了严重的负面影响, 对致灾海域的生态环境、群落结构、生物多样性以及水产养殖也造成极大的破坏性影响(Lyons et al, 2014; Liu et al, 2020)。绿潮暴发的原因种主要包括绿藻门石莼科的石莼属(Ulva)(Smetacek et al, 2013)、硬毛藻属(Chaetomorpha)(De Paula Silva et al, 2008)和刚毛藻属(Cladophora)(De Paula Silva et al, 2013), 其中石莼属绿藻占主要部分(Fletcher, 1996; Smetacek et al, 2013; Fort et al, 2020)。石莼属绿潮物种中, 部分物种能够形成跨境侵袭的大规模绿潮灾害, 譬如中国黄海绿潮; 部分仅在局部海域暴发, 譬如美国纳拉甘塞特海湾连年发生的扁浒苔(Ulva compressa)-硬石莼(U. rigida)绿潮(Guidone et al, 2013)、韩国济州岛东海岸绿潮(Lee et al, 2019), 以及我国渤海自2015年连续暴发的浒苔绿潮(Song et al, 2019)。我国黄海浒苔绿潮在2007年暴发之前, 浒苔物种或绿潮事件在我国并未引起广泛关注, 暴发之后投入大量的科研工作致力于浒苔绿潮的相关研究, 这恰恰警示我们对潜在的绿潮物种和局地规模的绿潮事件应给予高度关注。调查和了解石莼属绿潮物种生物地理分布和局地规模的绿潮事件是预防和监测大规模绿潮暴发的首要工作之一。
我国在局地绿潮方面的研究报道相对较少。马元庆等(2010)报道了2007年烟台金沙滩海域石莼(U. lactuca)绿潮事件相关的理化条件; Song等(2019)研究发现秦皇岛海域自2015年连年暴发的绿潮原因种是羽藻(Bryopsis plumosa)和浒苔(U. prolifera), 经巡航观测和对沿岸定生样本的分子标记rbcL分析发现, 这两个原因种只在海草床中被发现, 最终确定海草床中的羽藻和浒苔是渤海绿潮的原因种。总体而言, 我国在局地绿潮的相关研究工作正处于起步阶段。
我国海域石莼属物种的记录自20世纪60年代由曾呈奎等生态学前辈系统整理开始, 一直处于不断地补充和修正中。1963年董美龄(1963)报道我国海域的“浒苔属”(Enteromorpha)绿藻有11个种。2015年丁兰平等(2015)整理的中国海洋绿藻门新分类系统中“浒苔属”有10个种, “石莼属”有14个种。2020年Xie等(2020)报道了我国海域的6个新记录物种U. simplex、U. splitiana、U. partita、U. meridionalis、U. tepida和U. chaugulli, 后三者为绿潮物种。此外, 还有一株淡水石莼物种U. shanxiensis (Chen et al, 2015)和一株浒苔亚种U. prolifera subsp. Qingdaoensis (Cui et al, 2018)被鉴定。这说明我国海域可能还有更多的石莼属物种(包括石莼属绿潮物种)有待发现。由于石莼属部分近缘物种种间形态十分相近, 并且具有形态可塑性, 基于形态进行石莼属物种鉴定难度较大。随着分子生物技术的发展, 分子标记的开发和利用对石莼属物种的鉴定起了重要的推动作用(Hayden et al, 2003)。目前, 应用于石莼属物种鉴定的通用单基因分子标记是ITS、rbcL和tufA (Gary et al, 2010; Mudassar et al, 2016; Cui et al, 2018)。然而, 尽管这些通用分子标记使用广泛, 但是由于它们的分辨率比较有限, 在部分情况下无法做到准确定种。相对单基因分子标记, 细胞器基因组可以作为串联基因标记提供更可靠的分类依据。
本研究针对2020年11月初烟台第一海水浴场局地绿潮事件, 采用分子标记和叶绿体基因组比较分析对此次绿潮事件的原因种进行物种鉴定和分析, 确定该局地绿潮原因种为U. laetevirens, 系该物种在我国海域的首次绿潮记录。同时, 探索了三者之间的遗传进化关系。
1 材料与方法 1.1 样品采集与处理2020年11月2日于山东省烟台市第一海水浴场的石莼绿潮事件(121°24′59″E, 37°32′6″N, 图 1a)中随机采集了11株石莼属绿藻, 分别编号CNS00533、CNS00592—CNS00601。现场采集的藻体置于内含无菌海水的离心管, 无菌海水中含有f/2培养基、0.5‰ GeO2、50 µg/mL敌百虫和五种复合抗生素(50 µg/mL链霉素, 66.6 µg/mL庆大霉素, 20 µg/mL环丙杀星, 2.2 µg/mL氯霉素, 100 µg/mL氨苄青霉素)(Shibl et al, 2020)。采样后低温保存, 24 h内运输回实验室。藻体样本到达实验室后, 立即用无菌海水冲洗藻体表面。最后, 将藻体置于含有30 mL培养液的培养皿中, 内含f/2培养基、0.5‰ GeO2、50 µg/mL敌百虫和上述五种复合抗生素, 置于光照培养箱进行培养, 培养条件18 ℃, 100—120 μmol photons/(m2·s), L: D = 12 h: 12 h。待镜检无其他微藻、浮游动物等杂质, 即可用于后续实验。
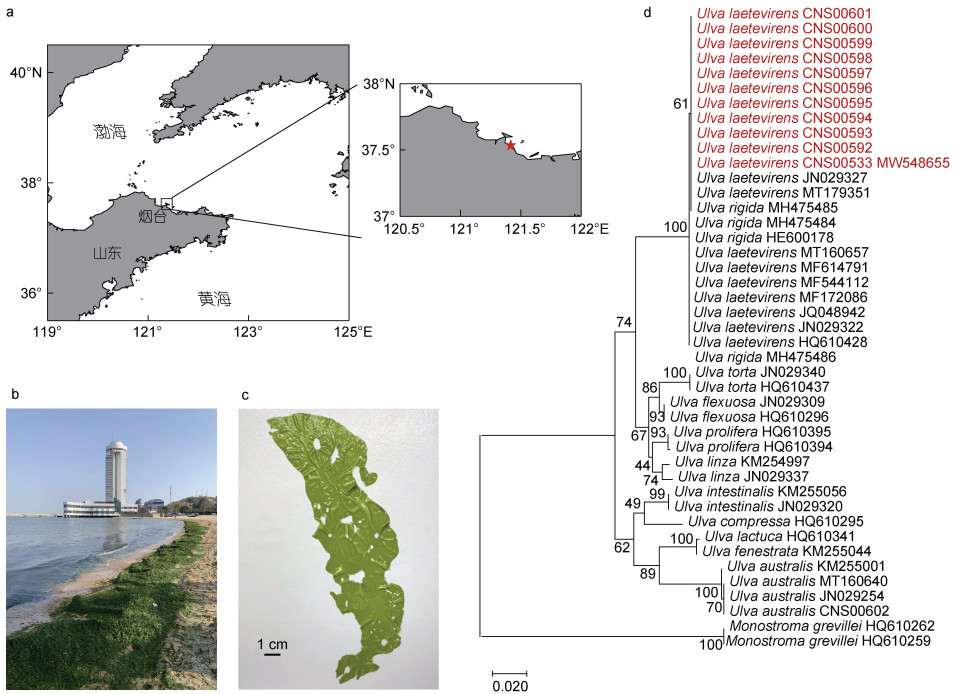 |
| 图 1 烟台局地绿潮及其致灾物种的形态和分子鉴定 Fig. 1 A green tide in Yantai coast and the morphological and molecular analysis of the causative species 注: a. 烟台局地绿潮暴发地, 星号标记为绿潮暴发地点; b. 2020年11月烟台第一海水浴场绿潮现场; c. 物种形态; d. 基于tufA基因构建的系统进化树; 红色字体为本文采集的11株U. laetevirens |
藻体DNA采用植物基因组DNA提取试剂盒(DP305, 北京天根)进行提取, 所提取的DNA经Covaris S220超声破碎仪进行片段化, 获得读长为350 bp的DNA序列; 经末端修复、加A尾、加测序接头、纯化、PCR及产物富集。文库质量分别采用NGS3K/Caliper和real-time PCR (Qubit 3.0 fluorometer, 美国Invitrogen)进行文库大小分布评估和定量分析。质量合格的文库采用美国Illumina NovaSeq PE150 (北京诺禾致源科技股份有限公司)进行测序。
1.3 分子标记tufA的序列获取及系统发育树构建烟台绿潮11个藻体样本的分子标记tufA序列信息通过软件BWA v0.7.17 (Li et al, 2010)、samtools v1.9 (Li et al, 2009)和SPAdes v3.14.0 (Bankevich et al, 2012)从上述全基因组测序数据中筛选组装获得, 所采用的参考序列来自于U. laetevirens叶绿体基因组MT179351注释的tufA基因全长序列。将11个样品和来自GenBank的tufA序列通过Clustal W进行多重比对分析(Thompson et al, 1997), 并辅以手工校正。通过Mega 7.0软件的最大似然法构建进化树(Kumar et al, 2016)。Bootstrap值为1 000。
1.4 叶绿体基因组组装与注释采用GetOrganelle软件(Jin et al, 2020)从头组装获得叶绿体基因组序列, 其中使用的组装软件为SPAdes (3.10.1)(Bankevich et al, 2012), 分析过程中使用的seed参考序列为NCBI登录序列MT179351 (Ulva rigida叶绿体基因组)(Fort et al, 2021)。组装后的叶绿体基因组序列通过BWA v0.7.17软件的MEM运算法对所构建的叶绿体基因组序列进行质检(Li et al, 2010), 并通过IGV v2.8.12进行可视化比对分析(Robinson et al, 2011)。本研究采用在线软件MFannot (https://megasun.bch.umontreal.ca/RNAweasel/)和ORF finder (https://www.ncbi.nlm.nih.gov/orffinder)对叶绿体基因组进行注释。
1.5 共线性分析将CNS00533分别与U. laetevirens MT179351、U. rigida MT179352和U. australis MT179348进行共线性分析, 采用circos-0.69进行作图(Krzywinski et al, 2009)。
1.6 遗传进化分析对19株石莼属绿藻叶绿体基因组的71个蛋白编码基因的氨基酸序列进行遗传进化分析。氨基酸序列采用MAFFT(默认参数)比对(Katoh et al, 2013), 然后通过PhyloSuite进行串联(Smith et al, 2008), 以基因的位置对串联后的序列进行分区, 每个分区的最适氨基酸替代模型通过ModelFinder确定(Kalyaanamoorthy et al, 2017), 以IQ-TREE(默认参数)构建基于氨基酸序列的遗传进化树(Trifinopoulos et al, 2016), bootstrap值设置为1 000。硅藻Lithodesmium undulatum(KC509525)和Odontella sinensis(Z67753)的叶绿体基因组作为外类群。
2 结果 2.1 2020年11月烟台局地绿潮原因种的鉴定2020年11月2日于山东省烟台市第一海水浴场暴发了局地绿潮, 浴场沿岸水体充斥绿色藻体碎片, 在海浪的作用下大量的藻体积留海滩(图 1b)。该绿藻藻体呈叶片状, 多孔、双层基质; 因藻体破碎无法辨别不同部位且未见假根, 难以从形态水平鉴别物种(Mao et al, 2014)(图 1c)。
为鉴定此次烟台绿潮物种, 随机采集了12株藻体样本, 对其构建了基于tufA的单基因遗传进化树(图 1d)。结果显示, 其中11株CNS00533、CNS00592—CNS00601序列完全一致, 且与U. rigida和U. laetevirens的参考序列聚类在一个clade中; 在NCBI数据库中, 11株藻的tufA全长序列(1 224 bp)与U. laetevirens (MT179351)(Fort et al, 2021)的tufA序列(1 224 bp) 100%相同。另外1株CNS00602与U. australis参考序列聚类在一个clade中, 且与U. australis MT160640 (Fort et al, 2021)完全一致。综上所述, 除CNS00602株系以外的11株可以初步鉴定为同一物种——U. laetevirens, 在烟台绿潮的物种组成中占绝对优势, 是造成此次局地绿潮的原因种; 而CNS00602可以初步鉴定为U. australis。
2.2 烟台绿潮物种U. laetevirens CNS00533叶绿体基因组特征相较于单基因分子标记, 集合多基因的叶绿体基因组信息可能提供更高的分辨率, 能够更准确地反映烟台绿潮原因种的分类地位, 进一步确定U. laetevirens、U. rigida以及U. australis的遗传进化关系。为此, 本研究构建并深入解析了烟台绿潮物种的叶绿体基因组。本研究共完成了4个株系CNS00533、CNS00392、CNS00396和CNS00398叶绿体基因组的组装, 4个叶绿体基因组碱基组成完全一致。以CNS00533叶绿体基因组(图 2)为参考, 比较分析了11个株系之间的遗传多样性, 仅发现一个单碱基变异位点, 进一步表明从烟台绿潮中分离得到的这11个株系是同一个株系克隆繁殖的结果。后续分析仅以CNS00533为代表。
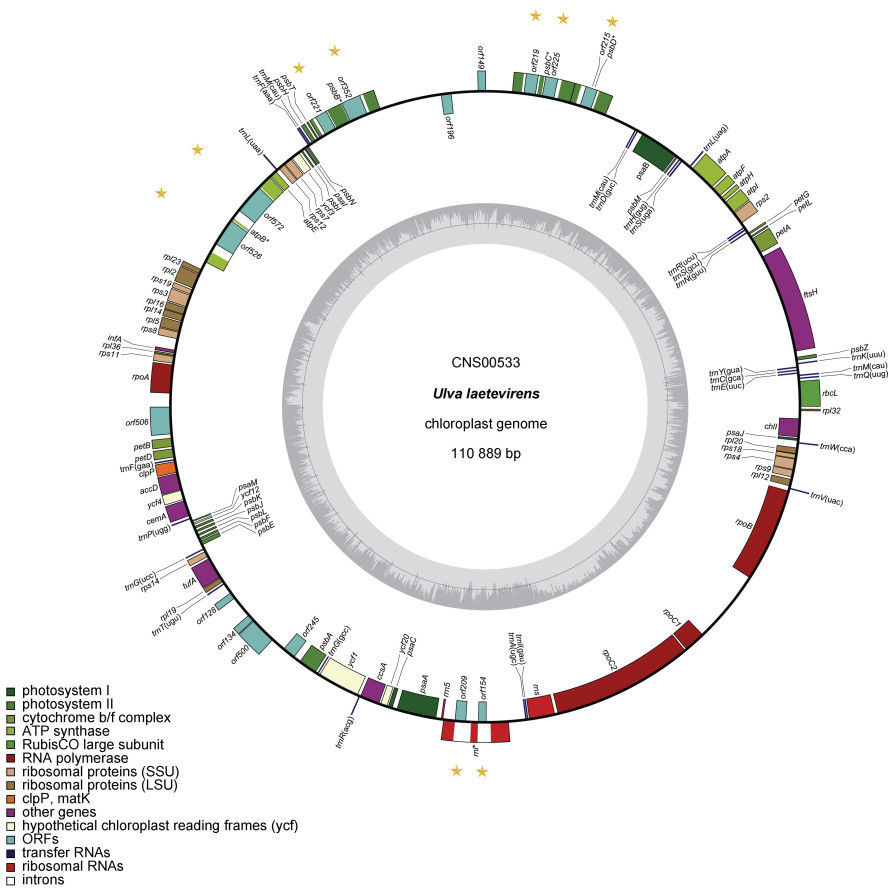 |
| 图 2 烟台绿潮物种U. laetevirens CNS00533叶绿体基因组 Fig. 2 The chloroplast genome of U. laetevirens CNS00533 in the local green tide in Yantai 注: 黄色星号标记指示基因组中内含子所在位置。 |
CNS00533的叶绿体基因组为110 889 bp, 与U. laetevirens (MT179351, 103 444 bp)、U. rigida (MT179352, 96 673 bp)和U. australis (MT179348, 99 820 bp)三个叶绿体基因组相比略长(表 1)。CNS00533叶绿体基因组的AT含量与U. laetevirens (MT179351)比较相近, 74.4%—74.6%, 比U. australis与U. rigida的AT含量(分别为74.8%和75.4%)略低。四个叶绿体基因组共有71个蛋白编码基因、3个rRNA基因(rrl、rrs和rrn5)和26个tRNA基因。CNS00533与U. laetevirens (MT179351)的叶绿体基因组的tRNA基因完全相同, 它们比U. australis多了一个tRNA基因trnF (aaa)。同时, U. rigida相比其他三个叶绿体基因组多了一个trnR (ucu)。总体而言, CNS00533与U. laetevirens (MT179351)的叶绿体基因组的基因组成完全一致, 进一步支持上述对CNS00533的物种鉴定。
| 物种株系 | U. laetevirens
CNS00533 MW531676 |
U. laetevirens MT179351 |
U. rigida MT179352 |
U. australis MT179348 |
| 基因组大小(bp) | 110 889 | 103 444 | 96 673 | 99 820 |
| AT含量(%) | 74.4 | 74.6 | 75.4 | 74.8 |
| 共有/特异蛋白编码基因 | 71/0 | 71/0 | 71/0 | 71/0 |
| 共有/特异tRNA基因 | 26/1 | 26/1 | 26/2 | 26/0 |
| rRNA基因 | rrl-rrs-rrn5 | rrl-rrs-rrn5 | rrl-rrs-rrn5 | rrl-rrs-rrn5 |
| 参考文献 | 本文 | Fort, 2020 | Fort, 2020 | Fort, 2020 |
为比较分析CNS00533与其他3个近缘物种叶绿体基因组的相似度, 本研究将烟台U. laetevirens株系CNS00533 (MW53 1676)分别与U. laetevirens (MT179351)、U. rigida (MT179352)、U. australis (MT179348)进行基因组共线性分析(图 3)。结果显示, CNS00533与U. laetevirens (MT179351)的叶绿体基因组基因数目和基因排列顺序完全一致(图 3a)。CNS00533与U. rigida的叶绿体基因组相比, trnR基因为U. rigida的叶绿体基因组特有(图 3b)。CNS00533与U. australis的叶绿体基因组存在显著的差异, 二者之间存在一个包括45个基因的基因组反转(inversion)事件(图 3c)。同时, 相比于U. australis的叶绿体基因组, CNS00533还多一个基因trnF (aaa)。因此, 共线性分析也支持CNS00533作为U. laetevirens的鉴定。
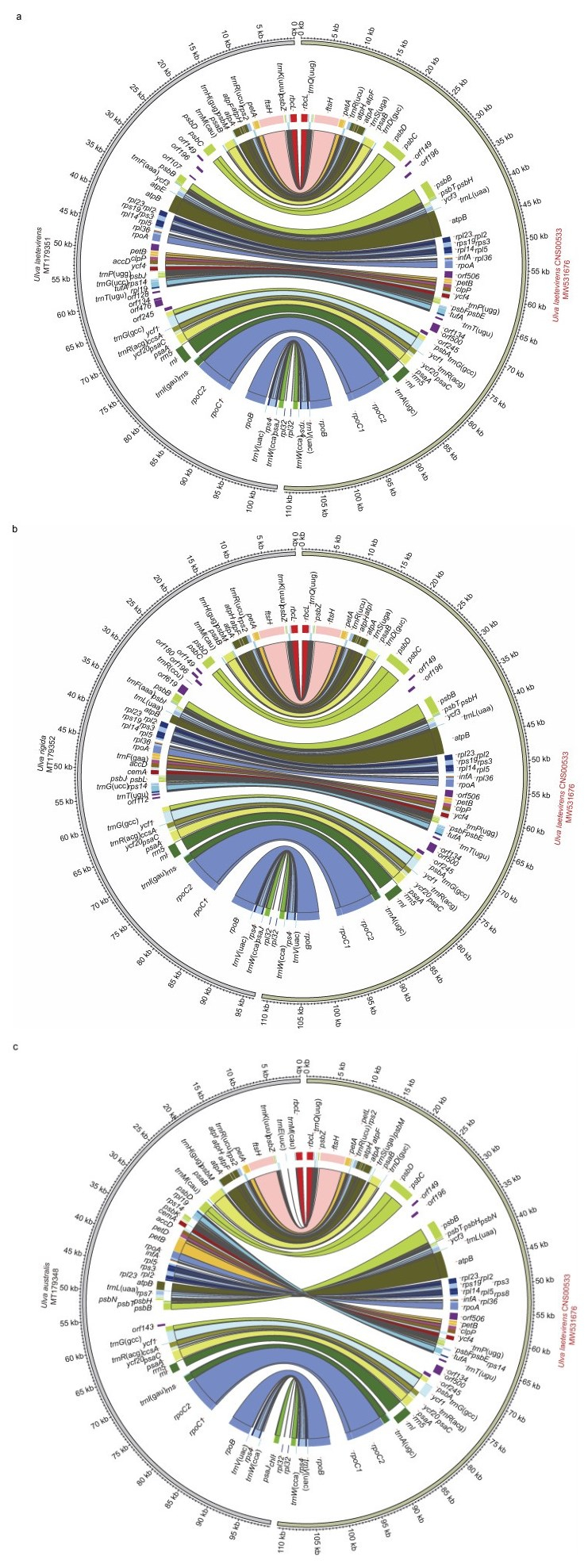 |
| 图 3 U. laetevirens CNS00533 (MW53 1676)与U. laetevirens (MT179351)(a)、U. rigida (MT179352)(b)、U. australis (MT179348)(c)叶绿体基因组共线性分析 Fig. 3 Synteny analysis of chloroplast genomes of U. laetevirens CNS00533 MW531676 vs. U. laetevirens MT179351 (a), U. laetevirens CNS00533 MW531676 vs. U. rigida MT179352 (b), and U. laetevirens CNS00533 MW531676 vs. U. australis MT179348 (c) |
为了探究烟台绿潮物种U. laetevirens CNS00533的遗传进化关系, 本研究针对已发表的19株石莼绿藻和CNS00533的叶绿体基因组, 构建了基于蛋白质编码基因氨基酸序列的遗传进化树(图 4)。结果显示, 石莼属物种聚类为4个大的clade, 分别是Ulva lactuca-ohnoi-laetevirens-rigida-gigantea、Ulva linza- prolifera-flexuosa、Ulva australis-fenestrata- pseudorotundata和Ulva compressa。烟台绿潮原因种CNS00533聚类到Ulva lactuca-ohnoi-laetevirens- rigida-gigantea, 且与U. laetevirens遗传进化距离更近。同时, 该结果也表明U. laetevirens与Ulva australis遗传进化关系较远, 不是同一物种, 与Hughey等(2021)的结论相反。
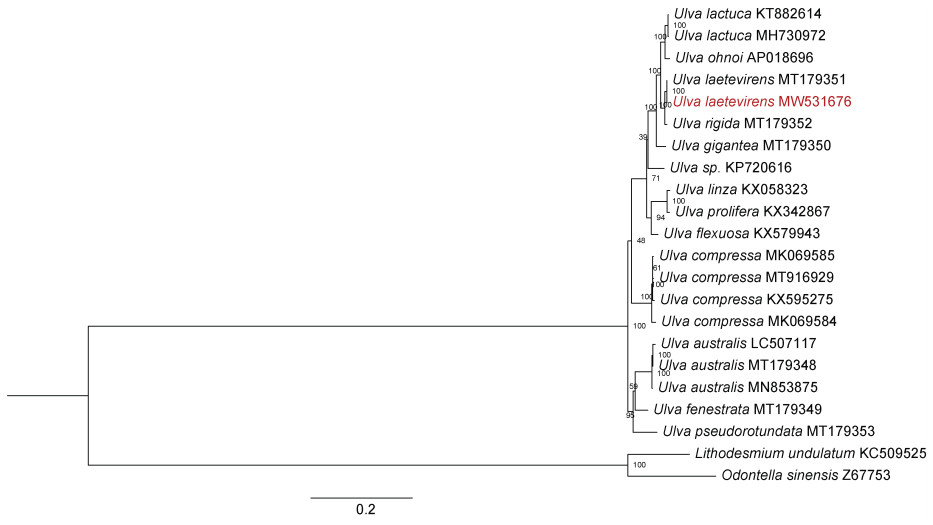 |
| 图 4 针对U. laetevirens CNS00533和其他石莼属叶绿体蛋白质编码基因氨基酸序列的遗传进化树 Fig. 4 The phylogenetic tree based on concatenated amino acid sequences of chloroplast protein-coding genes of U. laetevirens CNS00533 and other Ulva species. 注: 基于71个蛋白编码基因氨基酸水平建立的最大似然法遗传进化树, 以硅藻Lithodesmium undulatum和Odontella sinensis为外类群; 红色字体为本文的研究对象U. laetevirens CNS00533 MW531676 |
上述关于分子标记(tufA)、基因组成、共线性分析和遗传进化关系等多方面的分析表明CNS00533是U. laetevirens的一个株系。比较分析CNS00533和U. laetevirens (MT179351)的叶绿体基因组DNA序列发现, 相较于U. laetevirens MT179351, CNS00533叶绿体基因组(MW53 1676)多出6个较长的插入序列(表 2, 序号1—6), 长度范围1—2 kb。这6个插入序列分别对应6个完整的内含子(表 3, 5个I型和1个II型内含子)。比较分析CNS00533和U. rigida (MT179352)的叶绿体基因组发现, CNS00533叶绿体基因组多了10个插入序列(表 2)。其中1—6号插入序列与上面的6个插入序列相同, 其他4个插入序列分别是基因组atpB、rrl基因内的1个II型和1个I型内含子。为了验证不同物种间非插入序列的相似性, 本研究分析发现CNS00533 MW531676与U. laetevirens MT179351插入序列之外的序列相似性达到99.8%。与此相比, CNS00533 MW531676与U. rigida MT179352的DNA序列相似性只有93.1%。
| 序号 | 插入位置 | 长度/bp | 覆盖区域类型 |
| 1 | 21 550—22 605 | 1 056 | psbD内含子 |
| 2 | 23 615—24 602 | 988 | psbC内含子 |
| 3 | 24 778—25 704 | 927 | psbC内含子 |
| 4 | 35 832—37 130 | 1 299 | psbB内含子 |
| 5 | 37 883—38 822 | 940 | psbB内含子 |
| 6 | 45 740—47 944 | 2 205 | atpB内含子 |
| 7 | 41 7 79—44 142 | 2 346 | atpB内含子 |
| 8 | 54 877—57 108 | 2 232 | orf506及其邻近非编码区 |
| 9 | 66 132—67 019 | 888 | orf128及其邻近非编码区 |
| 10 | 82 733—83 495 | 763 | rrl内含子 |
| 物种株系 | 内含子个数 | 包含内含子的基因 | 内含子 长度 |
内含子 类型 |
内含子内的ORF | ORF长度 |
| U. laetevirens CNS00533 MW531676 |
9 | psbD | 1 055 | I | orf215 | 647 |
| psbC | 987 | I | orf225 | 677 | ||
| 926 | I | orf219 | 659 | |||
| psbB | 1 298 | I | orf352 | 1058 | ||
| 939 | I | orf221 | 665 | |||
| atpB | 2 363 | II | orf572 | 1718 | ||
| 2 204 | II | orf526 | 1580 | |||
| rrl | 952 | I | orf209 | 629 | ||
| 762 | I | orf154 | 464 | |||
| U. laetevirens MT179351 |
3 | atpB | 2 383 | II | orf572 | 1718 |
| rrl | 952 | I | orf209 | 629 | ||
| 762 | I | orf154 | 464 | |||
| U. rigida MT179352 |
1 | rrl | 952 | I | orf209 | 629 |
本研究进一步统计分析了U. laetevirens CNS00533、U. laetevirens (MT179351)和U. rigida (MT179352)叶绿体基因组内含子的分布情况(表 3)。结果显示, U. rigida (MT179352)只有1个I型内含子, 位于其rrl基因内; U. laetevirens (MT179351)有1个位于atpB基因内的II型内含子和2个位于rrl基因内的I型内含子; 相对而言, CNS00533含有7个I型内含子(位于psbD、psbC、psbB和rrl基因内)和2个II型内含子(位于atpB基因内), 远远多于前两者。
综上, CNS00533与U. rigida (MT179352)叶绿体基因组的差异体现在内含子组成和基因编码区, 差异较大; 而CNS00533与U. laetevirens (MT179351)叶绿体基因组基因编码区差异较小, 内含子组成是其种内差异的主要方面。
3 讨论 3.1 U. laetevirens的形态和分子鉴定根据文献报道, U. laetevirens具有掌状叶片、浅裂、不规则分叉、不规则的边缘突起和二层细胞基质等等, 这些形态特征与其他石莼属叶状藻体有不同程度的相似性, 特别是与其近缘的U. rigida极易混淆(Steinhagen et al, 2019b)。Sfriso发现U. rigida的根状体细胞相比U. laetevirens更厚、更暗(Sfriso, 2010)。Sfriso (2010)的研究还发现U. laetevirens的假根和基部边缘没有突起。此外, 石莼属物种具有形态可塑性, 不同的营养环境、光照以及共附生菌均会影响其形态(Taylor et al, 2001; Fu et al, 2008)。总体而言, 仅仅基于形态学的描述, 难以实现对包括U. laetevirens在内的石莼属物种进行准确的鉴定。
分子学方法已成为物种分类的一个有效手段, 可以在形态学鉴定的基础上, 更加准确地鉴定石莼属物种。但是, 分子学方法的准确鉴定仍然依赖于分子标记的选择。在已报道的研究中, U. laetevirens、U. rigida和U. australis分类存在争议。Batista等(2018)基于分子标记ITS和rbcL的分析结果认为U. laetevirens和U. rigida区分不开。Steinhagen等(2019b)甚至基于此在其研究中将U. laetevirens和U. rigida作为同一物种。Hughey等(2021)根据分子标记rbcL分析结果确定U. laetevirens与U. australis为同一物种。Lee等(2019)在分析韩国济州岛的样品时, 尽管采用tufA和rbcL联合建树分析, 但由于其参考序列中没有U. rigida相关序列作为参考, 所以分析结果并没有说明其建树方式能有效区分U. laetevirens和U. rigida。Fort等(2021)的研究中对欧洲海域采集的110株叶状石莼进行单核苷酸多态性(single nucleotide polymorphism, SNP)分析, 结果明确聚类为6个clade, 基于分子标记ITS和rbcL的结果, 6个SNP-clade分别对应6个物种, 其中包括U. laetevirens和U. rigida, 其后续研究均视U. laetevirens和U. rigida为两个物种, 且细胞器基因组均显示二者种间差异极大, 种内差异极小。
此外, Fort等(2021)比较了tufA、rbcL的单基因进化树以及线粒体、叶绿体的全部蛋白编码序列的进化树, 结果显示这四种方式的分类效果基本一致, tufA的bootstrap支持率相对高于rbcL, 认为单分子标记tufA足以区分叶状石莼属物种。本研究为了鉴定烟台株系CNS00533, 对其tufA进行分析, 结果(图 1d)显示CNS00533与U. laetevirens和U. rigida的参考序列聚类在一个clade中, NCBI比对发现仅与U. laetevirens (MT179351)(Fort et al, 2021)的tufA序列(1 224 bp) 100%一致。另外, 根据石莼属属内物种的叶绿体基因组蛋白编码基因的进化树(图 4), CNS00533与U. laetevirens有更近的遗传进化关系。此外, 为了排除叶绿体基因组中可能的插入序列的影响, 本研究剔除了CNS00533与U. laetevirens (MT179351)、U. rigida (MT179352)参考叶绿体基因组彼此间的长片段插入序列后发现, CNS00533与U. laetevirens相似度达到99.8%, 与U. rigida相似度只有93.1%。Fort等(2021)在进行6个石莼属物种种间、种内叶绿体基因组多样性分析时, 发现种间蛋白编码区单核苷酸变异(single nucleotide variant, SNV)位点至少391个, 而U. laetevirens种内只有36个SNV位点。本研究比较分析了CNS00533与爱尔兰U. laetevirens的叶绿体基因组全序列, 发现仅有51个SNV位点, 进一步说明二者为种内差异。以上分子标记、叶绿体蛋白编码基因遗传进化分析和全序列比对的结果, 均表明烟台株系CNS00533为U. laetevirens。
3.2 U. laetevirens烟台株系与爱尔兰株系间的异同同一物种的不同株系之间可能存在遗传变异, 包括大尺度的遗传变异。截至目前已公布的记录中, 仅有爱尔兰海域和中国烟台海域的U. laetevirens株系的叶绿体基因组[MT179351 (Fort et al, 2021)和MW531676]被构建和解析。经比较发现, 二者的叶绿体基因组的基因种类、数量和排列顺序完全一致, 包括71个蛋白编码基因、27个tRNA基因和3个rRNA基因基因(图 2, 3)。Fort等(2021)的研究结果显示欧洲海域U. laetevirens种内差异极低, 主要体现在种内仅0.6 SNVs/kb编码序列。本研究的结果显示烟台株系与爱尔兰株系之间SNV也极低。同时, 烟台株系的叶绿体基因组比爱尔兰株系长7 445 bp, 这个长度差异在于二者内含子的组成差异, 前者比后者多了6个完整的内含子序列(表 2, 3)。种内内含子差异显著的现象并不例外, 本研究在整理NCBI数据库的所有石莼属物种内含子分布情况时发现, U. compressa不同株系的叶绿体基因组中内含子相差较大, MK069584 [U. mutabilis更名为U. compressa (Steinhagen et al, 2019a)]含有10个内含子, 而KX595275只有4个内含子。另外, 在已报道的两株Metschnikowia santaceciliae线粒体中, 二者之间仅cob基因含有的内含子个数就相差6个(Lee et al, 2020)。对于U. laetevirens烟台株系和爱尔兰株系间的内含子差异, 可能是不同地理环境压力下自然选择的结果。
3.3 U. laetevirens是我国新记录石莼属(绿潮)物种截至2021年1月, 根据已有记载信息, 我国海域共记录29个石莼属物种(丁兰平等, 2015; Xie et al, 2020), 其中11个物种已在全球有引发绿潮的报道, 包括U. australis、U. chaugulli、Ulva clathrata、U. compress、U. flexuosa、U. intestinalis、U. linza、U. meridionalis、U. prolifera、Ulva ridiga、U. tepida (Guidone et al, 2013; Smetacek et al, 2013; Gao et al, 2018; Golubkov et al, 2018; Xie et al, 2020)。本研究报道的烟台U. laetevirens是我国绿潮事件中该物种的首次记录。该物种于1854年在澳大利亚维多利亚的菲利普港首次采集并记录, 现已在北美长岛湾、加拿大、爱尔兰、法国、荷兰、葡萄牙、突尼斯和韩国均有采集记录(Kirkendale et al, 2013; Mao et al, 2014; Miladi et al, 2018; Lee et al, 2019; Fort et al, 2021)。此外, 本研究发现在NCBI数据库中U. laetevirens CNS00533的ITS序列与我国研究者Miao等提交的8个标记“Ulva sp.”的ITS序列(KY446828等, 未发表)一致, 说明该物种在我国其他地区也有分布。U. laetevirens在澳洲、美洲、欧洲和亚洲海域均已有绿潮暴发记录, 该物种快速增殖和分布广泛的特征需要我们持续关注和监管。
4 结论本研究聚焦了2020年11月初在烟台第一海水浴场发生的绿潮事件, 通过比较叶绿体基因组学分析证明该石莼属绿藻为U. laetevirens, 该物种在我国海域尚属首次记录。本研究构建了烟台株系U. laetevirens CNS00533的叶绿体基因组(MW53 1676), 相比于爱尔兰株系U. laetevirens (MT179351)二者之间蛋白编码基因、tRNA基因和rRNA基因的种类和数目一致, 仅有51个SNV位点, 序列组成差异在于烟台株系的叶绿体基因组的4个基因psbD、psbC、psbB和atpB中共多了6个内含子。U. laetevirens烟台株系与爱尔兰株系可能存在其他潜在的差异, 包括核基因组和线粒体基因组的组成差异等, 这些需要进一步解析以期为理解我国U. laetevirens绿潮暴发机制提供分子理论支持。对我们海域石莼属绿潮物种(包括新记录种)的全面调查和生物地理分布研究, 将是对我国石莼绿潮实施有效监管和可靠预测的必要工作之一。
丁兰平, 黄冰心, 栾日孝. 2015. 中国海洋绿藻门新分类系统. 广西科学, 22(2): 201-210 DOI:10.3969/j.issn.1005-9164.2015.02.013 |
董美龄. 1963. 中国浒苔属植物地理学的初步研究. 海洋与湖沼, 4(1): 46-51 |
马元庆, 邢红艳, 宋秀凯, 等. 2010. 烟台金沙滩绿潮爆发的基本状况与应对措施. 齐鲁渔业, 27(4): 10-12 |
Bäck S, Lehvo A, Blomster J, 2000. Mass occurrence of unattached Enteromorpha intestinalis on the Finnish Baltic Sea coast. Annales Botanici Fennici, 37: 155-161 |
Bankevich A, Nurk S, Antipov D et al, 2012. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology, 19(5): 455-477 DOI:10.1089/cmb.2012.0021 |
Batista M B, Cunha R L, Castilho R et al, 2018. Sea lettuce systematics: lumping or splitting?. BioRxiv, https://doi.org/10.1101/413450
|
Chen L, Feng J, Xie S L, 2015. Ulva shanxiensis (Ulvaceae), a new species from Shanxi, China. Novon: A Journal for Botanical Nomenclature, 23(4): 397-405 DOI:10.3417/2013021 |
Cui J J, Monotilla A P, Zhu W R et al, 2018. Taxonomic reassessment of Ulva prolifera (Ulvophyceae, Chlorophyta) based on specimens from the type locality and Yellow Sea green tides. Phycologia, 57(6): 692-704 DOI:10.2216/17-139.1 |
De Paula Silva P H, Paul N A, de Nys R et al, 2008. Integrating filamentous 'green tide' algae into tropical pond-based aquaculture. Aquaculture, 284(1/4): 74-80 |
De Paula Silva P H, Paul N A, de Nys R et al, 2013. Enhanced production of green tide algal biomass through additional carbon supply. PLoS One, 8(12): e81164 DOI:10.1371/journal.pone.0081164 |
Fletcher R L, 1996. The Occurrence of "green tides"-a review. In: Schramm W, Nienhuis P H eds. Marine Benthic Vegetation: Recent Changes and the Effects of Eutrophication. Berlin, Heidelberg: Springer, 7-43
|
Fort A, Mannion C, Fariñas-Franco J M et al, 2020. Green tides select for fast expanding Ulva strains. Science of the Total Environment, 698: 134337 DOI:10.1016/j.scitotenv.2019.134337 |
Fort A, McHale M, Cascella K et al, 2021. Foliose Ulva species show considerable inter-specific genetic diversity, low intra-specific genetic variation, and the rare occurrence of inter-specific hybrids in the wild. Journal of Phycology, 57(1): 219-233 DOI:10.1111/jpy.13079 |
Fu G, Yao J T, Liu F L et al, 2008. Effect of temperature and irradiance on the growth and reproduction of Enteromorpha prolifera J. Ag. (Chlorophycophyta, Chlorophyceae). Chinese Journal of Oceanology and Limnology, 26(4): 357-362 DOI:10.1007/s00343-008-0357-0 |
Gao G, Beardall J, Bao M L et al, 2018. Ocean acidification and nutrient limitation synergistically reduce growth and photosynthetic performances of a green tide alga Ulva linza. Biogeosciences, 15(11): 3409-3420 DOI:10.5194/bg-15-3409-2018 |
Gary W, Hana K, 2010. An evaluation of rbcL, tufA, UPA, LSU and ITS as DNA barcode markers for the marine green macroalgae. Cryptogamie Algologie, 31: 487-528 |
Golubkov S M, Berezina N A, Gubelit Y I et al, 2018. A relative contribution of carbon from green tide algae Cladophora glomerata and Ulva intestinalis in the coastal food webs in the Neva Estuary (Baltic Sea). Marine Pollution Bulletin, 126: 43-50 DOI:10.1016/j.marpolbul.2017.10.032 |
Guidone M, Thornber C S, 2013. Examination of Ulva bloom species richness and relative abundance reveals two cryptically co-occurring bloom species in Narragansett Bay, Rhode Island. Harmful Algae, 24: 1-9 DOI:10.1016/j.hal.2012.12.007 |
Hayden H S, Blomster J, Maggs C A et al, 2003. Linnaeus was right all along: Ulva and Enteromorpha are not distinct genera. European Journal of Phycology, 38(3): 277-294 DOI:10.1080/1364253031000136321 |
Hughey J R, Gabrielson P W, Maggs C A et al, 2021. Taxonomic revisions based on genetic analysis of type specimens of Ulva conglobata, U. laetevirens, U. pertusa and U. spathulata (Ulvales, Chlorophyta). Phycological Research, 69(2): 148-153 DOI:10.1111/pre.12450 |
Jin J J, Yu W B, Yang J B et al, 2020. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes. Genome Biology, 21(1): 241 DOI:10.1186/s13059-020-02154-5 |
Kalyaanamoorthy S, Minh B Q, Wong T K F et al, 2017. ModelFinder: fast model selection for accurate phylogenetic estimates. Nature Methods, 14(6): 587-589 DOI:10.1038/nmeth.4285 |
Katoh K, Standley D M, 2013. MAFFT multiple sequence alignment software version 7:improvements in performance and usability. Molecular Biology and Evolution, 30(4): 772-780 DOI:10.1093/molbev/mst010 |
Kirkendale L, Saunders G W, Winberg P, 2013. A molecular survey of Ulva (Chlorophyta) in temperate Australia reveals enhanced levels of cosmopolitanism. Journal of Phycology, 49(1): 69-81 DOI:10.1111/jpy.12016 |
Kolbe K, Kaminski E, Michaelis H et al, 1995. Macroalgal mass development in the Wadden sea: first experiences with a monitoring system. Helgoländer Meeresuntersuchungen, 49(1): 519-528 DOI:10.1007/BF02368379 |
Krzywinski M, Schein J, Birol İ et al, 2009. Circos: an information aesthetic for comparative genomics. Genome Research, 19(9): 1639-1645 DOI:10.1101/gr.092759.109 |
Kumar S, Stecher G, Tamura K, 2016. MEGA7:Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 33(7): 1870-1874 DOI:10.1093/molbev/msw054 |
Largo D B, Sembrano J, Hiraoka M et al, 2004. Taxonomic and ecological profile of 'green tide' species of Ulva (Ulvales, Chlorophyta) in central Philippines. Hydrobiologia, 512(1): 247-253 DOI:10.1023%2FB%3AHYDR.0000020333.33039.4b |
Lee D K, Hsiang T, Lachance M A et al, 2020. The strange mitochondrial genomes of Metschnikowia yeasts. Current Biology, 30(14): R800-R801 DOI:10.1016/j.cub.2020.05.075 |
Lee H W, Kang J C, Kim M S, 2019. Taxonomy of Ulva causing blooms from Jeju Island, Korea with new species, U. pseudo-ohnoi sp. nov. (Ulvales, Chlorophyta). Algae, 34(4): 253-266 DOI:10.4490/algae.2019.34.12.9 |
Li H, Durbin R, 2010. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics, 26(5): 589-595 DOI:10.1093/bioinformatics/btp698 |
Li H, Handsaker B, Wysoker A et al, 2009. The sequence alignment/map format and SAMtools. Bioinformatics, 25(16): 2078-2079 DOI:10.1093/bioinformatics/btp352 |
Liu F, Pang S J, Chopin T et al, 2013. Understanding the recurrent large-scale green tide in the Yellow Sea: Temporal and spatial correlations between multiple geographical, aquacultural and biological factors. Marine Environmental Research, 83: 38-47 DOI:10.1016/j.marenvres.2012.10.007 |
Liu C Y, Xu G B, Deng X et al, 2020. Changes in concentrations of biogenic sulfur compounds in coastal waters off Qingdao, China during an Ulva prolifera bloom. Marine Pollution Bulletin, 152: 110940 DOI:10.1016/j.marpolbul.2020.110940 |
Lyons D A, Arvanitidis C, Blight A J et al, 2014. Macroalgal blooms alter community structure and primary productivity in marine ecosystems. Global Change Biology, 20(9): 2712-2724 DOI:10.1111/gcb.12644 |
Mao Y X, Kim J K, Wilson R et al, 2014. The appearance of Ulva laetevirens (Ulvophyceae, Chlorophyta) in the northeast coast of the United States of America. Journal of Ocean University of China, 13(5): 865-870 DOI:10.1007/s11802-014-2419-6 |
Miladi R, Manghisi A, Armeli Minicante S et al, 2018. A DNA barcoding survey of Ulva (Chlorophyta) in Tunisia and Italy reveals the presence of the overlooked alien U. ohnoi. Cryptogamie Algologie, 39(1): 85-107 DOI:10.7872/crya/v39.iss1.2018.85 |
Mudassar A K, Monica G K, Veerendra V S, 2016. Morphological and molecular characterization of Ulva chaugulii sp. nov., U. lactuca and U. ohnoi (Ulvophyceae, Chlorophyta) from India. Phycologia, 55(1): 45-54 DOI:10.2216/15-11.1 |
Nelson T A, Lee D J, Smith B C, 2003. Are "green tides" harmful algal blooms? Toxic properties of water-soluble extracts from two bloom-forming macroalgae, Ulva fenestrata and Ulvaria Obscura (Ulvophyceae). Journal of Phycology, 39(5): 874-879 DOI:10.1046/j.1529-8817.2003.02157.x |
Robinson J T, Thorvaldsdóttir H, Winckler W et al, 2011. Integrative genomics viewer. Nature Biotechnology, 29(1): 24-26 DOI:10.1038/nbt.1754 |
Runca E, Bernstein A, Postma L et al, 1996. Control of macroalgae blooms in the Lagoon of Venice. Ocean & Coastal Management, 30(2/3): 235-257 |
Sfriso A, 2010. Coexistence of Ulva rigida and Ulva laetevirens (Ulvales, Chlorophyta) in Venice Lagoon and other Italian transitional and marine environments. Botanica Marina, 53(1): 9-18 |
Shibl A A, Isaac A, Ochsenkühn M A et al, 2020. Diatom modulation of select bacteria through use of two unique secondary metabolites. Proceedings of the National Academy of Sciences of the United States of America, 117(44): 27445-27455 DOI:10.1073/pnas.2012088117 |
Shimada S, Hiraoka M, Nabata S et al, 2003. Molecular phylogenetic analyses of the Japanese Ulva and Enteromorpha (Ulvales, Ulvophyceae), with special reference to the free-floating Ulva. Phycological Research, 51(2): 99-108 DOI:10.1111/j.1440-1835.2003.tb00176.x |
Smetacek V, Zingone A, 2013. Green and golden seaweed tides on the rise. Nature, 504(7478): 84-88 DOI:10.1038/nature12860 |
Smith S A, Dunn C W, 2008. Phyutility: a phyloinformatics tool for trees, alignments and molecular data. Bioinformatics, 24(5): 715-716 DOI:10.1093/bioinformatics/btm619 |
Song W, Wang Z L, Li Y et al, 2019. Tracking the original source of the green tides in the Bohai Sea, China. Estuarine, Coastal and Shelf Science, 219: 354-362 DOI:10.1016/j.ecss.2019.02.036 |
Steinhagen S, Barco A, Wichard T et al, 2019a. Conspecificity of the model organism Ulva mutabilis and Ulva compressa (Ulvophyceae, Chlorophyta). Journal of Phycology, 55(1): 25-36 DOI:10.1111/jpy.12804 |
Steinhagen S, Karez R, Weinberger F, 2019b. Cryptic, alien and lost species: molecular diversity of Ulva sensu lato along the German coasts of the North and Baltic Seas. European Journal of Phycology, 54(3): 466-483 DOI:10.1080/09670262.2019.1597925 |
Taylor R, Fletcher R L, Raven J A, 2001. Preliminary studies on the growth of selected 'green tide' algae in laboratory culture: effects of irradiance, temperature, salinity and nutrients on growth rate. Botanica Marina, 44(4): 327-336 |
Thompson J D, Gibson T J, Plewniak F et al, 1997. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 25(24): 4876-4882 DOI:10.1093/nar/25.24.4876 |
Trifinopoulos J, Nguyen L T, von Haeseler A et al, 2016. W-IQ-TREE: a fast online phylogenetic tool for maximum likelihood analysis. Nucleic Acids Research, 44(W1): W232-W235 DOI:10.1093/nar/gkw256 |
Xie W F, Wu C H, Zhao J et al, 2020. New records of Ulva spp. (Ulvophyceae, Chlorophyta) in China, with special reference to an unusual morphology of U. meridionalis forming green tides. European Journal of Phycology, 55(4): 412-425 DOI:10.1080/09670262.2020.1740946 |
 2021, Vol. 52
2021, Vol. 52


