中国海洋湖沼学会主办。
文章信息
- 李碧瀚, 于洋, 刘桂嘉, 罗正, 李富花. 2021.
- LI Bi-Han, YU Yang, LIU Gui-Jia, LUO Zheng, LI Fu-Hua. 2021.
- 基于三代测序技术的凡纳滨对虾(Litopenaeus vannamei)高通量候选基因关联分析方法的建立
- A HIGH-THROUGHPUT METHOD FOR CANDIDATE GENE ASSOCIATION ANALYSIS IN LITOPENAEUS VANNAMEI BASED ON THE THIRD-GENERATION SEQUENCING TECHNOLOGY
- 海洋与湖沼, 52(5): 1255-1264
- Oceanologia et Limnologia Sinica, 52(5): 1255-1264.
- http://dx.doi.org/10.11693/hyhz20210300071
文章历史
-
收稿日期:2021-03-19
收修改稿日期:2021-05-08
2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
水产动物的经济性状大多是由微效多基因控制的数量性状, 如生长速率、抗病性等(Yue, 2014; Gjedrem, 2015)。然而数量性状易受环境等因素影响, 导致了遗传评估准确率偏低。抗性性状由于表型度量困难、遗传力低, 加之与生长或其他性状之间存在负相关, 使得利用传统育种技术对抗病性状进行选育的效率较低(Cock et al, 2009)。随着分子标记, 特别是以单核苷酸多态性(SNP)标记为主的基因分型技术的发展, 分子标记辅助选择(MAS)和基因组选择(GS)等分子育种技术在水产动物抗病育种中彰显了巨大优势, 比如个体评估的准确性高, 可在繁殖周期的早期进行选择等。尽管分子育种技术优势大, 但是在大多数水产动物的商业化育种中的应用仍然较少。在水产动物中开展高效分子育种面临着两大难点, 一者是缺少高密度、高精度的性状连锁标记(Meuwissen et al, 2004), 二者是对标记和性状之间关联程度的评估有待优化(VanRaden et al, 2009)。因此鉴定与性状紧密相关的基因或基因位点是水产动物分子育种亟需解决的问题。
鉴定性状相关位点的主要方法包括全基因组关联分析(GWAS)和候选基因关联分析(Xu et al, 2009)。其中, GWAS已经在人类疾病相关基因的鉴定和农作物、畜禽经济性状相关SNP鉴定中得到广泛应用, 而水产养殖物种中GWAS研究起步较晚, 主要应用于罗非鱼、大西洋鲑鱼、鲤鱼和太平洋牡蛎等几种研究基础较好的水产物种(Houston et al, 2020)。与GWAS相比, 候选基因关联分析更加便捷、高效, 在鱼类、贝类与虾蟹类等多种水产经济物种中得到了较好的应用(Yue, 2014; Yu et al, 2020)。随着测序技术的不断发展, 科研人员已成功破译了多个水产动物的全基因组(Lien et al, 2016; Zhang et al, 2019b; Houston et al, 2020), 并构建了高密度遗传连锁图谱(Yu et al, 2015; Yue et al, 2017), 筛选到大量分子标记, 为候选基因关联分析和性状相关标记的筛选奠定了重要基础。
然而对水产动物候选基因进行关联分析仍然缺乏高效、低成本的分型方法, 制约了相关技术的应用。目前, 主要的基因分型方法包括基因芯片、SNaPshot法、Taqman分型、MassArray飞行质谱法、PCR产物一代测序法、二代靶向测序法等。芯片法需要单独定制芯片, 造价高; SNaPshot法、Taqman分型、MassArray飞行质谱法可以对候选基因进行分型, 但是只能分析已知的SNP位点, 且单个标记的分型成本仍然较高; 一代测序分型是指通过PCR产物直接测序分型, 优势在于精确度高、速度快, 但是其分型结果受DNA序列结构影响大、读长有限(< 1 000 bp), 且测序通量低、成本高; 二代靶向测序指通过探针捕获或者PCR扩增目标片段, 之后对该片段进行高通量测序, 该技术能够发掘新的SNP位点, 且价格相对较低, 但是对于长的序列片段捕获探针设计成本高, 且PCR建库过程复杂。近年来, 随着三代测序技术的发展, 基于CCS的测序技术准确率不断提高, 加大测序深度至15×后, HIFI数据准确率可达99.3% (Eid et al, 2009), 且由于三代测序具有长度长(平均5 000 bp), 无偏好性、准确性高等特点, 使得利用三代测序进行基因分型成为可能(Rhoads et al, 2015), 尤其是利用其长读长以及无系统偏差和无GC偏好性的特点, 无需对目的序列进行分段扩增以及花费大量精力组装拼接测序结果, 因此在解决复杂基因组的基因分型方面具有明显优势。
凡纳滨对虾(Litopenaeus vannamei)具有生长速度快、抗逆能力强、肉质鲜美且出肉率高等特点, 是世界公认的优良对虾养殖品种。然而, 随着养殖环境的恶化, 细菌病、病毒病的频发, 严重制约了对虾产业的健康可持续发展。其中, 带有毒素质粒的副溶血弧菌(Vibrio parahaemolyticus)所引发的对虾早期死亡综合征(Early Mortality Syndrome, EMS), 又称为急性肝胰腺坏死症(Acute Hepatopancreatic Necrosis Disease, AHPND) (Lee et al, 2015), 具有发病迅速、传染性强、致死率高的特点, 给对虾养殖业造成了巨大损失。抗病品种的选育被认为是解决病害问题的根本途径。然而由于抗病性状的表型难以度量, 且抗病性状的遗传力通常较低, 受环境的影响较大, 利用传统选育技术进行选育的准确性低。分子育种在抗病品种的选育中具有其独特优势, 抗病分子标记和基因的筛选是进行分子育种的基础, 目前有关对虾抗病性状相关基因和标记的筛选进展相当缓慢, 缺乏高通量、低成本的基因分型技术是一个重要的限制因素。本团队前期通过对目标序列进行混池测序分型的方式, 筛选到了多个与对虾抗弧菌性状相关的SNP标记和基因, 其中LvPI3K的一个SNP标记与抗弧菌性状呈显著相关关系(Zhang et al, 2019a)。
本研究基于三代测序技术, 在凡纳滨对虾中建立了一种基于三代靶向测序的候选基因关联分析技术, 并对前期筛选的抗弧菌性状相关基因LvPI3K进行了标记发掘和关联分析, 以筛选抗弧菌性状相关标记。本研究不仅为水产动物提供了一种高效、低成本的候选基因关联分析方法, 也为凡纳滨对虾的抗弧菌品种选育提供了有效的分子标记。
1 材料与方法 1.1 实验材料 1.1.1 候选基因关联分析材料的获得凡纳滨对虾遗传材料来自渤海水产育种(海南)有限公司(海南, 文昌), 分析用的群体为经多代选育的多个弧菌抗性家系和敏感家系的杂交F2代群体。
随机选择500尾个体, 利用副溶血弧菌进行浸泡感染。感染所用的副溶血弧菌为从患病个体中分离出的副溶血弧菌菌株, 在含有2%氯化钠的胰蛋白酶大豆肉汤(TBS)液体培养基中30 ℃恒温培养过夜。经对毒素质粒PirA、PirB的PCR扩增检测为阳性, 确定其为致病性的副溶血弧菌(Han et al, 2015)。采用血球计数板在光学显微镜下进行计数, 计算菌液浓度。设定弧菌浸泡感染浓度为5×106 CFU/mL。弧菌感染实验持续8 d, 取最先死亡的96尾虾为敏感组样品, 最后存活的96尾虾为抗性组样品, 记录其存活和死亡时间, 并将样品保存在–80 ℃直至DNA提取。采用TIANGEN植物DNA提取试剂盒(TIANGEN, 北京)逐个提取DNA, 采用NanoDrop 1000分光光度计(NanoDrop, 美国)和琼脂糖凝胶电泳以检查DNA的浓度和质量。
1.1.2 验证群体材料的获得验证群体为经多代选育的多个抗弧菌家系和敏感家系的杂交F2代群体, 验证群体抗性与敏感材料的获得方法与1.1.1所示相同, 并且扩大了验证群体样本, 选择弧菌感染后最先死亡的160尾虾为敏感材料, 最后死亡的160尾虾为抗性材料。DNA提取方式同1.1.1。
1.2 LvPI3K基因组DNA扩增根据凡纳滨对虾基因组参考序列(Zhang et al, 2019b), 获得LvPI3K的基因组全长序列, 共计7 378 bp, 使用primer 3设计LvPI3K特异性扩增引物。为实现在一个单分子实时测序单元(Single Molecule Real Time cell, SMRT cell)中区分每一个抗性个体和敏感个体的序列信息, 通过两步PCR的方式进行扩增。
第1步PCR: 以提取的抗弧菌和敏感个体DNA为模板, 在LvPI3K的特异引物的5'端加上通用引物序列作为第一步PCR的复合引物(图 1, 表 1), 并将该引物的5'末端用5AmMC6修饰封闭。采用PrimeSTAR GXL聚合酶(TaKaRa, 日本)进行扩增, 扩增体系为: 50 ng模板DNA, 10 pmol引物F/R, 4 μL dNTP混合物, 10 μL 5×buffer, 1 μL GXL酶, 灭菌水补充体系至50 μL。PCR循环参数为: 98 ℃ 10 s, 60 ℃ 15 s, 68 ℃ 80 s, 共30个循环。
 |
| 图 1 两步PCR将barcode整合进扩增产物 Fig. 1 Incorporation of barcodes into the PCR amplicon via a two-step PCR approach |
| 步骤 | 引物序列 | 引物序列结构 | PCR产物长度 | |
| 第一轮PCR | F | 5′-gcagtcgaacatgtagctgactcaggtcac GCTTGCATATAACGAATGTACAGACA-3′ |
5′-/5AmMC6/F接头序列-F特异序列-3′ | 7 378 bp |
| R | 5′-tggatcacttgtgcaagcatcacatcgtag AAATCTGTTTGACTGCACTGTATAGC-3′ |
5′-/5AmMC6/R接头序列-R特异序列-3′ | ||
| 第二轮PCR | F | 5′-GCATCCACATATCAGAGTGCG gcagtcgaacatgtagctgactcaggtcac-3′ |
5′-barcode-F接头序列-3′ | 7 480 bp |
| R | 5′-GCATCCACATATCAGAGTGCG tggatcacttgtgcaagcatcacatcgtag-3′ |
5′-barcode-R接头序列-3′ | ||
第2步PCR: 引入每个样本专属的barcode, 以便在PacBio RSⅡ上进行多样品平行测序。以第1步PCR产物稀释50倍为模板, 引物为5′端带有独特barcode序列的通用引物序列(图 1)。使用PrimeSTAR GXL聚合酶(TaKaRa, 日本)进行扩增, 体系为: 1 μL稀释50×的第一步PCR的产物, 10 pmol引物F/R, 4 μL dNTP混合物, 10 μL 5×buffer, 1 μL GXL酶, 灭菌水补充体系至50 μL。PCR循环参数为: 98 ℃ 10 s, 60 ℃ 15 s, 68 ℃ 80 s, 共18个循环。
1.3 SMRT文库的构建及PacBio测序将192个个体的第二轮PCR扩增产物等摩尔混合后, 样品送至天津生物芯片有限公司进行SMRT文库的构建及PacBio测序, 文库构建方式参考SMRT bell Express Template Prep Kit 2.0的建库方案。
1.4 数据分析依据PacBio的数据分析流程, 使用SMRT link9.0对测序原始数据进行过滤, 获得CCS的原始数据。之后使用Lima对FASTA格式的原始CCS数据进行barcode拆分, 输出FASTQ格式文件, 使用DeepVariant将该文件与参考基因组文件比对, 并进行变异位点的发掘(Poplin et al, 2018), 使用WhatsHap构建单倍型后(Martin et al, 2016), 再次使用DeepVariant软件根据构建的单倍型发掘变异位点, 获得结构变异(图 2)。分型数据使用VCFtools转换为plink格式, 之后结合抗性和敏感的表型数据, 使用R软件进行Logistic关联分析并作图, 使用Haploview进行单倍型分析(Barrett et al, 2005)。
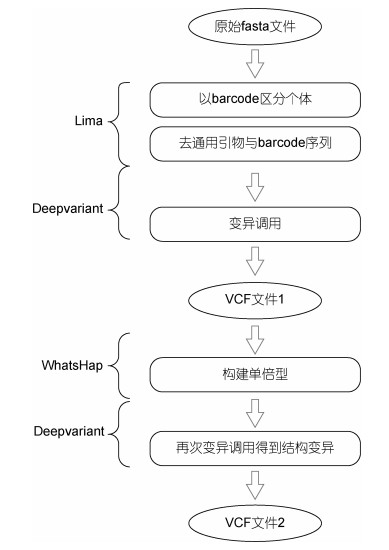 |
| 图 2 SMRT测序数据分析流程图 Fig. 2 Flowchart of the SMRT sequencing analysis |
使用FastQC与MultiQC软件对SMRT测序数据进行整体的质量分析。使用SAMtools及PLINK软件对FASTQ文件进行位点测序深度与reads长度的统计, 并使用R软件进行做图。
1.5 对虾抗弧菌性状相关候选位点的验证抗性群体和敏感群体等实验材料的选择, 对于突变位点的筛选与关联分析有着直接的影响。为了排除群体差异, 本研究选择了另外的遗传背景丰富的群体作为验证材料, 对三代测序的分型结果加以验证, 主要针对两个与抗弧菌性状极显著相关(P < 0.01)的位点(PI3K_5366, PI3K_2205), 基于SNP的侧翼序列设计引物, 在320个验证个体中进行PCR扩增和测序。使用PrimeSTAR GXL聚合酶(TaKaRa, 日本)进行扩增, 体系组成参考1.2所述, PCR扩增参数为: 98 ℃ 10 s, 58 ℃ 15 s, 68 ℃ 45 s, 共30个循环。PCR产物送青岛睿博生物技术有限公司进行Sanger测序, 基于测序峰图确定每个样品的基因型, 分型结果使用R软件进行关联分析。
2 结果 2.1 PacBio测序数据分析目的片段LvPI3K基因全长扩增子序列长度为7 378 bp (图 3a), 采用本实验室构建的针对三代测序数据的专用分析流程, 对测序结果进行了全面分析。统计显示有99.89%的位点测序深度大于20 000×, 位点平均测序深度为22 012×。对样品的测序reads总数进行统计, 结果显示, 测序的192个个体共产生了22 089条reads, 其中有21 990条(99.6%) reads长度在目的片段长度周围(7.4—7.6 kb) (图 3b)。通过FastQC与MultiQC软件对测序数据质量进行分析, 发现除极少数序列外, 三代测序数据均符合质控标准(Q30 > 90), 并且全部样品的平均测序深度为139×, 最高达902×, 测序质量和测序深度均可以满足后续变异发掘的要求(Edge et al, 2019; Wenger et al, 2019)。
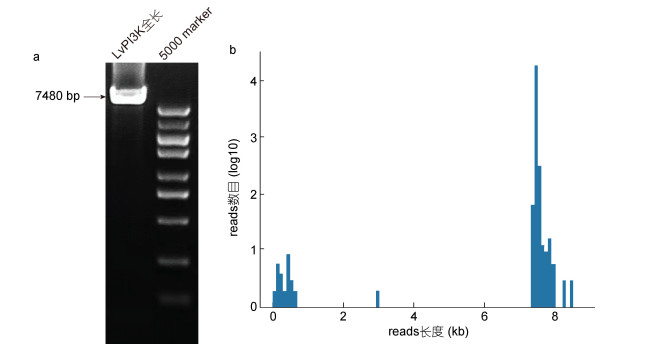 |
| 图 3 LvPI3K基因PCR扩增产物的琼脂糖凝胶电泳图(a)以及测序reads长度分布特征图(b) Fig. 3 Detection of PCR amplification products of LvPI3K by agarose gel electrophoresis (a) and length distribution of sequencing reads (b) |
使用变异调用软件DeepVariant在LvPI3K基因内共筛选到91个突变位点, 最小等位基因频率结果如图 4所示, 以最小等位基因频率(Minor Allele Frequency, MAF)小于0.05作为标准进行稀有突变位点过滤, 最后得到65个突变位点。这些位点中包括1个三等位SNP位点, 11个indel, 其余均为二等位SNP位点。对SNP的分布情况进行分析, 发现内含子区域内的突变有36个, 在5'-UTR区域的突变有24个, 其余4个位于外显子上。
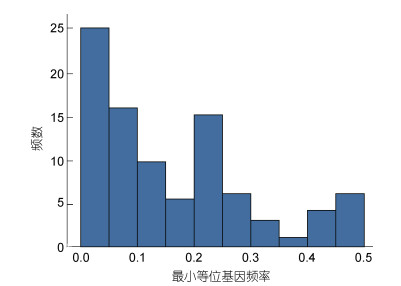 |
| 图 4 质控前筛选到的所有突变位点MAF分布直方图 Fig. 4 The minor allele frequency (MAF) distribution of the SNPs before quality control |
为了降低由于不同关联分析策略而导致的分析结果差异, 我们利用存活情况(阈值性状)与存活时间(数量性状)两个指标对筛选到的突变位点进行关联分析。经过Logistic关联分析, 最终筛选到21个与抗弧菌性状显著相关(P < 0.05)的位点(表 2), 其中有5个SNPs位于5′-UTR, 1个SNP位于外显子区域, 12个SNPs位于内含子, 3个Indel突变也均位于内含子区域。在21个抗性相关位点中, 有两个位点PI3K_5366和PI3K_2205与抗弧菌性状呈极显著相关(P < 0.01) (图 5a, 5b)。
| 标记 | 所在序列结构 | 所在位置 | 基因型 | 不同基因型个体数 | 基因型P值 | 抗性优势等位基因 | 抗性等位基因频率 | 卡方(χ2) | 等位基因P值 | ||
| R | S | R | S | ||||||||
| PI3K_254_T/C | 5′-UTR | pos.254 | TT: TC: CC | 77:7:0 | 60:10:2 | 0.042 9 | T | 0.097 | 0.042 | 3.812 | 0.050 9 |
| PI3K_258_T/A | 5′-UTR | pos.258 | TT: TA: AA | 39:34:0 | 42:20:1 | 0.031 7 | T | 0.828 | 0.730 | 3.816 | 0.050 8 |
| PI3K_264_T/C | 5′-UTR | pos.264 | TT: TC: CC | 77:7:0 | 60:10:2 | 0.042 9 | T | 0.097 | 0.042 | 3.812 | 0.050 9 |
| PI3K_1929_T/A | 5′-UTR | pos.1929 | TT: TA: AA | 40:34:4 | 41:17:1 | 0.016 3 | T | 0.842 | 0.731 | 4.845 | 0.027 7 |
| PI3K_1932_A/T | 5′-UTR | pos.1932 | AA: AT: TT | 39:37:4 | 40:22:1 | 0.042 3 | A | 0.812 | 0.719 | 3.431 | 0.064 0 |
| PI3K_2112_T/G | 内含子 | pos.2112 | TT: TG: GG | 54:25:1 | 37:19:6 | 0.036 6 | T | 0.250 | 0.169 | 2.838 | 0.092 1 |
| PI3K_2134_C/T | 内含子 | pos.2134 | CC: CT: TT | 52:20:1 | 37:16:6 | 0.030 4 | C | 0.250 | 0.156 | 3.633 | 0.056 6 |
| PI3K_2205_GC/G | 内含子 | pos.2205 | GC/GC: GC/C: GG | 38:36:5 | 23:43:1 | 0.009 2** | GC | 0.816 | 0.696 | 5.636 | 0.017 6 |
| PI3K_2298_G/GT | 内含子 | pos.2298 | G/G: G/GT: GT/GT | 77:8:0 | 61:10:2 | 0.048 2 | G | 0.096 | 0.042 | 3.680 | 0.055 1 |
| PI3K_2731_C/T | 内含子 | pos.2731 | CC: CT: TT | 75:6:0 | 61:10:2 | 0.027 0 | C | 0.096 | 0.036 | 4.743 | 0.029 4 |
| PI3K_2778_C/T | 内含子 | pos.2778 | CC: CT: TT | 77:7:0 | 60:11:2 | 0.024 3 | C | 0.108 | 0.042 | 5.147 | 0.023 3 |
| PI3K_3149_C/G | 内含子 | pos.3149 | CG: CC: GG | 50:20:9 | 37:24:5 | 0.044 4 | C | 0.649 | 0.570 | 1.926 | 0.165 2 |
| PI3K_3568_A/T | 内含子 | pos.3568 | AA: AT: TT | 51:29:3 | 36:24:9 | 0.024 0 | A | 0.304 | 0.211 | 3.483 | 0.062 0 |
| PI3K_3669_C/T | 内含子 | pos.3669 | CC: CT: TT | 36:21:4 | 38:15:1 | 0.045 8 | C | 0.865 | 0.762 | 3.873 | 0.049 1 |
| PI3K_3958_T/C | 内含子 | pos.3958 | TT: TC: CC | 43:34:4 | 28:24:13 | 0.013 5 | T | 0.385 | 0.259 | 5.252 | 0.021 9 |
| PI3K_3977_T/C | 内含子 | pos.3977 | TT: TC: CC | 77:7:0 | 60:7:5 | 0.012 1 | T | 0.123 | 0.042 | 7.102 | 0.007 7** |
| PI3K_4828_T/C | 内含子 | pos.4828 | TT: TC: CC | 16:28:22 | 26:13:19 | 0.044 1 | T | 0.568 | 0.455 | 3.197 | 0.073 8 |
| PI3K_5031_A/T | 内含子 | pos.5031 | AT: TT: AA | 43:19:15 | 23:14:26 | 0.014 8 | T | 0.602 | 0.474 | 4.567 | 0.032 6 |
| PI3K_5366_T/C | 外显子 | pos.5366 | CT: CC: TT | 40:20:15 | 18:16:30 | 0.006 6** | C | 0.609 | 0.467 | 5.650 | 0.017 5 |
| PI3K_5989_A/C | 内含子 | pos.5989 | AA: AC: CC | 75:9:0 | 59:9:4 | 0.033 3 | A | 0.123 | 0.054 | 4.831 | 0.028 0 |
| PI3K_6016_ACT/A | 内含子 | pos.6016 | AA: A/ACT: ACT/ACT | 41:31:5 | 46:18:1 | 0.013 8 | A | 0.848 | 0.734 | 5.575 | 0.018 2 |
| 注: **表示呈极显著(P < 0.01)相关 | |||||||||||
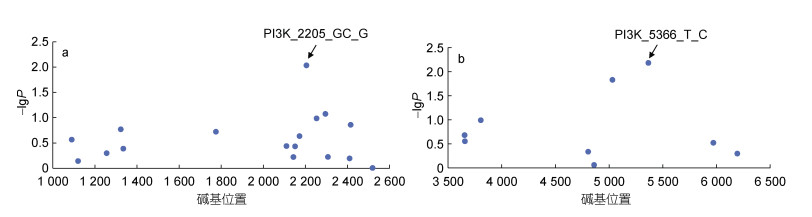 |
| 图 5 LvPI3K基因上与抗弧菌性状呈极显著相关(P < 0.01)的标记及周边标记的P值分布图 Fig. 5 The P values of very significant markers related to the resistance of shrimp against V. parahaemolyticus (P < 0.01) and surrounding markers in LvPI3K 注: a. PI3K_2205; b. PI3K_5366 |
为进一步了解抗性标记之间的关系, 对鉴定到的SNP标记进行了单倍型分析, 通常R2 > 0.8的连锁才被认为是可信的单倍域(De Bakker et al, 2005), 以此为标准, 在LvPI3K基因中鉴定出了5个具有高LD (D' > 0.9, R2 > 0.8)的区域(图 6)。之后对这5个紧密连锁的单倍域与抗病性状进行关联分析, 发现Block3、Block4与Block5的部分基因型在抗弧菌/敏感群体存在显著差异(P < 0.05) (表 3)。尤其是由PI3K_5031_A/T和PI3K_5366_T/C两个位点构成的Block5, 基因型AT/TC在群体中的累计频率达0.977, 可以作为凡纳滨对虾弧菌抗病性状分子育种中一个潜在可用的单倍型。
 |
| 图 6 LvPI3K基因上标记间连锁不平衡分析图 Fig. 6 Linkage disequilibrium of markers in LvPI3K gene 注: 黑色(R2 = 1), 白色(R2 = 0), 灰色(0 < R2 < 1) |
| 单倍域名称及标记组成 | 单倍型 | 频率 | 在抗性/敏感个体中出现频率 | 卡方(χ2) | P值 |
| Block 1 (PI3K_1862_T/C, PI3K_1929_T/A, PI3K_1932_A/T, PI3K_2110_TC/T) | TTA(T) | 0.655 | 0.686, 0.629 | 1.095 | 0.295 4 |
| CAT(TC) | 0.238 | 0.190, 0.280 | 3.392 | 0.065 5 | |
| CTA(TC) | 0.056 | 0.071, 0.043 | 1.112 | 0.291 6 | |
| CTA(T) | 0.033 | 0.033, 0.034 | 0.003 | 0.960 0 | |
| TTA(TC) | 0.013 | 0.012, 0.013 | 0.004 | 0.946 6 | |
| Block 2 (PI3K_2112_T/G, PI3K_2134_C/T) | TC | 0.798 | 0.758, 0.831 | 2.435 | 0.118 7 |
| GT | 0.202 | 0.242, 0.169 | 2.435 | 0.118 7 | |
| Block 3 (PI3K_2295_A/AG, PI3K_2298_G/GT) | (AG)G | 0.924 | 0.892, 0.952 | 4.119 | 0.042 4* |
| A(GT) | 0.072 | 0.107, 0.042 | 5.066 | 0.024 4* | |
| Block 4 (PI3K_2524_GTTGT/T, PI3K_2731_C/T, PI3K_2778_C/T) | (GTTGT)CC | 0.927 | 0.892, 0.958 | 5.147 | 0.023 3* |
| (T)TT | 0.06 | 0.087, 0.036 | 3.639 | 0.056 4 | |
| Block 5 (PI3K_5031_A/T, PI3K_5366_T/C) | AT | 0.528 | 0.596, 0.467 | 4.977 | 0.025 7* |
| TC | 0.449 | 0.372, 0.518 | 6.482 | 0.010 9* | |
| AC | 0.019 | 0.024, 0.014 | 0.415 | 0.519 4 | |
| 表示存在显著(P < 0.05)相关 | |||||
在320个验证样本中, 分别对两个最显著的位点PI3K_2205和PI3K_5366进行了验证, 其中位点PI3K_2205为插入缺失突变, 位点PI3K_5366位于第6个外显子上, 为同义突变。Sanger测序分型结果显示PI3K_2205在验证群体中存在插入缺失, 但是关联分析显示其与抗弧菌性状不相关。PI3K_5366标记的基因型与三代测序基因型分布一致(表 4), 其中, PI3K_5366标记的TT基因型在敏感群体中出现的频率显著高于抗性群体, CC基因型则反之; 进一步对T、C的基因频率进行统计的结果显示, 在验证材料中, T、C等位基因在抗性群体与敏感群体中出现的频率存在极显著差异(P < 0.01), 等位基因型C为抗性优势基因, T则为敏感等位基因型。
| SNP | 分析群体 | 突变类型 | 基因型 | 个体数 | P值 | |
| R | S | |||||
| PI3K_2205_GC/G | 验证群体 | indel | G/GC: GC/GC: GG | 22:76:2 | 20:68:1 | 0.851 8 |
| 三代测序分型群体 | indel | G/GC: GC/GC: GG | 38:36:5 | 23:43:1 | 0.009 2** | |
| PI3K_5366_T/C | 验证群体 | sSNP | TT: TC: CC | 8:96:23 | 32:58:0 | 0.003 0** |
| 三代测序分型群体 | sSNP | TT: TC: CC | 15:40:20 | 30:16:18 | 0.006 6** | |
| 注: **表示存在极显著(P < 0.01)相关 | ||||||
随着PacBio三代测序技术的发展, 测序准确率日渐提高, 三代测序在基因组学和基因分型中的应用也越来越广泛。本研究针对水产动物性状相关标记高通量筛选的需求, 建立了基于PacBio SMRT靶向测序的候选基因关联分析方法, 并在凡纳滨对虾抗弧菌性状相关标记的筛选中进行了应用, 证实了该方法是一种高通量、准确可靠的方法, 本研究为水产动物的性状相关基因精细定位和分子育种研究提供了一种高效工具。
随着高通量SNP分型技术的发展, 利用全基因组关联分析(GWAS)定位性状关键基因成为了重要的技术手段, 在通过GWAS获得性状相关QTL后, 需要进一步对QTL进行精细分析, 以鉴定与性状紧密连锁的分子标记或者因果突变, 因此针对QTL区域开展候选基因关联分析是精细定位的关键步骤。然而目前水产动物中对性状进行精细定位的研究较少, 一方面水产动物GWAS的研究起步较晚, 目前较多处于QTL或者标记定位阶段; 另一方面缺乏针对靶向区域进行高效关联分析的方法。在凡纳滨对虾分子育种研究方面, 目前已鉴定出一些与生长(Andriantahina et al, 2012)、耐氨氮(Lu et al, 2018)和WSSV抗性(Liu et al, 2014)等性状相关的QTL或者SNP标记等, 并通过精细定位鉴定了PKC、MMD2、SRC等生长候选基因(Wang et al, 2019), 在这些研究中, 针对候选基因的关联分析采用的是PCR扩增后一代测序或二代测序的方法。该方法需要针对目标基因的外显子区域分段设计引物, 分别扩增后测序, 工作量大、费用高, 对于序列比较长的基因分析难度大。凡纳滨对虾基因组十分复杂, 是公认的复杂基因组之一, 其重复序列约占基因组的23.93%, SSR的平均长度为72.21 bp, 是其他节肢动物(20.11—31.91 bp)的两倍以上(Zhang et al, 2019b)。重复序列在全基因组上出现频率高以及长度长的特点, 使得利用一代和二代测序技术在进行对虾基因分型面临较大困难(Sulovari et al, 2019)。虽然对重复序列区域测序的难度较大, 但是这些区域通常显示出较高的突变率, 这些突变通常与疾病和进化相关(Huddleston et al, 2017)。一代测序通常难以跨越连续的SSR序列, 二代测序由于其读长限制, 需要对测序结果进行拼接, 而高重复序列又会影响拼接的准确性。相较于一代和二代测序技术、三代测序兼具通量高、测序长度长的特点, 通过长片段靶向捕获结合三代测序成为开展凡纳滨对虾候选基因关联分析的最佳选择(Yue et al, 2017)。本研究建立了一套基于PacBio SMRT测序技术进行候选基因关联分析的实验流程和数据分析流程, 对长片段靶向序列进行全长测序的同时, 也通过增加测序深度, 实现了对候选基因的精准分型。三代测序数据的准确性很大程度上受SMRT建库质量的影响, 且扩增子混合建库本身也存在混合不均匀、扩增子本身质量不佳等问题(DePristo et al, 2011)。采用三代测序数据适配的分析软件和流程, 对于得到更精准的基因分型结果至关重要。本研究采用IGV等可视化工具, 对测序数据的连续性进行了评估, 发现测序结果连续性较好, 在7 378 bp的全长中无测序导致的断点, 测序reads连续性将大大减少后续单倍型分析的难度和工作量。
根据所建立的候选基因关联分析方法, 结合团队前期发掘的与LvPI3K紧密连锁的抗弧菌性状相关分子标记(Unigene19157_All__1806_348_223) (Zhang et al, 2019a), 本研究将LvPI3K基因作为凡纳滨对虾对抗弧菌性状相关候选基因, 进一步鉴定与抗弧菌性状相关的突变位点。在候选基因LvPI3K共计7 378 bp的序列内发现91个突变位点, 平均每81 bp出现一个SNP, 其中有95.6%位于内含子或者5'-UTR区域, 有4个突变发生在外显子区域, 由此推测, 非编码区中的突变发生频率要高于编码区中的发生率。内含子区域的多态性对于性状相关性的解释较为困难, 目前主要被当作与抗病功能基因紧密连锁的分子标记来应用。本研究中与抗弧菌性状呈极显著相关的PI3K_5366_T/C位点发生在PI3K的第六个外显子区域, 其T > C突变会导致mRNA的ACU突变为ACC, 然而由于密码子的简并性, 氨基酸序列并没有变化。但是同义突变依旧具有巨大的研究价值, 其可以从核酸和蛋白两个方面影响基因的表达。在核酸层面, 同义突变通过调控剪接位点序列进而产生多种剪接形式, 或者通过影响DNA序列内的GC含量, 从而影响序列稳定性等。在蛋白层面, 虽然同义突变并不改变其编码的氨基酸序列, 但不同物种对于不同密码子的偏好性不同, 从而影响mRNA的翻译效率。同义突变还会对蛋白质的折叠产生影响, 从而改变蛋白自身的理化性质。因此, 同义突变在分子功能层面并不是沉默突变, 可以通过参与基因表达调控的各个阶段对基因功能产生影响(Plotkin et al, 2011)。
4 结论本研究开发了一种适用于凡纳滨对虾等水产生物的基于三代测序的高通量靶向测序基因分型技术, 并发掘了LvPI3K基因上91个多态性位点, 其中有21个位点与对虾的抗弧菌性状呈显著相关(P < 0.05), 有2个位点(PI3K_2205_GC/G, PI3K_5366_T/C)与抗弧菌性状呈极显著相关(P < 0.01), 这些位点的开发对于对虾抗弧菌性状的分子育种有重要指导意义。
Andriantahina F, Liu X L, Huang H et al, 2012. Erratum to: Response to selection, heritability and genetic correlations between body weight and body size in Pacific white shrimp, Litopenaeus vannamei. Chinese Journal of Oceanology and Limnology, 30(3): 506 DOI:10.1007/s00343-012-1004-3 |
Barrett J C, Fry B, Maller J et al, 2005. Haploview: analysis and visualization of LD and haplotype maps. Bioinformatics, 21(2): 263-265 DOI:10.1093/bioinformatics/bth457 |
Cock J, Gitterle T, Salazar M et al, 2009. Breeding for disease resistance of Penaeid shrimps. Aquaculture, 286(1): 1-11 |
De Bakker P I W, Yelensky R, Pe'er I et al, 2005. Efficiency and power in genetic association studies. Nature Genetics, 37(11): 1217-1223 DOI:10.1038/ng1669 |
DePristo M A, Banks E, Poplin R et al, 2011. A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nature Genetics, 43(5): 491-498 DOI:10.1038/ng.806 |
Edge P, Bansal V, 2019. Longshot enables accurate variant calling in diploid genomes from single-molecule long read sequencing. Nature Communications, 10(1): 4660 DOI:10.1038/s41467-019-12493-y |
Eid J, Fehr A, Gray J et al, 2009. Real-Time DNA sequencing from single polymerase molecules. Science, 323(5910): 133-138 DOI:10.1126/science.1162986 |
Gjedrem T, 2015. Disease resistant fish and shellfish are within reach: a review. Journal of Marine Science and Engineering, 3(1): 146-153 DOI:10.3390/jmse3010146 |
Han J E, Tang K F J, Tran L H et al, 2015. Photorhabdus insect-related (Pir) toxin-like genes in a plasmid of Vibrio parahaemolyticus, the causative agent of acute hepatopancreatic necrosis disease (AHPND) of shrimp. Diseases of Aquatic Organisms, 113: 33-40 DOI:10.3354/dao02830 |
Houston R D, Bean T P, Macqueen D J et al, 2020. Harnessing genomics to fast-track genetic improvement in aquaculture. Nature Reviews Genetics, 21(7): 389-409 DOI:10.1038/s41576-020-0227-y |
Huddleston J, Chaisson M J P, Steinberg K M et al, 2017. Discovery and genotyping of structural variation from long-read haploid genome sequence data. Genome Research, 27: 677-685 DOI:10.1101/gr.214007.116 |
Lee C T, Chen I T, Yang Y T et al, 2015. The opportunistic marine pathogen Vibrio parahaemolyticus becomes virulent by acquiring a plasmid that expresses a deadly toxin. Proceedings of the National Academy of Sciences of the United States of America, 112(34): 10798-10803 DOI:10.1073/pnas.1503129112 |
Lien S, Koop B F, Sandve S R et al, 2016. The Atlantic salmon genome provides insights into rediploidization. Nature, 533(7602): 200-205 DOI:10.1038/nature17164 |
Liu J W, Yu Y, Li F H et al, 2014. A new anti-lipopolysaccharide factor (ALF) gene with its SNP polymorphisms related to WSSV-resistance of Litopenaeus vannamei. Fish & Shellfish Immunology, 39(1): 24-33 |
Lu X, Kong J, Meng X H et al, 2018. Identification of SNP markers associated with tolerance to ammonia toxicity by selective genotyping from de novo assembled transcriptome in Litopenaeus vannamei. Fish & Shellfish Immunology, 73: 158-166 |
Martin M, Patterson M, Garg S et al, 2016. WhatsHap: fast and accurate read-based phasing. BioRxiv, 18 |
Meuwissen T H E, Goddard M E, 2004. Mapping multiple QTL using linkage disequilibrium and linkage analysis information and multitrait data. Genetics Selection Evolution, 36(3): 261-279 DOI:10.1186/1297-9686-36-3-261 |
Plotkin J B, Kudla G, 2011. Synonymous but not the same: the causes and consequences of codon bias. Nature Reviews Genetics, 12(1): 32-42 DOI:10.1038/nrg2899 |
Poplin R, Chang P C, Alexander D et al, 2018. A universal SNP and small-indel variant caller using deep neural networks. Nature Biotechnology, 36(10): 983-987 DOI:10.1038/nbt.4235 |
Rhoads A, Au K F, 2015. PacBio sequencing and its applications. Genomics, Proteomics & Bioinformatics, 13(5): 278-289 |
Sulovari A, Li R Y, Audano P A et al, 2019. Human-specific tandem repeat expansion and differential gene expression during primate evolution. Proceedings of the National Academy of Sciences of the United States of America, 116(46): 23243-23253 DOI:10.1073/pnas.1912175116 |
VanRaden P M, Van Tassell C P, Wiggans G R et al, 2009. Invited review: reliability of genomic predictions for North American Holstein bulls. Journal of Dairy Science, 92(1): 16-24 DOI:10.3168/jds.2008-1514 |
Wang Q C, Yu Y, Zhang Q et al, 2019. A novel candidate gene associated with body weight in the pacific white shrimp Litopenaeus vannamei. Frontiers in Genetics, 10: 520 DOI:10.3389/fgene.2019.00520 |
Wenger A M, Peluso P, Rowell W J et al, 2019. Accurate circular consensus long-read sequencing improves variant detection and assembly of a human genome. Nature Biotechnology, 37: 1155-1162 DOI:10.1038/s41587-019-0217-9 |
Xu Z L, Taylor J A, 2009. SNPinfo: integrating GWAS and candidate gene information into functional SNP selection for genetic association studies. Nucleic Acids Research, 37(2): 600-605 |
Yu Y, Luo Z, Wang Q C et al, 2020. Development of high throughput SNP genotyping approach using target sequencing in Pacific white shrimp and its application for genetic study. Aquaculture, 528: 735549 DOI:10.1016/j.aquaculture.2020.735549 |
Yu Y, Zhang X J, Yuan J B et al, 2015. Genome survey and high-density genetic map construction provide genomic and genetic resources for the Pacific White Shrimp Litopenaeus vannamei. Scientific Reports, 5(1): 15612 DOI:10.1038/srep15612 |
Yue G H, 2014. Recent advances of genome mapping and marker-assisted selection in aquaculture. Fish and Fisheries, 15(3): 376-396 DOI:10.1111/faf.12020 |
Yue G H, Wang L, 2017. Current status of genome sequencing and its applications in aquaculture. Aquaculture, 468: 337-347 DOI:10.1016/j.aquaculture.2016.10.036 |
Zhang Q, Yu Y, Wang Q C et al, 2019a. Identification of single nucleotide polymorphisms related to the resistance against acute hepatopancreatic necrosis disease in the pacific white shrimp Litopenaeus vannamei by target sequencing approach. Frontiers in Genetics, 10: 700 DOI:10.3389/fgene.2019.00700 |
Zhang X J, Yuan J B, Sun Y M et al, 2019b. Penaeid shrimp genome provides insights into benthic adaptation and frequent molting. Nature Communications, 10(1): 356 DOI:10.1038/s41467-018-08197-4 |
 2021, Vol. 52
2021, Vol. 52


