
中国海洋湖沼学会主办。
文章信息
- 董正, 曾启繁, 刘亮洁, 王师. 2021.
- DONG Zheng, ZENG Qi-Fan, LIU Liang-Jie, WANG Shi. 2021.
- 栉孔扇贝Smad基因家族的表达特征和对鳗弧菌侵染的免疫应答分析
- EXPRESSION CHARACTERISTICS OF Smad GENE FAMILY AND ITS IMMUNE RESPONSE TO VIBRIO ANGUILLARUM INFECTION IN CHLAMYS FARRERI
- 海洋与湖沼, 52(5): 1273-1283
- Oceanologia et Limnologia Sinica, 52(5): 1273-1283.
- http://dx.doi.org/10.11693/hyhz20210200029
文章历史
-
收稿日期:2021-02-01
收修改稿日期:2021-04-10
2. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室 青岛 266237
2. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China
在脊椎动物中, Smad基因通过调节T细胞、中性粒细胞等免疫细胞功能, 在机体的免疫调节中发挥着重要作用(Massagué et al, 2000; Weinstein et al, 2000; Derynck et al, 2003)。
Smad基因家族从结构和功能上主要可以分为3个亚族: 受体调节Smad(R-Smad)、通用性Smad (Co-Smad)和抑制型Smad (I-Smad), 其中R-smad可分为TGF-β通路Smad和BMP通路Smad。Smad蛋白含有高度保守的N端结构域mad homologous domain1 (MH1)和C端结构域mad homologous domain2 (MH2) (Grishin, 2001; Wu et al, 2001)。近年来研究发现, 在脊椎动物中Smad3基因通过调控增强子结合蛋白(CEBP/ε)来促进中性粒细胞的成熟和功能极化, 发挥免疫调节作用(吴婷婷等, 2014)。中性粒细胞是动物体内天然免疫系统的重要组成部分, 作为效应细胞抗击病原菌入侵, 在介导先天免疫防御和获得性免疫反应方面都有非常重要的作用(Datto et al, 1999)。贝类的免疫机制包括细胞免疫和体液免疫, 主要靠血淋巴细胞来完成。与脊椎动物相比, 缺乏获得性免疫功能(刘世良等, 2003)。近些年来研究暗示, Smad基因家族也参与了软体动物的免疫应答, 但其在贝类免疫反应中的作用机制仍不清晰(Hu et al, 2017)。
栉孔扇贝是我国山东一带重要的经济贝类, 也是我国重要的海水养殖品种(李成林等, 2011)。自20世纪90年代以来, 高密度的人工养殖加上海洋水质资源的恶化, 经常引发大规模的细菌性疾病。其中, 鳗弧菌(Vibrio anguillarum)病是主要的病原之一, 广泛存在于海洋动物体内及沿岸海水沉积物中(窦海鸽等, 2005)。目前, 在双壳贝类中关于Smad基因家族的研究大多局限于鉴定和克隆(郭慧慧, 2012; Liu et al, 2014; 钱雪骏等, 2015), 对Smad基因家族的系统性分析以及免疫调控功能的相关理解仍然有限。因此, 我们对栉孔扇贝进行了Smad基因家族的系统发生和表达模式分析, 以深入理解栉孔扇贝中Smad基因的共表达网络和调控机制。此外, 我们也开展了鳗弧菌侵染实验, 以分析Smad基因的免疫应答反应, 为研究双壳贝类的免疫应答机制提供参考。
1 材料与方法 1.1 Smad基因的全基因组筛查与鉴定为了筛查和鉴定Smad基因, 本研究使用脊椎动物和无脊椎动物Smad同源蛋白质序列对栉孔扇贝的全基因组数据库(Li et al, 2017)进行检索, 使用的物种包括人(Homo sapiens)、小鼠(Mus musculus)、斑点雀鳝(Lepisosteus oculatus)、果蝇(Drosophilidae melanogaster)、海葵(Nematostella vectensis)、紫海胆(Strongylocentrotus purpuratus)、长牡蛎(Crassostrea gigas)、福寿螺(Pomacea canaliculata)和虾夷扇贝(Patinopecten yessoensis) (表 1)。利用TBLASTN将这些物种的Smad蛋白序列与栉孔扇贝的转录组和基因组数据进行比对(e值小于1e-5), 得到栉孔扇贝候选Smad基因(e值小于1e-5)。通过查询基因组的结构注释文件, 获得上述所选物种的Smad基因在染色体或scaffold上的位置信息。使用SMART (Letunic et al, 2018)识别Smad蛋白的保守结构域, 并进一步使用Compute pI/Mw工具(Bjellqvist et al, 1993)计算Smad蛋白的等电点(pI)和分子量。选用MEME (Bailey et al, 1994)的默认参数对Smad蛋白的基序进行识别和分析。使用IBS1.0.3 (Liu et al, 2015)展示Smad蛋白结构域和基序的分布。
| 物种 | 基因 | 检索号 | 物种 | 基因 | 检索号 | |
| 人 H. sapiens |
Smad1 | AAC50790.1 | 斑点雀鳝 L. oculatus |
Smad1 | XP_006629559.1 | |
| Smad2 | AAC39657.1 | Smad2 | XP_006627302.1 | |||
| Smad3 | AAL68976.1 | Smad3 | XP_006628801.1 | |||
| Smad4 | Q13485.1 | Smad4 | XP_015224305.1 | |||
| Smad5 | AAB92396.1 | Smad5 | XP_006631964.1 | |||
| Smad6 | AAC82331.1 | Smad6 | XP_006628799.1 | |||
| Smad7 | AAL68977.1 | Smad7 | XP_015224305.1 | |||
| Smad9 | AAI04763.1 | Smad9 | XP_006628242.1 | |||
| 小鼠 M. musculus |
Smad1 | AAG41407.1 | 果蝇 D. melanogaster |
Mad | NP_477017.1 | |
| Smad2 | AAH89184.1 | Smox | NP_477017.1 | |||
| Smad3 | AAB81755.1 | Med | NP_524610.1 | |||
| Smad4 | AAM74472.1 | Dad | AAN13728.1 | |||
| Smad5 | AAC83580.1 | 长牡蛎 C. gigas |
Smad1/5/8 | AHB37076.1 | ||
| Smad6 | AAB81351.1 | Smad3 | XP_011441244.1 | |||
| Smad7 | AAB81353.1 | Smad4 | AHB37077.1 | |||
| Smad9 | AAI04763.1 | Smad6 | NP_001295807.1 | |||
| 海葵 N. vectensis |
Smad1/5 | ABC88374.1 | 虾夷扇贝 P. yessoensis |
Smad3 | OWF37852.1 | |
| Smad3 | XP_001631657.1 | Smad4 | OWF37845.1 | |||
| Smad4 | EDO31382.1 | Smad5 | OWF47058.1 | |||
| Smad6 | EDO39628.1 | Smad6 | OWF38654.1 | |||
| 福寿螺 P. canaliculata |
Smad3 | XP_025084166.1 | 紫海胆 S. purpuratus |
Smad3 | BAF45857.1 | |
| Smad4 | XP_025081820.1 | Smad4 | XP_030853090.1 | |||
| Smad5 | XP_025086090.1 | Smad5 | XP_030836187.1 | |||
| Smad6 | XP_025080867.1 | Smad6 | XP_798238.2 |
将人(H. sapiens)、小鼠(M. musculus)、斑点雀鳝(L. oculatus)、果蝇(D. melanogaster)、海葵(N. vectensis)、紫海胆(S. purpuratus)、长牡蛎(C. gigas)、福寿螺(P. canaliculata)、虾夷扇贝(P. yessoensis)以及栉孔扇贝的Smad蛋白序列进行序列比对和系统发生分析。使用ClustalX (Larkin et al, 2007)进行多重序列比对, 导入MEGA7 (Kumar et al, 2016), 运用最大似然法(Maximum Likelihood)构建进化树, Bootstrap 1000次检验。使用iTOL展示进化树(Letunic et al, 2007)。
1.3 Smad基因的表达量计算、共表达网络的构建和GO富集用STAR (Dobin et al, 2013)软件将栉孔扇贝的成体组织(横纹肌、血、肝胰腺、眼、性腺、足、鳃、肾、外套膜、平滑肌)转录组数据(Li et al, 2017)比对到栉孔扇贝参考基因组序列, 用Samtools (Li et al, 2009)软件处理比对结果件, 用featureCounts (Liao et al, 2014)软件统计基因的counts值, 最后基因的表达量用TPM (Transcripts Per Million)表示。
将23 506个在栉孔扇贝各组织中TPM > 1的基因, 导入“WGCNA”包(Langfelder et al, 2008)构建共表达网络(WGCNA)。根据之前构建贝类共表达网络的方法, 选择权重系数softpower=13, 网络类型参数networkType=“unsigned”, 最小模块大小minModuleSize=450, 设定切树高度detectCut et al, 2020)。使用“pheatmap”包绘制表达热图, 对均一化的表达量进行展示。
利用EnrichPipeline软件(Huang et al, 2009)对Smad基因所在模块的基因进行功能富集分析, FDR校正后的P值(adjusted P-value) < 0.05为阈值, 筛选富集的GO term。
1.4 鳗弧菌侵染实验鳗弧菌(Vibrio anguillarum)的培养和侵染方法参考Cong等(2008)所述。在28 ℃下利用2216E液体培养基(5 g/L胰蛋白胨, 1 g/L酵母提取物和0.1 g C6H5Fe·5H2O, pH=7.6)培养鳗弧菌, 通过2000 g离心5 min进行收集, 将沉淀物悬浮于过滤海水中(0.22 µm滤膜), 将鳗弧菌浓度调节至1×107 CFU/mL用于进行侵染实验。选取健康1龄栉孔扇贝, 在实验室暂养3 d后, 用于革兰氏阴性细菌鳗弧菌侵染实验。随后挑选50只, 随机分成对照组与实验组, 在利用鳗弧菌侵染后0、5、24、48、72 h进行取样, 每个时间点取5个个体做为生物学重复。使用注射器从横纹肌中收集血淋巴。收集完血淋巴样品后, 立即将其在800 g, 4 ℃条件下离心10 min以收集血细胞。然后将血细胞立即在液氮中冷冻, 在使用前用–80 ℃冰箱冷藏。
1.5 RNA提取和实时定量PCR分析按照本课题组前期发表的方法提取总RNA (Hu et al, 2006), 使用Nanovue Plus分光光度计(GE Healthcare, NJ, USA)测定RNA浓度和纯度, 并通过琼脂糖凝胶电泳评估RNA完整性。使用莫洛尼鼠白血病病毒(MMLV)逆转录酶(Thermo, USA)合成第一链cDNA用作实时定量PCR的模板。Smad基因的实时定量PCR扩增按照说明书体系使用SsoFast™EvaGreen®试剂进行, 引物序列见表 2, 在LightCycler480进行实时定量PCR分析(Roche Diagnostics公司, 曼海姆, 德国)。PCR运行程序如下: 50 ℃ 2 min, 94 ℃ 10 min, 40个循环, 94 ℃ 15 s, 62 ℃ 1 min, 选取EF1A基因作为鳗弧菌侵染实验中实时定量PCR分析的内参基因(Li et al, 2016)。所有引物设计均使用Primer Premier5.0软件, 所设计引物首先通过BLASTN (1e-10)比对栉孔扇贝基因组来评估特异性。采用2–ΔΔCt方法基于对照组计算鳗弧菌侵染后各Smad成员的相对表达量。使用SPSS20.0软件(IBM, 2013)中独立t检验的统计软件包, 分析各Smad各成员的表达变化。
| 基因名称 | 引物序列 |
| Smad3-F | 5′-CTCAAGAGGGATGAAGTGTGTGTA-3′ |
| Smad3-R | 5′-CTGTGGAAATTTGTCTGGAACC-3′ |
| Smad4-F | 5′-CACCAAGTCTGGCTATTACAGGAAG-3′ |
| Smad4-R | 5'-GACATGATCAAGCCCCTAGGT-3' |
| Smad5-F | 5′-CTAGAACTGGAGGAAGCACTGG-3′ |
| Smad5-R | 5′-CCACGAACTGAAACCTCTTGAAT-3′ |
| Smad6-F | 5′-CGCTGTCGTAACTCCCAATAG-3′ |
| Smad6-R | 5′-GAAACGGGAAACACACCTACC-3′ |
| EF1A-F | 5′-AGCAATGTGAGCGGTATGGC-3′ |
| EF1A-R | 5′-TGATCCCCCTGCTGGAGTAG-3′ |
我们在栉孔扇贝总共鉴定出Smad3、Smad4、Smad5和Smad6四个Smad基因(表 3)。四个基因全长差异较大, 其中Smad3基因最长, 达50 088 bp, Smad5基因最短, 为13 759 bp。在外显子数量方面, Smad3最多, 有13个外显子, Smad6数量最小, 有5个外显子。这些Smad的预测分子量范围从41.28到62.46 kDa, 预测的等电点(pI)范围从6.34到8.37 (表 3)。通过Smad蛋白序列在SMRT上进行结构域预测(图 1), 4个Smad基因都具有N端结构域MH1和C端结构域MH2。同一亚族的成员含有大致相同的基序组成和排列顺序, 例如R-Smad成员均具有Motif1-13。不同亚族中的蛋白序列Motif组成和分布存在较大差异, 仅Motif1、Motif2、Motif4和Motif8是三个亚家族共有的。
| 基因名称 | 基因长度(bp) | 开放阅读框长度(bp) | 外显子数量 | 蛋白长度(aa) | 等电点pI | 蛋白分子量(kDa) |
| Smad3 | 50 088 | 1 286 | 10 | 431 | 6.39 | 48.57 |
| Smad4 | 23 264 | 1 718 | 13 | 575 | 7.34 | 62.46 |
| Smad5 | 13 759 | 1 376 | 6 | 461 | 8.15 | 51.44 |
| Smad6 | 39 942 | 1 079 | 5 | 362 | 8.37 | 41.28 |
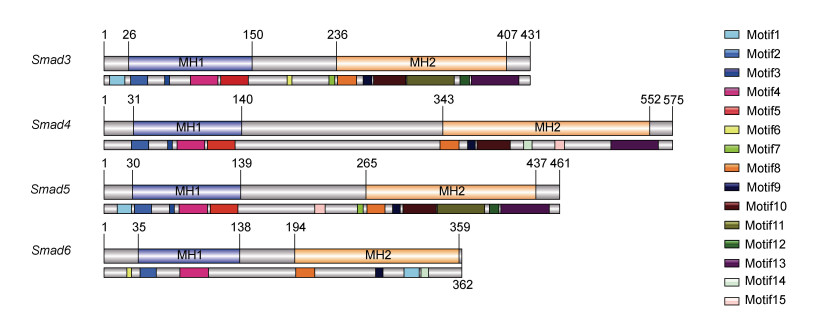 |
| 图 1 栉孔扇贝Smad蛋白结构域和基序的分布 Fig. 1 Domains and motifs of Smad proteins 注: 蓝色框为MH1结构域, 黄色框为MH2结构域, 上面为蛋白结构域分布图, 下面为基序分布图 |
系统发生分析表明, Smad基因主要聚为4个支, 即TGF-β通路R-Smad、BMP通路R-Smad、Co-Smad和I-Smad (图 2)。其中TGF-β通路R-Smad和BMP通路R-Smad同属R-Smad亚家族。栉孔扇贝中的Smad3、Smad4、Smad5、Smad6分别聚到TGF-β通路R-Smad、Co-Smad、BMP通路R-Smad和I-Smad四个亚族中。在本文所分析的所有无脊椎动物中, Smad数量都为4个。软体动物Smad基因家族成员基本类似。其中栉孔扇贝、虾夷扇贝和牡蛎享有完全相同的Smad基因家族成员, 这与它们高度相近的亲缘关系情况相一致。栉孔扇贝的Smad3与其他物种的Smad2和Smad3聚在一起; Smad5与其他物种的Smad1、Smad5、Smad9聚类, 两者所在的亚族分别调控不同的信号通路, TGF-β通路R-Smad和BMP通路R-Smad的同源性高于其与Co-Smad和I-Smad的同源性。栉孔扇贝的Smad4与其他物种的Smad4聚类, Co-Smad亚族中只有一种Smad4, 负责与R-Smad蛋白形成异源复合物。栉孔扇贝Smad6与其他物种的Smad6、Smad7聚类。脊椎动物含有完备的Smad基因家族成员, 数量都为8个。其中, Smad1、Smad2、Smad7和Smad9为脊椎动物特异性Smad基因。Smad1、Smad5和Smad9聚为一支; Smad2和Smad3聚为一支; Smad6和Smad7具有更近的共同祖先, 暗示这些Smad基因可能由基因或染色体加倍产生。这一发现也得到了共线性分析的支持(图 3)。软体动物的Smad3、Smad4和Smad6分布在同一条染色体上。而在本文所分析的脊椎动物中Smad3和Smad6在同一条染色体上, Smad2、Smad4和Smad7在同一条染色体。暗示Smad基因家族在脊椎动物的演化中发生了加倍。
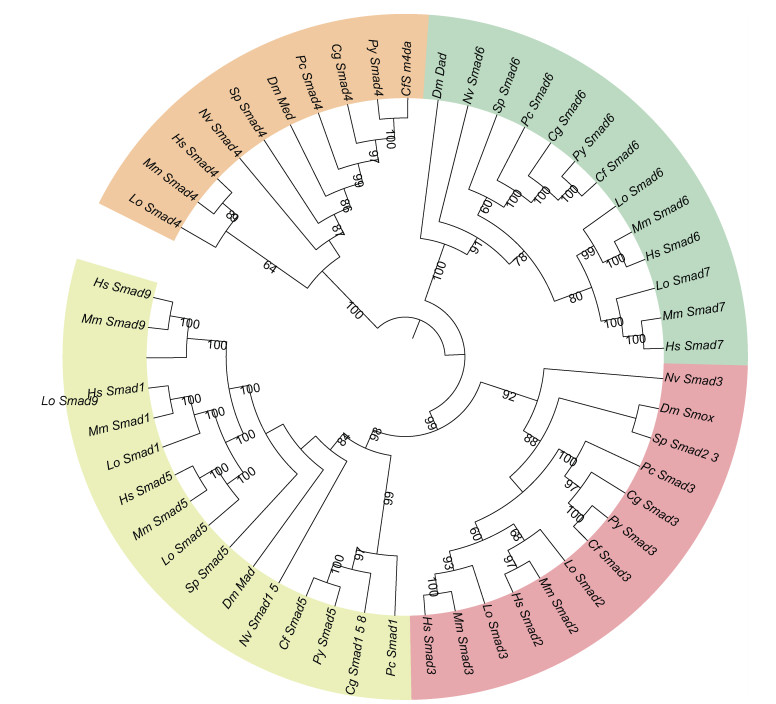 |
| 图 2 Smad家族系统进化分析 Fig. 2 Phylogenetic tree of Smad genes 注: Hs. 人; Mm. 小鼠; Lo. 斑点雀鳝; Dm. 果蝇; SP. 紫海胆; Cg. 牡蛎; Py. 虾夷扇贝; Nv. 海葵; Ps. 鹦鹉螺; Cf. 栉孔扇贝。绿色: I-Smad亚群; 粉色: 介导TGF-β通路的R-Smad亚群; 黄色: 介导BMP通路的R-Smad亚群; 橘黄色: Co-Smad亚群 |
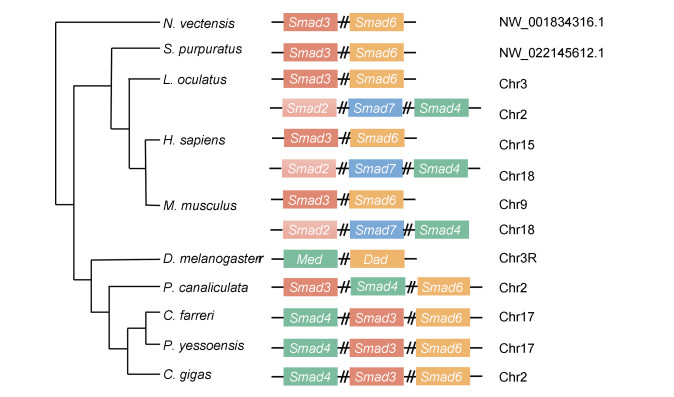 |
| 图 3 Smad基因在染色体或scaffold上的分布 Fig. 3 Distribution of Smad genes on chromosomes and scaffolds |
共表达网络结果发现, 栉孔扇贝Smad3、Smad4、Smad5和Smad6分别富集在M14、M12、M9和M6模块中(图 4a)。M14、M12、M9和M6模块分别包含2 025、2 709、2 369和2 564个基因。Smad基因在10种成体组织中的表达具有明显特异性, Smad3在横纹肌和平滑肌中表达量最高, 暗示其参与了肌肉的形成和发育(图 4b)。Smad3基因所在的模块主要富集到细胞增殖和蛋白质复合物等GO功能(表 4)。Smad4在栉孔扇贝各个成体组织中均有表达, 其中性腺中表达量最高, 表明其可能在性腺的形成和发育中起重要作用(图 4c)。Smad4所在的基因模块主要富集到细胞器组织和信号受体活性等GO功能(表 5)。Smad5在血液中特异性高表达, 暗示其调控血细胞的生成(图 4d)。另外, 我们发现与Smad5共表达的基因中包含大量Toll样受体信号通路的关键基因(表 6)。Smad5所在的基因模块主要富集到蛋白质结合和蛋白质复合物等GO功能(表 7)。Smad6在鳃中表达量最高, 暗示其可能在鳃中起调控作用(图 4e)。Smad6所在的基因模块主要富集到对外界刺激的反应调节和蛋白质结合等GO功能(表 8)。
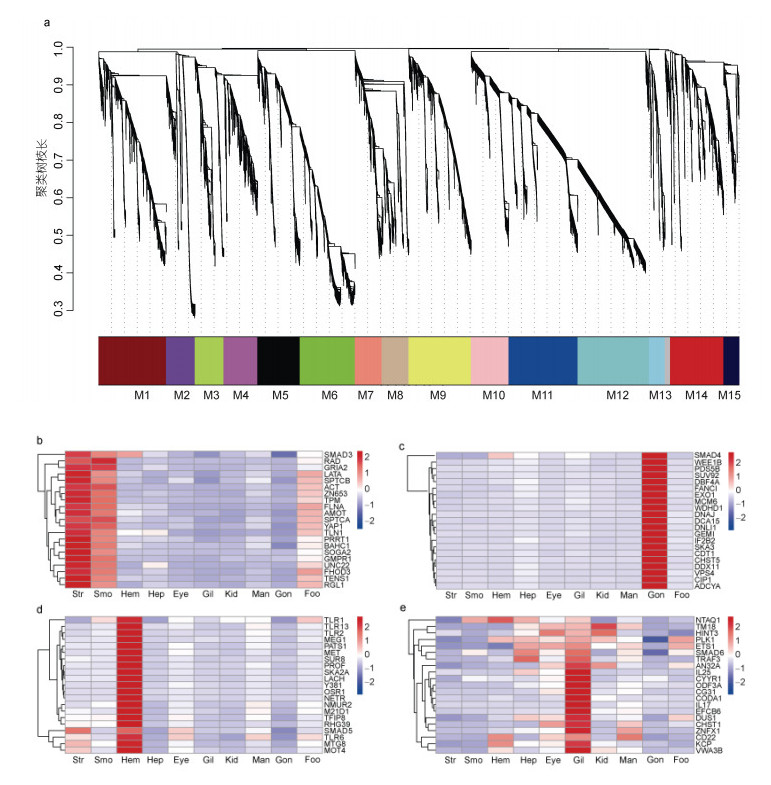 |
| 图 4 共表达网络和Smad及其共表达基因在栉孔扇贝成体组织中的表达量热图 Fig. 4 WGCNA and heatmaps of Smad and their co-expressed genes among adult tissues in C. farreri 注: a. 共表达网络; b. Smad3及其共表达基因表达热图; c. Smad4及其共表达基因表达热图; d. Smad5及其共表达基因表达热图; e. Smad6及其共表达基因表达热图; 绘制热图基因来自与Smad基因共表达系数最高的前20个基因。Str: 横纹肌; Smo: 平滑肌; Hem: 血; Hep: 腺肝胰; Eye: 眼; Gil: 鳃; Kid: 肾; Man: 外套膜; Gon: 性腺; Foo: 足 |
| GO号 | GO项 | 校正后P值 | 基因数 |
| GO: 0048522 | Positive regulation of cellular process | 0.006 | 6 |
| GO: 0008283 | Cell proliferation | 0.022 | 2 |
| GO: 1902578 | Single-organism localization | 0.022 | 4 |
| GO: 0009719 | Response to endogenous stimulus | 0.026 | 6 |
| GO: 0009893 | Positive regulation of metabolic process | 0.027 | 3 |
| GO: 0019222 | Regulation of metabolic process | 0.035 | 44 |
| GO: 0042221 | Response to chemical | 0.038 | 7 |
| GO: 0043021 | Ribonucleoprotein complex binding | 0.041 | 2 |
| GO号 | GO项 | 校正后P值 | 基因数 |
| GO: 0006996 | Organelle organization | 0.000 | 37 |
| GO: 0065008 | Regulation of biological quality | 0.000 | 21 |
| GO: 0044237 | Cellular metabolic process | 0.001 | 298 |
| GO: 0016043 | Cellular component organization | 0.001 | 51 |
| GO: 0044238 | Primary metabolic process | 0.006 | 337 |
| GO: 0007155 | Cell adhesion | 0.007 | 6 |
| GO: 0006807 | Nitrogen compound metabolic process | 0.007 | 175 |
| GO: 0071704 | Organic substance metabolic process | 0.008 | 357 |
| GO: 0051641 | Cellular localization | 0.025 | 36 |
| GO: 0051649 | Establishment of localization in cell | 0.042 | 34 |
| 基因 | 主要功能 |
| TLR1 | TLR1可与TLR2和TLR6协同作用识别酰基脂肽 |
| TLR2 | 促进细菌脂蛋白的凋亡 |
| TLR6 | 主要识别细菌的肽和多脂糖 |
| TLR13 | 激活NF-kB通路 |
| TRAF2 | 调节TNF-R1介导的NF-kB通路 |
| TRAF3 | 参与TLR非依赖性的调节抗病毒反应 |
| MyD88 | 调节许多促炎基因的激活 |
| NFkB2 | 氧化细菌或病毒抗原 |
| GO号 | GO项 | 校正后P值 | 基因数 |
| GO: 0051716 | Cellular response to stimulus | 0.003 | 129 |
| GO: 0023051 | Regulation of signaling | 0.007 | 17 |
| GO: 0048583 | Regulation of response to stimulus | 0.012 | 17 |
| GO: 0044765 | Single-organism transport | 0.015 | 74 |
| GO: 0043234 | Protein complex | 0.020 | 25 |
| GO: 0043227 | Membrane-bounded organelle | 0.030 | 38 |
| GO: 0008047 | Enzyme activator activity | 0.001 | 12 |
| GO: 0060589 | Nucleoside-triphosphatase regulator activity | 0.002 | 12 |
| GO: 0005515 | Protein binding | 0.004 | 305 |
| GO: 0043167 | Ion binding | 0.018 | 233 |
| GO: 0038023 | Signaling receptor activity | 0.034 | 58 |
| GO: 0016787 | Hydrolase activity | 0.043 | 109 |
| GO号 | GO项 | 校正后P值 | 基因数 |
| GO: 0006955 | Immune response | 0.000 | 9 |
| GO: 0044238 | Primary metabolic process | 0.002 | 193 |
| GO: 0051707 | Response to other organism | 0.003 | 3 |
| GO: 0071704 | Organic substance metabolic process | 0.004 | 212 |
| GO: 0048584 | Positive regulation of response to stimulus | 0.008 | 3 |
| GO: 0002682 | Regulation of immune system process | 0.010 | 2 |
| GO: 0051606 | Detection of stimulus | 0.010 | 2 |
| GO: 0006807 | Nitrogen compound metabolic process | 0.010 | 87 |
| GO: 0009605 | Response to external stimulus | 0.015 | 3 |
| GO: 0044237 | Cellular metabolic process | 0.033 | 171 |
| GO: 0009607 | Response to biotic stimulus | 0.042 | 4 |
为研究Smad基因响应鳗弧菌侵染的表达模式, 本研究监测了侵染72 h内栉孔扇贝的Smad基因表达变化。结果显示, Smad3基因在侵染24 h时上调表达, 48 h之后与0 h的表达量基本一致。Smad5基因在侵染24 h时, 表达量上调1.74倍, 显著上调。在侵染48 h时, Smad5基因的表达量较24 h有所降低, 但仍显著高于0 h时。直至侵染后的72 h, Smad5基因的表达逐步恢复正常, 与0 h的表达量基本一致(图 5)。暗示Smad3和Smad5基因参与了栉孔扇贝的免疫应答。
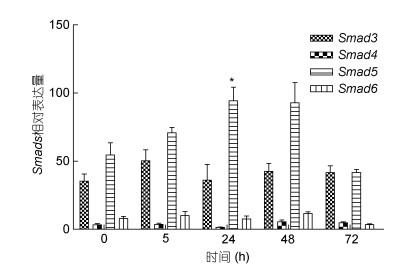 |
| 图 5 鳗弧菌感染后Smad基因在栉孔扇贝血淋巴中的表达模式 Fig. 5 Expression of Smad genes in the hemolymph of C. farreri after V. anguillarum challenge 注: 竖线表示平均值±SE (n=3), 星号表示显着差异(P < 0.05) |
本研究通过检测Smad基因的表达量以及构建共表达网络, 分析了Smad基因及其共表达基因在栉孔扇贝不同成体组织中的表达模式。Smad基因家族各基因在栉孔扇贝成体各组织均产生了不同程度的表达, Smad3在横纹肌中表达量最高, 暗示其在肌肉组织生长和发育过程中具有重要作用。在哺乳动物中, 成肌细胞中的Smad3可以与转录因子Myod1结合, 促进成肌细胞的肌源性分化(Huang et al, 2016)。在三角帆蚌中, 敲降Smad3可加速伤口的愈合(Hu et al, 2017)。Smad4是目前发现的哺乳动物中唯一一个Co-Smad成员, 可以介导所有TGF-β超家族成员信号的传递(Nakao et al, 1997)。Smad4在栉孔扇贝肌肉、眼、鳃、肾、性腺、肝胰腺、外套膜、血和足中均有不同程度的表达, 这说明与哺乳动物中Smad4广泛的生物学作用一致。其中, 性腺中Smad4表达量最高, 暗示了该基因可能参与了扇贝的性腺发育。在哺乳动物中, Smad4对于卵巢颗粒细胞的增殖与分化、颗粒细胞和膜细胞的增殖与凋亡、卵母细胞的成熟、排卵、繁殖性能等方面有着重要作用。Smad4在水牛和绵羊的卵巢中高度表达。有实验证明, 特异性敲除小鼠卵巢颗粒细胞中的Smad4基因, 会导致雌性小鼠生育能力下降甚至不育; 特异性敲除卵母细胞中的Smad4基因, 也会导致小鼠不育(秦智娟等, 2016)。Smad5在血液中特异性高表达, 暗示其参与调控了血细胞的生成与发育。在哺乳动物中, Smad5调控胚胎的造血发育机制并且介导由中胚层细胞向造血特化的负向调控。Smad5缺失小鼠的原始红系祖细胞增殖形成集落的能力减弱。Smad6在鳃中的表达量最高, 推测其在鳃中发挥调控作用。鳃是贝类滤食、呼吸的重要器官, 其与外界接触最多, 需要对外部环境的变化及时做出调节反应(郭海燕等, 2007)。
贝类只存在天然免疫, 其主要通过血淋巴细胞行使天然免疫功能(刘世良等, 2003)。为探究Smad基因家族在贝类免疫调节中的作用机制, 本研究分析了鳗弧菌侵染72 h内血细胞中Smad基因家族各成员的表达情况。结果发现, Smad3和Smad5均上调表达。其中, Smad5在侵染后的24 h时, 发生显著上调。这与Hu等(2017)在用嗜水气单胞菌感染三角帆蚌后, 血细胞中Smad3和Smad5的表达变化情况基本相一致。在哺乳动物中, Smad3可通过调控增强子结合蛋白(CEBP/ε)来促进中性粒细胞的成熟和功能极化, 进而增强机体的抗感染能力, 发挥天然免疫调节作用。我们推测, Smad3在扇贝中也发挥天然免疫调控功能。另外我们发现与Smad5共表达的基因中包含大量Toll样受体信号通路的关键基因(表 8)。Toll样受体(TLRs)是一类在脊椎动物和无脊椎动物中发挥天然免疫反应极为重要的蛋白质分子。Toll样受体对病原体的病原相关分子模式(PAMPs)进行识别, 是天然免疫发挥防御作用的关键。到目前为止, 已发现11个TLR家族成员(胡宁克等, 2011)。邱丽梅(2006)在栉孔扇贝中克隆得到了CfToll-1、CfMyD88、CfTRAF6、CfNFkB和CfIkB基因, 表明贝类中确实存在TLR信号通路。据此, 可以推测Smad5与TLR家族相互作用, 共同调控扇贝的免疫应答。
此外, 我们还分析了各Smad成员的共线性情况。结果发现, 在所有分析物种中Smad3和Smad6均在分布同一条染色体上。考虑到软体动物的早期起源及其相对原始的基因组, 这意味着Smad3和Smad6的共染色体现象可能早已存在于两侧对称动物的共同祖先中(Li et al, 2017)。
4 结论综上所述, 本研究中通过对栉孔扇贝Smad基因家族进行系统发生、共表达网络构建、以及鳗弧菌侵染的免疫应答分析, 解析了贝类Smad基因家族的表达调控机制和在免疫应答中的作用。结果表明, Smad3和Smad6的共染色体现象可能早已存在于两侧对称动物的共同祖先中; Smad5基因可能通过与Toll样受体信号通路相互作用, 参与机体的免疫应答。本研究将为贝类Smad基因在天然免疫系统中的作用提供进一步参考。
刘世良, 麦康森. 2003. 贝类免疫系统和机理的研究进展. 海洋学报, 25(2): 95-105 DOI:10.3321/j.issn:0253-4193.2003.02.011 |
李成林, 宋爱环, 胡炜, 等. 2011. 山东省扇贝养殖产业现状分析与发展对策. 海洋科学, 35(3): 92-98 |
吴婷婷, 赵阳, 赵勇, 2014. Smad3对中性粒细胞分化及功能极化的调控作用. 见: 中国免疫学会主编. 第九届全国免疫学学术大会论文集. 济南: 山东省免疫学会
|
邱丽梅, 2006. 栉孔扇贝(Chlamys farreri) Toll样受体及其信号传递相关基因的克隆与表达. 青岛: 中国科学院研究生院(海洋研究所)博士学位论文, 18-30
|
胡宁克, 陆欢平. 2011. Toll样受体功能研究进展. 现代检验医学杂志, 26(1): 14-16 DOI:10.3969/j.issn.1671-7414.2011.01.006 |
秦智娟, 杨晓清. 2016. 基因敲除在雌性小鼠生殖系统研究中的应用. 国际生殖健康/计划生育杂志, 35(1): 51-55 |
钱雪骏, 董迎辉, 姚韩韩, 等. 2015. 泥蚶Smad1/5基因cDNA全长克隆及时空表达特征分析. 水产学报, 39(9): 1302-1312 |
郭海燕, 叶嘉, 陈雷, 等. 2007. 瓣鳃纲贝类摄食器官组织学研究进展. 邯郸学院学报, 17(3): 74-76 DOI:10.3969/j.issn.1673-2030.2007.03.021 |
郭慧慧, 2012. 栉孔扇贝TGF-β/Smad信号通路基因的克隆、表达分析及生长性状相关SNP位点筛查. 青岛: 中国海洋大学博士学位论文, 50-51
|
窦海鸽, 黄倢, 王秀华, 等. 2005. 工厂化海水养殖中刺激隐核虫病的综合防治新技术. 北京水产, (2): 34-35 |
Bailey T L, Elkan C, 1994. Fitting a mixture model by expectation maximization to discover motifs in biopolymers. Proceedings of International Conference on Intelligent Systems for Molecular Biology, 2: 28-36 |
Bjellqvist B, Hughes G J, Pasquali C et al, 1993. The focusing positions of polypeptides in immobilized pH gradients can be predicted from their amino acid sequences. Electrophoresis, 14(1): 1023-1031 DOI:10.1002/elps.11501401163 |
Cong M, Song L S, Wang L L et al, 2008. The enhanced immune protection of Zhikong scallop Chlamys farreri on the secondary encounter with Listonella anguillarum. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 151(2): 191-196 DOI:10.1016/j.cbpb.2008.06.014 |
Datto M B, Frederick J P, Pan L et al, 1999. Targeted disruption of Smad3 reveals an essential role in transforming growth factor β-mediated signal transduction. Molecular and Cellular Biology, 19(4): 2495-2504 DOI:10.1128/MCB.19.4.2495 |
Derynck R, Zhang Y E, 2003. Smad-dependent and Smad-independent pathways in TGF-β family signalling. Nature, 425(6958): 577-584 DOI:10.1038/nature02006 |
Dobin A, Davis C A, Schlesinger F et al, 2013. STAR: ultrafast universal RNA-seq aligner. Bioinformatics, 29(1): 15-21 DOI:10.1093/bioinformatics/bts635 |
Grishin N V, 2001. Mh1 domain of Smad is a degraded homing endonuclease. Journal of Molecular Biology, 307(1): 31-37 DOI:10.1006/jmbi.2000.4486 |
Hu X L, Bao Z M, Hu J J et al, 2006. Cloning and characterization of tryptophan 2, 3-dioxygenase gene of Zhikong scallop Chlamys farreri (Jones and Preston 1904). Aquaculture Research, 37(12): 1187-1194 DOI:10.1111/j.1365-2109.2006.01546.x |
Hu B Q, Yi P P, Li Z F et al, 2017. Molecular characterization of two distinct Smads gene and their roles in the response to bacteria change and wound healing from Hyriopsis cumingii. Fish & Shellfish Immunology, 67: 129-140 |
Huang Q K, Qiao H Y, Fu M H et al, 2016. MiR-206 attenuates denervation-induced skeletal muscle atrophy in rats through regulation of satellite cell differentiation via TGF-β1, Smad3, and HDAC4 signaling. Medical Science Monitor: International Medical Journal of Experimental and Clinical Research, 22: 1161-1170 |
Huang D W, Sherman B T, Lempicki R A, 2009. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research, 37(1): 1-13 DOI:10.1093/nar/gkn923 |
IBM, 2013. IBM, SPSS statistics for windows, version 20.0. New York: IBM
|
Kumar S, Stecher G, Tamura K, 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 33(7): 1870-1874 DOI:10.1093/molbev/msw054 |
Langfelder P, Horvath S, 2008. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics, 9: 559 DOI:10.1186/1471-2105-9-559 |
Larkin M A, Blackshields G, Brown N P et al, 2007. Clustal W and Clustal X version 2.0. Bioinformatics, 23(21): 2947-2948 DOI:10.1093/bioinformatics/btm404 |
Letunic I, Bork P, 2007. Interactive Tree of Life (iTOL): an online tool for phylogenetic tree display and annotation. Bioinformatics, 23(1): 127-128 DOI:10.1093/bioinformatics/btl529 |
Letunic I, Bork P, 2018. 20 years of the SMART protein domain annotation resource. Nucleic Acids Research, 46(D1): D493-D496 DOI:10.1093/nar/gkx922 |
Li H, Handsaker B, Wysoker A et al, 2009. The sequence alignment/map format and SAMtools. Bioinformatics, 25(16): 2078-2079 DOI:10.1093/bioinformatics/btp352 |
Li Y L, Sun X Q, Hu X L et al, 2017. Scallop genome reveals molecular adaptations to semi-sessile life and neurotoxins. Nature Communications, 8(1): 1721 DOI:10.1038/s41467-017-01927-0 |
Li Y P, Zhang L L, Sun Y et al, 2016. Transcriptome sequencing and comparative analysis of ovary and testis identifies potential key sex-related genes and pathways in scallop Patinopecten yessoensis. Marine Biotechnology, 18(4): 453-465 DOI:10.1007/s10126-016-9706-8 |
Liao Y, Smyth G K, Shi W, 2014. FeatureCounts: an efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics, 30(7): 923-930 DOI:10.1093/bioinformatics/btt656 |
Liu G, Huan P, Liu B Z, 2014. Cloning and expression patterns of two Smad genes during embryonic development and shell formation of the Pacific oyster Crassostrea gigas. Chinese Journal of Oceanology and Limnology, 32(6): 1224-1231 DOI:10.1007/s00343-014-3360-7 |
Liu W Z, Xie Y B, Ma J Y et al, 2015. IBS: an illustrator for the presentation and visualization of biological sequences. Bioinformatics, 31(20): 3359-3361 DOI:10.1093/bioinformatics/btv362 |
Massagué J, Wotton D, 2000. Transcriptional control by the TGF-β/Smad signaling system. The EMBO Journal, 19(8): 1745-1754 DOI:10.1093/emboj/19.8.1745 |
Nakao A, Imamura T, Souchelnytskyi S, 1997. TGF-β receptor-mediated signalling through Smad2, Smad3 and Smad4. The EMBO Journal, 16(17): 5353-5362 DOI:10.1093/emboj/16.17.5353 |
Wang J, Yu H W, Yao L J et al, 2020. Genomic and transcriptomic landscapes and evolutionary dynamics of molluscan glycoside hydrolase families with implications for algae-feeding biology. Computational and Structural Biotechnology Journal, 18: 2744-2756 DOI:10.1016/j.csbj.2020.09.021 |
Weinstein M, Yang X, Deng C X, 2000. Functions of mammalian Smad genes as revealed by targeted gene disruption in mice. Cytokine & Growth Factor Reviews, 11(1/2): 49-58 |
Wu J W, Hu M, Chai J J et al, 2001. Crystal structure of a phosphorylated Smad2. recognition of phosphoserine by the MH2 domain and insights on Smad function in TGF-β signaling. Molecular Cell, 8(6): 1277-1289 DOI:10.1016/S1097-2765(01)00421-X |
 2021, Vol. 52
2021, Vol. 52


