中国海洋湖沼学会主办。
文章信息
- 任翊卓, 韩学凯, 左佳俊, 欧阳雪梅, 段虎, 隋丽英. 2021.
- REN Yi-Zhuo, HAN Xue-Kai, ZUO Jia-Jun, OUYANG Xue-Mei, DUAN Hu, SUI Li-Ying. 2021.
- Piwi基因参与两性卤虫(Artemia franciscana)的生殖调控研究
- THE REPRODUCTIVE REGULATION OF Piwi IN BISEXUAL ARTEMIA FRANCISCANA
- 海洋与湖沼, 52(6): 1567-1576
- Oceanologia et Limnologia Sinica, 52(6): 1567-1576.
- http://dx.doi.org/10.11693/hyhz20210300069
文章历史
-
收稿日期:2021-03-16
收修改稿日期:2021-04-29
Piwi (P-element induced wimpy testis)蛋白属于Argonaute蛋白家族, 是RNA沉默通路中的主要蛋白。Argonaute蛋白具有PAZ和Piwi两个主要结构域(Höck et al, 2008), 通过小RNAs介导基因调控从而在不同生理过程中促进细胞稳态(Sheu-Gruttadauria et al, 2017)。Piwi基因在果蝇(Drosophila)生殖腺中被首次发现, 在生殖干细胞的自我更新中发挥着重要作用(Cox et al, 1998), 主要由Piwi蛋白与小RNA (piRNAs)相互关联形成复合体来实现其功能(Ozata et al, 2019)。研究发现Piwi蛋白在果蝇(Marie et al, 2017)、斑马鱼(Barchydanio rerio) (Houwing et al, 2007)和小鼠(Mus musclus) (Xu et al, 2008)生殖系转座子沉默中起关键作用, 是多种生物生殖系发育和干细胞自我更新过程的关键调控因子(Ramat et al, 2021)。Piwi蛋白与piRNAs形成piRNA通路, 在生殖细胞发育、性别决定、剂量补偿等与有性生殖相关的发育过程中起着至关重要的作用(Ramat et al, 2021)。Piwi或Piwi同源物基因的缺失会导致线虫(Caenorhabditis elegans) (Wang et al, 2008)、蝇类(Li et al, 2009)及哺乳动物(Kim et al, 2012)的生育能力严重受损。在甲壳动物中研究发现Piwi蛋白可在斑节对虾(Penaeus monodon)生殖细胞发育中发挥作用, PmPiwi1可能参与了精子成熟的后期阶段(Sukthaworn et al, 2020)。此外, Piwi蛋白对piRNAs的生物合成(Czech et al, 2016)、表观遗传调控(Huang et al, 2013)以及癌细胞生存和转移(Halajzadeh et al, 2020)都是至关重要的。
卤虫(Artemia)属小型甲壳动物, 广泛分布于世界各地沿海盐场和内陆盐湖高盐水体中, 是卤水生态系统食物链的重要组成部分和生物调节者(Dattilo et al, 2005), 无节幼体是水产养殖育苗中重要的生物饵料(Lopes-dos-Santos et al, 2019)。卤虫具有世代周期短、子代产量高和易于培养等优点, 是基础和应用生物学研究的良好实验动物(Manfra et al, 2015)。按照生殖方式, 卤虫可以分为两性(Bisexual)卤虫和孤雌(Parthenogenetic)卤虫两种类型, 并且均具有卵生(Oviparity)和卵胎生(Ovoviviparity)两种繁育方式, 而卤虫生殖和繁育方式的特殊性也逐渐成为研究者关注的热点。在卤虫生殖及性别决定方面, 鳞翅类性别决定基因Masc的同源基因已被证实是调控卤虫性别分化的途径之一(Li et al, 2017); p90 RSK被证实参与卤虫卵母细胞成熟及休眠胚胎细胞周期等过程(段如冰, 2014); 而小热休克蛋白p26影响卤虫的胚胎发育并在休眠生殖途径中发挥重要作用(King et al, 2012)。Piwi基因在生殖方面起着重要的作用, Dung等(2019)在卤虫中发现了Piwi蛋白家族成员Ago, 但未对其功能进行研究, 而关于Piwi基因在卤虫中的研究尚未见报道。本研究以两性卤虫为研究对象, 获得该基因的ORF序列并对其进行生物信息学分析, 运用qPCR检测生殖腺不同发育时期Piwi基因的表达特征, 利用RNAi技术探究其功能, 为揭示两性卤虫的生殖发育调控机制提供重要参考。
1 材料与方法 1.1 实验材料本实验选取两性卤虫A. franciscana (VinhChau strain), 在28 ℃光照条件下(24L : 0D)孵化培养。选取6个1L锥形瓶, 每个锥形瓶饲养200只两性卤虫并投喂新鲜盐生杜氏藻Dunaliella salina。根据孙瑜霞(2014)关于两性卤虫胚胎发育的描述, 收集雌性卤虫卵黄发生早期(孵化后约16 d)、卵黄发生晚期(孵化后约19 d)、早期胚胎(孵化后约20 d)、晚期胚胎(孵化后约25 d)各个时期的卵巢。通过观察雄性卤虫精巢的形态特征及成熟程度, 将雄虫分为未成熟期(孵化后约8 d)、成熟早期(孵化后约13 d)、成熟中期(孵化后约19 d)、成熟晚期(孵化后约25 d)并收集各个时期的精巢, 将解剖后的组织迅速置于液氮中, 于–80 ℃冰箱中保存。
1.2 总RNA的提取及反转录取两性卤虫卵巢和精巢组织, 采用Trizol法提取组织总RNA, 用1.2%琼脂糖凝胶电泳检测RNA纯度, 并用核酸蛋白测定仪(Eppendorf BioPhotometer plus)测定RNA浓度, 后置于–80 ℃冰箱保存。选用A260/A280范围在1.8—2.0的RNA作为模板, 通过TaKaRa试剂盒进行反转录, 将得到的cDNA分别作为常规PCR和qPCR模板, 并存于–20 ℃冰箱备用。
1.3 两性卤虫Piwi基因的获得及分析从前期构建的两性卤虫A. franciscana转录组数据库中筛选获得Piwi基因片段, 该基因序列包括完整的开放阅读框(Open reading frame, ORF)。为保证基因序列的准确性, 利用Primer Premier5软件设计3对Piwi基因特异性引物(表 1), 拼接得到其完整ORF用于比对。本实验涉及的引物均由北京华大基因科技有限公司合成。以两性卤虫cDNA为模板进行扩增, PCR体系(25 μL): ddH2O 10.5 μL、Premix Ex Taq 12.5 μL、上下游引物各0.5 μL、cDNA模板1 μL。PCR反应条件: 94 ℃预变性5 min; 94 ℃变性1 min, 54 ℃退火1 min, 72 ℃延伸1 min 30 s, 35个循环; 72 ℃延伸10 min, PCR产物用1.2%琼脂糖凝胶电泳检测条带扩增结果, 将含有目的片段的PCR产物送到北京华大基因科技有限公司测序。
| 引物名称 | 序列(5′—3′) | 退火温度(℃) | 片段大小(bp) | 用途 |
| Piwi-F1 | ATTCGGATGTTGAAGCAG | 53 | 854 | 基因扩增 |
| Piwi-R1 | CCCGGCCATACCTCTAT | |||
| Piwi-F2 | GAAGAGTTATGGCGAAGAT | 60 | 1 107 | 基因扩增 |
| Piwi-R2 | CGCAGGGTTTGGAGACA | |||
| Piwi-F3 | AAACCCTGCGCCTCCCA | 63 | 927 | 基因扩增 |
| Piwi-R3 | AAGCCACTACAATCACCAGTACAAA | |||
| Piwi-F | GCAGCACCGTTTCGTTC | 60 | 114 | qPCR |
| Piwi-R | CCCATTTCCGTTACCATT | |||
| GAPDH-F | GGTCGTGACTTGACGGACTATCT | 57 | 120 | 内参基因 |
| GAPDH-R | AGCGGTTGCCATTTCTTGTT | |||
| dsPiwi-F | T7-TTGTCGCTCAGATTCCTC | 70 | 487 | dsRNA合成 |
| dsPiwi-R | T7-GCCCTCATTTCGTCAGT | |||
| dsEGFP-F | T7-CAGTGCTTCAGCCGCTACCC | 58 | 350 | dsRNA合成 |
| dsEGFP-R | T7-AGTTCACCTTGATGCCGTTCTT |
通过在线软件(表 2)对两性卤虫Piwi基因进行生物信息学分析, 包括对测序拼接得到的序列进行ORF、理化性质、亲/疏水性、信号肽以及亚细胞定位预测分析, 并进行跨膜结构、保守结构域、二级结构和三级结构预测; 采用NCBI (http://www.ncbi.nlm.nih.gov)数据库对两性卤虫Piwi基因的氨基酸序列进行BLAST比对, 分析与其他物种的相似性; 利用MEGA5.0软件将不同物种的氨基酸序列进行多重比对, 构建系统进化树。
| 在线软件 | 用途 | 网址 |
| ORF Finder-NCBI | 开放阅读框分析 | https://www.ncbi.nlm.nih.gov/orffinder/ |
| ExPASy-ProtParam | 理化性质分析 | https://web.expasy.org/protparam/ |
| ExPASy-ProtScale | 亲水性/疏水性分析 | https://web.expasy.org/protscale/ |
| TMHMM Server V 2.0 | 跨膜结构预测 | https://www.cbs.dtu.dk/services/TMHMM/ |
| SignallP-5.0 Server | 信号肽分析 | https://www.cbs.dtu.dk/services/SignalP/ |
| CDD | 保守结构域预测 | https://www.ncbi.nlm.nih.gov/Structure/cdd/docs/cdd_search.html |
| PSORT II Prediction | 亚细胞定位 | https://psort.hgc.jp/form2.html |
| SOPMA | 二级结构预测 | https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html |
| SWISS-MODEL | 三级结构预测 | https://swissmodel.expasy.org/ |
根据测序得到的两性卤虫Piwi基因序列设计qPCR特异性引物Piwi-F和Piwi-R (表 1), 以GAPDH作为内参基因对两性卤虫卵巢和精巢不同发育时期的mRNA表达量进行qPCR检测(美国伯乐Bio-rad)。反应条件(20 μL): TB Green Premix Ex Taq II 10 μL、上下游引物各0.8 μL、ddH2O 6.4 μL、cDNA模板2 μL。反应程序为: 95 ℃预变性3 min, 95 ℃变性10 s, 退火60 ℃ 30 s, 40个循环。每个样本设置3个生物学重复和3个技术重复, 采用2–△△Ct法计算目的基因在生殖腺不同发育时期的相对表达量, 通过SPSS 25.0软件对其进行单因素方差分析, P < 0.05为差异显著, P < 0.01为差异极显著。
1.6 dsRNA的合成及显微注射RNAi利用Primer Premier5软件设计并合成Piwi基因dsRNA引物dsPiwi-F和dsPiwi-R及阴性对照增强型绿色荧光蛋白EGFP基因dsRNA引物dsEGFP-F和dsEGFP-R (表 1), 引物的5′端包含T7启动子序列(TAATACGACTCACTATAGGG)。通过PCR扩增Piwi基因的ORF片段并进行纯化回收, 获得的基因片段连接到pGM-T载体上并提取质粒, 使用TranscriptAid T7 High Yield Transcription Kit体外转录试剂盒从质粒中体外转录dsRNA, 纯化dsRNA并进行消化反应检测其质量(王志伟, 2017)。阴性对照EGFP基因dsRNA的合成同上。
分别设置对照组(dsEGFP)和干扰组(dsPiwi), 每组60只雌性卤虫, 采用显微注射(日本Nikon NARISHIGE)对活力较好并处于卵黄发生早期的雌性卤虫进行注射, 注射部位为雌性卤虫外骨骼和肠道之间的开放循环体腔。将dsRNA溶于磷酸缓冲液中并与0.1%酚红混合(1︰1, 体积分数)得到dsRNA显微注射溶液, 每只雌性卤虫注射1 μL含1 000 ng dsRNA的溶液, 注射成功的雌性卤虫放入50 mL试管中单独培养并进行雌雄配对。待雌性卤虫发育到卵黄发生晚期、早期胚胎和晚期胚胎时分别收集卵囊, 每个时期设置3个平行, 每个平行收集5个卵囊, 利用qPCR测定干扰效果。剩余的对照组和干扰组雌性卤虫各15只继续培养并观察产后代情况, 通过SPSS 25.0软件对数据进行卡方检验分析。
2 结果 2.1 两性卤虫Piwi基因ORF序列的扩增及测序经1.2%琼脂糖凝胶电泳对Piwi基因的PCR产物进行检测, 得到清晰单一的条带(图 1), 对测序结果进行拼接比对后, 发现得到的基因序列与预期目的基因片段大小一致, 验证后得到两性卤虫Piwi基因ORF大小为2 619 bp, 编码872个氨基酸。
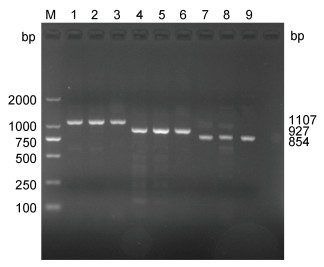 |
| 图 1 两性卤虫Piwi基因PCR扩增结果 Fig. 1 Results of PCR amplification of Piwi gene of A. franciscana 注: M. Marker DL 2000; 1—3. 引物Piwi-F2/R2的扩增条带; 4—6. 引物Piwi-F3/R3的扩增条带; 7—9. 引物Piwi-F1/R1的扩增条带 |
两性卤虫Piwi基因氨基酸序列与昆虫纲、甲壳纲(节肢动物门)和双壳纲、瓣鳃纲(软体动物门)相关物种的Piwi基因氨基酸序列相对较近, 相似性范围为41.05%—43.02% (表 3)。使用MEGA5.0软件构建Piwi基因系统进化树(图 2), 表明两性卤虫Piwi基因与蚤状溞和大型溞的Piwi序列相似性最高。
| 序列号 | 物种名称 | 序列相似性 |
| XP_022341466.1 | 美洲牡蛎(Crassostrea virginica) | 43.02% |
| XP_031328334.1 | 萤火虫(Photinus pyralis) | 42.99% |
| KXJ68137.1 | 白纹伊蚊(Aedes albopictus) | 42.82% |
| XP_023703405.1 | 白蚁(Cryptotermes secundus) | 42.76% |
| QID20005.1 | 果蝇(Drosophila koepferae) | 42.39% |
| RVE42096.1 | 二化螟(Chilo suppressalis) | 42.36% |
| AMN88361.1 | 紫贻贝(Mytilus galloprovincialis) | 42.26% |
| AFW04603.1 | 短翅鸣螽(Gampsocleis gratiosa) | 42.03% |
| XP_028523889.1 | 中华蜜蜂(Apis cerana) | 41.99% |
| QAW56456.1 | 马氏珠母贝(Pinctada fucata) | 41.87% |
| EFX71843.1 | 蚤状溞(Daphnia pulex) | 42.29% |
| RZF43882.1 | 灰飞虱(Laodelphax striatellus) | 41.07% |
| KZS2095.1 | 大型溞(Daphnia magna) | 41.05% |
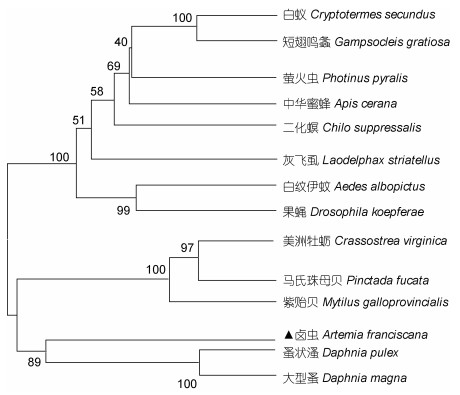 |
| 图 2 两性卤虫Piwi基因氨基酸系统进化树 Fig. 2 Amino acid phylogenetic tree of Piwi gene of A. franciscana |
两性卤虫Piwi基因编码蛋白的分子式为C4362H6911N1237O1255S42, 分子质量为98.11 kDa, 氨基酸总数为872, 含量最多的是甘氨酸Gly (8.1%)和缬氨酸Val(8.0%), 其次是精氨酸Arg (7.2%)和亮氨酸Leu (7.0%), 带负电荷的残基总数(Asp+Glu)为89, 带正电荷的残基总数(Arg+Lys)为122, 总平均亲水性(GRAVY)为–0.386, 理论等电点为9.50, 不稳定指数为33.97。
2.4 两性卤虫Piwi基因编码蛋白的信号肽、亲/疏水性及亚细胞定位预测分析信号肽分析发现Piwi基因编码蛋白无信号肽(图 3a)。亲/疏水性分析得到748位点处的半胱氨酸(Cys)有最大值为2.222, 115位点处的天冬氨酸(Asp)有最小值为–2.844 (图 3b), 该蛋白为亲水性蛋白。亚细胞定位发现该蛋白在细胞质内分布最多(47.8%), 其次为细胞核(30.4%), 线粒体(13%)、细胞骨架(4.3%)和质膜(4.3%)分布最少。
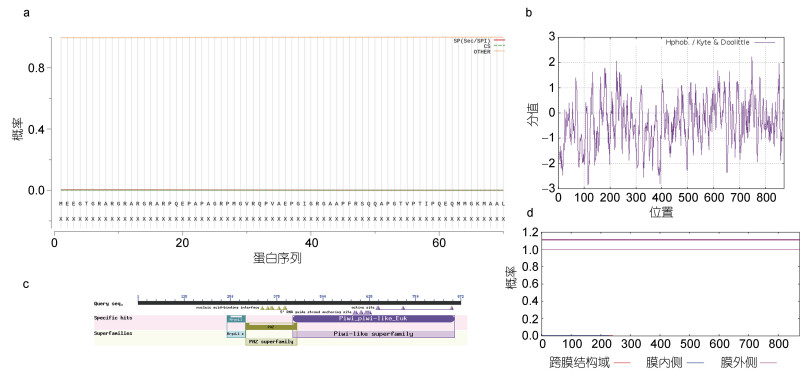 |
| 图 3 两性卤虫Piwi基因编码蛋白信号肽、亲水性、保守结构域、跨膜结构预测 Fig. 3 Prediction of signal peptide, hydrophilicity, conserved domain, and transmembrane structure of the protein encoded by Piwi gene in A. franciscana 注: a. Piwi蛋白信号肽分析; b. Piwi蛋白亲水性分析; c. Piwi蛋白保守结构域预测; d. Piwi蛋白跨膜结构预测 |
对Piwi蛋白的保守结构域预测发现该蛋白共包含3个结构域, 其中第241—291位氨基酸为ArgoL1结构域, 第292—430位氨基酸为PAZ结构域, 第418—855位氨基酸为Piwi结构域(图 3c)。对跨膜结构域预测发现Piwi蛋白无跨膜结构域(图 3d)。
2.6 两性卤虫Piwi基因编码蛋白的高级结构预测对Piwi蛋白的二级结构预测发现该蛋白主要由4种折叠方式构成, 282个α-螺旋占比32.34%, 177个延伸链占比20.30%, 39个β-转角占比4.47%, 374个无规则卷占比42.87%(图 4a)。蛋白的三级结构模型与二级结构预测相符, 无规则卷、α-螺旋和延伸链是该蛋白结构的主体(图 4b)。
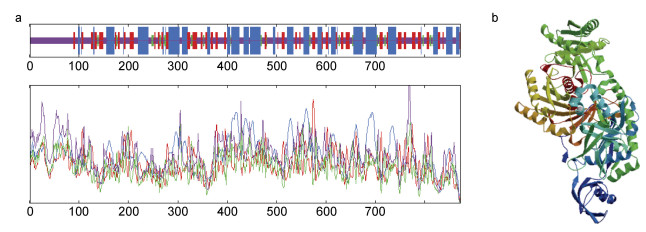 |
| 图 4 两性卤虫Piwi基因编码蛋白高级结构预测结果 Fig. 4 The results of advanced structure prediction of the protein encoded by Piwi gene in A. franciscana 注: a. 两性卤虫Piwi蛋白二级结构预测; b. 两性卤虫Piwi蛋白三级结构预测 |
通过qPCR检测Piwi基因在两性卤虫卵巢和精巢不同发育时期的表达量, 结果表明Piwi基因的表达普遍存在于卵巢和精巢中。在卵巢发育过程中, Piwi基因在卵黄发生晚期和晚期胚胎表达量最高, 显著高于卵黄发生早期和早期胚胎(P < 0.01); 在精巢发育过程中, Piwi基因在精巢未成熟期表达量最高, 显著高于精巢成熟早期、成熟中期和成熟晚期(P < 0.01) (图 5)。
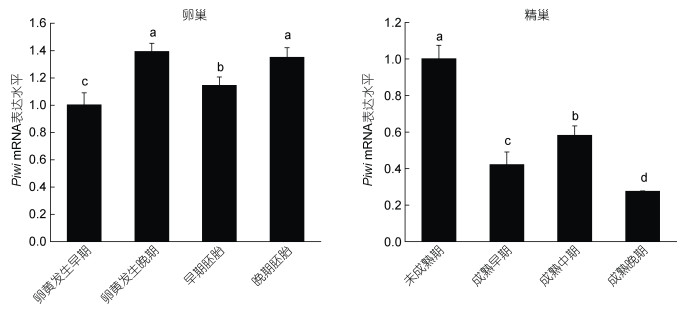 |
| 图 5 Piwi基因在两性卤虫不同发育时期卵巢和精巢中的表达 Fig. 5 Expression of Piwi gene in ovary and testis of A. franciscana at different developmental stages 注: 相同字母表示差异不显著(P > 0.05); 不同字母表示差异极显著(P < 0.01) |
与对照组dsEGFP相比, 雌性卤虫在注射dsPiwi后的卵黄发生晚期、早期胚胎、晚期胚胎均检测到Piwi基因的表达量极显著降低(P < 0.01), 干扰效率分别为91%、82%、92%, 说明显微注射实验是成功的(图 6)。观察RNAi干扰后雌性卤虫产后代的情况, 发现显微注射后大约10 d对照组(dsEGFP)和干扰组(dsPiwi)均有后代产出。15只注射dsPiwi的雌性卤虫中有10只雌性卤虫存活并全部产休眠卵; 15只注射dsEGFP的雌性卤虫中有11只雌性卤虫存活, 其中9只产无节幼体, 2只产休眠卵, 卡方检验显示对照组与干扰组产卵和产幼上存在极显著差异(P < 0.01), 因此Piwi基因的敲降诱导两性卤虫产休眠卵。
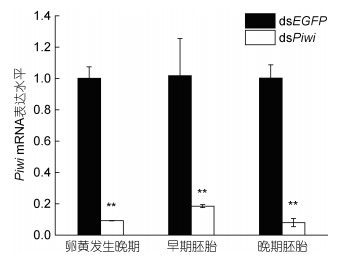 |
| 图 6 显微注射后Piwi基因在两性卤虫不同时期卵巢中的表达 Fig. 6 Expression of Piwi gene in ovary of A. franciscana at different developmental stages after microinjection 注: **表示与对照有极显著性差异(P < 0.01) |
本研究选取两性卤虫为研究对象, 得到了Piwi基因完整的ORF为2 619 bp, 编码872个氨基酸, 与甲壳动物中的中华绒螯蟹(Eriocheir sinensis) (王敏, 2015)以及昆虫中的家蚕(Bombyx mori) (廖珍, 2009)、小菜蛾(Plutella xylostella) (Hameed et al, 2018)等物种的Piwi基因序列长度相似。生物信息学分析显示, 两性卤虫Piwi蛋白分子质量为98.11 kDa, 总平均亲水性(GRAVY)为–0.386, 推测该蛋白为亲水性蛋白, 与亲/疏水性预测结果一致; 正电荷残基总数(Arg+Lys)大于负电荷残基总数(Asp+Glu), 故该蛋白带正电荷; 理论等电点为9.50, 为偏碱性蛋白; 不稳定指数为33.97, 属于稳定蛋白, 故该蛋白属于稳定的碱性亲水性蛋白。两性卤虫Piwi蛋白含有保守的PAZ和Piwi结构域, 属于Argonaute蛋白家族, 这与Dung等(2019)关于两性卤虫ArAgo蛋白结构域的研究结果一致。在动物中, Argonaute蛋白家族有AGO亚家族和PIWI亚家族两个分支, 其中PIWI亚家族由N端结构域、PAZ结构域、MID结构域和Piwi结构域组成, 而PAZ结构域和Piwi结构域是最保守的(Sheu-Gruttadauria et al, 2017)。PAZ结构域能够与piRNA的3′结合, Piwi结构域中心有引导单链裂解的RNaseH, 具有核酸内切酶活性(Kwon et al, 2014)。
piRNAs是一类主要在动物生殖系中表达的非编码小RNA, 与Piwi蛋白形成piRNA通路, 参与转座子沉默和基因表达调控, 在生物发育过程中发挥关键作用(Ramat et al, 2021)。该通路依赖piRNAs序列的特异性来识别转座子靶点, 参与转座子的转录后抑制和表观遗传来保护基因组的完整性; 通过Piwi蛋白提供效应功能, 在动物生殖细胞防御系统中发挥重要作用(Zhou et al, 2015)。Piwi蛋白是生殖腺发育过程中piRNA合成以及转录和转录后调控所必需的, 该蛋白成员定位于不同的细胞区域(Tamtaji et al, 2020)。本研究通过生物信息学亚细胞定位预测, 发现Piwi蛋白主要分布在细胞质(47.8%)和细胞核(30.4%)中, 推测Piwi蛋白在细胞核和细胞质中发挥着重要作用。果蝇(Klattenhoff et al, 2008)和小鼠(Ishizu et al, 2012)的研究结论也证实了这一观点, 果蝇的Argonaute蛋白家族成员Aubergine (Aub)蛋白和Argonaute 3 (Ago3)蛋白位于细胞质和生殖细胞颗粒中, 它们在piRNA的产生中发挥着重要作用, 而Piwi蛋白主要在生殖细胞核的转录沉默中起作用。此外, 小鼠中含有三种Piwi蛋白, Mili和Miwi在细胞质内参与piRNA合成, 调节细胞质转录后沉默, 而Miwi2位于细胞核, 在调控细胞核沉默中发挥作用。本研究通过相似性比对可知, Piwi基因在昆虫中的保守性较高, 且两性卤虫与昆虫的Piwi基因氨基酸序列相对较近。Li等(2017)的研究证实了鳞翅类性别决定基因Masc的同源基因是调控两性卤虫性别分化的途径之一, 也有报道称家蚕卵巢培养细胞系BmN4特异性表达两种PIWI亚家族蛋白Siwi和BmAgo3以及大量的piRNAs (Kawaoka et al, 2009), 此后又有研究发现家蚕中Fem、Masc基因可与Piwi蛋白组成“乒乓”性别级联通路, 从而进一步完善了piRNA通路(Shoji et al, 2017), 这些结果表明两性卤虫中Piwi基因可能与家蚕中Piwi基因行使着相似的功能。
为了进一步研究Piwi基因对两性卤虫的调控功能, 本研究通过qPCR检测Piwi基因在两性卤虫生殖腺中不同发育时期的表达量, 结果显示Piwi基因在两性卤虫卵巢和精巢各个阶段均有表达, 但表达量有显著差异。在两性卤虫受精前, Piwi基因在卵巢中的表达量逐渐上升, 到卵黄发生晚期Piwi基因表达量最高; 卵巢受精后, Piwi基因表达量骤降, 在之后4—5 d的胚胎发育过程中, Piwi基因表达量再次呈现上升的趋势, 这种表达趋势与家蚕和西方蜜蜂(Apis mellifera)卵巢发育中Piwi基因表达相似(廖珍, 2009)。在卵巢整个发育过程中, Piwi基因在卵黄发生晚期与晚期胚胎表达量最高, 推测Piwi基因在两性卤虫准备受精的过程和胚胎发育即将进入不同生殖途径过程中发挥着重要作用。
目前没有雄性卤虫精巢相关的报道, 本研究首次完成了对雄性卤虫的生殖发育周期的探究。与虾蟹等甲壳动物不同, 卤虫发育周期短, 本研究根据精巢发育程度将精巢发育分为四个时期。雄性卤虫精巢从未成熟期到成熟期的发育过程中, Piwi基因表达水平呈现逐渐下降的趋势, 且四个时期均存在极显著差异, 推测Piwi基因在雄性卤虫精巢发育前期起着重要作用。相比于其他甲壳动物, Piwi基因在中华绒螯蟹精巢中的表达与本研究结果相似, 即随着精巢的逐渐发育, Piwi表达量降低(王敏, 2015)。而在三疣梭子蟹(Portunus trituberculatus)中Piwi基因虽在精子细胞的不同发育阶段均有表达, 但其在精子细胞早期、中期和晚期的表达没有显著差异, 进一步研究发现Piwi蛋白主要分布在顶体小管和膜复合物上, 表明其可能在精子发生过程中发挥功能(Xiang et al, 2014)。
piRNAs和Piwi蛋白对于生殖系的形成和维持是必不可少的, piRNA通路受损会导致转座子的过表达, 显著破坏基因组结构, 并导致生殖细胞死亡和不育(Tóth et al, 2016)。斑马鱼的Ziwi (Houwing et al, 2007)和Zili (Houwing et al, 2008)两种Piwi蛋白对维持生殖系至关重要, Ziwi在生殖细胞发育的有丝分裂和早期减数分裂阶段大量表达, Ziwi缺失使幼鱼生殖细胞衰竭, 并导致成鱼生殖细胞异常凋亡。卤虫作为一种良好的实验动物, 其多种生物学机制如抗逆性和滞育(Liu et al, 2009)、细胞分裂(Chen et al, 2016)、发育分化(Copf et al, 2004)和繁殖(Dai et al, 2011)等方面研究已开展较多。本研究利用RNAi技术探究两性卤虫Piwi基因的功能, 对处于卵黄发生早期的雌性卤虫进行显微注射, 利用qPCR检测注射后雌性卤虫的干扰效率, 结果显示Piwi基因的干扰效率均在90%左右, 显微注射很成功。观察RNAi干扰后雌性卤虫产后代的情况, 发现对照组(dsEGFP)和干扰组(dsPiwi)胚胎发育周期都很正常, 然而干扰组由于Piwi基因的敲降导致两性卤虫胚胎滞育产生休眠卵。这说明Piwi基因的表达量减少可促使雌性卤虫胚胎发育进入卵生生殖途径, Piwi基因在两性卤虫的生殖发育过程中起到了重要作用, 这可能与两性卤虫的生殖方式决定机制相关。而在斑节对虾中, Piwi同源物PmPiwi1 (Sukthaworn et al, 2019)和PmPiwi2 (Sukthaworn et al, 2020)在斑节对虾雌雄生殖腺不同发育阶段均有表达, 且PmPiwi1或PmPiwi2的敲降都会导致精子数量的显著减少, 同样表明Piwi基因可能在斑节对虾生殖系发育中发挥重要作用。本研究为两性卤虫Piwi/piRNA分子机制和功能的后续研究以及两性卤虫生殖方式方面的研究提供了基础数据, 同时为甲壳动物生殖发育调控研究奠定了基础。
4 结论本研究成功获得两性卤虫Piwi基因完整的开放阅读框, 其总长2 619 bp, 编码872个氨基酸; 生物信息学分析显示, Piwi蛋白包含PAZ结构域、Piwi结构域和ArgoL1结构域, 二级结构的主体为无规则卷、α-螺旋和延伸链, 且该蛋白主要分布在细胞质和细胞核中; 进化树分析表明两性卤虫与大型溞和蚤状溞的Piwi序列相似性最高; qPCR结果显示两性卤虫在生殖腺中均有表达且在发育不同阶段存在差异表达。注射dsPiwi后能够有效抑制两性卤虫Piwi基因的表达, 且与对照组相比干扰组后代均产休眠卵。说明Piwi基因在两性卤虫的生殖发育及生殖方式决定过程中起到重要作用。
王敏, 2015. 中华绒螯蟹(Eriocheir sinensis)生殖相关基因的克隆及其表达模式研究. 苏州: 苏州大学硕士学位论文, 37-44
|
王志伟, 2017. 对虾VEGF信号通路在WSSV感染过程中的功能研究. 北京: 中国科学院大学博士学位论文, 77-79
|
孙瑜霞, 2014. 卤虫休眠胚胎形成过程中相关基因分子克隆及功能研究. 杭州: 浙江大学硕士学位论文, 2-3
|
段如冰, 2014. 卤虫两种p90 RSK在调节减数分裂及有丝分裂停滞中功能的研究. 杭州: 浙江大学博士学位论文, 39-44
|
廖珍, 2009. 家蚕和蜜蜂中Piwi蛋白基因的克隆与表达谱分析. 南京: 南京农业大学硕士学位论文, 28-63
|
Chen D F, Lin C, Wang H L et al, 2016. An La-related protein controls cell cycle arrest by nuclear retrograde transport of tRNAs during diapause formation in Artemia. BioMed Central Biology, 14(1): 16 |
Copf T, Schröder R, Averof M, 2004. Ancestral role of caudal genes in axis elongation and segmentation. Proceedings of the National Academy of Sciences of the United States of America, 101(51): 17711-17715 DOI:10.1073/pnas.0407327102 |
Cox D N, Chao A, Baker J et al, 1998. A novel class of evolutionarily conserved genes defined by piwi are essential for stem cell self-renewal. Genes & Development, 12(23): 3715-3727 |
Czech B, Hannon G J, 2016. One loop to rule them all: the Ping-Pong cycle and piRNA-guided silencing. Trends in Biochemical Sciences, 41(4): 324-337 DOI:10.1016/j.tibs.2015.12.008 |
Dai Z M, Li R, Dai L et al, 2011. Determination in oocytes of the reproductive modes for the brine shrimp Artemia parthenogenetica. Bioscience Reports, 31(1): 17-30 DOI:10.1042/BSR20090141 |
Dattilo A M, Bracchini L, Carlini L et al, 2005. Estimate of the effects of ultraviolet radiation on the mortality of Artemia franciscana in naupliar and adult stages. International Journal of Biometeorology, 49(6): 388-395 DOI:10.1007/s00484-005-0255-5 |
Dung N V, Christiaens O, Le Van Bao D et al, 2019. Identification of RNAi-related genes and transgenerational efficiency of RNAi in Artemia franciscana. Aquaculture, 501: 285-292 DOI:10.1016/j.aquaculture.2018.09.042 |
Halajzadeh J, Dana P M, Asemi Z et al, 2020. An insight into the roles of piRNAs and PIWI proteins in the diagnosis and pathogenesis of oral, esophageal, and gastric cancer. Pathology Research and Practice, 216(10): 153112 DOI:10.1016/j.prp.2020.153112 |
Hameed M S, Wang Z B, Vasseur L et al, 2018. Molecular characterization and the function of Argonaute3 in RNAi pathway of Plutella xylostella. International Journal of Molecular Sciences, 19(4): 1249 DOI:10.3390/ijms19041249 |
Höck J, Meister G, 2008. The Argonaute protein family. Genome Biology, 9(2): 210 DOI:10.1186/gb-2008-9-2-210 |
Houwing S, Berezikov E, Ketting R F, 2008. Zili is required for germ cell differentiation and meiosis in zebrafish. European Molecular Biology Organization Journal, 27(20): 2702-2711 DOI:10.1038/emboj.2008.204 |
Houwing S, Kamminga L M, Berezikov E et al, 2007. A role for Piwi and piRNAs in germ cell maintenance and transposon silencing in Zebrafish. Cell, 129(1): 69-82 DOI:10.1016/j.cell.2007.03.026 |
Huang X A, Yin H, Sweeney S et al, 2013. A major epigenetic programming mechanism guided by piRNAs. Developmental Cell, 24(5): 502-516 DOI:10.1016/j.devcel.2013.01.023 |
Ishizu H, Siomi H, Siomi M C, 2012. Biology of PIWI-interacting RNAs: new insights into biogenesis and function inside and outside of germlines. Genes & Development, 26(21): 2361-2373 |
Kawaoka S, Hayashi N, Suzuki Y et al, 2009. The Bombyx ovary-derived cell line endogenously expresses PIWI/PIWI-interacting RNA complexes. RNA, 15(7): 1258-1264 DOI:10.1261/rna.1452209 |
Kim T H, Yun T W, Rengaraj D et al, 2012. Conserved functional characteristics of the PIWI family members in chicken germ cell lineage. Theriogenology, 78(9): 1948-1959 DOI:10.1016/j.theriogenology.2012.07.019 |
King A M, MacRae T H, 2012. The small heat shock protein p26 aids development of encysting Artemia embryos, prevents spontaneous diapause termination and protects against stress. PLoS One, 7(8): e43723 DOI:10.1371/journal.pone.0043723 |
Klattenhoff C, Theurkauf W, 2008. Biogenesis and germline functions of piRNAs. Development, 135(1): 3-9 DOI:10.1242/dev.006486 |
Kwon C, Tak H, Rho M et al, 2014. Detection of PIWI and piRNAs in the mitochondria of mammalian cancer cells. Biochemical and Biophysical Research Communications, 446(1): 218-223 DOI:10.1016/j.bbrc.2014.02.112 |
Li C J, Vagin V V, Lee S et al, 2009. Collapse of germline piRNAs in the absence of Argonaute3 reveals somatic piRNAs in flies. Cell, 137(3): 509-521 DOI:10.1016/j.cell.2009.04.027 |
Li D R, Ye H L, Yang J S et al, 2017. Identification and characterization of a Masculinizer (Masc) gene involved in sex differentiation in Artemia. Gene, 614: 56-64 DOI:10.1016/j.gene.2017.03.010 |
Liu Y L, Zhao Y, Dai Z M et al, 2009. Formation of diapause cyst shell in brine shrimp, Artemia parthenogenetica, and its resistance role in environmental stresses. Journal of Biological Chemistry, 284(25): 16931-16938 DOI:10.1074/jbc.M109.004051 |
Lopes-dos-Santos R M A, Groot R, Sui L Y et al, 2019. Halophilic bacteria as a food source for the brine shrimp Artemia. Aquaculture, 500: 631-639 DOI:10.1016/j.aquaculture.2018.10.068 |
Manfra L, Savorelli F, Di Lorenzo B et al, 2015. Intercalibration of ecotoxicity testing protocols with Artemia franciscana. Ecological Indicators, 57: 41-47 DOI:10.1016/j.ecolind.2015.04.021 |
Marie P P, Ronsseray S, Boivin A, 2017. From embryo to adult: piRNA-mediated silencing throughout germline development in Drosophila. G3:Genes, Genomes, Genetics, 7(2): 505-516 |
Ozata D M, Gainetdinov I, Zoch A et al, 2019. PIWI-interacting RNAs: small RNAs with big functions. Nature Reviews Genetics, 20(2): 89-108 DOI:10.1038/s41576-018-0073-3 |
Ramat A, Simonelig M, 2021. Functions of PIWI proteins in gene regulation: new arrows added to the piRNA quiver. Trends in Genetics, 37(2): 188-200 DOI:10.1016/j.tig.2020.08.011 |
Sheu-Gruttadauria J, MacRae I J, 2017. Structural foundations of RNA silencing by Argonaute. Journal of Molecular Biology, 429(17): 2619-2639 DOI:10.1016/j.jmb.2017.07.018 |
Shoji K, Suzuki Y, Sugano S et al, 2017. Artificial "Ping-Pong" cascade of PIWI-interacting RNA in silkworm cells. RNA, 23(1): 86-97 DOI:10.1261/rna.058875.116 |
Sukthaworn S, Panyim S, Udomkit A, 2019. Functional characterization of a cDNA encoding Piwi protein in Penaeus monodon and its potential roles in controlling transposon expression and spermatogenesis. Comparative Biochemistry and Physiology, Part A: Molecular & Integrative Physiology, 229: 60-68 |
Sukthaworn S, Panyim S, Udomkit A, 2020. Homologues of Piwi control transposable elements and development of male germline in Penaeus monodon. Comparative Biochemistry and Physiology, Part A: Molecular & Integrative Physiology, 250: 110807 |
Tamtaji O R, Behnam M, Pourattar M A et al, 2020. PIWI-interacting RNAs and PIWI proteins in glioma: molecular pathogenesis and role as biomarkers. Cell Communication and Signaling, 18(1): 168 DOI:10.1186/s12964-020-00657-z |
Tóth K F, Pezic D, Stuwe E et al, 2016. The piRNA pathway guards the germline genome against transposable elements. Non-coding RNA and the Reproductive System, Advances in Experimental Medicine and Biology, 886: 51-77 |
Wang G L, Reinke V, 2008. A C. elegans Piwi, PRG-1, regulates 21U-RNAs during spermatogenesis. Current Biology, 18(12): 861-867 DOI:10.1016/j.cub.2008.05.009 |
Xiang D F, Zhu J Q, Hou C C et al, 2014. Identification and expression pattern analysis of Piwi genes during the spermiogenesis of Portunus trituberculatus. Gene, 534(2): 240-248 DOI:10.1016/j.gene.2013.10.050 |
Xu M G, You Y, Hunsicker P et al, 2008. Mice deficient for a small cluster of Piwi-interacting RNAs implicate Piwi-interacting RNAs in transposon control. Biology of Reproduction, 79(1): 51-57 DOI:10.1095/biolreprod.108.068072 |
Zhou X, Battistoni G, El Demerdash O et al, 2015. Dual functions of Macpiwi1 in transposon silencing and stem cell maintenance in the flatworm Macrostomum lignano. RNA, 21(11): 1885-1897 DOI:10.1261/rna.052456.115 |
 2021, Vol. 52
2021, Vol. 52


