 )(Seriola dumerili)线粒体全基因组测定及结构特征分析
)(Seriola dumerili)线粒体全基因组测定及结构特征分析

中国海洋湖沼学会主办。
文章信息
- 王开杰, 徐永江, 崔爱君, 柳学周, 姜燕, 王滨. 2022.
- WANG Kai-Jie, XU Yong-Jiang, CUI Ai-Jun, LIU Xue-Zhou, JIANG Yan, WANG Bin. 2022.
- 高体(
 )(Seriola dumerili)线粒体全基因组测定及结构特征分析
)(Seriola dumerili)线粒体全基因组测定及结构特征分析 - COMPLETE SEQUENCE AND GENE ORGANIZATION OF THE MITOCHONDRIAL GENOME OF SERIOLA DUMERILI
- 海洋与湖沼, 53(1): 120-132
- Oceanologia et Limnologia Sinica, 53(1): 120-132.
- http://dx.doi.org/10.11693/hyhz20210700160
文章历史
-
收稿日期:2021-07-09
收修改稿日期:2021-08-27
 )(Seriola dumerili)线粒体全基因组测定及结构特征分析
)(Seriola dumerili)线粒体全基因组测定及结构特征分析
2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东青岛 266237;
3. 浙江海洋大学国家海洋设施养殖工程技术研究中心 浙江舟山 316022
 )属鱼类物种、加强(
)属鱼类物种、加强(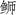 )鱼遗传多样性管理与种质资源保护,通过Illumina测序技术,获得了东海海域养殖高体(
)鱼遗传多样性管理与种质资源保护,通过Illumina测序技术,获得了东海海域养殖高体(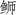 )(Seriola dumerili)线粒体基因组全序列(16 530 bp),碱基组成为A(26.83%)、G(17.6%)、C(30.04%)和T(25.53%),A+T含量为52.36%,且非编码控制区(D-loop)A+T富含61.64%,表现明显的AT偏好性。与其他硬骨鱼一样,高体(
)(Seriola dumerili)线粒体基因组全序列(16 530 bp),碱基组成为A(26.83%)、G(17.6%)、C(30.04%)和T(25.53%),A+T含量为52.36%,且非编码控制区(D-loop)A+T富含61.64%,表现明显的AT偏好性。与其他硬骨鱼一样,高体(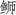 )线粒体基因组包含13条蛋白编码基因,22个tRNA基因,2个rRNA基因,除ND6、tRNAGln、tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer、tRNAGlu、tRNAPro基因外,其余均位于H链编码;蛋白编码基因中,除COⅠ、COⅡ和ND5的起始密码子分别为ATC、ATA和ATA外,其余10个蛋白编码基因的起始密码子均为ATG,以典型的TAA和TAG为终止密码子,在ND4和Cytb中存在不完全密码子T;除tRNASer-GCT外,其余21个tRNA均为典型三叶草二级结构。比较中国和日本海域高体(
)线粒体基因组包含13条蛋白编码基因,22个tRNA基因,2个rRNA基因,除ND6、tRNAGln、tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer、tRNAGlu、tRNAPro基因外,其余均位于H链编码;蛋白编码基因中,除COⅠ、COⅡ和ND5的起始密码子分别为ATC、ATA和ATA外,其余10个蛋白编码基因的起始密码子均为ATG,以典型的TAA和TAG为终止密码子,在ND4和Cytb中存在不完全密码子T;除tRNASer-GCT外,其余21个tRNA均为典型三叶草二级结构。比较中国和日本海域高体(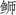 )线粒体基因组发现,CO I、CO II和ND5蛋白编码基因在起止位置、片段长度及起止密码子上存在显著差异。此外,与同属的黄条(
)线粒体基因组发现,CO I、CO II和ND5蛋白编码基因在起止位置、片段长度及起止密码子上存在显著差异。此外,与同属的黄条( )(Seriola aureovittata)和五条(
)(Seriola aureovittata)和五条( )(Seriola quinqueradiata)的线粒体基因组13个蛋白编码基因进行两两对比分析,结果表明3种(
)(Seriola quinqueradiata)的线粒体基因组13个蛋白编码基因进行两两对比分析,结果表明3种( )属鱼类的蛋白编码基因的相似性在85%~100%之间。基于线粒体基因组全序列构建的系统发育树,成功将高体(
)属鱼类的蛋白编码基因的相似性在85%~100%之间。基于线粒体基因组全序列构建的系统发育树,成功将高体( )与其他(
)与其他( )属鱼类有效区分,高体(
)属鱼类有效区分,高体(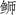 )与长鳍(
)与长鳍(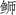 )同属一支,亲缘关系最近;黄条(
)同属一支,亲缘关系最近;黄条(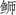 )和五条(
)和五条(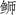 )聚为一支,亲缘关系最近。
)聚为一支,亲缘关系最近。 ) 线粒体基因组 序列比较分析 系统发育分析
) 线粒体基因组 序列比较分析 系统发育分析 2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China;
3. National Engineering Research Center For Marine Aquaculture, Zhejiang Ocean University, Zhoushan 316022, China
线粒体DNA (Mitochondria DNA)具有结构简单、高拷贝数, 分子量小、偏母性遗传且进化速度快等优点(Curole et al, 1999)。鱼类线粒体DNA大小一般为15~20 kb, 多为双链闭合的环状结构, 排列紧密, 不同物种间线粒体基因组存在很大差异, 序列中存在串联重复序列以及少数的散在重复序列(周传江等, 2019), 与其他高等动物相似, 鱼类线粒体基因组也是由13个蛋白质编码基因, 2个rRNA, 22个tRNA, 以及非编码区重链复制起始相关的控制区(D-loop)和轻链复制起始区(OL)组成(Satoh et al, 2016)。随着DNA测序技术的逐渐成熟, 鱼类线粒体基因组已被作为分子标记广泛应用于鱼类种质资源保护, 群体多态性以及系统进化发育等领域(陈四海等, 2011)。黄小林等(2018)探讨了线粒体基因组在6种篮子鱼(Siganus)系统发育中的适用性; 程佩琳等(2021)通过线粒体全基因组对6种鲟形目鱼类(Acipenseriformes)进行了种群划分; Bernal-Ramírez等(2003)利用线粒体基因组控制区分析了新西兰金赤鲷(Pagrus auratus)群体遗传结构的稳定性。
高体
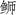
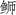
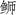
本研究测定了东海海域养殖高体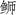

试验髙体
委托青岛欧易生物科技有限公司进行测序, 流程如下: 将提取的DNA样品参照TruSeqTM Nano DNA Sample Prep Kit试剂盒方法构建文库, 采用Covaris M220超声波破碎仪分割成长度为300~500 bp左右的片段并在两端加接头, 进行桥式PCR扩增, 采用Illumina NovaSeq 6000测序技术对样品DNA进行paired-end测序。
1.3 序列组装与分析使用SPAdes v3.14.1 (http://bioinf.spbau.ru/spades)软件对Clean Data进行校对、排列、拼接后获得高体 的线粒体基因组序列。通过Trimmomatic v0.39 (http://www.usadellab.org/cms/index.php?page=trimmomati)软件对其修剪后, 使用MITOS (http://mitos.bioinf.uni-leipzig.de/index.py) 软件对线粒体基因组预测编码蛋白、tRNA和rRNA基因, 然后对MITOS预测的初始基因去冗余, 并人工校正基因的起始、终止密码子位置, 获得高准确性的保守基因集。利用tRNA scan-SE在线软件(http://www.Genetics.Wustl.Edu./eddy/tRNAscan-SE)进行tRNA定位与RNAstructure预测, 利用软件CGView (http://stothard.afns.ualberta.ca/cgview_server/)对样品基因组进行圈图展示, 最后将蛋白编码基因通过Blast比对, 进行NR、Swiss-Prot、eggNOG、KEGG、GO注释分析。
的线粒体基因组序列。通过Trimmomatic v0.39 (http://www.usadellab.org/cms/index.php?page=trimmomati)软件对其修剪后, 使用MITOS (http://mitos.bioinf.uni-leipzig.de/index.py) 软件对线粒体基因组预测编码蛋白、tRNA和rRNA基因, 然后对MITOS预测的初始基因去冗余, 并人工校正基因的起始、终止密码子位置, 获得高准确性的保守基因集。利用tRNA scan-SE在线软件(http://www.Genetics.Wustl.Edu./eddy/tRNAscan-SE)进行tRNA定位与RNAstructure预测, 利用软件CGView (http://stothard.afns.ualberta.ca/cgview_server/)对样品基因组进行圈图展示, 最后将蛋白编码基因通过Blast比对, 进行NR、Swiss-Prot、eggNOG、KEGG、GO注释分析。
通过MEGA 7.0软件对高体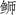
| 物种名 | 拉丁文 | GenBank登录号 | 总长/bp |
| 拉巴若鲹 | Carangoides malabaricus | KJ174514 | 16 561 |
| 甲若鲹 | Carangoides armatus | AP004444 | 16 556 |
| 游鳍叶鲹 | Atule mate | KM522838 | 16 565 |
| 丽叶鲹 | Alepes kleinii | KF728081 | 16 571 |
| 大甲鲹 | Megalaspis cordyl | KM522836 | 16 566 |
| 金带细鲹 | Selaroides leptolepis | KM522839 | 16 560 |
| 乌鲹 | Parastromateus niger | KJ192332 | 16 561 |
| 高体若鲹 | Carangoides equula | KM201334 | 16 588 |
| 竹荚鱼 | Trachurus trachurus | AB108498 | 16 559 |
| 白舌尾甲鲹 | Uraspis secunda | KT819204 | 16 554 |
| 长身圆鲹 | Decapterus macrosoma | KF841444 | 16 545 |
| 脂眼凹肩鲹 | Selar crumenophthalmus | KJ148633 | 16 610 |
| 小甘鲹 | Seriolina nigrofasciata | KT591876 | 16 531 |
日本黄条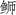 |
Seriola lalandi | AB517557 | 16 532 |
中国黄条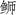 |
Seriola aureovittata | MH211123 | 16 609 |
五条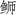 |
Seriola quinqueradiata | AB517556 | 16 537 |
日本高体 |
Seriola dumerili | AB517558 | 16 530 |
长鳍 |
Seriola rivoliana | KP347126 | 16 530 |
| 纺缍鲹 | Elagatis bipinnulata | KT824759 | 16 542 |
| 日本花鲈 | Lateolabrax japonicus | AP006789 | 16 593 |
本研究获得的髙体
高体
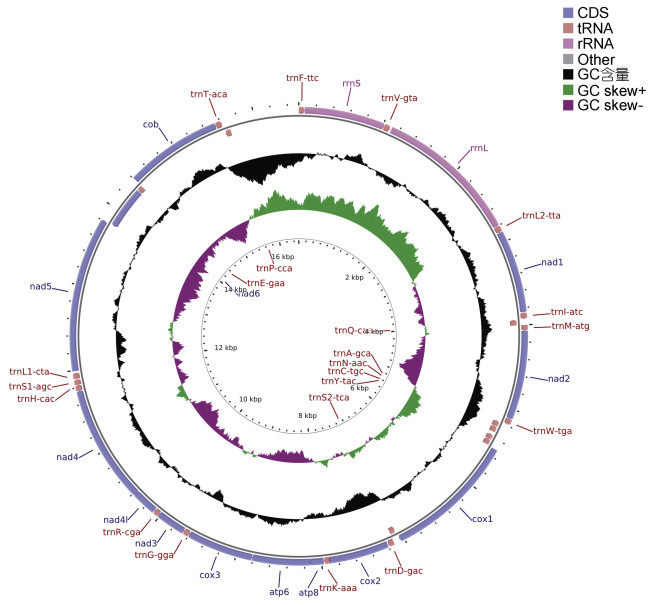 |
图 1 高体 |
| 基因名称 | 起止位置 | 长度/bp | 编码链 | 间隔长度 | 起始密码子 | 终止密码子 |
| tRNAPhe | 1~68 | 68 | H | — | — | — |
| 12S rRNA | 69~1 018 | 950 | H | 0 | — | — |
| tRNAVal | 1 019~1 090 | 72 | H | 0 | — | — |
| 16S rRNA | 1 109~2 808 | 1 700 | H | 18 | — | — |
| tRNALeu | 2 809~2 883 | 75 | H | 0 | — | — |
| ND1 | 2 884~3 858 | 975 | H | 0 | ATG | TAA |
| tRNAIle | 3 863~3 932 | 70 | H | 4 | — | — |
| tRNAGln | 3 932~4 002 | 71 | L | –1 | — | — |
| tRNAMet | 4 002~4 070 | 69 | H | –1 | — | — |
| ND2 | 4 071~5 117 | 1 047 | H | 0 | ATG | TAA |
| tRNATrp | 5 117~5 187 | 71 | H | –1 | — | — |
| tRNAAla | 5 189~5 257 | 69 | L | 1 | — | — |
| tRNAAsn | 5 259~5 331 | 73 | L | 1 | — | — |
| tRNACys | 5 366~5 431 | 66 | L | 34 | — | — |
| tRNATyr | 5 432~5 501 | 70 | L | 0 | — | — |
| CO I | 5 509~7 053 | 1 545 | H | 7 | ATC | TAA |
| tRNASer | 7 054~7 124 | 71 | L | 0 | — | — |
| tRNAAsp | 7 129~7 199 | 71 | H | 4 | — | — |
| CO II | 7 205~7 912 | 708 | H | 5 | ATA | TAA |
| tRNALys | 7 899~7 973 | 75 | H | –14 | — | — |
| ATP8 | 7 975~8 142 | 168 | H | 1 | ATG | TAA |
| ATP6 | 8 133~8 816 | 684 | H | –10 | ATG | TAA |
| CO III | 8 816~9 601 | 786 | H | –1 | ATG | TAA |
| tRNAGly | 9 601~9 671 | 71 | H | –1 | — | — |
| ND3 | 9 672~10 022 | 351 | H | 0 | ATG | TAG |
| tRNAArg | 10 021~10 089 | 69 | H | –2 | — | — |
| ND4L | 10 090~10 386 | 297 | H | 0 | ATG | TAA |
| ND4 | 10 380~11 760 | 1 381 | H | –7 | ATG | T— |
| tRNAHis | 11 761~11 829 | 69 | H | 0 | — | — |
| tRNASer | 11 830~11 896 | 67 | H | 0 | — | — |
| tRNALeu | 11 901~11 973 | 73 | H | 4 | — | — |
| ND5 | 11 992~13 812 | 1 821 | H | 18 | ATA | TAA |
| ND6 | 13 809~14 330 | 522 | L | –4 | ATG | TAG |
| tRNAGlu | 14 332~14 400 | 69 | L | 1 | — | — |
| Cytb | 14 406~15 546 | 1 141 | H | 5 | ATG | T-- |
| tRNAThr | 15 547~15 618 | 72 | H | 0 | — | — |
| tRNAPro | 15 618~15 688 | 71 | L | –1 | — | — |
| D-loop | 15 689~16 530 | 842 | H | 0 | — | — |
22个tRNA的序列总长度为1 552 bp, 长度范围为66~75 bp, 长度最短的为tRNACys, 最长的为tRNALeu和tRNALys。其中tRNASer和tRNALeu基因各有两个, 除tRNAGln、tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer、tRNAGlu、tRNAPro位于L链外, 其余14个tRNA皆位于H链上。22个tRNA结构均可通过tRNAscan-SE在线预测, 除tRNASer-GCT基因缺失二氢尿嘧啶臂(DHU臂)外, 其余21个tRNA均含有典型的三叶草二级结构(图 2)。
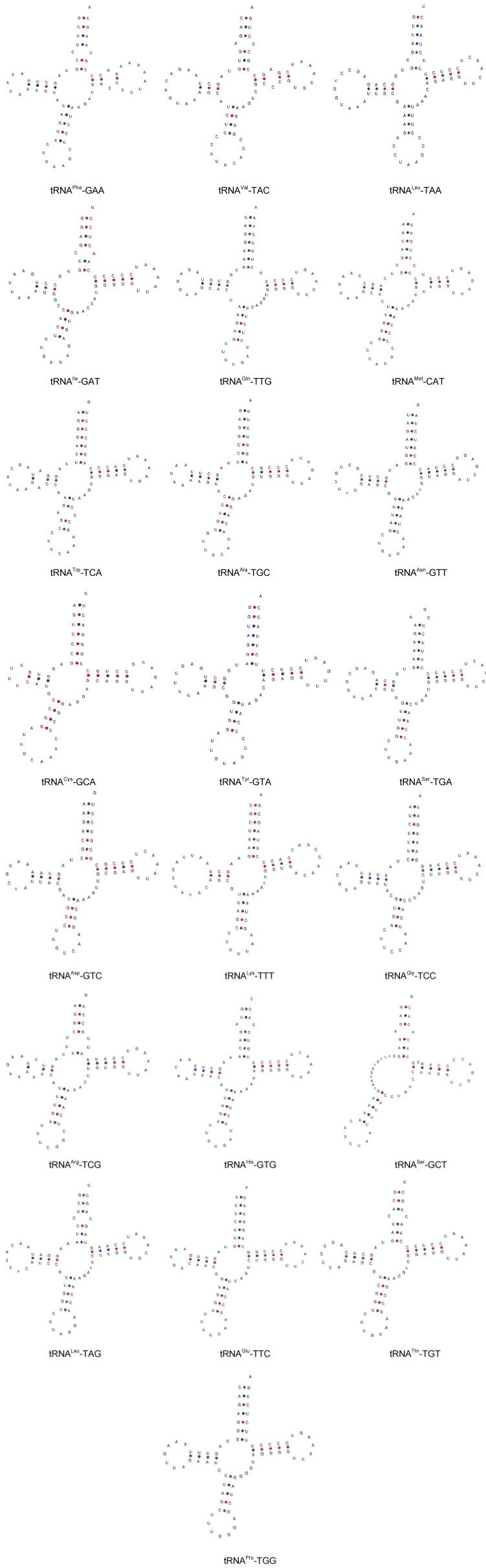 |
图 2 高体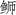 |
在氨基酸臂中由于C-T转换造成的tRNAVal、tRNAIle和tRNAHis中A-C不配对, 还导致tRNAGln、tRNAAla、tRNATyr和tRNAPro基因中G-T发生错配。且在氨基酸臂中tRNASer-GCT存在1对A-A不配对, tRNAPhe、tRNALeu、tRNAThr存在1对C-C不配对, tRNAArg存在2对T-T不配对。在反密码子茎中也存在由于C-T转换造成的tRNATrp中A-C不配对。在TΨC茎上, 同样也存在C-T转换现象, 除此之外, tRNAMet有1对U-U不配对。DHU环也存在G-T错配现象, tRNATrp有1对A-A不配对, 且tRNASer-GCT基因该环缺失。
高体
本研究对高体
| 密码子 | 出现次数 | RSCU | 密码子 | 出现次数 | RSCU | 密码子 | 出现次数 | RSCU | 密码子 | 出现次数 | RSCU | |||
| UUU(F) | 88 | 0.8 | UCU(S) | 32 | 0.7 | UAU(Y) | 29 | 0.55 | UGU(C) | 7 | 0.4 | |||
| UUC(F) | 154 | 1.2 | UCC(S) | 73 | 2.21 | UAC(Y) | 81 | 1.3 | UGC(C) | 17 | 0.99 | |||
| UUA(L) | 79 | 0.77 | UCA(S) | 50 | 1.11 | UAA(*) | 9 | 0.47 | UGA(*) | 100 | 2.44 | |||
| UUG(L) | 25 | 0.28 | UCG(S) | 25 | 0.62 | UAG(*) | 2 | 0.08 | UGG(W) | 20 | 0.85 | |||
| CUU(L) | 148 | 1.41 | CCU(P) | 59 | 1.3 | CAU(H) | 25 | 0.72 | CGU(R) | 8 | 0.55 | |||
| CUC(L) | 174 | 1.51 | CCC(P) | 103 | 1.62 | CAC(H) | 82 | 1.28 | CGC(R) | 13 | 1.13 | |||
| CUA(L) | 169 | 1.32 | CCA(P) | 43 | 0.78 | CAA(Q) | 77 | 1.55 | CGA(R) | 46 | 3.01 | |||
| CUG(L) | 65 | 0.72 | CCG(P) | 19 | 0.3 | CAG(Q) | 17 | 0.3 | CGG(R) | 12 | 0.79 | |||
| AUU(I) | 127 | 1.11 | ACU(T) | 53 | 0.91 | AAU(N) | 28 | 0.59 | AGU(S) | 9 | 0.22 | |||
| AUC(I) | 124 | 1.1 | ACC(T) | 105 | 1.27 | AAC(N) | 90 | 1.41 | AGC(S) | 43 | 1.15 | |||
| AUA(I) | 77 | 0.79 | ACA(T) | 115 | 1.39 | AAA(K) | 61 | 1.28 | AGA(R) | 1 | 0.07 | |||
| AUG(M) | 75 | 1 | ACG(T) | 23 | 0.43 | AAG(K) | 15 | 0.41 | AGG(R) | 0 | 0 | |||
| GUU(V) | 69 | 1.18 | GCU(A) | 62 | 0.71 | GAU(D) | 17 | 0.59 | GGU(G) | 34 | 0.5 | |||
| GUC(V) | 83 | 1.45 | GCC(A) | 166 | 1.79 | GAC(D) | 62 | 1.26 | GGC(G) | 80 | 1.16 | |||
| GUA(V) | 56 | 0.94 | GCA(A) | 115 | 1.3 | GAA(E) | 71 | 1.46 | GGA(G) | 73 | 1.09 | |||
| GUG(V) | 25 | 0.44 | GCG(A) | 17 | 0.21 | GAG(E) | 29 | 0.54 | GGG(G) | 52 | 1.05 | |||
| 注: *表示终止密码子; 括号内的字母表示各氨基酸名称的缩写; 表中加粗字体为氨基酸偏好密码子 | ||||||||||||||

本研究发现东海海域与日本海域高体

| 基因 | 中国海域高体 |
日本海域高体 |
|||||||
| 起止位置(bp) | 长度(bp) | 起始密码 | 终止密码 | 起止位置(bp) | 长度(bp) | 起始密码 | 终止密码 | ||
| ND1 | 2 884~3 858 | 975 | ATG | TAA | 2 884~3 858 | 975 | ATG | TAA | |
| ND2 | 4 071~5 117 | 1 047 | ATG | TAA | 4 071~5 117 | 1 047 | ATG | TAA | |
| CO I | 5 509~7 053 | 1 545 | ATC | TAA | 5 503~7 053 | 1 551 | GTG | TAA | |
| CO II | 7 205~7 912 | 708 | ATA | TAA | 7 208~7 898 | 691 | ATG | T-- | |
| ATP8 | 7 975~8 142 | 168 | ATG | TAA | 7 975~8 142 | 168 | ATG | TAA | |
| ATP6 | 8 133~8 816 | 684 | ATG | TAA | 8 133~8 816 | 684 | ATG | TAA | |
| CO III | 8 816~9 601 | 786 | ATG | TAA | 8 816~9 601 | 786 | ATG | TAA | |
| ND3 | 9 672~10 022 | 351 | ATG | TAG | 9 672~10 022 | 351 | ATG | TAG | |
| ND4L | 10 090~10 386 | 297 | ATG | TAA | 10 090~10 386 | 297 | ATG | TAA | |
| ND4 | 10 380~11 760 | 1 381 | ATG | T-- | 10 380~11 760 | 1 381 | ATG | T-- | |
| ND5 | 11 992~13 812 | 1 821 | ATA | TAA | 11 974~13 812 | 1 839 | ATG | TAA | |
| ND6 | 13 809~14 330 | 522 | ATG | TAG | 13 809~14 330 | 522 | ATG | TAG | |
| Cytb | 14 406~15 546 | 1 141 | ATG | T-- | 14 406~15 546 | 1 141 | ATG | T-- | |
比较分析了三种


| 基因 | 黄条 |
高体 |
五条 |
密码子 | ||||||||||
| 起始 | 终止 | 长度/bp | 起始 | 终止 | 长度/bp | 起始 | 终止 | 长度/bp | 起始 | 终止 | ||||
| tRNAPhe | 1 | 68 | 68 | 1 | 68 | 68 | 1 | 68 | 68 | |||||
| 12S rRNA | 69 | 1 019 | 951 | 69 | 1 018 | 950 | 69 | 1 023 | 955 | |||||
| tRNAVal | 1 020 | 1 092 | 72 | 1 019 | 1 090 | 72 | 1 024 | 1 095 | 72 | |||||
| 16S rRNA | 1 110 | 2 808 | 1 699 | 1 109 | 2 808 | 1 700 | 1 096 | 2 813 | 1 718 | |||||
| tRNALeu(UUR) | 2 809 | 2 883 | 75 | 2 809 | 2 883 | 75 | 2 814 | 2 888 | 75 | |||||
| ND1 | 2 884 | 3 858 | 975 | 2 884 | 3 858 | 975 | 2 889 | 3 863 | 975 | ATG | TAA | |||
| tRNAIle | 3 863 | 3 932 | 70 | 3 863 | 3 932 | 70 | 3 868 | 3 937 | 70 | |||||
| tRNAGln(L) | 3 932 | 4 002 | 71 | 3 932 | 4 002 | 71 | 3 937 | 4 007 | 71 | |||||
| tRNAMet | 4 002 | 4 070 | 69 | 4 002 | 4 070 | 69 | 4 007 | 4 075 | 69 | |||||
| ND2 | 4 071 | 5 117 | 1 047 | 4 071 | 5 117 | 1 047 | 4 076 | 5 122 | 1 047 | ATG | TAA | |||
| tRNATrp | 5 117 | 5 187 | 71 | 5 117 | 5 187 | 71 | 5 122 | 5 192 | 71 | |||||
| tRNAAla(L) | 5 189 | 5 257 | 69 | 5 189 | 5 257 | 69 | 5 194 | 5 262 | 69 | |||||
| tRNAAsn(L) | 5 259 | 5 331 | 73 | 5 259 | 5 331 | 73 | 5 264 | 5 336 | 73 | |||||
| O L | 5 332 | 5 365 | 33 | 5 332 | 5 365 | 33 | 5 337 | 5 370 | 34 | |||||
| tRNACys(L) | 5 366 | 5 431 | 66 | 5 366 | 5 431 | 66 | 5 371 | 5 436 | 66 | |||||
| tRNATyr(L) | 5 432 | 5 501 | 70 | 5 432 | 5 501 | 70 | 5 437 | 5 506 | 70 | |||||
| CO I | 5 584 | 7 053 | 1 470 | 5 509 | 7 053 | 1 545 | 5 508 | 7 058 | 1 551 | ATG/ATC/GTG | TAA | |||
| tRNASer (UCN)(L) | 7 054 | 7 124 | 71 | 7 054 | 7 124 | 71 | 7 059 | 7 129 | 71 | |||||
| tRNAAsp | 7 129 | 7 199 | 71 | 7 129 | 7 199 | 71 | 7 134 | 7 204 | 71 | |||||
| CO II | 7 208 | 7 898 | 691 | 7 205 | 7 912 | 708 | 7 213 | 7 903 | 691 | ATG/ATA/ATG | T/TAA/T | |||
| tRNALys | 7 899 | 7 973 | 75 | 7 899 | 7 973 | 75 | 7 904 | 7 978 | 75 | |||||
| ATP8 | 7 975 | 8 142 | 168 | 7 975 | 8 142 | 168 | 7 980 | 8 147 | 168 | ATG | TAA | |||
| ATP6 | 8 133 | 8 816 | 684 | 8 133 | 8 816 | 684 | 8 138 | 8 821 | 684 | TAA | ||||
| CO III | 8 816 | 9 601 | 786 | 8 816 | 9 601 | 786 | 8 821 | 9 606 | 786 | ATG | TAA | |||
| tRNAGly | 9 601 | 9 671 | 71 | 9 601 | 9 671 | 71 | 9 606 | 9 676 | 71 | |||||
| ND3 | 9 672 | 10 022 | 351 | 9 672 | 10 022 | 351 | 9 677 | 10 027 | 351 | ATG | TAG | |||
| tRNAArg | 10 021 | 10 089 | 69 | 10 021 | 10 089 | 69 | 10 026 | 10 094 | 69 | |||||
| ND4L | 10 090 | 10 386 | 297 | 10 090 | 10 386 | 297 | 10 095 | 10 391 | 297 | ATG | TAA | |||
| ND4 | 10 380 | 11 760 | 1 381 | 10 380 | 11 760 | 1 381 | 10 385 | 11 765 | 1 381 | ATG | T | |||
| tRNAHis | 11 761 | 11 829 | 69 | 11 761 | 11 829 | 69 | 11 766 | 11 834 | 69 | |||||
| tRNASer(AGY) | 11 830 | 11 896 | 67 | 11 830 | 11 896 | 67 | 11 835 | 11 901 | 67 | |||||
| tRNALeu(CUN) | 11 901 | 11 973 | 73 | 11 901 | 11 973 | 73 | 11 906 | 11 978 | 73 | |||||
| ND5 | 11 992 | 13 812 | 1 821 | 11 992 | 13 812 | 1 821 | 11 979 | 13 817 | 1 839 | ATA/ATA/ATG | TAA | |||
| ND6 (L) | 13 809 | 14 330 | 522 | 13 809 | 14 330 | 522 | 13 814 | 14 335 | 522 | ATG | TAG | |||
| tRNAGlu(L) | 14 332 | 14 400 | 69 | 14 332 | 14 400 | 69 | 14 337 | 14 405 | 69 | |||||
| Cytb | 14 405 | 15 545 | 1 141 | 14 406 | 15 546 | 1 141 | 14 410 | 15 550 | 1 141 | ATG | T | |||
| tRNAThr | 15 546 | 15 617 | 72 | 15 547 | 15 618 | 72 | 15 551 | 15 622 | 72 | |||||
| tRNAPro(L) | 15 617 | 15 687 | 71 | 15 618 | 15 688 | 71 | 15 622 | 15 692 | 71 | |||||
| Control region | 15 689 | 16 609 | 921 | 15 689 | 16 530 | 842 | 15 693 | 16 537 | 845 | |||||
三种
| 种类 | 碱基 | ND1 | ND2 | CO I | CO II | ATP8 | ATP6 | CO III | ND3 | ND4L | ND4 | ND5 | ND6 | Cytb |
| A+T | 48.10 | 48.62 | 52.24 | 57.16 | 52.98 | 52.63 | 50.89 | 50.14 | 48.82 | 50.62 | 52.39 | 49.43 | 49.78 | |
黄条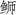 |
T | 26.36 | 24.83 | 28.30 | 28.08 | 23.81 | 27.63 | 26.97 | 29.63 | 30.64 | 24.55 | 26.03 | 36.02 | 26.73 |
| S. aureovittata | C | 33.44 | 36.77 | 28.84 | 26.63 | 32.74 | 32.46 | 31.04 | 32.48 | 35.02 | 32.95 | 32.24 | 16.86 | 34.36 |
| A | 21.74 | 23.78 | 23.95 | 29.09 | 29.17 | 25.00 | 23.92 | 20.51 | 18.18 | 26.07 | 26.36 | 13.41 | 23.05 | |
| G | 18.46 | 14.61 | 18.91 | 16.21 | 14.29 | 14.91 | 18.07 | 17.38 | 16.16 | 16.44 | 15.38 | 33.72 | 15.86 | |
| A+T | 49.95 | 50.15 | 52.16 | 56.77 | 51.78 | 51.61 | 51.40 | 49.29 | 49.50 | 51.48 | 52.83 | 51.72 | 51.00 | |
高体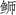 |
T | 27.18 | 25.60 | 28.41 | 27.82 | 22.02 | 27.78 | 27.23 | 27.35 | 30.64 | 25.63 | 26.03 | 36.97 | 26.99 |
| S. dumerili | C | 32.92 | 35.05 | 29.19 | 26.84 | 34.52 | 32.75 | 30.92 | 34.47 | 35.35 | 32.37 | 31.96 | 15.33 | 33.39 |
| A | 22.77 | 24.55 | 23.75 | 28.95 | 29.76 | 23.83 | 24.17 | 21.94 | 18.86 | 25.85 | 26.80 | 14.75 | 24.01 | |
| G | 17.13 | 14.80 | 18.64 | 16.38 | 13.69 | 15.64 | 17.68 | 16.24 | 15.15 | 16.15 | 15.21 | 32.95 | 15.60 | |
| A+T | 48.51 | 47.27 | 52.22 | 56.29 | 51.19 | 50.81 | 51.53 | 50.99 | 48.82 | 49.46 | 53.12 | 50.19 | 49.96 | |
五条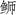 |
T | 26.77 | 24.16 | 28.69 | 27.35 | 23.21 | 27.01 | 27.10 | 29.34 | 30.64 | 24.48 | 26.26 | 13.41 | 26.73 |
| S. quinqueradiata | C | 32.72 | 37.15 | 28.24 | 27.35 | 33.33 | 33.43 | 30.92 | 32.76 | 34.68 | 33.02 | 31.65 | 33.91 | 33.83 |
| A | 21.74 | 23.11 | 23.53 | 28.94 | 27.98 | 23.80 | 24.43 | 21.65 | 18.18 | 24.98 | 26.86 | 36.78 | 23.23 | |
| G | 18.77 | 15.57 | 19.54 | 16.35 | 15.48 | 15.77 | 17.56 | 16.24 | 16.50 | 17.52 | 15.23 | 15.90 | 16.21 |
对三种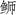
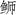
 |
图 3 三种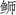  ; JSD: 日本高体 ; JSD: 日本高体 ; SA: 中国黄条 ; SA: 中国黄条 ; SL: 日本黄条 ; SL: 日本黄条 ; SQ: 五条 ; SQ: 五条 |
以日本花鲈为外类群, 构建了高体
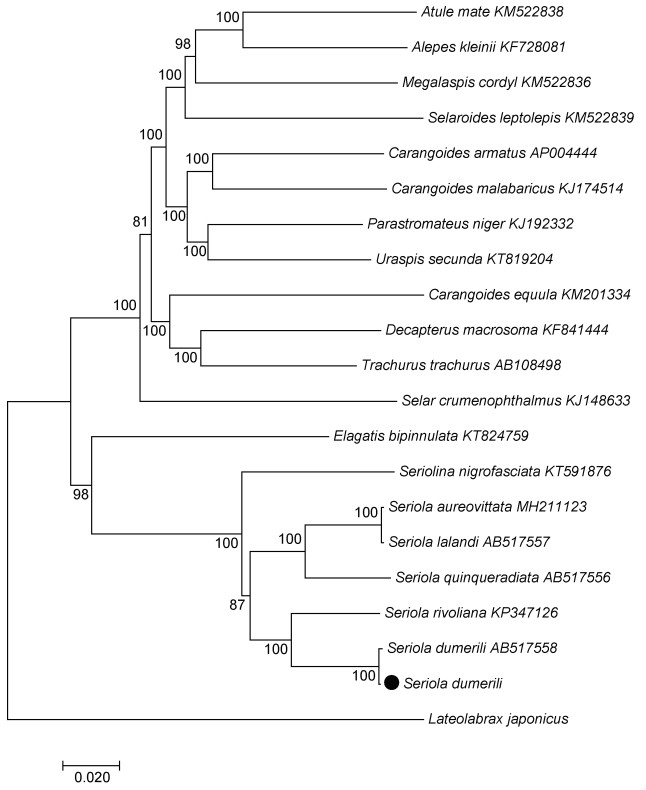 |
| 图 4 基于线粒体基因组全序列构建的NJ系统进化树 Fig. 4 Molecular phylogenetic tree constructed by NJ method based on mitochondrial genome |
本研究测定了高体

本研究通过对中国海域高体
此外, 本研究对三种
本研究基于线粒体基因组全序列通过邻接法构建了部分鲹科鱼类系统发育树, 

本研究基于线粒体基因组结构特征比较, 推测中国海域高体
马江茹, 2020. 西里伯斯青鳉线粒体基因组全序列和中华青鳉群体遗传结构的研究[D]. 湛江: 广东海洋大学: 29-30.
|
王尚洪, 2015. 鳑鲏亚科三种鱼类线粒体全基因组测定及其比较基因组学分析[D]. 南昌: 南昌大学: 54-76.
|
史宝, 柳学周, 刘永山, 等. 2019. 黄条  线粒体全基因组测序及结构特征分析. 中国水产科学, 26(3): 405-415 线粒体全基因组测序及结构特征分析. 中国水产科学, 26(3): 405-415 |
肖家光, 2015. 基于线粒体基因组全序列的鱚属鱼类系统发育研究[D]. 青岛: 中国海洋大学: 24-25.
|
张方, 米志勇. 1998. 动物线粒体DNA的分子生物学研究进展. 生物工程进展, 18(3): 25-31 |
陈大刚, 张美昭. 2015. 中国海洋鱼类. 青岛: 中国海洋大学出版社, 1096-1097
|
陈四海, 区又君, 李加儿. 2011. 鱼类线粒体DNA及其研究进展. 生物技术通报, (3): 13-20 |
周传江, 马爱喆, 汪曦, 等. 2019. 鱼类线粒体基因组研究进展. 河南师范大学学报(自然科学版), 47(2): 74-82 |
郑文娟, 朱世华, 邹记兴, 等. 2008. 基于16S rRNA部分序列探讨12种鲹科鱼类的分子系统进化关系. 水产学报, 32(6): 847-854 |
孟乾, 张志勇, 张志伟, 等. 2020. 斑石鲷和条石鲷线粒体基因组密码子使用分析. 水产科学, 39(5): 702-709 |
郭新红, 刘少军, 刘巧, 等. 2004. 鱼类线粒体DNA研究新进展. 遗传学报, 31(9): 983-1000 |
黄小林, 杨育凯, 林黑着, 等. 2018. 篮子鱼属线粒体基因组序列系统发育信息分析. 生物学杂志, 35(5): 33-36 DOI:10.3969/j.issn.2095-1736.2018.05.033 |
程佩琳, 俞丹, 刘焕章, 等. 2021. 基于线粒体基因组全序列的鲟形目鱼类(Pisces: Acipenseriformes)的分子系统发育重建. 水生生物学报, 45(3): 487-494 |
赖瑞芳, 张秀杰, 李艳和, 等. 2014. 鲂属鱼类线粒体基因组的比较及其系统发育分析. 水产学报, 38(1): 1-14 |
廖志强. 2003. 高体  网箱养殖技术. 中国水产, (12): 60-61 网箱养殖技术. 中国水产, (12): 60-61 |
BERNAL-RAMÍREZ J H, ADCOCK G J, HAUSER L et al, 2003. Temporal stability of genetic population structure in the New Zealand snapper, Pagrus auratus, and relationship to coastal currents. Marine Biology, 142(3): 567-574 DOI:10.1007/s00227-002-0972-9 |
BROUGHTON R E, MILAM J E, ROE B A, 2001. The complete sequence of the zebrafish (Danio rerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA. Genome Research, 11(11): 1958-1967 DOI:10.1101/gr.156801 |
CUROLE J P, KOCHER T D, 1999. Mitogenomics: digging deeper with complete mitochondrial genomes. Trends in Ecology & Evolution, 14(10): 394-398 |
IGUCHI J, TAKASHIMA Y, NAMIKOSHI A et al, 2012. Species identification method for marine products of Seriola and related species. Fisheries Science, 78(1): 197-206 |
NUGROHO E, TANIGUCHI N, KATO K et al, 2000. Genetic difference among seed populations of greater amberjack used in aquaculture farm of Japan. Aquaculture Science, 48(4): 665-674 |
PREMACHANDRA H K A, LAFARGA-DE LA CRUZ F, TAKEUCHI Y et al, 2017. Genomic DNA variation confirmed Seriola lalandi comprises three different populations in the Pacific, but with recent divergence. Scientific Reports, 7(1): 9386 |
SATOH T P, MIYA M, MABUCHI K et al, 2016. Structure and variation of the mitochondrial genome of fishes. BMC Genomics, 17(1): 719 |
ZARDOYA R, MEYER A, 1996. Phylogenetic performance of mitochondrial protein-coding genes in resolving relationships among vertebrates. Molecular Biology and Evolution, 13(7): 933-942 |
ZHU Y X, CHEN Y, CHENG Q Q et al, 2013. The complete mitochondrial genome sequence of Schizothorax macropogon (Cypriniformes: Cyprinidae). Mitochondrial DNA, 24(3): 237-239 |
 2022, Vol. 53
2022, Vol. 53


