
中国海洋湖沼学会主办。
文章信息
- 李迎娣, 孙志宾, 马爱军, 杨敬昆, 刘志峰, 于宏, 赵亭亭, 常浩文, 朱理光, 刘诗颖, 曲江波. 2022.
- LI Ying-Di, SUN Zhi-Bin, MA Ai-Jun, YANG Jing-Kun, LIU Zhi-Feng, YU Hong, ZHAO Ting-Ting, CHANG Hao-Wen, ZHU Li-Guang, LIU Shi-Ying, QU Jiang-Bo. 2022.
- 双锯鱼(Amphiprion)胚胎不同发育阶段和成鱼不同组织中内参基因的筛选及应用
- SCREENING AND APPLICATION OF REFERENCE GENE IN ANEMONE FISH (AMPHIPRION) EMBRYOS IN DIFFERENT DEVELOPMENT STAGES AND ADULT TISSUES
- 海洋与湖沼, 53(1): 150-160
- Oceanologia et Limnologia Sinica, 53(1): 150-160.
- http://dx.doi.org/10.11693/hyhz20210800196
文章历史
-
收稿日期:2021-08-31
收修改稿日期:2021-10-06
2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室 青岛市海水鱼类种子工程与生物技术重点实验室 山东青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室 山东青岛 266237;
4. 中国-东盟海水养殖技术"一带一路"联合实验室 广东广州 510275;
5. 烟台开发区天源水产有限公司 山东烟台 264003
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs; Qingdao Key Laboratory for Marine Fish Breeding and Biotechnology, Qingdao 266071, China;
3. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China;
4. China-ASEAN Belt and Road Joint Laboratory on Marine Aquaculture Technology, Guangzhou 510275, China;
5. Yantai Tianyuan Aquatic Co. Ltd., Yantai 264003, China
双锯鱼隶属于鲈形目(Perciformes)、雀鲷科(Pomacentridae)、双锯鱼属(Amphiprion) (白海锋等, 2011)。其与海葵共生, 为海葵去除坏死组织, 海葵为其提供保护, 亦被称为海葵鱼(叶乐等, 2008)。双锯鱼体表都有一条或两条白色条纹, 神似戏剧中的丑角, 因此俗称“小丑鱼”。双锯鱼色彩绚丽、性情温和, 且经济价值高, 是海水观赏鱼的首选品种(章霞等, 2015; 张薇等, 2018)。国内在其人工繁育和饲养、形态学观察、性别分化等方面取得了较多成就, 如滕力平等(2005)对二线小丑鱼(Amphiprion clarkii)的发育进行了生态学和形态学两方面的研究, 发现胚后仔、稚、幼鱼阶段的二线小丑鱼体表色素会逐渐增加并积累; Amonrat等(2013)用富含蛋白质、脂质和虾青素的饲料对眼斑双锯鱼进行投喂, 结果表明, 在一定范围内, 蛋白质含量较高的饲料更适合双锯鱼的生长, 而虾青素和脂质则有利于色素的积聚。但目前在分子水平上对双锯鱼体色相关基因表达模式的研究还比较匮乏。
TYR (酪氨酸酶, tyrosinase)基因参与生物体黑色素(melanin)的合成, 其表达量决定了生物的体色变化, 在鱼类的胚胎时期就已经开始表达(蒋燕玲, 2016)。通过实时定量PCR (quantitative real-time polymerase chain reaction, qPCR)技术对TYR的表达进行分析有助于了解基因的功能、阐明相关基因在胚胎发育和体色变异中的调控机制(杨爱馥等, 2010; Tang et al, 2017; 何丽斌等, 2018)。且生物体内mRNA水平的变化是生理变化的最早信号, 会因为处于不同的发育时期或实验条件的不同而表现出不同的浓度, 因此可以用来预测生物组织更高层次的变化(Franzellitti et al, 2015)。
内参基因(reference gene)又称管家基因(house-keeping gene), 其表达水平受环境因素影响较小, 在所有细胞中都能够相对稳定的表达, 不同实验条件下选用的内参基因有所不同(邓红平等, 2006; 刘颖等, 2013; Szczygieł et al, 2021)。常用的内参基因有18S rRNA基因、重组蛋白基因(rps29)、肌动蛋白基因(ACT)、甘油醛-3-磷酸脱氢酶基因(GAPDH)、转录延伸因子基因(Ef-1α)、多聚泛素酶基因(UBQ)、β-肌动蛋白基因(β-actin)、α-微管蛋白基因(TUA)和β-微管蛋白基因(TUB) (蒋素华等, 2018)。内参基因在qPCR技术分析基因表达的过程中非常关键, 其稳定表达是目标基因表达数据标准化的先决条件, 可以作为数据分析的参考标准(Pfaffl et al, 2004; Mitter et al, 2009; 吴萍等, 2017)。研究表明, 在同一物种中, 即使内参基因在某一条件下能够相对稳定的表达(Mughal et al, 2018), 但不一定在所有实验条件下都适用, 因此在qPCR之前需要筛选出合适的内参基因(Dheda et al, 2005)。若内参基因有较大的表达误差, 实验结果中存在的干扰就会增加, 从而限制双锯鱼分子生物学方面的研究(Iwata et al, 2008; 吕梁等, 2019)。
目前对双锯鱼内参基因筛选的研究较少, 为了进一步探索TYR基因的表达机制, 本实验以白条双锯鱼和眼斑双锯鱼的组织及不同发育阶段的胚胎作为样品, 综合利用Norm Finder和geNorm软件对GAPDH、18S rRNA、Ef-1α和β-actin这4个候选内参基因的稳定性进行分析, 并选出相对稳定的内参基因, 以TYR基因作为目的基因检测其表达规律, 为获得最优内参基因提供理论依据, 为双锯鱼相关基因的分析奠定应用基础。
1 材料与方法 1.1 实验材料供试胚胎、组织均取自黄海水产研究所实验室内经驯化后的野生白条双锯鱼(番茄小丑鱼) (Amphiprion frenatus)和眼斑双锯鱼(公子小丑鱼) (Amphiprion ocellaris) (图 1)。实验期间将双锯鱼成对饲养在养殖缸中, 即一对白条双锯鱼, 一对眼斑双锯鱼, 水温用加热棒维持在(27±1) ℃, 盐度为30~35, 光照周期为14L : 10D, 每天在8:00和14:00投喂人工饵料。白条双锯鱼通常在12:00~13:00产卵, 孵化期为8~9 d; 眼斑双锯鱼通常在15:00~16:00产卵, 孵化期为9~10 d。分别取这两种双锯鱼的6个胚胎发育时期(25个胚胎/时期, 三个重复): 2 hpf (hours post fertilization, 受精后小时数) (卵裂期)、33 hpf (体节期, 出现黑色素细胞, 图 2)、57 hpf (翻转期)、81 hpf (血管形成期)、105 hpf (器官形成期)、225 hpf (孵化期) (图 3, 图 4)。取这两种双锯鱼的成鱼组织(三月龄, 三个重复): 心脏、脑、脾脏、眼、肠、鳃、白皮、红皮、肌肉、尾(图 5, 以眼斑双锯鱼为例)。将取得的胚胎和组织样品迅速浸泡在RNA保护液中, 然后储存在–80 ℃冰箱中, 用于后续总RNA的提取。
 |
| 图 1 眼斑双锯鱼和白条双锯鱼外观 Fig. 1 Picture of Fales-Clown anemone fish (A. ocellaris) and tomato clownfish (A. frenatus) 注: a、c: 眼斑双锯鱼; b、d: 白条双锯鱼 |
 |
| 图 2 两种双锯鱼体节期胚胎的黑色素细胞 Fig. 2 Melanocytes of the somite embryos of two anemone fish (A. frenatus and A. ocellaris) 注: a. 白条双锯鱼; b. 眼斑双锯鱼 |
 |
| 图 3 白条双锯鱼胚胎不同发育时期 Fig. 3 Different stages of embryonic development of tomato clownfish (A. frenatus) 注: a. 卵裂期; b. 体节期; c. 翻转期; d. 血管形成期; e. 器官形成期; f. 孵化期 |
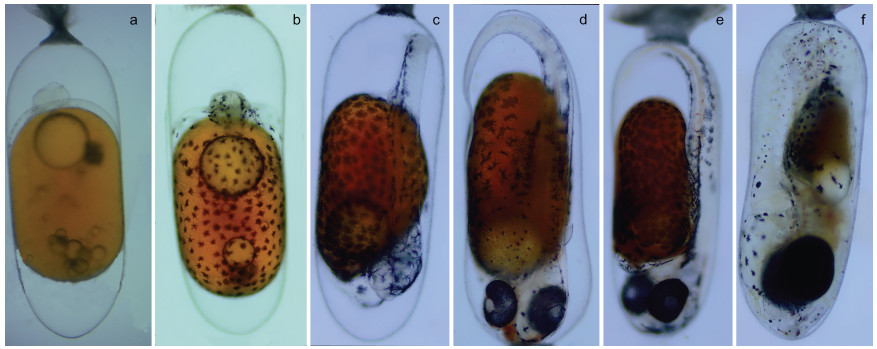 |
| 图 4 眼斑双锯鱼胚胎不同发育时期 Fig. 4 Different stages of embryonic development of Fales-Clown anemone fish (A. ocellaris) 注: a. 卵裂期; b. 体节期; c. 翻转期; d. 血管形成期; e. 器官形成期; f. 孵化期 |
 |
| 图 5 成鱼组织(以眼斑双锯鱼为例) Fig. 5 Different tissue of adult fish (taking A. ocellaris as an example) 注: a. 眼; b. 肌肉; c. 红皮; d. 尾; e. 鳃; f. 心; g. 肠; h. 白皮 |
根据总RNA提取试剂盒(TIANGEN)中的操作说明, 从胚胎和组织样品中提取总RNA。由于RNA易污染, 提取完成后迅速将其储存在–80 ℃中。随后, 用1%的琼脂糖凝胶电泳检测总RNA的完整性, 并使用分光光度计(260 nm)检测总RNA的浓度和纯度(王玉成等, 2006)。取5 μL总RNA, 根据TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix试剂盒(TransGen, 北京)提供的说明书将mRNA反转录成cDNA, 然后用无菌去离子水稀释5倍(1︰5), 置于–20 ℃中保存备用。
1.2.2 引物设计从NCBI (National Center for Biotechnology Information)数据库中获得候选内参基因的编码序列, 用Primer Premier 5.0设计出上、下游引物(表 1)。
| 基因 | 基因功能 | 引物序列(5′~3′) | 产物大小/bp | Tm /℃ | NCBI序列号 |
| 18S rRNA | 组成核糖体 | F: CGGTCGGCGTCCAACTTCTTAG R: TCTCGGCGAAGGGTAGACACAC | 183 | 60 | XR_002748043.1 |
| GAPDH | 参与糖酵解反应 | F: ACCGCCACACAGAAGACAGTT R: GGGGACACGGAAAGCCATAC | 146 | 60 | XM_023263785.2 |
| Ef-1α | 蛋白质翻译 | F: CCGCCTTACAGCCAGAAAC R: CAGCCAGAAATGGGAACAAAC | 179 | 61 | XM_023293606.1 |
| β-actin | 细胞骨架蛋白 | F: GCCTCCCCTCAGTAAAGCCAGAT R: CGCCTTCCTCCTCAAGTCAAACA | 145 | 61 | XM_023294531.1 |
以双锯鱼组织样品逆转录得到的cDNA混合液为模板, 对这4个候选内参基因进行普通PCR反应, 检测引物的特异性。反应体系(20 μL)为: ddH2O 10 μL, 2×TSINGKE® Master Mix 7.4 μL, 上游引物0.8 μL, 下游引物0.8 μL, cDNA 1 μL。
PCR反应程序设置为: 94 ℃预变性5 min; 94 ℃变性30 s, 60 ℃退火30 s, 72 ℃延伸30 s, 35个循环; 72 ℃延伸10 min。普通PCR扩增得到的产物用1%的琼脂糖凝胶电泳检测, 观察得到的条带是否单一。
1.2.4 qPCR试验以及数据分析使用TOROGreen® qPCR Master Mix试剂盒进行qPCR, 反应用StepOnePlusTM Real-Time PCR System仪器完成, 每份样品重复三次。反应体系(20 μL)为: 2×TSINGKE® Master Mix 10 μL, ddH2O 6.4 μL, 上游引物0.8 μL, 下游引物0.8 μL, cDNA 2 μL。
qPCR反应程序设置为: 95 ℃预变性1 min; 95 ℃变性10 s, 60 ℃退火30 s, 40个循环。
将qPCR得到的数据进行标准化处理, 然后运用geNorm和Norm Finder这两种软件对以上4个候选内参基因进行统计学分析, 从而筛选出在双锯鱼胚胎发育和不同组织中表达相对稳定的内参基因。
1.2.5 内参基因的应用本研究选用18S作为内参基因, 对TYR基因的表达进行分析。TYR基因的NCBI序列号为XP_023132182.1, 利用Primer Premier 5.0设计得到正、反向引物, 与内参基因的引物一并交由上海生工生物工程有限公司合成。
正向引物: TGGTTCCCTTCTTCCCTCTC
反向引物: GGTTACGCTTCCACTTCCTTC
2 结果与分析 2.1 RNA的质量评估及普通PCR产物检测利用分光光度计对提取的双锯鱼胚胎和成鱼组织样品的总RNA进行测定, 其中, OD260/280值为1.8~2.0, 浓度为340~590 ng/μL。数据表明样品的总RNA质量较好, 可以用于后续实验。
通过1%的琼脂糖凝胶电泳对普通PCR的产物进行检测, 这4个候选内参基因的引物与组织cDNA结合之后均得到一个单一的条带, 没有引物二聚体(图 6)。
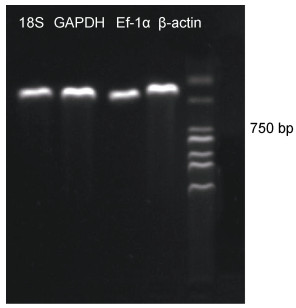 |
| 图 6 候选内参基因PCR产物 Fig. 6 PCR products of candidate reference genes |
利用qPCR反应得到的这4个候选内参基因的熔解曲线, 均为单一峰且重复性好(图 7), 进一步说明引物的特异性好。
 |
| 图 7 候选内参基因熔解曲线分析 Fig. 7 The melting curve analysis of candidate reference genes |
利用qPCR反应对这4个候选内参基因进行扩增, 模板分别取自双锯鱼的胚胎和成鱼组织, 数据见表 2, Ct值分布范围为13.67~36.71。其中, 最小的Ct值出现在18S rRNA基因的表达中, 表明其在胚胎和组织中的表达水平相对较高; 最大的Ct值出现在Ef-1α的表达中, 表明其表达水平较低, 分布范围为28.00~ 36.00 (图 8)。
| 样品 | 18S | β-actin | GAPDH | Ef-1α | |
| 白条双锯鱼胚胎不同时期 | 卵裂期 | 20.233 203 89 | 25.674 508 41 | 29.277 252 20 | 32.196 454 37 |
| 体节期 | 16.748 215 36 | 20.474 146 53 | 27.209 703 45 | 32.359 635 67 | |
| 翻转期 | 15.737 508 14 | 16.256 411 87 | 19.087 019 60 | 32.642 943 06 | |
| 血管形成期 | 15.722 440 08 | 16.599 465 69 | 17.217 384 97 | 32.366 768 14 | |
| 器官形成期 | 19.776 355 11 | 19.285 518 65 | 18.475 953 42 | 33.355 667 11 | |
| 孵化期 | 16.910 668 69 | 22.922 412 24 | 17.811 621 35 | 36.706 408 18 | |
| 眼斑双锯鱼胚胎不同时期 | 卵裂期 | 20.222 445 17 | 26.838 653 56 | 31.984 596 89 | 28.165 008 54 |
| 体节期 | 16.829 250 34 | 18.672 692 62 | 20.899 373 37 | 34.057 744 34 | |
| 翻转期 | 17.921 131 77 | 17.694 208 15 | 20.695 796 33 | 34.300 362 90 | |
| 血管形成期 | 18.422 943 12 | 17.104 501 72 | 20.288 960 77 | 34.078 220 37 | |
| 器官形成期 | 19.897 961 30 | 16.444 176 99 | 18.930 776 60 | 34.195 220 95 | |
| 孵化期 | 18.943 976 72 | 17.059 791 56 | 18.780 651 73 | 36.153 385 16 | |
| 白条双锯鱼成鱼组织 | 脑 | 19.604 449 80 | 31.802 319 42 | 32.193 656 07 | 34.485 516 23 |
| 白皮 | 17.555 663 85 | 22.446 880 34 | 24.226 675 03 | 33.309 556 54 | |
| 红皮 | 14.895 516 29 | 24.355 391 82 | 24.157 561 62 | 33.661 858 45 | |
| 眼 | 14.895 516 29 | 24.355 391 82 | 24.157 561 62 | 33.661 858 45 | |
| 肠 | 20.018 682 48 | 29.740 699 56 | 33.973 227 83 | 34.305 686 38 | |
| 心 | 20.598 351 37 | 31.882 079 44 | 23.165 037 58 | 29.639 093 19 | |
| 脾 | 17.419 445 36 | 32.643 692 65 | 34.429 874 84 | 32.211 217 46 | |
| 鳃 | 22.472 355 10 | 21.329 067 23 | 29.698 423 39 | 33.661 944 71 | |
| 尾 | 22.074 438 73 | 21.784 748 08 | 29.626 211 80 | 33.197 558 72 | |
| 肌肉 | 23.921 159 74 | 29.890 197 97 | 23.689 308 38 | 34.933 262 61 | |
| 眼斑双锯鱼成鱼组织 | 脑 | 20.417 102 81 | 30.651 219 26 | 31.172 313 37 | 34.264 852 77 |
| 白皮 | 16.068 778 36 | 19.582 672 75 | 20.008 410 14 | 33.236 911 99 | |
| 红皮 | 14.664 040 46 | 19.894 173 94 | 19.722 485 86 | 32.842 303 38 | |
| 眼 | 13.666 399 53 | 23.348 739 84 | 22.207 817 08 | 34.954 510 79 | |
| 肠 | 17.942 029 95 | 24.017 220 60 | 33.303 523 59 | 32.495 918 91 | |
| 心 | 21.746 465 47 | 28.551 180 73 | 20.381 635 45 | 35.072 257 15 | |
| 脾 | 19.750 886 71 | 29.513 195 04 | 33.019 158 26 | 33.210 472 53 | |
| 鳃 | 15.100 910 93 | 16.546 852 75 | 21.012 755 92 | 31.778 099 27 | |
| 尾 | 15.541 249 17 | 18.285 227 46 | 23.272 449 28 | 34.287 048 34 | |
| 肌肉 | 16.406 237 60 | 27.077 861 57 | 19.316 722 66 | 32.947 932 56 | |
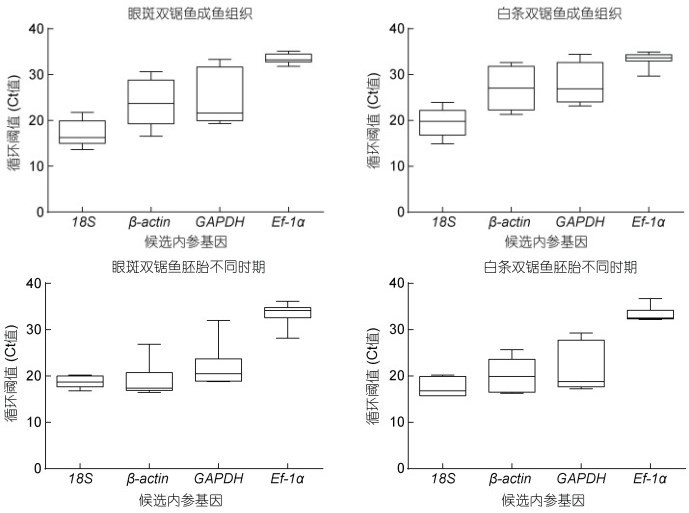 |
| 图 8 双锯鱼胚胎不同发育时期以及成鱼组织中4个候选内参基因的Ct值分布 Fig. 8 The Ct distribution of 4 candidate reference genes in the embryos and in the adult tissues of two anemone fish (A. frenatus and A. ocellaris) |
向内参基因筛选软件中输入标准化后的定量数据, geNorm会得出相应的表达稳定值(M值); M值越小, 基因表达越稳定(吴建阳等, 2017); 还有配对变异分析Vn/Vn+1结果, 若Vn/Vn+1 > 0.15, 则表明适合内参基因的数量有n+1个(胡金川, 2008)。Norm Finder会给出候选内参基因表达稳定值且得到一个最佳选择(滕彦娇等, 2021)。
geNorm分析结果表明, 在眼斑双锯鱼和白条双锯鱼的胚胎中, 18S和β-actin的M值最小; 在眼斑双锯鱼和白条双锯鱼的成鱼组织中, 18S、GAPDH和β-actin的M值最小(图 9)。又因为配对变异分析结果V2/3=0.904 > 0.15 (图 10), 因此在眼斑双锯鱼和白条双锯鱼的成鱼组织、胚胎中, 适合的内参基因有3个, 即18S、GAPDH和β-actin。
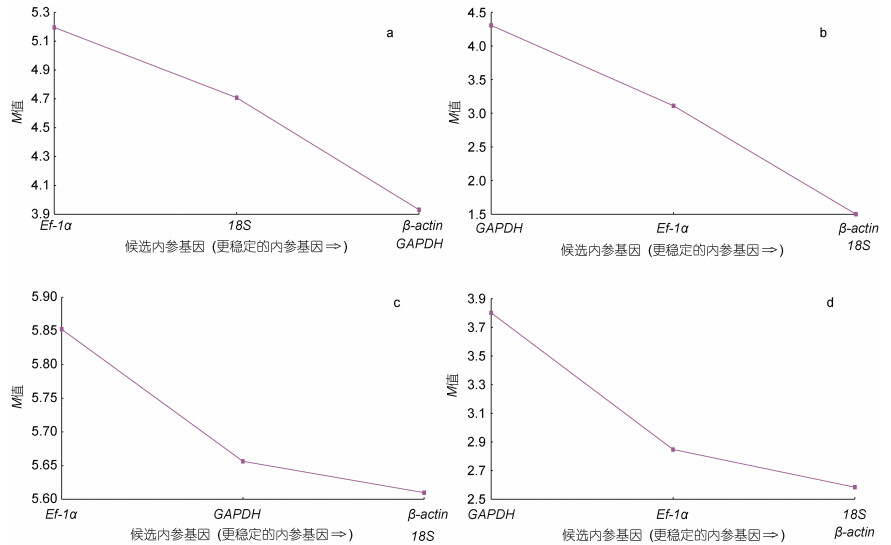 |
| 图 9 4个候选参考基因的平均表达稳定值 Fig. 9 The mean expression stability values of candidate reference genes 注: a. 眼斑双锯鱼成鱼组织; b. 眼斑双锯鱼胚胎不同发育时期; c. 白条双锯鱼成鱼组织; d. 白条双锯鱼胚胎不同发育时期 |
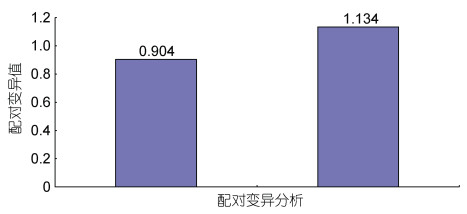 |
| 图 10 标准化控制基因数量的确定 Fig. 10 Determination of the optimal number of control genes for normalization |
利用Norm Finder软件对标准化后的数据进行分析, 在白条双锯鱼和眼斑双锯鱼的胚胎、成鱼组织样品中, 候选内参基因的稳定性排序为18S rRNA > β-actin > Ef-1α > GAPDH (稳定性值见表 3), 这与geNorm分析结果相符, 表明18S和β-actin在这4个候选基因中较为稳定。
| 基因 | 白条双锯鱼胚胎 | 白条双锯鱼成鱼组织 | 眼斑双锯鱼胚胎 | 眼斑双锯鱼成鱼组织 |
| 18S | 1.38 | 3.15 | 1.22 | 2.07 |
| β-actin | 1.49 | 3.17 | 1.84 | 2.86 |
| Ef-1α | 2.41 | 3.31 | 2.67 | 3.27 |
| GAPDH | 3.42 | 3.38 | 3.07 | 3.56 |
因此, 综合geNorm和NormFinder对候选内参基因稳定性的分析结果, 18S和β-actin的表达相对稳定, 且18S的M值最小, 更适合作为双锯鱼qPCR分析实验中的内参基因。
2.3 TYR基因表达分析利用qPCR技术, 以18S作为内参基因检测TYR基因在白条双锯鱼和眼斑双锯鱼的胚胎及成鱼组织中的表达情况, 数据见表 4。结果显示TYR基因在双锯鱼胚胎时期就已经开始表达, 表达水平呈先上升后下降的趋势, 在体节期达到最高。且在胚胎观察过程中发现, 黑色素细胞亦出现在白条双锯鱼和眼斑双锯鱼的体节期(参考图 3, 图 4); 在白条双锯鱼和眼斑双锯鱼成鱼的眼、红皮以及尾中表达量相对较高(图 11)。
| 样品 | 表达值 | 方差 | |
| 白条双锯鱼胚胎不同时期 | 卵裂期 | 0.064 700 329 | 0.014 031 195 |
| 体节期 | 20.985 859 680 | 4.366 306 772 | |
| 翻转期 | 8.080 906 002 | 3.139 288 718 | |
| 血管形成期 | 1.829 702 667 | 0.220 074 797 | |
| 器官形成期 | 0.684 392 815 | 0.068 085 951 | |
| 孵化期 | 0.086 264 321 | 0.020 710 141 | |
| 眼斑双锯鱼胚胎不同时期 | 卵裂期 | 0.006 143 738 | 0.000 527 067 |
| 体节期 | 25.406 642 290 | 3.618 796 182 | |
| 翻转期 | 0.552 635 445 | 0.085 370 738 | |
| 血管形成期 | 1.173 101 338 | 0.164 337 016 | |
| 器官形成期 | 5.204 407 300 | 1.545 504 665 | |
| 孵化期 | 1.556 491 153 | 0.414 764 253 | |
| 白条双锯鱼成鱼组织 | 脑 | 1.979 600 353 | 0.839 208 295 |
| 白皮 | 2.086 598 358 | 1.447 061 984 | |
| 红皮 | 17.873 852 740 | 6.024 160 485 | |
| 眼 | 20.769 397 450 | 3.064 902 810 | |
| 心 | 1.129 389 057 | 0.946 304 624 | |
| 肠 | 0.098 956 221 | 0.120 741 096 | |
| 脾 | 0.005 908 475 | 0.002 985 394 | |
| 鳃 | 3.499 220 193 | 3.091 848 081 | |
| 尾 | 29.834 047 950 | 5.862 954 725 | |
| 肌肉 | 0.004 298 665 | 1.546 804 270 | |
| 眼斑双锯鱼成鱼组织 | 脑 | 0.553 707 659 | 0.396 591 927 |
| 白皮 | 4.339 093 184 | 2.904 411 319 | |
| 红皮 | 16.485 693 880 | 8.766 300 220 | |
| 眼 | 8.937 691 557 | 2.699 426 528 | |
| 心 | 0.913 546 080 | 0.818 018 113 | |
| 肠 | 0.969 164 233 | 0.924 197 013 | |
| 脾 | 0.024 136 951 | 0.012 803 594 | |
| 鳃 | 0.403 210 861 | 0.337 350 766 | |
| 尾 | 10.851 223 170 | 3.826 271 414 | |
| 肌肉 | 0.024 572 363 | 0.000 053 000 |
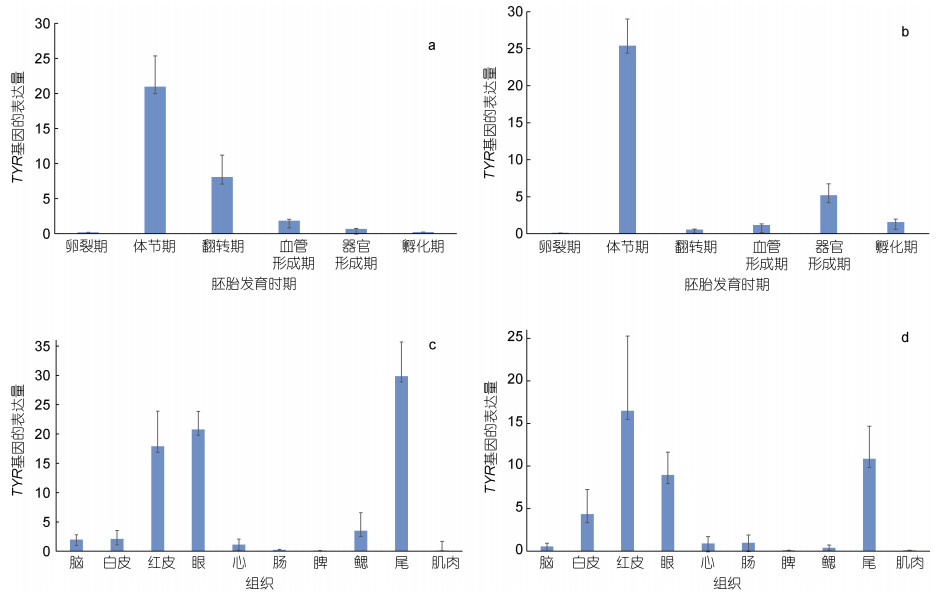 |
| 图 11 TYR基因在白条双锯鱼和眼斑双锯鱼的胚胎及成鱼组织中的表达 Fig. 11 The expression of TYR gene in the embryos and adult tissues of tomato clownfish (A. frenatus) and Fales-Clown anemone fish (A. ocellaris) 注: a. 白条双锯鱼胚胎不同发育时期; b. 眼斑双锯鱼胚胎不同发育时期; c. 白条双锯鱼成鱼组织; d. 眼斑双锯鱼成鱼组织 |
实时定量PCR (qPCR)是目标基因准确表达的首选工具, 内参基因则是qPCR数据分析的参考标准, 但不是所有内参基因在实验中都能够稳定表达(Vandesompele et al, 2002)。因此, 在进行qPCR之前需要根据样品和实验条件选择合适的内参基因。geNorm和Norm Finder软件是专门用来筛选内参基因的, 但分析结果略有不同。其中, Norm Finder是根据内参基因的稳定值大小对其进行排序, 并得到每个实验条件下最稳定的内参基因; geNorm则是可以得出适合实验的最佳内参基因组合(吴建阳等, 2017)。
用于鱼类胚胎不同发育阶段以及不同组织研究的内参基因种类很多, 常见的有Ef-1α基因, 在真核生物中参与蛋白质的翻译, 是总蛋白含量的1%~2% (唐永凯等, 2008); 18S rRNA是一类编码核糖体小亚基的rRNA (周晓馥等, 2016); GAPDH基因参与糖降解反应(Pei et al, 2007); β-actin基因是肌肉及细胞骨架的主要成分, 广泛分布于各组织中(唐永凯等, 2013)。更多的研究为了真实地反映目标基因的表达水平, 会筛选出两个或两个以上的内参基因作为数据分析过程中的参考, 如张宁等(2018)通过实验选择了rpl7和rpl7α作为多鳞鱚研究中的内参基因; 在对不同处理条件下的异育银鲫(Carassius auratus gibelio)组织和尾鳍细胞的研究中, 实验结果表明β-actin可以作为肾脏组织和细胞研究的适合内参基因, 而Ef-1α适合在病毒刺激下作为数据分析的内参基因(费越越等, 2020); 茅华华等(2016)对斑鳢(Channa maculata)的胚胎不同时期和成鱼组织进行分析, 结果得出β-actin和tmem104适合作为组织相关基因表达分析的内参基因, 而sep15和tmem256可以作为胚胎相关研究的内参基因。
目前, 关于双锯鱼的研究多倾向于胚胎发育和饲养、繁殖, 而对内参基因筛选的研究少之又少。本研究通过geNorm和Norm Finder软件对β-actin、18S rRNA、Ef-1α和GAPDH的稳定性进行分析, 最终选择18S作为双锯鱼基因表达研究的合适内参基因。这与陈张帆等(2018)在牙鲆胚胎中的筛选结果相似。TYR是黑色素合成的关键酶, TYR基因的表达决定了生物体色的变化程度(张植元等, 2017)。结果表明, 在双锯鱼胚胎发育过程中就已经出现了黑色素细胞(图 2), 即TYR基因开始表达, 在体节期表达量最高; 在成鱼的眼、尾和红皮中表达量最高。只有筛选出合适的内参基因, qPCR技术在目标基因的研究中才能发挥重要作用, 否则就会出现一定的误差(吕梁等, 2019)。旨在为后期研究双锯鱼相关基因的表达机制提供应用基础, 使数据分析结果更有效、可靠。
4 结论为了筛选出合适的内参基因, 本实验以眼斑双锯鱼和白条双锯鱼的胚胎、组织为研究对象, 对β-actin、18S rRNA、Ef-1α和GAPDH的表达稳定性进行分析, 虽然geNorm和Norm Finder显示的结果略有不同, 但18S rRNA相对于其他内参基因稳定值更高, 并选其作为TYR基因相对定量数据分析的参考标准, 以期为研究双锯鱼胚胎、个体发育过程中基因的表达水平提供有效的数据和应用基础。
王玉成, 张国栋, 姜静. 2006. 一种适用范围广的总RNA提取方法. 植物研究, 26(1): 84-87 DOI:10.3969/j.issn.1673-5102.2006.01.018 |
邓红平, 何继亮. 2006. 几种管家基因突变试验及其研究进展. 中华劳动卫生职业病杂志, 24(11): 702-704 DOI:10.3760/cma.j.issn.1001-9391.2006.11.025 |
叶乐, 王雨, 杨其彬, 等. 2008. 小丑鱼规模化繁育技术研究. 中国水产, (12): 59-60 DOI:10.3969/j.issn.1002-6681.2008.12.030 |
白海锋, 李晓光, 张霖, 等. 2011. 八种小丑鱼与五种海葵相容性的初步研究. 水产养殖, 32(11): 11-13 DOI:10.3969/j.issn.1004-2091.2011.11.05 |
吕梁, 张子平, 万海付, 等. 2019. 拟穴青蟹不同发育时期胚胎基因表达的内参基因的筛选. 中国水产科学, 26(3): 457-464 |
刘颖, 王双耀, 高乔, 等. 2013. 栉孔扇贝内参基因稳定性研究. 生态毒理学报, 8(4): 616-622 |
杨爱馥, 周遵春, 董颖, 等. 2010. 仿刺参cytb和β-actin基因表达稳定性比较. 中国农业科技导报, 12(1): 79-84 DOI:10.3969/j.issn.1008-0864.2010.01.14 |
吴建阳, 何冰, 杜玉洁, 等. 2017. 利用geNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法. 现代农业科技, (5): 278-281 DOI:10.3969/j.issn.1007-5739.2017.05.174 |
吴萍, 张方亮, 杨程涌, 等. 2017. 翘嘴鳜per1 mRNA表达量分析中采用的内参基因稳定性比较. 激光生物学报, 26(4): 379-384 DOI:10.3969/j.issn.1007-7146.2017.04.015 |
何丽斌, 陈芳, 朱志煌, 等. 2018. 基于线粒体16S rRNA、COX1和Cytb基因探讨11种小丑鱼的系统发育关系. 福建农业学报, 33(3): 230-235 |
张宁, 杜文文, 王中铎, 等. 2018. 多鳞鱚不同组织荧光定量PCR内参基因筛选. 广东海洋大学学报, 38(5): 8-14 DOI:10.3969/j.issn.1673-9159.2018.05.002 |
张植元, 魏东, 乔秀亭, 等. 2017. 饲料浮萍水平对大正三色锦鲤TYR及MC1R基因表达的影响. 水产科学, 36(2): 172-177 |
张薇, 孙志宾, 马爱军, 等. 2018. 双锯鱼属(Amphiprion)四种鱼种比较胚胎学研究和进化探讨. 海洋与湖沼, 49(3): 671-681 |
陈张帆, 宋莉妮, 王鹏飞, 等. 2018. 牙鲆胚胎低温处理下内参基因筛选及CIRP和Hsp70基因表达分析. 农业生物技术学报, 26(8): 1382-1391 |
茅华华, 陈昆慈, 赵建, 等, 2016. 斑鳢实时定量PCR内参基因的分离鉴定[C]. // 2016年中国水产学会学术年会论文摘要集. 成都: 中国水产学会.
|
周晓馥, 王晶, 史宏伟, 等. 2016. 18S rRNA作为植物实时荧光定量PCR内参基因的探究. 吉林师范大学学报(自然科学版), 37(2): 115-119 |
胡金川. 2008. 如何利用geNorm软件筛选基因表达测定的内参基因?. 中华检验医学杂志, 31(8): 918 DOI:10.3321/j.issn:1009-9158.2008.08.033 |
费越越, 南星羽, 余路, 等. 2020. 异育银鲫内参基因的筛选. 水产科学, 39(3): 306-315 |
唐永凯, 李建林, 李红霞, 等. 2008. 奥利亚罗非鱼EF-1α基因的克隆与结构分析. 动物学杂志, 43(5): 7-12 DOI:10.3969/j.issn.0250-3263.2008.05.002 |
唐永凯, 俞菊华, 徐跑, 等. 2013. 建鲤内参基因β-actin的实时荧光定量PCR方法的建立. 浙江农业学报, 25(1): 31-36 DOI:10.3969/j.issn.1004-1524.2013.01.06 |
章霞, 李伟业, 傅荣兵, 等. 2015. 观赏小丑鱼人工繁育技术的研究. 科学养鱼, (8): 78 |
蒋素华, 马杰, 梁芳, 等, 2018. 一个白及内参基因EF1α及其应用: CN109022448A [P]. [2018-12-18].
|
蒋燕玲, 2016. 橘色双冠丽鱼体色发育变化及体色相关基因TYR的克隆与表达研究[D]. 上海: 上海海洋大学.
|
滕力平, 杨担光, 李晓光, 等. 2005. 二线小丑鱼的人工繁殖. 水产科学, 24(2): 26-28 |
滕彦娇, 王宏鹏, 王菁, 等. 2021. 大花红景天内参基因筛选与POD基因表达分析. 中药材, 44(6): 1341-1347 |
AMONRAT K, RAWIWAN W, VORATHEP M et al, 2013. Effects of dietary protein, lipid and astaxanthin levels on growth and carotenoid accumulation in anemone fish, Amphiprion ocellaris. Journal of Science, Technology, and Humanities, 11(2): 95-103 |
DHEDA K, HUGGETT J F, CHANG J S et al, 2005. The implications of using an inappropriate reference gene for real-time reverse transcription PCR data normalization. Analytical Biochemistry, 344(1): 141-143 DOI:10.1016/j.ab.2005.05.022 |
FRANZELLITTI S, KIWAN A, VALBONESI P et al, 2015. Selection of best-performing reference gene products for investigating transcriptional regulation across silvering in the European eel (Anguilla anguilla). Scientific Reports, 5(1): 16966 DOI:10.1038/srep16966 |
IWATA E, NAGAI Y, HYOUDOU M et al, 2008. Social environment and sex differentiation in the false clown anemonefish, Amphiprion ocellaris. Zoological Science, 25(2): 123-128 DOI:10.2108/zsj.25.123 |
MITTER K, KOTOULAS G, MAGOULAs A, et al, 2009. Evaluation of housekeeping genes for quantitative expression studies in the European seabass D. labrax [C]. // Proceedings of the 9th Symposium on Oceanography & Fisheries, 2nd: 1178-1182.
|
MUGHAL B B, LEEMANS M, SPIRHANZLOVA P et al, 2018. Reference gene identification and validation for quantitative real-time PCR studies in developing Xenopus laevis. Scientific Reports, 8(1): 496 DOI:10.1038/s41598-017-18684-1 |
PEI D S, SUN Y H, CHEN S P et al, 2007. Zebrafish GAPDH can be used as a reference gene for expression analysis in cross-subfamily cloned embryos. Analytical Biochemistry, 363(2): 291-293 DOI:10.1016/j.ab.2006.12.005 |
PFAFFL M W, TICHOPAD A, PRGOMET C et al, 2004. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper- excel-based tool using pair-wise correlations. Biotechnology Letters, 26(6): 509-515 DOI:10.1023/B:BILE.0000019559.84305.47 |
SZCZYGIEŁ J, KAMIŃSKA-GIBAS T, PETIT J et al, 2021. Re-evaluation of common carp (Cyprinus carpio L.) housekeeping genes for gene expression studies - considering duplicated genes. Fish & Shellfish Immunology, 115: 58-69 |
TANG X, ZHANG N, SI H J et al, 2017. Selection and validation of reference genes for RT-qPCR analysis in potato under abiotic stress. Plant Methods, 13: 85 DOI:10.1186/s13007-017-0238-7 |
VANDESOMPELE J, DE PRETER K, PATTYN F et al, 2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biology, 3(7): RESEARCH0034 DOI:10.1186/gb-2002-3-7-reports0034 |
 2022, Vol. 53
2022, Vol. 53


