
中国海洋湖沼学会主办。
文章信息
- 王菁, 李桂英, 林楷琪, 欧静, 张淑瓶, 王忠良. 2022.
- WANG Jing, LI Gui-Ying, LIN Kai-Qi, OU Jing, ZHANG Shu-Ping, WANG Zhong-Liang. 2022.
- 基于马氏珠母贝(Pinctada fucata)血细胞全长转录组数据补体样组分的鉴定与分析
- IDENTIFICATION AND ANALYSIS OF COMPLEMENT-LIKE COMPONENTS IN PINCTADA FUCATA BASED ON THE FULL-LENGTH TRANSCRIPTOME SEQUENCE DATASETS
- 海洋与湖沼, 53(1): 176-186
- Oceanologia et Limnologia Sinica, 53(1): 176-186.
- http://dx.doi.org/10.11693/hyhz20210700158
文章历史
-
收稿日期:2021-07-08
收修改稿日期:2021-08-20
与其他无脊椎动物一样, 马氏珠母贝(Pinctada fucata)缺乏适应性免疫系统, 其机体的正常生命活动主要依靠先天免疫系统以抵御各种病原和外来物质的入侵(焦钰等, 2020)。先天免疫是机体抵抗和消灭外来病原入侵的第一道防线(吴勇等, 2019)。补体系统作为先天免疫的组成部分及免疫效应系统, 是连接先天免疫和适应性免疫的枢纽, 具有识别、清除病原体的作用(刘先军等, 2011; Li et al, 2019)。
补体系统是一种复杂的生物效应系统, 自19世纪末被发现以来, 在哺乳动物中展开了广泛的研究(Fujita et al, 2004)。哺乳动物补体系统是由30多种蛋白组成的复杂蛋白水解系统, 具有诱导炎症、调理并溶解病原体的作用(Suckale et al, 2005)。补体系统的激活途径主要包括经典途径(classic pathway)、凝集素途径(lectin pathway)和替代途径(alternative pathway) (Fujita et al, 2004)。经典途径和凝集素途径通过利用不同的蛋白质C1q (C1q domain containing protein)、MBL (mannose-binding lectin)、Ficolins识别靶标从而激活蛋白酶C1r、C1s、MASP (mannan-binding lectin serine protease); 而替代途径中激活的C3 (complement 3)可与Bf (Factor B)结合, 在Factor D作用下形成C3转化酶(Nonaka et al, 2006)。三条补体途径激活后最终均进入终末途径(terminal pathway), 形成膜攻击复合物(membrane attack complex, MAC)裂解靶细胞(Suckale et al, 2005)。此外, 缺乏C3基因的小鼠可通过凝血酶替代C3依赖型C5转化酶介导补体途径的激活(Huber-Lang et al, 2006)。
事实上, 补体系统是一种古老的防御系统, 其起源早于脊椎动物的发育, 在许多无脊椎动物门中均发现了与哺乳动物相似的补体成分, 包括尾索动物(Al-Sharif et al, 1998; Suckale et al, 2005)、头索动物(Suzuki et al, 2002)、刺胞动物(Ji et al, 1997)、节肢动物(Dishaw et al, 2005; Liu et al, 2019)、软体动物(Gerdol et al, 2015; Wang et al, 2019)等。早期在海胆(sea urchins)和被囊类动物中鉴定到的与脊椎动物C3同源的分子具有调理活性以及潜在的C3转化酶作用。因此, 研究者普遍认为补体系统起源于后口动物(Nonaka et al, 2004a, 2004b)。然而, 在节肢动物鲎(Carcinoscorpius rotundicauda)中发现了脊椎动物C3和C2/Bf的同源物, 表明在原口动物谱系中存在着以C3为中心分子的复杂的调理防御系统(Zhu et al, 2005)。从海鞘(Halocynthia roretzi)分离得到甘露糖凝集素, 可结合相关的甘露糖结合型凝集素关联丝氨酸蛋白酶, 进而激活C3启动补体系统(Sekine et al, 2001)。关于补体系统的研究在双壳贝类中同样展开了广泛的研究, 且相关补体分子在机体的免疫响应中发挥着重要作用, 包括栉孔扇贝(Chlamys farreri) (Yang et al, 2011)、菲律宾蛤仔(Ruditapes philippinarum) (Takahashi et al, 2008)、海湾扇贝(Argopecten irradians) (Yang et al, 2014)、光滑双脐螺(Biomphalaria glabrata) (Lu et al, 2020)、太平洋牡蛎(Crassostrea gigas) (Wang et al, 2017)、厚壳贻贝(Mytilus coruscus) (陈永霞, 2018)等。对太平洋牡蛎的研究发现, 其体内存在C3、含C1q结构域蛋白(C1qDC)、凝集素样蛋白(C-lectin like protein)、纤维蛋白原相关蛋白(fibrinogen-related protein, FREP)、丝氨酸蛋白酶(Serine protease)和补体受体样蛋白, 表明双壳类中存在一个多组分的补体系统(Wang et al, 2017)。文蛤(Ruditapes decussatus)中C3样分子和Bf样分子的存在, 表明双壳类补体系统可能通过替代途径激活(Prado-Alvarez et al, 2009)。缢蛏(Sinonovacula constricta)中C3、C1qDC、C-type lectin基因在先天免疫中发挥重要的免疫作用(Peng et al, 2017; Li et al, 2019; Lan et al, 2020)。此外, 补体系统中存在具有相似结构的同源分子, 如C3/C4/C5、C2/Bf和C1s/C1r/MASP, 被认为是基因复制的产物(Nonaka et al, 2004a)。因此, Nonaka等(2004a)认为哺乳动物的补体系统似乎是通过基因复制从一个更简单的补体系统逐步发展进化形成的。
全长转录组测序技术的兴起为绝大部分非模式生物的基因研究提供了更全面的基因序列信息(Chao et al, 2019), 进而可以全面、系统地开展补体系统进化、功能和作用机制研究。本文基于马氏珠母贝全长转录组测序数据, 系统地检索马氏珠母贝血细胞中存在的潜在补体样组分基因; 同时, 使用溶藻弧菌(Vibrio alginolyticus)刺激马氏珠母贝后的血细胞样品进行实时定量PCR检测相关补体基因的表达水平变化, 有助于加强对马氏珠母贝免疫相关基因的系统认知, 进一步理解马氏珠母贝补体系统的组成及作用机制, 对丰富和发展海洋无脊椎动物补体系统也具有重要理论意义。
1 材料与方法 1.1 马氏珠母贝的暂养、细菌刺激及总RNA提取马氏珠母贝(平均壳长70 mm)购自广东省湛江市雷州养殖场, 暂养于室内过滤海水桶中, 水温25 ℃, 饲养期间保持充气, 定时投喂螺旋藻粉; 每天100%换水一次。室内暂养一周后将马氏珠母贝随机分为两组, 实验组采用闭壳肌注射的方法注射100 µL浓度为5×107 CFU/mL的溶藻弧菌悬浮液; 对照组于闭壳肌处注射等体积的PBS溶液(pH 7.0) (Wang et al, 2016)。注射4 h后, 于闭壳肌处采集血淋巴, 每只约0.5 mL, 采集的血淋巴立即分装至1.5 mL离心管中, 4 ℃, 800×g条件下离心10 min收集血细胞, 并立即提取总RNA。
1.2 全长转录组文库构建及测序使用马氏珠母贝健康血细胞样品, 利用Agilent 2100和NanoDrop 2000分光光度计检测总RNA的完整性和浓度。按照制造商说明书进行SMRTbell文库构建, 采用MagBead Loading上机测序获得原始数据, 使用SMRT Link v6.0对原始数据进行分析提取CCS (Circular Consensus Sequence)序列, 对全长序列进行去冗余聚类、校正, 得到的全长转录组数据已提交至NCBI数据库(https://www.ncbi.nlm.nih.gov/sra/SRR9644402)。
1.3 生物信息学分析Unigene在Nt、Nr、Swiss-Prot、KEGG、COG及Pfam数据库中比对并获得基因注释信息(E值< 1.0E–5)。使用unigene在Nr库中比对结果最好(E值最低)的序列为对应同源序列, 确定同源序列所属物种并统计比对到各个物种的同源序列数量。
1.4 补体样组分筛选基于功能基因注释信息检索含C1q结构域蛋白(C1q、C1qDC)、凝集素(凝集素、C型凝集素、甘露糖结合凝集素)、纤维胶凝蛋白、C3、纤维蛋白原相关蛋白、丝氨酸蛋白酶、含硫酯蛋白(thioester- containing protein, TEP)、补体因子等补体样组分。采用NCBI保守结构域数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)及SMART server (http://smart.embl-heidelberg.de/)进行补体样组分的结构域检测。
1.5 实时定量PCR检测补体相关基因表达水平从全长转录组数据中筛选的补体相关基因中随机选择12个基因进行实时定量PCR检测其表达水平, 使用One-Step gDNA Removal试剂盒(北京全式金生物公司)合成cDNA第一链。按照PerfectStart® Green qPCR SuperMix (北京全式金生物公司)说明书进行实时定量PCR, 实验获得数据使用2–ΔΔCt方法进行计算, 并使用SPSS 24进行独立样本T检验, P < 0.05为显著差异, P < 0.01为极显著差异。
2 结果与分析 2.1 全长转录组测序数据利用SMRT测序技术, 共获得14 048 434 585总碱基数(bp), 原始平均序列长度为1 838 bp, N50为2 563 bp。使用Permissive CCS2软件进行序列提取和质量过滤后, 共得到915 060 427个循环一致序列(CCS), 其中全长非嵌合序列(full-length non-chimeric, FLNC)具有636 357 960个CCS, 去掉5′primer、3′primer和polyA后获得原始的全长转录本序列。序列去冗余后获得82 381 unigenes, N50为2861 bp (表 1)。
| 转录本 | SMRT结果 |
| 数据总长/bp | 14 048 434 585 |
| 平均原始数据长度/bp | 1 838 |
| 原始数据N50/bp | 2 563 |
| CCS | 915 060 427 |
| FLNC | 636 357 960 |
| Unigene | 82 381 |
| N50/bp | 2 861 |
使用BLAST工具将所有的Unigene与NR、NT、Pfam、Swiss-Prot等蛋白序列数据库进行比对, 结果共得到59 621个注释基因和22 760个未注释基因(图 1), 表明全长转录组基因注释的水平较好。Unigene注释到同源基因的物种分布如图 2所示, 其中匹配到太平洋牡蛎的基因数量最多, 有42 014条基因。其他同源序列匹配较高的物种有马氏珠母贝、鸭嘴舌形贝(Lingula anatina)、美国加州海兔(Aplysia californica)等物种。
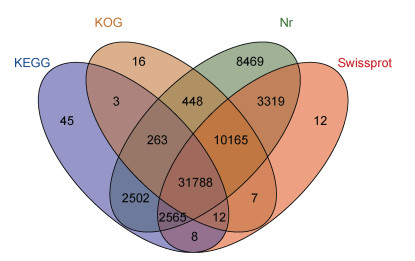 |
| 图 1 马氏珠母贝基因功能注释venn图 Fig. 1 The Venn diagram of gene functional annotation in P. fucata |
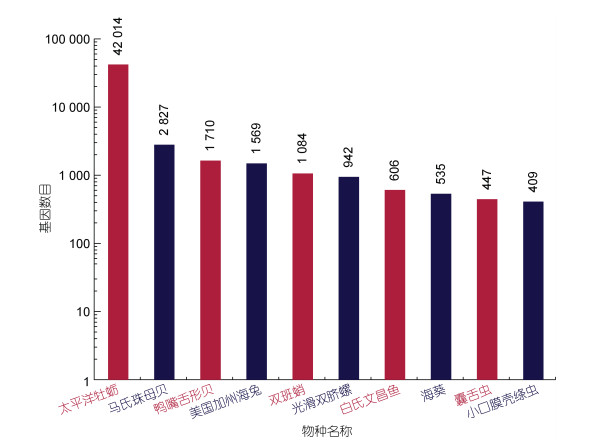 |
| 图 2 马氏珠母贝全长转录组unigene与NR数据库匹配物种分布 Fig. 2 NR annotated species distribution of transcriptome unigenes of P. fucata |
通过检索马氏珠母贝全长转录组测序数据, 共获得212条补体样成分unigene, 分别编码89个含C1q结构域蛋白、57个C型凝集素蛋白、33个纤维胶凝蛋白、11个纤维蛋白原相关蛋白、8个甘露糖结合型凝集素关联丝氨酸蛋白酶(MASP)、2个含硫酯蛋白(1个C3分子, 1个TEP分子)、1个补体受体、2个补体因子、9个丝氨酸蛋白酶。
2.4 含C1q结构域蛋白在检索到的89个含C1q结构域蛋白(C1qDC)中, 有81个C1qDC含有C末端球状结构域, 10个C1qDC蛋白还具有smc结构域; 6个C1q亚成分结合蛋白含有MAM33结构域, 该结构域可与C1q的球状结构域结合, 参与线粒体的氧化磷酸化和细胞核-线粒体的相互作用, 但均未能检测到N末端的胶原结构域(表 2)。
| 基因ID | 注释信息 | 保守结构域 |
| Isoform0004975 | Complement C1q-like protein 4 | — |
| Isoform0072432 | Complement C1q-like protein 4 | — |
| Isoform0002888 | Complement component 1 Q subcomponent-binding protein | MAM33 |
| Isoform0008172 | Complement component 1 Q subcomponent-binding protein | MAM33 |
| Isoform0008699 | Complement component 1 Q subcomponent-binding protein | MAM33 |
| Isoform0011565 | Complement C1q-like protein 4 | C-球状结构域、smc |
| Isoform0015471 | Complement C1q-like protein 4 | C-球状结构域、smc |
| Isoform0016222 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0019455 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0023169 | Complement component 1 Q subcomponent-binding protein | MAM33 |
| Isoform0025205 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0025376 | Complement component 1 Q subcomponent-binding protein | MAM33 |
| Isoform0026227 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0031059 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0047238 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0047673 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0049451 | Complement component 1 Q subcomponent-binding protein | MAM33 |
| Isoform0075709 | Complement C1q -like protein 4 | C-球状结构域、smc |
| Isoform0055352 | complement 1 q protein | C-球状结构域 |
甘露糖结合凝集素(MBL)和纤维胶凝蛋白(ficolin)分子的结构类似, 仅在C端的放射状球形结构域上各自有差别。在本文中, 共检索到33个ficolin, 其中31个分子具有C末端FBG结构域。同时, 检测到11个具有FBG结构域的纤维蛋白原相关蛋白, 57个C型凝集素基因(C-type lectin, CTL)中有26个基因具有CTLD结构域(表 3)。且在全长转录组数据中未检索到甘露糖结合凝集素。
| 基因ID | 注释信息 | 保守结构域 |
| Isoform0000057 | C-type lectin-1 | CTLD结构域 |
| Isoform0000588 | C-type lectin 8 | CTLD结构域 |
| Isoform0000836 | C-type lectin 8 | CTLD结构域 |
| Isoform0016929 | C-type lectin 7 | CTLD结构域 |
| Isoform0028928 | C-type lectin lectoxin-Lio3-like | CTLD结构域 |
| Isoform0050423 | C-type lectin domain family 17 | CTLD结构域 |
| Isoform0066707 | C-type lectin isoform 3 | REJ |
| Isoform0080026 | C-type lectin isoform 1 | CTLD结构域 |
| Isoform0015097 | C-type lectin domain family 17 | FTP |
| Isoform0001111 | C-type lectin | CTLD结构域 |
| Isoform0006712 | C-type lectin | CTLD结构域 |
| Isoform0009334 | C-type lectin | CTLD结构域 |
| Isoform0009711 | C-type lectin | — |
| Isoform0010194 | C-type lectin | — |
| Isoform0010594 | C-type lectin | CTLD结构域 |
| Isoform0010611 | C-type lectin | CTLD结构域 |
| Isoform0011410 | C-type lectin | — |
| Isoform0011681 | C-type lectin | CUB结构域 |
| Isoform0013319 | C-type lectin | CTLD结构域 |
| Isoform0013683 | C-type lectin | — |
| Isoform0014936 | C-type lectin | CUB结构域 |
| Isoform0016790 | C-type lectin | CTLD结构域 |
| Isoform0017277 | C-type lectin | CCP结构域 |
| Isoform0017960 | C-type lectin | CTLD结构域 |
| Isoform0018879 | C-type lectin | CUB结构域 |
| Isoform0019968 | C-type lectin | CCP结构域 |
| Isoform0020006 | C-type lectin | CTLD结构域 |
| Isoform0021924 | C-type lectin | CCP结构域 |
| Isoform0028029 | C-type lectin | CTLD结构域 |
| Isoform0031196 | C-type lectin | CTLD结构域 |
| Isoform0032523 | C-type lectin | CTLD结构域 |
| Isoform0034067 | C-type lectin | CUB结构域 |
| Isoform0034757 | C-type lectin | CUB结构域 |
| Isoform0036042 | C-type lectin | CTLD结构域 |
| Isoform0041354 | C-type lectin | CTLD结构域 |
| Isoform0042024 | C-type lectin | CCP结构域 |
| Isoform0042186 | C-type lectin | CA结构域 |
| Isoform0042950 | C-type lectin | CCP结构域 |
| Isoform0047481 | C-type lectin | CTLD结构域 |
| Isoform0048787 | C-type lectin | — |
| Isoform0049720 | C-type lectin | CTLD结构域 |
| Isoform0050311 | C-type lectin | CTLD结构域 |
| Isoform0050510 | C-type lectin | — |
| Isoform0051990 | C-type lectin | GAIN结构域 |
| Isoform0053723 | C-type lectin | CUB结构域 |
| Isoform0053790 | C-type lectin | CUB结构域 |
| Isoform0054956 | C-type lectin | CUB结构域 |
| Isoform0057321 | C-type lectin | CCP结构域 |
| Isoform0057716 | C-type lectin | CUB结构域 |
| Isoform0058976 | C-type lectin | CUB结构域 |
| Isoform0061957 | C-type lectin | CUB结构域 |
| Isoform0064154 | C-type lectin | CCP、EGF结构域 |
| Isoform0068521 | C-type lectin | CCP结构域 |
| Isoform0074531 | C-type lectin | CTLD结构域 |
| Isoform0077736 | C-type lectin | CTLD结构域 |
| Isoform0080116 | C-type lectin | CCP结构域 |
| Isoform0082337 | C-type lectin | — |
补体系统三条激活途径具有不同的丝氨酸蛋白酶分子, MASP/C1r/C1s在凝集素途径和经典途径中发挥作用, 而替代途径中则是Bf/C2。在马氏珠母贝全长转录组数据中共检索到17个丝氨酸蛋白酶基因, 其中, 6个甘露糖结合型凝集素丝氨酸蛋白酶含有IG_like、Tryp_SPc结构域; 2个甘露糖结合型丝氨酸蛋白酶含有CUB、IG_like、Tryp_SPc结构域; 2个丝氨酸蛋白酶含有Trypsin_2结构域; 7个丝氨酸蛋白酶基因含有N端β-螺旋结构域(表 4)。
| 基因ID | 注释信息 | 结构域 |
| Isoform0009961 | mannan-binding lectin serine protease 1 | IG_like、Tryp_SPc |
| Isoform0017290 | mannan-binding lectin serine protease 1 | IG_like、Tryp_SPc |
| Isoform0021114 | mannan-binding lectin serine protease 1 | IG_like、Tryp_SPc |
| Isoform0026673 | mannan-binding lectin serine protease 1 | IG_like、Tryp_SPc |
| Isoform0038356 | mannan-binding lectin serine protease 1 | IG_like、Tryp_SPc |
| Isoform0058600 | mannan-binding lectin serine protease 1 | CUB、IG_like、Tryp_SPc |
| Isoform0059099 | mannan-binding lectin serine protease 1 | IG_like、Tryp_SPc |
| Isoform0080823 | mannan-binding lectin serine protease 1 | CUB、IG_like、Tryp_SPc |
| Isoform0031455 | Serine protease | Trypsin_2 |
| Isoform0032197 | Serine protease | Trypsin_2 |
| Isoform0044225 | Serine protease | N端β-螺旋结构域 |
| Isoform0049848 | Serine protease | N端β-螺旋结构域 |
| Isoform0054163 | Serine protease | N端β-螺旋结构域 |
| Isoform0065400 | Serine protease | N端β-螺旋结构域 |
| Isoform0073592 | Serine protease | N端β-螺旋结构域 |
| Isoform0074112 | Serine protease | N端β-螺旋结构域 |
| Isoform0075574 | Serine protease | N端β-螺旋结构域 |
马氏珠母贝全长转录组数据中共检索到2个含硫酯结构域蛋白, 包括C3分子和TEP (thioester- containing protein)分子。保守结构域分析发现C3具有complement_C3_C4_C5、A2M_comp结构域; TEP分子具有A2M_2、A2M_comp、A2M_recep结构域(表 5)。D因子具有Tryp_SPc结构域, 而H因子和补体受体2 (Cr2)未检测到相应的结构域(表 5)。
| 基因ID | 注释信息 | 结构域 |
| Isoform0018832 | Complement C3 | Complement_C3_C4_C5、A2M_comp |
| Isoform0081916 | thioester-containing protein 1 | A2M_2、A2M_comp、A2M_recep |
| Isoform0001492 | Complement factor D | Tryp_SPc |
| Isoform0050510 | Complement factor H | — |
| Isoform0065377 | Complement receptor type 2 | — |
随机挑选12个补体样相关基因进行实时荧光定量PCR, 检测其在溶藻弧菌刺激前后的表达水平变化(图 3)。结果显示, 与对照组相比, 11个基因的表达水平上调, 其中10个基因表现出极显著差异, 包括C1q、C-lectin、MBL、ficolin、MASP、Hf-like等补体基因, 暗示马氏珠母贝的补体系统是一个复杂的免疫效应系统, 包括并不限于这些基因参与到马氏珠母贝的免疫响应过程中。
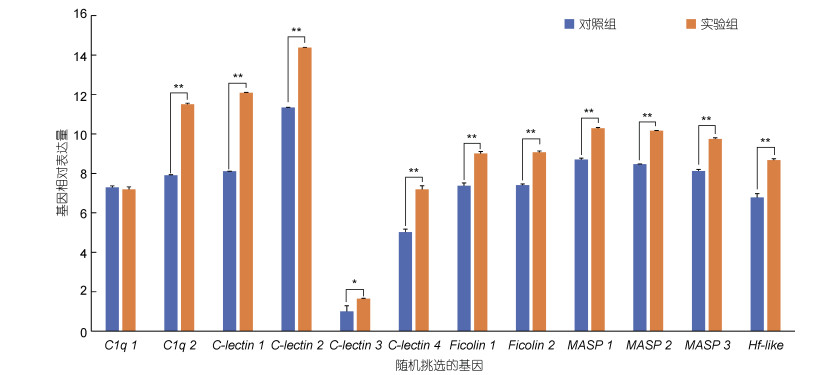 |
| 图 3 马氏珠母贝补体样组分基因表达水平 Fig. 3 Expression level of complement component gene of P. fucata |
补体系统是一种古老的免疫防御机制, 在无脊椎动物抵御病原入侵中发挥重要的免疫作用(Nonaka et al, 2004a)。补体组分是具有特定结构域的分子, 研究表明在哺乳动物补体成分中发现的大多数结构域普遍存在于后口动物, 这表明后口动物体内已经存在由补体分子共同发挥免疫作用的效应系统(Patthy, 2003)。研究表明无脊椎动物的补体系统与脊椎动物的补体系统存在一定差异(Nonaka et al, 2006)。
目前, 关于无脊椎动物补体系统的研究主要集中在海胆(Al-Sharif et al, 1998)、海鞘(Ji et al, 1997)、鲎(Zhu et al, 2005)、珊瑚(Coral) (Dishaw et al, 2005)、太平洋牡蛎(C. gigas) (Wang et al, 2017)等物种中, 而关于马氏珠母贝补体系统的研究尚且处于起步阶段。本研究基于马氏珠母贝全长转录组数据, 系统检索补体相关组分, 结果显示共挖掘到212条补体样成分unigene, 分别编码89个含C1q结构域蛋白、57个C型凝集素蛋白、33个纤维胶凝蛋白、11个纤维蛋白原相关蛋白、8个甘露糖结合型凝集素关联丝氨酸蛋白酶、2个含硫酯蛋白(1个C3分子, 1个TEP分子)、1个补体受体、2个补体因子、9个丝氨酸蛋白酶。这些补体同源分子的发现暗示它们参与构成马氏珠母贝原始的补体系统, 可能具有类似于脊椎动物补体组分相似的免疫学功能。
补体C3作为补体系统的中心组分, 在经典途径、凝集素途径和替代途径中具有枢纽作用, 因此大部分研究人员将C3分子或类似物的出现作为补体系统产生的主要标志(Nonaka et al, 2004b)。在无脊椎动物海参(Apostichopus japonicus) (Zhou et al, 2011)、珊瑚(Swiftia exserta) (Dishaw et al, 2005)、海葵(Nematostella vectensis) (Kimura et al, 2009)、文蛤(Prado-Alvarez et al, 2009)中发现的C3分子具有原始C3的结构特征。补体C3具有独特的链内硫酯键, 可与入侵微生物形成共价键, 促进吞噬作用(Wang et al, 2021)。节肢动物鲎、乌贼、珊瑚中C3样分子的发现, 将其起源推至13亿年前的寒武纪, 表明这类分子可能在后生动物进化的最早期就已具备调理作用(张峘, 2010)。在鲎中发现的C3属于脊椎动物C3的同源分子, 其硫酯键水解后能够促进与金黄色葡萄球菌(Staphylococcus aureus)、大肠杆菌(Escherichia coli)等微生物表面结合(Zhu et al, 2005)。南美白对虾(Litopenaeus vannamei) C3在细菌溶解和溶血方面起着至关重要的免疫作用, 激活包括C3在内的补体途径可能会限制WSSV在对虾体内的复制(Liu et al, 2019)。在前期研究中, 我们成功从马氏珠母贝中克隆得到完整的C3分子, 其能够在体外条件下激活NF-kB信号通路, 特异性的siRNA敲降C3的表达能够显著降低马氏珠母贝血细胞对溶藻弧菌的吞噬作用(Wang et al, 2021)。
替代途径可通过C3分子的激活而启动, 在没有抗体的情况下可通过补充调节因子(H因子和P因子)的相互作用直接结合病原体表面(Wang et al, 2017)。哺乳动物Bf和C2在补体激活过程中具有重要的作用, 其具有相似结构域, 是通过基因复制产生的嵌合蛋白, 包括short consensus repeat (SCR), von Willebrand factor type A (vWA), serine protease domain-containing (SPC) (张峘, 2010)。研究发现Bf和C2仅在两栖动物和哺乳动物中同时存在(Ohta et al, 2006)。目前在无脊椎动物体内发现的具有类似结构域的分子均是Bf和C2的同源分子, 在结构上同哺乳动物存在一定差异。文蛤中Bf因子相比脊椎动物缺少1个SCR结构域, 而海胆Bf具有5个SCR结构域(Smith et al, 1998); 玻璃海鞘(Ciona intestinalis) Bf因子除了具有脊椎动物典型的结构域, 还额外具有2个Low-density lipoprotein receptor (LDLR)和1个SCR (Yoshizaki et al, 2005)。文昌鱼(Branchiostoma belcheri) Bf/C2分子在SCR重复结构域中包含1个表皮生长因子(EGF)结构域, LPS刺激后在肝脏中的表达量显著上升(He et al, 2008)。虽然在马氏珠母贝全长转录组数据中发现了大量的SCR、SPC结构域, 但是没有发现同时具有SCR、SPC结构域的基因, 表明没有典型的Bf/C2分子存在于马氏珠母贝中, 其分子功能仍需要进一步验证。
原始补体组分的功能进化及整合促进了补体系统调理作用的产生(Zhu et al, 2005)。凝集素途径中MASP代替替代途径中Bf/C2的作用直接激活C3 (Fujita et al, 2004)。纤维胶凝蛋白(Ficolin)和甘露糖结合型凝集素(MBL)是识别外源病原体并激活凝集素途径中的关键分子(Gadjeva et al, 2001)。光滑双脐螺和文昌鱼的FREP具有凝集素的活性, 能识别并结合病原微生物, 在先天免疫中发挥着PRR的作用(Fan et al, 2008; Lu et al, 2020)。本研究中从全长转录组中检索到57个C-lectin和8个MASP。C-lectin含有一个或多个C型凝集素结构域(CTLD), 能够与多种配体结合, 包括碳水化合物、脂质、蛋白质、CaCO3等(Huang et al, 2008)。MBL的C型凝集素结构域可识别病原体表面上的甘露糖或N-乙酰氨基葡糖结构, 与活化的MASP形成复合酶, 进而激活补体系统(Ikeda et al, 1987)。缢蛏C型凝集素具有介导凝集与吞噬作用的免疫功能(Lan et al, 2020)。缢蛏C-type lectin具有促进血细胞吞噬细菌的能力, 且这种促吞噬活性是依靠CTLD结构域对细菌的结合实现的(蓝天一, 2018)。凝集素途径在缺乏免疫球蛋白的无脊椎动物中具有重要的免疫作用, 海鞘补体系统的凝集素途径的激活产生C3片段, 有助于吞噬细胞上结合受体进行吞噬作用(Miyazawa et al, 2001)。事实上, 凝集素-MASP复合物在结构和功能上类似于C1复合物, 基于凝集素的补体系统已逐步演变形成经典途径(Matsushita et al, 2004)。
在全长转录组数据中共鉴定到89个C1qDC基因, 其中有81个C1qDC基因含有C末端球状结构域(Globular C1q domain, gC1q), 该结构域作为C1qDC的核心组成部分, 主要功能是作为PRR直接与细胞外膜上的受体相互作用, 介导细胞吞噬和凝集(Gerdol et al, 2011)。大量的基因复制事件导致了C1qDC家族成员的扩增, 这可能是促进经典途径产生的重要原因(Nonaka, 2001)。C1qDC蛋白是经典补体经典途径的起始分子, 是构成C1复合物的重要组分; 当C1qDC分子与免疫球蛋白IgM或IgG的Fc段结合后, C1qDC结构发生变化, 进而导致C1r和C1s的活化, 启动补体经典激活途径(Kishore et al, 2004; Yan et al, 2020)。研究显示C1qDC出现在脊椎动物进化的早期阶段, 在适应性免疫建立和免疫球蛋白出现之前, 原始的C1qDC作为补体系统的初始识别分子发挥免疫作用(Matsushita et al, 2004; Nonaka et al, 2006)。七鳃鳗(Lampetra japonicum) C1qDC分子能够结合广泛分布于细菌的细胞壁中的N-乙酰葡糖胺, 其功能上可能类似于凝集素, 是一种模式识别受体(Matsushita et al, 2004)。太平洋牡蛎C1qDC-5可以作为调理素来识别和结合细菌, 并能与血细胞表面的整联蛋白相互作用, 增强牡蛎血细胞的吞噬作用(Lv et al, 2019); C1qDC-6可以作为模式识别受体, 并介导血细胞的吞噬作用和迁移, 以消除入侵的病原体(Lv et al, 2019); C1qDC-7对LPS和PGN以及革兰阴性细菌具有很高的结合亲和力(Zong et al, 2019)。七鳃鳗C1q-MASP-A复合物可结合病原体上的碳水化合物导致C3的活化及C3b的形成, 沉积在病原体的表面上的C3b具有调理素的作用(Nonaka et al, 1984)。
随机挑选12个补体相关基因进行荧光定量PCR检测, 结果显示其中10个基因的表达水平经溶藻弧菌刺激后显著上升, 包括C1q、C-lectin、MBL、ficolin、MASP、Hf-like等补体基因, 暗示马氏珠母贝的补体系统是一个复杂的免疫效应系统, 包括并不限于这些基因参与到马氏珠母贝的免疫响应过程中。
4 结论本研究基于马氏珠母贝全长转录组数据检索到多种C1qDC、C型凝集素、纤维胶凝蛋白、甘露糖凝集素、甘露糖结合型凝集素关联丝氨酸蛋白酶以及C3等补体相关分子。经实时定量PCR检测显示, 多种补体样基因参与到溶藻弧菌刺激后的免疫响应, 暗示马氏珠母贝补体系统是一个复杂的多组分效应系统。C1q、C-lectin、MBL、ficolin、MASP等基因呈现出显著差异表达, 表明马氏珠母贝可能倾向于通过凝集途径或类似于凝集素途径执行补体系统的先天免疫作用。以上研究结果为探讨马氏珠母贝体内可能存在的补体系统的激活机制提供了研究基础, 对于进一步加深对马氏珠母贝的免疫防御机制的理解、丰富和发展无脊椎动物补体系统的进化过程具有理论意义。
刘先军, 杨丽诗, 黄建华, 等. 2011. 斑节对虾C1q结合蛋白(PmC1qBP)的克隆及表达特征分析. 中国水产科学, 18(4): 774-781 |
吴勇, 张野, 许尤厚, 等. 2019. 马氏珠母贝(Pinctada fucata)血细胞RNA-Seq转录组数据中补体样组分分析. 海洋与湖沼, 50(6): 1343-1353 |
张峘, 2010. 扇贝补体样成分的基因及其功能研究[D]. 青岛: 中国科学院研究生院(海洋研究所): 32-34.
|
陈永霞, 2018. 脂多糖及铜离子胁迫下厚壳贻贝补体C3分子表达研究[D]. 舟山: 浙江海洋大学: 8-12.
|
焦钰, 梁金婵, 谷泽丰, 等. 2020. 脂多糖刺激对马氏珠母贝血细胞DNA甲基化修饰的影响. 广东海洋大学学报, 40(3): 7-13 DOI:10.3969/j.issn.1673-9159.2020.03.002 |
蓝天一, 2018. 缢蛏C型凝集素基因ScCTL-1和ScCTL-2的克隆鉴定与免疫功能研究[D]. 上海: 上海海洋大学: 5-6.
|
AL-SHARIF W Z, SUNYER J O, LAMBRIS J D et al, 1998. Sea urchin coelomocytes specifically express a homologue of the complement component C3. Journal of Immunology, 160(6): 2983-2997 |
CHAO Y H, YUAN J B, GUO T et al, 2019. Analysis of transcripts and splice isoforms in Medicago sativa L. by single-molecule long-read sequencing. Plant Molecular Biology, 99(3): 219-235 DOI:10.1007/s11103-018-0813-y |
DISHAW L J, SMITH S L, BIGGER C H, 2005. Characterization of a C3-like cDNA in a coral: phylogenetic implications. Immunogenetics, 57(7): 535-548 DOI:10.1007/s00251-005-0005-1 |
FAN C X, ZHANG S C, LI L et al, 2008. Fibrinogen-related protein from amphioxus Branchiostoma belcheri is a multivalent pattern recognition receptor with a bacteriolytic activity. Molecular Immunology, 45(12): 3338-3346 DOI:10.1016/j.molimm.2008.04.019 |
FUJITA T, ENDO Y, NONAKA M, 2004. Primitive complement system-recognition and activation. Molecular Immunology, 41(2/3): 103-111 |
GADJEVA M, THIEL S, JENSENIUS J C, 2001. The mannan-binding-lectin pathway of the innate immune response. Current Opinion in Immunology, 13(1): 74-78 DOI:10.1016/S0952-7915(00)00185-0 |
GERDOL M, MANFRIN C, DE MORO G et al, 2011. The C1q domain containing proteins of the Mediterranean mussel Mytilus galloprovincialis: a widespread and diverse family of immune-related molecules. Developmental & Comparative Immunology, 35(6): 635-643 |
GERDOL M, VENIER P, 2015. An updated molecular basis for mussel immunity. Fish & Shellfish Immunology, 46(1): 17-38 |
HE Y N, TANG B, ZHANG S C et al, 2008. Molecular and immunochemical demonstration of a novel member of Bf/C2 homolog in amphioxus Branchiostoma belcheri: implications for involvement of hepatic cecum in acute phase response. Fish & Shellfish Immunology, 24(6): 768-778 |
HUANG S F, YUAN S C, GUO L et al, 2008. Genomic analysis of the immune gene repertoire of amphioxus reveals extraordinary innate complexity and diversity. Genome Research, 18(7): 1112-1126 DOI:10.1101/gr.069674.107 |
HUBER-LANG M, SARMA J V, ZETOUNE F S et al, 2006. Generation of C5a in the absence of C3: a new complement activation pathway. Nature Medicine, 12(6): 682-687 DOI:10.1038/nm1419 |
IKEDA K, SANNOH T, KAWASAKI N et al, 1987. Serum lectin with known structure activates complement through the classical pathway. Journal of Biological Chemistry, 262(16): 7451-7454 DOI:10.1016/S0021-9258(18)47587-4 |
JI X, AZUMI K, SASAKI M et al, 1997. Ancient origin of the complement lectin pathway revealed by molecular cloning of mannan binding protein-associated serine protease from a urochordate, the Japanese ascidian, Halocynthia roretzi. Proceedings of the National Academy of Sciences of the United States of America, 94(12): 6340-6345 DOI:10.1073/pnas.94.12.6340 |
KIMURA A, SAKAGUCHI E, NONAKA M, 2009. Multi-component complement system of Cnidaria: C3, Bf, and MASP genes expressed in the endodermal tissues of a sea anemone, Nematostella vectensis. Immunobiology, 214(3): 165-178 DOI:10.1016/j.imbio.2009.01.003 |
KISHORE U, GABORIAUD C, WATERS P et al, 2004. C1q and tumor necrosis factor superfamily: modularity and versatility. Trends in Immunology, 25(10): 551-561 DOI:10.1016/j.it.2004.08.006 |
LAN T Y, LI Z, PENG M X et al, 2020. A four-CRD C-type lectin from razor clam Sinonovacula constricta mediates agglutination and phagocytosis. Gene, 728: 144287 DOI:10.1016/j.gene.2019.144287 |
LI Y, NIU D H, BAI Y Q et al, 2019. Identification of a novel C1q complement component in razor clam Sinonovacula constricta and its role in antibacterial activity. Fish & Shellfish Immunology, 87: 193-201 |
LIU Y C, SONG Q Z, LI D L et al, 2019. A novel complement C3 like gene (Lv-C3L) from Litopenaeus vannamei with bacteriolytic and hemolytic activities and its role in antiviral immune response. Fish & Shellfish Immunology, 91: 376-387 |
LU L J, LOKER E S, ADEMA C M et al, 2020. Genomic and transcriptional analysis of genes containing fibrinogen and IgSF domains in the schistosome vector Biomphalaria glabrata, with emphasis on the differential responses of snails susceptible or resistant to Schistosoma mansoni. Plos Neglected Tropical Diseases, 14(10): e0008780 DOI:10.1371/journal.pntd.0008780 |
LV Z, WANG L L, JIA Z H et al, 2019. Hemolymph C1qDC promotes the phagocytosis of oyster Crassostrea gigas hemocytes by interacting with the membrane receptor β-integrin. Developmental & Comparative Immunology, 98: 42-53 |
MATSUSHITA M, MATSUSHITA A, ENDO Y et al, 2004. Origin of the classical complement pathway: lamprey orthologue of mammalian C1q acts as a lectin. Proceedings of the National Academy of Sciences of the United States of America, 101(27): 10127-10131 DOI:10.1073/pnas.0402180101 |
MIYAZAWA S, AZUMI K, NONAKA M, 2001. Cloning and characterization of integrin α subunits from the solitary ascidian, Halocynthia roretzi. The Journal of Immunology, 166(3): 1710-1715 DOI:10.4049/jimmunol.166.3.1710 |
NONAKA M, 2001. Evolution of the complement system. Current Opinion in Immunology, 13(1): 69-73 DOI:10.1016/S0952-7915(00)00184-9 |
NONAKA M, FUJII T, KAIDOH T et al, 1984. Purification of a lamprey complement protein homologous to the third component of the mammalian complement system. Journal of Immunology, 133(6): 3242-3249 |
NONAKA M, KIMURA A, 2006. Genomic view of the evolution of the complement system. Immunogenetics, 58(9): 701-713 DOI:10.1007/s00251-006-0142-1 |
NONAKA M, YOSHIZAKI F, 2004a. Evolution of the complement system. Molecular Immunology, 40(12): 897-902 DOI:10.1016/j.molimm.2003.10.009 |
NONAKA M, YOSHIZAKI F, 2004b. Primitive complement system of invertebrates. Immunological Reviews, 198(1): 203-215 DOI:10.1111/j.0105-2896.2004.00118.x |
OHTA Y, GOETZ W, HOSSAIN M Z et al, 2006. Ancestral organization of the MHC revealed in the amphibian Xenopus. Journal of Immunology, 176(6): 3674-3685 DOI:10.4049/jimmunol.176.6.3674 |
PATTHY L, 2003. Modular assembly of genes and the evolution of new functions. Genetica, 118(2/3): 217-231 DOI:10.1023/A:1024182432483 |
PENG M X, NIU D H, CHEN Z Y et al, 2017. Expression of a novel complement C3 gene in the razor clam Sinonovacula constricta and its role in innate immune response and hemolysis. Developmental & Comparative Immunology, 73: 184-192 |
PRADO-ALVAREZ M, ROTLLANT J, GESTAL C et al, 2009. Characterization of a C3 and a factor B-like in the carpet-shell clam, Ruditapes decussatus. Fish & Shellfish Immunology, 26(2): 305-315 |
SEKINE H, KENJO A, AZUMI K et al, 2001. An ancient lectin-dependent complement system in an ascidian: novel lectin isolated from the plasma of the solitary ascidian, Halocynthia roretzi. Journal of Immunology, 167(8): 4504-4510 DOI:10.4049/jimmunol.167.8.4504 |
SMITH L C, SHIH C S, DACHENHAUSEN S G, 1998. Coelomocytes express SpBf, a homologue of factor B, the second component in the sea urchin complement system. Journal of Immunology, 161(12): 6784-6793 |
SUCKALE J, SIM R B, DODDS A W, 2005. Evolution of innate immune systems. Biochemistry and Molecular Biology Education, 33(3): 177-183 DOI:10.1002/bmb.2005.494033032466 |
SUZUKI M M, SATOH N, NONAKA M, 2002. C6-like and C3-like molecules from the cephalochordate, amphioxus, suggest a cytolytic complement system in invertebrates. Journal of Molecular Evolution, 54(5): 671-679 DOI:10.1007/s00239-001-0068-z |
TAKAHASHI K G, KURODA T, MUROGA K, 2008. Purification and antibacterial characterization of a novel isoform of the Manila clam lectin (MCL-4) from the plasma of the Manila clam, Ruditapes philippinarum. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 150(1): 45-52 DOI:10.1016/j.cbpb.2008.01.006 |
WANG L L, ZHANG H, WANG L L et al, 2017. The RNA-seq analysis suggests a potential multi-component complement system in oyster Crassostrea gigas. Developmental & Comparative Immunology, 76: 209-219 |
WANG N, QIN M T, CHEN X H et al, 2019. Molecular cloning of complement component C3 gene from pearl mussel, Hyriopsis cumingii and analysis of the gene expression in response to tissue transplantation. Fish & Shellfish Immunology, 94: 288-293 |
WANG Z L, LIANG X R, LI G Y et al, 2021. Molecular characterization of complement component 3 (C3) in the pearl oyster Pinctada fucata improves our understanding of the primitive complement system in bivalve. Frontiers in Immunology, 12: 652805 DOI:10.3389/fimmu.2021.652805 |
WANG Z L, WANG B, CHEN G et al, 2016. Transcriptome analysis of the pearl oyster (Pinctada fucata) hemocytes in response to Vibrio alginolyticus infection. Gene, 575(2): 421-428 DOI:10.1016/j.gene.2015.09.014 |
YAN F F, ZHOU E X, LIU S et al, 2020. Complement C1q subunit molecules from Xenopus laevis possess conserved function in C1q-immunoglobulin interaction. Developmental & Comparative Immunology, 103: 103532 |
YANG C Y, WANG L L, ZHANG H et al, 2014. A new fibrinogen-related protein from Argopecten irradians (AiFREP-2) with broad recognition spectrum and bacteria agglutination activity. Fish & Shellfish Immunology, 38(1): 221-229 |
YANG J L, WANG L L, ZHANG H et al, 2011. C-type lectin in Chlamys farreri (CfLec-1) mediating immune recognition and opsonization. PLoS One, 6(2): e17089 DOI:10.1371/journal.pone.0017089 |
YOSHIZAKI F Y, IKAWA S, SATAKE M et al, 2005. Structure and the evolutionary implication of the triplicated complement factor B genes of a urochordate ascidian, Ciona intestinalis. Immunogenetics, 56(12): 930-942 DOI:10.1007/s00251-004-0752-4 |
ZHOU Z C, SUN D P, YANG A F et al, 2011. Molecular characterization and expression analysis of a complement component 3 in the sea cucumber (Apostichopus japonicus). Fish & Shellfish Immunology, 31(4): 540-547 |
ZHU Y, THANGAMANI S, HO B et al, 2005. The ancient origin of the complement system. The EMBO Journal, 24(2): 382-394 DOI:10.1038/sj.emboj.7600533 |
ZONG Y N, LIU Z Q, WU Z J et al, 2019. A novel globular C1q domain containing protein (C1qDC-7) from Crassostrea gigas acts as pattern recognition receptor with broad recognition spectrum. Fish & Shellfish Immunology, 84: 920-926 |
 2022, Vol. 53
2022, Vol. 53


