中国海洋湖沼学会主办。
文章信息
- 张旨轩, 王子言, 王泽, 刘岩, 刘松怡, 钱鹏宇, 叶欢, 韩姣姣, 周君, 苏秀榕. 2022.
- ZHANG Zhi-Xuan, WANG Zi-Yan, WANG Ze, LIU Yan, LIU Song-Yi, QIAN Peng-Yu, YE Huan, HAN Jiao-Jiao, ZHOU Jun, SU Xiu-Rong. 2022.
- 基于全基因组重测序技术的浙江近岸海域耐盐金黄色葡萄球菌(Staphylococcus aureus)耐药机制解析
- Analysis of the drug resistance mechanism of Staphylococcus aureus based on whole-genome resequencing technology
- 海洋与湖沼, 53(2): 394-404
- Oceanologia et Limnologia Sinica, 53(2): 394-404.
- http://dx.doi.org/10.11693/hyhz20210900218
文章历史
-
收稿日期:2021-09-24
收修改稿日期:2021-10-25
2. 浙江正合谷生物科技有限公司 浙江宁波 315048
2. Zhejiang Zhenghegu Biotechnology Co., Ltd, Ningbo 315048, China
金黄色葡萄球菌(Staphylococcus aureus)是葡萄球菌科(Staphylococcaceae)最具侵袭性的物种, 近年来成为近岸海域的主要病原菌之一。研究显示近岸海域中的各项指标受到人类活动的影响(王小静等, 2015), 金黄色葡萄球菌可能经由污水排放、地表径流和粪便、表皮脱落等途径进入海域(何艺科, 2018)。海水中的金黄色葡萄球菌被认为是皮肤、眼睛和耳朵疾病的风险指标(Cheung et al, 1990), 体表伤口接触被金黄色葡萄球菌污染的海水可能引起皮肤感染、败血症等疾病的发生(Tong et al, 2015), 还可能通过吞咽等方式感染浴场游客, 因此一直被推荐作为一个新的参数添加到海水质量评估体系中(Ramcharitar et al, 2006)。
金黄色葡萄球菌能够在选择压力下快速适应并传播, 除盐度适应外, 包括耐甲氧西林金黄色葡萄球菌(Methicillin-resistant S. aureus, MRSA)在内的越来越多的耐药型金黄色葡萄球菌被分离出来(Obaidat et al, 2015; Vaiyapuri et al, 2019)。MRSA是当前最常见的耐药病原体之一, 染色体上的mecA基因可产生一种与β-内酰胺类抗生素低结合力的青霉素结合蛋白PBP2α, 因此MRSA在β-内酰胺类抗生素存在的情况下能够继续生长, 从而表现出耐药性(Ibrahim et al, 2021); 有些则是质粒介导获得的耐药性, 通过耐药基因blaZ等产生大量β-内酰胺酶, 使青霉素缓慢失活。耐药性的出现给金黄色葡萄球菌的治疗带来了重大阻碍, 因此深入探究金黄色葡萄球菌的耐药机制和致病机制并找到准确的治疗靶位, 寻找除抗生素之外的治疗手段具有重要意义。
细菌耐药性常采用纸片扩散法、稀释法、E-test法、PCR技术、质谱技术等手段进行研究, 但以上方法均存在一定的局限性, 本研究采用全基因组重测序的方法, 对耐盐和标准金黄色葡萄球菌耐药性相关位点进行检测, 既可以掌握已知的耐药基因还能够预测到耐药性相关的潜在基因, 从而解析耐盐菌株与标准菌株耐药机制的异同(Zankari et al, 2013)。
1 材料与方法 1.1 材料耐盐菌株S. aureus ZS01和不耐盐菌株S. aureus 502A为实验室保存于–80 ℃的菌种, 牛肉膏培养基购自青岛海博生物技术有限公司, 细菌DNA提取试剂盒购于北京全氏金生物技术有限公司, 红霉素、氯霉素和万古霉素购买于上海源叶生物科技有限公司。
1.2 方法 1.2.1 菌株复苏及鉴定冷冻保存的S. aureus ZS01和S. aureus 502A菌株接种于海水牛肉膏液体培养基37 ℃进行复苏, 10 000 r/min离心5 min收集对数生长前期的菌沉淀, 用等量生理盐水稀释后取200 μL菌悬液加入5 mL的海水牛肉膏培养基中。取沉淀用细菌DNA提取试剂盒提取DNA, 16S rRNA通用引物(27F: 5′-AGAGTTTGATCCTGGCTCAG-3′; 1492R: 5′-GGTTACCTTGTTACGACTT-3′)用于菌株鉴定, 测序结果在NCBI数据库(http://www.ncbi.nlm.nih.gov)进行比对, 并采用基因芯片检测试剂盒进一步验证。
1.2.2 抗生素处理分别用400 μg/mL的红霉素、氯霉素和万古霉素对两株菌的转接菌液进行胁迫处理, 于37 ℃培养24 h。根据菌株生长状况以10倍为梯度调整抗生素处理浓度, 最终找到菌株对各抗生素的最高耐受浓度。收集S. aureus ZS01和S. aureus 502A最高耐受抗生素浓度胁迫下的菌沉淀作为重测序样品。
1.2.3 重测序及生物信息学分析将上述样品进行基因组DNA的提取, 利用超声法打断基因组DNA, 然后修复DNA片段末端, 将A碱基添加到DNA片段的3′末端, 完成测序接头拼接, 选择合适的片段进行PCR扩增完成建库, 对文库进行质检。基因组重测序采用HiSeq 4000平台进行, 测序模式为PE 150。对原始数据进行预处理获得有效数据, 利用BWA软件(Li et al, 2009)将读长(reads)比对到参考基因组(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/016/805/GCF_000016805.1_ASM1680v1/GCF_000016805.1_ASM1680v1_genomic.fna.gz), 用SnpEff软件(Cingolani et al, 2012)对检测到的SNPs (单核苷酸多态性)、InDels (插入缺失)以及SVs (结构变异)进行功能注释, 并进行相关基因的KEGG富集分析(https://www.kegg.jp/)。
2 结果 2.1 菌株鉴定及样品制备NCBI数据库比对结果显示两菌株均为金黄色葡萄球菌, 基因芯片检测验证了此结果。S. aureus ZS01和S. aureus 502A在海水牛肉膏固体培养基上生长状态良好, 表面光滑凸起, 颜色为金黄或者白色, 符合金黄色葡萄球菌的生长特征。最终选定320 μg/mL红霉素、40 μg/mL氯霉素和3 μg/mL万古霉素处理S. aureus ZS01和S. aureus 502A, 所得样本分别记为E1、C1、V1和E2、C2、V2。
2.2 重测序 2.2.1 重测序数据统计对S. aureus ZS01及经特定浓度抗生素处理后的S. aureus ZS01 DNA基因组重测序并进行质量控制后, 获得了总长度为7.59 G的数据, 共50 320 502个reads。对S. aureus 502A及经特定浓度抗生素处理后的S. aureus 502A DNA基因组重测序并进行质量控制后, 获得了总长度为7.52 G的数据, 共50 009 758个reads。预处理后, 各样本获得的有效数据占原始数据的比值均大于98.53%, 数据质量≥Q30的部分均在96.65%以上, 测序数据质量良好。通过BWA (Burrows-Wheeler Aligner)软件(v0.7.0)将有效数据与参考基因组进行比对, 比对到参考基因组上的总reads平均比例为71.91%, 覆盖深度不低于30×的碱基所占比例均高于89.22%, 样品有效目标区域的测序平均覆盖度超过435倍。
2.2.2 SNPs的检测及注释S. aureus ZS01和E1、C1、V1与参考基因组进行比较, 分别得到19 203、19 235、19 198、19 233个SNPs位点, S. aureus 502A和E2, C2, V2与参考基因组进行比较, 分别得到19 256、19 173、20 270、20 177个SNPs位点, 所有样本约83%的SNPs突变分布于外显子区域; 13%的SNPs位点位于转录起始位点上游500 bp; 位于转录终止位点下游500 bp的SNPs位点约占3.4%~3.6%, 所有SNPs共导致2 403个基因的突变。每个样品检测到的SNPs位点约有55%是同义突变, 发生起始密码子丢失的SNPs均为6个左右, 终止密码子区域检测到的SNPs数目为71~75。
基于各组的SNPs数据进行主成分分析评估抗生素处理前后菌株的突变程度(图 1)。从PCA图中可以看出, 在经三种抗生素处理后, 不耐盐菌株S. aureus 502A和耐盐菌株S. aureus ZS01均发生了一定程度的突变, 其中S. aureus 502A发生的突变更加显著。经红霉素和万古霉素处理的S. aureus ZS01接近于原始菌株, 经氯霉素处理后则产生了较大程度的突变; 不耐盐菌株S. aureus 502A同样是经氯霉素处理之后发生了较大程度的突变; 且经万古霉素处理后S. aureus ZS01与S. aureus 502A发生的突变有较大差异。
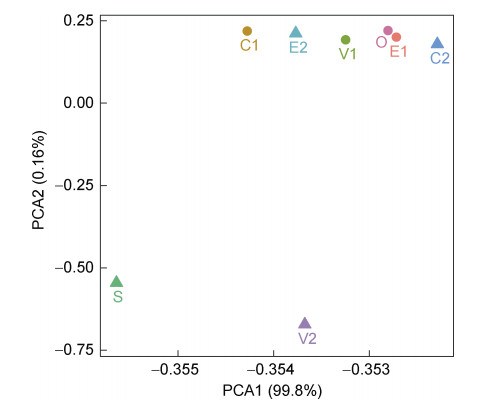 |
| 图 1 基于SNPs的主成分分析图 Fig. 1 Diagram of the principal component analysis based on SNPs 注: O表示耐盐菌株S. aureus ZS01; S表示不耐盐菌株S. aureus 502A; E1, C1, V1, E2, C2, V2依次表示经红霉素、氯霉素、万古霉素处理后的S. aureus ZS01和S. aureus 502A |
S. aureus ZS01经抗生素处理后, 检测到E1发生了53个非同义SNPs, 涉及35个基因, 独有的突变基因6个。C1检测到非同义SNPs位点62个, 涉及43个突变基因, 独有的突变基因有15个。V1处理组发生的非同义SNPs突变位点61个, 涉及36个突变基因, 其中独有突变基因8个。三种抗生素处理后都发生了非同义SNPs突变的基因有19个(图 2), 其中有3个TIGR01741家族蛋白和2个假设蛋白的编码基因, 另外包含了串联型脂蛋白(7个)、聚集因子B、膜蛋白和凝固酶等与金黄色葡萄球侵染宿主相关蛋白的编码基因以及编码青霉素水解A类β-内酰胺酶的耐药基因BlaZ等。
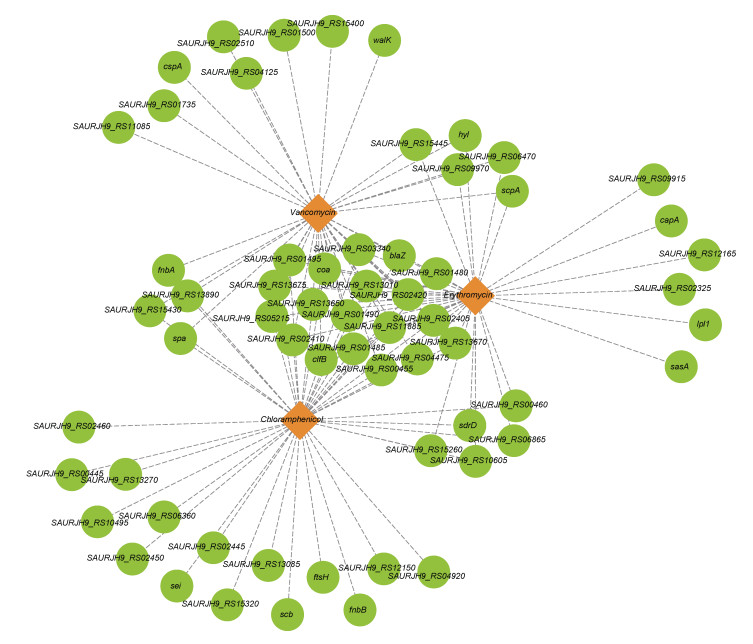 |
| 图 2 抗生素处理后S. aureus ZS01发生非同义SNPs突变的基因 Fig. 2 Genes occurred non-synonymous SNPs in S. aureus ZS01 after antibiotic treatment |
S. aureus 502A经抗生素处理后, 检测到E2发生了50个非同义SNPs, 涉及33个基因, 独有的突变基因18个。C2检测到非同义SNPs位点43个, 涉及31个突变基因, 独有的突变基因有13个。V2处理组发生的非同义SNPs突变位点92个, 涉及63个突变基因, 独有的突变基因43个。三种抗生素处理后都发生了非同义SNPs突变的基因有11个(图 3), 其中有1个TIGR01741家族蛋白和3个假设蛋白的编码基因, 另外包含了串联型脂蛋白、聚集因子A、聚集因子B和β-溶血素等与金黄色葡萄球侵染宿主相关蛋白的编码基因以及编码葡萄球菌蛋白A、DNA指导的RNA聚合酶亚基β的耐药基因rpoB。
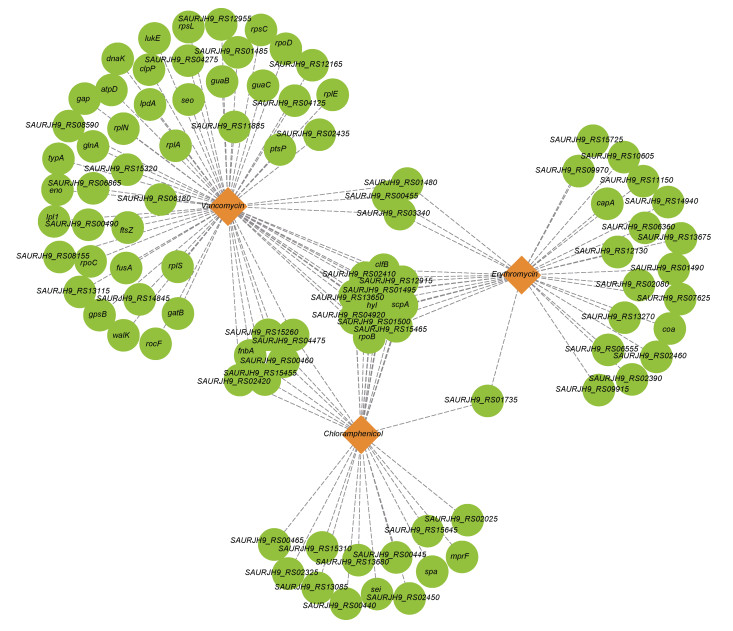 |
| 图 3 抗生素处理后S. aureus 502A发生非同义SNPs突变的基因 Fig. 3 Genes occurred non-synonymous SNPs in S. aureus 502A after antibiotic treatment |
S. aureus ZS01和E1、C1、V1分别检测到622、627、621、625个InDels位点, S. aureus 502A和E2、C2、V2分别检测到630、620、641、658个InDels位点, 主要分布于转录起始位点上游500 bp、转录终止位点下游500 bp, 所占比例约75%, 发生在外显子区域的InDels位点占13%~15%。InDels突变类型中移码突变占比较大, 破坏性编码删除和插入均各有14~18个, 所有InDels突变位点共导致455个基因的突变。
S. aureus ZS01经抗生素处理后, 检测到E1发生了5个基因上的5个InDels突变, 没有独有的基因; C1检测到8个基因的8个InDels突变, 独有的InDels突变基因分别编码荚膜多糖5型生物合成蛋白capA、N-乙酰转移酶、凝集因子B和假设蛋白; V1检测到8个基因的8个InDels突变, 其中MBL折叠金属水解酶、包含CHAP域的蛋白和2个假设蛋白基因上的InDels突变是万古霉素处理组独有的。三种处理均检测到的InDels突变基因有3个, 分别编码串联型脂蛋白、膜蛋白和DNA结合反应调控因子(图 4)。
 |
| 图 4 抗生素处理后S. aureus ZS01发生InDels突变的基因 Fig. 4 Genes occurred InDels in S. aureus ZS01 after antibiotic treatment |
S. aureus 502A经抗生素处理后, 检测到E2发生了5个基因上的6个InDels突变, 4个独有突变基因分别编码TIGR01741家族蛋白、假设蛋白、MSCRAMM家族黏附素SdrE和凝集因子A; C2检测到4个基因的4个InDels突变, 2个独有的InDels突变基因分别编码荚膜多糖5型生物合成蛋白cap5A和MBL折叠金属水解酶; V2检测到4个基因上的4个InDels突变, 其中N-乙酰基转移酶和生物膜操纵子icaADBC HTH型负转录调节因子IcaR蛋白基因上的InDels突变是万古霉素处理组独有的。三种处理均检测到的1个InDels突变基因编码TIGR01741家族蛋白(图 5)。
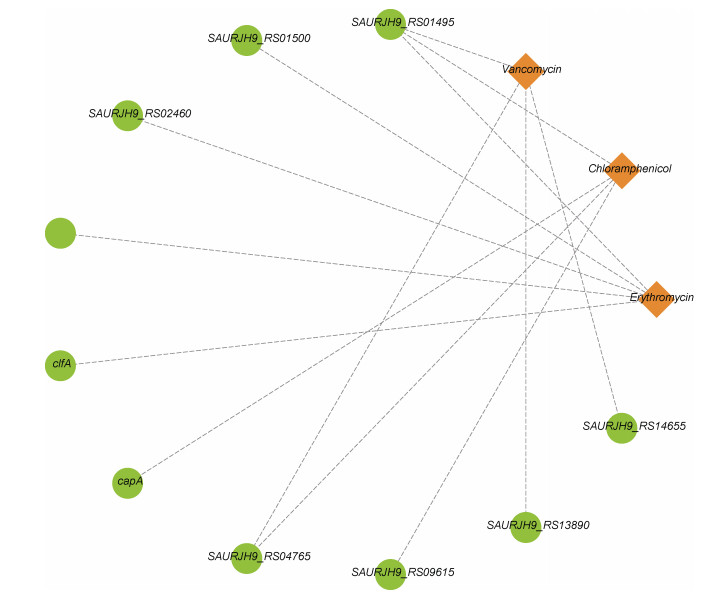 |
| 图 5 抗生素处理后S. aureus 502A发生InDels突变的基因 Fig. 5 Genes occurred InDels in S. aureus 502A after antibiotic treatment |
S. aureus ZS01和E1、C1、V1分别检测到153、154、152、154个SVs位点, S. aureus 502A和E2、C2、V2分别检测到157、152、150、158个SVs位点, 其中缺失类型的结构变异占比范围在53%~58%, 插入类型占21%~24%, 另外各样品分别存在21%~24%的易位类型。所有SVs位点共导致了144个基因的突变。
S. aureus ZS01经抗生素处理后, E1检测到的13个SVs突变涉及11个基因, 其中3个独有突变基因分别编码HPr激酶/磷酸化酶、磷酸烯醇丙酮酸羧激酶和假设蛋白; C1检测到12个基因上发生了13个SVs突变, 其中独有的5个突变基因编码6-磷酸-β-葡糖苷酶、过氧化物酶抑制剂、糖结合转录调节因子、二酰甘油激酶和假设蛋白。V1检测到10个基因上的11个SVs突变, 其中2个独有SVs突变基因编码ISL3家族转座酶和拓扑异构酶。三种抗生素处理都发生SVs突变的基因有5个, 分别编码黄酮素家族蛋白、酰基载体蛋白、DNA结合反应调控因子和2个假设蛋白(图 6)。
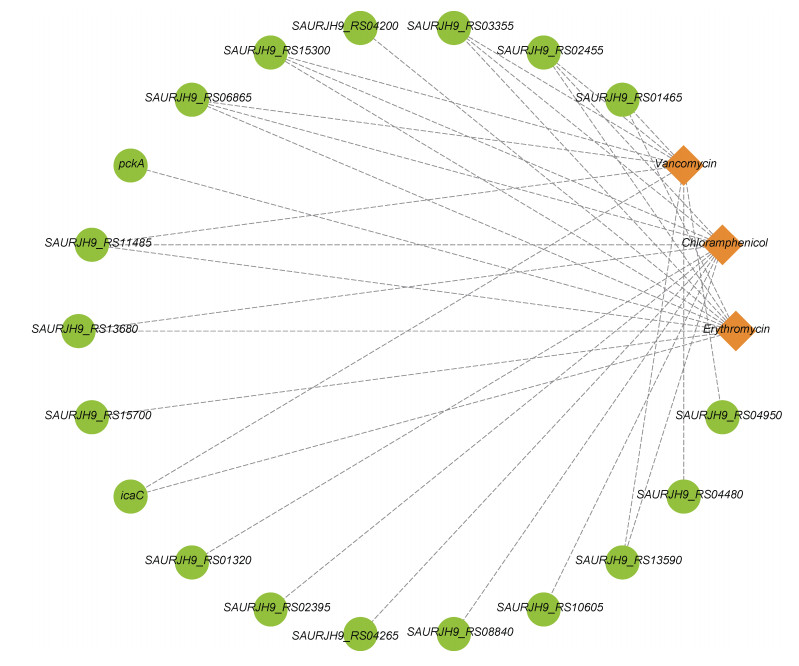 |
| 图 6 抗生素处理后S. aureus ZS01发生SVs突变的基因 Fig. 6 Genes occurred SVs in S. aureus ZS01 after antibiotic treatment |
S. aureus 502A经抗生素处理后, E2检测到的8个SVs突变涉及8个基因, 其中3个独有突变基因分别编码糖结合转录调节子、MutS家族DNA错配修复蛋白和聚集因子B; C2检测到6个基因上的6个SVs突变, 没有独有的SVs突变基因。V2检测到7个基因上的7个SVs突变, 2个独有SVs突变基因编码串联型脂蛋白和假设蛋白。三种抗生素处理都发生SVs突变的基因有2个, 分别编码二肽酶PepV和羧酸酯酶/脂肪酶家族蛋白(图 7)。
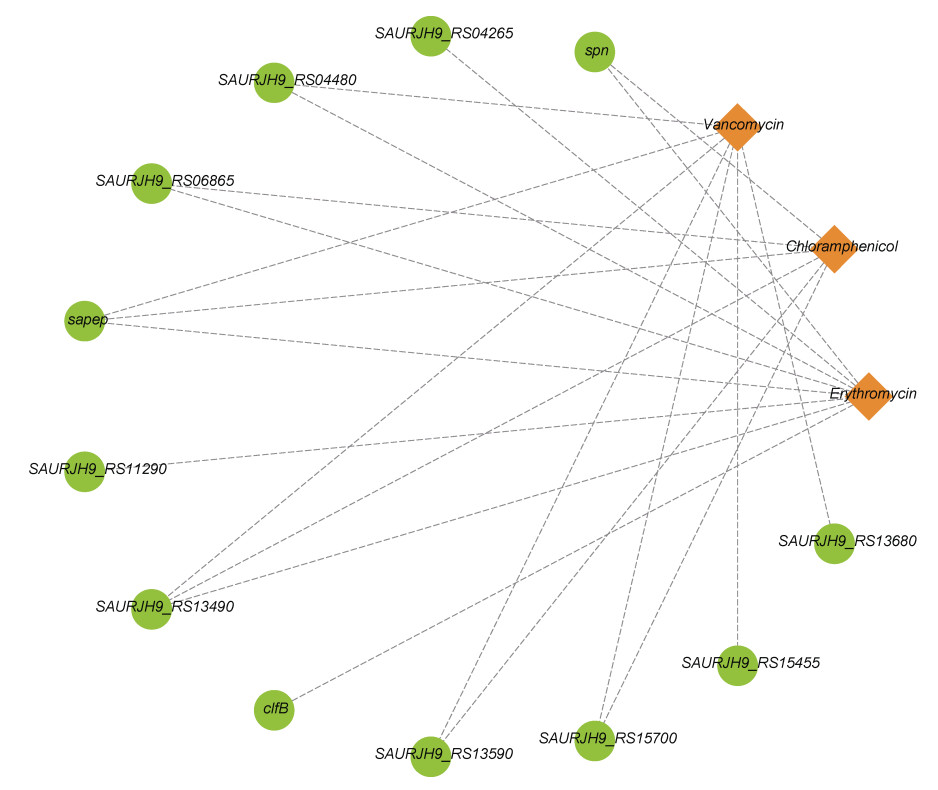 |
| 图 7 抗生素处理后S. aureus 502A发生SVs突变的基因 Fig. 7 Genes occurred SVs in S. aureus 502A after antibiotic treatment |
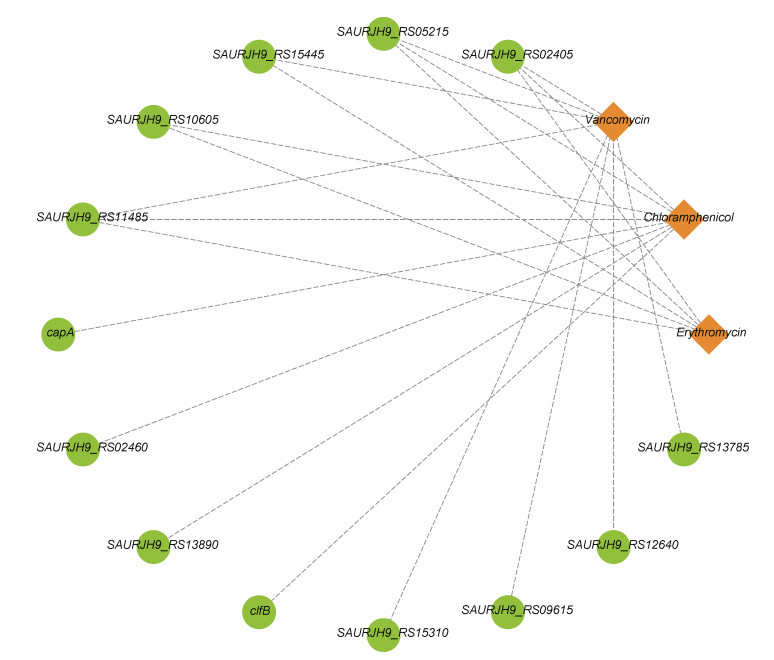
对各处理组检测到的SNPs、InDels和SVs突变涉及的所有基因进行统计, 存在多种类型的突变发生在同一基因上的情况。E1共涉及45个基因的突变, E2涉及42个基因的突变, 其中17个基因是二者共有的。其中串联型脂蛋白和TIGR01741家族蛋白相关突变基因达41%, 另外包含了与致病性相关的荚膜多糖合成蛋白、聚集因子、凝固酶和β-通道形成溶细胞素基因的突变等。与E2相比, E1独有的突变基因中有很多是编码串联型脂蛋白和MSCRAMM家族黏附素的, 且有ABC转运蛋白相关的蛋白基因突变; 而E2独有突变基因中没有用于编码串联型脂蛋白的。C1发生的所有类型突变共涉及54个基因, C2涉及40个基因, 其中二者共有的基因20个, 包含了串联型脂蛋白、膜蛋白、纤连蛋白结合蛋白、聚集因子和荚膜多糖等多个与金黄色葡萄球菌致病性相关的基因; 与C2相比, C1突变中包含了更多编码膜蛋白、纤连蛋白结合蛋白、凝固酶以及MSCRAMM家族黏附素等与金黄色葡萄球菌侵染宿主有关的独有基因。V1发生的突变涉及50个基因, V2涉及72个基因, 其中21个基因是二者共有的, 包含了细胞壁代谢传感器组氨酸激酶WalK以及串联型脂蛋白、纤连蛋白结合蛋白、凝集因子、β-通道形成溶细胞素等致病相关蛋白的编码基因。相比于V1, V2独有的突变基因更多, 且有谷氨酰胺合成酶、RNA聚合酶sigma因子SigA等与细胞壁成分相关的蛋白和多个核糖体结合蛋白编码基因上的突变(图 8)。
 |
| 图 8 编码不同蛋白的突变基因发生突变频次的统计 Fig. 8 Statistics on the frequency of mutations in mutant genes encoding different proteins |
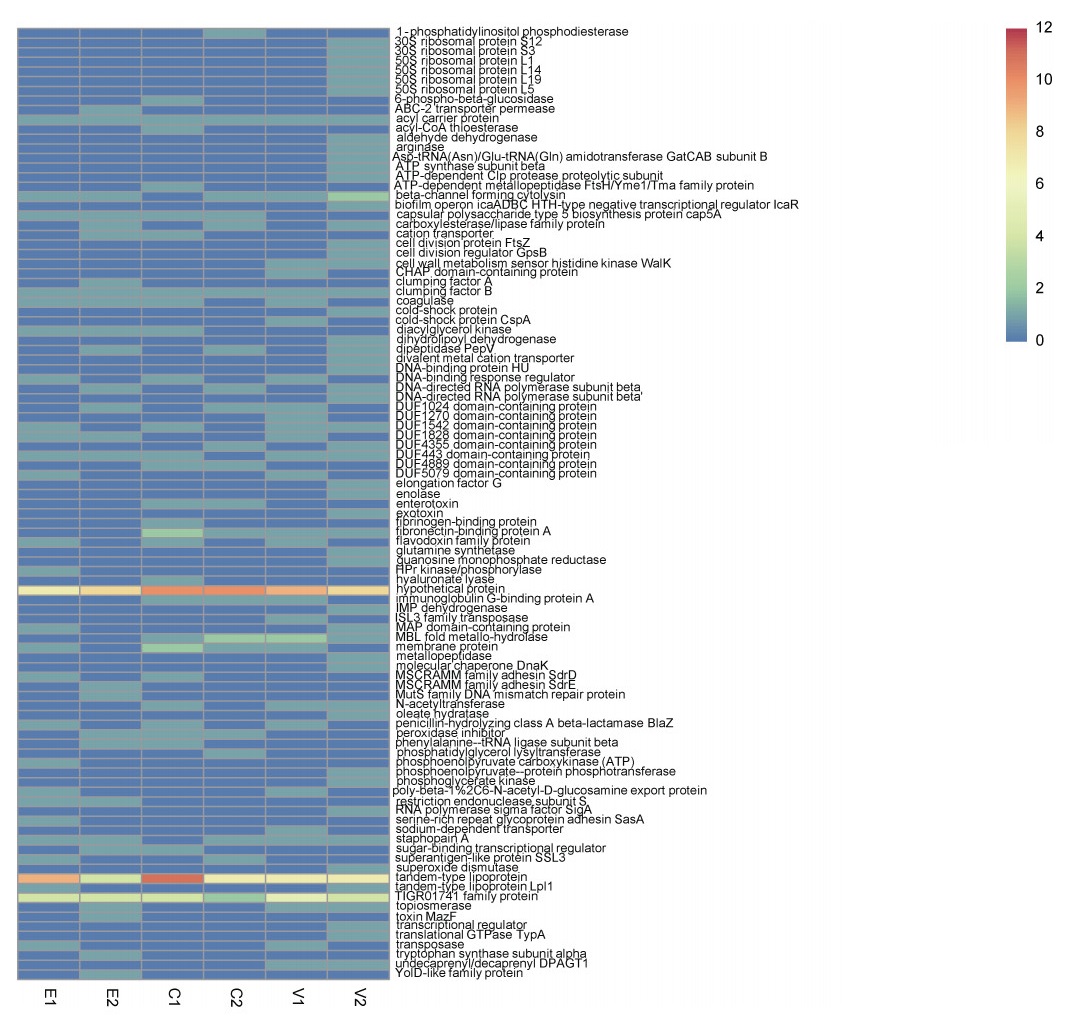
对E1、C1、V1和E2、C2、V2的SNPs、InDels、SVs突变基因进行KEGG富集分析, 分别注释了11、18、11、12、14、37个突变基因, 存在多种突变发生在同一基因上的情况, V2统计到最多的KEGG注释突变基因。分析结果显示突变基因富集于人类疾病, 代谢, 细胞过程, 环境信息处理和基因信息处理5个一级分类, 其中人类疾病和代谢相关通路占有较大比例; 在二级分类中, 6个处理组都有很多突变基因富集在疾病感染通路上, 红霉素和氯霉素处理组有较多突变基因与脂代谢相关(图 9)。
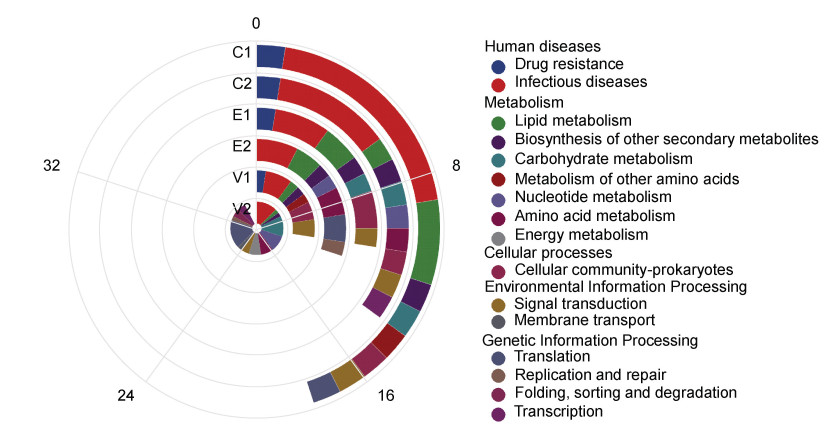 |
| 图 9 E1、E2、C1、C2、V1、V2突变基因的KEGG富集分析 Fig. 9 KEGG enrichment analysis of mutant genes in E1, E2, C1, C2, V1, and V2 |
红霉素处理组中的β-通道形成溶细胞素hyl、MSCRAMM家族黏附素SdrD突变使金黄色葡萄球菌更具有感染性; 氯霉素处理组的纤连蛋白结合蛋白fnbA、fnbB等富集在细菌入侵上皮细胞的通路上, 葡萄球菌蛋白A spA、凝集因子clfA、clfB等也与金黄色葡萄球菌感染相关。万古霉素处理后MBL折叠金属水解酶、spA、凝集因子clfB等基因富集在金黄色葡萄球菌感染通路上。
3 讨论本研究对耐盐菌株S. aureus ZS01和不耐盐菌株S. aureus 502A分别进行红霉素、氯霉素和万古霉素的胁迫处理, 获得最高耐受浓度下的菌沉淀进行重测序, 从而得到耐盐菌株与不耐盐菌株在抗生素耐药机制方面的相似和差异之处。结果表明, S. aureus 502A经抗生素处理后发生突变的程度大于S. aureus ZS01, 说明耐盐菌株本身耐受抗生素的能力可能强于不耐盐菌株; 且相较于红霉素和万古霉素, 在经过氯霉素处理后两株菌发生了较大程度的突变, 出现这种现象的原因可能是氯霉素的使用多于其余两种抗生素, 在氯霉素长期使用过程中, 金黄色葡萄球菌容易发生适应性变化; 万古霉素处理后的两株菌发生突变差异较大。6个处理组都出现了较多串联型脂蛋白基因相关的突变, 引起菌株致病性的变化, 与尚伟龙的研究中阐述的抗生素诱导串联型脂蛋白表达从而增强MRSA的致病性(尚伟龙, 2019)类似。已有研究表明MRSA的耐药性与生物膜的形成能力可能存在相关性(张伟松, 2014), 本研究6个处理组的突变基因中均有部分与脂代谢通路相关, 尤其在红霉素和氯霉素处理组中占比更大, 表明红霉素和氯霉素可能通过影响金黄色葡萄球菌脂类的代谢引起其耐药性的变化, 且处理组都出现了TIGR01741家族蛋白和假设蛋白基因的突变, 因此推测二者与金黄色葡萄球菌的致病性和耐药性相关, 其准确功能有待进一步研究。除此之外, 凝集因子B、纤连结合蛋白结合蛋白、凝固酶、膜蛋白等与金黄色葡萄球菌侵染细胞相关蛋白的基因突变在S. aureus ZS01抗生素处理后均有检出(Liu et al, 2017; Lacey et al, 2019; Li et al, 2019), 这些突变导致了金黄色葡萄球菌毒力状态的改变(图 10), S. aureus 502A经过几种抗生素处理后均检测到DNA依赖性RNA聚合酶rpoB基因上的突变, 这种突变可以抑制一系列抗生素的活性。
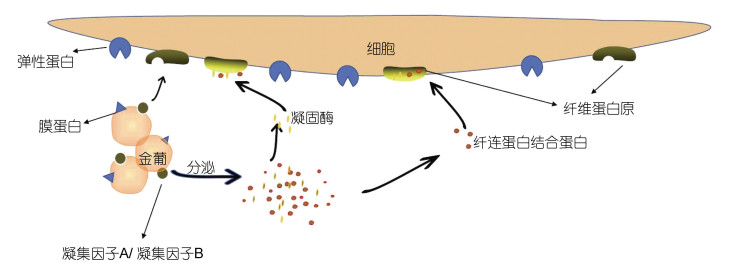 |
| 图 10 金黄色葡萄球菌侵染细胞重要途径 Fig. 10 Important ways of S. aureus infecting cells 注: 聚集因子A、B可与宿主细胞表面的纤维蛋白原特异性结合; 凝固酶等分泌蛋白与细胞表面的纤维蛋白原有较弱的结合能力; 膜蛋白则可与宿主细胞表面的弹性蛋白特异性结合 |
红霉素处理后, 耐盐菌株检测到编码ABC转运蛋白的突变基因, ABC转运蛋白是一种外排系统, 可将抗生素排出体外, 从而产生耐药性(丁兆凤, 2010), 不耐盐菌株则没有检测到相关突变。氯霉素处理后, 耐盐菌株检测到更多编码膜蛋白、纤连蛋白结合蛋白、凝固酶以及MSCRAMM家族黏附素等与金黄色葡萄球菌侵染宿主有关的独有基因, 可能说明氯霉素胁迫后, 耐盐菌株的致病性受到了更大程度的影响。万古霉素处理后, 除毒力因子编码基因的突变外, 还出现了细胞壁代谢传感器组氨酸激酶WalK的突变, 该突变可能导致金黄色葡萄球菌对万古霉素敏感性的降低, 从而增强菌株对万古霉素的耐受能力(Zhu et al, 2021)。V2还检测到谷氨酰胺合成酶、RNA聚合酶sigma因子SigA等与细胞壁成分相关的编码基因突变, 已有研究表明二者在万古霉素耐药性中起着重要作用(代洪等, 2003), V1未检测到相关突变, 所以推测万古霉素处理影响了耐盐菌株和不耐盐菌株侵染宿主的能力, 不耐盐菌株还因细胞壁成分的相关突变提高了万古霉素耐受能力。
4 结论耐盐菌株S. aureus 502A经抗生素处理后发生突变的程度大于标准菌株S. aureus ZS01; 相较于红霉素和万古霉素, 在经过氯霉素处理后两株菌发生了更大程度的突变。红霉素、氯霉素和万古霉素主要影响了金黄色葡萄球菌侵染细胞的能力, 红霉素和氯霉素可能通过影响金黄色葡萄球菌脂类的代谢引起其耐药性的变化。另外, 三种抗生素处理均出现了较多TIGR01741家族蛋白和假设蛋白基因的突变, 推测与菌株的耐药性和致病性相关。耐盐金黄色葡萄球菌可通过外排系统产生红霉素耐药性; 不耐盐菌株还会因为细胞壁成分相关基因的突变提高万古霉素耐受性。
丁兆凤, 2010. 奶牛乳房炎金黄色葡萄球菌红霉素耐药机制分析[D]. 大庆: 黑龙江八一农垦大学: 2-4.
|
王小静, 李力, 高晶晶, 等, 2015. 渤海西南部近岸功能区表层沉积物重金属形态分析及环境评价. 海洋与湖沼, 46(3): 517-525 |
代洪, 范学工, 2003. 金黄色葡萄球菌对万古霉素耐药现状及耐药机制. 国外医学·流行病学传染病学分册, 30(5): 301-304 |
何艺科, 2018. 秦皇岛海滨浴场病原菌分布及其风险评估[D]. 天津: 天津大学: 1.
|
张伟松, 2014. 食源性耐甲氧西林金黄色葡萄球菌的耐药性及生物膜形成能力的研究[D]. 杨凌: 西北农林科技大学: 31-34.
|
尚伟龙, 2019. β-内酰胺类抗生素通过SarA促进串联脂蛋白表达的分子机制及对MRSA致病性的影响[D]. 重庆: 中国人民解放军陆军军医大学: 9.
|
CHEUNG W H S, CHANG K C K, HUNG R P S, et al, 1990. Health effects of beach water pollution in Hong Kong. Epidemiology & Infection, 105(1): 139-162 |
CINGOLANI P, PLATTS A, WANG L L, et al, 2012. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain W1118; iso-2; iso-3. Fly, 6(2): 80-92 DOI:10.4161/fly.19695 |
IBRAHIM M A A, ABDELJAWAAD K A A, ABDELRAHMAN A H M, et al, 2021. Non-β-lactam allosteric inhibitors target methicillin-resistant Staphylococcus aureus: an in silico drug discovery study. Antibiotics, 10(8): 934 DOI:10.3390/antibiotics10080934 |
LACEY K A, MULCAHY M E, TOWELL A M, et al, 2019. Clumping factor B is an important virulence factor during Staphylococcus aureus skin infection and a promising vaccine target. PLoS Pathogens, 15(4): e1007713 DOI:10.1371/journal.ppat.1007713 |
LI H, DURBIN R, 2009. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 25(14): 1754-1760 DOI:10.1093/bioinformatics/btp324 |
LI J N, WANG H, HAN Y X, et al, 2019. Novel peptides screened by phage display peptide library can mimic epitopes of the FnBPA-A protein and induce protective immunity against Staphylococcus aureus in mice. MicrobiologyOpen, 8(10): e910 DOI:10.1002/mbo3.910 |
LIU B, PARK S, THOMPSON C D, et al, 2017. Antibodies to Staphylococcus aureus capsular polysaccharides 5 and 8 perform similarly in vitro but are functionally distinct in vivo. Virulence, 8(6): 859-874 DOI:10.1080/21505594.2016.1270494 |
OBAIDAT M M, SALMAN A E B, LAFI S Q, 2015. Prevalence of Staphylococcus aureus in imported fish and correlations between antibiotic resistance and enterotoxigenicity. Journal of Food Protection, 78(11): 1999-2005 DOI:10.4315/0362-028X.JFP-15-104 |
Ramcharitar J, Gannon D P, Popper A N, 2006. Bioacoustics of fishes of the family Sciaenidae (Croakers and Drums). Transactions of the American Fisheries Society, 135(5): 1409-1431 DOI:10.1577/T05-207.1 |
TONG S Y C, DAVIS J S, EICHENBERGER E, et al, 2015. Staphylococcus aureus infections: epidemiology, pathophysiology, clinical manifestations, and management. Clinical Microbiology Reviews, 28(3): 603-661 DOI:10.1128/CMR.00134-14 |
VAIYAPURI M, JOSEPH T C, RAO B M, et al, 2019. Methicillin-resistant Staphylococcus aureus in seafood: prevalence, laboratory detection, clonal nature, and control in seafood chain. Journal of Food Science, 84(12): 3341-3351 DOI:10.1111/1750-3841.14915 |
ZANKARI E, HASMAN H, KAAS R S, et al, 2013. Genotyping using whole-genome sequencing is a realistic alternative to surveillance based on phenotypic antimicrobial susceptibility testing. Journal of Antimicrobial Chemotherapy, 68(4): 771-777 DOI:10.1093/jac/dks496 |
ZHU J D, LIU B H, SHU X Q, et al, 2021. A novel mutation of walK confers vancomycin-intermediate resistance in methicillin-susceptible Staphylococcus aureus. International Journal of Medical Microbiology, 311(2): 151473 DOI:10.1016/j.ijmm.2021.151473 |
 2022, Vol. 53
2022, Vol. 53


