中国海洋湖沼学会主办。
文章信息
- 栗洋洋, 贾梦雪, 王锦乙, 吴紫琪, 羊妍珂, 严小军, 廖智, 何建瑜. 2022.
- LI Yang-Yang, JIA Meng-Xue, WANG Jin-Yi, WU Zi-Qi, YANG Yan-Ke, YAN Xiao-Jun, LIAO Zhi, HE Jian-Yu. 2022.
- 厚壳贻贝(Mytilus coruscus)中抗生素耐受细菌多样性研究
- DIVERSITY OF ANTIBIOTIC RESISTANT BACTERIA IN MYTILUS CORUSCUS
- 海洋与湖沼, 53(2): 405-413
- Oceanologia et Limnologia Sinica, 53(2): 405-413.
- http://dx.doi.org/10.11693/hyhz20210900199
文章历史
-
收稿日期:2021-09-04
收修改稿日期:2021-11-15
我国是世界上主要的海水养殖大国, 但由于近年来近海环境污染、高密度养殖等因素, 海水养殖生物中细菌性疾病频繁暴发, 造成了重大经济损失(张骞月等, 2015)。由于抗生素被广泛用于水产养殖业中(陈昌福, 2013), 水生动物的细菌性疾病得到抑制, 极大提高了养殖生物在细菌性疾病中的生存率。然而, 抗生素的大量使用以及养殖废水的无序排放致使残余的抗生素在土壤、河口、海洋中不断迁移富集, 导致环境介质中的耐药性细菌出现或者丰度增高(Depaola et al, 1995), 例如嗜水气单胞菌(朱芝秀等, 2012)、鳗弧菌(赵鲁宁等, 2015)、哈维氏弧菌(曾德乾等, 2015)等。河口中的抗生素通常来源于水产养殖废水和未经处理过的生活污水(Han et al, 2020)。其中, 由于长江口特殊的地缘环境, 该海域受人为以及自然因素影响极大, 很容易造成抗生素的累积。同时该区域内存在多个渔业养殖区, 水体或沉积物中抗生素可能对养殖生态造成一定影响(Huang et al, 2021)。既往的研究多集中在环境样本的抗生素抗性基因的调查(郭行磐, 2019), 包括磺胺类、四环素类、大环内酯类等在内的20种抗生素及其抗性基因普遍存在长江口及其邻近水域环境的微生物被膜、沉积物和表层水中。然进一步对抗生素如何影响宿主微生物群落结构的研究仍然比较匮乏。
具有开放式循环系统以及强大滤食性特征的双壳贝类的组织表面常附着大量微生物, 它们不仅参与宿主的生理代谢活动(黄道芬等, 2017; 杨娜, 2018), 还会影响区域内的关键元素循环(Pfister et al, 2010)。因此, 双壳贝类是研究宿主微生物群落结构的一个良好的模型系统。目前已有研究关注贝类体内的耐药性微生物, 例如, 江艳华等(2015)从山东和辽宁沿海地区的养殖海水贝类中分离得到少量致病且具耐药性的副溶血性弧菌。暴露于磺胺甲恶唑(sulfamethoxazole, SMX)的紫贻贝(Mytilus edulis)体内微生物会出现特异的抗生素抗性基因(Serra-Compte et al, 2019)。另一方面, 多重抗生素的联合可能会加速细菌耐药性的产生(Liu et al, 2020)。然耐药性微生物的出现如何影响宿主微生态尤未可知。在此情况下, 利用联合抗生素研究贝类体内的微生物群落结构俨然成为了研究耐药性微生物的一部分。
厚壳贻贝(Mytilus coruscus)隶属于双壳纲(Bivalvia)、贻贝目(Mytiloida)、壳菜蛤科(Mytilidae)、贻贝属(Mytilus), 是中国沿海主要的经济养殖贝类。浙江沿岸的贻贝养殖区主要集中在长江口及其毗邻海域(长江口向东至125°E, 27°30′~33°30′N之间)。由于具有较强的免疫耐受性和环境适应性, 厚壳贻贝不仅成为重要的经济养殖贝类, 更成为一种环境指示生物(阎铁等, 1993; 刘硕博, 2019)。目前对于贻贝微生物的研究多集中在养殖海区的微生物群落分析(李斯远等, 2021), 以及不同组织的微生物种群结构解析(Musella et al, 2020), 对于贻贝体内抗生素耐受微生物类群尚缺乏足够的认识。根据前期研究发现, 贻贝组织微生物以变形菌门为主(Musella et al, 2020; 李斯远等, 2021), 且氨基糖苷类抗生素对变形菌有较强的作用(谢惠民, 1986)。因此为更好地了解多重抗生素影响下厚壳贻贝体内抗性微生物的群落结构变化, 本研究拟采用两种针对革兰氏阴性菌的氨基糖苷类抗生素(链霉素和卡那霉素)和一种针对革兰氏阳性菌的青霉素G类抗生素(青霉素)对厚壳贻贝进行联合刺激。将厚壳贻贝充分暴露于含有多重抗生素的水体中, 以获取其体内微生物的群落结构。为系统了解整体组织微生物的分布情况, 本研究采用超声方式获得贻贝组织超声液, 再利用Zobell 2216E培养基对超声液进行液体培养, 通过16S rDNA高通量测序获得可培养与未培养细菌多样性。最终研究结果将有助于了解厚壳贻贝组织中抗生素耐受细菌群落结构, 服务于我国贻贝养殖业的健康发展。
1 材料与方法 1.1 实验材料2020年12月从浙江省舟山市嵊泗海域采集厚壳贻贝60只[约2年龄, 体长: (98.74±5.86) mm; 体重: (73.6±9.1) g], 立即返回实验室后进行个体分离, 清理贻贝表面附着物后置于洁净的海水中(盐度为25, 温度为18 ℃)进行暂养48 h后, 开展如下实验。
1.2 厚壳贻贝组织超声液的获取与微生物培养为系统了解厚壳贻贝体内组织微生物的多样性, 本研究采用超声法获取厚壳贻贝体内组织表面附着的微生物。在超净工作台上, 用无菌水刷洗贻贝壳表面并去除附着的杂质。用解剖剪将贻贝(N=3)开壳去除体内多余海水, 经无菌水冲洗后分别置于三个无菌烧杯中, 加入无菌海水浸没贻贝, 利用超声波清洗仪(SB-5200DT)进行梯度超声处理(20 ℃, 40 kHz)。梯度超声累计时间分别为1、2、5、10、20、30 min, 每种超声强度下3个生物学重复。为了防止超声引起水体发热, 每超声1 min后暂停1 min。设定超声时间结束后, 将三个烧杯中的超声液混合, 置于离心管中进行多次离心(4 ℃, 10 000 r/min, 5 min), 最终收集约10 mL厚壳贻贝组织超声液, 分别标记后开展液体培养实验和DNA提取。为进一步探究厚壳贻贝组织中可培养微生物的物种分布, 将1 mL上述获取的厚壳贻贝组织超声液加入至5 mL含有Zobell 2216E培养基(ZoBell, 1941)的EP管中, 在恒温培养摇床(QYC-200)中进行恒温震荡培养(160 r/min, 28 ℃), 培养16 h后获得组织微生物培养液, 并在600 nm下测量其吸光度值(OD600)。
1.3 多重抗生素共培养为阐明厚壳贻贝历经抗生素刺激后组织微生物的群落结构变化, 本研究将厚壳贻贝充分暴露于链霉素、卡那霉素和青霉素联合培养的养殖水体中。由于前期文献已知厚壳贻贝体内主要为变形菌门(李斯远等, 2021), 而变形菌门对链霉素更为敏感(谢惠民, 1986)。因此, 多重抗生素组合以链霉素浓度梯度进行, 分别为20 μg/mL (L组)、40 μg/mL (M组)、80 μg/mL (H组), 而青霉素与卡那霉素浓度固定为20 μg/mL。经共培养48 h后, 获取贻贝组织超声液并转入液体培养, 根据最低OD值确定最适抗生素浓度(王敏等, 2020)。随后, 采用确定的抗生素浓度进行刺激, 期间每天换一次含有相同抗生素浓度的海水。刺激6 d后, 换成不添加任何抗生素的无菌海水进行微生物的恢复, 每48 h换一次无菌海水。参考相关文献(孔辉, 2020; Xu et al, 2021)以及前期预实验, 为避免长时间饥饿效应影响宿主微生物群落结构, 本实验中厚壳贻贝组织微生物的恢复时间设置为10 d。以洁净海水养殖的厚壳贻贝作为对照组。每个测量时间点分别取3只贻贝进行解剖获取组织超声液和微生物培养液, 方法同上。
1.4 微生物总DNA提取、文库构建和16S rDNA高通量测序为了解厚壳贻贝组织中抗生素耐受细菌群落结构特征, 采用16S rDNA高通量测序方法研究可培养与未培养细菌多样性。样品总DNA提取参照TIANamp Genomic DNA Kit (DP304, TIANGEN, 中国)试剂盒说明书进行, 并经1.2%琼脂糖凝胶电泳和Nanodrop 2000 (THERMO, 美国)检验合格后, 进行16S rDNA V3~V4区测序文库构建。文库构建采用细菌通用引物B341F (5′-CCTACGGGNGGCWGCAG-3′)和B785R (5′-ACTACHVGGGTATCTAATCC-3′)进行, 扩增体系和反应条件参照KAPA HiFi HotStart PCR Kit (KAPA Biosystems, Roche, 美国)试剂盒说明书。PCR产物经2%琼脂糖凝胶回收纯化后进行文库DNA质量浓度检测, 胶回收方法参照QIAquick Gel Extraction Kit试剂盒(QIAGEN, 德国)说明书进行。DNA浓度采用Qubit Fluorometer (美国)进行, 测序文库的摩尔浓度以KAPA Library Quantification Kit试剂盒(KAPA Biosystems, Roche, 美国)进行定量。测序文库经检验合格后, 采用Illumina MiSeq测序平台(美国)进行两端并行测序(PE250)。微生物16S rDNA高通量测序在浙江杭州开泰生物技术有限公司进行。
1.5 生物信息学分析将获得的原始序列使用软件Vsearch v2.13.6 (Rognes et al, 2016)的fastq_mergepairs模块进行拼接, 利用Cutadapt v2.4去除序列中的标签Barcode, 同时利用Vsearch的fastq_filter模块去除低质量序列、嵌合体序列以及长度小于100 bp的序列, 对得到的高质量序列进行统计。利用MOTHUR (Schloss et al, 2009)对获得的高质量序列按照97%相似度进行OTU (operational taxonomic unite)聚类。OTU的物种注释采用SILVA_132数据库进行。去除低丰度OTU (Reads < 2)后进行抽平, 获得最终OTU_TAX表后分析细菌Alpha多样性, 包括Observed OTU和Shannon指数。菌属重要性利用随机森林(Random forest)回归分析。所有分析均在R v4.0.3环境下调用ggplot2、phyloseq (McMurdie et al, 2013)、microeco (Liu et al, 2021)和vegan (Dixon et al, 2003)软件包进行。
1.6 统计分析培养液OD值的显著性差异和Alpha多样性指数的显著性差异主要采用单因素方差分析(One-way ANOVA), 组间多重比较采用Student-Newman-Keuls (SNK)检验进行。P < 0.05表示具有显著差异。
2 结果 2.1 超声处理后厚壳贻贝组织微生物分布情况通过对厚壳贻贝全组织进行时间梯度超声, 所获得的超声液利用Zobell 2216E培养基进行液体培养, 结果显示OD600值随着超声时间的延长而明显增加, 最高峰值(0.26)出现在累计超声10 min组, 随后在30 min后下降至0.2 (图 1a, ANOVA: df=5, F-value=36.85, P < 0.001)。进一步对10 min和30 min的超声液和微生物培养液进行16S rDNA高通量测序, 结果显示可培养获得的OTU数显著低于未培养获得OTU数(P < 0.01), 仅约20% (图 1b)。在未培养模式下, 超声10 min获得的OTU数和香农指数显著高于超声30 min (P < 0.05); 在可培养模式下, 两种超声时间强度获得的细菌群落的OTU数和香农指数并无明显差别(图 1b)。通过超声方式获得的微生物类群主要为拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia)。利用Zobell 2216E培养后获得的主要微生物群落为变形菌门(图 1c)。
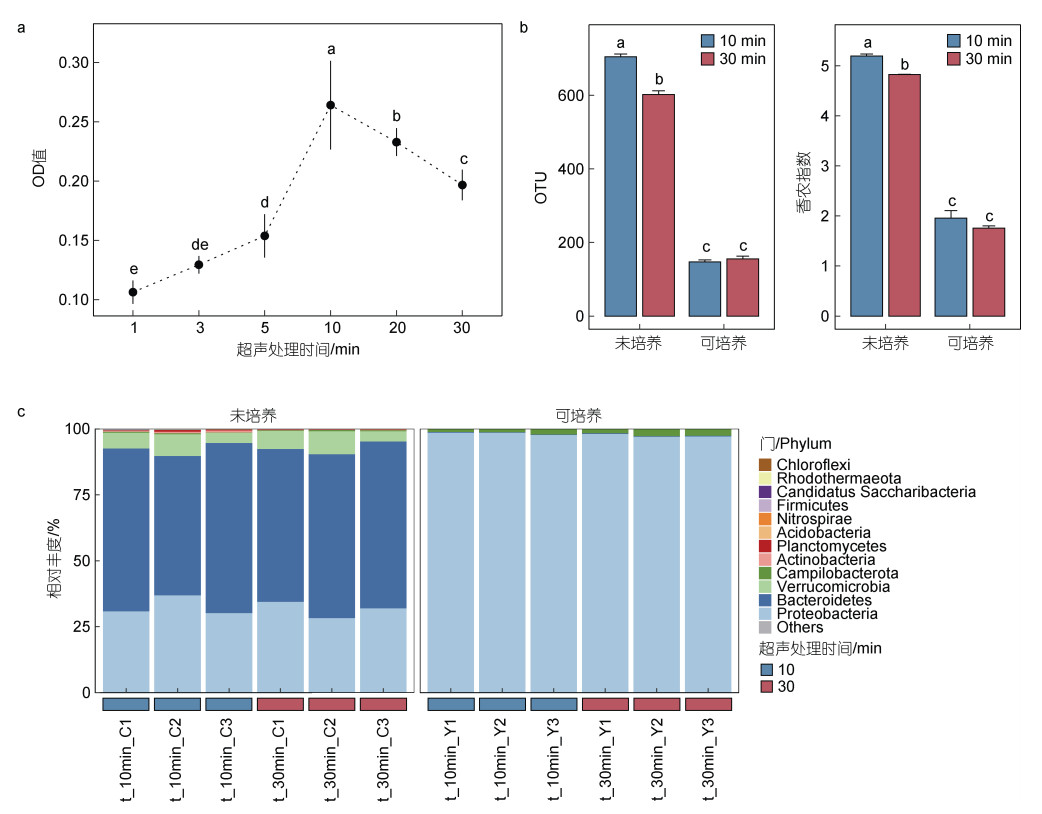 |
| 图 1 超声处理后组织微生物变化情况 Fig. 1 Changes of microorganism in tissue after ultrasonic treatment 注: a. 超声时间梯度下, 超声液在Zobell 2216E培养后的OD值; b. 两种模式下, 可观察到的OTU数和香农多样性指数; c. 微生物门水平下的群落结构组成 |
利用青霉素、链霉素和卡那霉素组成的多重抗生素刺激厚壳贻贝。结果显示(图 2), 相比于对照组, L组(链霉素20 μg/mL)和M组(链霉素40 μg/mL)获得的可培养菌液的OD值显著降低, 而H组(链霉素80 μg/mL)与对照组无显著差异。考虑到链霉素对变形菌门的影响较大, 因此将青霉素20 μg/mL、链霉素40 μg/mL和卡那霉素20 μg/mL作为联抗实验的刺激浓度, 将超声10 min作为获取厚壳贻贝组织微生物超声液的时间强度。
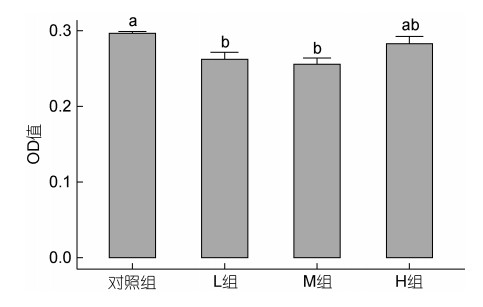 |
| 图 2 多重抗生素处理后可培养菌液的OD值变化情况 Fig. 2 The change of OD value of culturable bacteria solution after combined antibiotic treatment 注: L组: 青霉素20 μg/mL, 链霉素20 μg/mL, 卡那霉素20 μg/mL; M组: 青霉素20 μg/mL, 链霉素40 μg/mL, 卡那霉素20 μg/mL; H组: 青霉素20 μg/mL, 链霉素80 μg/mL, 卡那霉素20 μg/mL |
在实验室养殖过程中, 对照组和联抗组的可培养的微生物菌液OD值都呈现下降趋势(图 3a, 对照组ANOVA: df =3, F-value=106.1, P < 0.001; 联抗组ANOVA: df =4, F-value =38.91, P < 0.001)。在刺激阶段(0~144 h), 对照组和联抗组48 h前的可培养的微生物菌液OD值都呈现下降趋势, 随后, 联抗组的可培养微生物菌液OD值继续下降, 在144 h时OD值达到最低, 但对照组未呈现明显差异。在恢复阶段(144~408 h), 联抗组的可培养微生物菌液OD值呈上升趋势。通过对联抗组细菌16S rDNA高通量测序, 无论是可培养还是未培养模式, 联抗处理(0~144 h) 都显著下调厚壳贻贝组织微生物多样性, 经过264 h恢复后, 细菌多样性有所升高(图 3b)。通过对组织超声液的菌落结构分析可知, 开始时厚壳贻贝组织微生物主要为变形菌门(约75%), 多重抗生素清除了大量的变形菌类群, 致使变形菌门丰度下降至约25% (144 h时), 而拟杆菌门类群的丰度由15%上升至75%。停止抗生素使用后(408 h), 变形菌门的丰度又上升至70%。可培养的微生物基本都为变形杆菌类群(图 3c)。由此可见, 多重抗生素可以改变厚壳贻贝组织微生物的群落结构, 并在受到刺激后自行恢复。
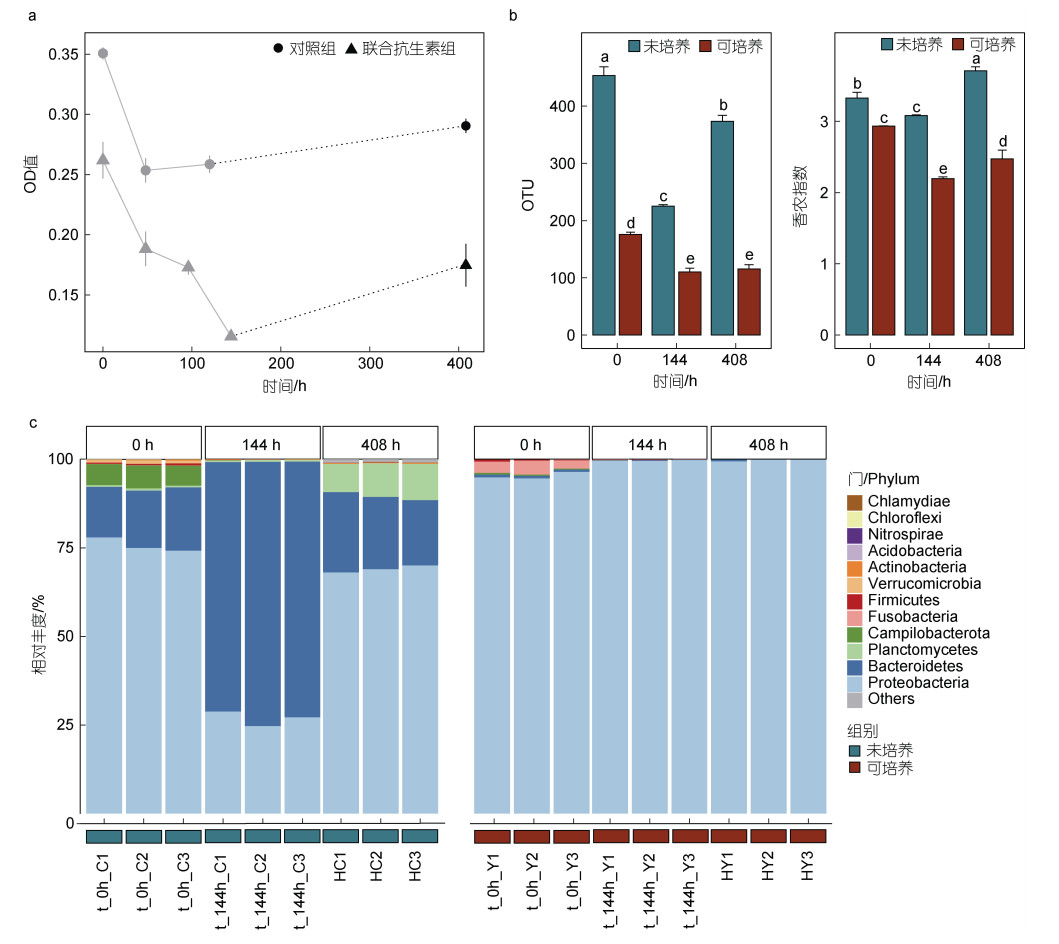 |
| 图 3 多重抗生素处理后组织微生物变化情 Fig. 3 Tissue microorganism changes after combined antibiotic treatment 注: a. 多重抗生素处理时间梯度下, 超声液在Zobell 2216E培养后的OD值。灰色表示对照组, 黑色表示联抗组。b. 两种模式下, 可观察到的OTU数和香农多样性指数。c. 微生物在门水平下的群落结构组成 |
通过随机森林回归模型分析发现在样本中存在显著差异且相对丰度前20的细菌物种(图 4)。在未经培养的超声液中, 假交替单胞菌属(Pseudoalteromonas)在联抗处理144 h后丰度显著上调, 约占全部菌属的0.5%, 当移除抗生素的影响后, 该属的相对丰度下降至0.15%左右。相反, 弧菌属(Vibrio)在未用抗生素处理前相对丰度约为0.18%, 联抗处理后其相对丰度下降(< 0.1%)。当移除抗生素的影响后, 弧菌属的相对丰度显著增高, 超过0.3%。联抗处理144 h后的贻贝超声液经过Zobell 2216E培养, 主要为黄杆菌属(Tenacibaculum, ~0.35%)和Bizionia属(~0.15%)。在洁净海水中恢复264 h后, 可培养的菌属主要为盐单胞菌属(Halomonas)、假单胞菌属(Pseudomonas)和Rheinheimera属, 丰度均大于0.1%。
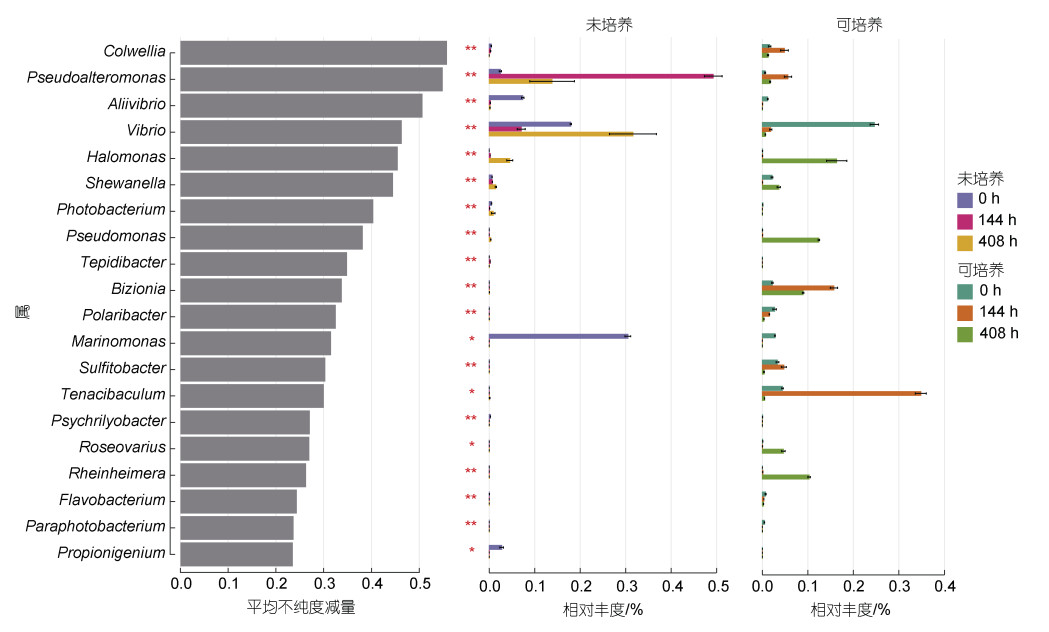 |
| 图 4 基于随机森林分析厚壳贻贝体内对抗生素敏感的重要细菌标志菌属 Fig. 4 The biomarker genus of antibiotic-sensitive bacteria evaluated by Random Forests regression in M. coruscus |
通过对可培养微生物菌液OD值和其多样性进行相关分析, 结果显示菌液OD值和OTU数呈现显著的正相关(R=0.61, P < 0.01, 图 5), 但与香农多样性指数无明显相关关系(P > 0.05, 图 5)。
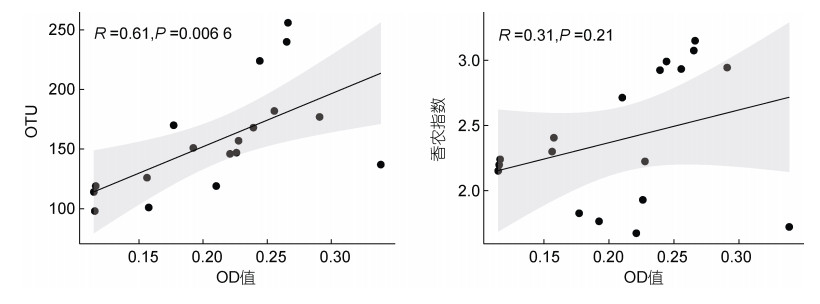 |
| 图 5 可培养细菌多样性指数与培养液OD值的相关性分析 Fig. 5 Correlation analysis between diversity index and OD value of culture medium |
本文主要探讨了多重抗生素刺激下厚壳贻贝体内微生物的群落结构变化。对于动物组织微生物的群落结构研究, 一般采用直接提取组织DNA后, 进行标记基因的高通量测序(韩毛振, 2019)。16S rDNA基因仍然是目前微生物多样性研究中最为广泛使用的标记基因(Yarza et al, 2014)。尽管该方法简单高效, 但存在一定局限性, 主要是受到取样的限制, 通常只能反映出取样位点的微生物结构组成等, 而无法反映整体的组织微生物群落结构。本文基于16S rDNA基因高通量测序方法, 对贻贝超声液和液体培养液进行群落结构分析, 结果显示经Zobell 2216E液体培养, 获得的微生物类群主要是变形菌门。可培养模式下的细菌OTU数约为未培养模式的25%, 且多样性水平也显著低于培养模式。通常可培养模式获得的微生物多样性低于未培养模式, 对液体培养后直接进行高通量测序, 尽管可以获得更多群落信息, 但仍受制于培养条件和培养基(邢磊等, 2017)。
为简单有效地获取厚壳贻贝体内较为全面的微生物群落信息, 本文利用超声波对厚壳贻贝进行原位超声处理, 将获取的超声液进行液体培养, 通过16S rDNA高通量测序揭示厚壳贻贝体内可培养与未培养细菌群落多样性。培养液的OD值随着超声时间强度的增大呈先上升后下降趋势, 并于10 min时达到峰值。16S rDNA高通量测序结果, 相比于超声10 min, 在超声30 min之后细菌多样性显著下降。这可能是因为超声波水中传播会产生空化共振效应, 处理时间过长会对厚壳贻贝表面附着微生物具有破坏作用, 可能破坏微生物细胞壁结构和功能(张婧男等, 2021), 也可能造成微生物菌体被膜脂质结构过氧化和物理断裂(赵伟睿等, 2014), 最终致使微生物死亡。结合OD值与OTU数呈正相关关系, 因此本文选取10 min作为最佳超声时间。由此说明, 通过对超声方式获得贻贝超声液进行测序分析, 可以对厚壳贻贝体内整体群落结构进行简单有效的分析, 但超声时间不宜过长。拟杆菌门、变形菌门和疣微菌门是贻贝超声液中的主要微生物类群, 这与李斯远等(2021)的研究结果基本一致。
为分析厚壳贻贝组织体内抗生素耐受细菌的群落结构, 通过将厚壳贻贝暴露于含有3种抗生素的养殖水体中, 进而研究贻贝体内微生物群落结构变化。在实验初始阶段(0~48 h), 对照组和联抗组的可培养液OD值都存在下降趋势, 这可能与贻贝生存环境改变有关。在洁净海水养殖后, 体内微生物丰度被稀释, 导致部分机会菌群丧失。经过144 h的连续抗生素刺激, 组织超声液的可培养OD值达到最低。利用16S rDNA高通量测序, 结果显示超声液中的细菌OTU数和多样性水平显著下降, 且细菌群落结构发生明显变化, 优势菌群由变形菌门(Proteobacteria)转变为拟杆菌门(Bacteroidetes)。这说明了多种抗生素的联合使用改变了厚壳贻贝体内微生物的群落结构。根据文献资料已知, 青霉素类抗生素主要抑制革兰氏阳性菌细胞壁的合成; 链霉素和卡那霉素作为氨基糖苷类抗生素, 主要抑制革兰氏阴性菌的蛋白质合成(徐进等, 2005)。由于变形菌门主要为革兰氏阴性菌, 在多重抗生素的作用下, 菌群的相对丰度发生明显的变化。Pindling等(2018)将斑马鱼幼体暴露于含链霉素的水体中, 结果发现会链霉素会增加斑马鱼幼体的死亡率, 改变体内微生物的群落结构, 减弱多样性水平, 而且会促使微生物在个体中的差异减小。环境中抗生素或可作为微生物群落之间的信号, 致使微生物种群发生趋同进化(Aminov, 2009), 或将抗生素抗性基因水平转移至其他菌株(Mishra et al, 2021), 最终致使单一类群(拟杆菌门)丰度显著上升。
进一步通过随机森林回归分析, 结果发现假交替单胞菌属在抗生素刺激后丰度显著上升, 而弧菌属出现相反的趋势。假交替单胞菌属是水产养殖生物和环境中常见的一类菌群, 且比较容易发生抗生素耐受。例如黄志坚等(2012)从湛江、珠海、湖南、海南等地的水产养殖生物和环境中分离得到的139株细菌进行抗生素抗性基因鉴定, 发现链霉素抗性基因(str)阳性占比率为10.07%。因此, 尽管受三种抗生素持续胁迫约一周, 贻贝体内依然存在一定数量的微生物。Kerry等(1996)比较了水产养殖池和未使用抗生素的河流, 结果发现养殖池中抗四环素和氯霉素的抗性细菌数量显著增加。这从侧面反映了环境或贻贝组织中存在抗生素耐受细菌, 可能是由于微生物存在内在抗性基因, 或随机突变表达潜在抗性基因而导致耐受现象的出现(Davies et al, 2010; 苏建强等, 2013)。当环境中没有抗生素胁迫压力后, 厚壳贻贝体内微生物丰度重新上升, 多样性水平增加, 在门水平的群落结构趋向于初始状态, 这可能暗示贻贝体内微生物具有一定韧性, 可以自发恢复到初始群落状态。虽然本文并未对微生物的抗性基因进行解析, 因而无法判断相关抗生素抗性基因的种类和丰度, 但贻贝组织微生物群落重建的内在机制是一个值得研究的方向。这将有助于理解贝类微生物定植以及群落发生的机理, 同时有助于理解水产动物细菌耐药性的起源和扩散机制。
4 结论与展望本文采用抗生素胁迫策略, 结合超声方式获取贻贝组织附生微生物菌群, 通过16S rDNA测序和生物信息学手段, 对厚壳贻贝组织微生物对抗生素的耐受性及其群落结构在抗生素处理条件下的动态变化进行了研究。研究结果证实了贻贝组织中存在抗生素耐受菌群; 表明了贻贝组织微生物菌落具有较强的自我重构和自我恢复能力。上述研究结果一方面为了解贻贝在当前环境水体抗生素残留背景下, 其体内微生物群落结构的变化以及由此带来对贻贝养殖业和食品安全的可能影响奠定了基础; 另一方面, 也为深入了解贻贝-微生物之间的生态关联以及这种关联对贻贝生存的重要作用提供了新的研究思路。
王敏, 黄晨, 隋蕾, 等, 2020. 兴庆湖中典型抗生素污染特征及其生态风险评价. 西安理工大学学报, 36(1): 17-24 |
孔辉, 2020. 海洋酸化和缺氧对贻贝性腺功能和幼体发育的交互影响作用[D]. 上海: 上海海洋大学: 12.
|
邢磊, 赵圣国, 郑楠, 等, 2017. 未培养微生物分离培养技术研究进展. 微生物学通报, 44(12): 3053-3066 |
朱芝秀, 何后军, 邓舜洲, 等, 2012. 嗜水气单胞菌江西地区分离株耐药性及耐药质粒分析. 江西农业大学学报, 34(6): 1262-1268 DOI:10.3969/j.issn.1000-2286.2012.06.031 |
刘硕博, 2019. 厚壳贻贝含硒GPx克隆及在Cu2+和LPS胁迫下的表达[D]. 舟山: 浙江海洋大学: 8.
|
江艳华, 姚琳, 李君, 等, 2015. 养殖海水贝类中副溶血性弧菌的致病性及耐药性分析. 食品安全质量检测学报, 6(9): 3474-3479 |
苏建强, 黄福义, 朱永官, 2013. 环境抗生素抗性基因研究进展. 生物多样性, 21(4): 481-487 |
李斯远, 何治江, 吕泓玥, 等, 2021. 厚壳贻贝(Mytilus coruscus)养殖海域与天然生长海域的微生物群落比较研究. 海洋与湖沼, 52(1): 196-205 |
杨娜, 2018. 厚壳贻贝肠道细菌群落组成及肠道细菌对稚贝附着的作用研究[D]. 上海: 上海海洋大学: 43.
|
张婧男, 刘昊天, 陈倩, 等, 2021. 超声技术抑制微生物生物膜污染: 机制、影响因素及其在肉及肉制品中的应用. 肉类研究, 35(5): 50-59 |
张骞月, 赵婉婉, 吴伟, 2015. 水产养殖环境中抗生素抗性基因污染及其研究进展. 中国农业科技导报, 17(6): 125-134 |
陈昌福, 2013. 我国抗微生物水产用兽药使用情况、存在问题与解决途径(中). 当代水产, 38(8): 68-69 DOI:10.3969/j.issn.1674-9049.2013.08.018 |
赵伟睿, 胡升, 黄俊, 等, 2014. 微生物细胞通透性改善方法与策略. 中国生物工程杂志, 34(3): 125-131 |
赵鲁宁, 李贵阳, 李杰, 等, 2015. 海水养殖鱼类鳗弧菌(Vibrio anguillarum)临床分离株的血清型和抗生素耐药谱. 海洋与湖沼, 46(5): 1109-1118 |
徐进, 刘秀梅, 2005. 国内保健食品常用益生菌株的耐药性分析. 中国食品卫生杂志, (2): 108-112 DOI:10.3969/j.issn.1004-8456.2005.02.005 |
郭行磐, 2019. 长江口滨岸水环境中抗生素抗性基因的赋存特征[D]. 上海: 华东师范大学: 1.
|
黄志坚, 陈旭凌, 路晓峰, 等, 2012. 水产养殖生物和养殖环境细菌鉴定及抗生素抗性基因检测. 中山大学学报(自然科学版), 51(6): 92-96 |
黄道芬, 梁箫, 彭莉华, 等, 2017. 不同来源海洋弧菌微生物被膜对厚壳贻贝稚贝附着的影响. 水产学报, 41(7): 1140-1147 |
阎铁, 吕海晶, 1993. 贻贝在海洋污染生物监测中的应用. 海洋通报, 12(3): 117-125 |
韩毛振, 2019. 微生物组大数据的时空动态建模和应用[D]. 武汉: 华中科技大学: 1-3.
|
曾德乾, 冯娟, 徐力文, 等, 2015. 海水养殖鱼哈维弧菌分离株的耐药谱型分析. 中国水产科学, 22(1): 129-138 |
谢惠民, 1986. 氨基甙类抗生素的合理应用. 山东医药, (1): 30-31 |
AMINOV R I, 2009. The role of antibiotics and antibiotic resistance in nature. Environmental Microbiology, 11(12): 2970-2988 DOI:10.1111/j.1462-2920.2009.01972.x |
DAVIES J, DAVIES D, 2010. Origins and evolution of antibiotic resistance. Microbiology and Molecular Biology Reviews, 74(3): 417-433 DOI:10.1128/MMBR.00016-10 |
DEPAOLA A, PEELER J T, RODRICK G E, 1995. Effect of oxytetracycline-medicated feed on antibiotic resistance of gram-negative bacteria in catfish ponds. Applied and Environmental Microbiology, 61(6): 2335-2340 DOI:10.1128/aem.61.6.2335-2340.1995 |
DIXON P, 2003. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science, 14(6): 927-930 DOI:10.1111/j.1654-1103.2003.tb02228.x |
HAN Q F, ZHAO S, ZHANG X R, et al, 2020. Distribution, combined pollution and risk assessment of antibiotics in typical marine aquaculture farms surrounding the Yellow Sea, North China. Environment International, 138: 105551 DOI:10.1016/j.envint.2020.105551 |
HUANG A R, YAN M T, LIN J J, et al, 2021. A review of processes for removing antibiotics from breeding wastewater. International Journal of Environmental Research and Public Health, 18(9): 4909 DOI:10.3390/ijerph18094909 |
KERRY J, COYNE R, GILROY D, et al, 1996. Spatial distribution of oxytetracycline and elevated frequencies of oxytetracycline resistance in sediments beneath a marine salmon farm following oxytetracycline therapy. Aquaculture, 145(1/4): 31-39 |
LIU C, CUI Y M, LI X Z, et al, 2021. Microeco: an R package for data mining in microbial community ecology. FEMS Microbiology Ecology, 97(2): fiaa255 DOI:10.1093/femsec/fiaa255 |
LIU J F, GEFEN O, RONIN I, et al, 2020. Effect of tolerance on the evolution of antibiotic resistance under drug combinations. Science, 367(6474): 200-204 DOI:10.1126/science.aay3041 |
MCMURDIE P J, HOLMES S, 2013. Phyloseq: an R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One, 8(4): e61217 DOI:10.1371/journal.pone.0061217 |
MISHRA S, KLÜMPER U, VOOLAID V, et al, 2021. Simultaneous estimation of parameters governing the vertical and horizontal transfer of antibiotic resistance genes. Science of the Total Environment, 798: 149174 DOI:10.1016/j.scitotenv.2021.149174 |
MUSELLA M, WATHSALA R, TAVELLA T, et al, 2020. Tissue-scale microbiota of the Mediterranean mussel (Mytilus galloprovincialis) and its relationship with the environment. Science of the Total Environment, 717: 137209 DOI:10.1016/j.scitotenv.2020.137209 |
PFISTER C A, MEYER F, ANTONOPOULOS D A, 2010. Metagenomic profiling of a microbial assemblage associated with the California mussel: a node in networks of carbon and nitrogen cycling. PLoS One, 5(5): e10518 DOI:10.1371/journal.pone.0010518 |
PINDLING S, AZULAI D, ZHENG B, et al, 2018. Dysbiosis and early mortality in zebrafish larvae exposed to subclinical concentrations of streptomycin. FEMS Microbiology Letters, 365(18): fny188 |
ROGNES T, FLOURI T, NICHOLS B, et al, 2016. VSEARCH: a versatile open source tool for metagenomics. PeerJ, 4: e2584 DOI:10.7717/peerj.2584 |
SCHLOSS P D, WESTCOTT S L, RYABIN T, et al, 2009. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 75(23): 7537-7541 DOI:10.1128/AEM.01541-09 |
SERRA-COMPTE A, SÁNCHEZ-MELSIÓ Á, ÁLVAREZ- MUÑOZ D, et al, 2019. Exposure to a subinhibitory sulfonamide concentration promotes the spread of antibiotic resistance in marine blue mussels (Mytilus edulis). Environmental Science & Technology Letters, 6(4): 211-215 |
XU G E, KONG H, CHANG X Q, et al, 2021. Gonadal antioxidant responses to seawater acidification and hypoxia in the marine mussel Mytilus coruscus. Environmental Science and Pollution Research International, 28(38): 53847-53856 DOI:10.1007/s11356-021-14584-0 |
YARZA P, YILMAZ P, PRUESSE E, et al, 2014. Uniting the classification of cultured and uncultured bacteria and archaea using 16S rRNA gene sequences. Nature Reviews Microbiology, 12(9): 635-645 DOI:10.1038/nrmicro3330 |
ZOBELL C E, 1941. Studies on marine bacteria.I. The cultural requirements of heterotrophic aerobes. Journal of Marine Research, 4: 42-75 |
 2022, Vol. 53
2022, Vol. 53


