
中国海洋湖沼学会主办。
文章信息
- 陈辙聿, 陶怡曦, 叶勤, 王果, 王文波, 范士琦, 何春, 李云. 2022.
- CHEN Zhe-Yu, TAO Yi-Xi, YE Qin, WANG Guo, WANG Wen-Bo, FAN Shi-Qi, HE Chun, LI Yun. 2022.
- 三峡库区短颌鲚(Coilia brachygnathus)新建种群的年龄结构、生长特征及遗传多样性分析
- AGE STRUCTURE, GROWTH CHARACTERISTICS AND GENETIC DIVERSITY OF NEWLY-ESTABLISHED POPULATION OF SHORTJAW ANCHOVY (COILIA BRACHYGNATHUS) IN THE THREE GORGES RESERVOIR AREA
- 海洋与湖沼, 53(2): 430-439
- Oceanologia et Limnologia Sinica, 53(2): 430-439.
- http://dx.doi.org/10.11693/hyhz20210900217
文章历史
-
收稿日期:2021-09-22
收修改稿日期:2021-11-29
2. 西南大学淡水鱼类资源与生殖发育教育部重点实验室 重庆市水产科学重点实验室 重庆 400715;
3. 西南大学化学化工学院 重庆 400715
2. Key Laboratory of Freshwater Fish Reproduction and Development (Ministry of Education), Key Laboratory of Aquatic Science of Chongqing, Southwest University, Chongqing 400715, China;
3. School of Chemistry and Chemical Engineering, Southwest University, Chongqing 400715, China
短颌鲚(Coilia brachygnathus)隶属鲱形目(Clupeiforms)、鳀科(Engraulidae)、鲚属(Coilia), 在长江流域主要分布在中下游地区。过去曾将短颌鲚视为洄游型刀鲚的淡水生态型种群(郭弘艺等, 2010; 周晓犊等, 2010), 最近有研究认为两者间分化显著, 互为有效种(Cheng et al, 2019; Xuan et al, 2021)。在淡水生态系统中, 短颌鲚承接着食物网的上下级, 以浮游动物、虾及小型鱼类为食, 同时又是大型食鱼性鱼类的食物, 对维持生态系统稳定起关键作用(Zhang et al, 2013)。2014~2018年, 课题组在长江上游三峡库区巫山江段采集到24尾短颌鲚个体(Yang et al, 2019), 2019~2020年, 我们在三峡库区采集到153尾短颌鲚个体。依据历史文献, 长江上游地区无鲚属鱼类分布的记录, 库区发现的短颌鲚群体应该是长江上游的新建种群。短短数年时间, 短颌鲚种群规模不断扩张, 目前已成为三峡水库常见鱼类。
目前, 关于短颌鲚的报道大多集中在物种有效性(Yang et al, 2010; 姜涛等, 2020)、生活史(徐钢春等, 2014; 陈婷婷等, 2016)、生理学(范海霞等, 2016)和资源现状(胡茂林等, 2011)上。先前有研究报道了长江水系短颌鲚种群的年龄组成(王旭歌等, 2016)、生长特征(Qin et al, 2017)和遗传参数(汪焕等, 2020; 轩中亚等, 2020)等, 但这些研究的样品均采自长江中下游水域, 无法反映上游新出现短颌鲚种群的种质资源现状。微卫星DNA标记(simple sequence repeat, SSR)属共显性标记, 具有多态性高、重复稳定性好等特点, 已被广泛应用于水生野生生物群体间的遗传多样性比较和群体发展趋势研究。马克异等(2011)运用10个微卫星位点对钱塘江流域的野生日本沼虾(Macrobrachium nipponense)群体进行研究, 发现新安江电站和富春江电站的阻隔已使钱塘江上游和中下游的日本沼虾群体分化为2个种群, 且中下游水域日本沼虾的遗传多样性有高于上游的趋势。Zhang等(2010)通过11个微卫星位点对长江干流和支流雅砻江的圆口铜鱼(Coreius guichenoti)群体进行研究, 发现二滩水电站、三峡水电站和葛洲坝水电站的阻隔可能加快了圆口铜鱼群体间的分化。
为探究长江上游新建短颌鲚种群的来源, 推测其未来发展趋势, 本研究对三峡库区采集到短颌鲚个体的年龄结构、生长特征进行研究, 通过微卫星DNA标记分析三峡库区野生短颌鲚种群的遗传多样性, 探讨种群遗传结构, 以期为三峡库区水生生态系统演变和长江上游短颌鲚资源生物学研究与保护提供基础数据。
1 材料与方法 1.1 样本采集短颌鲚各采样点详细信息如表 1所示。2019~ 2020年, 在三峡库区共采集到153尾短颌鲚个体。使用量鱼板和电子天平分别测量短颌鲚体长L (精确至0.1 mm)和体重W (精确至0.01 g), 取背鳍基部下方鳞片作为年龄鉴定材料, 并剪取背鳍下方肌肉于95%乙醇中保存备用。
| 采样点 | 缩写 | 纬度(N) | 经度(E) | 个体数 | 体长范围/mm |
| 涪陵 | FL | 29°42′46″ | 107°24′11″ | 19 | 172.1~283.4 |
| 丰都 | FD | 29°53′19″ | 107°44′35″ | 14 | 134.9~235.5 |
| 万州 | WZ | 30°49′07″ | 108°23′24″ | 55 | 102.1~289.0 |
| 巫山 | WS | 31°04′11″ | 109°52′55″ | 65 | 128.5~301.6 |
鳞片在盛有清水的烧杯中浸泡12 h后取出, 用镊子刮除表皮黏膜并洗净, 夹在两块载玻片之间保存。将制备好的载玻片置于Nikon SMZ800体式显微镜下观察, 若年轮数为n, 且年轮外侧无新的环片出现, 则计年龄为n, 若年轮外侧有新环片出现则计年龄为n+。年龄0+~1计为1龄鱼, 1+~2计为2龄鱼, 依此类推。全部鳞片由2名观察者分别读数, 若二者读数一致则采用此结果, 否则舍弃该个体。
1.3 DNA提取采用传统苯酚-氯仿抽提法对153尾短颌鲚的肌肉组织进行基因组DNA提取。使用NanoPhotometer N50分光光度计检测所提取DNA的纯度和浓度, 并使用1 %琼脂糖凝胶电泳检测其完整性, 随后转入–20 ℃保存备用。
1.4 微卫星DNA标记实验使用的10个微卫星DNA标记见表 2, 其中标记DJ41、DJ516、DJ578来自陈芳(2012)为刀鲚(Coilia nasus)设计的引物, 另外7个标记来自Yang等(2014)为刀鲚(Coilia nasus)设计的引物。微卫星DNA标记的引物由上海生工生物工程有限公司合成, 所有上游引物的5′端添加FAM或HEX荧光修饰合成荧光引物。利用合成的荧光引物和153尾短颌鲚的基因组DNA进行PCR扩增, 反应体系为25 μL, 包括DNA模板1 μL (50 ng/μL), 上下游引物各1 μL (10 μmol/L), 22 μL 1.1×T3 Super PCR Mix (TSINGKE公司)。PCR反应程序: 98 ℃预变性2 min; 98 ℃变性10 s, 退火10 s, 72 ℃延伸10 s, 共计35个循环; 最后72 ℃延伸2 min。PCR产物利用ABI3730XL遗传分析系统进行基因型分型, 片段大小内标为LIZ500。
| 位点 | 引物序列(5′~3′) | 退火温度/℃ | 等位基因长度/bp | 5′端修饰 |
| DJ41 | F: GTGATGTTGTTGAACCTGAG | 55 | 126~186 | FAM |
| R: CCGTCCTTTACTCTTTGTCT | ||||
| DJ516 | F: GCTGTCCACAATAGAATTACC | 59 | 124~182 | HEX |
| R: CTCTGTCTTTGCTGTCCAT | ||||
| DJ578 | F: TGTCACACCGAGTAATGTC | 59 | 140~198 | FAM |
| R: AGAGGATGAGAGATAGAAGGA | ||||
| Cnas01 | F: TGCTATCTGTGATGATGCTACG | 60 | 127~161 | HEX |
| R: GAATACCTGCCCTTGTTTCTTG | ||||
| Cnas04 | F: GTATGTTTGTGAGGGTATGGGA | 60 | 173~207 | FAM |
| R: GTGAGCAAGGAACGGATAGTAA | ||||
| Cnas07 | F: CATTCTTTCCTCCCATCCCATA | 60 | 247~291 | HEX |
| R: GACTCCTTCCAAGACCCACTGA | ||||
| Cnas10 | F: ACTTGGTGAATTACAATGGGAA | 55 | 181~227 | FAM |
| R: ATGGAAAATAAGAAGCAGGAGA | ||||
| Cnas11 | F: AGGTGGTAAAAGACCCAAAGAA | 55 | 155~181 | HEX |
| R: TGTTTTCCCTCAATCCATCATA | ||||
| Cnas15 | F: TGGTAATGGCACTAATGGTATG | 55 | 282~316 | FAM |
| R: TAAATCAACCAACCAAGAAACA | ||||
| Cnas17 | F: GGCCCATATTGACCTCCAGACAGA | 60 | 245~279 | HEX |
| R: CACAGAGACTGGTGCTGCATTGCT |
用Excel 2016软件进行数据处理和作图。体长L (mm)与体重W (g)关系用幂函数拟合:
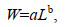 (1)
(1)式中, a为生长条件因子, b为幂函数指数。PopGen32软件计算各位点等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Shannon-Weiner指数(I)和F-统计量, 以及Nei’s标准遗传距离(Ds)等指标, 并进行Hardy-Weinberg平衡检验; PIC CALC 6.0软件计算各位点多态信息含量(PIC); Arlequin3.11软件检验各位点连锁不平衡(LD)状况; GenAlEx 6.51b2软件进行分子方差分析(AMOVA)确定变异来源; MEGA5.1软件基于Nei’s标准遗传距离绘制聚类分析图。
2 结果 2.1 体长、体重和年龄结构153尾短颌鲚种群体长分布范围为102.1~301.6 mm, 平均体长(196.6±40.0) mm, 优势体长组为145.0~ 234.9 mm, 占总个体数的77.12% (图 1); 个体体重分布范围1.90~79.70 g, 平均体重(17.70±13.06) g, 优势体重组为0.00~29.90 g, 占总个体数的84.97% (图 2)。
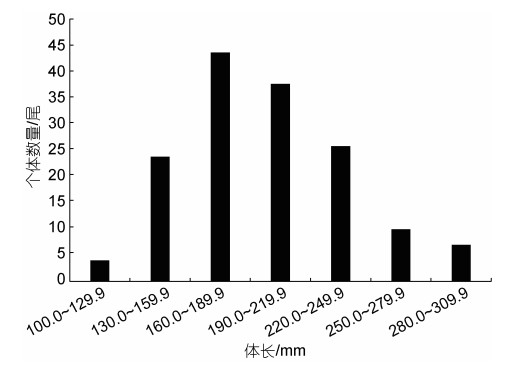 |
| 图 1 短颌鲚的体长分布 Fig. 1 Distribution of body length of C. brachygnathus |
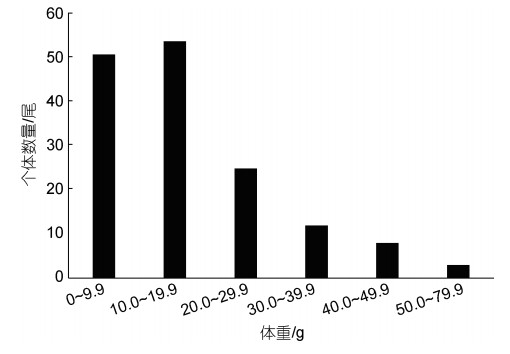 |
| 图 2 短颌鲚的体重分布 Fig. 2 Distribution of body weight of C. brachygnathus |
短颌鲚的年龄组成及个体分布见表 3、表 4。个体由1~5龄组成, 其中2龄和3龄为优势年龄组, 占总个体数的82.35%; 5龄鱼最少, 仅发现1尾。1~4龄个体的平均体长分别为(148.9±24.9)、(184.4±28.6)、(218.4±36.8)、(242.4±36.1) mm, 平均体重分别为(5.85±2.74)、(13.61±8.55)、(23.93±14.58)、(32.26±13.80) g, 短颌鲚的平均体长和体重随着年龄的增加而逐渐增长。
| 体长/mm | 1龄 | 2龄 | 3龄 | 4龄 | 5龄 | 总计/尾 |
| 100.0~114.9 | 2 | — | — | — | — | 2 |
| 115.0~129.9 | 1 | 1 | — | — | — | 2 |
| 130.0~144.9 | 3 | 2 | 1 | — | — | 6 |
| 145.0~159.9 | 3 | 14 | 1 | — | — | 18 |
| 160.0~174.9 | 4 | 12 | 2 | — | — | 18 |
| 175.0~189.9 | 1 | 18 | 6 | 1 | — | 26 |
| 190.0~204.9 | — | 16 | 8 | 1 | — | 25 |
| 205.0~219.9 | — | 3 | 9 | 1 | — | 13 |
| 220.0~234.9 | — | 9 | 6 | 3 | — | 18 |
| 235.0~249.9 | — | 3 | 4 | 1 | — | 8 |
| 250.0~264.9 | — | 1 | 5 | 1 | 1 | 8 |
| 265.0~279.9 | — | — | 2 | — | — | 2 |
| 280.0~294.9 | — | — | 2 | 3 | — | 5 |
| 295.0~309.9 | — | — | 1 | 1 | — | 2 |
| 平均体长/mm | 148.9 | 184.4 | 218.4 | 242.4 | 258.5 | — |
| 标准差(SD) | 24.9 | 28.6 | 36.8 | 36.1 | 0 | — |
| 总计/尾 | 14 | 79 | 47 | 12 | 1 | — |
| 百分比 | 9.15% | 51.63% | 30.72% | 7.84% | 0.66% | — |
| 体重/g | 1龄 | 2龄 | 3龄 | 4龄 | 5龄 | 总计/尾 |
| 0.00~4.99 | 7 | 8 | — | — | — | 15 |
| 5.00~9.99 | 6 | 25 | 5 | — | — | 36 |
| 10.00~14.99 | 1 | 17 | 7 | — | — | 25 |
| 15.00~19.99 | — | 14 | 12 | 3 | — | 29 |
| 20.00~24.99 | — | 6 | 4 | 1 | — | 11 |
| 25.00~29.99 | — | 5 | 7 | 2 | — | 14 |
| 30.00~34.99 | — | — | 2 | 2 | — | 4 |
| 35.00~39.99 | — | 3 | 4 | 1 | — | 8 |
| 40.00~44.99 | — | 1 | 1 | — | 1 | 3 |
| 45.00~49.99 | — | — | 3 | 2 | — | 5 |
| 50.00~54.99 | — | — | 1 | — | — | 1 |
| 55.00~59.99 | — | — | — | — | — | — |
| 60.00~64.99 | — | — | — | 1 | — | 1 |
| 65.00~69.99 | — | — | — | — | — | — |
| 70.00~74.99 | — | — | — | — | — | — |
| 75.00~79.99 | — | — | 1 | — | — | 1 |
| 平均体重/g | 5.85 | 13.61 | 23.93 | 32.26 | 41.60 | — |
| 标准差(SD) | 2.74 | 8.55 | 14.58 | 13.80 | 0 | — |
| 总计(尾) | 14 | 79 | 47 | 12 | 1 | |
| 百分比 | 9.15% | 51.63% | 30.72% | 7.84% | 0.66% |
基于样本的体长、体重数据拟合所得体长-体重关系式为:
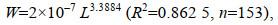 (2)
(2)式中, 幂函数指数b值为3.388 4, 表明库区短颌鲚属正异速生长型, 生长速度较快(图 3)。
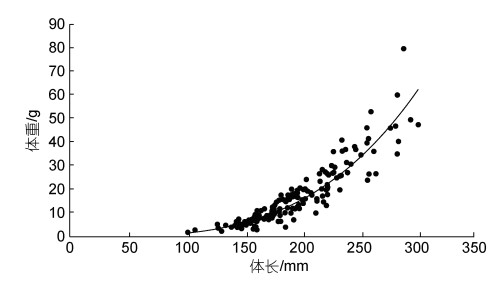 |
| 图 3 短颌鲚体长和体重的关系 Fig. 3 Relationship between body length and body weight of C. brachygnathus |
如表 5所示, 4个样本群体整体来看, 10个微卫星位点共检测到82个等位基因, 等位基因数(Na)介于4~16, 有效等位基因数(Ne)介于1.802 4~6.851 7, 观测杂合度(Ho)和期望杂合度(He)范围分别是0.451 0~ 0.849 7和0.446 7~0.856 9, 每个位点的多态信息含量(PIC)介于0.371 1~0.837 3之间, 除位点DJ516和Cnas04为中度多态位点外(0.25 < PIC < 0.5), 其余位点均为高度多态位点(PIC > 0.5)。
| 位点 | 等位基因数Na | 有效等位基因数Ne | 观测杂合度Ho | 期望杂合度He | 多态信息含量PIC |
| DJ41 | 12 | 3.968 7 | 0.756 6 | 0.750 5 | 0.710 9 |
| DJ516 | 4 | 1.885 5 | 0.496 7 | 0.471 2 | 0.371 1 |
| DJ578 | 6 | 2.676 8 | 0.575 2 | 0.628 5 | 0.568 5 |
| Cnas01 | 5 | 4.468 2 | 0.764 7 | 0.778 7 | 0.740 7 |
| Cnas04 | 6 | 1.802 4 | 0.451 0 | 0.446 7 | 0.405 6 |
| Cnas07 | 16 | 6.851 7 | 0.849 7 | 0.856 9 | 0.837 3 |
| Cnas10 | 8 | 5.062 5 | 0.562 1 | 0.805 1 | 0.780 7 |
| Cnas11 | 6 | 3.192 1 | 0.692 8 | 0.689 0 | 0.633 8 |
| Cnas15 | 6 | 3.579 6 | 0.725 5 | 0.723 0 | 0.677 7 |
| Cnas17 | 13 | 4.545 4 | 0.751 6 | 0.782 6 | 0.748 9 |
10个微卫星位点在4个短颌鲚样本群体中的遗传多样性信息见表 6。可以看出, 4个群体的平均等位
| 位点 | 参数 | 群体 | |||
| FL | FD | WS | WZ | ||
| DJ41 | Na | 6 | 7 | 11 | 9 |
| Ne | 3.628 1 | 3.500 0 | 3.856 7 | 3.940 5 | |
| Ho | 0.736 8 | 0.500 0 | 0.830 8 | 0.740 7 | |
| He | 0.744 0 | 0.740 7 | 0.746 5 | 0.753 2 | |
| PIC | 0.677 0 | 0.681 4 | 0.701 4 | 0.708 7 | |
| P | 0.248 4 | 0.074 8 | 0.976 4 | 0.913 3 | |
| I | 1.437 4 | 1.540 7 | 1.633 5 | 1.583 9 | |
| FIS | –0.017 2 | 0.300 0 | –0.121 6 | 0.007 4 | |
| DJ516 | Na | 3 | 3 | 3 | 3 |
| Ne | 1.875 3 | 1.742 2 | 1.906 2 | 1.884 1 | |
| Ho | 0.473 7 | 0.428 6 | 0.507 7 | 0.509 1 | |
| He | 0.479 4 | 0.441 8 | 0.479 1 | 0.473 6 | |
| PIC | 0.379 7 | 0.360 8 | 0.369 5 | 0.367 4 | |
| P | 0.916 9 | 0.936 0 | 0.844 6 | 0.834 2 | |
| I | 0.735 2 | 0.705 9 | 0.700 1 | 0.698 0 | |
| FIS | –0.014 8 | –0.006 0 | –0.068 0 | –0.084 9 | |
| DJ578 | Na | 4 | 4 | 4 | 5 |
| Ne | 2.292 1 | 2.992 4 | 2.681 7 | 2.654 7 | |
| Ho | 0.631 6 | 0.714 3 | 0.600 0 | 0.490 9 | |
| He | 0.578 9 | 0.690 5 | 0.632 0 | 0.629 0 | |
| PIC | 0.510 2 | 0.608 3 | 0.563 1 | 0.568 0 | |
| P | 0.760 2 | 0.000 1** | 0.775 5 | 0.002 9** | |
| I | 1.019 1 | 1.210 3 | 1.118 6 | 1.151 4 | |
| FIS | –0.120 4 | –0.072 8 | 0.043 2 | 0.212 4 | |
| Cnas01 | Na | 5 | 5 | 5 | 5 |
| Ne | 4.456 8 | 3.959 6 | 4.309 0 | 4.330 7 | |
| Ho | 0.789 5 | 0.785 7 | 0.800 0 | 0.709 1 | |
| He | 0.799 6 | 0.775 1 | 0.773 9 | 0.776 1 | |
| PIC | 0.739 9 | 0.710 6 | 0.729 8 | 0.731 7 | |
| P | 0.217 5 | 0.681 1 | 0.882 9 | 0.118 3 | |
| I | 1.548 5 | 1.492 4 | 1.522 3 | 1.530 4 | |
| FIS | –0.017 9 | –0.051 2 | –0.041 8 | 0.078 0 | |
| Cnas04 | Na | 5 | 4 | 5 | 4 |
| Ne | 2.867 0 | 1.979 8 | 1.787 2 | 1.689 0 | |
| Ho | 0.473 7 | 0.571 4 | 0.400 0 | 0.472 7 | |
| He | 0.534 9 | 0.513 2 | 0.443 9 | 0.411 7 | |
| PIC | 0.479 7 | 0.452 9 | 0.401 8 | 0.367 3 | |
| P | 0.751 2 | 0.969 0 | 0.000 0** | 0.680 3 | |
| I | 1.015 5 | 0.929 1 | 0.850 2 | 0.760 5 | |
| FIS | 0.090 4 | –0.154 6 | 0.091 9 | –0.158 8 | |
| Cnas07 | Na | 9 | 8 | 14 | 11 |
| Ne | 6.504 5 | 6.644 1 | 6.204 1 | 6.843 9 | |
| Ho | 0.947 4 | 0.642 9 | 0.923 1 | 0.781 8 | |
| Cnas07 | He | 0.869 1 | 0.881 0 | 0.845 3 | 0.861 7 |
| PIC | 0.827 5 | 0.831 4 | 0.819 0 | 0.837 9 | |
| P | 0.523 8 | 0.000 1** | 0.000 0** | 0.001 5** | |
| I | 1.986 4 | 1.970 8 | 2.036 2 | 2.099 5 | |
| FIS | –0.119 5 | 0.243 2 | –0.100 5 | 0.084 4 | |
| Cnas10 | Na | 7 | 4 | 8 | 8 |
| Ne | 4.813 3 | 3.111 1 | 5.732 7 | 3.883 2 | |
| Ho | 0.736 8 | 0.357 1 | 0.676 9 | 0.418 2 | |
| He | 0.813 7 | 0.703 7 | 0.832 0 | 0.749 3 | |
| PIC | 0.765 3 | 0.613 7 | 0.805 5 | 0.716 8 | |
| P | 0.092 7 | 0.035 1* | 0.000 0** | 0.000 0** | |
| I | 1.732 0 | 1.197 4 | 1.907 2 | 1.677 2 | |
| FIS | 0.069 9 | 0.473 7 | 0.180 0 | 0.436 8 | |
| Cnas11 | Na | 4 | 4 | 6 | 4 |
| Ne | 2.481 1 | 3.136 0 | 3.538 5 | 2.999 5 | |
| Ho | 0.684 2 | 0.928 6 | 0.692 3 | 0.636 4 | |
| He | 0.613 1 | 0.706 3 | 0.723 0 | 0.672 7 | |
| PIC | 0.525 0 | 0.630 3 | 0.668 5 | 0.612 2 | |
| P | 0.899 4 | 0.061 3 | 0.000 0** | 0.460 2 | |
| I | 1.060 2 | 1.255 7 | 1.387 4 | 1.224 5 | |
| FIS | –0.146 2 | –0.363 3 | 0.035 0 | 0.045 4 | |
| Cnas15 | Na | 6 | 6 | 6 | 6 |
| Ne | 3.421 8 | 4.041 2 | 3.325 5 | 3.707 1 | |
| Ho | 0.789 5 | 0.785 7 | 0.723 1 | 0.690 9 | |
| He | 0.726 9 | 0.780 4 | 0.704 7 | 0.736 9 | |
| PIC | 0.659 4 | 0.718 1 | 0.651 9 | 0.688 4 | |
| P | 0.059 6 | 0.059 6 | 0.514 5 | 0.029 7* | |
| I | 1.429 3 | 1.560 6 | 1.419 8 | 1.502 4 | |
| FIS | –0.115 5 | –0.044 1 | –0.034 0 | 0.053 9 | |
| Cnas17 | Na | 7 | 8 | 11 | 9 |
| Ne | 4.079 1 | 4.307 7 | 5.008 9 | 4.025 3 | |
| Ho | 0.736 8 | 0.857 1 | 0.753 8 | 0.727 3 | |
| He | 0.775 2 | 0.796 3 | 0.806 6 | 0.758 5 | |
| PIC | 0.721 6 | 0.740 2 | 0.773 5 | 0.711 1 | |
| P | 0.545 4 | 0.958 7 | 0.000 0** | 0.000 0** | |
| I | 1.603 5 | 1.721 3 | 1.850 4 | 1.598 5 | |
| FIS | 0.023 9 | –0.116 3 | 0.058 1 | 0.032 3 | |
| 平均 | Na | 5.6 | 5.3 | 7.3 | 6.4 |
| Ne | 3.563 9 | 3.541 4 | 3.835 0 | 3.595 8 | |
| Ho | 0.700 0 | 0.657 1 | 0.690 8 | 0.617 7 | |
| He | 0.693 2 | 0.702 9 | 0.698 7 | 0.682 3 | |
| PIC | 0.628 5 | 0.634 7 | 0.648 4 | 0.630 9 | |
| I | 1.356 2 | 1.358 4 | 1.442 6 | 1.382 6 | |
| FIS | –0.037 1 | 0.030 5 | 0.003 7 | 0.086 3 | |
| 注: *表示显著偏离Hardy-Weinberg平衡(P < 0.05), **表示极显著偏离Hardy-Weinberg平衡(P < 0.01) | |||||
基因数(Na)由高到低依次为WS群体(7.3)、WZ群体(6.4)、FL群体(5.6)、FD群体(5.3); 平均有效等位基因数(Ne)的顺序与平均等位基因数排序一致。各群体的平均观测杂合度(Ho)和平均期望杂合度(He)差异较小, 分别为0.617 7~0.700 0和0.682 3~0.702 9。4个群体的平均多态信息含量(PIC)介于0.628 5~0.648 4之间, 平均香农指数(I)由高到低依次为WS群体(1.442 6)、WZ群体(1.382 6)、FD群体(1.358 4)、FL群体(1.356 2), 平均近交系数(FIS)分别为FL群体(–0.037 1)、FD群体(0.030 5)、WS群体(0.003 7)、WZ群体(0.086 3)。以上结果表明, 4个短颌鲚群体的遗传多样性较为丰富, 其中WS群体的遗传多样性在4个群体中最高, 而FD群体和FL群体较低。
基于Hardy-Weinberg平衡定律对4个样本群体中每个位点的基因平衡状态进行检测(表 6), 在WS群体中, 有5个位点显著偏离Hardy-Weinberg平衡, 分别为Cnas04、Cnas07、Cnas10、Cnas11、Cnas17; 在WZ群体中, 有5个位点显著偏离, 分别为DJ578、Cnas07、Cnas10、Cnas15、Cnas17; 在FD群体中, 有3个位点显著偏离, 分别为DJ578、Cnas07、Cnas10。连锁不平衡检验结果表明, 各位点间无连锁不平衡状况。
2.4 群体遗传结构分析FD群体和FL群体的个体数量较少, 相距约50 km, 地理位置相近, 可将二者合并为一个采样群体进行分析。3个群体间的遗传分化指数(FST)介于0.000 07~0.003 24, 群体间遗传分化程度很低(FST < 0.05), P值显著性检验结果表明, 3个群体间两两比较均无显著分化(P > 0.05)。从Nei’s标准遗传距离来看, FL、FD合并群体与其他群体间遗传距离较远, WZ和WS群体遗传距离最近(Ds=0.025 6), 说明两群体间亲缘关系较近(表 7)。基于Nei’s标准遗传距离的UPGMA系统树(图 4)显示, WS群体和WZ群体先聚为一类, 然后与FD群体和FL群体的合并群体聚为一类。分子方差分析(AMOVA)结果显示, 95.44%的遗传变异来自个体内, 群体间的遗传变异仅为0.09%, 群体间遗传差异极小(表 8)。
| 群体 | FL & FD | WS | WZ |
| FL & FD | — | 0.000 07 | 0.000 34 |
| WS | 0.026 20 | — | 0.003 24 |
| WZ | 0.027 10 | 0.025 60 | — |
 |
| 图 4 三个短颌鲚群体UPGMA聚类图 Fig. 4 The UPGMA clustering tree of C. brachygnathus among three populations |
| 变异来源 | 自由度(df) | 方差(SS) | 平方和(MS) | 变异组成/% | 遗传分化指数(FST) |
| 群体间 | 3 | 11.533 | 3.844 | 0.09 | 0.000 9 |
| 群体内 | 149 | 539.594 | 3.621 | 4.47 | |
| 个体内 | 153 | 506.500 | 3.310 | 95.44 | |
| 总变异 | 305 | 1 057.627 | 10.775 | 100.00 |
体长-体重关系式中的幂函数指数b常用于反映鱼类在不同阶段环境中的生长状况, 若b=3表明鱼类等速生长, b > 3表明鱼类正异速生长, b < 3则为负异速生长。本研究的幂函数指数b值(3.388 4)高于鄱阳湖短颌鲚群体(b=3.022) (吴斌等, 2015)和长湖短颌鲚群体(b=2.855) (王旭歌等, 2016), 表明三峡水库广阔的水域和丰富的营养沉积为短颌鲚生长提供了充足的饵料, 生长速度较快。
20世纪70年代, 袁传宓等(1978)曾对包括长颌鲚(Coilia nasus)、短颌鲚和湖鲚(Coilia nasus taihuensis)在内的长江流域刀鲚资源状况进行调查, 发现长江刀鲚中存在6龄鱼个体, 并认为长江流域刀鲚以3~4龄鱼为主。万全等(2009)对长江下游地区刀鲚生殖洄游群体的研究结果表明, 该群体仍存在5~6龄个体。本研究采样过程中仅发现1尾5龄鱼个体, 且1~3龄鱼占总个体数的91.5%。根据文献报道, 短颌鲚属r选择生活史类型, 三峡库区短颌鲚种群增长模式与湖北长湖短颌鲚群体相似, 种群扩散能力强(王旭歌, 2016), 年龄组成表明该种群处于快速增长期。三峡库区建成蓄水后, 原有的生境发生改变, 圆口铜鱼、长鳍吻鮈(Rhinogobio ventralis)等喜急流鱼类离开库区静水江段向上游流水江段迁移, 库区生态位出现空缺(杨志等, 2012)。短颌鲚作为三峡库区的新建种群, 处于快速增长期, 填补库区空缺的生态位, 因此呈现出低龄鱼个体较多, 高龄鱼数量较少的年龄结构特点。
3.2 群体遗传多样性分析多态信息含量(PIC)是反映微卫星DNA变异程度高低的指标, PIC > 0.5为高度多态性, 0.25 < PIC < 0.5为中度多态性, PIC < 0.25表示多态性低(Botstein et al, 1980)。本研究中短颌鲚群体的平均多态信息含量在0.628 5~0.648 4之间, 表明三峡库区短颌鲚具有较高的遗传多样性。根据微卫星位点的选择标准(Barker, 1994), 等位基因数(Na) > 4时位点才能较好的反映出群体的遗传变异水平。本研究中4个样本群体的平均Na在5.3~7.3之间, 等位基因保存较好, 其原因可能是库区广阔的水域为坝下短颌鲚的大量迁入提供了条件, 迁入库区的建群群体保留了坝下群体较高的遗传多样性。
期望杂合度(He)的数值与样本量大小无显著相关性, 相比观测杂合度(Ho)能够更准确的反映群体遗传多样性, 常将其视为衡量群体变异的最适参数(闫路娜等, 2004)。本研究中4个样本群体的平均Ho为0.617 7~0.700 0, 平均He为0.682 3~0.702 9, 与马春艳(2010)调查的长江中游武汉短颌鲚群体多样性相近(Ho=0.665 5; He=0.676 3)。从等位基因数(Na)和有效等位基因数(Ne)来看, 4个样本群体的遗传多样性依次为: WS > WZ > FL > FD; 从Shannon-Weiner指数(I)反映的遗传多样性来看, 4个群体的多样性依次为: WS > WZ > FD > FL。这与Na和Ne反映的结果基本一致, 均呈现出由下游至上游遗传多样性逐渐降低的趋势。根据Yang等(2019)的报道, 作为三峡库区的新建种群, 短颌鲚最早出现在巫山水域。我们近几年的调查数据显示, 短颌鲚在建群时间最早的巫山水域的渔获物中占比最高, 在丰都、涪陵水域的渔获物中占比较低, 在渔获物中的数量由库区下游至上游逐渐减少。水坝修建对鱼类的影响主要来源于河流生境“湖泊化”发展和地理隔离两方面(Neraas et al, 2001)。水库建成后的蓄水倒灌使库区生境发生显著改变(Taylor et al, 2001), 同时大坝造成的地理隔离也阻碍了鱼类的产卵和进食, 导致鱼类数量锐减(Barthem et al, 1991; Nehlsen et al, 1991)。短颌鲚开始在三峡库区建群之际, 库区蓄水后的“湖泊化”生境迫使喜急流鱼类向上游迁移, 原有鱼类的繁殖和摄食也同样受到影响, 造成库区生态位空缺, 为短颌鲚的迁入和扩张创造了有利条件。然而近年来水库万州以下江段生态系统逐渐恢复, 食物网已得到重建, 同时由于库区水位提升, 水库中营养物质含量增加, 鱼类的生物量也得以恢复(杨志等, 2012; He et al, 2020)。受三峡库区下游生态系统恢复和建群时间的综合作用, 库区短颌鲚群体的遗传多样性呈现出由下游至上游逐渐减小的特点, 各采样点的个体数量也呈现从下游向上游逐渐减少的趋势, 同时库区短颌鲚的遗传多样性与长江中游短颌鲚群体相近, 综合以上结果提示, 三峡库区的群体很可能是坝下短颌鲚通过三峡大坝船闸迁入库区水域而繁殖建群。在生态系统中, 鲚属鱼类作为顶级捕食者通常占据较高的生态位(胡忠军等, 2019), 我们推测溯河而上的短颌鲚会改变整个地区的营养级结构, 逐渐成为三峡库区的优势种, 而且有逐渐向库区上游扩散的趋势。
本研究使用遗传分化指数(FST)作为评价种群分化程度的指标。根据Balloux等(2002)提出的标准, FST < 0.05表示群体间分化程度较小。本研究短颌鲚群体间的遗传分化指数为0.000 07~0.003 24, 远低于0.05, 说明4个样本群体间分化水平微弱。分子方差分析(AMOVA)结果显示, 群体间的遗传变异主要源于个体内部(95.44%), 群体间变异程度很低(0.09%), 与近年来长江上游圆筒吻
综上所述, 本研究表明长江上游三峡水库短颌鲚新建种群处于快速增长期, 微卫星遗传多样性水平较高, 与中游短颌鲚群体相近, 且各样本群体呈现出从三峡水库下游至上游多样性逐渐下降的趋势。结果提示, 三峡库区的短颌鲚群体很可能来源于三峡大坝下游种群过坝扩散而来, 而且其种群可能发展为三峡库区优势种并且有进一步向库区上游扩散的趋势。本研究结果可为三峡库区鱼类群落结构演变和长江上游短颌鲚资源现状和保护提供基础数据。
万全, 赖年悦, 李飞, 等, 2009. 安徽无为长江段刀鲚生殖洄游群体年龄结构的变化分析. 水生态学杂志, 30(4): 60-65 |
马克异, 冯建彬, 谢楠, 等, 2011. 钱塘江日本沼虾野生群体遗传变异的SSR分析. 动物学研究, 32(4): 363-370 |
马春艳, 2010. 鳀科鱼类分子系统进化及凤鲚、刀鲚遗传多样性研究[D]. 上海: 华东师范大学: 84-85.
|
王旭歌, 2016. 长湖短颌鲚种群生态学及遗传多样性[D]. 上海: 上海海洋大学: 31.
|
王旭歌, 何勇凤, 李昊成, 等, 2016. 长湖短颌鲚的年龄与生长特性. 淡水渔业, 46(3): 29-33 DOI:10.3969/j.issn.1000-6907.2016.03.005 |
龙安雨, 田辉伍, 汪登强, 等, 2020. 长江上游中华金沙鳅和短身金沙鳅线粒体遗传多样性研究. 淡水渔业, 50(3): 34-41 |
闫路娜, 张德兴, 2004. 种群微卫星DNA分析中样本量对各种遗传多样性度量指标的影响. 动物学报, 50(2): 279-290 |
杨志, 陶江平, 唐会元, 等, 2012. 三峡水库运行后库区鱼类资源变化及保护研究. 人民长江, 43(10): 62-67 DOI:10.3969/j.issn.1001-4179.2012.10.017 |
轩中亚, 姜涛, 刘洪波, 等, 2020. 基于线粒体Cyt-b序列的太湖湖鲚与短颌鲚种群遗传分析. 渔业科学进展, 41(4): 33-40 |
吴斌, 方春林, 傅培峰, 等, 2015. 鄱阳湖通江水道短颌鲚生长特性初探. 水生态学杂志, 36(3): 51-55 |
汪焕, 胡玉婷, 江河, 等, 2020. 安徽省淮河水系短颌鲚群体遗传多样性. 动物学杂志, 55(6): 760-767 |
陈芳, 2012. 刀鲚和凤鲚遗传多样性的微卫星分析[D]. 上海: 上海海洋大学: 19-21.
|
陈婷婷, 姜涛, 卢明杰, 等, 2016. 基于耳石微化学的长江靖江段长颌鲚与短颌鲚生境履历重建. 湖泊科学, 28(1): 149-155 |
范海霞, 刘遂飞, 胡茂林, 2016. 鄱阳湖短颌鲚营养成分分析. 水产科学, 35(1): 77-80 DOI:10.3969/j.issn.2095-0780.2016.01.011 |
周晓犊, 杨金权, 唐文乔, 等, 2010. 基于线粒体COⅠ基因DNA条形码的中国鲚属物种有效性分析. 动物分类学报, 35(4): 819-826 |
胡茂林, 吴志强, 刘引兰, 2011. 鄱阳湖湖口水域鲚鱼的物种属性及资源现状. 水产科技情报, 38(5): 223-226 DOI:10.3969/j.issn.1001-1994.2011.05.002 |
胡忠军, 史先鹤, 吴昊, 等, 2019. 上海青草沙水库食物网结构特征分析. 水生态学杂志, 40(2): 47-54 |
姜涛, 刘洪波, 轩中亚, 等, 2020. 长江中下游流域刀鲚(Coilia nasus)生态表型的划分. 湖泊科学, 32(2): 518-527 |
袁传宓, 林金榜, 刘仁华, 等, 1978. 刀鲚的年龄和生长. 水生生物学集刊, 6(3): 285-298 |
徐钢春, 顾若波, 刘洪波, 等, 2014. 长江短颌鲚耳石Sr/Ca值变化特征及其江海洄游履历. 水产学报, 38(7): 939-945 |
郭弘艺, 魏凯, 唐文乔, 等, 2010. 基于矢耳石形态特征的中国鲚属鱼类种类识别. 动物分类学报, 35(1): 127-134 |
蒲艳, 田辉伍, 陈大庆, 等, 2019. 长江中上游圆筒吻鮈群体线粒体Cyt b遗传多样性分析. 淡水渔业, 49(1): 14-19 DOI:10.3969/j.issn.1000-6907.2019.01.003 |
BALLOUX F, LUGON-MOULIN N, 2002. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 11(2): 155-165 DOI:10.1046/j.0962-1083.2001.01436.x |
BARKER J S F, 1994. Sequential gel electrophoretic analysis of esterase-2 in two populations of Drosophila buzzatii. Genetica, 92(3): 165-175 DOI:10.1007/BF00132535 |
BARTHEM R B, DE BRITO RIBEIRO M C L, PETRERE M JR, 1991. Life strategies of some long-distance migratory catfish in relation to hydroelectric dams in the Amazon Basin. Biological Conservation, 55(3): 339-345 DOI:10.1016/0006-3207(91)90037-A |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 32(3): 314-331 |
CHENG F Y, WANG Q, DELSER P M, et al, 2019. Multiple freshwater invasions of the tapertail anchovy (Clupeiformes: Engraulidae) of the Yangtze River. Ecology and Evolution, 9(21): 12202-12215 DOI:10.1002/ece3.5708 |
HE C, DENG H T, BA J W, et al, 2020. Food chain length associated with environmental factors affected by large dam along the Yangtze River. Water, 12(11): 3157 DOI:10.3390/w12113157 |
NEHLSEN W, WILLIAMS J E, LICHATOWICH J A, 1991. Pacific salmon at the crossroads: stocks at risk from California, Oregon, Idaho, and Washington. Fisheries, 16(2): 4-21 DOI:10.1577/1548-8446(1991)016<0004:PSATCS>2.0.CO;2 |
NERAAS L P, SPRUELL P, 2001. Fragmentation of riverine systems: the genetic effects of dams on bull trout (Salvelinus confluentus) in the Clark Fork River system. Molecular Ecology, 10(5): 1153-1164 DOI:10.1046/j.1365-294X.2001.01269.x |
QIN X C, WANG T, LIN P C, et al, 2017. Age, growth, mortality and movement patterns of shortjaw tapertail anchovy, Coilia brachygnathus, in the channel connecting Dongting Lake and the Yangtze River in central China. Aquatic Living Resources, 31: 3 |
TAYLOR C A, KNOUFT J H, HILAND T M, 2001. Consequences of stream impoundment on fish communities in a small North American drainage. Regulated Rivers: Research & Management, 17(6): 687-698 |
XUAN Z Y, JIANG T, LIU H B, et al, 2021. Mitochondrial DNA and microsatellite analyses reveal strong genetic differentiation between two types of estuarine tapertail anchovies (Coilia) in Yangtze River Basin, China. Hydrobiologia, 848(6): 1409-1431 DOI:10.1007/s10750-021-04541-w |
YANG Q L, GAO T X, LIU J X, 2014. Development and characterization of 17 microsatellite loci in an anadromous fish Coilia nasus. Conservation Genetics Resources, 6(2): 357-359 DOI:10.1007/s12686-013-0093-4 |
YANG Q L, HAN Z Q, SUN D R, et al, 2010. Genetics and phylogeny of genus Coilia in China based on AFLP markers. Chinese Journal of Oceanology and Limnology, 28(4): 795-801 DOI:10.1007/s00343-010-9093-3 |
YANG F, TAO Y X, DUAN B C, et al, 2019. Complete mitochondrial genome of the Yangtze grenadier anchovy, Coilia brachygnathus (Clupeiformes: Engraulidae) from the upper Yangtze River. Mitochondrial DNA Part B, 4(1): 1140-1141 DOI:10.1080/23802359.2019.1591181 |
ZHANG F T, TAN D Q, 2010. Genetic diversity in population of largemouth bronze gudgeon (Coreius guichenoti Sauvage et Dabry) from Yangtze River determined by microsatellite DNA analysis. Genes & Genetic Systems, 85(5): 351-357 |
ZHANG H, WU G G, 2013. Role of body size and temporal hydrology in the dietary shifts of shortjaw tapertail anchovy Coilia brachygnathus (Actinopterygii, Engraulidae) in a large floodplain lake. Hydrobiologia, 703(1): 247-256 DOI:10.1007/s10750-012-1370-z |
 2022, Vol. 53
2022, Vol. 53


