中国海洋湖沼学会主办。
文章信息
- 练佳莹, 吕丽媛, 姚韩韩, 董迎辉, 林志华. 2022.
- LIAN Jia-Ying, LYU Li-Yuan, YAO Han-Han, DONG Ying-Hui, LIN Zhi-Hua. 2022.
- 缢蛏HAT基因家族的全基因组鉴定及在环境因子、细菌胁迫下的表达分析
- GENOME-WIDE IDENTIFICATION AND EXPRESSION ANALYSIS OF THE HISTONE ACETYLTRANSFERASE (HAT) GENE FAMILY IN SINONOVACULA CONSTRICTA UNDER ENVIRONMENTAL AND BACTERIAL STRESSES
- 海洋与湖沼, 53(2): 475-485
- Oceanologia et Limnologia Sinica, 53(2): 475-485.
- http://dx.doi.org/10.11693/hyhz20211000239
文章历史
-
收稿日期:2021-10-10
收修改稿日期:2021-11-17
2. 浙江万里学院宁海海洋生物种业研究院 浙江宁波 315604
2. Ninghai Institute of Mariculture Breeding and Seed Industry, Zhejiang Wanli University, Ningbo 315604, China
在真核生物中, 染色质结构的动态调节影响各种生物过程(Ho et al, 2010)。核小体是染色质的基本结构单位, 由四个核心组蛋白(H2A, H2B, H3, H4)组成的八聚体和其周围包裹着147 bp的DNA共同构成(Kouzarides, 2007), 每个组蛋白均包含一个结构化的球形结构域和一个从核小体核心延伸出来的非结构化N端尾部(Patel et al, 2013), 其尾部受到组蛋白乙酰化、甲基化、磷酸化、泛素化等各种翻译后修饰(Kouzarides, 2007)。组蛋白乙酰化是由组蛋白乙酰化酶(histone acetyltransferases, HATs)和组蛋白去乙酰化酶(histone deacetylases, HDACs)共同调控的动态可逆过程(Bannister et al, 2011)。HAT可将乙酰辅酶A的乙酰基转移至核心组蛋白(主要为H3和H4)N末端特定赖氨酸的ε-残基上, 乙酰基通过中和赖氨酸残基上的正电荷, 减弱组蛋白与DNA的结合能力, 使染色质结构松散, 有利于转录因子与DNA结合, 从而促进基因转录与表达(Struhl, 1998; Eberharter et al, 2002)。根据氨基酸序列和结构相似性, HAT至少可分为3类亚家族: (1) GNAT, 与GCN5相关的N端乙酰转移酶, 包括KAT1 (Hat1)、KAT2B (PCAF)、KAT9 (Elp3)等; (2) MYST, 包括KAT5 (Tip60)、KAT6A (MOZ)、KAT7 (Hbo1)、KAT8 (Sas2)等; (3) p300/CREB结合蛋白(CBP)家族(Hodawadekar et al, 2007; Bannister et al, 2017)。
目前, HAT已在多种生物中被鉴定, 如动物中的小鼠(Yao et al, 1998)、秀丽隐杆线虫(Caenorhabditis elegans) (Goodman et al, 2000)、果蝇(Carre et al, 2005)、弓形虫(Padgett et al, 2018)、马氏珠母贝(Pinctada martensi) (杨帅, 2020)等, 植物中的拟南芥(Latrasse et al, 2008)、水稻(Liu et al, 2012)、棉花(Imran et al, 2019)、小麦(Gao et al, 2021)等。研究表明, HAT在植物生长发育(Latrasse et al, 2008)、响应生物和非生物胁迫方面发挥着重要作用(Yin et al, 2019; Gan et al, 2021), 其应激响应很大程度上依赖于翻译后的组蛋白乙酰化(Gao et al, 2021)。但目前关于贝类组蛋白乙酰化修饰鲜有报道。
缢蛏(Sinonovacula constricta)是我国重要的海水养殖贝类, 由于其广泛分布于潮间带, 极易受到高温、高氨氮和病原微生物等极端环境和生物压力的影响(郑余琦等, 2017; Dong et al, 2020), 从而导致生长变缓、免疫力降低等问题, 严重时暴发大规模死亡, 造成巨大的经济损失, 因此开展缢蛏抗逆、抗病研究对其养殖业的可持续发展具有重要意义。本研究对缢蛏HAT基因家族进行全基因组鉴定, 并对其保守结构域、保守基序、系统发育关系及其在不同发育时期、环境因子(氨氮和高温胁迫)和副溶血弧菌(Vibrio parahaemolyticus)胁迫下的表达特征进行分析, 旨在为深入探索HAT在贝类中的功能及调控机制奠定理论基础。
1 材料与方法 1.1 实验动物及细菌2021年3月, 从宁波市海洋与渔业科技创新基地取健康缢蛏成贝, 平均壳长(58.43±1.75) mm、壳高(19.93±0.46) mm。缢蛏处理前, 在实验室配制海水中(温度25 ℃, 盐度20)暂养3 d。实验用副溶血弧菌为实验室保存菌种, 采用2216E培养基于28 ℃恒温摇床上培养后测定OD600值, 所需终浓度为1×108 CFU/mL。
1.2 数据库资源缢蛏基因组和转录组数据均为本实验室前期测序获得(Dong et al, 2020)。其余的人(Homo sapiens)、白氏文昌鱼(Branchiostoma belcheri)、秀丽隐杆线虫、太平洋牡蛎(Crassostrea gigas)、虾夷扇贝(Mizuhopecten yessoensis)、加州双斑蛸(Octopus bimaculoides)和紫色球海胆(Strongylocentrotus purpuratus)基因组数据均来源于NCBI数据库(https://www.ncbi.nlm.nih.gov/)。
1.3 ScHAT基因家族的鉴定为鉴定ScHAT基因家族成员, 先保留与注释高度匹配的序列, 将所获得的序列作为目标序列通过Blastp方式检索缢蛏基因组数据库, 手动剔除冗余序列, 然后通过SMART、Pfam、Batch-CD Search在线工具结合预测候选序列的保守结构域进行确认, 并根据HAT各家族的相似性进一步分类并命名。使用ProtParam (https://web.expasy.org/protparam/)分析HAT蛋白等电点(pI)、分子量(MW)和总疏水性(GRAVY)等理化性质。同时采用相同的方法鉴定出其他物种的HAT基因, 并进行基因数目的分类统计。
1.4 ScHAT基因家族的系统进化分析采用MAFFT 7 (Katoh et al, 2013)对缢蛏和其他7个物种(人、白氏文昌鱼、秀丽隐杆线虫、太平洋牡蛎、虾夷扇贝、加州双斑蛸和紫色球海胆)的HAT蛋白序列进行多重序列比对。用MrBayes 3.2.6 (Ronquist et al, 2003)进行系统发育分析, 4条马尔可夫链运行1.5×107代, 取样频率(sampling frequency)设为5 000, 样本舍弃比率(burn-in fraction)设为0.25, 分裂频率平均标准差(average standard deviation of split frequencies)小于0.01视为达到收敛。最后用FigTree 1.4.3对贝叶斯系统树进行可视化。
1.5 ScHAT基因家族的结构域及motif分析通过在线工具SMART (http://smart.embl-heidelberg.de/smart/set_mode.cgiGENOMIC=1)、Pfam(https://meme-suite.org/meme/tools/meme)预测HAT蛋白的保守结构域。利用MEME (_aaaaaa_paichu__)预测HAT蛋白的保守基序(motif), motif数目参数设置为18, 长度设置为21~50 aa, 并用TBtools (Chen et al, 2020)进行可视化。
1.6 ScHAT基因在不同发育时期及环境胁迫下的表达分析利用已发表的缢蛏不同发育时期、氨氮胁迫(180 mg/L, 72 h)和高温(32 ℃, 96 h)胁迫的RNA-seq数据集中检索11个ScHAT基因的FPKM (fragments per kilobase per million mapped reads)值, 分析它们在不同发育时期和环境胁迫下的表达模式。其中, 卵子、4细胞、囊胚、原肠胚、担轮幼虫、D形幼虫、壳顶幼虫和稚贝8个时期的SRA登录号分别为: SRR10097413~SRR10097424; 氨氮胁迫和高温胁迫的转录组SRA登录号分别为: SRR9943679~ SRR9943690和SRR13594753~SRR13594764。利用TBtools绘制基因表达热图。
1.7 ScHAT基因在副溶血弧菌胁迫下的表达分析将150粒缢蛏成贝随机分成3组, 用浓度为1×108 CFU/mL的副溶血弧菌浸泡胁迫。于浸泡后0、3、6、12、24、48、72和96 h, 每个时间点随机选6粒缢蛏, 取其鳃置于液氮速冻后保存在–80 ℃。以0 h未胁迫组为对照组。用Trizol (TaKaRa, Japan)试剂提取鳃的总RNA, 并对RNA浓度和完整性进行检测。利用Prime-ScriptTM RT (TaKaRa, Japan)试剂盒合成cDNA。用LightCycler®480II仪器进行荧光定量反应, 检测ScHATs在抗菌免疫中的表达规律。实验所用引物如表 1所示, 利用Primer 5软件对ScHATs的保守结构域进行引物设计, 通过溶解曲线验证引物特异性。以18S rRNA为内参基因。qRT-PCR反应体系(20 μL)为: 上下游引物各1 μL、cDNA (稀释100倍) 8 μL、SYBR® Green Supermix 10 μL。反应程序为: 95 ℃ 30 s, 40个循环; 95 ℃变性15 s, 60 ℃退火1 min。每组3个生物平行和3个技术重复。用2–△△CT法计算ScHATs的相对表达量, 利用SPSS软件统计分析其差异显著性(P < 0.05), GraphPad Prism 8.0.1绘图。
| 引物名称 | 序列(5′~3′) |
| ScKAT1F | CACAAACATTGATGACTTCTCTGC |
| ScKAT1R | GCTCCATTTATCATCATCCACATC |
| ScKAT2BF | TGAAAACACAGCGGAAGAAGA |
| ScKAT2BR | ATGGAGGATTGCCAAGAGGA |
| ScKAT5F | CTAACCGAAGGGTGTAGGCTG |
| ScKAT5R | CGAGGGTACTCCAACTTGCTG |
| ScKAT6AF | AACAAACGGGAGACGGAG |
| ScKAT6AR | ACACATAGCAGTCATCAGGAAGT |
| ScKAT6BF | AGAGCAGGGAGTAAAGCGTG |
| ScKAT6BR | CTGAGATGGTAGCAGCGTAGA |
| ScKAT7F | GTCTCAAAATGCCCCCTGCC |
| ScKAT7R | TCATAGCGGGTCTTACACTCG |
| ScKAT8F | TGGGAAGTATGAGATTGACACCTG |
| ScKAT8R | CCCATCCACCTCAAACAAAGA |
| ScKAT9-1F | TGCCAGACTTGCCAAATGTAG |
| ScKAT9-1R | TCAGGGTGGAAGGGGAATAA |
| ScKTA9-2F | AGGACCTTGGAACGCAGTGT |
| ScKTA9-2R | GTAGGCGTAACAGACCAATCAAA |
| Scp300F | AACCAACAGAGGCACCAAGG |
| Scp300R | TCCTTTAGATGTGTGGGCTCA |
| ScCBPF | CCGAGTGTGCCTTCCCAAAT |
| ScCBPR | ACAAGCCCAGATGTGTGCCC |
| 18SF | TCGGTTCTATTGCGTTGGTTTT |
| 18SR | CAGTTGGCATCGTTTATGGTCA |
在缢蛏基因组中共鉴定出11个ScHAT家族成员, 根据其序列同源性可分为3类亚家族, GNAT类(ScKAT1, ScKAT2B, ScKAT9-1, ScKAT9-2)、MYST类(ScKAT5, ScKAT6A, ScKAT6B, ScKAT7, ScKAT8)和p300/CBP类(Scp300, ScCBP)。对其家族成员的理化性质分析结果显示, ScHAT家族所有成员均为亲水性蛋白; 除Scp300为酸性蛋白外, 其余均为碱性蛋白(表 2)。不同成员外显子数量在5~26个之间, 氨基酸长度在187~2 261 aa之间, 蛋白分子量在19.99~92.40 kDa之间, 等电点(pI)在5.95~9.30, 不稳定系数介于34.44~62.72之间。染色体定位发现, 11个ScHATs均成功定位在缢蛏的7条染色体上。另外, 其他7个物种的全基因组鉴定结果如表 3所示, 太平洋牡蛎和紫色球海胆HAT基因数目最多为13个, 其次人和缢蛏分别为12个和11个; 其中MYST亚家族为最大的一类, 在不同物种中基因数目均为最多; 缢蛏和人HAT各亚家族基因数最相似。
| 基因ID | 基因名 | 亚家族 | 氨基酸数量 | 染色体号 | 染色体位置 | 等电点/pI | 分子量/kDa | 外显子数量 | 亲水性 | 不稳定系数 |
| evm.model.ctg169.36 | ScKAT1 | GNAT | 379 | Hic_asm_8 | 19899672:19907780 | 8.65 | 44.65 | 9 | –0.599 | 54.55 |
| evm.model.ctg313.2 | ScKAT2B | 806 | Hic_asm_10 | 42250573:42264982 | 8.89 | 92.40 | 18 | –0.534 | 50.17 | |
| evm.model.ctg56.31 | ScKAT9-1 | 390 | Hic_asm_0 | 9015516:9025304 | 9.3 | 44.32 | 13 | –0.396 | 36.33 | |
| evm.model.ctg56.32 | ScKAT9-2 | 187 | Hic_asm_0 | 9026664:9031490 | 7.71 | 21.23 | 5 | –0.416 | 34.33 | |
| evm.model.ctg367.24 | ScKAT5 | MYST | 473 | Hic_asm_1 | 4261871:4279716 | 8.29 | 53.97 | 14 | –0.592 | 44.51 |
| evm.model.ctg52.16 | ScKAT6A | 584 | Hic_asm_0 | 53282557:53297791 | 7.23 | 66.57 | 10 | –0.785 | 53.89 | |
| evm.model.ctg122.31 | ScKAT6B | 1 806 | Hic_asm_4 | 73085863:73111539 | 8.07 | 19.99 | 17 | –0.846 | 59.97 | |
| evm.model.ctg77.48 | ScKAT7 | 616 | Hic_asm_0 | 32365113:32372689 | 9.11 | 70.54 | 12 | –0.872 | 50.04 | |
| evm.model.ctg55.51 | ScKAT8 | 415 | Hic_asm_13 | 22940213:22957896 | 7.56 | 48.76 | 10 | –0.643 | 40.29 | |
| evm.model.ctg75.43 | Scp300 | p300/CBP | 670 | Hic_asm_6 | 65986389:66001279 | 5.95 | 73.33 | 10 | –0.606 | 55.83 |
| evm.model.ctg53.44 | ScCBP | 2 261 | Hic_asm_6 | 62838874:62869874 | 8.81 | 25.08 | 26 | –0.767 | 62.72 |
| 物种 | GNAT | MYST | p300/CBP | 总计 |
| 人(H. sapiens) | 5 | 5 | 2 | 12 |
| 白氏文昌鱼(B. belcheri) | 2 | 6 | 2 | 10 |
| 秀丽隐杆线虫(C. elegans) | 0 | 4 | 1 | 5 |
| 紫色球海胆(S. purpuratus) | 3 | 9 | 1 | 13 |
| 加州双斑蛸(O. bimaculoides) | 2 | 6 | 2 | 10 |
| 缢蛏(S. constricta) | 4 | 5 | 2 | 11 |
| 太平洋牡蛎(C. gigas) | 2 | 6 | 5 | 13 |
| 虾夷扇贝(M. yessoensis) | 2 | 6 | 1 | 9 |
用MrBayes构建了缢蛏与其他物种HAT基因家族的系统发育树(图 1)。该进化树共分成7个独立的分支, KAT2A/2B和KAT9分支形成独立的一支(GNAT); KAT5、KAT6A/6B、KAT7和KAT8四个分支形成最大的一支(MYST)。不同物种HAT蛋白具有高度同源性, ScHAT各成员均与太平洋牡蛎、虾夷扇贝、加州双斑蛸同源基因形成独立分支, 再依次与紫色球海胆、白氏文昌鱼、秀丽隐杆线虫和人聚类。
 |
| 图 1 缢蛏与其他物种HAT基因家族的系统发育分析 Fig. 1 Phylogenetic analysis of HAT gene family in S. constricta and other species 注: 右上角为3类亚家族成员的不同颜色区块, 红色字体代表缢蛏基因。Hs: 人; Bb: 白氏文昌鱼; Ce: 秀丽隐杆线虫; Sc: 缢蛏; Cg: 太平洋牡蛎; My: 虾夷扇贝; Ob: 加州双斑蛸; Sp: 紫色球海胆 |
基于SMART结构域预测, 比较了缢蛏、太平洋牡蛎和人HAT蛋白的结构域(表 4)。结果显示, GNAT亚家族的KAT2成员大多含有PCAF_N结构域和Bromo结构域; KAT9成员大多包含一个Radical_SAM结构域特征; 除ScKAT9-1之外, 其余GNAT亚家族成员均含有N-Acetyltransf_1或C-Acetyltransf_1结构域。MYST亚家族成员中, 除ScKAT6A和CgKAT6B-2只包含1个PHD-SAM_1结构域外, 其余成员均含有zf-MYST和MOZ_SAS结构域; 另外KAT5和KAT8还有一个Tudor-knot结构域(HsKAT8除外); KAT6成员存在一个PHD结构域特征, 以及KAT7成员包含一个C2HC-zf结构域。p300/CBP亚家族成员含有zf-TAZ、KIX、Bromo等结构域。
| 基因ID | 基因名 | KAT分类 | 基因家族 | 结构域组成 |
| evm.model.ctg169.36 | ScKAT1 | KAT1 | GNAT | Hat1_N |
| NP_066564.2 | HsKAT2A-1 | KAT2A | PCAF_N-Acetyltransf_1-Bromo | |
| XP_006721881.1 | HsKAT2A-2 | Acetyltransf_1-Bromo | ||
| XP_011432298.2 | CgKAT2A | PCAF_N-Acetyltransf_1-Bromo | ||
| evm.model.ctg313.2 | ScKAT2B | KAT2B | PCAF_N-Acetyltransf_1-Bromo | |
| NP_003875.3 | HsKAT2B-1 | PCAF_N-Acetyltransf_1-Bromo | ||
| XP_005265585.1 | HsKAT2B-2 | PCAF_N-Acetyltransf_1 | ||
| evm.model.ctg56.31 | ScKAT9-1 | KAT9 | Radical_SAM-Radical_SAM_C | |
| evm.model.ctg56.32 | ScKAT9-2 | Acetyltransf_1 | ||
| NP_060561.3 | HsKAT9 | Radical_SAM-Radical_SAM_C-Acetyltransf_1 | ||
| XP_011425652.2 | CgKAT9 | Radical_SAM-Radical_SAM_C-Acetyltransf_1 | ||
| evm.model.ctg367.24 | ScKAT5 | KAT5 | MYST | Tudor-knot-zf-MYST-MOZ_SAS |
| XP_006718484.1 | HsKAT5 | Tudor-knot-zf-MYST-MOZ_SAS | ||
| XP_034324845.1 | CgKAT5 | Tudor-knot-zf-MYST-MOZ_SAS | ||
| evm.model.ctg52.16 | ScKAT6A | KAT6A | PHD-SAM_1 | |
| XP_011542958.1 | HsKAT6A | PHD-zf-MYST-MOZ_SAS | ||
| evm.model.ctg122.31 | ScKAT6B | KAT6B | PHD-zf-C2HC-zf-MYST-MOZ_SAS | |
| XP_016871492.1 | HsKAT6B | PHD-zf-MYST-MOZ_SAS | ||
| XP_034318334.1 | CgKAT6B-1 | Linker_histone-PHD-zf-C2HC-zf-MYST-MOZ_SAS | ||
| XP_011442343.2 | CgKAT6B-2 | PHD-SAM_1 | ||
| evm.model.ctg77.48 | ScKAT7 | KAT7 | zf-C2HC-zf-MYST-MOZ_SAS | |
| NP_008998.1 | HsKAT7 | zf-C2HC-zf-MYST-MOZ_SAS | ||
| XP_034330903.1 | CgKAT7-1 | zf-C2HC-zf-MYST-MOZ_SAS | ||
| XP_034329657.1 | CgKAT7-2 | zf-C2HC-zf-MYST-MOZ_SAS | ||
| evm.model.ctg55.51 | ScKAT8 | KAT8 | Tudor-knot-zf-MYST-MOZ_SAS | |
| XP_011544272.1 | HsKAT8 | zf-MYST-MOZ_SAS | ||
| XP_011434277.2 | CgKAT8 | Tudor-knot-zf-MYST-MOZ_SAS | ||
| evm.model.ctg75.43 | Scp300 | p300/CBP | zf-TAZ-KIX | |
| XP_011438001.2 | Cgp300-1 | zf-TAZ-zf-TAZ-KIX | ||
| XP_034303589.1 | Cgp300-2 | zf-TAZ | ||
| XP_034303586.1 | Cgp300-3 | zf-TAZ-KIX | ||
| XP_034303592.1 | Cgp300-4 | KIX | ||
| evm.model.ctg53.44.1 | ScCBP | zf-TAZ-KIX-Bromo-DUF902-HAT_KAT11-ZZ-zf-TAZ-Creb_binding | ||
| NP_001420.2 | Hsp300 | zf-TAZ-KIX-Bromo-DUF902-HAT_KAT11-ZZ-zf-TAZ-Creb_binding | ||
| XP_016878433.1 | HsCBP | zf-TAZ-KIX-Bromo-DUF902-HAT_KAT11-ZZ-zf-TAZ-Creb_binding | ||
| XP_034303595.1 | CgCBP | zf-TAZ-KIX-Bromo-DUF902-HAT_KAT11-ZZ-zf-TAZ-Creb_binding |
缢蛏、太平洋牡蛎和人HAT家族基因的motif分析结果如图 2所示。HAT不同亚家族之间序列分化明显, 共鉴定出18个保守的motif。除ScKAT6A和CgKAT6B-2外, MYST亚家族的成员从5′到3′方向依次含有motif 8、motif 5、motif 9、motif 1、motif 2、motif 6, 其中KAT6成员具有1或2个motif 3; 在GNAT亚家族中, 除KAT1和KAT9只含有1或2个保守motif外, motif 16、motif 7、motif 4、motif 14、motif 15存在于所有KAT2成员中; p300/CBP亚家族也具有其独特的motif序列。此外, 同一亚家族成员存在motif新增或丢失的情况, 如CgKAT5与ScKAT8与其同源基因KAT5和KAT8分别新增了1个motif 4和motif 10; 而HsKAT2B-2与其同源基因KAT2则丢失了motif 12。
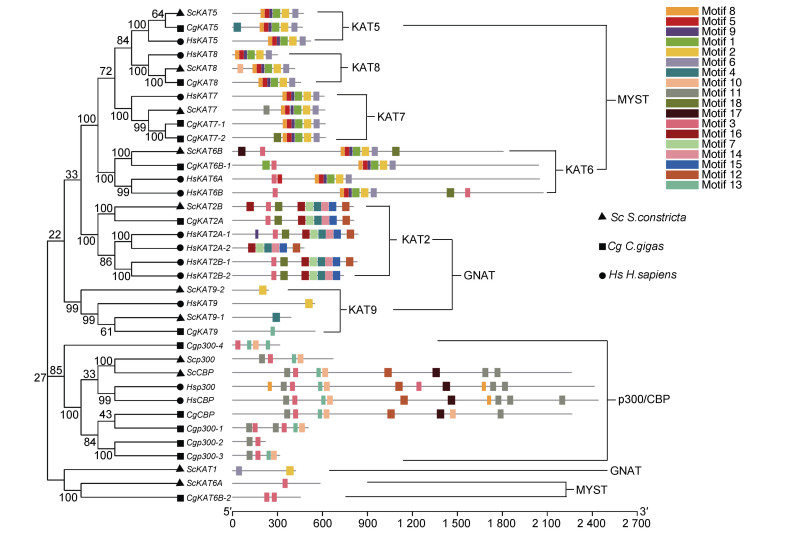 |
| 图 2 缢蛏、太平洋牡蛎和人HAT基因家族motif分析 Fig. 2 Motif analysis of HAT gene family in S. constricta, C. gigas and H. sapiens |
基于转录组分析ScHATs在缢蛏早期发育时期的表达规律, 结果显示, 它们在不同发育时期的表达趋势呈现多变性(图 3)。GNAT亚家族中, ScKAT1和ScKAT9-1均在4细胞期表达量最高, 而ScKAT2B和ScKAT9-2则在壳顶幼虫时期表达量最高。MYST亚家族中, ScKAT5随着缢蛏的生长发育表达量逐渐升高, 在稚贝期达到最高; 其他4个基因呈现出相似的表达模式, 均在发育初期(D形幼虫时期之前)表达量较高, 而在壳顶幼虫和稚贝期表达量明显下降。p300/CBP亚家族的2个成员也表现出不同的表达情况, Scp300在稚贝期高表达, 而ScCBP在D形幼虫期表达量最高。
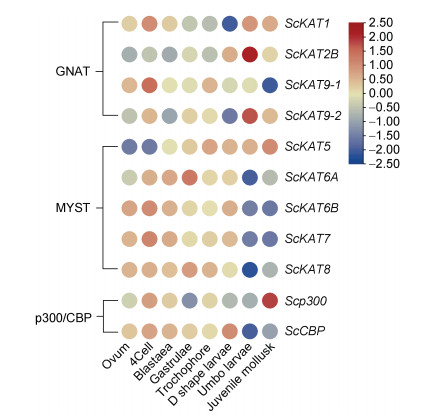 |
| 图 3 ScHATs在不同发育时期中的表达谱 Fig. 3 Expression profiles of ScHATs in different developmental stages 注: 8个发育时期为卵子(Ovum)、4细胞(4 cell)、囊胚(Blastaea)、原肠胚(Gastrulae)、担轮幼虫(Trochophore)、D形幼虫(D-shape larvae)、壳顶幼虫(Umbo larvae)、稚贝(Juvenile mollusk)。图例表示FPKM对数归一化 |
对ScHATs在高氨氮、高温胁迫下的表达模式进行分析(图 4)。在氨氮胁迫下, ScHATs在鳃组织中的表达响应高于肝胰腺, 其中ScKAT9-2、ScKAT8和Scp300三个基因的表达显著升高(P < 0.05), 而ScKAT2B的表达显著降低(P < 0.05)。在高温胁迫后, ScHATs在鳃和肝胰腺中表达趋势几乎相同, 其中Scp300在肝胰腺中的表达量显著降低(P < 0.05)。
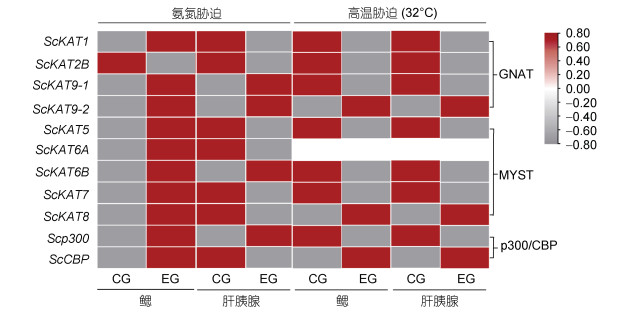 |
| 图 4 ScHATs在高氨氮、高温胁迫下鳃和肝胰腺的表达模式 Fig. 4 Expression profiles of ScHATs in gills and hepatopancreas under ammonia nitrogen and thermal stresses 注: CG: 对照组; EG: 实验组。图例表示FPKM对数归一化 |
用qRT-PCR方法, 对11个ScHATs基因在副溶血弧菌感染后鳃中的表达水平进行检测(图 5)。结果显示, 所有ScHATs的相对表达量在副溶血弧菌感染后均出现显著上调(P < 0.05), 并呈现时间依赖性的表达模式。多数基因在24 h达到较高水平(如ScKAT6A和ScKAT7), ScKAT1和ScKAT6A在48 h表达量最高, 其他基因多在96 h达到最高水平。其中Scp300有所不同, 在感染后3~12 h表达水平显著下降后, 于24 h后开始显著上升。
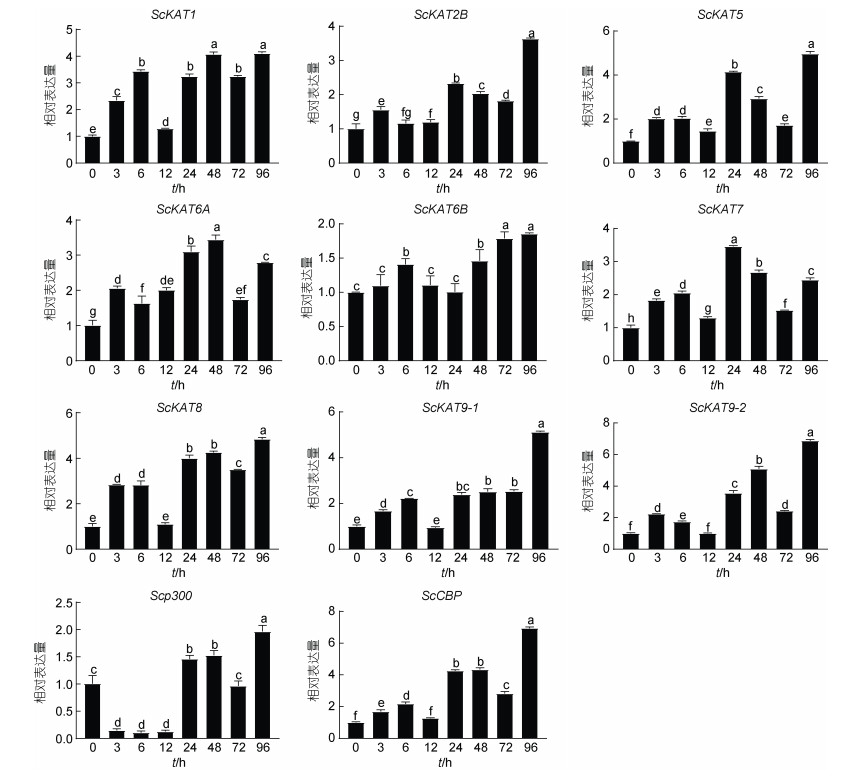 |
| 图 5 ScHATs在副溶血弧菌感染后鳃中的表达模式 Fig. 5 Relative expression of ScHATs in gills after V. parahemolyticus infection 注: 以对照组0 h的表达量为参照, 用18S rRNA归一化计算相对表达量。数据以平均值±S.E.表示(n=3)。不同字母表示时间点差异显著(P < 0.05) |
由HATs基因介导的组蛋白乙酰化修饰在转录激活、基因沉默、细胞周期调控、DNA复制修复、染色体组装等多个细胞过程中发挥至关重要的调控作用(Mai et al, 2009), 而组蛋白乙酰化与基因表达调控密切相关(Imran et al, 2019)。已有研究发现, HATs在棉花、玉米高等植物生长发育以及对环境胁迫的适应调节中发挥关键作用, 如同属不同种的3种棉花HAT基因数目差异巨大, 且HAT基因在纤维发育不同时期以及激素、干旱、重金属等不同因子胁迫下的表达差异显著(Imran et al, 2019); 在玉米基因组中鉴定出6个HAT家族基因, 它们在不同组织和生物/非生物胁迫下呈现出明显不同的表达规律(马宇馨等, 2020)。然而, 截至目前, 对动物HAT家族基因的研究鲜有报道。本研究通过全基因组分析在缢蛏基因组中鉴定出11个HAT家族基因, 根据序列同源性分为GNAT、MYST和p300/CBP三类亚家族, 这种多个亚家族的存在暗示其在缢蛏长期进化适应中的功能分化; 通过对缢蛏、太平洋牡蛎、人等8个物种HAT亚家族基因数目的比较发现, 每个亚家族在物种间分布不均匀, 其中MYST是最大的亚家族, 与以往研究一致(Hodawadekar et al, 2007)。此外, 保守基序和结构域预测结果表明, 在同一系统发育亚群中, 大多数成员具有各自保守的基因结构域, 因而推测ScHATs与其同源基因具有相似的功能, 在水稻、番茄的研究中也得到相似结论(Liu et al, 2012; 史建磊等, 2020)。
研究发现, HATs在机体发育中发挥关键性作用(Gan et al, 2021)。MYST亚家族主要作为一种发育调节剂, 参与干细胞的生长和自我修复过程(Voss et al, 2009); GCN5/PCAF和p300/CBP亚家族也被证实在脊椎动物的多个生长发育过程发挥重要的调控功能(Goodman et al, 2000; Koutelou et al, 2021)。GNAT亚家族的KAT2成员大多具有Bromo结构域, 可通过与乙酰化赖氨酸的特异性互作发挥乙酰化酶作用, 进而影响体内基因转录和细胞生长(Zeng et al, 2002; Ren et al, 2016)。缺失GCN5等位基因的果蝇突变体出现卵发生、变态以及成虫附肢等异常的现象(Carre et al, 2005)。与大多数双壳类一样, 缢蛏幼虫发育从细胞分化迅速的细胞期经卵裂、变态发育到浮游的D形幼虫不到24 h (林笔水等, 1984; 徐小伟等, 2015)。ScHATs在缢蛏不同发育时期的表达量呈现动态变化规律, 其在幼虫发育前期表达量普遍高于发育后期(壳顶幼虫和稚贝), 因此推测HAT基因对缢蛏的正常发育至关重要, 可能通过基因的乙酰化作用改变靶基因的表达水平, 进而参与早期发育的基因调控网络。
已有报道显示, HAT家族基因在响应环境胁迫中发挥重要功能。棉花HATs的相对表达量在温度、盐度和重金属等胁迫下发生显著变化(Imran et al, 2019); 油菜p300/CBP亚家族能够提高其应对外界环境变化的能力(Li et al, 2014); 拟南芥则通过调节GCN5基因的表达响应极端温度变化(Gan et al, 2021)。本研究中, 高浓度氨氮胁迫缢蛏后, 鳃中ScKAT9-2、ScKAT8和Scp300 3个基因的表达量显著升高, 而含有PCAF结构域的ScKAT2B的表达量显著降低(P < 0.05)。已有研究发现, 在金属镍环境暴露下会导致p300/CBP的相关蛋白PCAF低表达(Kim et al, 2012), 这与本研究结果相似。另外, 在缢蛏鳃和肝胰腺中HATs的表达模式差异较大, 反映这两个器官在氨氮代谢中的途径和机制有所不同(陈凯锋等, 2020)。在高温胁迫下, HATs在缢蛏鳃和肝胰腺中的表达趋势几乎相同, 这说明不同组织在适应高温响应的调节机制相似, 如通过增加抗氧化酶系统、提高细胞的渗透调节物质等提高耐热性, 在此过程中一些ScHATs基因表达量的显著变化表明组蛋白修饰的表观遗传在响应高温胁迫中发挥重要调控作用。在对仿刺参(Apostichopus japonicus)的高温胁迫下, 也发现组蛋白去乙酰化酶3 (HDAC3)和组蛋白甲基转移酶(histone methyltransferase, MLL5)等表观遗传调控基因表达量显著上调(李尚俊等, 2017)。然而, 目前HAT家族基因调控环境适应的机理尚需开展大量深入研究。
本研究中, 在副溶血弧菌胁迫下所有ScHATs基因在缢蛏鳃中的表达量均发生显著变化(P < 0.05)。与对照组相比, 一般呈现三个峰, 即3~6 h、24~48 h、96 h, 这种表达模式说明随着处理时间的延长, HATs家族基因可能通过调节组蛋白乙酰化水平参与了缢蛏在这些时间点的表达调控, 反映了缢蛏对环境的动态适应过程, 推测ScHATs的高表达变化可能是缢蛏应对细菌感染时参与调节免疫相关基因转录水平的结果。已有大量研究证明, 许多HAT家族基因参与免疫及抗病过程, 如敲除稻瘟病菌(Magnaporthe oryzae) HAT基因使突变体生长速度和孢子产量显著降低, 进而抑制其侵染能力(Yin et al, 2019); KAT6A中的PHD-zf结构域存在于一个自身免疫调节因子(AIRE)蛋白中(Musco et al, 2008), 通过与生物胁迫耐受性的其他蛋白质伴侣相互作用发挥功能(Waziri et al, 2020); 多数KAT7中包含C2HC-zf结构域, 而C2HC-zf结构域已在凡纳滨对虾(Litopenaeus vannamei)中被证实具有抗菌作用(Yang et al, 2021); KAT9中包含的Radical_SAM结构域可通过与病毒蛋白结合发挥抗病毒活性, 在病毒感染的先天性免疫反应中起着重要作用(Ghosh et al, 2020)。基于上述已有研究, 结合本研究的结构域分析, 不难推测分别包含PHD-zf、C2HC-zf和Radical_SAM结构域的ScKAT6A、ScKAT7、ScKAT9-1在缢蛏病原侵染的防御过程中发挥重要的抗菌作用, 而不同基因表达量和时间效应的差异也正反映了ScHATs家族基因不同成员的功能分化及其调控机制差别。
4 结论本研究在缢蛏基因组中鉴定出11个ScHAT基因, 比较分析了HAT家族成员的蛋白质理化性质、结构域、保守基序及系统发育关系, 并将其分为GNAT、MYST、p300/CBP三类。通过转录组表达谱分析和qRT-PCR验证, 探究了缢蛏在不同发育阶段、不同环境因子(高氨氮、高温)胁迫和副溶血弧菌感染条件下ScHAT家族成员的表达变化, 结果表明ScHATs基因功能具有多样性, 可能在早期发育及极端环境响应过程中发挥重要作用。该研究将丰富人们对贝类HAT家族基因系统进化的认识, 也为其功能研究提供重要参考。
马宇馨, 杜璇玥, 李肖慧, 等, 2020. 玉米组蛋白乙酰转移酶的鉴定与表达规律分析. 河北农业大学学报, 43(5): 20-26 |
史建磊, 熊自立, 李涛, 等, 2020. 番茄组蛋白乙酰转移酶(HAT)的全基因组鉴定与分析. 江苏农业学报, 36(3): 666-674 DOI:10.3969/j.issn.1000-4440.2020.03.019 |
李尚俊, 孙国华, 李雪燕, 等, 2017. 高温胁迫下仿刺参表观遗传调控相关基因的表达特征. 中国水产科学, 24(3): 470-476 |
杨帅, 2020. 组蛋白及其乙酰化修饰在珍珠贝移植免疫中的作用和机制[D]. 湛江: 广东海洋大学: 33-48
|
陈凯锋, 董迎辉, 姚韩韩, 等, 2020. 缢蛏(Sinonovacula constricta)氨氮胁迫应答miR-8245a-5p靶基因GOT验证及其表达特征分析. 海洋与湖沼, 51(2): 388-394 |
林笔水, 吴天明, 1984. 温度和盐度对缢蛏浮游幼虫发育的影响. 生态学报, 4(4): 385-392 |
郑余琦, 郑忠明, 秦文娟, 2017. 缢蛏(Sinonovacula constricta)生物扰动对养殖废水处理系统中沉积物磷赋存形态垂直分布的影响. 海洋与湖沼, 48(1): 161-170 |
徐小伟, 张鹏飞, 黄妙琴, 等, 2015. 波纹巴非蛤早期发育的扫描电镜观察. 渔业研究, 37(4): 263-269 |
BANNISTER A J, FALCÃO A M, CASTELO-BRANCO G, 2017. Histone modifications and histone variants in pluripotency and differentiation [M] // GÖNDÖR A. Chromatin Regulation and Dynamics. San Diego, CA: Academic Press: 35-64, doi: 10.1016/b978-0-12-803395-1.00002-2.
|
BANNISTER A J, KOUZARIDES T, 2011. Regulation of chromatin by histone modifications. Cell Research, 21(3): 381-395 DOI:10.1038/cr.2011.22 |
CARRE C, SZYMCZAK D, PIDOUX J, et al, 2005. The histone H3 acetylase dGcn5 is a key player in Drosophila melanogaster metamorphosis. Molecular and Cellular Biology, 25(18): 8228-8238 DOI:10.1128/MCB.25.18.8228-8238.2005 |
CHEN C J, CHEN H, ZHANG Y, et al, 2020. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 13(8): 1194-1202 DOI:10.1016/j.molp.2020.06.009 |
DONG Y H, ZENG Q F, REN J F, et al, 2020. The chromosome-level genome assembly and comprehensive transcriptomes of the razor clam (Sinonovacula constricta). Frontiers in Genetics, 11: 664 DOI:10.3389/fgene.2020.00664 |
EBERHARTER A, BECKER P B, 2002. Histone acetylation: a switch between repressive and permissive chromatin. EMBO Reports, 3(3): 224-229 DOI:10.1093/embo-reports/kvf053 |
GAN L, WEI Z Z, YANG Z R, et al, 2021. Updated mechanisms of GCN5-the monkey king of the plant kingdom in plant development and resistance to abiotic stresses. Cells, 10(5): 979 DOI:10.3390/cells10050979 |
GAO S Q, LI L Z, HAN X L, et al, 2021. Genome-wide identification of the histone acetyltransferase gene family in Triticum aestivum. BMC Genomics, 22(1): 49 DOI:10.1186/s12864-020-07348-6 |
GHOSH S, MARSH E N G, 2020. Viperin: An ancient radical SAM enzyme finds its place in modern cellular metabolism and innate immunity. Journal of Biological Chemistry, 295(33): 11513-11528 DOI:10.1074/jbc.REV120.012784 |
GOODMAN R H, SMOLIK S, 2000. CBP/p300 in cell growth, transformation, and development. Genes & Development, 14(13): 1553-1577 |
HO L, CRABTREE G R, 2010. Chromatin remodeling during development. Nature, 463(7280): 474-484 DOI:10.1038/nature08911 |
HODAWADEKAR S C, MARMORSTEIN R, 2007. Chemistry of acetyl transfer by histone modifying enzymes: structure, mechanism and implications for effector design. Oncogene, 26(37): 5528-5540 DOI:10.1038/sj.onc.1210619 |
IMRAN M, SHAFIQ S, FAROOQ M A, et al, 2019. Comparative genome-wide analysis and expression profiling of histone acetyltransferase (HAT) gene family in response to hormonal applications, metal and abiotic stresses in cotton. International Journal of Molecular Sciences, 20(21): 5311 DOI:10.3390/ijms20215311 |
KATOH K, STANDLEY D M, 2013. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Molecular Biology and Evolution, 30(4): 772-780 DOI:10.1093/molbev/mst010 |
KIM H L, SEO Y R, 2012. Molecular and genomic approach for understanding the gene-environment interaction between Nrf2 deficiency and carcinogenic nickel-induced DNA damage. Oncology Reports, 28(6): 1959-1967 DOI:10.3892/or.2012.2057 |
KOUTELOU E, FARRIA A T, DENT S Y R, 2021. Complex functions of Gcn5 and Pcaf in development and disease. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms, 1864(2): 194609 DOI:10.1016/j.bbagrm.2020.194609 |
KOUZARIDES T, 2007. Chromatin modifications and their function. Cell, 128(4): 693-705 DOI:10.1016/j.cell.2007.02.005 |
LATRASSE D, BENHAMED M, HENRY Y, et al, 2008. The MYST histone acetyltransferases are essential for gametophyte development in Arabidopsis. BMC Plant Biology, 8: 121 DOI:10.1186/1471-2229-8-121 |
LI H, YAN S H, ZHAO L, et al, 2014. Histone acetylation associated up-regulation of the cell wall related genes is involved in salt stress induced maize root swelling. BMC Plant Biology, 14: 105 DOI:10.1186/1471-2229-14-105 |
LIU X, LUO M, ZHANG W, et al, 2012. Histone acetyltransferases in rice (Oryza sativa L.): Phylogenetic analysis, subcellular localization and expression. BMC Plant Biology, 12: 145 DOI:10.1186/1471-2229-12-145 |
MAI A, ROTILI D, TARANTINO D, et al, 2009. Identification of 4-hydroxyquinolines inhibitors of p300/CBP histone acetyltransferases. Bioorganic & Medicinal Chemistry Letters, 19(4): 1132-1135 |
MUSCO G, PETERSON P, 2008. PHD finger of autoimmune regulator: an epigenetic link between the histone modifications and tissue-specific antigen expression in thymus. Epigenetics, 3(6): 310-314 DOI:10.4161/epi.3.6.7182 |
PADGETT L R, LENTINI J M, HOLMES M J, et al, 2018. Elp3 and RlmN: A tale of two mitochondrial tail-anchored radical SAM enzymes in Toxoplasma gondii. PLoS One, 13(1): e0189688 DOI:10.1371/journal.pone.0189688 |
PATEL D J, WANG Z X, 2013. Readout of epigenetic modifications. Annual Review of Biochemistry, 82(1): 81-118 DOI:10.1146/annurev-biochem-072711-165700 |
REN C, ZENG L, ZHOU M M, 2016. Preparation, biochemical analysis, and structure determination of the bromodomain, an acetyl-lysine binding domain. Methods in Enzymology, 573: 321-343 |
RONQUIST F, HUELSENBECK J P, 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models. Bioinformatics, 19(12): 1572-1574 DOI:10.1093/bioinformatics/btg180 |
STRUHL K, 1998. Histone acetylation and transcriptional regulatory mechanisms. Genes & Development, 12(5): 599-606 |
VOSS A K, THOMAS T, 2009. MYST family histone acetyltransferases take center stage in stem cells and development. BioEssays, 31(10): 1050-1061 DOI:10.1002/bies.200900051 |
WAZIRI A, SINGH D K, SHARMA T, et al, 2020. Genome-wide analysis of PHD finger gene family and identification of potential miRNA and their PHD finger gene specific targets in Oryza sativa indica. Non-coding RNA Research, 5(4): 191-200 DOI:10.1016/j.ncrna.2020.10.002 |
YANG L W, WANG Z A, ZUO H L, et al, 2021. The LARK protein is involved in antiviral and antibacterial responses in shrimp by regulating humoral immunity. Developmental & Comparative Immunology, 114: 103826 |
YAO T P, OH S P, FUCHS M, et al, 1998. Gene dosage-dependent embryonic development and proliferation defects in mice lacking the transcriptional integrator p300. Cell, 93(3): 361-372 DOI:10.1016/S0092-8674(00)81165-4 |
YIN Z Y, CHEN C, YANG J, et al, 2019. Histone acetyltransferase MoHat1 acetylates autophagy-related proteins MoAtg3 and MoAtg9 to orchestrate functional appressorium formation and pathogenicity in Magnaporthe oryzae. Autophagy, 15(7): 1234-1257 DOI:10.1080/15548627.2019.1580104 |
ZENG L, ZHOU M M, 2002. Bromodomain: an acetyl-lysine binding domain. FEBS Letters, 513(1): 124-128 DOI:10.1016/S0014-5793(01)03309-9 |
 2022, Vol. 53
2022, Vol. 53


