
中国海洋湖沼学会主办。
文章信息
- 李嘉华, 陈舜, 陈万东, 谢尚微, 倪孝品, 郑小东. 2022.
- LI Jia-Hua, CHEN Shun, CHEN Wan-Dong, XIE Shang-Wei, NI Xiao-Pin, ZHENG Xiao-Dong. 2022.
- 南麂列岛中华蛸(Octopus sinensis)形态与遗传多样性分析
- MORPHOLOGICAL AND GENETIC DIVERSITY ANALYSIS OF OCTOPUS SINENSIS IN NANJI ISLANDS
- 海洋与湖沼, 53(2): 486-495
- Oceanologia et Limnologia Sinica, 53(2): 486-495.
- http://dx.doi.org/10.11693/hyhz20210900225
文章历史
-
收稿日期:2021-09-29
收修改稿日期:2021-11-16
2. 中国海洋大学海水养殖教育部重点实验室山东青岛 266003;
3. 南麂列岛国家海洋自然保护区管理局 浙江温州 325400
2. Key Laboratory of Mariculture, Ministry of Education, Ocean University of China, Qingdao 266003, China;
3. Nanji Islands National Marine Nature Reserve Administration, Wenzhou 325400, China
中华蛸(Octopus sinensis d’Orbigny, 1841)隶属软体动物门(Mollusca)、头足纲(Cephalopoda)、八腕目(Octopoda)、蛸科(Octopodidae)、蛸属(Octopus), 广泛分布于日本、韩国以及中国沿海的岩礁、砂底, 是重要的经济蛸类。1834年, d’Orbigny整理了现存头足类图谱, 根据当时日本百科全书《Wakansansaizue》添加了中华蛸的相关插图, 于1841年描述并命名新种—中华蛸(d’Orbigny, 1835-1848)。Sasaki(1929)指出中华蛸为真蛸(O. vulgaris)的同物异名, 此后, 日本采集的样品均被称为“真蛸”。21世纪初, 日本学者仍沿用种名真蛸(Sakaguchi et al, 2000, 2005), Warnke等(2004)通过线粒体16S rRNA基因与COⅢ基因联合分析, 也认为在日本和中国台湾沿海采集的野生样本与真蛸为同一物种。然而, 由于存在地理隔离, Norman(2000)认为日本分布的所谓“真蛸”与真蛸模式种很可能不是同一物种。Norman等(2005)在蛸科分类修订中将“中华蛸”暂时归类于不确定种名。近年来, 基于形态学与分子标记的研究结果表明, 真蛸存在多个隐存种, 是一个大的复合物种(O. vulgaris complex species), 如O. insularis、O. tetricus、O. cf. tetricus、O. americanus (Amor et al, 2017a, 2017b; Van Nieuwenhove et al, 2019; Avendaño et al, 2020)。Gleadall(2016)对日本九州与本州沿海采集的“真蛸”进行了重描述, 发现与真蛸存在显著形态差异, 重新确定O. sinensis d’Orbigny, 1841为有效种名。Amor等(2017b)采用形态学与分子标记法得到的结果也支持上述结论。
国内已发表的论文专著多沿用物种名“真蛸”, 指出该物种为我国东南沿海重要经济蛸类, 其肉嫩味美, 营养价值高, 是妇女生乳的滋补品(董正之, 1988), 有关其亲体培育、胚胎发育、幼体生长、生理生态以及基因组学、转录组学等方面研究已有报道(蔡厚才等, 2007, 2009; 刘兆胜等, 2011; 郑小东等, 2011; 孙田田等, 2012; 冯雪等, 2013; 肖懿哲等, 2019; Li et al, 2020)。然而, 该物种个体大, 不易保存, 我国在其分类、遗传多样性方面鲜有文献记载。本文采用形态参数与分子标记相结合的分析方法研究南麂列岛重要经济贝类“真蛸”(叶鹏等, 2006)以确定其正确种名, 旨在为南麂列岛种质资源保护、合理开发利用以及丰富保护区物种基因库提供理论依据。
1 材料与方法 1.1 实验材料2020年6~12月, 从浙江省南麂列岛海域(大檑岛、马祖岙、上马鞍、下马鞍、三盘尾、柴屿)采捕活体样品共44只, 于–30 ℃冷冻保存。
1.2 形态学指标测量选取其中胴体完整、腕无残缺的样品37只(15雄、22雌)进行形态学测量。形态学测量指标及方法参见Roper等(1983)、Norman等(1997b)及Liao等(2009), 测量指标如下: 全长(TL, total length)、体重(TW, total weight)、胴背长(ML, mantle length)、胴背宽(MW, mantle width)、胴腹长(VML, ventral mantle length)、头宽(HW, head width)、外侧漏斗长(FL, funnel length)、内侧漏斗长(FFL, free funnel length)、腕间膜深(WD, web depth)、腕间膜式(WF, web formula)、腕长(AL, arm length)、腕式(AF, arm formula)、腕宽(AW, arm width)、右三腕吸盘数(SNR3, sucker number on 3rd arm of right side)、左三腕吸盘数(SNL3, sucker number on 3rd arm of left side)、吸盘直径(SD, sucker diameter)、舌叶长(LL, ligula length)、交接基长(CaL, calamus length)。
1.3 形态学数据处理与分析用于形态学分析的5个群体数据见表 1。除本文的南麂列岛数据, 其他中华蛸与真蛸数据引自Amor等(2017b)。
| 群体 | 物种 | 缩写 | 雄性 | 雌性 | |||
| 样本数 | 平均胴背长±标准差 | 样本数 | 平均胴背长±标准差 | ||||
| 南麂列岛 | 待定种 | NJLD | 15 | 97.47±24.45 | 22 | 116.95±32.00 | |
| 宁德 | 中华蛸 | ND | 4 | 136.57±23.29 | 2 | 118.76±4.54 | |
| 宜兰 | 中华蛸 | YL | 4 | 108.79±22.60 | 8 | 119.51±11.72 | |
| 九州 | 中华蛸 | JZ | 2 | 106.55±18.45 | 5 | 95.60±9.68 | |
| 西班牙 | 真蛸 | SP | 10 | 114.56±15.59 | 10 | 120.18±11.52 | |
| 注: 平均胴背长±标准差的单位为mm | |||||||
为消除不同规格个体对特征指标的影响, 对数据进行标准化处理, 标准化指数定义如下:
ALLI: 左腕长/胴背长×100; ALRI: 右腕长/胴背长×100; AWI: 腕宽/胴背长×100; CaLI: 交接基长/舌叶长×100; FLI: 外侧漏斗长/胴背长×100; FFLI: 内侧漏斗长/外侧漏斗长×100; HWI: 头宽/胴背长×100; LLI: 舌叶长/茎化腕长×100; MWI: 胴背宽/胴背长×100; OAI: 茎化腕长/左三腕长×100; SDIn: 普通吸盘直径/胴背长×100; SDIe: 扩大吸盘直径/胴背长×100; WDI: 腕间膜深/最长腕长×100。
主成分分析 根据雌性19个指标ML、VML、MWI、HWI、FLI、FFLI、ALLI1、ALLI2、ALLI3、ALLI4、ALRI1、ALRI2、ALRI3、ALRI4、AWI、SDIn、SNL3、SNR3、WDI, 雄性22个指标ML、VML、MWI、HWI、FLI、FFLI、WDI、ALLI1、ALLI2、ALLI3、ALLI4、ALRI1、ALRI2、ALRI3、ALRI4、AWI、SDIn、SDIe、SNL3、SNR3、LLI、CaLI, 计算出互不关联的主成分。主成分贡献率和累计贡献率的计算方法参照Brzeski等(1988)。
判别分析 采用逐步判别法(SPSS 25.0)对雌性和雄性指标(同主成分)进行判别分析, 建立5个群体的判别函数。利用判别函数得分绘制前两个判别函数系数散点图, 判别准确率的计算公式为:
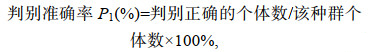 (1)
(1)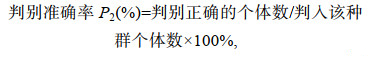 (2)
(2)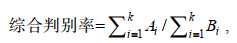 (3)
(3)式中, Ai和Bi分别为第i个群体中被判别正确的个体数和实际判别的个体数, k为群体数。
聚类分析 取雌性和雄性各群体指标(同主成分)的平均值, 采用欧式距离最短系统聚类法进行聚类分析(SPSS 25.0)。
1.4 COI序列分析分别从采集样品的胴体部剪取少许肌肉组织保存于无水乙醇中, 采用CTAB法提取总DNA, 用引物Primer-F: 5′-GGTCAACAAATCATAAAGATATTGG-3′, Primer-R: 5′-TAAACTTCAGGGTGACCAAAAAATCA-3′ (Folmer et al, 1994)扩增COI片段, PCR反应体系: H2O 19 μL, Mix酶25 μL, F与R引物各2.5 μL, DNA 1 μL。PCR反应程序: 94 ℃ 4 min, 然后94 ℃ 10 s, 55 ℃ 20 s, 72 ℃ 30 s, 共32个循环, 最后72 ℃ 5 min延伸。扩增产物经琼脂糖凝胶电泳检测, 获得23个体扩增产物用于双向测序。
测序完成后, 用DNASTAR软件包中的SeqMan对测序结果进行拼接, 使用MEGA X (Kumar et al, 2018)中的Clustal W将全部序列进行多重比对分析, 序列已上传至GenBank (序列号: OK001740~ OK00 1762)。利用DnaSP v5 (Librado et al, 2009)计算群体的遗传多样性参数, 并将23条COI序列与GenBank下载的真蛸(O. vulgaris)、中华蛸(O. sinensis)、O. americanus、O. insularis、O. tetricus、O. cf. tetricus的COI序列(表 2)构建系统发育树, 幽灵蛸Vampyroteuthis infernalis (AB385880)作为外群。建树前, 用ModelFinder (Kalyaanamoorthy et al, 2017)选择构建系统发育树的最佳模型, 用IQ-TREE (Nguyen et al, 2015)构建最大似然系统发育树。在计算真蛸复合体各物种间的遗传距离时, 利用MEGA X (Kumar et al, 2018)软件选择Kimura-2-Parameter (K2P)模型, 其他参数设置为默认值。
| 物种 | 序列数 | 序列号 |
| 南麂列岛待定种 | 23 | OK001740~OK001762 (本研究测序) |
| Octopus sinensis | 8 | AB430546~AB430548, HQ846154, KU525758, KU525759, HQ846110, NC006353 |
| Octopus vulgaris | 4 | KU525762~KU525765 |
| Octopus americanus | 13 | MT022413~MT022423, MT022425, MT022426 |
| Octopus insularis | 4 | KP056552~KP056555 |
| Octopus tetricus | 8 | KJ605247, KJ605249~KJ605253, KJ605255, KJ605256 |
| Octopus cf. tetricus | 15 | KJ605262~KJ605269, KJ605271~KJ605276, KJ605278 |
| Vampyroteuthis infernalis | 1 | AB385880 |
南麂列岛样品为中等至大型章鱼, 成熟个体胴背长117.5~133.9 mm, 全长593.1~998.0 mm, 最高体重达2 140.0 g; 活体皮肤呈灰色或红棕色, 皮肤表面有纹理; 胴背长大于胴体宽(MWI 60.3~75.0), 头部窄于胴体部(HWI 30.1~65.7); 眼上具乳头状突起; 漏斗中等长(FLI 30.8~58.2), 内侧漏斗长多为外侧漏斗长的70%左右(FFLI 51.5~80.6), 腕长中等(ALI 218.1~531.1), 腕式通常为2 > 3 > 4 > 1, 最长腕长约为胴背长的4倍, 最短腕长约为胴背长的3倍; 雄性右三腕为茎化腕, 成熟雄性茎化腕多为左三腕的80%(OAI 78.1~96.4), 茎化腕吸盘数范围123~151; 腕宽中等(AWI 14.6~29.5), 腕间膜中等深度(WDI 15.9~26.2), 腕间膜式通常为C > D > B > E > A; 吸盘中等大小(SDIn 6.0~13.9), 雄性在第二、第三对腕的第12~15吸盘位置具有1~2个扩大吸盘; 舌叶小, 呈锥形, 中央具有沟槽, 多为茎化腕长的1%左右(LLI 0.6~1.2), 交接基多为舌叶的50% (CaLI 34.2~65.5)。
2.2 主成分分析雌性主成分分析共构建5个主成分, 主成分的负荷值和贡献率见表 3, 主成分1的贡献率为46.565%, 主成分2的贡献率为13.152%, 主成分3贡献率为9.005%, 主成分4贡献率为6.164%, 主成分5贡献率为5.800%, 累计贡献率为80.686%。在主成分1中, 影响群体形态差异的指标为HWI、ALLI1、ALLI2、ALLI3、ALLI4、ALRI1、ALRI2、ALRI3、ALRI4、SDIn、SNL3、SNR3, 主成分2中影响群体差异的指标为ML、VML, 主成分3中影响群体差异的指标为FFLI和WDI, 主成分4中影响群体差异的指标为MWI, 主成分5中影响群体差异的指标为FLI和AWI。
| 性状 | 负荷值 | ||||
| 主成分1 | 主成分2 | 主成分3 | 主成分4 | 主成分5 | |
| ML | –0.014 | 0.909 | –0.307 | 0.101 | 0.125 |
| VML | –0.013 | 0.847 | –0.336 | 0.087 | 0.179 |
| MWI | 0.267 | –0.171 | –0.178 | 0.714 | 0.459 |
| HWI | 0.655 | –0.231 | 0.029 | –0.360 | 0.243 |
| FLI | 0.052 | –0.549 | –0.498 | –0.280 | 0.392 |
| FFLI | 0.084 | 0.013 | 0.739 | 0.396 | –0.086 |
| ALLI1 | 0.883 | –0.104 | –0.152 | 0.037 | –0.046 |
| ALLI2 | 0.906 | –0.120 | –0.110 | 0.178 | –0.006 |
| ALLI3 | 0.911 | –0.070 | –0.086 | –0.095 | –0.171 |
| ALLI4 | 0.941 | –0.047 | –0.022 | 0.035 | –0.167 |
| ALRI1 | 0.865 | –0.075 | –0.095 | 0.217 | 0.045 |
| ALRI2 | 0.853 | –0.102 | –0.205 | 0.217 | –0.111 |
| ALRI3 | 0.941 | –0.038 | –0.018 | –0.060 | –0.115 |
| ALRI4 | 0.934 | –0.019 | 0.052 | 0.029 | 0.002 |
| AWI | 0.216 | 0.170 | 0.556 | –0.163 | 0.657 |
| SDIn | 0.787 | 0.006 | 0.338 | –0.147 | 0.262 |
| SNL3 | 0.652 | 0.543 | 0.174 | –0.170 | –0.060 |
| SNR3 | 0.727 | 0.357 | 0.216 | –0.167 | –0.155 |
| WDI | –0.407 | –0.264 | 0.283 | 0.180 | –0.087 |
| 特征值 | 8.847 | 2.499 | 1.711 | 1.171 | 1.102 |
| 贡献率(%) | 46.565 | 13.152 | 9.005 | 6.164 | 5.800 |
| 累计贡献率(%) | 80.686 | ||||
雄性主成分分析共构建4个主成分, 主成分的负荷值和贡献率见表 4, 主成分1贡献率为47.060%, 主成分2贡献率为13.121%, 主成分3贡献率为9.461%, 主成分4贡献率为6.151%, 累计贡献率为75.794%。在主成分1中, 影响群体形态差异的指标为HWI、FLI、ALLI1、ALLI2、ALLI3、ALLI4、ALRI1、ALRI2、ALRI3、ALRI4、AWI、SDIn、SDIe、SNL3、SNR3, 主成分2中影响群体差异的指标为ML、VML、LLI, 主成分3中影响群体差异的指标为MWI、FFLI、WDI, 主成分4中影响群体差异指标为CaLI。
| 性状 | 负荷值 | |||
| 主成分1 | 主成分2 | 主成分3 | 主成分4 | |
| ML | –0.138 | 0.786 | –0.449 | 0.122 |
| VML | 0.074 | 0.765 | –0.465 | 0.073 |
| MWI | 0.095 | 0.543 | 0.626 | –0.069 |
| HWI | 0.682 | 0.110 | 0.203 | –0.300 |
| FLI | 0.505 | –0.547 | –0.387 | –0.388 |
| FFLI | –0.186 | 0.480 | 0.503 | –0.348 |
| WDI | –0.442 | 0.282 | 0.340 | –0.260 |
| ALL1 | 0.881 | –0.047 | –0.112 | –0.095 |
| ALLI2 | 0.931 | 0.036 | 0.035 | –0.128 |
| ALLI3 | 0.920 | 0.068 | –0.112 | –0.130 |
| ALLI4 | 0.959 | 0.019 | 0.023 | –0.064 |
| ALRI1 | 0.909 | –0.118 | –0.097 | –0.027 |
| ALRI2 | 0.900 | –0.040 | –0.131 | –0.130 |
| ALRI3 | 0.882 | 0.167 | 0.180 | –0.159 |
| ALRI4 | 0.910 | 0.113 | –0.078 | 0.004 |
| AWI | 0.807 | –0.217 | 0.178 | 0.185 |
| SDIn | 0.559 | –0.328 | –0.102 | 0.206 |
| SDIe | 0.777 | 0.289 | –0.065 | 0.072 |
| SNL3 | 0.767 | 0.371 | 0.079 | 0.289 |
| SNR3 | 0.713 | 0.285 | 0.311 | 0.403 |
| LLI | –0.171 | 0.346 | –0.563 | 0.211 |
| CaLI | 0.164 | –0.353 | 0.392 | 0.661 |
| 特征值 | 10.353 | 2.887 | 2.081 | 1.353 |
| 贡献率(%) | 47.060 | 13.121 | 9.461 | 6.151 |
| 累计贡献率(%) | 75.794 | |||
雌、雄各群体主成分1和主成分2的散点图如图 1所示。在雌性各群体中, 西班牙真蛸(SP)均分布在Y轴右侧, 可以跟其他群体分开, 而南麂列岛(NJLD)与中华蛸各群体(ND、YL和JZ)存在重叠; 在雄性各群体中, 西班牙真蛸(SP)分布于Y轴右侧, 其他四个群体(NJLD、ND、YL和JZ)集中分布在Y轴左侧并存在部分重叠。
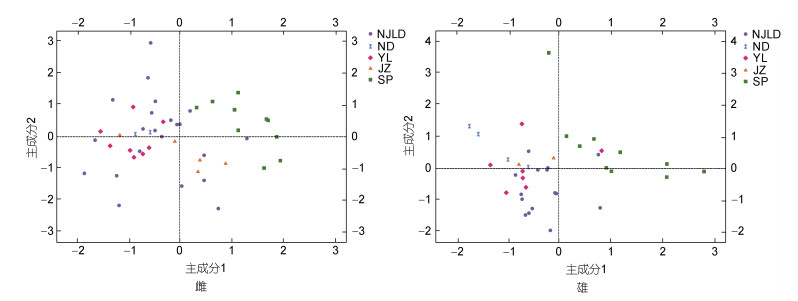 |
| 图 1 主成分分析散点图 Fig. 1 The scatter plot of principal component analysis 注: 图例中字母表示各群体。NJLD: 南麂列岛, ND: 宁德, YL: 宜兰, JZ: 九州, SP: 西班牙 |
利用逐步判别分析法分别对雌、雄群体进行费歇尔判别分析。
雌性各群体判别公式如下:

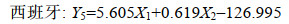
判别式中, X1为SDIn、X2为SNR3。
雄性各群体判别公式如下:
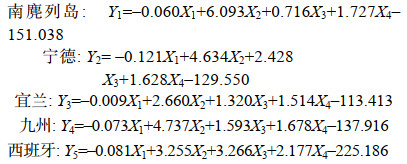
判别式中, X1为ALRI1、X2为SDIn、X3为SDIe、X4为SNR3。
判别分析散点图见图 2, 西班牙真蛸(SP)可与其余四个群体明显区分开, 而南麂列岛(NJLD)与中华蛸各群体(ND、YL和JZ)存在重叠。为检验判别结果, 对所有雌、雄群体进行预测分类(见表 5和表 6), 雌、雄综合判别分析结果分别为66.0%与84.2%, 其中, 雌、雄真蛸(SP)均被100%准确判别。
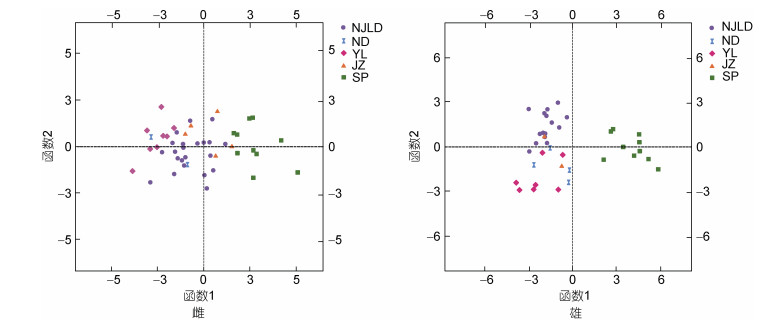 |
| 图 2 判别分析散点图 Fig. 2 Scatter plots of discriminant analysis 注: 图例中字母表示各群体。NJLD: 南麂列岛, ND: 宁德, YL: 宜兰, JZ: 九州, SP: 西班牙 |
| 群体 | 样本数 | 预测分类 | 判别准确率/% | |||||
| 南麂列岛 | 宁德 | 宜兰 | 九州 | 西班牙 | P1 | P2 | ||
| 南麂列岛 | 22 | 10 | 7 | 0 | 5 | 0 | 45.5 | 83.3 |
| 宁德 | 2 | 1 | 0 | 1 | 0 | 0 | 0.0 | 0.0 |
| 宜兰 | 8 | 0 | 0 | 8 | 0 | 0 | 100.0 | 88.9 |
| 九州 | 5 | 1 | 0 | 0 | 3 | 1 | 60.0 | 37.5 |
| 西班牙 | 10 | 0 | 0 | 0 | 0 | 10 | 100.0 | 100.0 |
| 综合判别率/% | 66.0 | |||||||
| 群体 | 样本数 | 预测分类 | 判别准确率/% | |||||
| 南麂列岛 | 宁德 | 宜兰 | 九州 | 西班牙 | P1 | P2 | ||
| 南麂列岛 | 15 | 12 | 0 | 0 | 3 | 0 | 80.0 | 92.3 |
| 宁德 | 4 | 0 | 3 | 0 | 1 | 0 | 75.0 | 100.0 |
| 宜兰 | 7 | 0 | 0 | 6 | 1 | 0 | 85.7 | 100.0 |
| 九州 | 2 | 1 | 0 | 0 | 1 | 0 | 50.0 | 50.0 |
| 西班牙 | 10 | 0 | 0 | 0 | 0 | 10 | 100.0 | 100.0 |
| 综合判别率/% | 84.2 | |||||||
对雌、雄5个群体的所有样本校正值进行聚类, 图 3结果显示5个群体聚为2大类, 西班牙真蛸单独成为一支, 其余四个群体聚成一支。在雌性聚类分析中, 南麂列岛(NJLD)、宁德(ND)、宜兰群体(YL)先聚成一支, 再与九州群体(JZ)群体聚在一起。在雄性聚类分析中, NJLD、YL与JZ群体先后聚在一起, 再与ND群体聚成一支。结果表明, NJLD与中华蛸亲缘关系更近, 与真蛸存在明显差异。
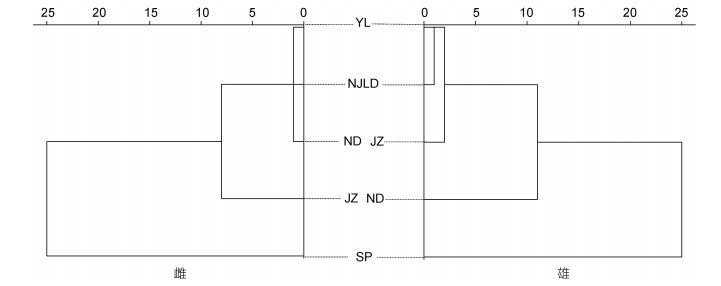 |
| 图 3 聚类分析图 Fig. 3 Diagram of cluster analysis 注: 图例中字母表示各群体地理位置。NJLD: 南麂列岛, ND: 宁德, YL: 宜兰, JZ: 九州, SP: 西班牙 |
南麂列岛群体的单倍型数(Nhap) 4个, 单倍型多样性(Hd)为0.320±0.121, 多态位点(S)为6个, 平均核苷酸差异数(K)为0.672, 核苷酸多样性指数(π)为0.001 11。南麂列岛群体与中华蛸的K2P遗传距离为0.14%, 而与真蛸复合种其他类型的遗传距离为2.96%~12.11% (表 7), 这一结果支持南麂列岛采集样品为中华蛸。
| 物种 | 南麂列岛待定种 | O. sinensis | O. vulgaris | O. americanus | O. insularis | O. tetricus |
| 南麂列岛待定种 | — | |||||
| O. sinensis | 0.001 4 | — | ||||
| O. vulgaris | 0.029 6 | 0.030 0 | — | |||
| O. americanus | 0.031 9 | 0.032 3 | 0.025 4 | — | ||
| O. insularis | 0.121 1 | 0.121 9 | 0.121 4 | 0.118 3 | — | |
| O. tetricus | 0.030 3 | 0.030 9 | 0.037 2 | 0.032 6 | 0.121 2 | — |
| O. cf. tetricus | 0.029 7 | 0.029 5 | 0.036 3 | 0.036 2 | 0.124 1 | 0.029 5 |
系统发育树(图 4)显示南麂列岛中华蛸与其他中华蛸序列聚成一支(序列OK001749除外), 这一支包括日本与中国沿海样本, 但未见明显遗传分化。其余真蛸物种复合体各类型均单独聚成一支, O. tetricus、O. cf. tetricus与O. vulgaris、O. americanus各聚为一支, 互为姊妹群, 然后与中华蛸聚在一起, O. insularis亲缘关系较远, 是其他五个真蛸复合种的姊妹群。
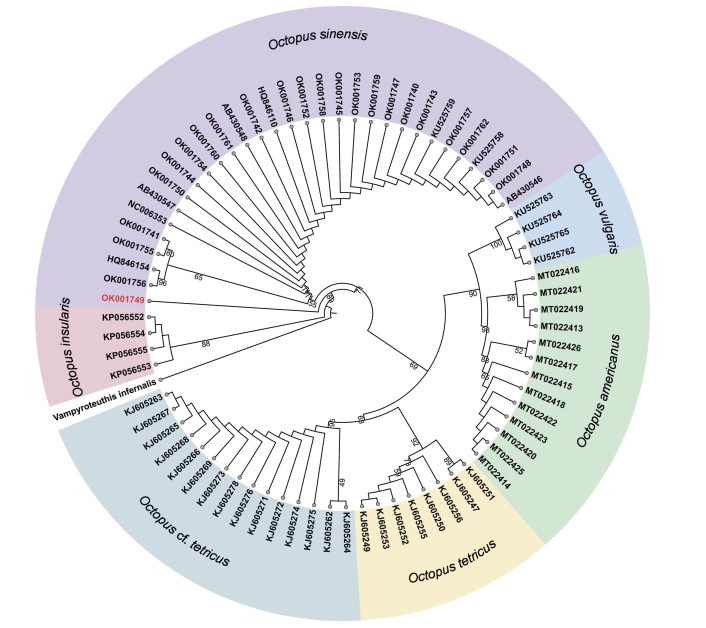 |
| 图 4 基于COI基因构建的ML系统发育树 Fig. 4 Maximum likelihood phylogenetic tree constructed using COI gene 注: 基于TIM2+F+G4进化模型, 节点数字为自举值 |
物种形态多元分析是划分不同群体、判断亲缘关系的有效方法, 物种形态学指标越多, 包含的鉴别特征信息就越多。头足类形态结构复杂, 可测量的形态学指标多达几十个。高晓蕾等(2019)利用形态多元分析方法表明中国沿海11个长蛸群体在形态上存在一定的地域差异, 陈唯(2018)研究了我国沿海短蛸6个群体的形态学特征, 结果显示群体间存在明显地理分化。在本研究中, 主成分分析、判别分析和聚类分析均将真蛸(SP)群体分离开来, 南麂列岛(NJLD)与其他三个中华蛸群体(ND、YL和JZ)存在重叠, 但雌性九州(JZ)与雄性宁德群体(ND)仍与其他中华蛸群体分离(图 1~图 3)。九州(JZ)中华蛸群体与中国沿海群体相距较远, 不同的生态与水文条件是导致其与中国沿海群体存在形态学差异的主要因素, 而宁德近海多港湾, 湾内风浪较小, 海水流速适中, 适宜海洋生物的生长, 这可能是造成该地区雄性中华蛸与其他群体存在形态差异的原因。
在头足类的物种鉴定中, 性别特征是重要的分类依据(Bello, 1995; Brakoniecki, 1996; Von Byern et al, 2010), 相较于其他可测量性状, 与性别相关的形态特征具有更加多样化的特点(Pomiankowski et al, 1995; Norman et al, 1997a; O’Dor et al, 1998), 雄性往往具有茎化腕、舌叶与扩大吸盘等特征, 因此, 基于形态学的物种多样性分析, 雄性的形态学差异分析结果可能更加可靠。本研究判别分析中, 雄性的综合判别分析准确率高于雌性(表 5和表 6), 雄性判别式共有4个参数, 分别为ALRI1、SDIn、SDIe与SNR3, 其中SDIe与SNR3为与雄性性别密切相关的形态指标。
Gleadall(2016)比较了中华蛸与真蛸的形态学异同, 重描述了日本九州与本州沿海采集的所谓“真蛸”, 恢复了中华蛸Octopus sinensis d’Orbigny, 1841种名的有效性。本文总结了南麂列岛章鱼的形态特征, 并将其与日本中华蛸标本(Gleadall, 2016)、真蛸(Norman et al, 2014; Gleadall, 2016)进行了比较(表 8), 指出中华蛸主要鉴别特征有三点: (1) 成熟雄性在第二对腕和第三对腕的第12~15个吸盘之间存在1~2个扩大吸盘(通常在第13和14个吸盘); (2) 茎化腕长约为左三腕的80%, 吸盘数量范围119~152个; (3) 末端舌叶较真蛸小。Toll(1988)认为, 茎化腕吸盘数是鉴定蛸类的重要指标, 雄性茎化腕吸盘数量在同一物种中相对稳定, 不同种之间多存在差异, 南麂列岛样品的茎化腕吸盘数量与真蛸明显不同, 其数量更少。根据以上形态学数据分析, 南麂列岛样品与Gleadall(2016)重描述的中华蛸为同一物种, 二者与真蛸存在明显差异。因此, 从形态上可以确定南麂列岛章鱼为中华蛸。任静等(2021)在浙江南麂列岛与福建连江样品的肠道与盲囊中发现了一种新的寄生虫——多刺丛集球虫Aggregata octaculeata, 该寄生虫与真蛸中发现的寄生虫在形态与分子上均存在很大差异, 由于蛸类丛集球虫具有宿主特异性, 因此, 从寄生虫角度也可佐证南麂列岛章鱼与真蛸为不同种。
| 特征指标 | 南麂列岛待定种 | 中华蛸标本(Gleadall, 2016) | 真蛸(Norman et al, 2014; Gleadall, 2016) |
| 第二、三对腕扩大吸盘位置 | 12th~14th | 12th~15th | 15th~19th |
| 茎化腕吸盘数(SNR3) | 123~151 | 119~152 | 154~192 |
| 左三腕吸盘数量(SNL3) | 151~263 | 207~273 | 320~350 |
| 舌叶指数(LLI) | 约1.0 | 1.2~1.8 | 3.1~3.5 |
利用线粒体COI序列发现南麂列岛章鱼的Nhap为4个, Hd为0.320±0.121, π为0.001 11显示其遗传多样性水平偏低。对于此结果, 需通过16S rRNA基因、微卫星等分子标记进一步验证, 同时建议当地有关部门对南麂列岛物种采取一定程度的保护措施, 如每年的5~7月为中华蛸繁殖期, 可设置禁捕或限捕期。
系统发育树显示序列OK001749未与其他中华蛸聚成一支(图 4), Avendaño等(2020)用COI序列构建了真蛸物种复合体系统发育树, 发现墨西哥采集的7号O. americanus样本与真蛸聚成一支, 而用16S rRNA构建的系统发育树显示所有O. ameiricanus聚成一支, 两种分子标记存在差异, 这可能也是序列OK001749单独分离的原因, 因此, 今后可用多基因联合构建更可靠的系统发育树。
南麂列岛章鱼和其他中华蛸的K2P遗传距离仅为0.14%, 与真蛸复合体其他类型的遗传距离均大于2% (表 7), 支持当地采集样品为中华蛸而非真蛸。此外, 中华蛸并未出现遗传分化(图 4), 说明不同地理群体间存在一定的基因交流, 可能与其浮游幼虫阶段随黑潮等洋流远距离扩散有关。
4 结论本研究表明南麂列岛采集的章鱼与真蛸在形态特征和遗传上均存在显著差异, 而与中华蛸鉴别特征一致, 支持该海域采集样品的正确种名为中华蛸, 为南麂列岛世界生物圈保护区种质资源保护提供了精准物种信息。
致谢 澳大利亚维多利亚博物馆卢重成教授对文章撰写提供了宝贵意见, 中国海洋大学贝类遗传育种研究室硕士研究生胡元海协助实验, 谨致谢忱。
叶鹏, 蔡厚才, 庄定根, 等, 2006. 南麂海区野生贝类增养殖种类初步筛选. 渔业现代化, (4): 26-28 DOI:10.3969/j.issn.1007-9580.2006.04.013 |
冯雪, 陈丕茂, 秦传新, 等, 2013. 不同温度和体质量对南海野生真蛸呼吸和排泄的影响. 中国水产科学, 20(5): 968-974 |
任静, 何卫彤, 王丽华, 等, 2021. 中华蛸寄生丛集球虫(顶复亚门: 丛集球虫科)新种的形态学与分子生物学研究. 海洋与湖沼, 52(5): 1323-1331 |
刘兆胜, 刘永胜, 郑小东, 等, 2011. 不同饵料对真蛸亲体产卵量、受精卵孵化率及初孵幼体大小的影响. 海洋科学, 35(10): 81-85 |
孙田田, 苏永全, 洪婧妮, 等, 2012. 真蛸热休克蛋白90基因(HSP90)的克隆及表达. 水产学报, 36(9): 1367-1375 |
肖懿哲, 姚成杰, 朱友芳, 等, 2019. 真蛸FAXDC2基因的克隆及其表达分析. 海洋科学, 43(8): 56-63 |
陈唯, 2018. 中国沿海短蛸(Octopus ocellatus)谱系地理格局与适应性分化研究[D]. 舟山: 浙江海洋大学: 14-19.
|
郑小东, 刘兆胜, 赵娜, 等, 2011. 真蛸(Octopus vulgaris)胚胎发育及浮游期幼体生长研究. 海洋与湖沼, 42(2): 317-323 |
高晓蕾, 许然, 张志新, 等, 2019. 中国沿海长蛸群体形态性状的差异. 水产学报, 43(7): 1593-1602 |
董正之, 1988. 中国动物志. 北京: 科学出版社, 182-184
|
蔡厚才, 庄定根, 叶鹏, 等, 2007. 浙江南麂岛真蛸网箱和水泥池养殖试验. 南方水产, 3(2): 66-70 DOI:10.3969/j.issn.2095-0780.2007.02.012 |
蔡厚才, 庄定根, 叶鹏, 等, 2009. 真蛸亲体培育、产卵及孵化试验. 海洋渔业, 31(1): 58-65 DOI:10.3969/j.issn.1004-2490.2009.01.009 |
AMOR M D, LAPTIKHOVSKY V, NORMAN M D, et al, 2017a. Genetic evidence extends the known distribution of Octopus insularis to the mid-Atlantic islands Ascension and St Helena. Journal of the Marine Biological Association of the United Kingdom, 97(4): 753-758 DOI:10.1017/S0025315415000958 |
AMOR M D, NORMAN M D, ROURA A, et al, 2017b. Morphological assessment of the Octopus vulgaris species complex evaluated in light of molecular-based phylogenetic inferences. Zoologica Scripta, 46(3): 275-288 DOI:10.1111/zsc.12207 |
AVENDAÑO O, ROURA Á, CEDILLO-ROBLES C E, et al, 2020. Octopus americanus: a cryptic species of the O. vulgaris species complex redescribed from the Caribbean. Aquatic Ecology, 54(4): 909-925 DOI:10.1007/s10452-020-09778-6 |
BELLO G, 1995. A key for the identification of the Mediterranean sepiolids (Mollusca: Cephalopoda). Bulletin de l'Institut Océanographique, Monaco, n° spécial, 16: 41-55 |
BRAKONIECKI T F, 1996. A revision of the genus Pickfordiateuthis Voss, 1953 (Cephalopoda; Myopsida). Bulletin of Marine Science, 58(1): 9-28 |
BRZESKI V J, DOYLE R W, 1988. A morphometric criterion for sex discrimination in tilapia [C] // The Second International Symposium on Tilapia in Aquaculture. ICLARM Conference Proceedings 15. Department of Fisheries, Bangkok, Thailand, and International Center of Living Aquatic Resources Management, Manila, Philippines: 439-444.
|
D'ORBIGNY A, 1835-1848. Histoire naturelle générale et particulière des céphalopodes acétabulifères [M] // DE FÉRUSSAC A E, D'ORBIGNY A. Histoire Naturelle Générale et Particulière des Céphalopodes Acétabulifères Vivants et Fossiles, Paris, Lacour.
|
FOLMER O, BLACK M, HOEH W, et al, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology, 3(5): 294-299 |
GLEADALL I G, 2016. Octopus sinensis d'Orbigny, 1841 (Cephalopoda: Octopodidae): valid species name for the commercially valuable East Asian common octopus. Species Diversity, 21(1): 31-42 DOI:10.12782/sd.21.1.031 |
KALYAANAMOORTHY S, MINH B Q, WONG T K F, et al, 2017. ModelFinder: fast model selection for accurate phylogenetic estimates. Nature Methods, 14(6): 587-589 DOI:10.1038/nmeth.4285 |
KUMAR S, STECHER G, LI M, et al, 2018. MEGA X: molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 35(6): 1547-1549 DOI:10.1093/molbev/msy096 |
LI F H, BIAN L, GE J L, et al, 2020. Chromosome-level genome assembly of the East Asian common octopus (Octopus sinensis) using PacBio sequencing and Hi-C technology. Molecular Ecology Resources, 20(6): 1572-1582 DOI:10.1111/1755-0998.13216 |
LIAO J X, Lu C C, 2009. A new species of Cistopus (Cephalopoda: Octopodidae) from Taiwan and morphology of mucous pouches. Journal of Molluscan Studies, 75(3): 502-509 |
LIBRADO P, ROZAS J, 2009. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 |
NGUYEN L T, SCHMIDT H A, VON HAESELER A, et al, 2015. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution, 32(1): 268-274 DOI:10.1093/molbev/msu300 |
NORMAN M D, 2000. Cephalopods: A World Guide [M]. Hackenheim, Germany: ConchBooks Press: 268.
|
NORMAN M D, FINN J K, HOCHBERG F G, 2014. Family octopodidae [M] // JEREB P, ROPER C F E, NORMAN M D, et al. Cephalopods of the World. An Annotated and Illustrated Catalogue of Cephalopod Species Known to Date. Vol 3 Octopods and Vampire Squids. FAO Species Catalogue for Fishery Purposes. Rome, Italy: FAO Press: 370.
|
NORMAN M D, HOCHBERG F G, 2005. The current state of octopus taxonomy. Phuket Marine Biological Center Research Bulletin, 66: 127-151 |
NORMAN M D, LU C C, 1997a. Redescription of the southern dumpling squid Euprymna tasmanica and a revision of the genus Euprymna (Cephalopoda: Sepiolidae). Journal of the Marine Biological Association of the United Kingdom, 77(4): 1109-1137 DOI:10.1017/S0025315400038662 |
NORMAN M D, SWEENEY M J, 1997b. The shallow-water octopuses (Cephalopoda: Octopodidae) of the Philippines. Invertebrate Taxonomy, 11(1): 89-140 DOI:10.1071/IT95026 |
O'DOR R K, LIPINSKI M R, 1998. The genus Illex (Cephalopoda; Ommastrephidae): characteristics, distribution and fisheries [M] // RODHOUSE P G, DAWE E G, O'DOR R K. Squid Recruitment Dynamics. Rome: FAO: 273.
|
POMIANKOWSKI A, MOLLER A P, 1995. A resolution of the lek paradox. Proceedings of the Royal Society B: Biological Sciences, 260(1357): 21-29 DOI:10.1098/rspb.1995.0054 |
ROPER C F E, VOSS G L, 1983. Guidelines for taxonomic descriptions of cephalopod species. Memoirs of Museum Victoria, 44: 48-63 DOI:10.24199/j.mmv.1983.44.03 |
SAKAGUCHI H, HAMANO T, NAKAZONO A, 2000. Population structure of Octopus vulgaris estimated from catch size composition in northeastern iyo-nada of the seto Inland Sea, Japan. Bulletin of the Japanese Society of Fisheries Oceanography, 64(4): 224-234 |
SAKAGUCHI H, HAMANO T, NAKAZONO A, 2005. Growth of Octopus vulgaris in the northeastern iyo-nada of the seto Inland Sea, Japan. Bulletin of the Japanese Society of Fisheries Oceanography, 66(1): 11-15 |
SASAKI M, 1929. A monograph of the dibranchiate cephalopods of the Japanese and adjacent waters. Journal of the College of Agriculture, Hokkaido Imperial University, 20(supplement): 1-357 |
TOLL R B, 1988. The use of arm sucker number in Octopodid systematics (Cephalopoda, Octopoda). American Malacological Bulletin, 6(2): 207-211 |
VAN NIEUWENHOVE A H M, RATSIMBAZAFY H A, KOCHZIUS M, 2019. Cryptic diversity and limited connectivity in octopuses: recommendations for fisheries management. PLoS One, 14(5): e0214748 DOI:10.1371/journal.pone.0214748 |
VON BYERN J, KLEPAL W, 2010. Re-evaluation of taxonomic characters of Idiosepius (Cephalopoda, Mollusca). Malacologia, 52(1): 43-65 DOI:10.4002/040.052.0104 |
WARNKE K, SÖLLER R, BLOHM D, et al, 2004. A new look at geographic and phylogenetic relationships within the species group surrounding Octopus vulgaris (Mollusca, Cephalopoda): indications of very wide distribution from mitochondrial DNA sequences. Journal of Zoological Systematics and Evolutionary Research, 42(4): 306-312 DOI:10.1111/j.1439-0469.2004.00277.x |
 2022, Vol. 53
2022, Vol. 53


