中国海洋湖沼学会主办。
文章信息
- 李彬, 王卫军, 王扬帆, 孙国华, 徐晓辉, 冯艳微, 李赞, 崔翠菊, 杨建敏. 2022.
- LI Bin, WANG Wei-Jun, WANG Yang-Fan, SUN Guo-Hua, XU Xiao-Hui, FENG Yan-Wei, LI Zan, CUI Cui-Ju, YANG Jian-Min. 2022.
- 棘皮动物育种数据管理与分析平台的开发及应用
- DEVELOPMENT AND APPLICATION OF DATA MANAGEMENT AND ANALYSIS PLATFORM FOR ECHINODERM BREEDING
- 海洋与湖沼, 53(5): 1225-1233
- Oceanologia et Limnologia Sinica, 53(5): 1225-1233.
- http://dx.doi.org/10.11693/hyhz20211200323
文章历史
-
收稿日期:2021-12-11
收修改稿日期:2021-12-30
2. 烟台海育海洋科技有限公司 山东烟台 264004;
3. 中国海洋大学 山东青岛 266003;
4. 山东华春渔业有限公司 山东东营 257200
2. Yantai Haiyu Marine Science and Technology Co., Ltd., Yantai 264004, China;
3. Ocean University of China, Qingdao 266003, China;
4. Shandong Huachun Fisheries Co., Ltd., Dongying 257200, China
棘皮动物(Echinoderm)作为一类重要的海洋经济动物, 在水产养殖中发挥着重要的作用。21世纪以来, 随着刺参、海胆等高价值海产品的不断开发利用, 以刺参为代表的高价值水产动物在我国的养殖规模不断扩大, 市场价值越来越高, 推动了我国第五次海水养殖浪潮(李成林等, 2017)。在国家重点研发计划“蓝色粮仓科技创新专项”、山东省农业良种工程、山东省现代农业产业技术体系等项目中均设置了刺参育种的研发内容。经过多年的科研努力, 棘皮动物遗传育种快速发展, 科研人员培育出了一系列棘皮动物新品种, 包括: 刺参新品种“水院1号”(大连海洋大学校科研处, 2010)、“崆峒岛1号”(杨建敏等, 2015)、“东科1号”(杨红生等, 2018)、“安源1号”(宋坚等, 2018)、“参优1号”(王印庚等, 2018)、“鲁海1号”(李成林, 2019), 以及中间球海胆新品种“大金”(常亚青等, 2015)。
基于最佳线性无偏估计(best linear unbiased prediction, BLUP)的遗传育种选择方法具有遗传评估准确度高的特点, 对于系谱复杂、数据量大的群体, 有更高的选择效率。我国从1972年就开始用BLUP法对中国荷斯坦牛的育种值进行了估算(黄冬维等, 2008)。目前BLUP技术被广泛应用于猪(李晓强等, 2001)、绒山羊(孟瑞强等, 2012, 2014)、山鸡(陆雪林等, 2015)、牛(刘丽元等, 2015)等禽畜物种的选择育种研究。动物的遗传育种是一个复杂且连续的系统工程——从基础群体的构建、养殖数据测量, 到遗传参数的评估, 再到亲本的选择, 然后制定选种配种方案等。近年来国内育种管理系统在各个物种的遗传育种中得到了广泛应用。徐东升等(2009)开发了一套陕北白绒山羊育种的辅助管理系统, 可以对选择性状的育种数据进行导入和导出。杨瑞飞等(2014)开发了对獭兔育种和生产进行系统高效管理的软件。陆雪林等(2015)根据山鸡生长和繁育特性, 设计开发了山鸡育种分析管理软件, 实现了育种信息的可追溯性, 实现了山鸡生产性状的数据育种值的高效计算。在水产动物的育种管理系统方面, 栾生等(2008)开发了一套在Windows环境下的水产动物育种分析与管理的系统, 并在鱼、虾等育种中得到应用; 李艳(2010)开发了贝类遗传育种分析体系, 用于贝类的数据库管理及遗传评估; 陆维(2011)对贝类遗传育种分析体系, 在育种工作中进行了应用; 苏海林等(2016)开发了贝类全基因组遗传育种评估与分析系统。
虽然国内已有针对贝类、虾蟹类、鱼类育种的水产动物育种分析与管理系统, 但水产经济动物的生物学习性差别很大, 针对棘皮动物育种数据管理与分析研究的平台系统尚未有报道。目前棘皮动物育种过程中, 大多数表型与基因组数据未进行系统化管理, 导致实际育种中育种数据管理系统和选种选配的计算脱节, 不能快速高效地查询调取育种数据信息, 制约了育种效率的进一步提高。因此, 结合计算机语言, 开发一套适合棘皮动物的育种管理与分析系统是一种行之有效的方法。本平台针对棘皮动物特有的生物学特点, 结合ASReml数据评估软件(Gilmour et al, 1995), 主要以BLUP遗传评估为核心, 建立了育种数据管理系统及遗传评估为一体的棘皮动物育种管理综合平台, 实现了棘皮动物育种流程的规范化和管理的智能化。该平台的建立将大大提高棘皮动物育种效率, 加快育种进程。
1 平台开发环境与工具本平台是在Linux环境下使用Mysql数据库(版本号: 5.1.30)和java、R语言, 利用maven、myeclipse 2017工具开发的一套基于B/S框架的育种数据管理与分析平台; 平台调用的是基于R语言的ASReml包, ASReml版本是4.1.0.151, 导入ASReml包后使用ASReml软件的计算功能。
2 平台功能平台采用模块化设计, 主要有系统管理、种质信息、表型数据库、表型数据分析、基因组数据库和基因组数据分析6个功能模块(图 1)。
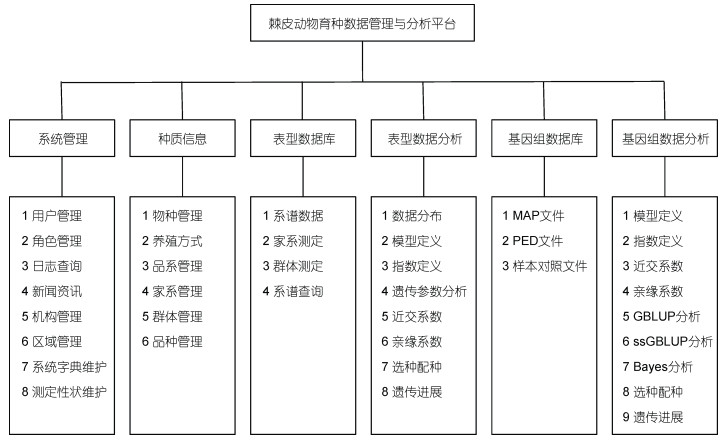 |
| 图 1 棘皮动物育种数据管理与分析平台各功能模块 Fig. 1 Modules of data management and analysis platform on Echinoderm breeding |
本模块主要功能是管理员对平台系统以及普通用户信息进行设置与维护; 主要包括: 用户管理、角色管理、日志查询、新闻资讯、机构管理、区域管理、系统字典维护、测定性状维护。以系统字典和测定性状维护为例, 进行功能描述。系统字典维护用来定义变量, 如: 棘皮动物的在场状态、测定阶段(幼体、成体)、存活状态(存活、死亡)等。测定性状维护是对所需测量的性状进行自定义维护, 按照性状类别分为三种: 连续性状、分类性状和阈性状。连续性状包括: 棘皮动物的体长、体重、棘刺总数、黏多糖含量、刺参皂苷含量等; 分类性状包括: 性腺颜色、体色、棘刺列数等; 阈性状主要是存活率。通过对其性状名称、英文名称、数据类型等信息的新建、修改和删除等, 形成相应数据报表用于数据库的构建。
2.2 种质信息该模块用于维护设定基础数据管理, 服务于其他模块。主要是维护棘皮动物的类别、养殖、管理等基本信息, 是系统登记和后期查询数据的基础。该模块包括: 物种管理、养殖方式、品系管理、家系管理、群体管理、品种管理等信息。以物种管理和测定家系管理为例, 进行功能描述。物种管理部分, 可以对棘皮动物物种进行新建、修改、删除、导出等操作, 输入物种名称(无字符限制, 如“仿刺参”)、物种编码(有字符限制, 为两位大写字母, 如“AJ”)、物种排序(数值型, 如“1”)、备注(如“棘皮类)”。家系管理包含: 所属品系名称、家系编码、家系名称、是否为核心群等信息。
2.3 表型数据库本模块主要功能是数据的录入、导入与查询, 将录入或导入的数据形成一个数据库。表型数据库包括: 系谱数据、家系选育数据、群体选育数据的上传及查询。其中, 系谱数据库主要包括: 个体编码、父本编码、母本编码、世代数及子代个体在世代中的位置; 系谱数据整理需先在系统中下载系谱档案模板, 将系谱数据整理到模板中后再上传至平台。家系选育数据、群体选育数据的表型数据按照性状类型分为连续性状、分类性状和阈性状三种, 在平台中进行模板下载, 将数据复制于模板中, 导入平台。个体ID是联系系谱数据和表型数据的纽带, 个体ID在数据库中至关重要, 其组成为15位数字和字母组成的代码, 其编码规则为: 物种代码两位大写字母, 如: AJ (刺参)+品种代码(两位数字, 如: 00)+个体出生场编号(四位字母与一位数字组成, 如: HYKJ1 (海育科技一厂)+选育代数(两位数字, 如: 01)+个体出生的年份后两位(两位, 如: 13)+家系(三位数字, 如: 004)+个体编号(两位数字, 如: 01)。系谱查询功能按个体ID编码进行查询, 能够分别向上追溯两代, 向下查询一代(图 2)。
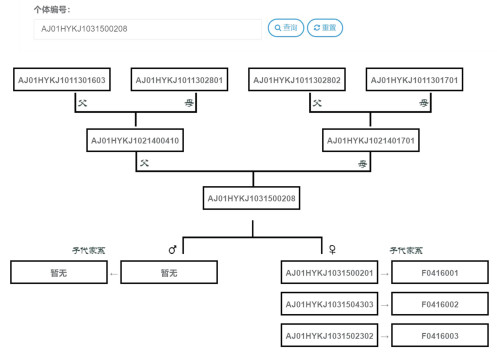 |
| 图 2 系谱查询 Fig. 2 Graph of pedigree inquire |
本模块主要功能是: 将采集的数据通过BLUP育种方法进行遗传参数分析和育种值估算。
2.4.1 测定数据分布通过设置测定时间、数据类别、性状类型进行特定数据的查看和统计。对符合条件的数据进行基本统计, 可展示该组数据的, 样本数、性状最大值与最小值、性状平均值、标准差、变异系数等; 并可对数据进行直方图展示、散点图展示、箱线图展示、Q-Q图展示, 用于直观展示性状分布情况。
2.4.2 模型定义通过设置固定效应、随机效应和因变量, 定义单性状或者多性状育种模型。根据数据分析需求, 构建相应的计算模型表达式, 举例: 点击左侧“选择变量”中的变量移动至已选择变量(如“群体来源JPqtly x”移动至固定效应项、“个体编号R_ANIMALID x”移动至随机效应项、“棘刺总数JPjczs x”移动至因变量因选), 定义单性状动物模型(图 3)。
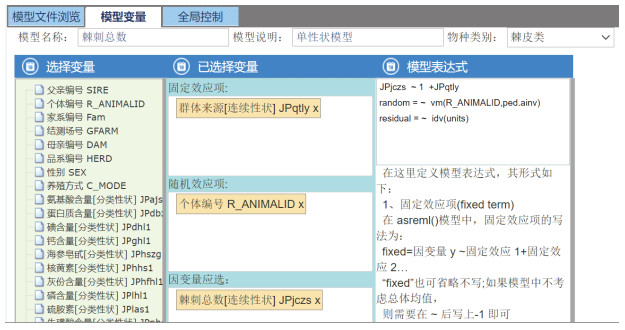 |
| 图 3 自定义遗传参数分析模型 Fig. 3 Customized model of genetic parameter analysis |
本模块调用ASReml遗传评估软件, 利用数据库中的表型数据和系谱数据, 调用定义的模型进行相关性状的育种值、遗传力、遗传相关计算以及结果查询, 为配种计划做准备。遗传参数分析分为以下4个步骤: 准备数据→数据浏览→BLUP计算→结果查询(图 4)。首先根据测定时间、测定机构、测定物种和分析模型进行数据的准备; 然后可查看满足条件的数据信息, 包含: 个体编码、父母本编码、性别和棘刺总数的数值等, 选择模型进行BLUP计算; 最后得出结果: 固定效应值、随机方差组分、育种值、遗传力和遗传相关等。
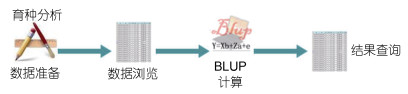 |
| 图 4 遗传参数分析步骤 Fig. 4 Steps of genetic parameter analysis |
设定测定日期和家系, 利用系谱信息, 计算有亲缘关系父母本产生的后代个体间的近交系数, 该参数用于分析从其双亲的共同祖先中得到相同基因的概率。
2.4.5 亲缘系数设定测定日期、家系、存留状态、机构名称、物种, 由于个体间共同祖先的不同, 根据其系谱信息, 计算两个个体在血缘上的亲缘系数。
2.4.6 指数定义用于多性状选择时, 将不同类型数据的育种值根据重要程度赋予权重, 然后计算出综合育种值, 最后根据综合育种值进行选种, 提高多性状育种的准确性。
设置指数育种值的所占权重时, 各性状权重的绝对值之和为1。如: 某一选择指数“DUOCI”设置为: 棘刺总数权重值为0.4, 体重权重值为0.6。其棘刺总数的估计育种值(Expected Breeding Value, EBV)为jczsEBV, 体重的估计育种值tzEBV。
 (1)
(1)式中, 
在遗传评估的基础上, 某个性状不同选育世代出生的个体的平均EBV或综合EBV的变化, 它反映了选育群体的遗传进展。
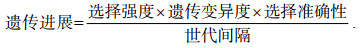 (2)
(2)根据某性状的个体育种值或综合育种值或家系育种值排名, 选定候选亲本, 并设定一定的亲缘系数值, 通过平台可制定相应的配种计划(图 5)。
 |
| 图 5 亲本的选配种方案 Fig. 5 Mating schemes of selected parents based on EBV |
不论是重测序基因分型数据还是SNP基因芯片分型数据, 格式都是其各自固定的格式, 且质量控制不统一, 导致育种分析软件无法进一步分析。本平台采用基因组数据文件上传的模式进行数据分析, 基因组数据上传前, 需对数据进行质控, 利用PLINK软件(Purcell et al, 2007), 对不同基因型文件格式转化为国际常用的MAP文件和PED文件格式。本平台可根据上传日期进行文件的查询、修改、删除等操作(图 6)。
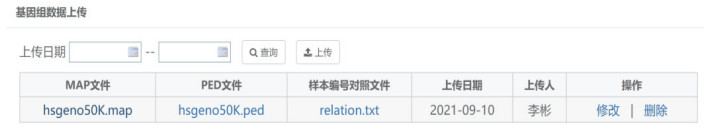 |
| 图 6 基因组数据上传 Fig. 6 Uploading of genomic data |
基因组选择育种需要首先建立参考群体, 需要既有表型数据, 又要有基因型数据, 因此需要建立一个对照文件把基因组数据和表型数据相关联。对照文件的建立: 创建txt文档, 由基因组数据PED文件第二列数据和系统个体编码组成。
2.5.3 参考群体SNP标记效应值的估计基因组选择是在群基因组范围内, 利用覆盖全基因组的高密度SNP标记进行标记辅助选择, 从而更加准确地评估个体间的遗传差异(吴常信, 2021)。本平台根据参考群体个体的表型数据, 以及使用SNP芯片或者重测序基因分型的SNP分型数据, 利用GBLUP (Genomic BLUP)、single step GBLUP (ssGBLUP)、Bayes等数学模型估计SNP标记的基因效应值。
2.6 候选群体全基因组选择分析本模块根据测定候选个体的SNP基因型, 估计计算个体的GEBV, 并依据GEBV进行选种。主要包含: 模型定义、指数定义、近交系数、亲缘系数计算等。基因组遗传参数分析的数学模型有三种方法供选择, GBLUP法(利用基因组标记信息计算亲缘关系构建G矩阵)、ssGBLUP法(利用系谱信息和基因组标记信息构建H矩阵)和Bayes法(在参考群中估计标记效应, 然后结合预测群的基因型信息将标记效应累加)。
以GBLUP分析为例, 介绍基因组选择育种: 点击“数据准备”选择测定日期、机构名称、物种、芯片文件、模型, 点击“开始准备”按钮准备数据, 提示“创建成功”后, 点击“数据浏览”, 即可显示在数据准备工作中满足条件的信息, 在选择模型后, 点击“GBLUP计算”, 提示“计算成功”后点击GBLUP分析页面的“结果查询”后显示: 基因GBLUP分析结果、分子遗传力等遗传参数。在“选种配种方案”子模块中, 按10%~20%的比例选取育种值高的个体作为亲本, 根据平台计算出的子代亲缘系数, 平台将给出最优的配种方案。
3 育种平台的应用为测试平台的表型数据分析模块, 构建了一组模拟数据。本研究模拟了215个F4代刺参的个体的表型数据, 其棘刺总数和体重是由个体均值为44.50个和19.84 g的正态分布数据的随机抽取得到的。采用个体模型, 运用单性状分析法, 估算棘刺总数性状与体重性状的遗传力和育种值。
个体模型如下:
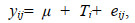 (3)
(3)式中, yij为第j个家系的第i个刺参个体的性状观测值, μ为棘刺总数与体重的均值, Ti为第i个刺参个体棘刺总数与体重的个体加性遗传效应, eij为第j个家系的第i个刺参的随机残差; 综合育种值设定为棘刺总数权重0.4与体重权重0.6。
表型数据分析过程中, 将群体来源作为固定效应, 家系内单个个体作为随机效应, 棘刺总数和体重作为因变量。表型数据分析结果显示: 棘刺总数遗传力0.853±0.142, 体重遗传力0.846±0.178; 并计算其综合育种值, 选取BLUP综合育种值排名前20的个体, 通过平台计算亲缘系数, 选取亲缘系数小于0.3的个体进行选种, 得到选配方案(图 7)。
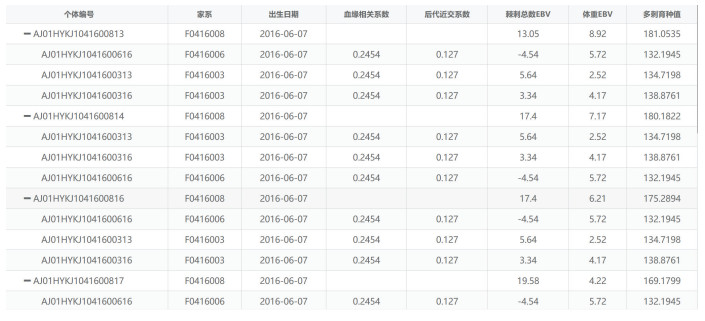 |
| 图 7 表型数据育种选配方案(部分) Fig. 7 Schemes (partial) of phenotypic data for breeding selection |
为测试平台的基因组数据分析模块, 构建了一组模拟数据。首先利用QU-GENE (Podlich et al, 1998)产生一个群体大小为200的初始群体做亲本, 50 K (50 000)个SNP位点均匀分布46个染色体上, 群体的基因频率均设定为0.5, 在50 K SNP中随机抽取100个SNP设置为QTL, 并赋值100个QTL基因效应值且符合正态分布N (100, 20) (均值是100, 标准差是20); 然后QuLine配置1 000个杂交组合, 经过4个世代的选择后, 得到215个选择个体; 根据215个体QTL基因方差, 增加环境方差使得遗传力为0.5。利用这215个刺参个体组成的群体来比较全基因组选择方法的准确性。分别采用GBLUP、ssGBLUP法、Bayes模型进行基因组数据分析。
GBLUP、ssGBLUP法模型:
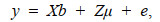 (4)
(4)式中, y为性状向量; b为固定效应; u为随机效应, 且服从均值为0, X和Z分别为b和u的关联矩阵; e为残差效应, 服从正态分布。
Bayes模型:
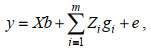 (5)
(5)式中, y为表型向量; X为固定效应系数矩阵; b为固定效应; Zi为第i个位点数字化基因型向量(如: 0, 1, 2); gi为第i个位点效应值; e为模型拟合残差, 服从正态分布。综合育种值设定为棘刺总数权重0.4与体重权重0.6。
基因组数据分析过程中, 将群体来源作为固定效应, SNP基因效应作为随机效应, 棘刺总数和体重作为因变量。分别运用GBLUP、ssGBLUP、Bayes三种数学模型算法进行基因组数据分析, 基因组数据分析结果: 棘刺总数遗传力0.518±0.448、体重遗传力0.736±0.421。计算各子代个体的综合育种值, 选取综合育种值排名前20的个体, 通过平台计算亲缘系数, 选取亲缘系数小于0.3的个体进行选种, 得到选配方案(图 8)。运行结果显示, 平台能够正确估算各种遗传参数和育种值, 根据育种值得出选种配种方案。
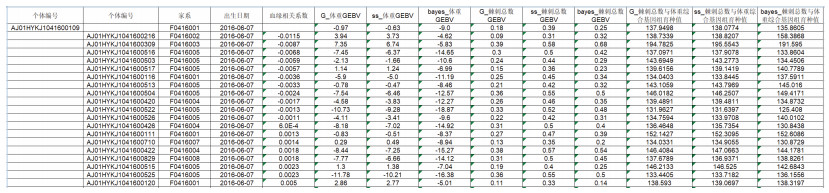 |
| 图 8 基因组数据育种选配方案(部分) Fig. 8 Schemes (partial) of genomic data for breeding selection |
种子是农业科技的“芯片”, 水产种业是渔业的战略性、基础性核心产业, 是保障水产品安全有效供给、促进现代水产业健康可持续发展的根本。在养殖业远远大于捕捞业的背景下, 水产育种在保障水产品产量和质量方面发挥着越来越重要的作用。我国水产遗传育种从传统育种、杂交育种和细胞工程育种, 逐步向传统育种与现代分子辅助育种技术、基因组选育技术相结合的方向发展(常亚青等, 2013)。不论是传统育种还是基因组选择育种, 均需要对遗传参数进行评估, 来确定适宜的选育技术路线, 并通过遗传分析软件进行育种值估算, 进行亲本的选择。
ASReml软件是拟合线性混合模型的数据分析软件, 可利用灵活的混合线性模型和广义线性模型来处理大规模的数据, 实现大数据高效、快速地分析。本平台选用ASReml软件作为内核进行遗传参数分析, 可对数量性状、阈值性状、分类性状、SNP标记效应等多个性状进行分析, 并可实现固定效应、随机效应值的预测以及显著性检验、遗传参数的估计、全基因组选择等。本平台可以计算个体近交系数、亲缘系数、遗传力、遗传相关、育种值等参数, 对于复杂模型和大数据的分析更具有优势。虽然SAS (Statistical Analysis System)数据分析软件是数据管理和统计分析的先锋(林元震等, 2017), 但是SAS模块(比如MIXED, GLIMMIX)在处理大规模遗传数据时效率低, 而且在拟合混合模型的复杂方差协方差结构不够灵活。随着数量遗传学不断地发展, 基于BLUP的选择方法在动物育种中普遍应用(Nielsen et al, 2009)。依据BLUP值选择, 使得选择是最佳的、无偏的, 从而能够有效提高选择的效率, 而且对于多个场次、系谱复杂、数据量大的育种群体, 选择效率更高。BLUP育种值是反映个体表现得好坏的指标, 育种值越高, 说明个体的表现越好。随着全基因组选择统计模型的不断优化, 模型的稳定性不断提高, 但是依然面临计算准确性和计算效率的挑战; 直接法(GBLUP和ssGBLUP)计算效率比间接法(BayesB等)高, 但是计算准确性略差; 间接法计算准确性较高, 由于计算量大, 无法做到高效指导育种实践。因此, 有学者认为如何优化模型, 与机器学习方法有效结合进行模型优化, 在保证准确性的同时, 又能提高计算效率, 是未来全基因组选择模型优化的方向(尹立林等, 2019)。
有研究表明, 为了获得持续、稳定的遗传进展, 需要严格控制繁育群体近交系数的升高、防止遗传变异的快速丢失(Sonesson et al, 2012)。邵学芬等(2021)在短角牛繁殖中应用选配分群技术, 不仅将群体近交系数降到了较低的水平, 还杜绝了极端近交现象的发生。近交系数高容易导致近交衰退, 因此在本平台开发过程中, 选择EBV或GEBV高的个体作为亲本, 同时将亲缘系数作为配种的重要指标控制近交系数, 以实现遗传育种的可持续性。本平台系统与“水产动物育种分析与管理的系统”、“贝类遗传育种分析评估系统”、“贝类全基因组遗传育种评估与分析系统”相比, 首次在棘皮动物中进行育种数据和育种分析, 而且能同时实现表型数据、系谱数据和基因组数据的管理与分析。系统采用B/S架构, 软件安装在服务器端, 网络管理人员只需要管理服务器(数据访问层), 而用户通过浏览器即可访问平台进行功能操作(表示层), 业务逻辑层用来链接表示层和数据访问层, 实现用户操作具体功能后端响应请求, 处理业务逻辑。这样形成数据访问层、业务逻辑层和表示层组成的三层结构, 大大减少客户端电脑负荷, 减轻了系统维护、升级的支出成本。只要有网络和浏览器, 可以随时随地进行数据上传、育种分析、选种选配等育种业务的操作。本系统使用GBLUP、ssGBLUP和Bayes三种算法对基因组芯片文件进行分析, 可以较为高效、准确的计算育种值、遗传力、遗传相关等。
5 结论本平台通过将育种数据管理和分析系统与计算机语言相结合, 以ASReml软件为遗传评估核心, 将育种流程科学化、简单化、快速化, 大大提高了育种效率。平台数据库的建立可以及时对数据进行平台上传, 实现数据的可追溯性, 并可对数据进行智能化管理和条件筛选。通过平台管理与数据分析结合, 最大程度实现数据的自动化分析, 而且本平台功能模块按照需求定制, 灵活性与适应性强, 有效解决数据管理与数据分析的难题。该平台的推广和应用将提升棘皮动物育种效率, 将对我国棘皮动物育种工作的发展具有大的促进作用。
大连海洋大学校科研处, 2010. 大连海洋大学《"水院1号"刺参新品种》项目被评为第八届中国·海峡项目成果交易会优秀参展项目二等奖. 中国渔业经济, 28(4): 177 |
王印庚, 廖梅杰, 李彬, 等, 2018. 刺参"参优1号". 海洋与渔业, (9): 70-71 |
尹立林, 马云龙, 项韬, 等, 2019. 全基因组选择模型研究进展及展望. 畜牧兽医学报, 50(2): 233-242 |
刘丽元, 周靖航, 叶东东, 等, 2015. 运用动物模型BLUP法估计荷斯坦牛育种值及制定总性能指数. 新疆农业科学, 52(4): 747-753 |
李成林, 2019. 刺参"鲁海1号"长速快成活率高. 海洋与渔业, (10): 56-57 DOI:10.3969/j.issn.1672-4046.2019.10.053 |
杨红生, 刘石林, 张思聪, 等, 2018. 刺参"东科1号". 海洋与渔业, (9): 68-69 |
李成林, 胡炜, 2017. 我国刺参产业发展状况、趋势与对策建议. 中国海洋经济, (1): 3-20 |
宋坚, 王增东, 2018. 刺参"安源1号". 海洋与渔业, (9): 66-67 |
邵学芬, 董正勖, 向必勇, 等, 2021. 短角牛选配分群技术及其应用效果. 云南畜牧兽医, (5): 19-22 DOI:10.3969/j.issn.1005-1341.2021.05.006 |
杨建敏, 宋志乐, 王卫军, 等, 2015. 刺参"崆峒岛1号". 中国水产, (12): 55-57 DOI:10.3969/j.issn.1002-6681.2015.12.028 |
李艳, 2010. 贝类遗传评估体系的建立研究[D]. 青岛: 中国海洋大学: 9-11.
|
苏海林, 王扬帆, 胡晓丽, 等, 2016. 贝类全基因组遗传育种评估与分析系统的开发. 中国海洋大学学报, 46(10): 65-72 |
李晓强, 张元, 高腾云, 2001. 种猪遗传育种分析系统的设计. 河南农业大学学报, 35(1): 18-21 DOI:10.3969/j.issn.1000-2340.2001.01.006 |
陆雪林, 周洪钧, 高杨, 等, 2015. 基于BLUP计算的山鸡育种分析管理软件开发. 特产研究, 37(4): 5-10 DOI:10.3969/j.issn.1001-4721.2015.04.002 |
陆维, 2011. 贝类遗传育种分析评估系统的应用研究[D]. 青岛: 中国海洋大学: 3-4.
|
吴常信, 2021. 动物比较育种学. 北京: 中国农业大学出版社, 105
|
杨瑞飞, 张凯, 汪平, 等, 2014. 獭兔育种与生产信息管理系统的研发. 四川农业大学学报, 32(1): 82-86 DOI:10.3969/j.issn.1000-2650.2014.01.014 |
林元震, 张卫华, 郭海, 2017. R与ASReml-R统计学. 北京: 中国林业出版社, 1-2
|
孟瑞强, 张燕军, 王瑞军, 等, 2012. 绒山羊育种信息管理系统的设计与实现. 中国草食动物科学, (S1): 132-134 |
孟瑞强, 刘慧, 高关岭, 等, 2014. 绒山羊育种信息管理系统的开发与应用. 中国畜牧杂志, 50(10): 31-35 DOI:10.3969/j.issn.0258-7033.2014.10.007 |
栾生, 孔杰, 王清印, 等, 2008. 水产动物育种分析与管理系统的开发和应用. 海洋水产研究, 29(3): 92-100 |
徐东升, 屈雷, 2009. 基于Web的陕北白绒山羊辅助育种管理系统的设计与实现. 家畜生态学报, 30(4): 57-60 |
黄冬维, 张元跃, 谭伟明, 2008. BLUP法在畜禽育种中的应用. 中国畜禽种业, 4(8): 66-68 |
常亚青, 田燚, 张伟杰, 2013. 我国海洋水产生物遗传育种技术进展. 中国农业科技导报, (6): 8-15 |
常亚青, 张伟杰, 冷晓飞, 等, 2015. 中间球海胆"大金". 中国水产, (12): 58-59 |
GILMOUR A R, THOMPSON R, CULLIS B R, 1995. Average information REML: an efficient algorithm for variance parameter estimation in linear mixed models. Biometrics, 51(4): 1440-1450 |
NIELSEN H M, SONESSON A K, YAZDI H, et al, 2009. Comparison of accuracy of genome-wide and BLUP breeding value estimates in sib based aquaculture breeding schemes. Aquaculture, 289(3/4): 259-264 |
PODLICH D W, COOPER M, 1998. QU-GENE: a simulation platform for quantitative analysis of genetic models. Bioinformatics, 14(7): 632-653 |
PURCELL S, NEALE B, TODD-BROWN K, et al, 2007. PLINK: a tool set for whole-genome association and population-based linkage analyses. The American Journal of Human Genetics, 81(3): 559-575 |
SONESSON A K, WOOLLIAMS J A, MEUWISSEN T H E, et al, 2012. Genomic selection requires genomic control of inbreeding. Genetics Selection Evolution, 44(1): 27 DOI:10.1186/1297-9686-44-27 |
 2022, Vol. 53
2022, Vol. 53


