中国海洋湖沼学会主办。
文章信息
- 张樱馨, 宋书群, 李才文. 2022.
- ZHANG Ying-Xin, SONG Shu-Qun, LI Cai-Wen. 2022.
- 夜光藻配子细胞实时荧光定量PCR检测方法的建立与应用
- APPLICATION OF QRT-PCR ASSAY IN DETECTION AND QUANTIFICATION OF NOCTILUCA SCINTILLANS GAMETES
- 海洋与湖沼, 53(6): 1385-1395
- Oceanologia et Limnologia Sinica, 53(6): 1385-1395.
- http://dx.doi.org/10.11693/hyhz20220300067
文章历史
-
收稿日期:2022-03-17
收修改稿日期:2022-05-13
2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室 山东青岛 266237;
3. 中国科学院大学 北京 100049;
4. 中国科学院海洋大科学研究中心 山东青岛 266071
2. Marine Ecology and Environmental Science laboratory, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China
夜光藻(Noctiluca scintillans)是我国重要的赤潮生物, 在近海海域广泛分布(Harrison et al, 2011)。1992~2020年间, 《中国海洋灾害公报》共报道了28次面积超过100 km2的夜光藻赤潮, 单次赤潮最大面积达到4 000 km2。赤潮期间, 暴发性增殖的夜光藻细胞引起海水变红, 并堵塞鱼、虾、贝类等经济水产生物的呼吸器官, 导致其大量窒息死亡(Huang et al, 1997; Shunmugaraj et al, 2020); 赤潮消亡过程会过度消耗溶解氧, 并释放胞内积累的铵、硫化氢等有毒物质(岡市友利等, 1976; Huang et al, 1997; Smayda, 1997; Baliarsingh et al, 2016), 对生态系统造成极大的危害。
夜光藻可以通过二分裂的方式进行无性繁殖(杞桑等, 1994; 周成旭等, 1994), 也可通过配子结合形成合子的方式进行有性繁殖(Elbrächter et al, 1998; 宋书群等, 2016)。随着研究的不断深入, 发现有性繁殖可能在夜光藻种群增长中发挥重要作用(Fukuda et al, 2006)。研究自然水体中夜光藻配子细胞的丰度及其时空分布特征对了解夜光藻的有性繁殖过程, 明确其种群增长模式及控制因素具有重要意义, 将有助于夜光藻赤潮的预警、预测和防治。然而, 夜光藻配子细胞较为脆弱, 不易长期保存, 且直径仅有10~15 μm, 形态类似裸甲藻, 在光学显微镜下不易鉴别(Elbrächter et al, 1998; Fukuda et al, 2006), 因此难以通过镜检进行准确的定量分析。
近年来, 实时荧光定量PCR (quantitative real-time PCR, qRT-PCR)技术被广泛用于海洋藻类的定量检测, 目前已经在铜绿微囊藻(Microcystis aeruginosa)、米氏凯伦藻(Karenia mikimotoi)、多环马格里夫藻(Margalefidinium polykrikoides)、圆海链藻(Thalassiosira rotula)等微藻及石莼属(Ulva)、萱藻属(Scytosiphon)等大型藻的生殖细胞中实现了应用(He et al, 2007; Yuan et al, 2012; 徐恒省等, 2013; 缪栋等, 2018; Hu et al, 2022)。与传统的镜检方法相比, qRT-PCR技术具有样品易保存, 检测耗时少、特异性强、灵敏度高等优势, 非常适用于大面调查或长期监测。核糖体RNA (ribosomal RNA, rRNA)基因具有高拷贝数, Godhe等(2008)估算9种甲藻的拷贝数在9×105~100×105之间, 基于rRNA基因序列设计的qRT-PCR引物的检测下限可以低至个位数细胞(Yuan et al, 2012; 高岩等, 2013)。其中核糖体内转录间隔区(internal transcribed spacer, ITS)是常用的分子标记之一, 在rRNA基因中有更快的进化速度, 具有丰富的遗传变异信息。基于此序列设计的引物具有种特异性, 能区分同一属内的不同物种(Blomster et al, 2000; Yuan et al, 2012)。
本研究设计了一对靶区域为夜光藻18S-ITS1的物种特异性引物, 建立了qRT-PCR定量检测水体中夜光藻配子细胞的方法, 并应用于2015年胶州湾逐月调查, 研究了夜光藻配子细胞和营养细胞的月度动态, 以评估本方法检测环境样品的可靠性和效果, 为深入研究夜光藻有性繁殖及其在种群增长中的作用奠定了基础。
1 材料与方法 1.1 夜光藻的培养及配子基因组DNA的获取实验所用夜光藻通过垂直拖曳浅水II型浮游生物网(网口面积: 0.08 m2, 网长: 140 cm, 孔径: 160 μm)的方式采自胶州湾, 经分离、纯化后, 培养于经0.22 μm混合纤维膜过滤的灭菌海水中, 每两天投喂一次青岛大扁藻(Tetraselmis helgolandica var. tsingtaoensis)至饵料终浓度不低于1×104 cells/mL, 培养温度(20.0±0.5) ℃, 平均光照强度55.0 μmol/(m2·s), 光暗比为14h : 10h。
配子母细胞的识别及其发育阶段的判断参考Fukuda等(2006)。使用毛细管挑取即将释放配子的母细胞, 并在灭菌海水中冲洗三次以去除饵料藻, 在×200倍下镜检确认没有饵料藻残留后, 将其转移至2 mL EP管中孵育24 h。孵育完成后, 先取500 μL含有配子细胞的水样, 加入鲁哥氏液固定(终浓度1%), 迅速在×200倍下镜检计数; 剩余水样滤至3 μm孔径的核孔滤膜上(Millipore, USA), 以收集夜光藻配子细胞。使用DNeasy PowerSoil试剂盒(QIAGEN, Germany)提取配子细胞的基因组DNA。
1.2 引物设计与特异性验证基于GeneBank中已知夜光藻rRNA基因的18S-ITS1序列信息(GQ380592.1), 使用Primer Premier 5.0软件设计, 经比对筛选, 获得了一对扩增产物为167 bp的引物(表 1)。引物由北京六合华大基因科技有限公司合成。
| 引物名 | 引物序列 |
| NS-F1742 | 5′-GAAGTCGTAACAAGGTTTCCGTAG-3′ |
| NS-R1908 | 5′-GCGAACTTTGCTG ACCGTAA-3′ |
用于引物特异性验证的11种海洋微藻来自中国科学院海洋研究所海洋生态与环境科学重点实验室藻种库, 于指数生长期各取50 mL藻液, 滤至3 μm孔径的核孔滤膜上(Millipore, USA), 使用DNeasy PowerSoil试剂盒提取其基因组DNA。以夜光藻配子基因组DNA为阳性对照, 无菌去离子水为阴性对照, 采用Labcycler PCR仪(SensoQuest, Germany)对11种微藻的基因组DNA进行PCR扩增(表 2), 实验设置两个平行。反应体系为2×PCR Mix 10 μL (东盛生物, 中国), 正反向引物(10 µmol/L)各0.8 µL, DNA模板1 µL, 无菌去离子水补足至20 µL。扩增程序为95 ℃预变性5 min; 95 ℃变性30 s, 60 ℃退火30 s, 72 ℃延伸30 s, 共38个循环; 72 ℃终延伸10 min。产物经琼脂糖凝胶(重量体积比1.2%)电泳, 使用Azure 200凝胶成像系统(Azure Biosystems, Inc., USA), 于365 nm波长紫外线下成像。
| 中文名 | 拉丁名 | 类群 |
| 抑食金球藻 | Aureococcus anophagefferens | 褐藻 |
| 红色赤潮藻 | Akashiwo sanguinea | 甲藻 |
| 米氏凯伦藻 | K. mikimotoi | 甲藻 |
| 东海原甲藻 | Prorocentrum donghaiense | 甲藻 |
| 海洋卡盾藻 | Chattonella marina | 针胞藻 |
| 球形棕囊藻 | Phaeocystis globosa | 定鞭藻 |
| 青岛大扁藻 | T. helgolandica var. tsingtaoensis | 绿藻 |
| 中肋骨条藻 | Skeletonema costatum | 硅藻 |
| 链状裸甲藻 | Gymnodinium catenatum | 甲藻 |
| 微小原甲藻 | Prorocentrum minimum | 甲藻 |
| 塔玛亚历山大藻 | Alexandrium tamarense | 甲藻 |
采用Rotor-Gene Q实时荧光定量PCR分析仪(QIAGEN, Germany)进行两步法qRT-PCR反应, 反应体系为SYBR Premix Ex Taq II (2×) (TaKaRa, Japan) 10 µL, 正反向引物(10 µmol/L)各1 µL, DNA模板1 µL, 无菌去离子水补足至20 µL。qRT-PCR程序为95 ℃预变性30 s; 95 ℃变性5 s, 60 ℃退火30 s, 共40个循环; 反应结束后在50 ℃预熔解90 s, 自50 ℃至99 ℃以0.2 ℃/s的熔解速率进行熔解曲线分析。
1.4 标准工作曲线的建立与验证以1.1节所述方法获得10000个夜光藻配子细胞的基因组DNA并洗脱为30 µL, 使用洗脱液稀释2、10、20、100、200、1 000、2 000、10 000、100 000倍, 各取1 µL进行qRT-PCR分析, 实验设置三个平行。所得定量循环值(quantification cycle, Cq)与每反应细胞数的对数值通过线性回归拟合为细胞数标准曲线, 以线性响应的最低细胞数代表其检出限。
依据1.2节所述方法, 以夜光藻配子细胞基因组DNA为模板进行PCR扩增, 使用E.Z.N.A® Gel Extraction Kit胶回收试剂盒(OMEGA, USA)回收长度在100~250 bp间的目的条带。胶回收产物连接pMD18-T载体(TaKaRa), 转化大肠杆菌DH5α感受态细胞(TaKaRa), 涂布于每100 mL含有48 µL IPTG (50 mg/mL)、200 µL X-Gal (20 mg/mL)和100 µL氨苄青霉素(100 mg/mL)的LB平板上。挑取白色单菌落进行菌落PCR验证, 选取阳性克隆扩大培养并测序, BLAST对比确认是否为目的片段。使用E.Z.N.A® Plasmid Mini Kit I质粒提取试剂盒(OMEGA)提取含有目的片段的质粒, 质粒经十倍梯度稀释为100~109 copies/µL后, 取1 µL进行qRT-PCR分析, 实验设置三个平行。所得Cq值与每反应拷贝数的对数值通过线性回归拟合为标准曲线, 以线性响应的最低拷贝数代表其检出限。
实验用天然海水采集自胶州湾, 经20 μm筛绢过滤处理, 且qRT-PCR检验无夜光藻配子细胞存在(Cq=35.43±0.17)。将10 000、1 000、100个配子细胞分别接种至100 mL海水中, 抽滤至3 μm孔径的核孔滤膜上, 提取其基因组DNA并洗脱为30 µL。各取1 µL进行qRT-PCR分析, 实验设置三个平行。
1.5 胶州湾现场调查 1.5.1 航次信息于2015年1月至12月, 搭载中国科学院海洋研究所“创新”号在胶州湾开展月度调查, 通常在每月上旬进行, 持续2~3天。在调查海域布设12个站位(图 1), 其中A3、A5、B2、C1、C4站位于湾内近岸, C3站位于湾中心, D1、D3、D5站位于湾口, D6、D7、D8站位于湾外, 每个站位的水深示于表 3。
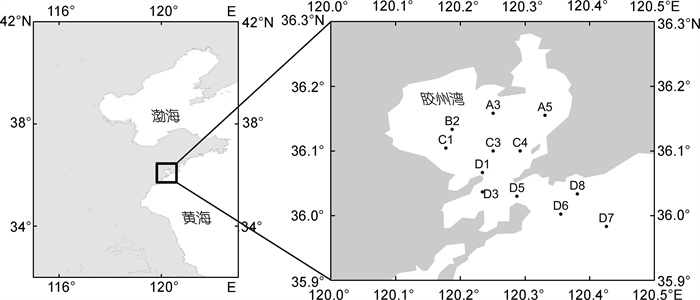 |
| 图 1 胶州湾调查站位 Fig. 1 Deployment of stations in Jiaozhou Bay |
| 站位 | 水深/m | 站位 | 水深/m | |
| A3 | 4.50±0.82 | D1 | 12.85±2.25 | |
| A5 | 7.46±0.93 | D3 | 10.56±2.63 | |
| B2 | 3.42±0.52 | D5 | 27.77±1.97 | |
| C1 | 4.41±1.07 | D6 | 26.54±1.99 | |
| C3 | 14.77±1.37 | D7 | 16.27±1.18 | |
| C4 | 6.33±0.47 | D8 | 13.15±1.87 |
夜光藻营养细胞: 通过由底至表垂直拖拽浅水II型浮游生物网采集夜光藻营养细胞, 置于广口塑料瓶中, 加入中性福尔马林溶液固定(终浓度5%)。将样品浓缩定容至50 mL, 取10 mL子样品置于体式显微镜(SZ61, Olympus, Japan)下进行鉴定和计数。夜光藻配子细胞: 使用Niskin采水器采集表层、中层和底层海水。每层取500 mL海水, 先用200 μm孔径筛绢滤去中型浮游动物和夜光藻营养细胞, 再用20 μm筛绢滤去小型浮游动物和较大的浮游植物, 最后抽滤至3 μm孔径的核孔滤膜上, 并立即液氮保存。对滤膜进行预破碎处理后, 采用DNeasy PowerSoil试剂盒提取样品中的DNA。随机挑取3份环境DNA样品进行PCR扩增, 回收100~250 bp间的条带并测序, BLAST对比确认是否为目的片段, 实验设置三个平行。依据1.3节所述方法, 对所有DNA样品进行qRT-PCR检测。
1.6 数据分析qRT-PCR扩增效率(E)与标准曲线斜率(s)的关系为:
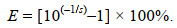 (1)
(1)夜光藻配子细胞的水柱平均丰度通过梯形法计算, 将配子丰度的垂直分布视为深度的函数, 对各水层的丰度进行积分后再除以水深得来。使用SPSS 19.0软件对配子细胞和营养细胞丰度进行t-test、Kruskal-Wallis One-Way ANOVA、Mann-Whitney U test、Spearman秩相关分析, 显著性水平P均设为0.05。标准工作曲线的拟合, 配子细胞和营养细胞丰度月度变化图及其他柱形图、箱线图均采用Origin 2019b软件绘制; 站位图采用Golden Software Surfer 17.0软件绘制。
2 结果 2.1 qRT-PCR方法引物特异性的验证使用本研究设计的引物对夜光藻配子及其他11种海洋微藻的基因组DNA进行PCR扩增。如图 2所示, 仅在夜光藻配子DNA样品中扩增到了长度在100~250 bp间的片段, 对片段进行测序并与目的序列进行BLAST对比发现, 相似性达到100%; 在其他微藻DNA样品中均未出现扩增产物。
以梯度稀释细胞的DNA为模板进行qRT-PCR分析, 熔解曲线在86.3~86.7 ℃显示单峰(图 3a)。以Cq值为纵坐标, 细胞数的对数值为横坐标, 通过线性回归拟合为细胞数标准工作曲线。如图 4a所示, 以稀释倍数2 000, 即每反应0.17个细胞为节点, 更低的稀释倍数与Cq值不再具有线性关系, 将该值视为qRT-PCR方法可检出的最低细胞数, 回归方程为y=−3.4373x+29.045 8, 回归系数R2=0.993 7, 扩增效率为95.40%。
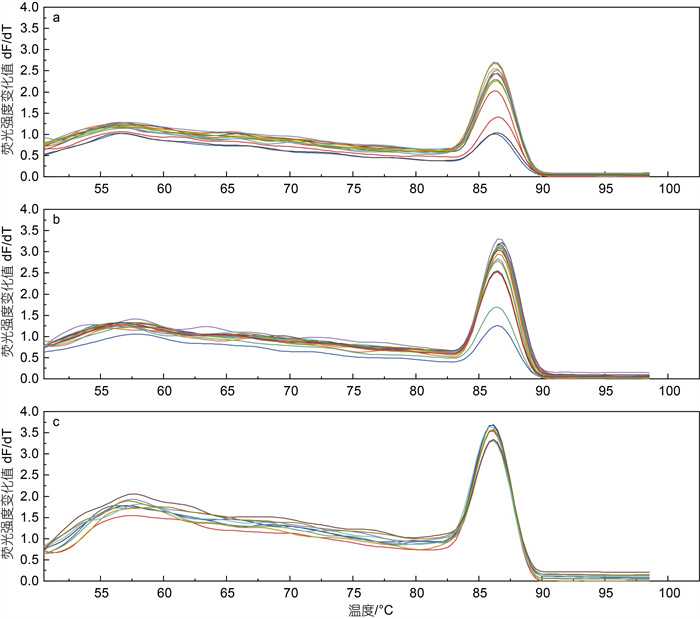 |
| 图 3 qRT-PCR方法的熔解曲线分析 Fig. 3 Curves of melting in the qRT-PCR assay 注: a. 基因组DNA标准品; b. 重组质粒标准品; c. 胶州湾环境DNA样品; 彩色曲线表示不同实验样品 |
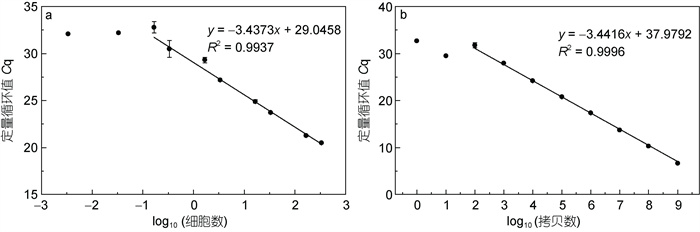 |
| 图 4 qRT-PCR方法标准工作曲线 Fig. 4 Standard curves of the qRT-PCR assay 注: a. 细胞数标准曲线; b. 拷贝数标准曲线 |
以梯度稀释的质粒DNA为模板进行qRT-PCR分析, 熔解曲线同上(图 3b)。以Cq值为纵坐标, 拷贝数的对数值为横坐标, 通过线性回归拟合为拷贝数标准工作曲线。如图 4b所示, 当每反应的质粒拷贝数高于102时, 其对数值与Cq值呈良好的线性关系, 回归方程为y=−3.441 6x+37.979 2, 回归系数R2=0.999 6, 扩增效率为95.23%。
qRT-PCR方法的验证如表 4所示, 接种至天然海水中的10 000、1 000、100个夜光藻配子细胞的计算值与实际值无显著性差异(n=6, P=0.085, 0.838, 0.150, t-test)。
| 镜检细胞数 | Cq值 | qRT-PCR检测细胞数 |
| 1×102 | 27.50±0.16 | (0.85±0.06)×102 |
| 1×103 | 23.80±0.08 | (1.01±0.06)×103 |
| 1×104 | 20.63±0.12 | (0.84±0.03)×104 |
采用本研究设计的引物对随机挑选的3个胶州湾环境DNA样品进行PCR扩增与qRT-PCR分析, 每组实验均只在100~250 bp间检测到扩增的PCR产物(图 5)。对产物进行回收测序并与目的序列进行BLAST对比, 序列相似性达到100 %, 且qRT-PCR熔解曲线同样在86.3~86.7 ℃显示单峰(图 3c), 说明该引物能够在环境DNA样品中特异性地扩增目的片段。
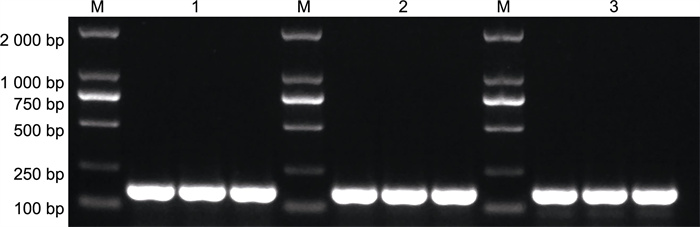 |
| 图 5 应用qRT-PCR引物对2015年胶州湾环境DNA样品扩增产物的电泳分析 Fig. 5 Electrophoresis analysis of the amplified products of environmental samples in Jiaozhou Bay, 2015 注: M: Marker; 1~3分别表示采集于3月D1站13 m水层, 7月D5站表层, 11月D1站9 m水层 |
应用本方法定量检测了2015年1月至12月间在胶州湾采集的环境DNA样品, 夜光藻配子细胞在全年均有检出, 其丰度呈现显著的季节变化(P < 0.01, Kruskal-Wallis One-Way ANOVA); 对各月份间进行两两比较发现, 在冬末春初的2~3月和夏季的7月出现两个显著的高峰(P < 0.05), 冬季的12月显著低于其他月份(P < 0.05)。1~5月, 配子丰度呈先升高后降低的趋势, 于2月达到全年最高水平, 其丰度范围为0.11×105~9.70×105 cells/L, 年最高值出现在D5站的表层; 6~9月, 配子丰度再次先升后降, 并于7月出现次高峰, 其丰度范围为0.07×105~1.20×105 cells/L, 最高值同样出现在D5站的表层; 此后, 配子丰度在11月略有回升后, 至12月降至全年最低, 此时除D8站外, 所有站位配子丰度都在100 cells/L以下, 在D3站的表层出现全年最低值, 为18.12 cells/L (图 6a)。
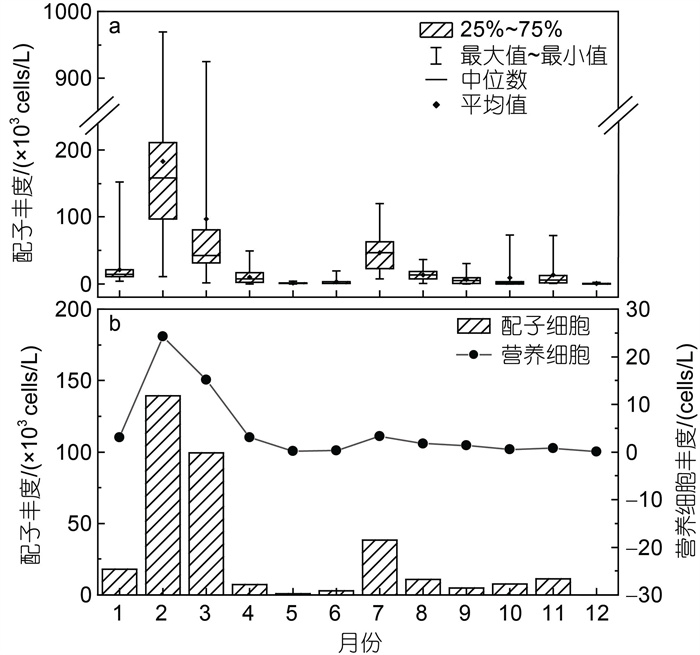 |
| 图 6 胶州湾夜光藻配子细胞与营养细胞丰度的周年变化 Fig. 6 Annual variation in abundances of N. scintillans gametes and of vegetative cells in Jiaozhou Bay 注: a. 配子丰度; b. 水柱平均配子丰度与营养细胞丰度的月平均值 |
选取湾内近岸的A5站、湾中心的C3站、湾口的D5站和湾外的D7站作为典型站位, 研究了夜光藻配子细胞的垂直分布(图 7)。A5站在全部月份的表底差异均不明显; 其他站位在2~3月、7月和11月呈现明显的表底差异。在2~3月, D5站配子丰度按表层、10 m层、10 m以下水层的顺序递减, C3站和D7站的底层丰度高于表层。在7月, D5站和D7站均为表层丰度最高, 表层以下水层差异不大。在11月, C3站的表层丰度略高于底层, D5站则在底层出现了非常明显的配子丰度高值。但从全年来看, 配子丰度与水深的Spearman相关性不显著(R=0.079, P > 0.05)。
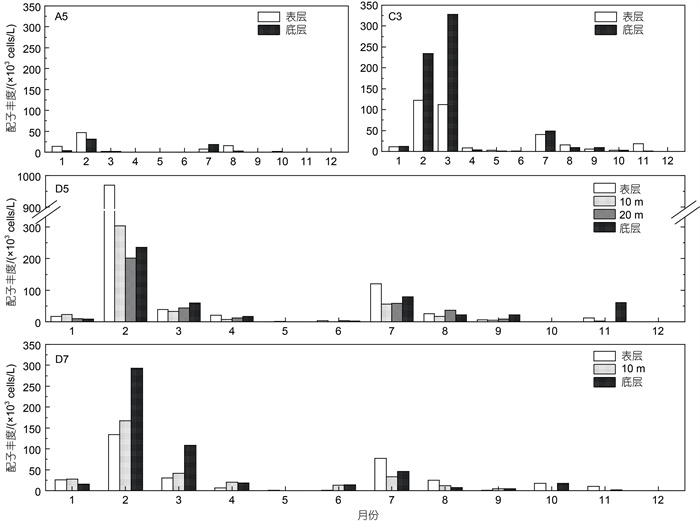 |
| 图 7 配子细胞在典型站位的垂直分布 Fig. 7 Vertical distribution of gametes in typical stations 注: A5: 湾内近岸; C3: 湾中心; D5: 湾口; D7: 湾内 |
水柱平均配子丰度在不同站位的年平均值示于图 8a, 配子细胞在湾内水深小于10 m的站位(A3、A5、B2、C1、C4站)丰度较低, 在湾中心、湾口和湾外水深大于10 m的站位丰度较高, 最高值出现在湾中心的C3站。全年来看, 水柱平均配子丰度在水深小于10 m的站位和大于10 m的站位存在显著差异(P < 0.01, Mann-Whitney U test); 在2~4月、7~9月和11月, 水深大于10 m站位的水柱平均配子丰度显著高于水深小于10 m的站位(P < 0.05), 其余月份则没有显著差异(P > 0.05) (图 8b)。
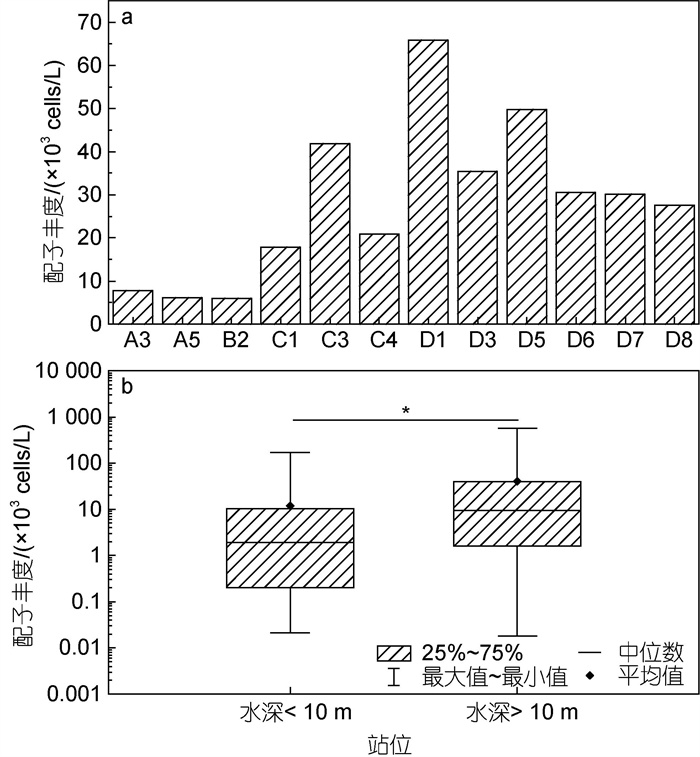 |
| 图 8 水柱平均配子丰度在不同站位的分布(*P < 0.05) Fig. 8 Distribution of gametes abundance in different stations (*P < 0.05) 注: a. 不同站位的年平均值; b. 水深 < 10 m和 > 10 m站位的比较 |
夜光藻营养细胞丰度同样呈现显著的季节变化(P < 0.01, Kruskal-Wallis One-Way ANOVA), 最高值出现在2月的C4站, 为31.37 cells/L; 最低值出现在11月的C4站和12月的A3、B2、C1站, 均未检出。对营养细胞丰度和水柱平均配子丰度进行分析, 发现营养细胞丰度与配子丰度的月度变化基本一致, 最高峰都出现在2月, 并在7月出现次高峰, 最低值都出现在12月(图 6b), Spearman相关性分析显示二者极显著正相关(R=0.759, P < 0.01)。在2月最高峰发生前后的1、4月, 虽然营养细胞丰度处于同一水平(P > 0.05, Mann-Whitney U test), 但4月的配子丰度显著低于1月(P < 0.05, Mann-Whitney U test), 其月平均值分别为(7.28±8.32)×103和(1.78±1.38)×104 cells/L; 在7月次高峰发生前后的6、9月, 营养细胞丰度与配子丰度均不存在显著差异(P > 0.05; Mann- Whitney U test)。
3 讨论有性繁殖可能在夜光藻的种群增长中发挥重要作用(Fukuda et al, 2006; Miyaguchi et al, 2008; Sriwoon et al, 2008; 宋书群等, 2016), 定量检测自然水体中的夜光藻配子, 研究其时空分布特征, 有助于阐明夜光藻有性繁殖与种群增长的关系。传统的浮游植物采集、保存、镜检技术只能计数自然水体中夜光藻的营养细胞、二分裂细胞和配子母细胞(周成旭等, 1994; Uhlig et al, 1995; Sriwoon et al, 2008)。然而, 夜光藻配子细胞粒径较小(10~15 μm), 难以固定保存, 且形态类似裸甲藻, 在光学显微镜下不易鉴别(Elbrächter et al, 1998; Fukuda et al, 2006), 传统的镜检技术无法对其进行鉴定和计数。近年来, 流式细胞术、荧光原位杂交和qRT-PCR等技术广泛应用于浮游植物的鉴定与计数。流式细胞术可以根据细胞粒径和荧光特性对粒径小于50 μm的细胞进行类群划分和计数, 常用于微微型浮游植物(picophytoplankton, < 2 μm)的检测(晁敏等, 2003), 但很难在环境样品中鉴定到种。荧光原位杂交技术能够利用特异性核酸探针对浮游植物进行定性分析, 但定量分析需要在固定、离心、裂解及杂交等步骤后保持细胞不变形、不出现因破碎导致的团聚现象, 否则会对后续的荧光显微计数造成很大的困难(张宝玉, 2005)。qRT-PCR技术具有高特异性与灵敏度, 广泛应用于多种海洋藻类的野外监测(He et al, 2007; 高岩等, 2013; Hu et al, 2022; Li et al, 2022), 能够区分同一属的不同物种(Blomster et al, 2000; Yuan et al, 2012), 其检出限甚至可以在个位数以下, Li等(2010)定量检测水体中血卵涡鞭虫(Hematodinium perezi)的检出限可低至每反应0.01个细胞; 另外, 该方法对样品保存的要求较低, 现场即可完成真空抽滤采样与液氮速冻保存工作, 很大程度上避免了细胞数量的损失。
Miyaguchi等(2008)在相模湾(Sagimi Bay)曾尝试建立夜光藻配子的qRT-PCR定量检测方法, 但其标准工作曲线没有体现出良好的线性响应(R2=0.722 5)。在本研究中, qRT-PCR熔解曲线显示单峰, PCR扩增产物的测序结果与目标序列完全匹配, 说明参照rRNA基因的18S-ITS1区域设计的引物能够从环境样品中精准地识别夜光藻的序列。两种标准品的qRT-PCR扩增效率(95.40%, 95.23%)及R2值(0.993 7, 0.999 6)均在理想范围内(90%~110%, R2 > 0.980 0) (Taylor et al, 2010), 能够实现对水体中配子细胞的准确计算。以500 mL的滤水体积、30 μL的DNA提取洗脱体积和1 μL的qRT-PCR分析体积计算, 本研究中检测到的最低配子丰度(18.12 cells/L)可换算为每反应0.30个细胞, 高于细胞数标准曲线的检出限(每反应0.17个细胞), 完全可以满足野外监测工作的需求。
应用qRT-PCR技术检测细胞丰度时, 标准工作曲线的构建至关重要。在过去的研究中, 检测标准品通常有两种, 即重组质粒标准品与基因组DNA标准品, 二者在不同的实验目的下各有其优势。重组质粒的制备便捷, 经测序验证后的标准品经长期保存, 反复冻融后仍可保证其稳定性(Yuan et al, 2012); 以重组质粒为标准品在彭宇科等(2011)与本研究中均表现出了更好的线性响应, 虽然无法直接获得细胞丰度, 但可用于比较夜光藻配子细胞这类不易保存、鉴定的细胞在不同时间、空间的拷贝数丰度。以基因组DNA为标准品能通过计算获得更为直观的细胞丰度数据, 其实是以标准品对应的细胞丰度为参照的相对值, 因此其准确性与细胞所含靶基因的拷贝数密切相关, 不同藻株、不同时期细胞所含拷贝数的差异会影响该方法的准确性(Galluzzi et al, 2004; 高岩等, 2013)。本研究专一地针对夜光藻生活史中的配子细胞阶段, 不需考虑不同细胞阶段间拷贝数的差异, 但夜光藻不同地理类群间的rRNA基因拷贝数是否存在差异尚不清楚, 建议在实际应用时以当地株的配子细胞建立标准工作曲线。
本研究应用qRT-PCR技术定量检测胶州湾的夜光藻配子细胞, 研究了其时空分布特征, 分析了其与营养细胞丰度的关系, 发现配子细胞具有促进种群增长的潜力。夜光藻的营养细胞与配子细胞在全年尺度上是紧密相关的, 两种细胞不仅呈现一致的月际变化趋势, 在相关性分析中也表现出显著的正相关关系(P < 0.01); 这与Miyaguchi等(2008)的研究结果相吻合, 在相模湾配子丰度高值多出现在营养细胞丰度高值之前或同时出现。在夜光藻种群大规模增长之前, 可能存在有性繁殖细胞发生的活跃期。营养细胞丰度在2~3月的峰值发生前后没有显著差异(P > 0.05), 但1月的配子丰度却显著高于4月(P < 0.05), 同期采集的配子母细胞在种群中的比例也高于4月(田达玮等, 2017)。Sriwoon等(2008)发现泰国湾(Gulf of Thailand)的绿色夜光藻无论是在自然水体还是培养于实验室内, 配子母细胞均在其快速增长期频繁出现。Miyaguchi等(2008)与本研究都发现配子细胞在水体中全年存在, 即使网采样品中没有检出夜光藻细胞, 该站位仍有配子细胞被检出, 说明在营养细胞丰度极低的水体中, 配子的存在可能是种群延续的关键之一。
胶州湾夜光藻的营养细胞与配子细胞呈现不同的时空分布模式。营养细胞的水平分布具有一定的季节规律: 1~2月, 细胞丰度在湾内东侧靠近河流入海口的站位最高; 3月, 仅C1站和D1站丰度略低, 其他站位差异不大; 4~9月, 细胞丰度的高值多出现在湾口和湾外; 10~12月, 高值分别出现在湾内、湾口和湾外(田达玮等, 2017)。配子细胞的水平分布没有明显的季节变化, 全年均表现出湾内近岸站位低于湾中心、湾口和湾外站位的分布规律。以水深是否超过10 m分组, 配子丰度在较深的站位总体上高于分布在湾内近岸的较浅站位, 这可能与10 m层的营养细胞活力较高有关, Tada等(2020)发现虽然表层水体中营养细胞丰度较高, 但活力极低, 较深的站位可能为夜光藻提供了适宜有性繁殖的水层。浅水站位受风、海流和潮汐等物理因素的驱使, 易导致营养细胞的聚集(Zhang et al, 2017, 2020), 但表层大量衰弱的营养细胞没有能力进行旺盛的繁殖活动, 可能造成湾内近岸站位的配子丰度呈现低值。
夜光藻配子细胞在水体中的形成与扩散可能受多种因素影响。在胶州湾, 配子丰度垂直分布总体上比较均匀。水深较浅时, 可能与水体垂直混合均匀有关(康美华, 2014)。同时, 夜光藻饵料的垂直分布也可能影响配子的形成。夜光藻的有性繁殖需要吞噬营养的支持, 在叶绿素a浓度低的月份, 观察不到配子母细胞的存在(田达玮等, 2017); 在不投喂食物的情况下, 绿色夜光藻不能形成配子母细胞(Sriwoon et al, 2008)。其次, 夜光藻在自然水体中存在昼夜迁移现象(张天文等, 2009), 其无性繁殖存在明显昼夜节律(陈汉辉等, 1991; Uhlig et al, 1995), 有性繁殖也可能存在节律性, 调查时采集到的样品只能代表该站位的瞬时状态, 可能影响对配子细胞垂直分布的整体描述。另外, 胶州湾局部海域在盛夏期间会形成温跃层(杨玉玲等, 1999), 水体的层化也可能影响配子细胞的垂直分布。
4 结论(1) 以夜光藻rRNA基因的18S-ITS1为靶区域, 建立了一种能快速、灵敏定量检测水体中夜光藻配子的qRT-PCR方法, 检出限为每反应0.17个细胞或102个拷贝, 在分析室内培养的其他海洋微藻和环境水样时均表现出良好的特异性。
(2) 2015年胶州湾水体中的夜光藻配子丰度呈双峰分布, 最高峰出现在2~3月, 次高峰出现在7月; 配子细胞的水平分布可能受存在于10 m水层的高活力细胞群体的影响, 呈现湾内近岸浅水站位低于湾中心、湾口和湾外深水站位的分布规律; 配子细胞总体在垂向分布均匀, 深水站位的个别月份存在垂直差异。
(3) 在营养细胞最高值出现的月份及前一个月, 水体中存在的大量配子细胞可能促进种群增长; 在营养细胞丰度极低时, 水体中的配子细胞对种群存续有潜在的积极意义。
致谢 数据在胶州湾海洋生态系统国家野外科学观测研究站帮助下产生, 谨致谢忱。
田达玮, 宋书群, 陈田田, 等, 2017. 胶州湾夜光藻种群动态及其大量繁殖的生态机制. 海洋与湖沼, 48(2): 276-284 |
杞桑, 李大勇, 1994. 夜光藻的不等分裂. 海洋与湖沼, 25(2): 158-161 DOI:10.3321/j.issn:0029-814X.1994.02.007 |
杨玉玲, 吴永成, 1999. 90年代胶州湾海域的温、盐结构. 黄渤海海洋, 17(3): 31-36 |
宋书群, 李才文, 孙军, 2016. 夜光藻有性繁殖研究进展. 生态学报, 36(9): 2451-2459 |
张天文, 朱丽岩, 徐培培, 等, 2009. 胶州湾夜光虫丰度的季节变化和分布特征. 中国海洋大学学报, 39(S1): 89-93 |
张宝玉, 2005. 赤潮藻的分子标识及荧光原位杂交(FISH)技术在检测赤潮藻中的应用[D]. 青岛: 中国科学院海洋研究所.
|
陈汉辉, 杞桑, 1991. 夜光藻摄食和营养繁殖的昼夜节律. 暨南大学学报, 12(3): 104-107 |
周成旭, 吴玉霖, 1994. 夜光藻的繁殖. 海洋与湖沼, 25(2): 162-164 DOI:10.3321/j.issn:0029-814X.1994.02.008 |
晁敏, 张利华, 张经, 2003. 流式细胞计在海洋浮游植物研究中的应用. 海洋科学, 27(4): 18-22 DOI:10.3969/j.issn.1000-3096.2003.04.005 |
徐恒省, 李继影, 刘孟宇, 等, 2013. 实时荧光定量聚合酶链反应快速检测产毒铜绿微囊藻. 中国环境监测, 29(4): 89-93 DOI:10.3969/j.issn.1002-6002.2013.04.019 |
高岩, 于仁成, 张清春, 等, 2013. 应用qPCR方法检测中国近海塔玛亚历山大藻复合种的研究. 环境科学学报, 33(8): 2256-2263 DOI:10.13671/j.hjkxxb.2013.08.026 |
康美华, 2014. 胶州湾生源要素的时空分布特征研究[D]. 青岛: 中国海洋大学.
|
彭宇科, 岳冬梅, 武俊, 等, 2011. 应用RT-qPCR技术定量检测湖泊水体中蓝藻方法的比较. 微生物学通报, 38(4): 460-467 DOI:10.13344/j.microbiol.china.2011.04.006 |
缪栋, 苏蕾, 张倩倩, 等, 2018. 养殖水体三种大型爆发性海洋藻类孢子/配子实时荧光定量PCR检测方法的建立与应用初探. 中国海洋大学学报, 48(8): 68-76 |
岡市友利, 西尾幸郎, 1976. 夜光虫(Noctiluca miliaris)の毒性について. 日本ブランクトン学会報, 23(2): 75-80 |
BALIARSINGH S K, LOTLIKER A A, TRAINER V L, et al, 2016. Environmental dynamics of red Noctiluca scintillans bloom in tropical coastal waters. Marine Pollution Bulletin, 111(1/2): 277-286 |
BLOMSTER J, HOEY E M, MAGGS C A, et al, 2000. Species-specific oligonucleotide probes for macroalgae: molecular discrimination of two marine fouling species of Enteromorpha (Ulvophyceae). Molecular Ecology, 9(2): 177-186 DOI:10.1046/j.1365-294x.2000.00850.x |
ELBRÄCHTER M, QI Y, 1998. Aspects of Noctiluca (Dinophyceae) population dynamics [M] // ANDERSON D M, CEMBELLA A D, HALLEGRAEFF G M. Physiological Ecology of Harmful Algal Blooms. Berlin, Germany: Springer: 315-336.
|
FUKUDA Y, ENDOH H, 2006. New details from the complete life cycle of the red-tide dinoflagellate Noctiluca scintillans (Ehrenberg) McCartney. European Journal of Protistology, 42(3): 209-219 DOI:10.1016/j.ejop.2006.05.003 |
GALLUZZI L, PENNA A, BERTOZZINI E, et al, 2004. Development of a real-time PCR assay for rapid detection and quantification of Alexandrium minutum (a dinoflagellate). Applied and Environmental Microbiology, 70(2): 1199-1206 DOI:10.1128/AEM.70.2.1199-1206.2004 |
GODHE A, ASPLUND M E, HARNSTROM K, et al, 2008. Quantification of diatom and dinoflagellate biomasses in coastal marine seawater samples by real-time PCR. Applied and Environmental Microbiology, 74(23): 7174-7182 DOI:10.1128/AEM.01298-08 |
HARRISON P J, FURUYA K, GLIBERT P M, et al, 2011. Geographical distribution of red and green Noctiluca scintillans. Chinese Journal of Oceanology and Limnology, 29(4): 807-831 DOI:10.1007/s00343-011-0510-z |
HE S Y, YU Z G, MI T Z, 2007. Development of a real-time PCR method for Thalassiosira rotula rapid detection. Acta Oceanologica Sinica, 26(2): 133-139 |
HU J R, GUO R Y, LU D D, et al, 2022. Detection and quantification of the harmful dinoflagellate Margalefidinium polykrikoides (east Asian ribotype) in the coastal waters of China. Toxins, 14(2): 95 DOI:10.3390/toxins14020095 |
HUANG C, QI Y, 1997. The abundance cycle and influence factors on red tide phenomena of Noctiluca scintillans (Dinophyceae) in Dapeng Bay, the South China Sea. Journal of Plankton Research, 19(3): 303-318 DOI:10.1093/plankt/19.3.303 |
LI C W, SHIELDS J D, MILLER T L, et al, 2010. Detection and quantification of the free-living stage of the parasitic dinoflagellate Hematodinium sp. in laboratory and environmental samples. Harmful Algae, 9(5): 515-521 DOI:10.1016/j.hal.2010.04.001 |
LI J J, XIAO X Y, GUO L Z, et al, 2022. A novel qPCR-based method to quantify seven phyla of common algae in freshwater and its application in water sources. Science of the Total Environment, 823: 153340 DOI:10.1016/j.scitotenv.2022.153340 |
MIYAGUCHI H, KUROSAWA N, TODA T, 2008. Real-time polymerase chain reaction assays for rapid detection and quantification of Noctiluca scintillans zoospore. Marine Biotechnology, 10(2): 133-140 DOI:10.1007/s10126-007-9031-3 |
SHUNMUGARAJ T, SADHUKHAN K, CHATRAGADDA R, et al, 2020. Impact of Noctiluca scintillans bloom on coastal fauna of mandapam region in gulf of mannar marine biosphere reserve, southeast coast of Tamil Nadu, India. International Journal of Ecology and Environmental Sciences, 46(2): 135-140 |
SMAYDA T J, 1997. What is a bloom? A commentary. Limnology and Oceanography, 42(5): 1132-1136 |
SRIWOON R, PHOLPUNTHIN P, LIRDWITAYAPRASIT T, et al, 2008. Population dynamics of green Noctiluca scintillans (Dinophyceae) associated with the monsoon cycle in the upper Gulf of Thailand. Journal of Phycology, 44(3): 605-615 DOI:10.1111/j.1529-8817.2008.00516.x |
TADA K, ASAHI T, KITATSUJI S, et al, 2020. Low-active high-density Noctiluca scintillans cells in surface seawater. Oceanologia, 62(3): 402-407 DOI:10.1016/j.oceano.2020.02.005 |
TAYLOR S, WAKEM M, DIJKMAN G, et al, 2010. A practical approach to RT-qPCR—publishing data that conform to the MIQE guidelines. Methods, 50(4): S1-S5 DOI:10.1016/j.ymeth.2010.01.005 |
UHLIG G, SAHLING G, 1995. Noctiluca scintillans: zeitliche verteilung bei helgoland und räumliche verbreitung in der deutschen bucht (langzeitreihen 1970-1993) [R]. Hamburg: Biologische Anstalt Helgoland: 1-127.
|
YUAN J, MI T Z, ZHEN Y, et al, 2012. Development of a rapid detection and quantification method of Karenia mikimotoi by real-time quantitative PCR. Harmful Algae, 17: 83-91 DOI:10.1016/j.hal.2012.03.004 |
ZHANG W J, DONG Z J, ZHANG C, et al, 2020. Effects of physical-biochemical coupling processes on the Noctiluca scintillans and Mesodinium red tides in October 2019 in the Yantai nearshore, China. Marine Pollution Bulletin, 160: 111609 DOI:10.1016/j.marpolbul.2020.111609 |
ZHANG S W, HARRISON P J, SONG S Q, et al, 2017. Population dynamics of Noctiluca scintillans during a bloom in a semi-enclosed bay in Hong Kong. Marine Pollution Bulletin, 121(1/2): 238-248 |
 2022, Vol. 53
2022, Vol. 53



