中国海洋湖沼学会主办。
文章信息
- 王彤彤, 张留所. 2022.
- WANG Tong-Tong, ZHANG Liu-Suo. 2022.
- 海洋线虫Litoditis marina早期发育的转录组分析
- TRANSCRIPTOME ANALYSIS OF MARINE NEMATODE LITODITIS MARINA IN EARLY DEVELOPMENTAL STAGES
- 海洋与湖沼, 53(6): 1558-1572
- Oceanologia et Limnologia Sinica, 53(6): 1558-1572.
- http://dx.doi.org/10.11693/hyhz20220300057
文章历史
-
收稿日期:2022-03-10
收修改稿日期:2021-04-05
2. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室 山东青岛 266237;
3. 中国科学院海洋大科学研究中心 山东青岛 266071;
4. 中国科学院大学 北京 100049
2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China;
3. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
全球气候变化影响海洋动物的生长发育、繁殖和代谢等生命过程, 变化的海洋环境如何影响海洋动物发育的分子机制还不清楚。本研究团队构建了潜在的海洋线虫Litoditis marina模式动物研究体系和平台(Xie et al, 2020; Zhao et al, 2021), 动物早期发育阶段对环境变化非常敏感(丛岩懿等, 2020; Xie et al, 2021), 但海洋线虫早期发育的分子调控网络还不清楚。
秀丽线虫Caenorhabditis elegans的发育起始于受精卵, 经过多轮的细胞分裂和分化, 孵化为幼虫, 依次经历L1、L2、L3和L4共四个幼虫阶段发育成为具有多种组织和细胞类型的成虫(Zhang et al, 2015)。在20 ℃的条件下, 秀丽线虫从L1幼虫发育到成虫约需3 d (Zhang et al, 2015)。Sulston等科学家首次绘制出了秀丽线虫从受精卵到成体的完整细胞谱系, 在20 ℃下, 从受精卵孵化为L1约需16 h, 刚孵化的L1幼虫含558个细胞, 幼虫通过细胞分裂和分化并最终发育成具有959个体细胞的成体, 不同个体间细胞发育谱系高度一致(Sulston et al, 1977, 1983)。
研究发现约3千万年前分化的两种同属线虫C. elegans和C. briggsae在发育过程中mRNA和蛋白质的变化高度保守, 但同一物种内mRNA和蛋白质的变化关联较弱, 该报道也对秀丽线虫胚胎期和L1幼虫间的转录组变化特征进行了详细描述(Grün et al, 2014)。另一项研究报道胚胎时期几乎一半的基因表达参与DNA复制和染色质形成, 以实现快速的细胞分裂和细胞类型增加(Boeck et al, 2016)。通过单细胞转录组测序, 已有报道量化了16细胞期前基因表达变化, 发现胚胎期转录本多样性随着时间推移而增加(Tintori et al, 2016)。通过全长转录组测序分析, 研究报道秀丽线虫胚胎转录本比幼虫阶段的转录本长, 进一步揭示了秀丽线虫发育过程中转录组的动态变化(Li et al, 2020)。
我们团队聚焦的海洋线虫L. marina与陆生模式动物秀丽线虫同属线虫动物门小杆线虫科, 属于海-陆线虫近缘物种(Xie et al, 2020; Zhao et al, 2021)。秀丽线虫发育机制研究已经比较深入, 而海洋线虫早期发育阶段的研究基本处于未知阶段。海洋线虫L. marina为雌雄异体, 其发育起始于受精卵, 受精卵在线虫子宫内完成早期胚胎发育, 产出体外的胚胎(eggs)孵化后为L1幼虫, 20 ℃下, 从L1幼虫发育为成体约需4~5 d (Xie et al, 2020, 2021)。L. marina从L1幼虫发育为成体依次经过L2、L3和L4幼虫阶段, 从L1幼虫发育为L2约需46 h, 从L2幼虫发育为L3约需19 h, 从L3幼虫发育为L4约需16 h, 而从L4幼虫发育为成体约需15 h (Zhao et al, 2021)。研究人员通过绘制L. marina的胚胎发育细胞谱系, 发现L. marina与秀丽线虫胚胎细胞谱系同源性高达95.5%, 但末端细胞分化命运的相似度只有76.4%, 表明了细胞分化和发育命运决定机制在海陆线虫近缘种间既有相似性又有物种特异性(Houthoofd et al, 2003)。为了初步探究海洋线虫与秀丽线虫早期发育的异同, 本研究对L. marina胚胎时期和孵化后L1发育阶段2 h、4 h和6 h的样品进行了转录组测序和分析, 研究结果表明, 海洋线虫与秀丽线虫早期发育过程中的调控机制高度相似, 秀丽线虫作为一种优秀的模式生物, 具有丰富的研究资源, 这些资源将极大地辅助海洋线虫的基础生物学研究。同时进一步通过基因编辑和转基因营救等技术和方法深入比较研究L. marina和C. elegans发育调控关键基因的功能将为海-陆近缘线虫间的发育进化机制、海洋线虫对潮间带环境适应以及全球气候变化应答的分子机制研究提供新认知。
1 材料与方法 1.1 品系的获得与培养本研究所用的海洋线虫L. marina野生型品系HQ1采集自青岛汇泉湾潮间带的表层泥沙, 已在实验室连续培养5年, 在20 ℃的条件下, 将其培养于90 mm SW-NGM平板(Xie et al, 2020)上, 以大肠杆菌E. coli OP50为食物。
1.2 海洋线虫大规模同步化将海洋线虫在20 ℃下扩大培养到30个板以上, 待其生长至成虫且交配产生了大量的卵时, 将平板上的全部线虫、卵以及菌用灭菌海水洗脱至15 mL离心管中, 静置5 min使大虫自然沉降, 卵及细菌在上清中, 将上清转移至新的15 mL离心管中, 3 000 g离心1 min, 弃上清。加入灭菌水, 混匀, 1 300 g离心1 min, 弃上清, 重复三次。加入3 mL灭菌水+ 3 mL裂解液[4 mL高乐式®漂白水(≥6.25%的次氯酸钠溶液) + 1 mL 10 mol/L NaOH + 5 mL灭菌水], 充分震荡75~90 s, 再加入灭菌水到12 mL, 1 300 g离心, 弃上清, 重复3次。此时得到大量的卵, 将其转移至海水中孵化18~20 h得到大量同步化的幼虫L1。
1.3 RNAseq样本制备将同步化后孵化前得到的卵通过400目网筛过滤, 转移至1.5 mL EP管, 8 000 g离心, 弃上清, 置于液氮中速冻30 min后保存在–80 ℃, 得到胚胎样品。将同步化的L1依次通过450目和500目网筛过滤后, 置于OP50的SW-NGM板上分别培养2 h、4 h、6 h后, 将平板上的L1用灭菌海水洗脱至15 mL离心管, 600 g离心1 min, 弃上清。加入灭菌水, 600 g离心2.5 min, 弃上清, 重复3次。将L1转移至1.5 mL离心管中, 加入灭菌水, 3 000 g离心1 min, 弃上清, 置于液氮中速冻30 min后保存至–80 ℃, 得到L1期2 h、4 h和6 h的样品。
1.4 RNAseq建库及分析提取样本RNA, 并使用NanoDrop 2000测量RNA浓度和纯度, 使用Agilent生物分析仪2100系统的RNA Nano 6000检测试剂盒对RNA完整性进行评估。使用Illumina的NEBNext UltraTM RNA Library Prep Kit (NEB, USA)试剂盒生成测序文库。使用TruSeq PE Cluster Kit v4-cBot-HS (Illumia)在cBot Cluster Generation System上对指标编码的样本进行聚类, 然后在Illumina平台上对文库进行测序生成双端reads, 最终获得文库。
原始fastq数据过滤掉含接头和低质量的reads后, 得到高质量Clean Data。使用HISAT2 (Kim et al, 2015)将其比对到参考基因组得到Mapped reads, 然后利用String Tie (Pertea et al, 2015)将比对上的reads进行组装和定量。对各样品中的Mapped reads的数目和转录本长度进行归一化, 采用FPKM (Florea et al, 2013)作为衡量基因表达水平的指标, 对每个样本分别进行基因表达水平定量。使用DESeq2 (Love et al, 2014)对两组样品进行差异表达分析, 将Fold Change≥1.5且FDR≤0.05作为差异表达基因筛选标准[差异倍数(Fold Change)表示两样品(组)间表达量的比值; 错误发现率(False Discovery Rate, FDR)是使用Benjamini和Hochberg的方法通过对差异显著性P值(P-value)进行校正得到的]。将差异表达基因注释到KEGG (Kanehisa et al, 2008)数据库(http://www.genome.jp/kegg/), 使用KOBAS (Mao et al, 2005)软件来检测KEGG通路中差异表达基因的统计富集, 筛选出两个样本间FDR≤0.05的显著性差异通路。
1.5 实时荧光定量PCR分析本研究以EVM0015995_esyt-2为内参基因, 随机选择11个关键基因(表 1)进行qPCR验证。使用ReverTra AceTM qPCR RT Master Mix with gDNA Remover试剂盒将RNA样品反转录为cDNA。cDNA中加入SYBR Green Realtime PCR Master Mix试剂和正反向引物, 在QuantStudioTM 6 Flex荧光实时定量PCR系统中检测, 每个样品设三个生物学重复。算出每个比较组合的ΔΔCt后取平均, 2–ΔΔCt即为该基因在两个比较组合之间的倍数变化。使用Graphpad对qPCR和RNAseq数据进行相关性分析和T检验, 得到两组数据的皮尔逊相关系数和P值。
| 基因名 | 正向引物 | 反向引物 |
| EVM0015995_esyt-2 | TTTCGTTCGTCATCCGGAAG | GTGAGCATGAAGGTGGTTTG |
| EVM0011486_aco-2 | TAGCTCAGGAAGCCGAGAAT | CGCGTTATCACCCAGTTTGT |
| EVM0011693_sdhb-1 | TGCCGACAAATATCTCGGTC | GGTGTGACATTTGAATGCGG |
| EVM0006431_lars-1 | ACCACTACGGGAGAAGTATG | CCTTCTTAGCCTCTTCCAGT |
| EVM0000938_nol-58 | TGCTGTTGCTCACTCACTTG | TCTCTGCAGCGCATGACATA |
| EVM0004991_asg-1 | ATGCCCCTTGTTAAAGCCGA | GACGACCAATCATTTCTCCG |
| EVM0015536_mcm-6 | CAAGACAGAAGGAACAACCC | CAGCAGCTGAAGAGGCTTTT |
| EVM0002773_rpa-2 | CGCCTACGATGTAATGGAGA | CGAATCAGCAGTACCCGATT |
| EVM0002807_hda-3 | CACCACGCAAAGAAAAGCGA | CACCCTATCGGTGGTGTAAA |
| EVM0006558_skp-1 | CCTCTCCATGCCTTATCGTA | TTCCGAGAAGATCGTGCTTG |
| EVM0008737_rnp-2 | AAGAGAATCTTGTTGGGCGG | CCAAGGGAACGTACGTTGAA |
| EVM0013266_bcas-2 | GTTCCAACTCCTTCAACTGG | AAAAGGAGTTGACCGAAGCC |
选取L. marina胚胎期(Embryo, EM)和L1幼虫2 h、4 h、6 h四个阶段, 每个阶段设三个生物学重复, 使用二代Illumina技术对其进行转录组测序。通过PCA主成分分析, 发现相同发育阶段的生物学重复高度相关, 不同发育阶段能够清晰区分(图 1)。
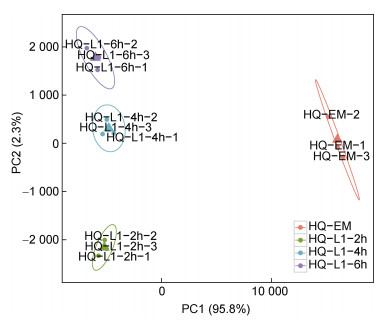 |
| 图 1 海洋线虫L. marina早期发育转录组主成分分析(PCA) Fig. 1 Principal component analysis of marine nematode L. marina in early developmental stages 注: 采用线性代数的计算方法, 对所有样本数以万计的基因表达量(FPKM)进行降维及主成分提取。横坐标为第一主成分, 纵坐标为第二主成分, 每个点代表一个样本, 每种颜色代表一个处理组 |
通过分析不同样品间的差异表达基因数目, 发现L1期2 h、4 h和6 h三个发育时期相较于EM, 均有超过9 000个基因的表达发生了显著变化, 而L1 2 h、4 h和6 h间的差异基因数目较少(表 2)。通过KEGG通路富集分析, 发现L1幼虫2 h、4 h和6 h相较于EM有超过30个通路发生显著上调(图 2), 而超过20个通路发生了显著下调(图 3)。
| 不同比较组合 | 差异表达基因数目 | 上调基因数目 | 下调基因数目 |
| L1-2 h VS EM | 9 701 | 5 004 | 4 697 |
| L1-4 h VS EM | 9 581 | 4 933 | 4 648 |
| L1-6 h VS -EM | 9 462 | 4 851 | 4 611 |
| L1-4 h VS L1-2 h | 280 | 139 | 141 |
| L1-6 h VS L1-2 h | 1 219 | 600 | 619 |
| L1-6 h VS L1-4 h | 103 | 57 | 46 |
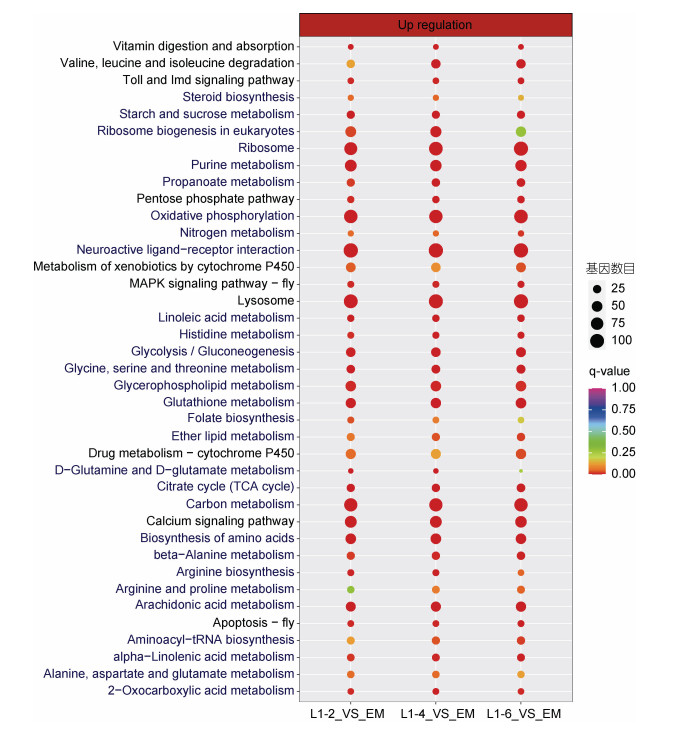 |
| 图 2 海洋线虫L. marina各比较组合中上调基因的KEGG富集情况 Fig. 2 KEGG enrichment of up-regulated genes in the marine nematode L. marina in each combination 注: 横坐标为不同比较组合, 纵坐标为通路名称, 点的大小表示基因数目, 点的颜色表示显著性差异q-value值 |
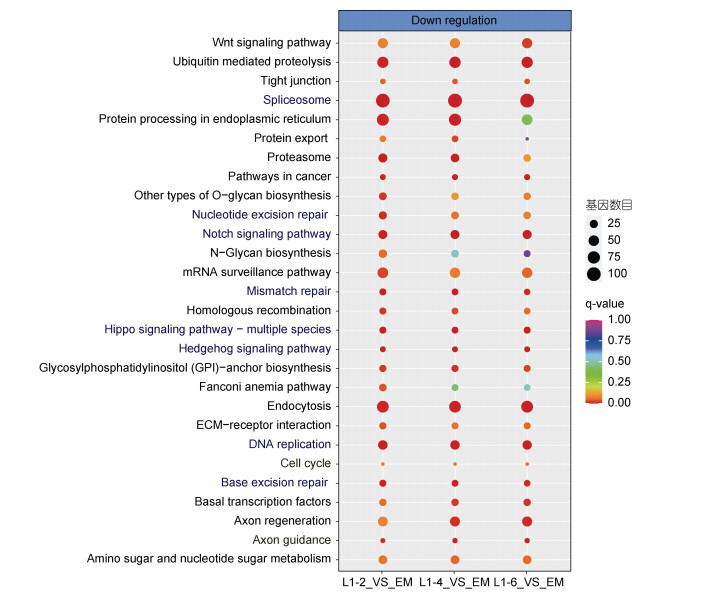 |
| 图 3 海洋线虫L. marina各比较组合中下调基因的KEGG富集情况 Fig. 3 KEGG enrichment of down-regulated genes in the marine nematode L. marina in each combination 注: 横坐标为不同比较组合, 纵坐标为通路名称, 点的大小表示基因数目, 点的颜色表示显著性差异q-value值 |
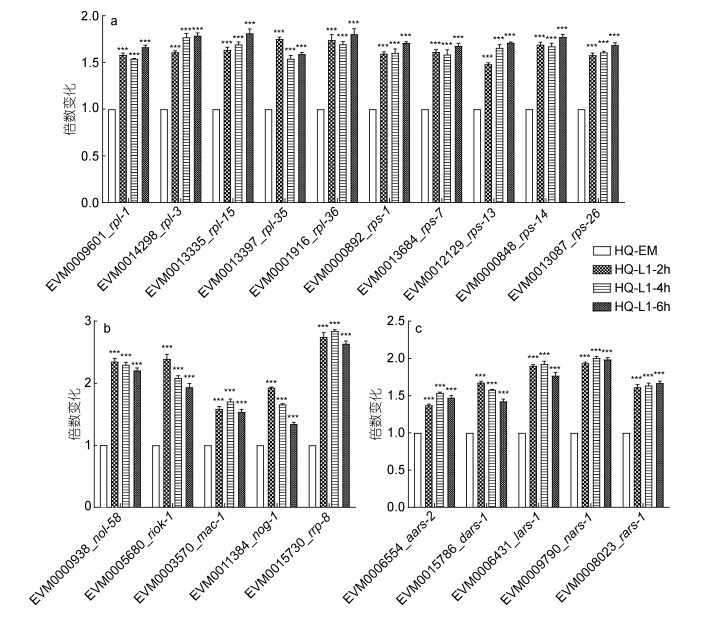
通过KEGG富集分析发现, 与胚胎期相比, L1发育时期2 h、4 h和6 h的多个核糖体蛋白大亚基编码基因(如EVM0009601_rpl-1和EVM0014298_rpl-3)和多个核糖体蛋白小亚基编码基因(EVM0000892_rps-1和EVM0013684_rps-7等)的表达均发生了显著上调(图 4a), 核糖体发生通路中的52个基因(如核仁蛋白EVM0010210_nol-6, Rio激酶EVM0005680_riok-1, 核糖体RNA加工蛋白质EVM0015730_rrp-8)在L1幼虫期发生了显著上调(图 4b), 氨酰基-rRNA生物合成通路中多个氨酰基-tRNA合成酶(如丙氨酰基tRNA合成酶EVM0006554_aars-2, 天冬氨酸氨酰基tRNA合成酶EVM0015786_dars-1, 精氨酸氨酰基tRNA合成酶EVM0008023_rars-1)在L1幼虫期发生了显著上调(图 4c)。
通过KEGG富集分析发现, 与胚胎期相比, 糖酵解和糖原异生通路中的多个基因(如醛脱氢酶EVM0001434_alh-4和二氢硫辛酰胺脱氢酶EVM0004862_dld-1)在L1发生显著上调(图 5a)。柠檬酸(TCA)循环通路中的多个基因(如顺乌头酸酶EVM0011486_aco-2、柠檬酸合成酶EVM0011427_ cts-1)在L1期发生显著上调(图 5b)。氧化磷酸化通路中的多个基因(如ATP合成酶EVM0008198_atp-1、液泡型H+ ATP酶EVM0002567_vha-2)在L1发生显著上调(图 5c)。
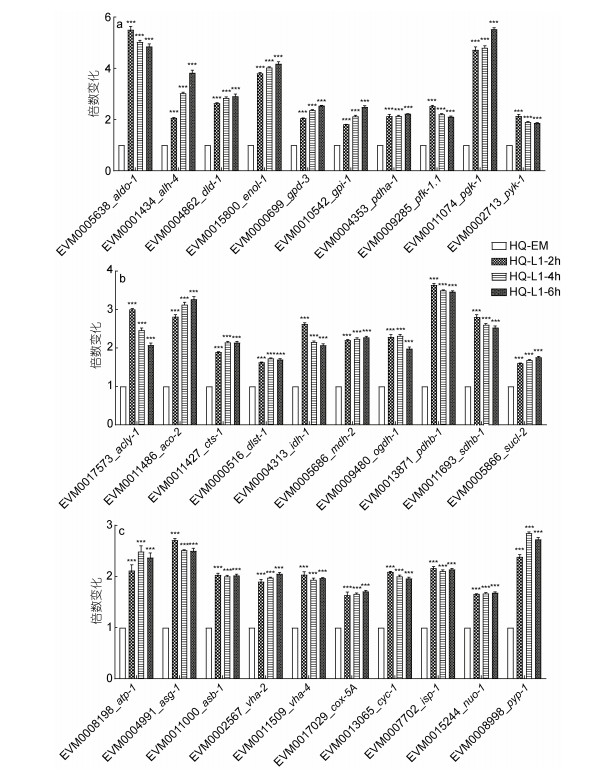 |
| 图 5 糖酵解/糖原异生(a)、TCA循环(b)和氧化磷酸化通路(c)中相关基因的表达变化情况 Fig. 5 Changes in expression of related genes in glycolysis / gluconeogenesis (a), citrate cycle (b), and oxidative phosphorylation (c) |
通过KEGG分析, 发现L1期相较于EM, 超过100个基因在神经活性配体−受体相互作用通路中上调(图 6)。EVM0000190_dop-1和EVM0009467_dop-2等多个多巴胺受体相关基因在L1期表达量上调(图 6a), GABA β受体相关基因如EVM0013292_gbb-2, EVM0015297_gbb-1和GABA受体基因EVM0014833_ gab-1 (图 6b), 多个神经肽受体家族基因如EVM0011355_npr-29和EVM0012055_npr-34等(图 6c), FMRFamide肽受体家族基因EVM0006577_frpr-3、EVM0008649_frpr-14等也在L1期表达量上调(图 6d)。
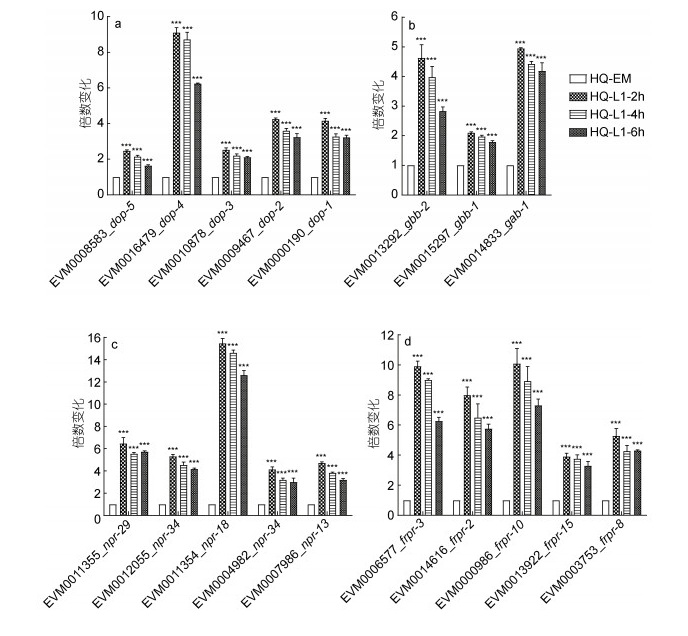 |
| 图 6 不同神经递质的基因表达变化情况 Fig. 6 Changes in gene expression of different neurotransmitters 注: a. 多巴胺受体相关基因的转录水平; b. GABA受体相关基因的转录水平; c. 神经肽受体相关基因的转录水平; d. FMRFamide肽受体家族相关基因的转录水平 |
通过KEGG分析, 发现DNA复制、错配修复、碱基切除修复和核苷酸切除修复通路在L1期下调。DNA复制通路中多个相关基因(如DNA解旋酶/核酸内切酶EVM0005762_dna-2、DNA连接酶EVM0006442_ pri-1)。错配修复通路中多个相关基因(DNA复制因子EVM0005944_rfc-1、复制蛋白A EVM0007970_ rpa-1)。碱基切除修复通路中多个相关基因(如DNA聚合酶EVM0014593_pole-1、无嘌呤/无嘧啶核糖内切酶EVM0009978_apn-1)和核苷酸切除修复通路中多个相关基因(如复制蛋白A EVM0002773_rpa-2、DNA复制因子EVM0013177_rfc-4)在L1时期显著下调(图 7)。
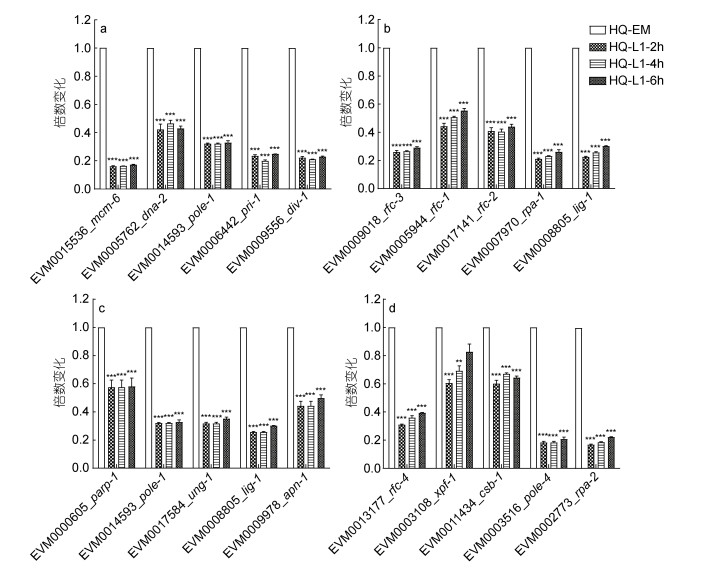 |
| 图 7 DNA复制(a)、错配修复(b)、碱基切除修复(c)和核苷酸切除(d)修复通路中相关基因表达变化情况 Fig. 7 Changes in expression of related genes in DNA replication (a), mismatch repair (b), base exicision repair (c) and nucleotide excision repair (d) |
Notch信号通路中30个基因(如EVM0011417_ lag-1, EVM0004706_bir-1, EVM0008121_hda-1)的转录水平在L1期显著下调(图 8a), Hippo信号通路中10多个基因(如EVM0004098_yap-1, EVM0016569_zyx-1)的转录水平在L1期显著下调(图 8b), Hedgehog信号通路中10个基因(如EVM0004427_ptc-3, EVM0015926_ wrt-8)的转录水平在L1期显著下调(图 8c)。
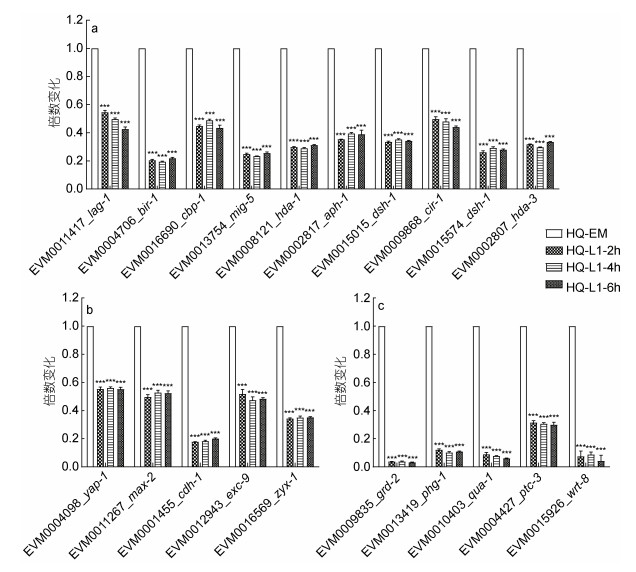 |
| 图 8 重要发育通路-Notch信号通路(a)、Hippo信号通路(b)和Hedgehog信号通路(c)的相关基因表达变化情况 Fig. 8 Changes in gene expression of important developmental pathways-Notch signaling pathway (a), Hippo signaling pathway (b), and Hedgehog signaling pathway (c) |
剪切因子对于RNA的加工至关重要(Morton et al, 2011)。本研究中, 多个剪切因子(如BCAS剪切因子EVM0013266_bcas-2、DBI1剪切因子EVM0005825_ dib-1、RSP蛋白EVM0003635_rsp-6、PRP剪切因子EVM0016298_prp-6、剪切因子3B亚基EVM0010128_ sftb-1和EVM0009161_sftb-2、SLU7剪切因子EVM0000329_sluh-7在L1期下调(图 9)。
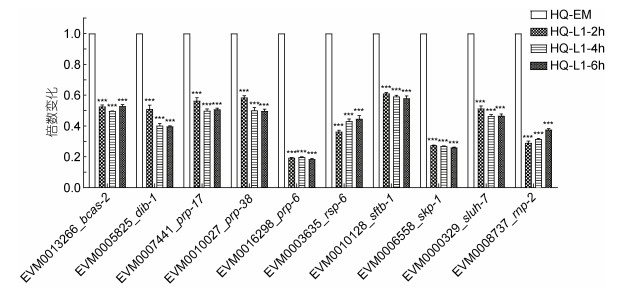 |
| 图 9 剪切体相关基因表达变化情况 Fig. 9 Changes in expression of spliceosomal related genes |
本研究随机选择11个基因进行qPCR验证, 得出倍数变化(图 10a)。采用皮尔逊相关系数r作为衡量转录组数据和qPCR数据相关性的指标, r越接近1, 相关性越好。通过Graphpad分析, 得出转录组测序结果和qPCR的结果相关性r为0.961 6, 显著性P值小于0.000 1 (图 10b), 结果显示两者相关性很高。
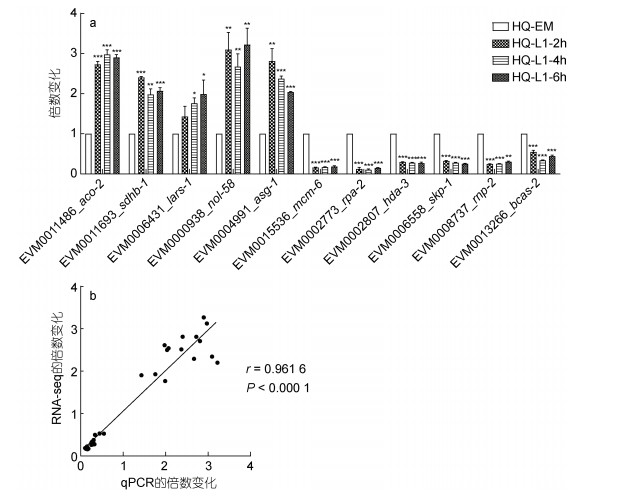 |
| 图 10 qPCR验证结果 Fig. 10 Verification results through qPCR analysis 注: a. qPCR得到的L1 (2 h、4 h、6 h)与胚胎期(EM)的表达倍数变化; b. qPCR和转录组分析间相关基因表达倍数变化的相关性分析 |
核糖体是蛋白质合成的场所, 与细胞生长和分裂密不可分(Fromont-Racine et al, 2003; Thomson et al, 2013), 对秀丽线虫全基因组RNAi筛查发现核糖体编码基因如rpl-4、rpl-35、rps-7、rps-13等基因的敲降会导致发育速率减慢(Sönnichsen et al, 2005)。研究发现RNAi敲降rps-14导致秀丽线虫生长缓慢和不育(Chan et al, 2009)。通过GFP标记秀丽线虫nog-1 (编码核仁GTP酶)启动子, 发现nog-1在胚胎早期到成虫均有表达, 使用RNAi抑制nog-1的功能导致秀丽线虫孵化数减少、生长减慢、寿命增加和脂肪储存增加(Kim et al, 2014)。秀丽线虫riok-1编码RIO激酶, RNAi抑制riok-1的功能会导致性腺形态异常, 从而使卵数量减少(Weinberg et al, 2014)。通过EMS化学诱变和核糖体图谱分析, 发现秀丽线虫rrp-8 (编码核糖体RNA加工蛋白)参与核糖体RNA加工, 对于核糖体的生物发生具有重要作用(Wu et al, 2018)。氨酰基-tRNA生物合成通路中的氨酰基-tRNA合成酶催化氨基酸与其同源tRNA的共价连接, 在翻译过程中具有至关重要的作用(Rajendran et al, 2018)。rrt-1 (rars-1)编码精氨酰tRNA合成酶, 通过RNAi抑制其功能发现, rrt-1对于秀丽线虫的发育、运动速度、缺氧敏感性、寿命有至关重要的作用(Fröhlich et al, 2017)。本研究发现多个核糖体、核糖体发生和氨酰基-tRNA生物合成通路相关基因在L1时期的表达相较于胚胎期发生了显著上调(图 4), 意味着在L. marina中, 相较于胚胎时期, L1期的发育可能需要更多蛋白质合成。据报道, 在秀丽线虫中, 相较于胚胎时期, L1期的多个核糖体编码基因rpl-和rps-发生了显著上调(Grün et al, 2014; Boeck et al, 2016; Li et al, 2020), 这种变化模式与本研究海洋线虫L. marina中发现的一致。然而在秀丽线虫中, 相比于胚胎期, 核糖体发生通路多个基因在L1期下调, 氨酰基tRNA合成通路中的基因在秀丽线虫EM和L1期之间变化不明显(Grün et al, 2014), 总之, 我们发现秀丽线虫这两个通路相关基因的表达变化特征和L. marina不同, 推测可能和两种线虫细胞发育过程存在的差异相关。
核糖体的生物发生是所有细胞中对能量要求最高的生命过程之一(Kressler et al, 2010; Thomson et al, 2013)。本研究中, L1期相较于胚胎期, 糖酵解/糖原异生通路、TCA循环通路和氧化磷酸化通路等与能量相关的通路中多个基因在L1期显著上调(图 5)。在糖酵解/糖原异生通路中, alh-4编码醛脱氢酶, 通过正向遗传筛选发现其缺失会增加脂肪醛, 减少脂肪的储存(Zeng et al, 2021)。RNAi抑制秀丽线虫中dld-1 (编码二氢硫辛酰胺脱氢酶)的功能, 可改变能量代谢, 激活与蛋白酶体降解相关的通路, 改善细胞间信号传导, 延长寿命(Ahmad et al, 2021)。TCA循环中, cts-1编码的柠檬酸合成酶是TCA循环的起始酶和限速酶, RNAi抑制秀丽线虫中cts-1的功能, 会导致早期发育停滞(Hada et al, 2019)。秀丽线虫aco-2编码乌头酸酶, idha-1编码异柠檬酸脱氢酶, RNAi抑制两者的功能会导致柠檬酸的积累, 并延迟秀丽线虫的生长和发育(Yang et al, 2022)。氧化磷酸化通路中: vha-8和vha-19编码液泡型H+ ATP酶, vha-8是PH动态平衡和幼虫发育所必需的, vha-19是胚胎发生所必需的, RNAi抑制秀丽线虫vha-8或vha-19的功能会致死(Ji et al, 2006; Knight et al, 2012)。因此, 我们推测在L1期, 这些与能量相关的基因转录水平的增加, 为核糖体的生物合成提供了必要的能量, 同时也为L1幼虫的发育提供了充足的能量供应。研究发现秀丽线虫从胚胎期发育到L1期, 上述三个能量通路中的多个基因的表达均发生显著上调(Grün et al, 2014; Boeck et al, 2016; Li et al, 2020)。因此, 我们推测L. marina早期发育的能量供应机制与秀丽线虫高度相似。
本研究通过KEGG富集分析, 发现在L1期, 17个代谢通路在L1期显著上调, 相关的通路是碳代谢, 嘌呤代谢, 花生四烯酸代谢, 亚油酸代谢, 淀粉和蔗糖代谢, 甘氨酸、丝氨酸、苏氨酸代谢, D-谷氨酰胺和D-谷氨酸代谢, 谷胱甘肽代谢, 2-氧代羧酸代谢, 甘油磷脂代谢代谢, 组氨酸代谢, 亚麻酸代谢, β-丙氨酸代谢, 丙酸酯代谢, 氮代谢(图 2)。4个生物合成通路在L1期显著上调, 相关的通路是氨基酸的生物合成, 精氨酸的生物合成, 叶酸的生物合成, 类固醇的生物合成(图 2)。与海洋线虫相似, 这些通路中的大部分基因在秀丽线虫L1期表达量也显著高于胚胎期(Grün et al, 2014; Boeck et al, 2016; Li et al, 2020), 据此推测L. marina早期发育的代谢调控机制与秀丽线虫高度保守。
3.2 海洋线虫L. marina L1幼虫神经受体基因表达显著上调通过KEGG富集分析, 发现L1期相较于EM, 超过100个基因在神经活性配体−受体相互作用通路中上调, 大多是神经递质或神经肽受体, 可能与L1幼虫的觅食和运动等行为调控相关。多巴胺受体和GABA受体在L1表达量较胚胎期显著上调(图 6a, 6b)。多巴胺和谷氨酸-氨基丁酸(γ-aminobuyric acid, GABA)对于线虫的基本运动功能至关重要(Jorgensen, 2005; Omura et al, 2012), 研究发现编码多巴胺受体的dop-2在多巴胺能神经元中表达, 可以调节多巴胺的合成和释放(Suo et al, 2004), 多巴胺能神经元在感觉到细菌食物时释放多巴胺从而引发运动减慢反应(Suo et al, 2004; Nagashima et al, 2016)。研究发现多巴胺受体编码基因dop-3缺失会导致秀丽线虫运动缺陷(Chase et al, 2004)。GABA是一种重要的抑制性神经递质, 在线虫运动和觅食时, 起到放松身体肌肉的作用(Jorgensen, 2005)。研究发现通过RNAi抑制gbb-1 (编码代谢型GABAβ受体)的功能增强抗氧化能力并延长秀丽线虫寿命(Yuan et al, 2019)。
多个神经肽受体家族基因在L1期表达量显著上升(图 6c, 6d)。在秀丽线虫中, 目前已鉴定出113个神经肽基因, 对于感觉和运动至关重要(Li et al, 2008)。比如, 通过RNAi敲降实验发现, 秀丽线虫运动和感觉唤醒需要frpr-3 (编码FMRFamide肽受体) (Chew et al, 2018)。在食物充足的情况下, npr-1 (编码神经肽受体)可以导致秀丽线虫在进食过程中聚集, 并在细菌边缘聚集(Li et al, 2008)。因此, 随着线虫孵化后开始运动, 这些神经递质表达量显著升高。除此之外, 速激肽受体家族TKR, 胆囊收缩素受体CKR, 神经调节肽NMUR, NMDA类谷氨酸受体NMR, 代谢型谷氨酸受体家族MGL等神经递质在L1期表达量较EM均发生显著上调。在秀丽线虫中, 相比于胚胎期, 多种神经递质和神经肽受体基因也均在L1期转录水平上调(Grün et al, 2014; Boeck et al, 2016; Li et al, 2020)。据此推测, 海洋线虫和秀丽线虫在L1幼虫发育阶段神经受体调控方面可能具有类似的机制。
3.3 海洋线虫L. marina胚胎期复制与修复和细胞分化调控相关基因表达显著上调DNA复制是细胞分裂周期的一个重要方面, 发生在细胞周期的合成阶段(S期), 准确无误的DNA复制对于保持基因组的完整性必不可少(Bell et al, 2002; Arias et al, 2007)。在海洋线虫中DNA复制、错配修复、碱基切除修复和核苷酸切除修复通路中的多个基因, 与胚胎期相比, 在L1幼虫期显著下调(图 7)。研究发现秀丽线虫RPA蛋白(编码复制蛋白A)参与有丝分裂复制、减数分裂、同源重组和修复等多个过程, 对于DNA复制过程至关重要, CRISPR/Cas9敲除rpa-1会导致秀丽线虫早期幼虫致死(Hefel et al, 2021)。在酵母和哺乳动物中, RFC蛋白(编码DNA复制因子)参与将某些DNA聚合酶加工因子加载到单链DNA上, 调控DNA复制过程(Venclovas et al, 2000; Griffith et al, 2002; Bermudez et al, 2003)。秀丽线虫中, 6个mcm-基因(mcm-2、mcm-3、mcm-4、mcm-5、mcm-6和mcm-7)编码的6个MCM蛋白(微小染色体维持蛋白)形成六聚体复合体, 作为DNA复制前起始复合体的关键组分, 在DNA复制中起到DNA解旋酶的作用(Korzelius et al, 2011)。RNAi抑制秀丽线虫dna-2的功能会降低胚胎发育速率、缩短寿命, 其编码的DNA解旋酶/核酸内切酶可以维持基因组的稳定性, 在细胞分裂和DNA复制中都具有非常重要的作用(Lee et al, 2011)。秀丽线虫div-1、pola-1、pri-1和pri-2编码的DNA聚合酶α-引发酶复合物启动增殖细胞的DNA复制, RNAi抑制这些基因的功能会使得胚胎致死率升高, 此外, 增殖细胞命运的维持需要pola-1、pri-1、pri-2 (Yoon et al, 2018)。我们推测, 与之相关基因在胚胎期的高表达可能反映了胚胎期较高的DNA复制率和细胞分裂, 因此, 相较于L1, 胚胎期细胞分裂速度可能更快, 从而需要复制与修复相关基因的高表达。这些基因在秀丽线虫中, 相较于胚胎期, 其转录水平在L1期也均显著下降(Rün et al, 2014; Boeck et al, 2016; Li et al, 2020), 因此L. marina和秀丽线虫类似, 胚胎时期复制和修复等多个通路基因表达显著上调可能用以支持快速、精准的细胞分裂和增殖。
KEGG富集分析发现海洋线虫在L1期, Notch、Hippo和Hedgehog信号通路中多个基因, 与胚胎期相比, 在L1期转录水平显著下调。lag-1编码Notch信号通路中的重要转录因子LAG-1, 在发育过程中介导细胞间的相互作用, RNAi敲降lag-1会导致孵化后的L1秀丽线虫发育停滞(Maicas et al, 2021)。Hippo信号通路中, yap-1 (编码Yes相关蛋白)参与秀丽线虫Wnt介导的神经元极化(Lee et al, 2018), 也与秀丽线虫的耐热性和衰老有关(Iwasa et al, 2013)。通过秀丽线虫EMS诱变发现, zyx-1 (编码含LIM结构域的黏着斑蛋白)调节秀丽线虫突触维持(Luo et al, 2014)。Hedgehog信号通路中, ptc-3 (编码PTC蛋白)在秀丽线虫中的表达是高度动态的, 通过RNAi抑制秀丽线虫ptc-3基因的功能会导致致死、不完全蜕皮、体型缩小、vulva发育缺陷等(Soloviev et al, 2011)。秀丽线虫wrt-8 (编码Hedgehog相关蛋白)的过度表达会导致轴突缺陷(Riveiro et al, 2017)。三个通路中在秀丽线虫中的大多数同源基因, 相较于胚胎期, L1期的转录表达也均发生了显著下调(Grün et al, 2014; Boeck et al, 2016; Li et al, 2020)。因此, 我们推测海洋线虫和秀丽线虫在胚胎期均需要更高的发育和细胞分化调控因子表达水平。
KEGG富集分析表明, 相比于胚胎期, 海洋线虫在L1期多个剪切体通路相关基因的转录水平显著下降。剪切因子对于RNA的加工过程至关重要(Morton et al, 2011)。研究表明prp-40调控秀丽线虫可变剪切, 其编码的PRP-40 mRNA前体加工因子是剪接体的重要组成之一(Choudhary et al, 2021)。通过RNAi抑制prp-8的功能导致秀丽线虫剪切异常, 其对于维持整个胚胎发生过程中的细胞分裂周期调控起重要作用(Hebeisen et al, 2008)。sftb-1编码剪切因子3B亚基, mCherry标记秀丽线虫sftb-1发现其是一种普遍表达的发育必需基因(Serrat et al, 2019)。在秀丽线虫中, 这些剪切因子的基因L1期表达量也均低于胚胎期(Grün et al, 2014; Boeck et al, 2016; Li et al, 2020)。最近报道发现秀丽线虫胚胎转录本的平均长度比胚胎后幼虫阶段的较长(Li et al, 2020), 因此推测海洋线虫与秀丽线虫在胚胎发育的过程中, 需要更高的剪切因子相关基因表达, 可能与胚胎期较长的转录本特征维持和细胞的快速分裂和分化等相关。
4 结论我们的研究结果表明, 与胚胎期相比, 孵化后早期L. marina L1幼虫的多个通路如核糖体、核糖体生物发生、糖酵解/糖原异生、TCA循环和氧化磷酸化通路相关基因发生了显著上调; 多个神经递质和神经肽受体基因如dop-、npr-和frpr-等在L1期转录水平也显著上调。与胚胎期相比, L1幼虫DNA复制和修复相关、Notch、Hippo和Hedgehog信号通路和剪切体调控相关基因发生了显著下调。本研究发现的海洋线虫早期发育转录组变化模式与陆生模式生物秀丽线虫高度相似, 但同一上调或下调通路中具体发生表达变化的基因有些不同。另外, L. marina L1幼虫显著上调的核糖体生物发生通路相关基因在秀丽线虫中发生了显著下调。因此, L. marina与秀丽线虫早期发育的表达调控机制高度保守, 秀丽线虫丰富的生物信息学和突变体库等资源和成熟的分子生物学和遗传学等研究手段将极大地辅助海洋线虫的基础生物学研究, 并推动模式海洋线虫体系和平台构建。结合秀丽线虫成熟的研究方法, 在L. marina中进一步优化基因编辑等遗传操作将会为海-陆近缘线虫间的发育进化机制, 海洋线虫对潮间带环境适应以及海洋动物如何应答和适应全球气候变化的分子机制研究提供新认知, 也可为蜕皮类经济物种如虾蟹的遗传育种与产业发展提供重要理论和技术支持。
丛岩懿, 谢玉素, 张留所, 2020. 海洋线虫Litoditis marina酸性pH胁迫响应的转录组分析. 海洋与湖沼, 51(6): 1472-1482 |
AHMAD W, EBERT P R, 2021. Suppression of a core metabolic enzyme dihydrolipoamide dehydrogenase (dld) protects against amyloid beta toxicity in C. elegans model of Alzheimer's disease. Genes & Diseases, 8(6): 849-866 |
ARIAS E E, WALTER J C, 2007. Strength in numbers: preventing rereplication via multiple mechanisms in eukaryotic cells. Genes & Development, 21(5): 497-518 |
BELL S P, DUTTA A, 2002. DNA replication in eukaryotic cells. Annual Review of Biochemistry, 71: 333-374 DOI:10.1146/annurev.biochem.71.110601.135425 |
BERMUDEZ V P, LINDSEY-BOLTZ L A, CESARE A J, et al, 2003. Loading of the human 9-1-1 checkpoint complex onto DNA by the checkpoint clamp loader hRad17-replication factor C complex in vitro. Proceedings of the National Academy of Sciences of the United States of America, 100(4): 1633-1638 DOI:10.1073/pnas.0437927100 |
BOECK M E, HUYNH C, GEVIRTZMAN L, et al, 2016. The time-resolved transcriptome of C. elegans. Genome Research, 26(10): 1441-1450 DOI:10.1101/gr.202663.115 |
CHAN S P, SLACK F J, 2009. Ribosomal protein RPS-14 modulates let-7 microRNA function in Caenorhabditis elegans. Developmental Biology, 334(1): 152-160 DOI:10.1016/j.ydbio.2009.07.011 |
CHASE D L, PEPPER J S, KOELLE M R, 2004. Mechanism of extrasynaptic dopamine signaling in Caenorhabditis elegans. Nature Neuroscience, 7(10): 1096-1103 DOI:10.1038/nn1316 |
CHEW Y L, TANIZAWA Y, CHO Y, et al, 2018. An afferent neuropeptide system transmits mechanosensory signals triggering sensitization and arousal in C. elegans. Neuron, 99(6): 1233-1246.e6 DOI:10.1016/j.neuron.2018.08.003 |
CHOUDHARY B, MARX O, NORRIS A D, 2021. Spliceosomal component PRP-40 is a central regulator of microexon splicing. Cell Reports, 36(5): 109464 DOI:10.1016/j.celrep.2021.109464 |
FLOREA L, SONG L, SALZBERG S L, 2013. Thousands of exon skipping events differentiate among splicing patterns in sixteen human tissues. F1000Research, 2: 188 DOI:10.12688/f1000research.2-188.v1 |
FRÖHLICH D, SUCHOWERSKA A K, SPENCER Z H T, et al, 2017. In vivo characterization of the aspartyl-tRNA synthetase DARS: homing in on the leukodystrophy HBSL. Neurobiology of Disease, 97: 24-35 DOI:10.1016/j.nbd.2016.10.008 |
FROMONT-RACINE M, SENGER B, SAVEANU C, et al, 2003. Ribosome assembly in eukaryotes. Gene, 313: 17-42 DOI:10.1016/S0378-1119(03)00629-2 |
GRIFFITH J D, LINDSEY-BOLTZ L A, SANCAR A, 2002. Structures of the human Rad17-replication factor C and checkpoint Rad 9-1-1 complexes visualized by glycerol spray/low voltage microscopy. Journal of Biological Chemistry, 277(18): 15233-15236 DOI:10.1074/jbc.C200129200 |
GRÜN D, KIRCHNER M, THIERFELDER N, et al, 2014. Conservation of mRNA and protein expression during development of C. elegans. Cell Reports, 6(3): 565-577 DOI:10.1016/j.celrep.2014.01.001 |
HADA K, HIROTA K, INANOBE A, et al, 2019. Tricarboxylic acid cycle activity suppresses acetylation of mitochondrial proteins during early embryonic development in Caenorhabditis elegans. Journal of Biological Chemistry, 294(9): 3091-3099 DOI:10.1074/jbc.RA118.004726 |
HEBEISEN M, DRYSDALE J, ROY R, 2008. Suppressors of the cdc-25.1(gf)-associated intestinal hyperplasia reveal important maternal roles for prp-8 and a subset of splicing factors in C. elegans. RNA, 14(12): 2618-2633 DOI:10.1261/rna.1168408 |
HEFEL A, HONDA M, CRONIN N, et al, 2021. RPA complexes in Caenorhabditis elegans meiosis; unique roles in replication, meiotic recombination and apoptosis. Nucleic Acids Research, 49(4): 2005-2026 DOI:10.1093/nar/gkaa1293 |
HOUTHOOFD W, JACOBSEN K, MERTENS C, et al, 2003. Embryonic cell lineage of the marine nematode Pellioditis marina. Developmental Biology, 258(1): 57-69 DOI:10.1016/S0012-1606(03)00101-5 |
IWASA H, MAIMAITI S, KUROYANAGI H, et al, 2013. Yes-associated protein homolog, YAP-1, is involved in the thermotolerance and aging in the nematode Caenorhabditis elegans. Experimental Cell Research, 319(7): 931-945 DOI:10.1016/j.yexcr.2013.01.020 |
JI Y J, CHOI K Y, SONG H O, et al, 2006. VHA-8, the E subunit of V-ATPase, is essential for pH homeostasis and larval development in C. elegans. FEBS Letters, 580(13): 3161-3166 DOI:10.1016/j.febslet.2006.04.067 |
JORGENSEN E M, 2005. GABA. WormBook: the Online Review of C. elegans Biology: 1-13 |
KANEHISA M, ARAKI M, GOTO S, et al, 2008. KEGG for linking genomes to life and the environment. Nucleic Acids Research, 36(S1): D480-D484 |
KIM Y I, BANDYOPADHYAY J, CHO I, et al, 2014. Nucleolar GTPase NOG-1 regulates development, fat storage, and longevity through insulin/IGF signaling in C. elegans. Molecules and Cells, 37(1): 51-57 DOI:10.14348/molcells.2014.2251 |
KIM D, LANGMEAD B, SALZBERG S L, 2015. HISAT: a fast spliced aligner with low memory requirements. Nature Methods, 12(4): 357-360 DOI:10.1038/nmeth.3317 |
KNIGHT A J, JOHNSON N M, BEHM C A, 2012. VHA-19 is essential in Caenorhabditis elegans oocytes for embryogenesis and is involved in trafficking in oocytes. PLoS One, 7(7): e40317 DOI:10.1371/journal.pone.0040317 |
KORZELIUS J, THE I, RUIJTENBERG S, et al, 2011. C. elegans MCM-4 is a general DNA replication and checkpoint component with an epidermis-specific requirement for growth and viability. Developmental Biology, 350(2): 358-369 DOI:10.1016/j.ydbio.2010.12.009 |
KRESSLER D, HURT E, BAΒLER J, 2010. Driving ribosome assembly. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research, 1803(6): 673-683 DOI:10.1016/j.bbamcr.2009.10.009 |
LEE M H, HOLLIS S E, YOO B H, et al, 2011. Caenorhabditis elegans DNA-2 helicase/endonuclease plays a vital role in maintaining genome stability, morphogenesis, and life span. Biochemical and Biophysical Research Communications, 407(3): 495-500 DOI:10.1016/j.bbrc.2011.03.045 |
LEE H, KANG J S, LEE J, 2018. Involvement of YAP-1, the homolog of yes-associated protein, in the wnt-mediated neuronal polarization in Caenorhabditis elegans. G3: Genes| Genomes. Genetics, 8(8): 2595-2602 |
LI C, KIM K, 2008. Neuropeptides. WormBook: the Online Review of C. elegans Biology: 1-36 |
LI R S, REN X L, DING Q T, et al, 2020. Direct full-length RNA sequencing reveals unexpected transcriptome complexity during Caenorhabditis elegans development. Genome Research, 30(2): 287-298 DOI:10.1101/gr.251512.119 |
LOVE M I, HUBER W, ANDERS S, 2014. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology, 15(12): 550 DOI:10.1186/s13059-014-0550-8 |
LUO S, SCHAEFER A M, DOUR S, et al, 2014. The conserved LIM domain-containing focal adhesion protein ZYX-1 regulates synapse maintenance in Caenorhabditis elegans. Development, 141(20): 3922-3933 DOI:10.1242/dev.108217 |
MAICAS M, JIMENO-MARTÍN Á, MILLÁN-TREJO A, et al, 2021. The transcription factor LAG-1/CSL plays a Notch-independent role in controlling terminal differentiation, fate maintenance, and plasticity of serotonergic chemosensory neurons. PLoS Biology, 19(7): e3001334 DOI:10.1371/journal.pbio.3001334 |
MAO X Z, CAI T, OLYARCHUK J G, et al, 2005. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary. Bioinformatics, 21(19): 3787-3793 DOI:10.1093/bioinformatics/bti430 |
MORTON J J, BLUMENTHAL T, 2011. RNA processing in C. elegans. Methods in Cell Biology, 106: 187-217 |
NAGASHIMA T, OAMI E, KUTSUNA N, et al, 2016. Dopamine regulates body size in Caenorhabditis elegans. Developmental Biology, 412(1): 128-138 DOI:10.1016/j.ydbio.2016.02.021 |
OMURA D T, CLARK D A, SAMUEL A D T, et al, 2012. Dopamine signaling is essential for precise rates of locomotion by C. elegans. PLoS One, 7(6): e38649 DOI:10.1371/journal.pone.0038649 |
PERTEA M, PERTEA G M, ANTONESCU C M, et al, 2015. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature Biotechnology, 33(3): 290-295 DOI:10.1038/nbt.3122 |
RAJENDRAN V, KALITA P, SHUKLA H, et al, 2018. Aminoacyl-tRNA synthetases: structure, function, and drug discovery. International Journal of Biological Macromolecules, 111: 400-414 DOI:10.1016/j.ijbiomac.2017.12.157 |
RIVEIRO A R, MARIANI L, MALMBERG E, et al, 2017. JMJD-1.2/PHF8 controls axon guidance by regulating Hedgehog-like signaling. Development, 144(5): 856-865 |
SERRAT X, KUKHTAR D, CORNES E, et al, 2019. CRISPR editing of sftb-1/SF3B1 in Caenorhabditis elegans allows the identification of synthetic interactions with cancer-related mutations and the chemical inhibition of splicing. PLoS Genetics, 15(10): e1008464 DOI:10.1371/journal.pgen.1008464 |
SOLOVIEV A, GALLAGHER J, MARNEF A, et al, 2011. C. elegans patched-3 is an essential gene implicated in osmoregulation and requiring an intact permease transporter domain. Developmental Biology, 351(2): 242-253 DOI:10.1016/j.ydbio.2010.12.035 |
SÖNNICHSEN B, KOSKI L B, WALSH A, et al, 2005. Full-genome RNAi profiling of early embryogenesis in Caenorhabditis elegans. Nature, 434(7032): 462-469 DOI:10.1038/nature03353 |
SULSTON J E, HORVITZ H R, 1977. Post-embryonic cell lineages of the nematode, Caenorhabditis elegans. Developmental Biology, 56(1): 110-156 DOI:10.1016/0012-1606(77)90158-0 |
SULSTON J E, SCHIERENBERG E, WHITE J G, et al, 1983. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology, 100(1): 64-119 DOI:10.1016/0012-1606(83)90201-4 |
SUO S, ISHIURA S, VAN TOL H H M, 2004. Dopamine receptors in C. elegans. European Journal of Pharmacology, 500(1/3): 159-166 |
THOMSON E, FERREIRA-CERCA S, HURT E, 2013. Eukaryotic ribosome biogenesis at a glance. Journal of Cell Science, 126(Pt 21): 4815-4821 |
TINTORI S C, NISHIMURA E O, GOLDEN P, et al, 2016. A transcriptional lineage of the early C. elegans embryo. Developmental Cell, 38(4): 430-444 DOI:10.1016/j.devcel.2016.07.025 |
VENCLOVAS C, THELEN M P, 2000. Structure-based predictions of Rad1, Rad9, Hus1 and Rad17 participation in sliding clamp and clamp-loading complexes. Nucleic Acids Research, 28(13): 2481-2493 DOI:10.1093/nar/28.13.2481 |
WEINBERG F, SCHULZE E, FATOUROS C, et al, 2014. Expression pattern and first functional characterization of riok-1 in Caenorhabditis elegans. Gene Expression Patterns, 15(2): 124-134 DOI:10.1016/j.gep.2014.05.005 |
WU J Y, JIANG X, LI Y M, et al, 2018. PHA-4/FoxA senses nucleolar stress to regulate lipid accumulation in Caenorhabditis elegans. Nature Communications, 9(1): 1195 DOI:10.1038/s41467-018-03531-2 |
XIE Y S, ZHANG P C, XUE B N, et al, 2020. Establishment of a marine nematode model for animal functional genomics, environmental adaptation and developmental evolution. bioRxiv DOI:10.1101/2020.03.06.980219 |
XIE Y S, ZHANG P C, ZHANG L S, 2021. Genome-wide transcriptional responses of marine nematode Litoditis marina to hyposaline and hypersaline stresses. Frontiers in Physiology, 12: 672099 DOI:10.3389/fphys.2021.672099 |
YANG R D, LI Y M, WANG Y L, et al, 2022. NHR-80 senses the mitochondrial UPR to rewire citrate metabolism for lipid accumulation in Caenorhabditis elegans. Cell Reports, 38(2): 110206 DOI:10.1016/j.celrep.2021.110206 |
YOON D S, CHA D S, ALFHILI M A, et al, 2018. Subunits of the DNA polymerase alpha-primase complex promote Notch-mediated proliferation with discrete and shared functions in C. elegans germline. The FEBS Journal, 285(14): 2590-2604 DOI:10.1111/febs.14512 |
YUAN F L, ZHOU J J, XU L X, et al, 2019. GABA receptors differentially regulate life span and health span in C. elegans through distinct downstream mechanisms. American Journal of Physiology Cell Physiology, 317(5): C953-C963 DOI:10.1152/ajpcell.00072.2019 |
ZENG L D, LI X S, PREUSCH C B, et al, 2021. Nuclear receptors NHR-49 and NHR-79 promote peroxisome proliferation to compensate for aldehyde dehydrogenase deficiency in C. elegans. PLoS Genetics, 17(7): e1009635 DOI:10.1371/journal.pgen.1009635 |
ZHANG L S, GUALBERTO D G, GUO X Y, et al, 2015. TMC-1 attenuates C. elegans development and sexual behaviour in a chemically defined food environment. Nature Communiations, 6: 6345 DOI:10.1038/ncomms7345 |
ZHAO L, GAO F, GAO S, et al, 2021. Biodiversity-based development and evolution: the emerging research systems in model and non-model organisms. Science China Life Sciences, 64(8): 1236-1280 |
 2022, Vol. 53
2022, Vol. 53