 (Sillago sihama)高密度遗传连锁图谱构建及生长性状QTL定位分析
(Sillago sihama)高密度遗传连锁图谱构建及生长性状QTL定位分析

中国海洋湖沼学会主办。
文章信息
- 田昌绪, 朱奕安, 钟键, 林星桦, 叶明慧, 黄洋, 张玉蕾, 朱春华, 李广丽. 2023.
- TIAN Chang-Xu, ZHU Yi-An, ZHONG Jian, LIN Xing-Hua, YE Ming-Hui, HUANG Yang, ZHANG Yu-Lei, ZHU Chun-Hua, LI Guang-Li. 2023.
- 多鳞
 (Sillago sihama)高密度遗传连锁图谱构建及生长性状QTL定位分析
(Sillago sihama)高密度遗传连锁图谱构建及生长性状QTL定位分析 - CONSTRUCTION OF A HIGH-DENSITY GENETIC LINKAGE MAP AND QTL DETECTION OF GROWTH TRAITS OF SILVER SILLAGO (SILLAGO SIHAMA)
- 海洋与湖沼, 54(1): 194-203
- Oceanologia et Limnologia Sinica, 54(1): 194-203.
- http://dx.doi.org/10.11693/hyhz20220500118
文章历史
-
收稿日期:2022-05-04
收修改稿日期:2022-07-04
 (Sillago sihama)高密度遗传连锁图谱构建及生长性状QTL定位分析
(Sillago sihama)高密度遗传连锁图谱构建及生长性状QTL定位分析
2. 南方海洋科学与工程广东省实验室(湛江) 广东湛江 524088;
3. 湛江海关技术中心 广东湛江 524022
 (Sillago sihama)遗传连锁图谱并鉴定生长等重要经济性状数量性状位点(QTL), 实验通过基因分型测序(GBS)技术对163个多鳞
(Sillago sihama)遗传连锁图谱并鉴定生长等重要经济性状数量性状位点(QTL), 实验通过基因分型测序(GBS)技术对163个多鳞 个体(2个亲本和161个全同胞家系F1代)进行测序及标记分型, 并通过复合区间定位法对该物种的体重、体高、体厚、眼径、体长和背鳍前长6个生长性状进行QTL定位分析。结果显示, 多鳞
个体(2个亲本和161个全同胞家系F1代)进行测序及标记分型, 并通过复合区间定位法对该物种的体重、体高、体厚、眼径、体长和背鳍前长6个生长性状进行QTL定位分析。结果显示, 多鳞 首张高密度遗传连锁图谱全长2 154.803 cM, 标记间平均遗传距离0.455 cM, 共有4 735个SNP标记分配到24个连锁群。QTL定位分析结果发现在6个生长性状中共检测到20个生长显著相关QTL位点, 分布在8个连锁群上, 单个QTL的LOD值范围为3.02~4.23, 可解释的表型变异范围为0.14%~8.42%。其中, 在连锁群LG08聚集了8个生长性状显著相关的QTL。通过对候选QTL区间内的基因进行功能注释, 共筛选到了19个潜在生长调控相关基因, 包含igf1、igf2、sstr5、sst1a、tgfbr2、gas1、igfals、gfg6、gfg20、bmp7、kdm5c、tti1以及rbm10等。实验获得的遗传标记及相关候选基因是多鳞
首张高密度遗传连锁图谱全长2 154.803 cM, 标记间平均遗传距离0.455 cM, 共有4 735个SNP标记分配到24个连锁群。QTL定位分析结果发现在6个生长性状中共检测到20个生长显著相关QTL位点, 分布在8个连锁群上, 单个QTL的LOD值范围为3.02~4.23, 可解释的表型变异范围为0.14%~8.42%。其中, 在连锁群LG08聚集了8个生长性状显著相关的QTL。通过对候选QTL区间内的基因进行功能注释, 共筛选到了19个潜在生长调控相关基因, 包含igf1、igf2、sstr5、sst1a、tgfbr2、gas1、igfals、gfg6、gfg20、bmp7、kdm5c、tti1以及rbm10等。实验获得的遗传标记及相关候选基因是多鳞 生长相关性状标记辅助选择(MAS)的有用基因资源, 为进一步研究鱼类生长调控机制提供了更多的理论依据。
生长相关性状标记辅助选择(MAS)的有用基因资源, 为进一步研究鱼类生长调控机制提供了更多的理论依据。 基因分型测序(GBS) 数量性状位点(QTL) 生长相关性状 候选基因
基因分型测序(GBS) 数量性状位点(QTL) 生长相关性状 候选基因 2. Southern Marine Science and Engineering Guangdong Laboratory(Zhanjiang), Zhanjiang 524088, China;
3. Zhanjiang Customs District Technology Center, Zhanjiang 524022, China
多鳞







基于分子标记技术构建遗传连锁图谱是实现重要经济性状QTL定位和分子标记辅助育种的有效途径(桂建芳等, 2016)。高通量测序技术简化了遗传标记分型方式, 降低了实验成本, 使得在众多水产养殖物种中通过构建高密度遗传图谱进行性状QTL精细定位成为可能(叶华等, 2011)。鱼类生长性状是重要的经济性状, 对生长性状进行遗传改良是最有价值的选育目标之一。借助简化基因组测序技术, 截至2020年已有40多种鱼类构建了高密度遗传连锁图谱, 并开展了经济性状QTL定位研究(You et al, 2020), 如鳙(Hypophthalmichehys nobilis) (Liu et al, 2016)、鲤(Cyprinus carpio) (Peng et al, 2016)、褐石斑鱼(Epinephelus bruneus) (Kessuwan et al, 2016)、大口黑鲈(Micropterus salmoides) (Dong et al, 2019)、锦鲤(Cyprinus carpio haematopterus) (Feng et al, 2018)以及斑点叉尾
目前, 多鳞


试验所用多鳞
多鳞
使用FASTP(0.18.0) (Chen et al, 2018)对测序后的raw data进行SNP过滤。筛选标准为: (1) 去除含有未知核苷酸(N)≥10%的reads; (2) 去除phred质量评分≤20及碱基≥50%的reads; (3) 删除含接头的reads。过滤后的clean reads用于组装分析。使用Burrows-Wheeler Aligner (BWA) (Li et al, 2009) (0.7.12; 比对参数为-k 32 -M)采用mem算法将过滤后的reads比对到参考基因组(SRA PRJNA642704); 比对后结果使用软件picard (http://sourceforge.net/projects/picard/.) (1.129)进行标记。使用变异检测软件GATK (Van Der Auwera et al, 2013) (3.4~46) (设置参数: -Window 4, -G_filter “QD < 2.0 || FS > 60.0 || MQ < 40.0”)进行群体SNP检测, SNP标记过滤标准为: 去除分型比例低于30%的位点, 去除杂合比例大于75%的位点, 按理论比对标记位点的基因型比例进行卡方检验, p值小于0.001的位点视为严重偏分离位点并去除, 保留分离类型为母本杂合型lmxll、父本杂合型nnxnp和双亲杂合型hkxhk的标记。使用ANNOVAR软件(Wang et al, 2010)进行功能注释SNP。
1.4 遗传图谱构建利用筛选出来的SNP标记, 使用Joinmap4.1划分连锁群, 构建遗传图谱。对划分好的连锁群采用最大似然法进行排序, 对排序后的结果进行校正, 然后再使用回归算法进行排序, 使用Perl SVG模型获得整合图谱(Van Ooijen, 2011)。整合图谱构建中, 以多鳞
使用R/qtl (Broman et al, 2003)的scanone()函数对6个生长性状进行复合区间定位(CIM), 扫描步长为1 cM, 使用LOD=3.0作为阈值筛选QTL。利用多鳞

本研究使用161尾多鳞



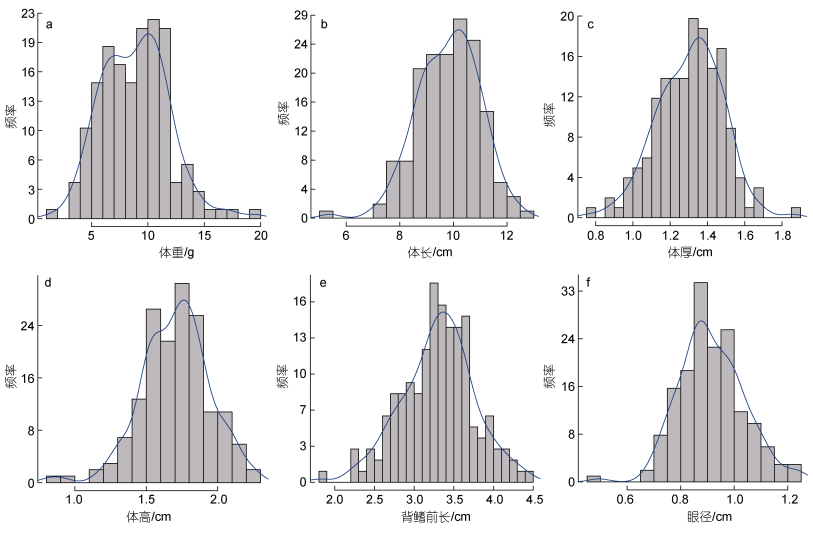 |
图 1 多鳞 |

对多鳞


群体变异检测共获得205 471个SNP位点, 这些位点中转换标记有120 302个, 占58.54%, 颠换标记有85 169个, 占41.46%。根据亲本的基因型确定标记的分离类型(亲本测序深度不低于4), 保留分离类型为母本杂合型lmxll、父本杂合型nnxnp和双亲杂合型hkxhk的标记, 共筛选获得143 886个多态性SNP位点(表 1); 随后, 对标记进一步过滤, 去除分型比例低于30%、或杂合率大于75%的位点、或严重偏分离的位点, 共保留107 406个高质量的SNP标记用于后续作图分析。

| 分离类型 | 父本基因型 | 母本基因型 | 标记数目 | 百分比/% |
| hk×hk | hk | hk | 22 364 | 16.00 |
| lm×ll | lm | ll | 59 104 | 41.00 |
| nn×np | nn | np | 62 418 | 43.00 |
| 总计 | 143 886 | 100.00 |

以筛选获得的107 406个SNP位点为基础, 根据标记的物理位置信息划分连锁群, 去除非染色体上的标记, 并在每个遗传位置的标记中保留完整度最高的标记, 最终获得4 735个SNP标记, 被分配到24个连锁群, 与多鳞

| 连锁群 | 标记数 | 连锁群长度/cM | 平均距离/cM | 大于5 cM的gap数 | 最大gap距离/cM |
| LG01 | 232 | 112.766 | 0.486 | 1 | 5.613 |
| LG02 | 203 | 99.516 | 0.490 | 0 | 4.021 |
| LG03 | 222 | 86.521 | 0.390 | 0 | 2.764 |
| LG04 | 218 | 83.448 | 0.383 | 0 | 1.228 |
| LG05 | 203 | 100.428 | 0.495 | 1 | 5.614 |
| LG06 | 199 | 94.002 | 0.472 | 0 | 4.337 |
| LG07 | 203 | 99.142 | 0.488 | 0 | 3.075 |
| LG08 | 230 | 114.276 | 0.497 | 0 | 4.654 |
| LG09 | 201 | 83.456 | 0.415 | 0 | 2.456 |
| LG10 | 168 | 80.460 | 0.479 | 0 | 4.337 |
| LG11 | 201 | 97.999 | 0.488 | 0 | 4.654 |
| LG12 | 188 | 82.541 | 0.439 | 0 | 2.461 |
| LG13 | 227 | 106.270 | 0.468 | 1 | 9.303 |
| LG14 | 198 | 89.941 | 0.454 | 0 | 3.380 |
| LG15 | 211 | 81.609 | 0.387 | 0 | 1.536 |
| LG16 | 189 | 79.469 | 0.420 | 0 | 2.456 |
| LG17 | 167 | 80.754 | 0.484 | 0 | 4.022 |
| LG18 | 223 | 92.019 | 0.413 | 1 | 8.264 |
| LG19 | 165 | 68.731 | 0.417 | 0 | 1.536 |
| LG20 | 212 | 98.225 | 0.463 | 0 | 3.997 |
| LG21 | 180 | 73.945 | 0.411 | 0 | 1.843 |
| LG22 | 174 | 96.466 | 0.554 | 1 | 9.303 |
| LG23 | 131 | 65.059 | 0.497 | 0 | 2.764 |
| LG24 | 190 | 87.760 | 0.462 | 0 | 2.153 |
| 总计 | 4735 | 2 154.803 | 0.455 | 5 | 9.303 |
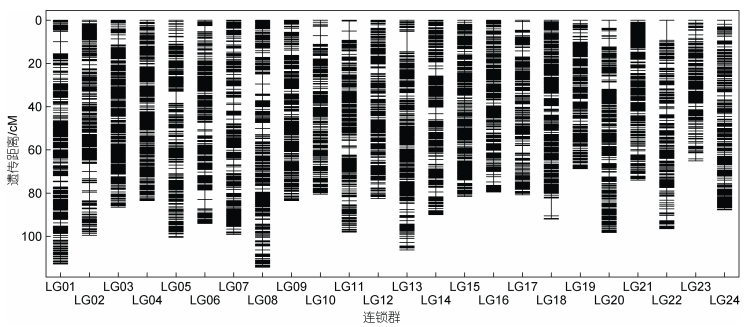 |
图 2 多鳞 |
如表 2所示, 24个连锁群长度介于65.059 cM (LG23)与114.276 cM (LG08)之间, 平均长度89.783 cM, 各连锁群标记平均遗传距离介于0.383 cM (LG04)至0.554 cM (LG22)之间。其中, LG01连锁群分布有最多的SNP标记(232个), 其连锁群长度为112.766 cM, 标记平均距离为0.486 cM; 而LG23上分布有最少的SNP标记(131个), 其长度为65.059 cM, 平均遗传距离为0.497 cM。
2.4 生长相关性状QTL的定位基于上述遗传图谱, 利用R/qtl软件对多鳞
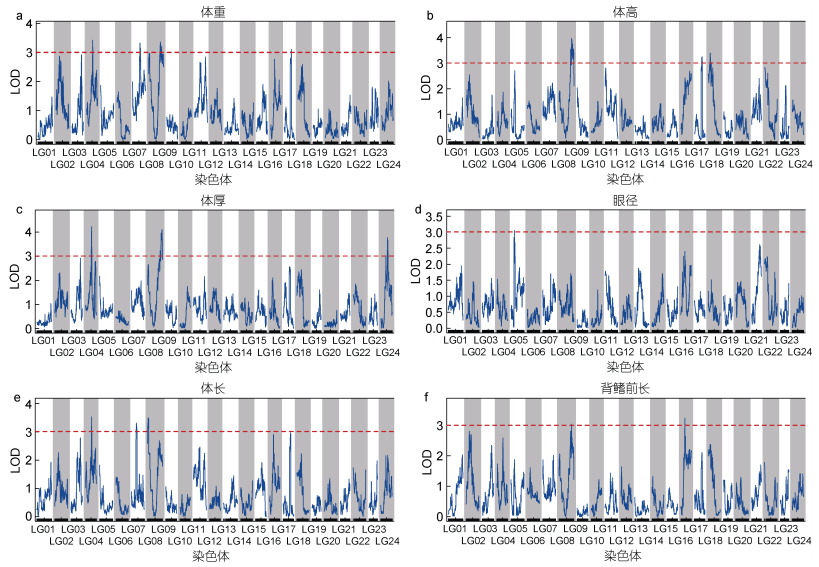 |
图 3 多鳞 |

| 性状 | QTL | 连锁群 | LOD | 贡献率/% | 遗传位置/cM | LOD峰值遗传位置/cM | LOD峰值对应标记 | 候选基因 | |
| 体重 | qGRT-1 | LG04 | 3.42 | 2.31 | 43.260~50.009 | 46.634 | LG3_10501634 | — | |
| qGRT-2 | LG07 | 3.33 | 4.36 | 52.497~66.925 | 57.713 | LG6_9129684 | — | ||
| qGRT-3 | LG08 | 3.37 | 0.85 | 79.908~98.625 | 88.498 | LG7_20534125 | kdm5c | ||
| qGRT-4 | LG08 | 3.21 | 1.47 | 90.031~110.290 | 100.158 | LG7_21679598 | — | ||
| qGRT-5 | LG17 | 3.11 | 3.23 | 45.763~57.115 | 55.000 | cLG17.loc55 | |||
| 体高 | qGRT-6 | LG08 | 3.97 | 1.01 | 87.269~106.297 | 95.864 | LG7_21084872 | tmem9 | |
| qGRT-7 | LG08 | 3.56 | 0.14 | 100.463~114.276 | 111.211 | LG7_22598757 | rbm10 | ||
| qGRT-8 | LG17 | 3.26 | 4.09 | 49.444~61.425 | 57.729 | LG16_15413357 | — | ||
| qGRT-9 | LG18 | 3.40 | 1.82 | 4.295~24.542 | 14.419 | LG17_2181895 | — | ||
| 背鳍前长 | qGRT-10 | LG08 | 3.04 | 5.74 | 86.041~106.297 | 96.170 | LG7_21093433 | tti1 | |
| qGRT-11 | LG16 | 3.23 | 6.00 | 25.157~40.498 | 30.373 | LG15_6742111 | — | ||
| 眼径 | qGRT-12 | LG05 | 3.06 | 8.42 | 21.789~32.219 | 27.005 | LG4_3754362 | — | |
| 体厚 | qGRT-13 | LG04 | 4.23 | 2.48 | 43.873~48.167 | 46.021 | LG3_10319846 | — | |
| qGRT-14 | LG08 | 4.11 | 7.08 | 100.463~114.276 | 111.211 | LG7_22598757 | rbm10 | ||
| qGRT-15 | LG24 | 3.02 | 0.58 | 32.222~50.025 | 39.286 | LG23_5155271 | — | ||
| qGRT-16 | LG24 | 3.77 | 2.02 | 50.025~57.388 | 51.559 | LG23_7622430 | — | ||
| 体长 | qGRT-17 | LG04 | 3.52 | 3.89 | 43.873~50.008 | 46.328 | LG3_10321212 | — | |
| qGRT-18 | LG07 | 3.31 | 4.27 | 33.142~43.592 | 38.369 | LG6_4597304 | — | ||
| qGRT-19 | LG08 | 3.21 | 1.74 | 0~10.738 | 0.614 | LG7_1159083 | — | ||
| qGRT-20 | LG08 | 3.49 | 1.98 | 0~17.182 | 6.000 | cLG08.loc6 | |||
本研究通过与多鳞


| 基因ID | 连锁群 | 基因名缩写 | 基因全称 |
| EVM0021738 | LG08 | igf1 | insulin-like growth factor-1 |
| EVM0006321 | LG05 | igf2 | insulin-like growth factor-2 |
| EVM0022219 | LG17 | tgfbr2 | TGF-beta receptor type-2-like |
| EVM0019806 | LG17 | gas1 | growth arrest-specific protein 1-like |
| EVM0017787 | LG16 | igfals | insulin-like growth factor-binding protein complex acid labile subunit |
| EVM0021175 | LG16 | sstr5 | somatostatin receptor type five |
| EVM0015404 | LG24 | sst1a | somatostatin-1A-like |
| EVM0019956 | LG05 | fgf6 | fibroblast growth factor 6 |
| EVM0011026 | LG04 | fgf20 | fibroblast growth factor 20-like |
| EVM0012898 | LG08 | mtor | serine/threonine-protein kinase mTOR |
| EVM0010667 | LG24 | slc2a4 | solute carrier family 2, facilitated glucose transporter member 4-like |
| EVM0008074 | LG08 | bmp7 | bone morphogenetic protein 7-like |
| EVM0002813 | LG08 | tti1 | TELO2-interacting protein 1 homolog |
| EVM0008475 | LG08 | wnt7a | wingless-type MMTV integration site family, member 7a |
| EVM0019971 | LG08 | wnt5a | wingless-type MMTV integration site family, member 5a |
| EVM0002580 | LG24 | p53 | tumor suppressor protein p53 |
作图群体的选择是构建遗传连锁图谱的先决条件。不同的作图家系类型各有利弊, 主要包括重组近交系(recombined inbred lines, RIL)、单倍体(haploid, HAP)和双单倍体(doubled haploid, DH)、子一代(F1)、子二代(F2)和回交(back crossing, BC)等家系群体(Peng et al, 2016)。通常在鱼类中构建RIL、HAP和DH家系较为困难, 而F2和BC家系的构建耗费时间长。基于养殖鱼类繁殖周期较长、基因组复杂、遗传多态性高、繁殖率高等特点, 目前大部分水产物种采用拟测交策略的F1作图家族构建遗传连锁图谱。除了作图家系的选择对遗传连锁图谱质量产生影响外, 作图标记类型的选择也是直接影响遗传图谱质量的原因之一。与其他遗传标记相比, SNP标记是基因组中最丰富的标记类型, 具有高多态性。然而, 在具有相对较大作图群体的非模式水产物种中, 很难开发足够数量的SNP (Tsigenopoulos et al, 2014; Zhu et al, 2014)。随着高通量测序技术的发展, 各种基于基因组序列的SNP基因分型技术(包括RAD、2b-RAD、GBS和SLAF等)被开发出来, 为构建高密度连锁作图提供了快速便捷的手段。GBS-seq是在第二代测序基础上发展而来的一种基于全基因组酶切位点的基因分型测序技术(Elshire et al, 2011)。GBS-seq不仅实验操作简单, 性价比高, 更为重要的是它一次测序即可获得数以万计的多态性遗传标记, 已经广泛用于群体遗传学、基因组学和遗传连锁图谱构建等研究领域(Guo et al, 2021; Weng et al, 2021)。本研究基于拟测交策略, 对多鳞






QTL分析有助于发现性状相关的连锁标记和预测候选基因。现有研究表明, 鱼类生长性状多受微效多基因的调控。本研究中, 在多鳞

位于连锁群L08上79.908~114.276 cM区间的6个显著QTL与体重、体高、体长以及背鳍前长4个生长性状显著相关, 定位到的生长相关QTL具有成簇分布的特点, 这表明连锁群LG08的上述区段可能与多鳞


现有研究结果表明, 鱼类生长与发育受多基因调控, 已有物种的研究成果尚不足以解释鱼类生长调控机理, 有必要在更多的物种中开展鱼类生长调控相关基因的发掘研究。本研究中, 在显著QTL区域内鉴定出22个生长相关候选基因, 其中包括胰岛素样生长因子1 (igf1)、胰岛素样生长因子2 (igf2)、类胰岛素生长因子酸不稳定亚基(igfals)、生长抑制特异性蛋白1 (gas1)、生长抑素1a (sst1a)、生长抑素受体5 (sstr5)、成纤维细胞因子6 (gfg6)、转化生长因子B受体2 (tgfbr2)、成纤维细胞因子20 (gfg20)以及骨形态发生蛋白7 (bmp7)等已知参与调控鱼类生长发育的功能基因。此外, 在连锁群L08 79.908~114.276 cM区间注释到组蛋白去甲基化酶(kdm5c)、Tel2相互作用蛋白1 (tti1)及RNA结合基序蛋白10 (rbm10)等候选基因。KDM5家族蛋白是激活大量促增殖细胞周期基因所必需的, 在神经发生、DNA损伤反应和癌症发展等方面发挥重要作用(Shen et al, 2021)。tti1基因参与促进哺乳动物组装、稳定和维持mTORC1和mTORC2复合物的活性, 这些复合物调节细胞生长和存活, 以响应营养和激素信号(Kaizuka et al, 2010)。rbm10基因参与调节前体mRNA的选择性剪接和mRNA稳定, 该基因的过度表达已被证明可抑制肺腺癌的恶性增殖(Li et al, 2020)。上述候选基因为研究多鳞
本研究基于多鳞

叶华, 王志勇, 2011. 水产动物遗传连锁图谱构建和QTL研究现状. 海洋科学, 35(1): 105-110 DOI:10.3969/j.issn.1671-6647.2011.01.013 |
李进波, 盛婧, 李想, 等, 2014. 五种DNA提取方法对鱼加工制品DNA提取效果的比较. 生物技术通报, (4): 43-49 DOI:10.13560/j.cnki.biotech.bull.1985.2014.04.020 |
余家旺, 王耀嵘, 林星桦, 等, 2022. 12月龄多鳞  形态性状对体质量的影响. 广东海洋大学学报, 42(1): 137-143 DOI:10.3969/j.issn.1673-9159.2022.01.018 形态性状对体质量的影响. 广东海洋大学学报, 42(1): 137-143 DOI:10.3969/j.issn.1673-9159.2022.01.018 |
陈军平, 胡玉洁, 王磊, 等, 2020. 鱼类遗传连锁图谱构建及QTL定位的研究进展. 水产科学, 39(4): 620-630 DOI:10.16378/j.cnki.1003-1111.2020.04.023 |
桂建芳, 包振民, 张晓娟, 2016. 水产遗传育种与水产种业发展战略研究. 中国工程科学, 18(3): 8-14 DOI:10.3969/j.issn.1009-1742.2016.03.003 |
黄洋, 杜涛, 黄海立, 2013. 多鳞  Sillago sihama Forskál人工繁殖研究. 广东海洋大学学报, 33(1): 15-21 DOI:10.3969/j.issn.1673-9159.2013.01.005 Sillago sihama Forskál人工繁殖研究. 广东海洋大学学报, 33(1): 15-21 DOI:10.3969/j.issn.1673-9159.2013.01.005 |
BROMAN K W, WU H, SEN Ś, et al, 2003. R/qtl: QTL mapping in experimental crosses. Bioinformatics, 19(7): 889-890 DOI:10.1093/bioinformatics/btg112 |
CHEN S F, ZHOU Y Q, CHEN Y R, et al, 2018. fastp: an ultra-fast all-in-one FASTQ preprocessor. Bioinformatics, 34(17): i884-i890 DOI:10.1093/bioinformatics/bty560 |
DONG C J, JIANG P, ZHANG J F, et al, 2019. High-density linkage map and mapping for sex and growth-related traits of largemouth bass (Micropterus salmoides). Frontiers in Genetics, 10: 960 DOI:10.3389/fgene.2019.00960 |
ELSHIRE R J, GLAUBITZ J C, SUN Q, et al, 2011. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species. PLoS One, 6(5): e19379 DOI:10.1371/journal.pone.0019379 |
FENG X, YU X M, FU B D, et al, 2018. A high-resolution genetic linkage map and QTL fine mapping for growth-related traits and sex in the Yangtze River common carp (Cyprinus carpio haematopterus). BMC Genomics, 19(1): 230 DOI:10.1186/s12864-018-4613-1 |
GUO W J, HE S, LIANG X F, et al, 2021. A high-density genetic linkage map for Chinese perch (Siniperca chuatsi) using 2.3K genotyping-by-sequencing SNPs. Animal Genetics, 52(3): 311-320 DOI:10.1111/age.13046 |
HUANG Q C, ZHANG S, DU T, et al, 2020. Modulation of growth, immunity and antioxidant-related gene expressions in the liver and intestine of juvenile Sillago sihama by dietary vitamin C. Aquaculture Nutrition, 26(2): 338-350 DOI:10.1111/anu.12996 |
KAIZUKA T, HARA T, OSHIRO N, et al, 2010. Tti1 and Tel2 are critical factors in mammalian target of rapamycin complex assembly. Journal of Biological Chemistry, 285(26): 20109-20116 DOI:10.1074/jbc.M110.121699 |
KESSUWAN K, KUBOTA S, LIU Q, et al, 2016. Detection of growth-related quantitative trait loci and high-resolution genetic linkage maps using simple sequence repeat markers in the kelp grouper (Epinephelus bruneus). Marine Biotechnology, 18(1): 57-84 DOI:10.1007/s10126-015-9673-5 |
LI H, DURBIN L R, 2009. Fast and accurate short read alignment with Burrows–Wheeler transform. Bioinformatics, 25(14): 1754-1760 DOI:10.1093/bioinformatics/btp324 |
LI Z Q, XUE Q, XU J X, et al, 2020. The role of RBM10 mutations in the development, treatment, and prognosis of lung adenocarcinoma. Cell Cycle, 19(21): 2918-2926 DOI:10.1080/15384101.2020.1829801 |
LIN X H, HUANG Y, JIANG D N, et al, 2021. Chromosomal-level genome assembly of silver sillago (Sillago sihama). Genome Biology and Evolution, 13(2): evaa272 DOI:10.1093/gbe/evaa272 |
LIU H Y, FU B D, PANG M X, et al, 2016. QTL fine mapping and identification of candidate genes for growth-related traits in bighead carp (Hypophthalmichehys nobilis). Aquaculture, 465: 134-143 DOI:10.1016/j.aquaculture.2016.08.039 |
LIU D, GUO Y S, WANG Z D, et al, 2012. Phylogenetics inferred from mitogenome and control region of Silver Sillago, Sillago sihama. Mitochondrial DNA, 23(4): 255-263 DOI:10.3109/19401736.2012.674118 |
PAN Y Y, LIN X H, CHEN F Y, et al, 2021. Genome-wide identification and expression profiling of glutathione S-transferase family under hypoxia stress in silver sillago (Sillago sihama). Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 40: 100920 DOI:10.1016/j.cbd.2021.100920 |
PENG W Z, XU J, ZHANG Y, et al, 2016. An ultra-high density linkage map and QTL mapping for sex and growth-related traits of common carp (Cyprinus carpio). Scientific Reports, 6(1): 26693 DOI:10.1038/srep26693 |
SHEN H F, ZHANG W J, HUANG Y, et al, 2021. The dual function of KDM5C in both gene transcriptional activation and repression promotes breast cancer cell growth and tumorigenesis. Advanced Science, 8(9): 2004635 DOI:10.1002/advs.202004635 |
TSIGENOPOULOS C S, LOURO B, CHATZIPLIS D, et al, 2014. Second generation genetic linkage map for the gilthead sea bream Sparus aurata L. Marine Genomics, 18: 77-82 DOI:10.1016/j.margen.2014.09.008 |
TIAN C X, LIN X H, SAETAN W, et al, 2020. Transcriptome analysis of liver provides insight into metabolic and translation changes under hypoxia and reoxygenation stress in silver sillago (Sillago sihama). Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 36: 100715 DOI:10.1016/j.cbd.2020.100715 |
VAN DER AUWERA G A, CARNEIRO M O, HARTL C, et al, 2013. From FastQ data to high-confidence variant calls: the genome analysis toolkit best practices pipeline. Current Protocols in Bioinformatics, 43(1110): 11 |
VAN OOIJEN J W, 2011. Multipoint maximum likelihood mapping in a full-sib family of an outbreeding species. Genetics Research, 93(5): 343-349 DOI:10.1017/S0016672311000279 |
WANG X H, FU B D, YU X M, et al, 2018. Fine mapping of growth-related quantitative trait loci in Yellow River carp (Cyprinus carpio haematoperus). Aquaculture, 484: 277-285 DOI:10.1016/j.aquaculture.2017.11.016 |
WANG K, LI M Y, HAKONARSON H, 2010. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Research, 38(16): e164 DOI:10.1093/nar/gkq603 |
WENG Z Y, YANG Y, WANG X, et al, 2021. Parentage analysis in giant grouper (Epinephelus lanceolatus) using microsatellite and SNP markers from genotyping-by-sequencing data. Genes, 12(7): 1042 DOI:10.3390/genes12071042 |
XU D D, ZHANG W C, CHEN R Y, et al, 2021. Chromosome-scale assembly and high-density genetic map of the yellow drum, Nibea albiflora. Scientific Data, 8(1): 268 DOI:10.1038/s41597-021-01045-z |
YANG W, WANG Y R, JIANG D N, et al, 2020. ddRADseq- assisted construction of a high-density SNP genetic map and QTL fine mapping for growth-related traits in the spotted scat (Scatophagus argus). BMC Genomics, 21(1): 278 DOI:10.1186/s12864-020-6658-1 |
YOU X X, SHAN X X, SHI Q, 2020. Research advances in the genomics and applications for molecular breeding of aquaculture animals. Aquaculture, 526: 735357 DOI:10.1016/j.aquaculture.2020.735357 |
ZHANG G S, LI J, ZHANG J J, et al, 2020. A high-density SNP-based genetic map and several economic traits-related loci in Pelteobagrus vachelli. BMC Genomics, 21(1): 700 DOI:10.1186/s12864-020-07115-7 |
ZHANG S Y, ZHANG X H, CHEN X H, et al, 2019. Construction of a high-density linkage map and QTL fine mapping for growth- and sex-related traits in channel catfish (Ictalurus punctatus). Frontiers in Genetics, 10: 251 DOI:10.3389/fgene.2019.00251 |
ZHANG G Q, ZHANG X H, YE H Z, et al, 2018. Construction of high-density genetic linkage maps and QTL mapping in the golden pompano. Aquaculture, 482: 90-95 DOI:10.1016/j.aquaculture.2017.09.011 |
ZHOU Y L, WANG Z W, GUO X F, et al, 2021. Construction of a high-density genetic linkage map and fine mapping of QTLs for growth and sex-related traits in red-tail catfish (Hemibagrus wyckioides). Aquaculture, 531: 735892 DOI:10.1016/j.aquaculture.2020.735892 |
ZHU C, TONG J, YU X, et al, 2014. A second-generation genetic linkage map for bighead carp (Aristichthys nobilis) based on microsatellite markers. Animal Genetics, 45(5): 699-708 DOI:10.1111/age.12194 |
 2023, Vol. 54
2023, Vol. 54


